Method Article
Screening auf Thermotoga maritima Membrane-Bound Pyrophosphatase Inhibitoren
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier stellen wir ein Screening-Verfahren für membrangebundene Pyrophosphatase (aus Thermotoga maritima) Inhibitoren auf Basis der Molybdän-Blaureaktion in einem 96-Well-Plattenformat vor.
Zusammenfassung
Membrangebundene Pyrophosphatasen (mPPases) sind dimere Enzyme, die in Bakterien, Archaeen, Pflanzen und protistischen Parasiten vorkommen. Diese Proteine spalten Pyrophosphat in zwei Orthophosphatmoleküle, die mit Proton und/oder Natriumionen gekoppelt sind, die über die Membran pumpen. Da bei Tieren und Menschen keine homologen Proteine vorkommen, sind mPPases gute Kandidaten bei der Gestaltung potenzieller Wirkstoffziele. Hier stellen wir ein detailliertes Protokoll zum Abschirmen für mPPase-Inhibitoren unter Verwendung der Molybdän-Blaureaktion in einem 96-Well-Plattensystem vor. Wir verwenden mPPase aus dem thermophilen Bakterium Thermotoga maritima (TmPPase) als Modellenzym. Dieses Protokoll ist einfach und kostengünstig und führt zu einem konsistenten und robusten Ergebnis. Es dauert nur etwa eine Stunde, um das Aktivitätstestprotokoll vom Beginn des Assays bis zur Absorptionsmessung abzuschließen. Da die in diesem Test erzeugte blaue Farbe über einen längeren Zeitraum stabil ist, können nachfolgende Assays unmittelbar nach der vorherigen Charge durchgeführt werden, und die Absorption kann später für alle Chargen auf einmal gemessen werden. Der Nachteil dieses Protokolls ist, dass es manuell durchgeführt wird und somit anstrengend sein kann und gute Fähigkeiten der Pipetten und Zeiterfassung erfordern. Darüber hinaus enthält die in diesem Test verwendete Arsenit-Citrat-Lösung Natriumarsenit, das giftig ist und mit den notwendigen Vorsichtsmaßnahmen behandelt werden sollte.
Einleitung
Ungefähr 25% der gesamten zellulären Proteine sind Membranproteine und etwa 60% von ihnen sind Arzneimittelziele1,2. Eines der potentiellen Wirkstoffziele3, membrangebundene Pyrophosphatasen (mPPases), sind dimerische Enzyme, die Proton und/oder Natriumion durch Hydrolyse von Pyrophosphat in zwei Orthophosphatepumpen 4. mPPases können in verschiedenen Organismen gefunden werden5 wie Bakterien, Archaeen, Pflanzen und protistische Parasiten, mit Ausnahme von Menschen und Tieren4. Bei protistischen Parasiten, z.B. Plasmodium falciparum, Toxoplasma gondii und Trypanosoma brucei, sind mPPases wesentlich für die Parasitenvirulenz6 und Knockout dieser Expression in den Parasiten führen zu einem Versagen bei der Aufrechterhaltung des intrazellulären pH bei Exposition gegenüber dem externen Grund-pH7. Aufgrund ihrer Bedeutung und des Mangels an homologem Protein, das bei Wirbeltieren vorhanden ist, können mPPases als potenzielle Wirkstoffziele für protistale Erkrankungen betrachtet werden3.
Das In-vitro-Screening von mPPase-Inhibitoren in dieser Arbeit basiert auf einem TmPPase-Modellsystem. TmPPase ist ein Natriumionenpumpen und kaliumionenabhängige mPPase von T. maritima und hat seine optimale Aktivität bei 71 °C8. Vorteile dieses Enzyms sind zum Beispiel seine einfache Produktion und Reinigung, gute thermische Stabilität und hohe spezifische Aktivität. TmPPase weist sowohl eine hohe Ähnlichkeit neben der vollständigen Erhaltung der Position als auch die Identität aller katalytischen Rückstände zu den protistischen mPPases3,9 und der gelösten Struktur von Vigna radiata10 mPPase auf. Die verfügbaren Strukturen von TmPPase in verschiedenen Konformationen sind auch für strukturbasierte Drogendesign-Experimente (als virtuelles Screening und de novo Design) nützlich.
Hier berichten wir über ein detailliertes Protokoll zum Screening von TmPPase-Inhibitoren in einem 96-Well-Plattenformat (Abbildung 1). Das Protokoll basiert auf der kolorimetrischen Methode der Molybdänblaureaktion, die zuerst von Fiske und Subbarow11entwickelt wurde. Bei dieser Methode wird 12-phosphomolybdische Säure aus Orthophosphat und Molybdat unter sauren Bedingungen entsteht, die dann reduziert wird, um charakteristische blau gefärbte Phosphomolybdänarten12zu geben.
Protokoll
1. Proteinzubereitung
HINWEIS: Der Ausdruck und die Reinigung von TmPPase wurde an anderer Stelle beschrieben13.
- Bereiten Sie 10 ml der Reaktivierungspufferlösung vor,die 20 mM 2-(N-Morpholino)Ethansulfonsäure (MES) pH 6,5, 3,5% (v/v) Glycerin, 2 mM Dithiothreitol (DTT) und 0,05% Dodecylmaltosid (DDM) enthält.
- Bereiten Sie 10 ml des Reaktionsgemisches mit 200 mM Tris-Cl pH 8.0, 8,0 mM MgCl2, 333 mM KCl und 67 mM M NaCl vor.
HINWEIS: Mg2+ ist erforderlich, um das Pyrophosphat als Substrat von mPPase zu chelatieren, K+ ist erforderlich, um die Enzymaktivität zu erhöhen, da TmPPase eine kaliumabhängige mPPase ist und Na+ für die Enzymaktivität während der Natriumionentranslokation durch TmPPase benötigt wird. - Bereiten Sie 30 mg/ml Liposomen für die Enzymreaktivierung vor.
- 10 ml 20 mM Tris-HCl pH 8.0 mit 1 mM DTT zu 0,3 g L-A-Phosphatidylcholin von Sojabohnen bis 10 ml 20 mM Tris-HCl pH 8.0 mit 1 mM DTT hinzufügen.
- Setzen Sie das Liposom auf Eis, und beschallen Sie mit 1 Sekunde Pulsintervall für 1 Minute, halten Sie für 1 Minute, und wiederholen Sie, bis die Lösung transparent gelb wird.
- Aliquot die Liposomen, gefrieren in flüssigem Stickstoff und lagern bei -80 °C, bis verwendet.
- Reaktivieren Sie das Enzym.
- Mischen Sie 40 l der Liposomenlösung mit 22,5 l dDM.
- Das Gemisch 15 min bei 55 °C erhitzen und auf Raumtemperatur abkühlen lassen.
- Fügen Sie 36,5 l der Reaktivierungspufferlösung hinzu, mischen Sie und fügen Sie 1 l konzentriertes Protein (13 mg/ml) hinzu, um eine Gesamtkonzentration von 0,13 mg/ml zu erzielen.
HINWEIS: Protein wird in der Regel nach der Reinigung in 10 L Aliquots eingefroren und vor der Anwendung auf Eis aufgetaut.
- Nehmen Sie 20 l des reaktivierten Enzyms und fügen Sie 1.480 l des Reaktionsgemisches hinzu, dann mischen Sie es vorsichtig.
HINWEIS: Die Zugabe des reaktivierten Enzyms zum Reaktionsgemisch sollte kurz vor der Verwendung erfolgen.
2. Compound-Vorbereitung
- Lösen Sie die Verbindungen in Dimethylsulfoxid (DMSO), um Lagerlösungen von 25 bis 100 mM in 50 bis 200 l zu bilden, basierend auf der Verfügbarkeit der Verbindungen.
HINWEIS: Alle hier verwendeten Verbindungen (Abbildung 2A) wurdenbereits9 veröffentlicht. Ist die Verbundlöslichkeit niedrig, kann die Lagerkonzentration entsprechend angepasst werden. - Bereiten Sie drei verschiedene Konzentrationen jeder Verbindung in Wasser vor.
HINWEIS: Die Endkonzentrationen im Reaktionsgemisch sind 1, 5 und 50 Mikromolar oder 1, 5 und 20 Mikromolar für lösliche bzw. sparsam lösliche Verbindungen.- Verdünnen Sie die Stammlösung mit Wasser auf 1 ml in Mikroröhrchen, um für lösliche Verbindungen 2 M, 10 M und 100 M oder alternativ 2 m, 10 m und 40 m für sparsam lösliche Verbindungen zu erhalten.
- Wirbel die zusammengesetzte Lösung sofort nach Verdünnung der Stammlösung für die richtige Mischung.
- Prüfen Sie die zusammengesetzte Aggregation mit einem Nephelometer.
HINWEIS: Dies wurde als Triplicate in drei Konzentrationen (1 M, 5 m und 20 m) untersucht und in einer 96-Well-Platte auf den Rohling normalisiert.- Geben Sie mit einer Mehrkanalpipette 75 l des Reaktionsgemisches in jeden Brunnen.
- Fügen Sie 75 l jeder Verbindung hinzu (für den Rohling, verwenden Sie stattdessen 75 l Wasser) und mischen Sie sie, indem Sie 5 € nach oben und unten pfeifen.
- Messen Sie jeden Brunnen bei 300 V mit einem Mikroplatten-Nephelometer.
3. Reagenzien für die Assay-Zubereitung
- Bereiten Sie die Arsenit-Citrat-Lösung vor.
- Wiegen Sie 5 g Natriumarsenit und 5 g Trinatriumcitratdihydrat.
VORSICHT: Natriumarsenit ist giftig, daher verwenden Sie die richtige Schutzausrüstung und behandeln mit besonderer Sorgfalt. Behandeln Sie vorsorglich nicht, bevor alle notwendigen Sicherheitsvorkehrungen gelesen und verstanden wurden. Behandeln Sie nur in einer Dunstabzugshaube, um Staub/Dämpfe der Verbindung oder ihrer Lösung(n) nicht einzuatmen. Wenn eingeatmet, an die frische Luft bewegen und ärztliche Hilfe erhalten. Tragen Sie eine geeignete Schutzbrille, Schutzhandschuhe und Kleidung, um Einnahme und Augen-/Hautkontakt zu vermeiden. Wenn Sie geschluckt werden, rufen Sie sofort ein Giftzentrum oder Arzt/Arzt an. Wenn es auf die Haut oder in die Augen gelangt, mit viel Wasser waschen und ärztliche Hilfe erhalten. - In 100 ml Wasser auflösen.
- 5 ml Eisessigsäure hinzufügen, mischen und 250 ml Wasser hinzufügen.
- Bei Raumtemperatur lichtgeschützt aufbewahren.
HINWEIS: Die Lösung ist seit mehr als einem Jahr stabil.
- Wiegen Sie 5 g Natriumarsenit und 5 g Trinatriumcitratdihydrat.
- Bereiten Sie Lösung A und Lösung B vor.
- Für Lösung A 10 ml eiskalte 0,5 M HCl auf 0,3 g Ascorbinsäure geben. Lösen Sie die Ascorbinsäure durch Wirbeln auf.
- Für Lösung B 1 ml eiskaltes Wasser zu 70 mg Ammoniumheptamolybdattetrahydrat und Wirbel hinzufügen, um sich aufzulösen.
HINWEIS: Bewahren Sie beide Lösungen bis zur Verwendung auf Eis auf. Für die Konsistenz des Testergebnisses können beide Lösungen maximal eine Woche auf Eis gelagert werden.
- Bereiten Sie den Phosphatstandard (Pi) mit einer Konzentration von 0 m, 62,5 m, 250 m und 500 m für die Kalibrierung vor.
- Fügen Sie vier Mikroröhrchen, die 370 l des Reaktionsgemisches enthalten, 0 L, 25 l, 50 l und 100 l mit 5 mM Na2HPO4 Dihydrat hinzugeben.
- Mit Wasser auf 1 ml aufladen.
4. Aktivitätstest für eine 96-Well-Platte
HINWEIS: Siehe Abbildung 1 für den schematischen Workflow des Assays.
- 1 ml Lösung B auf 10 ml Lösung A geben, durch Wirbeln mischen und auf Eis lagern.
HINWEIS: Diese Lösung sollte transparent und gelb sein. Lösung A + B vor der Anwendung mindestens 30 min auf Eis halten. Verwenden Sie die Lösung jedoch innerhalb von 3 h, da sie nach der Langzeitlagerung schlecht wird. - Fügen Sie die Röhrenstreifen in Dreifachausführung mit einer Mehrkanalpipette in Dreifachausführung zu den Rohrstreifen hinzu.
HINWEIS: Die Reaktionsmischung ohne Pi hinzugefügt wird als Leerzeichen verwendet werden. - Fügen Sie den Rohrstreifen mit einer Mehrkanalpipette eine Verbundlösung mit 25 l. L hinzu.
HINWEIS: Jede Verbindung hat drei verschiedene Konzentrationen in Triplicate, was für die anfängliche Schätzung der halbmaximalen hemmenden Konzentration (IC50) ausreicht. Für eine genauere IC50-Bestimmung können acht verschiedene Zusammengesetzte Konzentrationen verwendet werden. Für das hemmungslose Enzym wird die zusammengesetzte Lösung durch gleiche Menge Wasser ersetzt. Als Positivkontrollen wurden 2,5 M, 25 M und 250 M Natriumsalz (IDP) imidodiphosphat (IDP) verwendet. - Mit einer Mehrkanal-Pipette 15 l mPPase-Lösungsmischung in die Rohrstreifen (außer zu den Rohren, die Pi-Standard enthalten) hinzufügen.
- Versiegeln Sie die Rohrstreifen mit einer Klebedichtungsfolie. Schneiden Sie die Dichtplatte, um jeden Rohrstreifen zu trennen.
- Die Proben bei 71 °C vorinkubieren. Legen Sie die Proben auf den Heizblock mit 20 s Intervall zwischen den einzelnen Streifen, um den Zeitverbrauch während der nachfolgenden Schritte zu minimieren.
- Öffnen Sie für jeden Streifen die Klebedichtung. Fügen Sie 10 l von 2 mM Natriumpyrophosphat dibasic mit einer Mehrkanalpipette hinzu und mischen Sie sie, indem Sie für 5 " nach oben und unten pfeifen. Versiegeln Sie den Rohrstreifen erneut mit der gleichen Dichtung.
HINWEIS: Dieser Schritt könnte zunächst schwierig sein, in 20 s zu erreichen; jedoch wird es nach einigen Tests einfacher werden. - Bei 71 °C für 5 min inkubieren.
- Legen Sie die Proben auf das Kühlgerät mit 20 s Intervall zwischen den einzelnen Streifen. Lassen Sie sie 10 min abkühlen, zentrieren Sie aber jeden Streifen kurz nach 5 min Abkühlung, um Wassertropfen unter der Dichtplatte zu dekantieren, dann wieder an das Kühlgerät zu legen und die Abdichtung zu entfernen.
HINWEIS: Das Kühlgerät kann einfach hergestellt werden, indem man eine 96-Well-PCR-Platte auf eine mit Wasser gefüllte und mindestens 1 h gefrorene Polystyrol-Petrischale (Größe 150 mm x 15 mm) auflegt. Das Gerät sollte ca. 5 min vor Beginn des Testes aus dem Gefrierschrank herausgenommen werden. Nehmen Sie das Kühlgerät nicht direkt vor der Probenkühlung heraus, da es das Reaktionsgemisch einfrieren und die Farbentwicklung behindern wird. - Nach 10 min Abkühlung 60 l Lösung A + B hinzufügen, durch Pipettieren für 5" mischen und die Rohrleisten 10 min am Kühlgerät aufbewahren.
- Fügen Sie 90 l der Arsenit-Citrat-Lösung hinzu und halten Sie sie mindestens 30 min bei Raumtemperatur, um eine stabile blaue Farbe zu erzeugen.
VORSICHT: Aufgrund seiner Toxizität sollten alle Lösungen, die Natriumarsenit enthalten, jederzeit mit besonderer Sorgfalt behandelt werden. Daher sollte die Zugabe von Arsenit-Citrat-Lösung in einer Dunstabzugshaube erfolgen. - Geben Sie 180 l jedes Reaktionsgemisches in eine klare 96-Well-Polystyrol-Mikroplatte.
- Messen Sie die Absorption jedes Brunnens bei 860 nm mit einem Mikroplattenspektrophotometer.
5. Ergebnisanalyse
- Durchschnittlich die Triplicates jeder Probe und die P i-Standards. Subtrahieren Sie dann mit dem Rohling, um das Hintergrundsignal zu eliminieren.
- Erstellen Sie eine Kalibrierungskurve, indem Sie die Absorptionswerte (A860) mit der Menge des Pi-Standards (nmol) aufstellen und eine lineare Regression durchführen, um die Trendlinienfunktion mit der folgenden Formel zu erhalten:
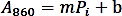
- Berechnen Sie die Phosphatmenge (nmol), die aus der enzymatischen Reaktion freigesetzt wird, basierend auf der oben genannten linearen Regressionsformel.
- Berechnen Sie die spezifische Aktivität mit der folgenden Formel:
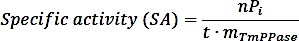
wobei nPi die Menge an Phosphat ist, die aus der Reaktion (nmol), t die Reaktionszeit (min) und mTmPPase die Menge des reinen TmPPase ist, die im Assay (mg) verwendet wird. - Berechnen Sie die prozentuale Aktivität für jede Inhibitorkonzentration nach folgender Formel:
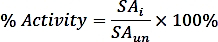
wenn SAidiespezifische Aktivität einer Probe mit Inhibitor und SAun ist die spezifische Aktivität der ungehemmten Probe. - Berechnen Sie die logIC50 (Schätzung) und IC50 (Schätzung) mit einer nichtlinearen Regressionsanpassung aus der Vier-Parameter-Dosis-Wirkungs-Kurve mit der folgenden Formel:
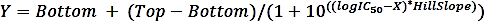
wobei X das Konzentrationsprotokoll (M), Y aktivität (%) ist: Oben und Unten sind Plateaus in der gleichen Einheit wie Y (100% bzw. 0%),e), logIC50 hat die gleichen Log-Einheiten wie X und HillSlope = Neigungsfaktor oder Hügelneigung, die einheitslos ist.
HINWEIS: Software (Materialtabelle) wird für das Fitting verwendet. Verwenden Sie für die Probe ohne Inhibitor die Konzentration von 0,01 m (anstelle von 0,00 m), da der Logarithmus von Null nicht definiert ist.
Ergebnisse
In diesem Protokoll wurden acht Verbindungen (1-8) zusammen mit IDP, einem häufigen Inhibitor von Pyrophosphatasen, als positiv zu kontrollieren getestet (Abbildung 2A). Jede Verbindung wurde in drei verschiedenen Konzentrationen (1 M, 5 m und 20 m) in DreifacherDikate getestet. Der Ablauf des Screenings ist in Abbildung 1dargestellt, beginnend mit der Proben- und Reagenzvorbereitung bis zur Absorptionsmessung bei 860 nm.
Am Ende dieses Protokolls entwickeln die Lösungen nach Zugabe von Lösung A + B und Arsenit-Citrat eine stabile blaue Farbe mit der maximalen Absorption bei 709 nm und 860 nm14 aufgrund der komplexen Bildung von Phosphationen mit Molybdat, die beobachtet werden können und das Auftreten der enzymatischen Reaktion zeigen. Für dieses Experiment verwenden wir die Absorption bei 860 nm für die Messung der freigesetzten P i-Menge, da sie eine bessere Nachweisgrenze und Empfindlichkeit hat als die Absorption bei 709 nm15. Die blaue Farbe ist in 30 min Inkubation bei Raumtemperatur voll entwickelt und stabil für mindestens 5 h14. Der Assay hat eine Sensitivität bis zu einer Pi-Konzentration von 10 m und die Absorption ist linear über einen Konzentrationsbereich von 10 bis 800 m14. Im repräsentativen Ergebnis enthalten hier die Brunnen E1-E3 (Abbildung 2C) das Reaktionsgemisch ohne Inhibitor und die blaue Lösung kann am Ende des Assays beobachtet werden. Dies kann auch bei niedrigen zusammengesetzten Konzentrationen beobachtet werden, bei denen keine vollständige Hemmung erreicht wurde, wie in den Brunnen F1-F3 für IDP und den Brunnen A4-A6 für Verbindung 1 (ATC, einem kürzlich bekannten nicht wettbewerbsfähigen Inhibitor von TmPPase9) bei einer Konzentration von 2,5 m bzw. 1 M. Die höhere Konzentration von IDP und Verbindung 1,desto weniger bis gar keine blaue Farbe kann beobachtet werden (G1-G3 und H1-H3 für IDP und B4-B6 und C4-C6 für Verbindung 1), was auf eine Hemmung der enzymatischen Aktivität hindeutet. Alle drei Konzentrationen von nicht hemmenden Verbindungen (2, 3und 8) zeigten die gleiche blaue Farbintensität wie auch E1-E3 ohne Inhibitor (Abbildung 2C).
Nach der Absorptionsmessung bei 860 nm können die Daten verarbeitet und analysiert werden (siehe Protokollabschnitt 5). Abbildung 2D zeigt das Kalibrierdiagramm des Pi-Standards mit seiner linearen Armatur (y = 0,0576x + 0,0019; r2 = 0,999). Abbildung 3 zeigt das Diagramm der enzymatischen Aktivität (%) gegen die Konzentration jeder getesteten Verbindung. Bei Verbindungen mit Hemmungsaktivität wird auch eine nichtlineare Kurvenanpassung angezeigt. Die IDP, die als Positivkontrolle verwendet wird, zeigt eindeutig einen Rückgang der Aktivität bei höherer Konzentration. Der IC50 (Schätzung), der auf der Grundlage von drei unterschiedlichen Konzentrationen berechnet wird, beträgt 88,2 m (Tabelle 1), was der vorherigen Messung (80,0 m) mit acht Konzentrationspunkten14ähnelt. Die Verbindungen 1, 4, 5, 6und 7 zeigten einen ähnlichen Trend wie IDP, da die Konzentration mit dem IC50 (Schätzung) von etwa 1,3 m, 7,4 m, 19,0 m, 37,4 m bzw. 156,1 m zunahm (Tabelle 1). Bei den Verbindungen 2, 3und 8 kann bei den Assaykonzentrationen keine Verringerung der Aktivität oder Hemmung beobachtet werden. Ein zusätzlicher Test mit acht Konzentrationspunkten kann durchgeführt werden, um präzise IC50zu erzeugen. Abbildung 4 zeigt die Hemmungskurve für die Verbindungen 1, 5, 6, 7 und 8 mit einem IC50 von 1,7 m, 21,4 m, 58,8 m, 239,0 m und >500 m bzw.9.
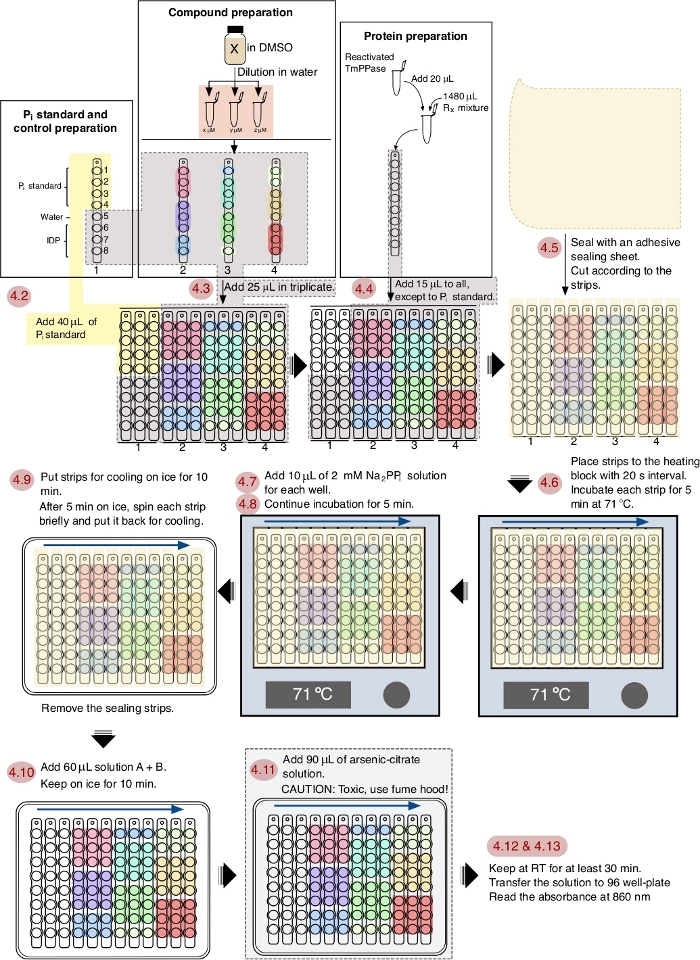
Abbildung 1: Ein schematischer Workflow des TmPPase-Inhibitions-Assays in einem 96-Well-Plattenformat. Die rote Nummerierung zeigt die Schritte des Assays entsprechend dem Protokoll und die blauen Pfeile zeigen die Intervallreihenfolge an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
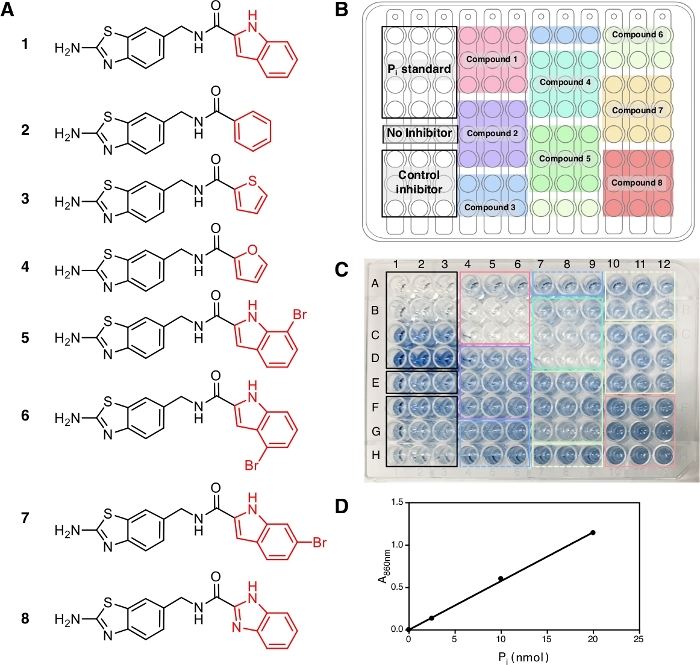
Abbildung 2: Proben, ihre Anordnung und Farbentwicklung in einer 96-Well-Platte. (A) Die Strukturen der Verbindungen 1x8, die für den Assay verwendet werden. Die Hemmungsaktivität dieser Verbindungen wurde in Vidilaseris et al.9berichtet. (B) Stichprobenanordnung. (C) Farbentwicklung, 30 min nach Zugabe von Arsenit-Citrat-Lösung. Die Konzentrationen von Kontrollinhibitoren (IDP) und proben, die von oben nach unten angeordnet sind, liegen bei der Konzentration von 2,5 m, 25 m und 250 m und der Konzentration von 1 m, 5 m bzw. 20 m. Die Intensität der blauen Farbe entspricht der Menge an Pi, die aufgrund der enzymatischen Reaktion freigesetzt wird, und der Mangel an Farbe entspricht keiner enzymatischen Reaktion. (D) Kalibrierkurve für Pi-Norm (nmol) gegen A860 mit linearer Passform (y = 0,0576x + 0,0019; r2 = 0,999). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
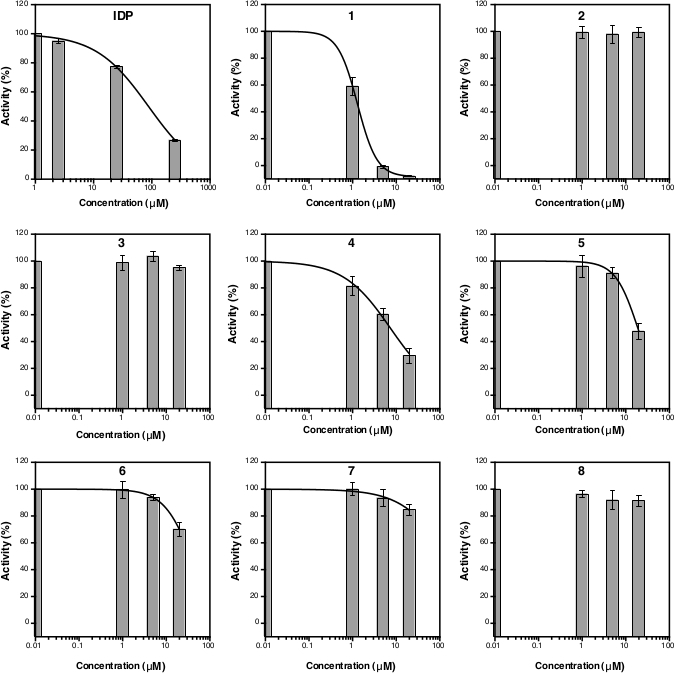
Abbildung 3: Kurve der TmPPase-Prozentaktivität für drei verschiedene Inhibitorkonzentrationen. Die nichtlinearen Regressionskurven zur Berechnung des IC50 (Schätzung) werden sowohl für IDP als auch für die Verbindungen 1, 4, 5, 6 und 7 angezeigt, jedoch nicht für die Verbindungen 2, 3und 8, da sie die TmPPase-Aktivität bei den Assay-Konzentrationen nicht hemmten. Die logIC50 und IC50 (Schätzung) jeder Verbindung sind in Tabelle 1dargestellt. Alle Daten werden als mittlere SD mit drei Replikationen angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Hemmungskurve von acht Konzentrationspunkten der Verbindungen 1, 5, 6, 7 und 8. Diese Figur stammt von Vidilaseris et al.9 mit leichter Modifikation. Alle Daten werden als mittlere SD mit drei Replikationen angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Beispiel | LogIC50 | IC50 (Schätzung) (M) |
| Idp | 1,95 € 0,0142 | 87,9 € 2,46 |
| 1 | 0,112 bei 0,0274 | 1,29 € 0,0816 |
| 2 | − | keine Hemmung |
| 3 | − | keine Hemmung |
| 4 | 0,870 bei 0,0447 | 7,39 € 0,760 |
| 5 | 1,28 € 0,0296 | 19,0 € 1,29 |
| 6 | 1,57 bei 0,0846 | 37,4 x 7,29 |
| 7 | 2,19 x 0,366 | 156 x 131 |
| 8 | − | keine Hemmung |
Tabelle 1: LogIC50 und IC50 (Schätzung) von IDP und Verbindungen 1-8 auf der Grundlage der Daten aus Abbildung 3.
Diskussion
Hier berichten wir über ein detailliertes Protokoll zum einfachen Screening von Inhibitoren auf membrangebundene Pyrophosphatase aus T. maritima in einem 96-Well-Plattenformat auf Basis von Vidilaseris et al.14. Dieses Protokoll ist kostengünstig und basiert auf 12-Phosphomolybdadsäure, die aus Orthophosphat und Molybdat unter sauren Bedingungen gebildet und auf Phosphomolybdän-Arten mit einer deutlichen blauen Farbe12reduziert wird. Diese Methode wird gegenüber anderen Protokollen bevorzugt, wie z. B. dem empfindlicheren Malachit-Grüntest16, da diese Methode keine Interferenz in Gegenwart einer hohen Phospholipidkonzentration aufweist, die für die TmPPase-Reaktivierung14erforderlich ist.
Der Workflow des Screening-Protokolls ist in Abbildung 1 dargestellt und dieser Vorgang kann in 1 h vollständig durchgeführt werden. Dieses Protokoll ist für TmPPase mit der optimalen Arbeitstemperatur bei 71 °C und einer Reaktionszeit von 5 min optimiert. Da Wasser bei dieser Temperatur aus dem Reaktionsgemisch verdunstet, wird ein Klebe-Dichtungsblech (geschnitten, um die Streifen zu montieren und zu bedecken) aufgebracht, um verdunsten14 zu verhindern, und das verdampfte Wasser wird einfach mit Zentrifugation wiederhergestellt. Die 5 min Inkubationszeit wird gewählt, da sie sich noch im linearen Bereich des enzymatisch freigesetzten Phosphats befindet und für ein zuverlässiges Screening ausreichend ist14. In diesem Protokoll sind die Timing- und Pipettierfähigkeiten wichtige Faktoren, um ein gutes und zuverlässiges Ergebnis zu erzielen. Die Zugabe von Reagenzien während des Assays mit 20 s Intervall zwischen den Streifen ist eine optimierte Timing-Option für die einfache Durchführung der nachfolgenden Schritte.
Bei verschiedenen mPPases sollte die optimale Temperatur und Inkubationszeit vor der Verwendung im Hemmungstest separat bestimmt werden. Das oben genannte Enzym-Reaktivierungsprotokoll ist für TmPPase optimiert und andere mPPases benötigen möglicherweise ein anderes Reaktivierungsprotokoll. Beispielsweise sollte DDM nicht zur Reaktivierung von mPPase von Pyrobaculum aerophilum hinzugefügt werden, da es seine enzymatische Aktivität verringert17. Da das Enzym weniger aktiv wird, wenn es rechtzeitig zubereitet wird, sollte dem Reaktionsgemisch kurz vor Beginn des Testes die Zugabe eines reaktivierten Enzyms zugesetzt werden. Nach Zugabe der Arsenit-Citrat-Lösung ist das Reaktionsprodukt für mindestens 5 h14stabil. Daher kann die nächste Charge des Assays sofort durchgeführt werden, und die Absorptionsmessung kann später für alle Chargen auf einmal durchgeführt werden.
Offenlegungen
Die Autoren haben nichts zu verraten.
Danksagungen
Diese Arbeit wurde durch die Stipendien der Jane and Aatos Erkko Foundation und der BBSRC (BB/M021610) an Adrian Goldman, die Akademie Finnlands (Nr. 308105) an Keni Vidilaseris,(Nr. 310297) an Henri Xhaard und (Nr. 265481) an Jari Yli-Kauhaluoma und die Forschungsfonds der Universität Helsinki an Gustav Boije af Gennäs. Die Autoren danken Bernadette Gehl für ihre technische Hilfe während des Projekts.
Materialien
| Name | Company | Catalog Number | Comments |
| Adhesive sealing sheet | Thermo Scientific | AB0558 | |
| Ammonium heptamolybdate tetrahydrate | Merck | F1412481 636 | |
| Ascorbic acid | Sigma-Aldrich | 95212-250G | |
| BioLite 96Well Multidish | Thermo Scientific | 130188 | |
| Dimethyl sulfoxide (DMSO) | Merck | 1167431000 | |
| 8-well PCR Tube Strips 0.2 ml without caps (120) | Nippon genetics | FG-028 | |
| Dodecyl maltoside (DDM) | Melford | B2010-100G | |
| Ethanol | Merck | 1009901001 | |
| Glacial acetic acid | Merck | 1000631011 | |
| Hydrochloric acid | Sigma-Aldrich | 258148-500ML | |
| Imidodiphosphate sodium salt | Sigma-Aldrich | I0631-1G | |
| L-α-Phosphatidyl choline from soybean lecithin | Sigma | 429415-100GM | |
| Magnesium chloride | Sigma-Aldrich | 8147330500 | |
| Multiplate 96-Well PCR Plates | Bio-Rad | MLL9651 | |
| MultiSkan Go | Thermo Scientific | 10680879 | |
| Nepheloskan Ascent (Type 750) | Labsystems | ||
| Polystyrene Petri dish (size 150 mm x 15 mm) | Sigma-Aldrich | P5981-100EA | |
| Potassium chloride | Merck | 104936 | |
| Prism 6 software | GraphPad | ||
| QBT2 Heating block | Grant Instruments | ||
| Sodium meta-arsenite | Fisher Chemical | 12897692 | |
| Sodium phosphate dibasic (Pi) | Sigma | S0876-1KG | |
| Sodium pyrophosphate dibasic | Fluka | 71501-100G | |
| Trisodium citrate dihydrate | Fluka | 71404-1KG |
Referenzen
- Terstappen, G. C., Reggiani, A. In silico research in drug discovery. Trends in Pharmacological Sciences. 22 (1), 23-26 (2001).
- Rask-Andersen, M., Almen, M. S., Schioth, H. B. Trends in the exploitation of novel drug targets. Nature Reviews Drug Discovery. 10 (8), 579-590 (2011).
- Shah, N. R., Vidilaseris, K., Xhaard, H., Goldman, A. Integral membrane pyrophosphatases: a novel drug target for human pathogens. AIMS Biophysics. 3 (1), 171-194 (2016).
- Baykov, A. A., Malinen, A. M., Luoto, H. H., Lahti, R. Pyrophosphate-Fueled Na+ and H+ Transport in Prokaryotes. Microbiology and Molecular Biology Reviews. 77 (2), 267-276 (2013).
- Serrano, A., Perez-Castineira, J. R., Baltscheffsky, M., Baltscheffsky, H. H+-PPases: yesterday, today and tomorrow. IUBMB Life. 59 (2), 76-83 (2007).
- Liu, J., et al. A vacuolar-H+-pyrophosphatase (TgVP1) is required for microneme secretion, host cell invasion, and extracellular survival of Toxoplasma gondii. Molecular Microbiology. 93 (4), 698-712 (2014).
- Lemercier, G., et al. A pyrophosphatase regulating polyphosphate metabolism in acidocalcisomes is essential for Trypanosoma brucei virulence in mice. Journal of Biological Chemistry. 279 (5), 3420-3425 (2004).
- Belogurov, G. A., et al. Membrane-bound pyrophosphatase of Thermotoga maritima requires sodium for activity. Biochemistry. 44 (6), 2088-2096 (2005).
- Vidilaseris, K., et al. Asymmetry in catalysis by Thermotoga maritima membrane-bound pyrophosphatase demonstrated by a nonphosphorus allosteric inhibitor. Science Advances. 5 (5), (2019).
- Lin, S. M., et al. Crystal structure of a membrane-embedded H+-translocating pyrophosphatase. Nature. 484 (7394), 399-403 (2012).
- Fiske, C. H., Subbarow, Y. The colorimetric determination of phosphorus. Journal of Biological Chemistry. 66 (2), 375-400 (1925).
- Nagul, E. A., McKelvie, I. D., Worsfold, P., Kolev, S. D. The molybdenum blue reaction for the determination of orthophosphate revisited: Opening the black box. Analytica Chimica Acta. 890, 60-82 (2015).
- Kellosalo, J., Kajander, T., Palmgren, M. G., Lopez-Marques, R. L., Goldman, A. Heterologous expression and purification of membrane-bound pyrophosphatases. Protein Expression and Purification. 79 (1), 25-34 (2011).
- Vidilaseris, K., Kellosalo, J., Goldman, A. A high-throughput method for orthophosphate determination of thermostable membrane-bound pyrophosphatase activity. Analytical Methods. 10 (6), 646-651 (2018).
- He, Z. Q., Honeycutt, C. W. A modified molybdenum blue method for orthophosphate determination suitable for investigating enzymatic hydrolysis of organic phosphates. Communications in Soil Science and Plant Analysis. 36 (9-10), 1373-1383 (2005).
- Martin, B., Pallen, C. J., Wang, J. H., Graves, D. J. Use of fluorinated tyrosine phosphates to probe the substrate specificity of the low molecular weight phosphatase activity of calcineurin. Journal of Biological Chemistry. 260 (28), 14932-14937 (1985).
- Strauss, J., Wilkinson, C., Vidilaseris, K., Harborne, S. P. D., Goldman, A. A simple strategy to determine the dependence of membrane-bound pyrophosphatases on K+ as a cofactor. Methods in Enzymology. 607, 131-156 (2018).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten