Method Article
Detección de inhibidores de la pirofosfatasa ligadas a la membrana Thermotoga maritima
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí presentamos un método de cribado para inhibidores de la pirofosfatasa ligada a la membrana (de Thermotoga maritima)basado en la reacción azul de molibdeno en un formato de placa de 96 pocillos.
Resumen
Las pirofosfasiasas ligadas a la membrana (mPPases) son enzimas dimericas que se producen en bacterias, arqueas, plantas y parásitos protistas. Estas proteínas clan el pirofosfato en dos moléculas de ortofosfato, que se combina con el bombeo de protones y/o iones de sodio a través de la membrana. Dado que no se producen proteínas homólogas en animales y humanos, los mPPases son buenos candidatos en el diseño de posibles objetivos farmacológicos. Aquí presentamos un protocolo detallado para detectar inhibidores de mPPase utilizando la reacción azul de molibdeno en un sistema de placa de pozo 96. Utilizamos mPPase de la bacteria termofílica Thermotoga maritima (TmPPase) como enzima modelo. Este protocolo es simple y económico, produciendo un resultado consistente y robusto. La medición de la absorbancia tarda sólo una hora en completar el protocolo de ensayo de actividad desde el inicio del ensayo hasta la medición de absorción. Dado que el color azul producido en este ensayo es estable durante un largo período de tiempo, los ensayos posteriores se pueden realizar inmediatamente después del lote anterior, y la absorbancia se puede medir más tarde para todos los lotes a la vez. El inconveniente de este protocolo es que se hace manualmente y por lo tanto puede ser agotador, así como requerir buenas habilidades de pipeteo y mantenimiento del tiempo. Además, la solución de arsénito-citrato utilizada en este ensayo contiene arsnito sódico, que es tóxico y debe tratarse con las precauciones necesarias.
Introducción
Aproximadamente el 25% del total de proteínas celulares son proteínas de membrana y alrededor del 60% de ellas son dianas de fármacos1,2. Uno de los fármacos potenciales se dirige a3, pirofosfatasas ligadas a la membrana (mPPases), son las enzimas dimericas que bombean protones y/o iones de sodio a través de la membrana por hidrólisis de pirofosfato en dos ortofosfatos4. mPPases se pueden encontrar en varios organismos5 tales como bacterias, arqueas, plantas, y parásitos protistas, con la excepción de los seres humanos y animales4. En los parásitos protistas, por ejemplo Plasmodium falciparum, Toxoplasma gondii y Trypanosoma brucei,las mPPases son esenciales para la virulencia del parásito6 y el nocaut de esta expresión en los parásitos conducen a la falla en el mantenimiento del pH intracelular tras la exposición al pH básico externo7. Debido a su importancia y falta de proteína homóloga presente en los vertebrados, las mPPases pueden considerarse como posibles dianas farmacológicas para enfermedades protistales3.
El cribado in vitro de inhibidores de mPPase en este trabajo se basa en un sistema modelo TmPPase. TmPPase es un mPPase dependiente del bombeo de iones de sodio y iones de potasio de T. maritima y tiene su actividad óptima a 71oC8. Los beneficios de esta enzima son, por ejemplo, su facilidad en la producción y purificación, una buena estabilidad térmica y una alta actividad específica. TmPPase muestra tanto una alta similitud además de la conservación completa de la posición como la identidad de todos los residuos catalíticos al protista mPPases3,9 y a la estructura resuelta de Vigna radiata10 mPPase. Las estructuras disponibles de TmPPase en diferentes conformaciones también son útiles para el experimento de diseño de fármacos basado en estructuras (como cribado virtual y diseño de novo).
Aquí informamos de un protocolo detallado para el cribado de inhibidores de TmPPase en un formato de placa de pozo 96 (Figura 1). El protocolo se basa en el método colorimétrico de la reacción azul de molibdeno, que fue desarrollado por primera vez por Fiske y Subbarow11. Este método implica la formación de ácido 12-fosfomolíbdico a partir de ortofosfato y molibdato en condiciones ácidas, que luego se reduce para dar especies características de fosfomolibdeno de color azul12.
Protocolo
1. Preparación de proteínas
NOTA: La expresión y purificación de TmPPase se ha descrito en otros lugares13.
- Preparar 10 ml de la solución tampón de reactivación que contiene 20 mM 2-(N-morpholino)ácido etanosulfónico (MES) pH 6,5, 3,5% (v/v) glicerol, ditiothreitol de 2 mM (TDT) y 0,05% de dodecil maltoside (DDM).
- Preparar 10 ml de la mezcla de reacción que contiene 200 mM Tris-Cl pH 8.0, 8.0 mM MgCl2, 333 mM KCl, y 67 mM NaCl.
NOTA: Se requiere Mg2+ para quelar el pirofosfato ya que el sustrato de mPPase, K+ es necesario para aumentar la actividad enzimática ya que TmPPase es un mPPase dependiente del potasio, y Na+ es necesario para la actividad enzimática durante la translocación de iones de sodio por TmPPase. - Preparar liposomas de 30 mg/ml para la reactivación enzimática.
- Añadir 10 mL de 20 mM Tris-HCl pH 8.0 con 1 mM de TDT a 0,3 g de L-a-fosfatidilcolina de soja a 10 mL de 20 mM Tris-HCl pH 8.0 con 1 mM de TDT.
- Ponga el liposoma en hielo, y sonicar con 1 segundo intervalo de pulso durante 1 minuto, haga una pausa durante 1 minuto y repita hasta que la solución se vuelva transparente de color amarillo.
- Alícuota los liposomas, congelar en nitrógeno líquido y almacenar a -80 oC hasta que se utilice.
- Reactivar la enzima.
- Mezclar 40 sl de la solución de liposomas con 22,5 s de 20% dDM.
- Calentar la mezcla a 55 oC durante 15 minutos y dejar que se enfríe a temperatura ambiente.
- Añadir 36,5 l de la solución tampón de reactivación, mezclar y añadir 1 l de proteína concentrada (13 mg/ml) para realizar una concentración total de 0,13 mg/ml.
NOTA: Las proteínas generalmente se congelan en alícuotas de 10 l después de la purificación y se descongelan sobre hielo antes de su uso.
- Tomar 20 s de la enzima reactivada y añadir a 1.480 s de la mezcla de reacción, luego mezclar suavemente.
NOTA: La adición de la enzima reactivada a la mezcla de reacción debe realizarse justo antes de su uso.
2. Preparación de compuestos
- Disolver los compuestos en dimetil sulfóxido (DMSO) para hacer soluciones de stock de 25 a 100 mM en 50-200 l, en función de la disponibilidad de los compuestos.
NOTA: Todos los compuestos utilizados aquí(Figura 2A)se han publicado previamente9. Si la solubilidad compuesta es baja, la concentración de stock se puede ajustar en consecuencia. - Preparar tres concentraciones diferentes de cada compuesto en agua.
NOTA: Las concentraciones finales en la mezcla de reacción serán 1, 5 y 50 micromolares o 1, 5 y 20 micromolares para compuestos solubles y ahorradormente solubles, respectivamente.- Diluir la solución en stock con agua a 1 ml en microtubos para dar 2 m, 10 m y 100 m para compuestos solubles, o, alternativamente, 2 m, 10 m y 40 m para compuestos escasamente solubles.
- Vortex la solución compuesta al instante después de la dilución de la solución de stock para una mezcla adecuada.
- Compruebe la agregación compuesta utilizando un nefélmetro.
NOTA: Esto se estudió como triplicados en tres concentraciones (1 M, 5 m y 20 m) y se normalizó al espacio en blanco en una placa de 96 pocillos.- Dispensar 75 l de la mezcla de reacción en cada poca utilizando una pipeta multicanal.
- Añadir 75 l de cada compuesto (para el espacio en blanco, utilizar 75 l de agua en su lugar) y mezclar pipeteando hacia arriba y hacia abajo 5o.
- Mida cada pocómetro a 300 V utilizando un nefélmetro de microplaca.
3. Reactivos para la preparación del ensayo
- Prepare la solución de arsénito-citrato.
- Pesar 5 g de arsenita sódica y 5 g de citrato de trisódico dihidrato.
ADVERTENCIA: El arsénito de sodio es tóxico, por lo tanto, utilice el equipo de protección adecuado y manipule con especial cuidado. Como precaución, no manipule antes de que se hayan leído y comprendido todas las precauciones de seguridad necesarias. Manipular sólo en una campana de humos con el fin de no inhalar polvo / vapores del compuesto o su(s) solución(es). Si se inhala, muévase al aire fresco y obtenga atención médica. Use gafas de seguridad químicas adecuadas, guantes de protección y ropa para evitar la ingestión y el contacto con los ojos y la piel. En caso de ingestión, llame inmediatamente a un centro de toxicología o a un médico/médico. Si se pone en la piel o en los ojos, lavar con abundante agua y obtener atención médica. - Disolver en 100 ml de agua.
- Añadir 5 ml de ácido acético glacial, mezclar y añadir agua a 250 ml.
- Conservar a temperatura ambiente protegido de la luz.
NOTA: La solución es estable durante más de un año.
- Pesar 5 g de arsenita sódica y 5 g de citrato de trisódico dihidrato.
- Preparar la solución A y la solución B.
- Para la solución A, añadir 10 ml de hielo frío 0,5 M HCl a 0,3 g de ácido ascórbico. Disolver el ácido ascórbico por vórtice.
- Para la solución B, añadir 1 ml de agua helada a 70 mg de tetrahidrato de heptamoybdato de amonio y vórtice para disolver.
NOTA: Almacene ambas soluciones en hielo hasta su uso. Para la consistencia del resultado del ensayo, ambas soluciones se pueden almacenar en hielo durante un máximo de una semana.
- Preparar el estándar de fosfato (Pi) con una concentración de 0 m, 62,5 m, 250 m y 500 m para la calibración.
- Añadir 0 sL, 25 l, 50 sl y 100 ml de 5 mM Na2HPO4 dihidrato a cuatro microtubos que contengan 370 ml de la mezcla de reacción.
- Cubra hasta 1 ml con agua.
4. Ensayo de actividad para una placa de 96 pocillos
NOTA: Consulte la Figura 1 para ver el flujo de trabajo esquemático del ensayo.
- Añadir 1 ml de solución B a 10 ml de solución A, mezclar por vórtice y almacenar la solución en hielo.
NOTA: Esta solución debe ser transparente y amarilla. Mantenga la solución A + B sobre hielo durante al menos 30 minutos antes de su uso. Sin embargo, utilice la solución dentro de 3 h, ya que irá mal después del almacenamiento a largo plazo. - Añadir 40 ml de 0 m, 62,5 m, 250 m y 500 m de pi estándar a las tiras de tubo en triplicado utilizando una pipeta multicanal.
NOTA: La mezcla de reacción sin Pi añadido se utilizará como blanco. - Añadir 25 l de solución compuesta a las tiras de tubo utilizando una pipeta multicanal.
NOTA: Cada compuesto tiene tres concentraciones diferentes en triplicado que es suficiente para la estimación inicial de la concentración inhibitoria media máxima (IC50). Para una determinación más precisa de IC50, se pueden utilizar ocho concentraciones compuestas diferentes. Para la enzima desinhibida, la solución compuesta se sustituye por la misma cantidad de agua. Como controles positivos se utilizaron 2,5 m, 25 m y 250 m de sal sódica de imidodifosfato (IDP). - Añadir 15 l de mezcla de solución de mPPase a las tiras de tubo (excepto a los tubos que contienen el estándar Pi) utilizando una pipeta multicanal.
- Sellar las tiras de tubo con una lámina de sellado adhesiva. Corte la hoja de sellado para separar cada tira de tubo.
- Pre-incubar las muestras durante 5 min a 71oC. Coloque las muestras en el bloque de calentamiento con un intervalo de 20 s entre cada tira para minimizar el consumo de tiempo durante los pasos posteriores.
- Para cada tira, abra el sellado adhesivo. Añadir 10 ml de pirofosfato sódico de 2 mM dibásico utilizando una pipeta multicanal y mezclar pipeteando hacia arriba y hacia abajo durante 5o. Vuelva a sellar la tira del tubo con el mismo sellado.
NOTA: Este paso podría ser inicialmente difícil de lograr en 20 s; sin embargo, será más fácil después de algunos ensayos. - Incubar a 71oC durante 5 min.
- Coloque las muestras en el aparato de refrigeración con un intervalo de 20 s entre cada tira. Dejar que se enfríen durante 10 minutos, pero centrifugar cada tira brevemente después de 5 minutos de enfriamiento, para decantar gotas de agua debajo de la hoja de sellado, luego ponerla de nuevo en el aparato de enfriamiento y quitar el sellado.
NOTA: El aparato de refrigeración se puede hacer simplemente colocando una placa PCR de 96 pocillos en una placa de poliestireno Petri (tamaño 150 mm x 15 mm) llena de agua y congelada durante al menos 1 h. El aparato debe extraerse del congelador unos 5 minutos antes del comienzo del ensayo. No saque el aparato de refrigeración justo antes de la refrigeración de la muestra, ya que congelará la mezcla de reacción y obstaculizará el desarrollo del color. - Después de 10 minutos de enfriamiento, añadir 60 sl de solución A + B, mezclar pipeteando hacia arriba y hacia abajo durante 5o y mantener las tiras de tubo en el aparato de refrigeración durante 10 minutos.
- Añadir 90 l de la solución de arsénito-citrato y mantener a temperatura ambiente durante al menos 30 minutos para producir un color azul estable.
ADVERTENCIA: Debido a su toxicidad, todas las soluciones que contienen arsenito sódico deben tratarse con especial cuidado en todo momento. Por lo tanto, la adición de solución de arsénito-citrato debe hacerse en una campana de humo. - Dispensar 180 ml de cada mezcla de reacción en una microplaca de poliestireno transparente de 96 pocillos.
- Mida la absorbancia de cada pocal a 860 nm utilizando un espectrofotómetro de microplacas.
5. Análisis de resultados
- Promedio de los triplicados de cada muestra y los estándares Pi. A continuación, reste con el espacio en blanco para eliminar la señal de fondo.
- Haga una curva de calibración trazando los valores de absorbancia (A860)con respecto a la cantidad de estándar Pi (nmol) y realice una regresión lineal para obtener la función de línea de tendencia utilizando la siguiente fórmula:
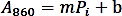
- Calcule la cantidad de fosfato (nmol) liberada de la reacción enzimática basándose en la fórmula de regresión lineal anterior.
- Calcule la actividad específica utilizando la siguiente fórmula:
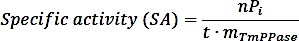
donde nPi es la cantidad de fosfato liberada de la reacción (nmol), t es el tiempo de reacción (min), y mTmPPase es la cantidad de la TmPPase pura utilizada en el ensayo (mg). - Calcule el porcentaje de actividad para cada concentración de inhibidores utilizando la siguiente fórmula:
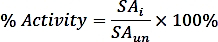
donde SAiis la actividad específica de una muestra con inhibidor y SAun es la actividad específica de la muestra desinhibida. - Calcule el logIC50 (estimación) y el IC50 (estimación) con un ajuste de regresión no lineal de la curva dosis-respuesta de cuatro parámetros utilizando la siguiente fórmula:
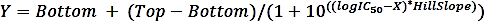
donde X es el registro de concentración (M), Y es actividad (%), Top e Bottom son mesetas en la misma unidad que Y (100% y 0%, respectivamente), logIC50 tiene las mismas unidades de tronco que X, y HillSlope - factor de pendiente o pendiente de la colina, que es sin unidad.
NOTA: El software(Tabla de materiales)se utiliza para el montaje. Utilice la concentración de 0,01 m (en lugar de 0,00 m) para la muestra sin inhibidor, ya que el laritmo de cero no está definido.
Resultados
En este protocolo, se probaron ocho compuestos (1-8)(Figura 2A)junto con IDP, un inhibidor común de las pirofosfalasas, como un control positivo. Cada compuesto se probó a tres concentraciones diferentes (1 M, 5 m y 20 m) en triplicado. El flujo de trabajo del cribado se muestra en la Figura 1,a partir de la preparación de muestras y reactivos hasta la medición de absorbancia a 860 nm.
Al final de este protocolo, tras la adición de la solución A + B y el arsénito-citrato, las soluciones desarrollan un color azul estable con la absorción máxima a 709 nm y 860 nm14 debido a la compleja formación de iones de fosfato con molibdato que se pueden observar y muestra la ocurrencia de la reacción enzimática. Para este experimento, utilizamos la absorbancia a 860 nm para la medición de la cantidad de Pi liberada ya que tiene un mejor límite de detección y sensibilidad en comparación con la absorbancia a 709 nm15. El color azul está completamente desarrollado en 30 min de incubación a temperatura ambiente y estable durante al menos 5 h14. El ensayo tiene la sensibilidad hasta una concentración de Pi de 10 m y la absorbancia es lineal en un rango de concentración de 10 a 800 m14. En el resultado representativo aquí, los pozos E1-E3(Figura 2C)contienen la mezcla de reacción sin inhibidor y la solución azul se puede observar al final del ensayo. Esto también se puede observar a concentraciones compuestas bajas donde no se ha alcanzado la inhibición completa, como en los pozos F1-F3 para el IDP y los pozos A4-A6 para el compuesto 1 (ATC, un inhibidor no competitivo recientemente conocido de TmPPase9) a la concentración de 2,5 m y 1 M, respectivamente. Se puede observar la mayor concentración de IDP y compuesto 1, el color azul menor o nula (G1-G3 y H1-H3 para IDP y B4-B6 y C4-C6 para el compuesto 1) que indican la inhibición de la actividad enzimática. Las tres concentraciones de compuestos no inhibidores (2, 3, y 8) mostraron la misma intensidad de color azul que los pozos E1-E3 sin ningún inhibidor (Figura 2C).
Después de la medición de la absorbancia a 860 nm, los datos pueden ser procesados y analizados (ver sección 5 del protocolo). La Figura 2D muestra la gráfica de calibración del estándar Pi con su ajuste lineal (y 0,0576x + 0,0019; r2 a 0,999). La Figura 3 muestra la gráfica de la actividad enzimática (%) contra la concentración de cada compuesto probado. Para compuestos con actividad de inhibición, también se muestra un empalme de curva no lineal. IdP, utilizado como un control positivo, muestra claramente una disminución en la actividad a mayor concentración. El IC50 (estimación) calculado sobre la base de tres concentraciones diferentes es de 88,2 m(Tabla 1),que es similar a la medición anterior (80,0 m) con ocho puntos de concentración14. Los compuestos 1, 4, 5, 6y 7 mostraron una tendencia similar a la de IDP, ya que la concentración aumentó con el IC50 (estimación) de aproximadamente 1,3 m, 7,4 m, 19,0 m, 37,4 m y 156,1 m, respectivamente(Tabla 1). Para los compuestos 2, 3, y 8 no se puede observar ninguna reducción en la actividad o inhibición en las concentraciones de ensayo. Se puede realizar un ensayo adicional con ocho puntos de concentración para generar IC50preciso. La Figura 4 muestra la curva de inhibición para los compuestos 1, 5, 6, 7 y 8 con un IC50 de 1,7 M, 21,4 m, 58,8 m, 239,0 m y >500 m, respectivamente9.
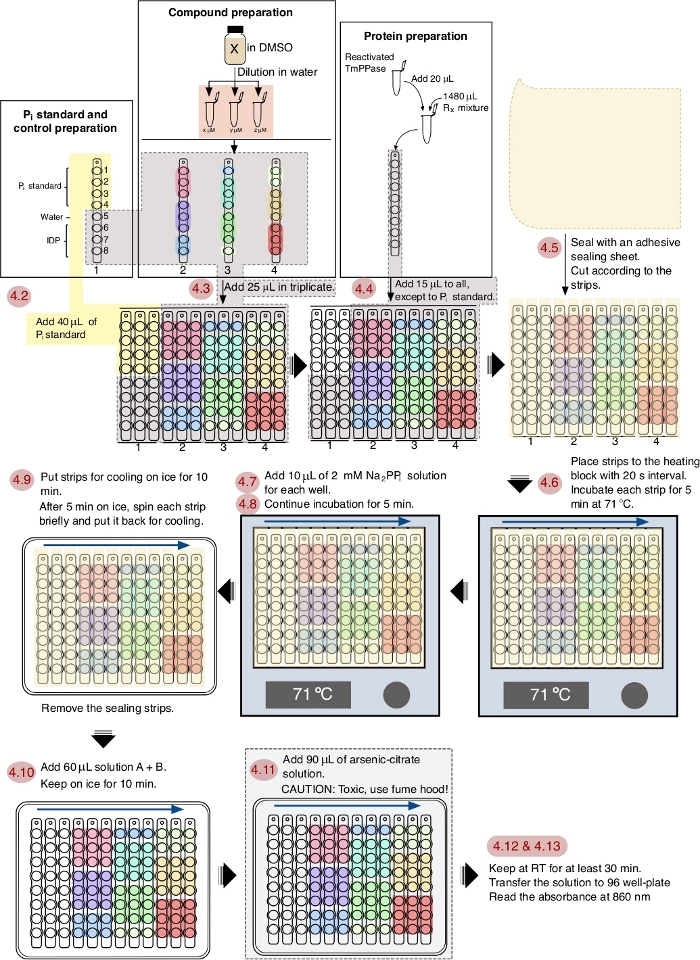
Figura 1: Un flujo de trabajo esquemático de ensayo de inhibición de TmPPase en un formato de placa de 96 pocillos. La numeración roja muestra los pasos del ensayo según el protocolo y las flechas azules muestran el orden del intervalo. Haga clic aquí para ver una versión más grande de esta figura.
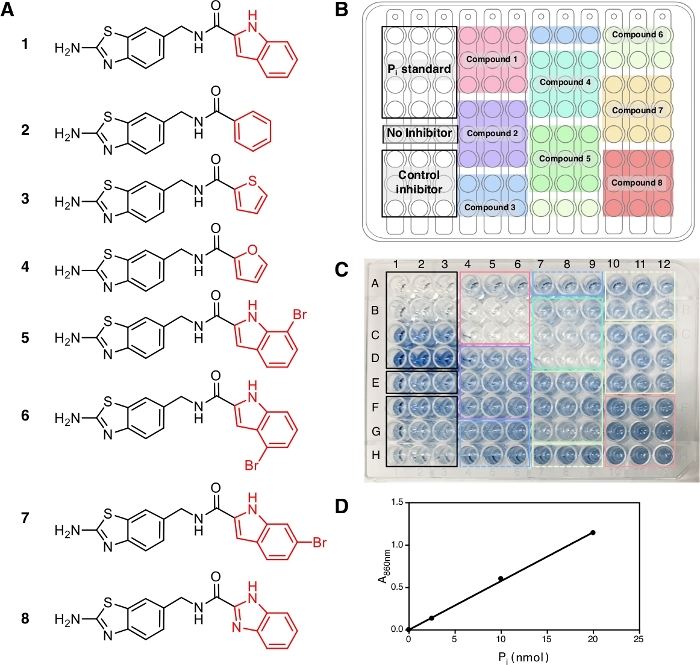
Figura 2: Muestras, su disposición y desarrollo de color en una placa de 96 pozos. (A) Las estructuras de los compuestos 1x8 utilizados para el ensayo. La actividad de inhibición de estos compuestos se ha divulgado en Vidilaseris et al.9. (B) Disposición de muestra. (C) Desarrollo de color, 30 min después de la adición de solución de arsénito-citrato. Las concentraciones de inhibidor de control (IDP) y muestras utilizadas, dispuestas de arriba a abajo, son de 2,5 oM, 25 m y 250 m de concentración y 1 m, 5 m y 20 m de concentración, respectivamente. La intensidad del color azul corresponde a la cantidad de Pi liberada debido a la reacción enzimática y la falta de color corresponde a ninguna reacción enzimática. (D) Curva de calibración para Pi estándar (nmol) contra A860 con empalme lineal (y a 0,0576x + 0,0019; r2 a 0,999). Haga clic aquí para ver una versión más grande de esta figura.
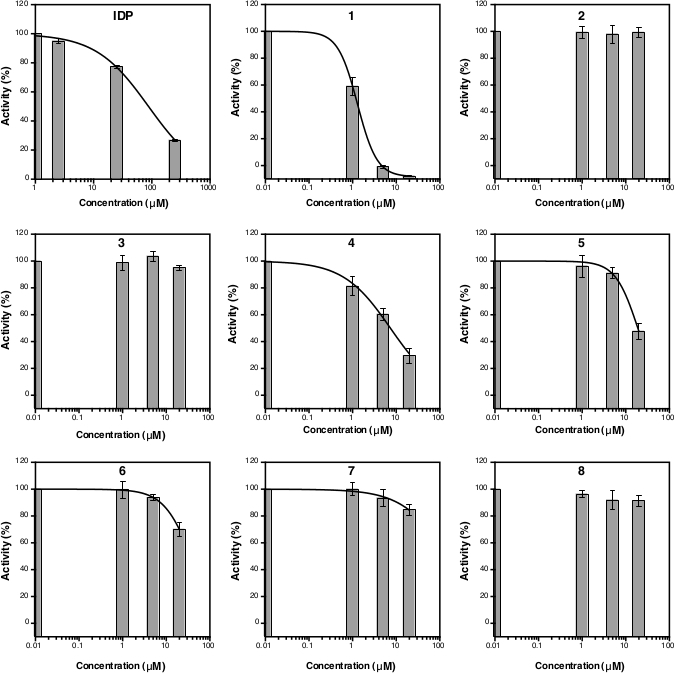
Figura 3: Curva de la actividad porcentual de TmPPase para tres concentraciones de inhibidores diferentes. Las curvas de regresión no lineales para calcular el IC50 (estimación) se muestran para el IDP, así como para los compuestos 1, 4, 5, 6 y 7 pero no para los compuestos 2, 3, y 8 ya que no estaban inhibiendo la actividad de TmPPase en las concentraciones de ensayo. El logIC50 y el IC50 (estimación) de cada compuesto se muestra nado en la Tabla 1. Todos los datos se muestran como medias - SD con tres réplicas. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Curva de inhibición a partir de ocho puntos de concentración de compuestos 1, 5, 6, 7 y 8. Esta figura está tomada de Vidilaseris et al.9 con ligera modificación. Todos los datos se muestran como medias - SD con tres réplicas. Haga clic aquí para ver una versión más grande de esta figura.
| Muestra | LogIC50 | IC50 (estimación) (M) |
| Idp | 1,95 á 0,0142 | 87,9 x 2,46 |
| 1 | 0,112 a 0,0274 | 1.29 a 0,0816 |
| 2 | − | sin inhibición |
| 3 | − | sin inhibición |
| 4 | 0,870 a 0,0447 | 7,39 á 0,760 |
| 5 | 1.28 a 0,0296 | 19,0 a 1,29 |
| 6 | 1.57 a 0,0846 | 37,4 x 7,29 |
| 7 | 2,19 a 0,366 | 156 x 131 |
| 8 | − | sin inhibición |
Tabla 1: LogIC50 e IC50 (estimación) de IDP y compuestos 1-8 basados en los datos de la Figura 3.
Discusión
Aquí informamos de un protocolo detallado para el cribado simple de inhibidores de la pirofosfatasa ligada a la membrana de T. maritima en un formato de placa de 96 pozos basado en Vidilaseris et al.14. Este protocolo es barato y se basa en ácido 12-fosfomolíbódico, que se forma a partir de ortofosfato y molibdato en condiciones ácidas y reducido a especies de fosfomolibdeno con un color azul distintivo12. Este método es preferido sobre otros protocolos, como el ensayo verde malaquita más sensible16,porque este método no muestra interferencia en presencia de alta concentración de fosfolípidos que se requiere para la reactivación de TmPPase14.
El flujo de trabajo del protocolo de cribado se muestra en la Figura 1 y este proceso se puede realizar completamente en 1 h. Este protocolo está optimizado para TmPPase con la temperatura de trabajo óptima a 71 oC y un tiempo de reacción de 5 minutos. Como el agua se evaporará a esta temperatura de la mezcla de reacción, se aplica una lámina de sellado adhesiva (cortada para encajar y cubrir las tiras) para evitar la evaporación14 y el agua evaporada simplemente se recoge con centrifugación. El tiempo de incubación de 5 minutos se elige ya que todavía está en el rango lineal del fosfato liberado enzimáticamente y suficiente para un cribado fiable14. En este protocolo, las habilidades de cronometraje y pipeteo son factores importantes para obtener un resultado bueno y confiable. La adición de reactivos durante el ensayo con un intervalo de 20 s entre tiras es una opción de sincronización optimizada para facilitar la realización de los pasos posteriores.
Para diferentes mPPases, la temperatura óptima y el tiempo de incubación deben determinarse por separado antes de su uso en el ensayo de inhibición. El protocolo de reactivación de enzimas anterior está optimizado para TmPPase y otros mPPases podrían necesitar un protocolo de reactivación diferente. Por ejemplo, no se debe añadir DDM para la reactivación de mPPase de Pyrobaculum aerophilum, ya que disminuirá su actividad enzimática17. Como la enzima se volverá menos activa si se prepara con suficiente antelación, la adición de enzima reactivada debe añadirse a la mezcla de reacción poco antes de que se inicie el ensayo. Después de la adición de la solución de arsénito-citrato, el producto de reacción es estable durante al menos 5 h14. Por lo tanto, el siguiente lote del ensayo se puede realizar inmediatamente, y la medición de la absorbancia se puede hacer más adelante a todos los lotes a la vez.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue apoyado por las subvenciones de la Fundación Jane y Aatos Erkko y la BBSRC (BB/M021610) a Adrian Goldman, la Academia de Finlandia (no 308105) a Keni Vidilaseris, (no 310297) a Henri Xhaard, y (no 265481) a Jari Yli-Kauhaluoma, y los Fondos de Investigación de la Universidad de Helsinki a Gustav Boije af Genns. Los autores agradecen a Bernadette Gehl por su ayuda técnica durante el proyecto.
Materiales
| Name | Company | Catalog Number | Comments |
| Adhesive sealing sheet | Thermo Scientific | AB0558 | |
| Ammonium heptamolybdate tetrahydrate | Merck | F1412481 636 | |
| Ascorbic acid | Sigma-Aldrich | 95212-250G | |
| BioLite 96Well Multidish | Thermo Scientific | 130188 | |
| Dimethyl sulfoxide (DMSO) | Merck | 1167431000 | |
| 8-well PCR Tube Strips 0.2 ml without caps (120) | Nippon genetics | FG-028 | |
| Dodecyl maltoside (DDM) | Melford | B2010-100G | |
| Ethanol | Merck | 1009901001 | |
| Glacial acetic acid | Merck | 1000631011 | |
| Hydrochloric acid | Sigma-Aldrich | 258148-500ML | |
| Imidodiphosphate sodium salt | Sigma-Aldrich | I0631-1G | |
| L-α-Phosphatidyl choline from soybean lecithin | Sigma | 429415-100GM | |
| Magnesium chloride | Sigma-Aldrich | 8147330500 | |
| Multiplate 96-Well PCR Plates | Bio-Rad | MLL9651 | |
| MultiSkan Go | Thermo Scientific | 10680879 | |
| Nepheloskan Ascent (Type 750) | Labsystems | ||
| Polystyrene Petri dish (size 150 mm x 15 mm) | Sigma-Aldrich | P5981-100EA | |
| Potassium chloride | Merck | 104936 | |
| Prism 6 software | GraphPad | ||
| QBT2 Heating block | Grant Instruments | ||
| Sodium meta-arsenite | Fisher Chemical | 12897692 | |
| Sodium phosphate dibasic (Pi) | Sigma | S0876-1KG | |
| Sodium pyrophosphate dibasic | Fluka | 71501-100G | |
| Trisodium citrate dihydrate | Fluka | 71404-1KG |
Referencias
- Terstappen, G. C., Reggiani, A. In silico research in drug discovery. Trends in Pharmacological Sciences. 22 (1), 23-26 (2001).
- Rask-Andersen, M., Almen, M. S., Schioth, H. B. Trends in the exploitation of novel drug targets. Nature Reviews Drug Discovery. 10 (8), 579-590 (2011).
- Shah, N. R., Vidilaseris, K., Xhaard, H., Goldman, A. Integral membrane pyrophosphatases: a novel drug target for human pathogens. AIMS Biophysics. 3 (1), 171-194 (2016).
- Baykov, A. A., Malinen, A. M., Luoto, H. H., Lahti, R. Pyrophosphate-Fueled Na+ and H+ Transport in Prokaryotes. Microbiology and Molecular Biology Reviews. 77 (2), 267-276 (2013).
- Serrano, A., Perez-Castineira, J. R., Baltscheffsky, M., Baltscheffsky, H. H+-PPases: yesterday, today and tomorrow. IUBMB Life. 59 (2), 76-83 (2007).
- Liu, J., et al. A vacuolar-H+-pyrophosphatase (TgVP1) is required for microneme secretion, host cell invasion, and extracellular survival of Toxoplasma gondii. Molecular Microbiology. 93 (4), 698-712 (2014).
- Lemercier, G., et al. A pyrophosphatase regulating polyphosphate metabolism in acidocalcisomes is essential for Trypanosoma brucei virulence in mice. Journal of Biological Chemistry. 279 (5), 3420-3425 (2004).
- Belogurov, G. A., et al. Membrane-bound pyrophosphatase of Thermotoga maritima requires sodium for activity. Biochemistry. 44 (6), 2088-2096 (2005).
- Vidilaseris, K., et al. Asymmetry in catalysis by Thermotoga maritima membrane-bound pyrophosphatase demonstrated by a nonphosphorus allosteric inhibitor. Science Advances. 5 (5), (2019).
- Lin, S. M., et al. Crystal structure of a membrane-embedded H+-translocating pyrophosphatase. Nature. 484 (7394), 399-403 (2012).
- Fiske, C. H., Subbarow, Y. The colorimetric determination of phosphorus. Journal of Biological Chemistry. 66 (2), 375-400 (1925).
- Nagul, E. A., McKelvie, I. D., Worsfold, P., Kolev, S. D. The molybdenum blue reaction for the determination of orthophosphate revisited: Opening the black box. Analytica Chimica Acta. 890, 60-82 (2015).
- Kellosalo, J., Kajander, T., Palmgren, M. G., Lopez-Marques, R. L., Goldman, A. Heterologous expression and purification of membrane-bound pyrophosphatases. Protein Expression and Purification. 79 (1), 25-34 (2011).
- Vidilaseris, K., Kellosalo, J., Goldman, A. A high-throughput method for orthophosphate determination of thermostable membrane-bound pyrophosphatase activity. Analytical Methods. 10 (6), 646-651 (2018).
- He, Z. Q., Honeycutt, C. W. A modified molybdenum blue method for orthophosphate determination suitable for investigating enzymatic hydrolysis of organic phosphates. Communications in Soil Science and Plant Analysis. 36 (9-10), 1373-1383 (2005).
- Martin, B., Pallen, C. J., Wang, J. H., Graves, D. J. Use of fluorinated tyrosine phosphates to probe the substrate specificity of the low molecular weight phosphatase activity of calcineurin. Journal of Biological Chemistry. 260 (28), 14932-14937 (1985).
- Strauss, J., Wilkinson, C., Vidilaseris, K., Harborne, S. P. D., Goldman, A. A simple strategy to determine the dependence of membrane-bound pyrophosphatases on K+ as a cofactor. Methods in Enzymology. 607, 131-156 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados