Method Article
Dépistage des inhibiteurs de la pyrophosphatase Thermotoga maritima Membrane-Bound
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Ici nous présentons une méthode de criblage pour la pyrophosphatase membran-liée (de Thermotoga maritima) inhibiteurs basés sur la réaction bleue de molybdène dans un format 96 de plaque de puits.
Résumé
Les pyrophosphatases membranaires (mPPases) sont des enzymes dimériques qui se produisent dans les bactéries, les archées, les plantes et les parasites protistes. Ces protéines censent le pyrophosphate en deux molécules d'orthophosphate, qui est couplée avec le proton et/ou le pompage d'ion de sodium à travers la membrane. Comme aucune protéine homologue ne se produit chez les animaux et les humains, les mPPases sont de bons candidats dans la conception de cibles médicamenteuses potentielles. Ici nous présentons un protocole détaillé pour examiner les inhibiteurs de mPPase utilisant la réaction bleue de molybdène dans un système de plaque de puits 96. Nous utilisons le mPPase de la bactérie thermophile Thermotoga maritima (TmPPase) comme enzyme modèle. Ce protocole est simple et peu coûteux, produisant un résultat cohérent et robuste. Il ne faut qu'environ une heure pour compléter le protocole d'etoile d'activité depuis le début de l'assiduité jusqu'à la mesure de l'absorption. Étant donné que la couleur bleue produite dans cet analyse est stable pendant une longue période de temps, l'analyse ultérieure peut être effectuée immédiatement après le lot précédent, et l'absorption peut être mesurée plus tard pour tous les lots à la fois. L'inconvénient de ce protocole est qu'il est fait manuellement et peut donc être épuisant ainsi que nécessitent de bonnes compétences de pipetage et de temps. De plus, la solution de citrate d'arsenit utilisée dans cet assay contient de l'arsénite de sodium, qui est toxique et doit être manipulée avec les précautions nécessaires.
Introduction
Environ 25 % des protéines cellulaires totales sont des protéines membranaires et environ 60 % d'entre elles sont des cibles médicamenteuses1,2. L'une des cibles médicamenteuses potentielles3, pyrophosphatases membranaires (mPPases), sont des enzymes dimériques qui pompent le proton et/ou l'ion de sodium à travers la membrane par hydrolyse de pyrophosphate en deux orthophosphates4. mPPases peut être trouvé dans divers organismes5 tels que les bactéries, archées, plantes, et les parasites protistes, à l'exception des humains et des animaux4. Chez les parasites protistes, par exemple Plasmodium falciparum, Toxoplasma gondii et Trypanosoma brucei, mPPases sont essentiels pour la virulence parasite6 et KO de cette expression dans les parasites conduire à l'échec dans le maintien du pH intracellulaire lors de l'exposition au pH de base externe7. En raison de leur importance et du manque de protéines homologues présentes chez les vertébrés, les mPPases peuvent être considérés comme des cibles médicamenteuses potentielles pour les maladies protistales3.
Le dépistage in vitro des inhibiteurs du mPPase dans ce travail est basé sur un système de modèle TmPPase. TmPPase est un pompage d'ion de sodium et de potassium ion dépendant mPPase de T. maritima et a son activité optimale à 71 c8. Les avantages de cette enzyme sont par exemple sa facilité de production et de purification, une bonne stabilité thermique et une activité spécifique élevée. TmPPase montre à la fois une forte similitude en plus de la conservation complète de la position ainsi que l'identité de tous les résidus catalytiques à la mPPases protiste3,9 et à la structure résolue de Vigna radiata10 mPPase. Les structures disponibles de TmPPase dans différentes conformations sont également utiles pour l'expérience de conception de médicaments basée sur la structure (comme le dépistage virtuel et la conception de novo).
Ici, nous rapportons un protocole détaillé pour le criblage des inhibiteurs de TmPPase dans un format de plaque de puits 96 (figure 1). Le protocole est basé sur la méthode colorimétrique de la réaction bleu molybdène, qui a d'abord été développé par Fiske et Subbarow11. Cette méthode implique la formation d'acide phosphomolybdique 12-phosphomolybd à partir d'orthophosphate et de molybdate dans des conditions acides, qui est ensuite réduite pour donner des espèces caractéristiques de phosphomolybdène de couleur bleue12.
Protocole
1. Préparation des protéines
REMARQUE: L'expression et la purification de TmPPase a été décrite ailleurs13.
- Préparer 10 ml de la solution tampon de réactivation contenant 20 mM 2-( N-morpholino)ethanesulfonic acid (MES) pH 6.5, 3.5% (v/v) glycerol, 2 mM dithiothreitol (DTT), et 0.05% dodecyl maltoside (DDM).
- Préparer 10 ml du mélange de réaction contenant 200 mM Tris-Cl pH 8,0, 8,0 mM MgCl2, 333 mM KCl, et 67 mM NaCl.
REMARQUE : Mg2est nécessaire pour chélater le pyrophosphate comme substrat de mPPase, K- est nécessaire pour augmenter l'activité enzymatique car TmPPase est un mPPase dépendant du potassium, et Naest nécessaire pour l'activité enzymatique pendant la translocation d'ion de sodium par TmPPase. - Préparer des liposomes de 30 mg/mL pour la réactivation des enzymes.
- Ajouter 10 ml de 20 mL de tris-HCl pH 8,0 avec 1 mM DTT à 0,3 g de L-phosphatidylcholine du soja à 10 ml de 20 mM Tris-HCl pH 8,0 avec 1 mM DTT.
- Mettre le liposome sur la glace, et sonicate avec 1 seconde d'intervalle d'impulsion pendant 1 minute, pause pendant 1 minute, et répéter jusqu'à ce que la solution devienne jaune transparent.
- Aliquot les liposomes, congeler dans l'azote liquide et stocker à -80 oC jusqu'à ce qu'ils soient utilisés.
- Réactiver l'enzyme.
- Mélanger 40 L de la solution liposomes avec 22,5 'L de 20% DDM.
- Chauffer le mélange à 55 oC pendant 15 min et laisser refroidir à température ambiante.
- Ajouter 36,5 L de la solution tampon de réactivation, mélanger et ajouter 1 l de protéines concentrées (13 mg/mL) pour obtenir une concentration totale de 0,13 mg/mL.
REMARQUE : Les protéines sont habituellement congelées dans des aliquots de 10 l après la purification et décongelées sur la glace avant utilisation.
- Prendre 20 ll de l'enzyme réactivée et ajouter à 1 480 l l du mélange de réaction, puis mélanger doucement.
REMARQUE : L'ajout de l'enzyme réactivée au mélange de réaction doit être effectué juste avant qu'il ne soit utilisé.
2. Préparation composée
- Dissoudre les composés dans le sulfoxyde de diméthyle (DMSO) pour faire des solutions de stock de 25 à 100 mm dans 50'200 'L, en fonction de la disponibilité des composés.
REMARQUE: Tous les composés utilisés ici (Figure 2A) ont été publiés précédemment9. Si la solubilité composée est faible, la concentration du stock peut être ajustée en conséquence. - Préparer trois concentrations différentes de chaque composé dans l'eau.
REMARQUE : Les concentrations finales dans le mélange de réaction seront 1, 5, et 50 micromolaires ou 1, 5, et 20 micromolar pour les composés solubles et avec parcimonie soluble, respectivement.- Diluer la solution de bouillon avec de l'eau à 1 ml dans des microtubes pour donner 2 M, 10 M et 100 M pour les composés solubles, ou alternativement 2 M, 10 M et 40 M pour les composés solubles avec parcimonie.
- Vortex la solution composée instantanément après dilution de la solution de stock pour un bon mélange.
- Vérifiez l'agrégation composée à l'aide d'un néphélomètre.
REMARQUE : Ceci a été étudié sous forme de triplitaux en trois concentrations (1 M, 5 M et 20 M) et normalisé au blanc dans une plaque de puits de 96.- Distribuer 75 l de mélange de réaction dans chaque puits à l'aide d'une pipette multicanal.
- Ajouter 75 lde de chaque composé (pour le blanc, utiliser 75 l'eau à la place) et mélanger par pipetting de haut en bas de 5 degrés.
- Mesurer chaque puits à 300 V à l'aide d'un néphélomètre microplaque.
3. Réactifs pour la préparation de l'essai
- Préparer la solution arsenit-citrate.
- Peser 5 g d'arsénite de sodium et 5 g de dihydrate de citrate trisodique.
CAUTION: L'arsénite de sodium est toxique, donc utiliser un équipement de protection approprié et manipuler avec un soin particulier. Par mesure de précaution, ne pas gérer avant que toutes les précautions de sécurité nécessaires ont été lus et compris. Manipuler uniquement dans un capot de fumée afin de ne pas inhaler la poussière / vapeurs du composé ou de sa solution. En cas d'inhalation, passer à l'air frais et obtenir des soins médicaux. Portez des lunettes de sécurité chimiques appropriées, des gants de protection et des vêtements pour éviter l'ingestion et le contact entre les yeux et la peau. S'il est avalé, appelez immédiatement un centre antipoison ou un médecin/médecin. Si elle obtient sur la peau ou dans les yeux, laver avec beaucoup d'eau et obtenir des soins médicaux. - Dissoudre dans 100 ml d'eau.
- Ajouter 5 ml d'acide acétique glaciaire, mélanger et ajouter de l'eau à 250 ml.
- Conserver à température ambiante à l'abri de la lumière.
REMARQUE : La solution est stable depuis plus d'un an.
- Peser 5 g d'arsénite de sodium et 5 g de dihydrate de citrate trisodique.
- Préparer la solution A et la solution B.
- Pour la solution A, ajouter 10 ml de glace froide de 0,5 M HCl à 0,3 g d'acide ascorbique. Dissoudre l'acide ascorbique par vortexing.
- Pour la solution B, ajouter 1 ml d'eau glacée à 70 mg d'hélimonium d'hélimolygène tétrahydrate et vortex pour se dissoudre.
REMARQUE : Conservez les deux solutions sur la glace jusqu'à l'utilisation. Pour la cohérence du résultat d'analyse, les deux solutions peuvent être stockées sur la glace pendant un maximum d'une semaine.
- Préparer la norme de phosphate (Pi) avec la concentration de 0 M, 62,5 M, 250 M et 500 M pour l'étalonnage.
- Ajouter 0 l, 25 l, 50 'L et 100 'L de 5 mM Na2HPO4 dihydrater à quatre microtubes contenant 370 'L du mélange de réaction.
- Garnir jusqu'à 1 ml d'eau.
4. Suivi d'activité pour une plaque de puits de 96
REMARQUE : Voir la figure 1 pour le flux de travail schématique de l'assay.
- Ajouter 1 ml de solution B à 10 ml de solution A, mélanger par vortex et stocker la solution sur la glace.
REMARQUE : Cette solution doit être transparente et jaune. Conserver la solution A et B sur la glace pendant au moins 30 minutes avant l'utilisation. Cependant, utilisez la solution dans les 3 h car elle ira mal après stockage à long terme. - Ajouter 40 'L de 0 'M, 62,5 'M, 250 'M et 500 'MP i standard aux bandes de tube en tripler à l'aide d'une pipette multicanal.
REMARQUE: Le mélange de réaction sans Pj'ai ajouté sera utilisé comme un blanc. - Ajouter 25 l de solution composée aux bandes de tube à l'aide d'une pipette multicanal.
REMARQUE : Chaque composé a trois concentrations différentes dans le triplicate qui est assez pour l'estimation initiale de la concentration inhibitrice moitié maximale (IC50). Pour une détermination IC50 plus précise, huit concentrations composées différentes peuvent être utilisées. Pour l'enzyme décomplexée, la solution composée est remplacée par une quantité égale d'eau. Comme contrôles positifs 2,5 M, 25 m, et 250 M de sel de sodium imidodiphosphate (IDP) ont été utilisés. - Ajouter 15 ll de mélange de solution mPPase aux bandes de tube (sauf aux tubes contenant la norme Pi) à l'aide d'une pipette multicanal.
- Scellez les bandes tubulaires à l'effile d'étanchéité adhésive. Couper la feuille d'étanchéité pour séparer chaque bande tubulaire.
- Pré-incuber les échantillons pendant 5 min à 71 oC. Placez les échantillons sur le bloc de chauffage avec un intervalle de 20 s entre chaque bande afin de minimiser la consommation de temps pendant les étapes suivantes.
- Pour chaque bande, ouvrir l'étanchéité adhésive. Ajouter 10 l de 2 mm de dibasic pyrophosphate de sodium à l'aide d'une pipette multicanal et mélanger en pipetting de haut en bas pour 5 degrés. Scellez à nouveau la bande tubulaire en utilisant le même scellement.
REMARQUE: Cette étape pourrait initialement être difficile à accomplir en 20 s; cependant, il deviendra plus facile après quelques essais. - Incuber à 71 oC pendant 5 min.
- Placer les échantillons sur l'appareil de refroidissement avec un intervalle de 20 s entre chaque bande. Laisser refroidir pendant 10 min, mais centrifuger chaque bande peu après 5 min de refroidissement, pour décander les gouttes d'eau sous la feuille d'étanchéité, puis le remettre à l'appareil de refroidissement et enlever l'étanchéité.
REMARQUE : L'appareil de refroidissement peut simplement être fabriqué en plaçant une plaque PCR de 96 puits sur un plat Petri en polystyrène (taille 150 mm et 15 mm) rempli d'eau et congelé pendant au moins 1 h. L'appareil doit être retiré du congélateur environ 5 minutes avant le début de l'assidu. Ne pas sortir l'appareil de refroidissement juste avant le refroidissement de l'échantillon car il va geler le mélange de réaction et entraver le développement des couleurs. - Après 10 min de refroidissement, ajouter 60 l de la solution A et B, mélanger par pipetting de haut en bas pendant 5 degrés et garder les bandes de tube sur l'appareil de refroidissement pendant 10 min.
- Ajouter 90 oL de la solution de citrate d'arsénite et conserver à température ambiante pendant au moins 30 min pour produire une couleur bleue stable.
CAUTION: En raison de sa toxicité, toutes les solutions contenant de l'arsénite de sodium doivent être manipulées avec une attention supplémentaire en tout temps. Ainsi, l'ajout de la solution de citrate d'arsénite devrait être fait dans une hotte de fumée. - Distribuer 180 l de chaque mélange de réaction dans une microplaque claire de 96 puits de polystyrène.
- Mesurer l'absorption de chaque puits à 860 nm à l'aide d'un spectrophotomètre microplaque.
5. Analyse des résultats
- Moyenne des tripliques de chaque échantillon et des normes Pi. Ensuite, soustrayez avec le blanc pour éliminer le signal d'arrière-plan.
- Faire une courbe d'étalonnage en traçant les valeurs d'absorption (A860) par rapport à la quantité de norme Pi (nmol) et effectuer une régression linéaire pour obtenir la fonction trendline en utilisant la formule suivante :
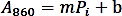
- Calculez la quantité de phosphate (nmol) libérée de la réaction enzymatique en fonction de la formule de régression linéaire ci-dessus.
- Calculez l'activité spécifique à l'aide de la formule suivante :
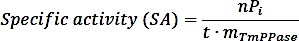
où nPi est la quantité de phosphate libérée par la réaction (nmol), t est le temps de réaction (min), et mTmPPase est la quantité de TmPPase pur utilisé dans l'assay (mg). - Calculez l'activité en pourcentage pour chaque concentration d'inhibiteurs à l'aide de la formule suivante :
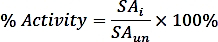
où SAiis l'activité spécifique d'un échantillon avec inhibiteur et SAun est l'activité spécifique de l'échantillon décomplexé. - Calculez le logIC50 (estimation) et ic50 (estimation) avec un ajustement de régression non linéaire de la courbe dose-réponse à quatre paramètres en utilisant la formule suivante :
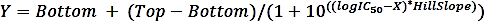
où X est log de concentration (M), Y est activité (%), Top et Bottom sont des plateaux dans la même unité que Y (100% et 0%, respectivement), logIC50 a les mêmes unités de notation que X, et HillSlope - facteur de pente ou pente de colline, qui est unitaire.
REMARQUE: Logiciel (Tableau des Matériaux) est utilisé pour le montage. Utilisez la concentration de 0,01 M (au lieu de 0,00 M) pour l'échantillon sans inhibiteur car le logarithme de zéro n'est pas défini.
Résultats
Dans ce protocole, huit composés (1 à 8) ont été testés (figure 2A) avec IDP, un inhibiteur commun des pyrophosphatases, comme un contrôle positif. Chaque composé a été testé à trois concentrations différentes (1 M, 5 M et 20 M) en triple. Le flux de travail de la présélection est représenté à la figure 1, à partir de la préparation de l'échantillon et du réactif jusqu'à la mesure de l'absorption à 860 nm.
À la fin de ce protocole, après l'ajout de la solution A et B et du citrate d'arsenit, les solutions développent une couleur bleue stable avec l'absorption maximale à 709 nm et 860 nm14 en raison de la formation complexe d'ions de phosphate avec du molybdate qui peut être observée et montre l'occurrence de la réaction enzymatique. Pour cette expérience, nous utilisons l'absorption à 860 nm pour la mesure de la quantité De Pi libérée car elle a une meilleure limite de détection et de sensibilité par rapport à l'absorption à 709 nm15. La couleur bleue est entièrement développée en 30 min d'incubation à température ambiante et stable pendant au moins 5 h14. L'analyse a la sensibilité jusqu'à la concentration de Pi de 10 M et l'absorption est linéaire sur une plage de concentration de 10 à 800 M14. Dans le résultat représentatif ici, les puits E1-E3(figure 2C) contiennent le mélange de réaction sans inhibiteur et la solution bleue peut être observée à la fin de l'analyse. Cela peut également être observé à de faibles concentrations composées où l'inhibition complète n'a pas été atteinte, comme dans les puits F1-F3 pour les iDP et les puits A4-A6 pour le composé 1 (ATC, un inhibiteur non compétitif récemment connu de TmPPase9) à la concentration de 2,5 M et 1 M, respectivement. La concentration plus élevée de PDI et de composé 1, la couleur moins à aucune couleur bleue peut être observée (G1-G3 et H1-H3 pour IDP et B4-B6 et C4-C6 pour le composé 1) indiquant l'inhibition de l'activité enzymatique. Les trois concentrations de composés non inhibants (2, 3, et 8) ont montré la même intensité de couleur bleue que les puits E1-E3 sans aucun inhibiteur (Figure 2C).
Après la mesure de l'absorption à 860 nm, les données peuvent être traitées et analysées (voir la section 5 du protocole). La figure 2D montre la parcelle d'étalonnage de la norme Pi avec son raccord linéaire (y à 0,0576x et 0,0019; r2 à 0,999). La figure 3 montre l'intrigue de l'activité enzymatique (%) contre la concentration de chaque composé testé. Pour les composés ayant une activité d'inhibition, un raccord de courbe non linéaire est également indiqué. Les PDI, utilisés comme un contrôle positif, montrent clairement une diminution de l'activité à une concentration plus élevée. L'IC50 (estimation) calculée sur la base de trois concentrations différentes est de 88,2 M (tableau 1), ce qui est similaire à la mesure précédente (80,0 M) avec huit points de concentration14. Les composés 1, 4, 5, 6, et 7 ont montré une tendance similaire à celle des PDI puisque la concentration a augmenté avec l'IC50 (estimation) d'environ 1,3 M, 7,4 M, 19,0 M, 37,4 M et 156,1 M, respectivement (tableau 1). Pour les composés 2, 3, et 8 aucune réduction de l'activité ou de l'inhibition ne peut être observée aux concentrations d'analyse. Un analyse supplémentaire avec huit points de concentration peut être fait pour générer précis IC50. La figure 4 montre la courbe d'inhibition pour les composés 1, 5, 6, 7 et 8 avec un IC50 de 1,7 M, 21,4 M, 58,8 M, 239,0 M et 500 M, respectivement9.
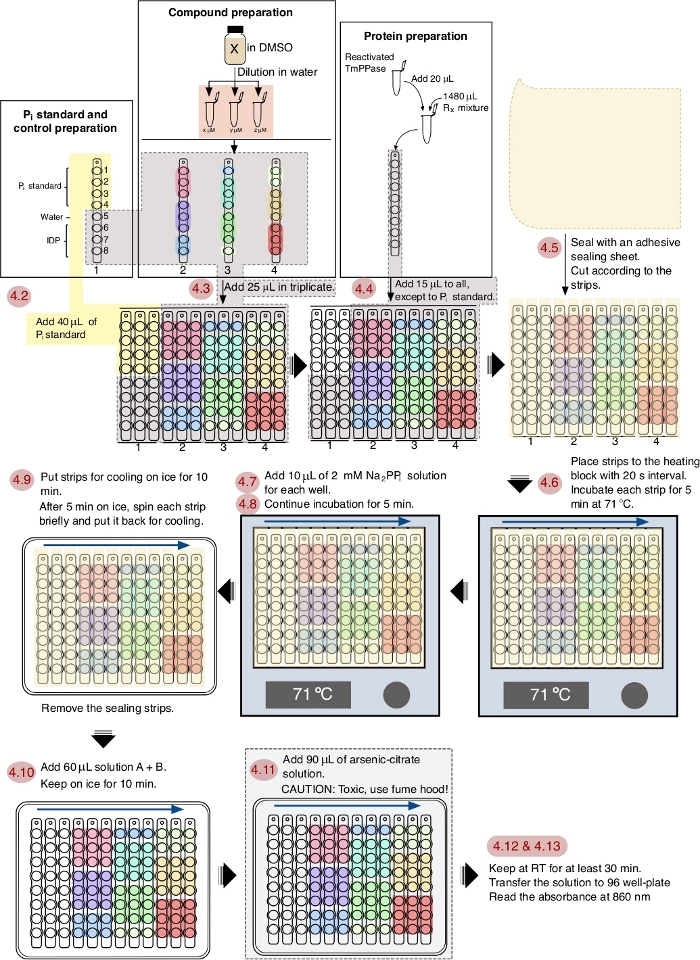
Figure 1 : Un flux de travail schématique d'inséditde de TmPPase dans un format de plaque de puits 96. La numérotrage rouge affiche les étapes de l'analyse selon le protocole et les flèches bleues montrent l'ordre d'intervalle. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
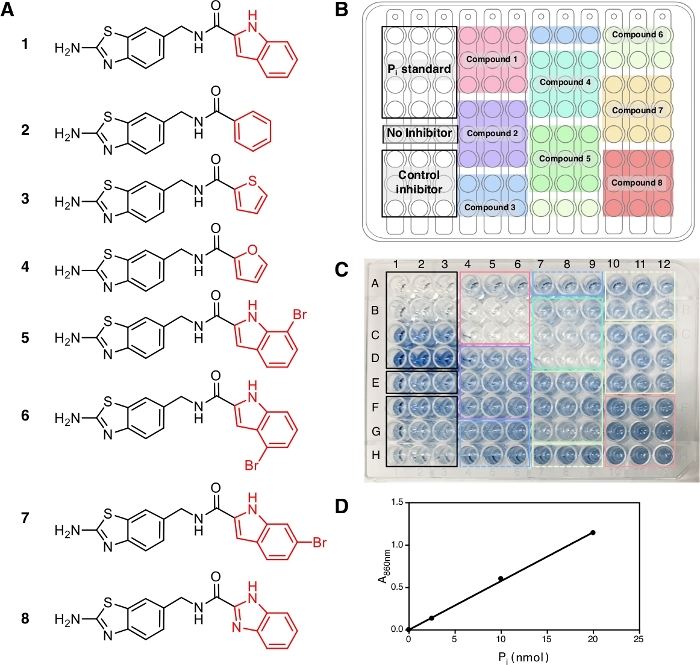
Figure 2 : Échantillons, leur arrangement et développement de couleur dans une plaque de puits de 96. (A) Les structures des composés 1à8 utilisés pour l'analyse. L'activité d'inhibition de ces composés a été rapportée dans Vidilaseris et autres9. (B) Arrangement d'échantillon. (C) Développement de couleur, 30 min après l'ajout de la solution d'arsénite-citrate. Les concentrations d'inhibiteurs de contrôle (IDP) et d'échantillons utilisés, disposés de haut en bas, sont de 2,5 M, 25 M, et 250 M de concentration et 1 M, 5 m, et 20 M de concentration, respectivement. L'intensité de la couleur bleue correspond à la quantité de Pi libéré en raison de la réaction enzymatique et le manque de couleur ne correspond à aucune réaction enzymatique. (D) Courbe de calibration pour la norme Pi (nmol) contre A860 avec raccord linéaire (y à 0,0576x et 0,0019; r2 à 0,999). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
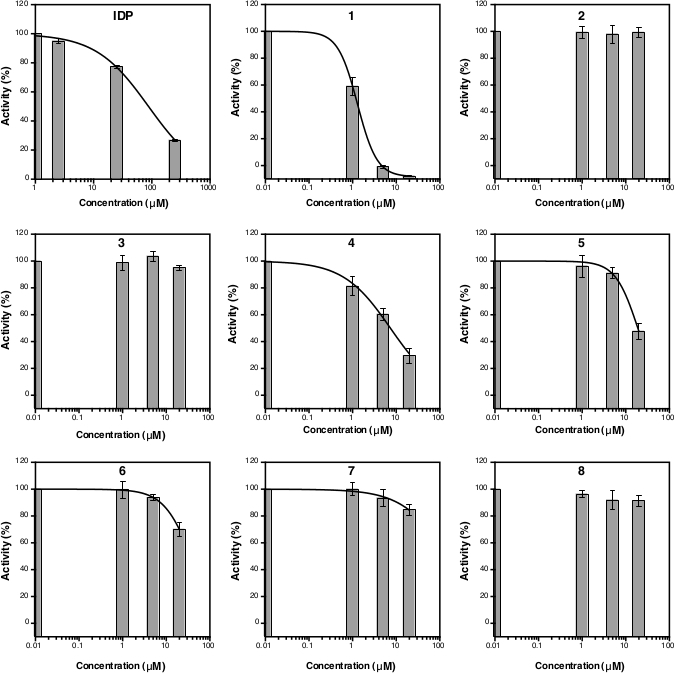
Figure 3 : Courbe de l'activité du pourcentage de TmPPase pour trois concentrations d'inhibiteurs différentes. Les courbes de régression non linéaires pour calculer l'IC50 (estimation) sont indiquées pour les PDI ainsi que pour les composés 1, 4, 5, 6 et 7, mais pas pour les composés 2, 3, et 8 car ils n'inhibaient pas l'activité tmPPase aux concentrations d'analyse. Le logIC50 et ic50 (estimation) de chaque composé est indiqué dans le tableau 1. Toutes les données sont affichées comme moyenne - SD avec trois répliques. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 4 : Courbe d'inhibition à partir de huit points de concentration des composés 1, 5, 6, 7 et 8. Ce chiffre est tiré de Vidilaseris et coll.9 avec une légère modification. Toutes les données sont affichées comme moyenne - SD avec trois répliques. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
| Échantillon | LogIC50 Annonces | IC50 (estimation) (M) |
| Les Personnes qui ont été pDI | 1,95 à 0,0142 | 87,9 à 2,46 |
| 1 | 0,112 à 0,0274 | 1,29 à 0,0816 |
| 2 | − | pas d'inhibition |
| 3 | − | pas d'inhibition |
| 4 | 0,870 à 0,0447 | 7,39 à 0,760 |
| 5 | 1,28 à 0,0296 | 19,0 à 1,29 |
| 6 | 1,57 à 0,0846 | 37,4 à 7,29 |
| 7 | 2,39 à 0,366 | 156 à 131 |
| 8 | − | pas d'inhibition |
Tableau 1 : LogIC50 et IC50 (estimation) des personnes pdiles et des composés 1 à 8 d'après les données de la figure 3.
Discussion
Ici, nous rapportons un protocole détaillé pour le criblage simple des inhibiteurs pour la pyrophosphatase membranaire-liée de T. maritima dans un format 96 de plaque de puits basé sur Vidilaseris et autres14. Ce protocole est peu coûteux et basé sur l'acide 12-phosphomolybd, qui est formé à partir d'orthophosphate et de molybdate dans des conditions acides et réduit à des espèces de phosphomolybdène avec une couleur bleue distincte12. Cette méthode est préférée à d'autres protocoles, tels que l'exemple plus sensible de vert malachite16,parce que cette méthode ne montre pas d'interférence dans la présence d'une concentration élevée de phospholipides qui est nécessaire pour la réactivation de TmPPase14.
Le flux de travail du protocole de présélection est représenté à la figure 1 et ce processus peut être entièrement réalisé en 1 h. Ce protocole est optimisé pour tmPPase avec une température de travail optimale à 71 oC et un temps de réaction de 5 min. Comme l'eau s'évapore à cette température du mélange de réaction, une feuille d'étanchéité adhésive (tranchée pour s'adapter et recouvrir les bandes) est appliquée pour empêcher l'évaporation14 et l'eau évaporée est simplement récupérée avec centrifugation. Le temps d'incubation de 5 min est choisi car il est encore dans la gamme linéaire du phosphate libéré enzymatiquement et suffisant pour un dépistage fiable14. Dans ce protocole, les compétences en matière de chronométrage et de tuyauterie sont des facteurs importants pour obtenir un résultat fiable et fiable. L'ajout de réactifs pendant l'analyse avec un intervalle de 20 s entre les bandes est une option de synchronisation optimisée pour faciliter l'exécution des étapes suivantes.
Pour les différents mPPases, la température optimale et le temps d'incubation doivent être déterminés séparément avant d'être utilisés dans l'inversion d'inhibition. Le protocole de réactivation enzymatique ci-dessus est optimisé pour TmPPase et d'autres mPPases pourraient avoir besoin d'un protocole de réactivation différent. Par exemple, DDM ne devrait pas être ajouté pour la réactivation de mPPase de Pyrobaculum aerophilum car il diminuera son activité enzymatique17. Comme l'enzyme deviendra moins active si elle est préparée bien à l'avance, l'ajout d'enzymerée devrait être ajouté au mélange de réaction peu de temps avant le début de l'essai. Après l'ajout de la solution d'arsénite-citrate le produit de réaction est stable pendant au moins 5 h14. Par conséquent, le prochain lot de l'analyse peut être effectué immédiatement, et la mesure d'absorption peut être faite plus tard à tous les lots à la fois.
Déclarations de divulgation
Les auteurs n'ont rien à révéler.
Remerciements
Ce travail a été soutenu par les subventions de la Fondation Jane et Aatos Erkko et du BBSRC (BB/M021610) à Adrian Goldman, l'Académie de Finlande (no 308105) à Keni Vidilaseris, (no 310297) à Henri Xhaard, et (no 265481) à Jari Yli-Kauhaluoma, et les fonds de recherche de l'Université d'Helsinki à Gustav Boije af Genns. Les auteurs remercient Bernadette Gehl pour son aide technique pendant le projet.
matériels
| Name | Company | Catalog Number | Comments |
| Adhesive sealing sheet | Thermo Scientific | AB0558 | |
| Ammonium heptamolybdate tetrahydrate | Merck | F1412481 636 | |
| Ascorbic acid | Sigma-Aldrich | 95212-250G | |
| BioLite 96Well Multidish | Thermo Scientific | 130188 | |
| Dimethyl sulfoxide (DMSO) | Merck | 1167431000 | |
| 8-well PCR Tube Strips 0.2 ml without caps (120) | Nippon genetics | FG-028 | |
| Dodecyl maltoside (DDM) | Melford | B2010-100G | |
| Ethanol | Merck | 1009901001 | |
| Glacial acetic acid | Merck | 1000631011 | |
| Hydrochloric acid | Sigma-Aldrich | 258148-500ML | |
| Imidodiphosphate sodium salt | Sigma-Aldrich | I0631-1G | |
| L-α-Phosphatidyl choline from soybean lecithin | Sigma | 429415-100GM | |
| Magnesium chloride | Sigma-Aldrich | 8147330500 | |
| Multiplate 96-Well PCR Plates | Bio-Rad | MLL9651 | |
| MultiSkan Go | Thermo Scientific | 10680879 | |
| Nepheloskan Ascent (Type 750) | Labsystems | ||
| Polystyrene Petri dish (size 150 mm x 15 mm) | Sigma-Aldrich | P5981-100EA | |
| Potassium chloride | Merck | 104936 | |
| Prism 6 software | GraphPad | ||
| QBT2 Heating block | Grant Instruments | ||
| Sodium meta-arsenite | Fisher Chemical | 12897692 | |
| Sodium phosphate dibasic (Pi) | Sigma | S0876-1KG | |
| Sodium pyrophosphate dibasic | Fluka | 71501-100G | |
| Trisodium citrate dihydrate | Fluka | 71404-1KG |
Références
- Terstappen, G. C., Reggiani, A. In silico research in drug discovery. Trends in Pharmacological Sciences. 22 (1), 23-26 (2001).
- Rask-Andersen, M., Almen, M. S., Schioth, H. B. Trends in the exploitation of novel drug targets. Nature Reviews Drug Discovery. 10 (8), 579-590 (2011).
- Shah, N. R., Vidilaseris, K., Xhaard, H., Goldman, A. Integral membrane pyrophosphatases: a novel drug target for human pathogens. AIMS Biophysics. 3 (1), 171-194 (2016).
- Baykov, A. A., Malinen, A. M., Luoto, H. H., Lahti, R. Pyrophosphate-Fueled Na+ and H+ Transport in Prokaryotes. Microbiology and Molecular Biology Reviews. 77 (2), 267-276 (2013).
- Serrano, A., Perez-Castineira, J. R., Baltscheffsky, M., Baltscheffsky, H. H+-PPases: yesterday, today and tomorrow. IUBMB Life. 59 (2), 76-83 (2007).
- Liu, J., et al. A vacuolar-H+-pyrophosphatase (TgVP1) is required for microneme secretion, host cell invasion, and extracellular survival of Toxoplasma gondii. Molecular Microbiology. 93 (4), 698-712 (2014).
- Lemercier, G., et al. A pyrophosphatase regulating polyphosphate metabolism in acidocalcisomes is essential for Trypanosoma brucei virulence in mice. Journal of Biological Chemistry. 279 (5), 3420-3425 (2004).
- Belogurov, G. A., et al. Membrane-bound pyrophosphatase of Thermotoga maritima requires sodium for activity. Biochemistry. 44 (6), 2088-2096 (2005).
- Vidilaseris, K., et al. Asymmetry in catalysis by Thermotoga maritima membrane-bound pyrophosphatase demonstrated by a nonphosphorus allosteric inhibitor. Science Advances. 5 (5), (2019).
- Lin, S. M., et al. Crystal structure of a membrane-embedded H+-translocating pyrophosphatase. Nature. 484 (7394), 399-403 (2012).
- Fiske, C. H., Subbarow, Y. The colorimetric determination of phosphorus. Journal of Biological Chemistry. 66 (2), 375-400 (1925).
- Nagul, E. A., McKelvie, I. D., Worsfold, P., Kolev, S. D. The molybdenum blue reaction for the determination of orthophosphate revisited: Opening the black box. Analytica Chimica Acta. 890, 60-82 (2015).
- Kellosalo, J., Kajander, T., Palmgren, M. G., Lopez-Marques, R. L., Goldman, A. Heterologous expression and purification of membrane-bound pyrophosphatases. Protein Expression and Purification. 79 (1), 25-34 (2011).
- Vidilaseris, K., Kellosalo, J., Goldman, A. A high-throughput method for orthophosphate determination of thermostable membrane-bound pyrophosphatase activity. Analytical Methods. 10 (6), 646-651 (2018).
- He, Z. Q., Honeycutt, C. W. A modified molybdenum blue method for orthophosphate determination suitable for investigating enzymatic hydrolysis of organic phosphates. Communications in Soil Science and Plant Analysis. 36 (9-10), 1373-1383 (2005).
- Martin, B., Pallen, C. J., Wang, J. H., Graves, D. J. Use of fluorinated tyrosine phosphates to probe the substrate specificity of the low molecular weight phosphatase activity of calcineurin. Journal of Biological Chemistry. 260 (28), 14932-14937 (1985).
- Strauss, J., Wilkinson, C., Vidilaseris, K., Harborne, S. P. D., Goldman, A. A simple strategy to determine the dependence of membrane-bound pyrophosphatases on K+ as a cofactor. Methods in Enzymology. 607, 131-156 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon