Method Article
Screening per inibitori di Pirofosofasi rilegati a Thermotoga
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui presentiamo un metodo di screening per gli inibitori di pyrophosphatase legati alla membrana (da Thermotoga maritima) basato sulla reazione blu molybdenum in un formato 96 pozzo piastra.
Abstract
I piifofosti legati alla membrana (mPPases) sono enzimi dimerici che si verificano nei batteri, negli arcai, nelle piante e nei parassiti protisti. Queste proteine si sciolgono il pirofosfato in due molecole di ortopofosde, che è accoppiato con protone e/o ione di sodio che pompa attraverso la membrana. Poiché non si verificano proteine omologhe negli animali e negli esseri umani, gli mPPasi e sono buoni candidati nella progettazione di potenziali bersagli farmacologici. Qui presentiamo un protocollo dettagliato per lo screening per gli inibitori mPPase utilizzando la reazione blu molybdenum in un sistema di piastra 96. Usiamo mPPase dal batterio termofilo Thermotoga maritima (TmPPase) come enzima modello. Questo protocollo è semplice ed economico, producendo un risultato coerente e robusto. Ci vuole solo circa un'ora per completare il protocollo di analisi dell'attività dall'inizio del saggio fino alla misurazione dell'assorbimento. Poiché il colore blu prodotto in questo saggio è stabile per un lungo periodo di tempo, il saggio successivo può essere eseguito immediatamente dopo il lotto precedente e l'assorbimento può essere misurato in un secondo momento per tutti i lotti contemporaneamente. Lo svantaggio di questo protocollo è che è fatto manualmente e quindi può essere estenuante così come richiedono buone abilità di pipettaggio e di mantenimento del tempo. Inoltre, la soluzione arsenite-citrate utilizzata in questo saggio contiene arsenite di sodio, che è tossico e deve essere maneggiato con le precauzioni necessarie.
Introduzione
Circa il 25% del totale delle proteine cellulari sono proteine di membrana e circa il 60% di esse sono bersagli farmacologici1,2. Uno dei potenziali bersagli farmacologici3, pirofofosasi legati alla membrana (mPPases), sono enzimi dimero che pompano protone e/o ione di sodio attraverso la membrana da idrolisi di piofosfato in due ortoposfati4. mPPases può essere trovato in vari organismi5 come batteri, archea, piante e parassiti protisti, con l'eccezione di esseri umani e animali4. Nei parassiti protisti, per esempio Plasmodium falciparum, Toxoplasma gondii e Trypanosoma brucei, mPPases sono essenziali per la virulenza del parassita6 e knockout di questa espressione nei parassiti portano al fallimento nel mantenimento del pH intracellulare dopo l'esposizione al pH di base esterno7. A causa della loro importanza e della mancanza di proteine omologhe presenti nei vertebrati, mPPases può essere considerato come potenziali bersagli farmacologici per le malattie protische3.
Lo screening in vitro degli inibitori di mPPase in questo lavoro si basa su un sistema modello TmPPase. TmPPase è un mPPasi dipendente dagli ioni di sodio e ioscio da T. maritima e ha la sua attività ottimale a 71 gradi centigradi8. I benefici di questo enzima sono ad esempio la sua facilità di produzione e purificazione, una buona stabilità termica e un'elevata attività specifica. TmPPase mostra sia un'elevata somiglianza oltre alla completa conservazione della posizione, sia l'identità di tutti i residui catalitici al protista mPPases3,9 e la struttura risolta di Vigna radiata10 mPPase. Le strutture disponibili di TmPPase in diverse conformazioni sono utili anche per l'esperimento di progettazione di farmaci basati sulla struttura (come screening virtuale e progettazione de novo).
Qui segnaliamo un protocollo dettagliato per lo screening degli inibitori di TmPPase in un formato 96 ben piatto (Figura 1). Il protocollo si basa sul metodo colorimetrico della reazione blu molybdenum, che è stato sviluppato per la prima volta da Fiske e Subbarow11. Questo metodo comporta la formazione di 12-ciclomolistici da ortopofosfato e molybdate in condizioni acide, che viene poi ridotto per dare caratteristiche specie di fosfomilybdenum di colore blu12.
Protocollo
1. Preparazione delle proteine
NOTA: l'espressione e la purificazione di TmPPase è stata descritta altrove13.
- Preparare 10 mL della soluzione di buffer di riattivazione contenente 20 mM 2-(N-morpholino)ethanesulfonico acid (MES) pH 6.5, 3,5% (v/v) glicerol, 2 mM dithiothreitol (DTT) e 0,05% dodecyl maltoside (DDM).
- Preparare 10 mL della miscela di reazione contenente 200 mM Tris-Cl pH 8.0, 8,0 mM MgCl2, 333 mM KCl e 67 mM NaCl.
NOTA: È necessario che il mg2 per masticare il pirofafato, poiché il substrato di mPPase, Kè necessario per aumentare l'attività degli enzimi poiché TmPPase è un mPPase dipendente dal potassio, e Naè necessaria per l'attività enzimatica durante la traslocazione di ioni di sodio da parte di TmPPase. - Preparare 30 liposomi mg/mL per la riattivazione degli enzimi.
- Aggiungere 10 mL di 20 mM Tris-HCl pH 8.0 con 1 mM DTT a 0,3 g di L-z-phosphatidylcolina da soia a 10 mL di 20 mM Tris-HCl pH 8.0 con 1 mM-DTT.
- Mettere il liposoma sul ghiaccio e sonicare con intervallo di impulso di 1 secondo per 1 minuto, mettere in pausa per 1 minuto e ripetere fino a quando la soluzione diventa giallo trasparente.
- Aliquota i liposomi, congelare in azoto liquido e conservare a -80 gradi centigradi fino a quando non viene utilizzato.
- Riattivare l'enzima.
- Mescolare 40 l della soluzione liposomi con 22,5 litri di 20% DDM.
- Riscaldare il composto a 55 gradi centigradi per 15 minuti e lasciarlo raffreddare a temperatura ambiente.
- Aggiungere 36,5 l della soluzione tampone di riattivazione, mescolare e aggiungere 1 l di proteine concentrate (13 mg/mL) per ottenere una concentrazione totale di 0,13 mg/mL.
NOTA: Le proteine sono solitamente congelate in 10 gradiNI dopo la purificazione e scongelate sul ghiaccio prima dell'uso.
- Prendere 20 l dell'enzima riattivato e aggiungere a 1.480 l della miscela di reazione, quindi mescolare delicatamente.
NOTA: L'aggiunta dell'enzima riattivato alla miscela di reazione deve essere eseguita appena prima che venga utilizzata.
2. Preparazione composta
- Sciogliere i composti in zolfo dimetilo (DMSO) per fare soluzioni di magazzino di 25-100 mM in 50-200 l, in base alla disponibilità dei composti.
NOTA: tutti i composti utilizzati qui (Figura 2A) sono stati pubblicati in precedenza9. Se la solubilità composto è bassa, la concentrazione di stock può essere regolata di conseguenza. - Preparare tre diverse concentrazioni di ogni composto in acqua.
NOTA: Le concentrazioni finali nella miscela di reazione saranno rispettivamente 1, 5 e 50 micromolar o 1, 5 e 20 micromolar per composti solubili e sobri, solubili.- Diluislo la soluzione di riserva con acqua a 1 mL in microtubi per dare 2 M, 10 M e 100 M per composti solubili, o in alternativa 2 M, 10 M e 40 M per composti spietatamente solubili.
- Vorticare la soluzione composta istantaneamente dopo la diluizione della soluzione di riserva per una corretta miscelazione.
- Verificare l'aggregazione composta utilizzando un nephelometro.
NOTA: Questo è stato studiato come triplicati in tre concentrazioni (1 M, 5 M e 20 M) e normalizzato nello spazio vuoto in un pozzo 96.- Distribuisci 75 -L della miscela di reazione in ogni pozzo utilizzando una pipetta multicanale.
- Aggiungere 75 l di ogni composto (per lo spazio vuoto, utilizzare invece 75 l'acqua) e mescolare convogliando su e giù per 5o.
- Misurare ogni pozzo a 300 V utilizzando un nephelometro a microplacca.
3. Reagenti per la preparazione del saggio
- Preparare la soluzione arsenite-citrate.
- Pesare 5 g di arsenite di sodio e 5 g di diidrato trisofato.
AVVISO: L'arsenite di sodio è tossica, quindi utilizzare attrezzature protettive adeguate e maneggiare con particolare cura. Per precauzione, non maneggiare prima che tutte le precauzioni di sicurezza necessarie siano state lette e comprese. Maneggiare solo in un cofano di fumi per non inalare polvere/vapori del composto o delle sue soluzioni. Se inalato, passare all'aria aperta e ottenere cure mediche. Indossare occhiali di sicurezza chimica, guanti protettivi e indumenti appropriati per evitare l'ingestione e il contatto con gli occhi e la pelle. Se ingerito, chiamare immediatamente un centro antiveleni o un medico/medico. Se arriva sulla pelle o negli occhi, lavare con molta acqua e ottenere cure mediche. - Sciogliere in 100 mL di acqua.
- Aggiungere 5 mL di acido acetico glaciale, mescolare e aggiungere acqua a 250 mL.
- Conservare a temperatura ambiente protetto dalla luce.
NOTA: La soluzione è stabile per più di un anno.
- Pesare 5 g di arsenite di sodio e 5 g di diidrato trisofato.
- Preparare la soluzione A e la soluzione B.
- Per la soluzione A, aggiungere 10 mL di ghiaccio freddo 0,5 M HCl a 0,3 g di acido ascorbico. Sciogliere l'acido ascorbico vortice.
- Per la soluzione B, aggiungere 1 mL di acqua fredda di ghiaccio a 70 mg di tetraidratte e vortice di ammonio eptamolybdate per sciogliere.
NOTA: Conservare entrambe le soluzioni su ghiaccio fino all'uso. Per la consistenza del risultato del saggio, entrambe le soluzioni possono essere conservate sul ghiaccio per un massimo di una settimana.
- Preparare lo standard fosfato (Pi)con la concentrazione di 0 M, 62,5 M, 250 M e 500 M per la calibrazione.
- Aggiungete 0 , 25 l, 50 l, e 100 ll di 5 mM Na2HPO4 disiscaricano a quattro microtubi contenenti 370 l della miscela di reazione.
- Top fino a 1 mL con acqua.
4. Anale di attività per una piastra da 96 pozzetto
NOTA: vedere la figura 1 per il flusso di lavoro schematico del saggio.
- Aggiungere 1 mL di soluzione B a 10 mL della soluzione A, mescolare vortice e conservare la soluzione sul ghiaccio.
NOTA: questa soluzione deve essere trasparente e gialla. Mantenere la soluzione A e B sul ghiaccio per almeno 30 minuti prima dell'uso. Tuttavia, utilizzare la soluzione entro 3 h in quanto andrà male dopo l'archiviazione a lungo termine. - Aggiungete alle strisce dei tubi in triplice le strisce in triplice copia utilizzando una pipetta multicanale.
NOTA: La miscela di reazione senza Pi aggiunto verrà utilizzata come vuoto. - Aggiungere 25 - L di soluzione composta alle strisce del tubo utilizzando una pipetta multicanale.
NOTA: Ogni composto ha tre diverse concentrazioni in triplice copia, il che è sufficiente per la stima iniziale della concentrazione inibitoria massima (IC50). Per una determinazione più accurata di IC50, è possibile utilizzare otto diverse concentrazioni composte. Per l'enzima disinibito la soluzione composta viene sostituita con la stessa quantità di acqua. Come controlli positivi 2,5 M, 25 M, e 250 M di imidodiphosfato (IDP) sale sono stati utilizzati. - Aggiungere 15 l-L di miscela di soluzione mPPase alle strisce del tubo (ad eccezione dei tubi contenenti Pi standard) utilizzando una pipetta multicanale.
- Sigillare le strisce del tubo con un foglio di sigillazione adesiva. Tagliare il foglio di saltellaggio per separare ogni striscia del tubo.
- Pre-incubare i campioni per 5 min a 71 gradi centigradi. Posizionare i campioni sul blocco di riscaldamento con intervallo di 20 s tra ogni striscia al fine di ridurre al minimo il consumo di tempo durante le fasi successive.
- Per ogni striscia, aprire la guarizione adesiva. Aggiungete 10 l of 2 mM di pirofosfato di sodio utilizzando una pipetta multicanale e mescolate con pipistrato su e giù per 5o. Sigillare nuovamente la striscia del tubo utilizzando la stessa guaritura.
NOTA: questo passaggio potrebbe essere inizialmente difficile da eseguire in 20 s; tuttavia, diventerà più facile dopo alcuni saggi. - Incubare a 71 gradi centigradi per 5 min.
- Posizionare i campioni sull'apparato di raffreddamento con un intervallo di 20 s tra ogni striscia. Lasciarli raffreddare per 10 min ma centrificare ogni striscia brevemente dopo 5 min di raffreddamento, per decantare le gocce d'acqua sotto il foglio di tenuta, quindi rimetterlo all'apparato di raffreddamento e rimuovere la tenuta.
NOTA: L'apparato di raffreddamento può essere semplicemente realizzato posizionando una piastra PCR da 96 pozzetti su una piastra Petri in polistirolo (dimensioni 150 mm x 15 mm) riempita con acqua e congelata per almeno 1 ora. L'apparecchio deve essere estratto dal congelatore circa 5 minuti prima dell'inizio del saggio. Non estrarre l'apparato di raffreddamento subito prima del raffreddamento del campione in quanto congela la miscela di reazione e ostacola lo sviluppo del colore. - Dopo 10 min di raffreddamento, aggiungere 60 -L di soluzione A e B, mescolare convogliando su e giù per 5 o più e mantenere le strisce del tubo sull'apparato di raffreddamento per 10 min.
- Aggiungere 90 l l della soluzione arsenite-citrate e mantenere a temperatura ambiente per almeno 30 min per produrre un colore blu stabile.
AVVISO: A causa della sua tossicità tutte le soluzioni contenenti arsenite al sodio devono essere gestite con particolare attenzione in ogni momento. Pertanto, l'aggiunta di soluzione arsenite-citrato dovrebbe essere fatta in una cappa di fumi. - Distribuisci 180 l di ogni miscela di reazione in una chiara microplacca di polistirolo 96 bene.
- Misurare l'assorbimento di ogni pozzo a 860 nm utilizzando uno spettrofotometro a microplacca.
5. Analisi dei risultati
- Media i triplicati di ogni campione e gli standard Pi. Quindi sottrarre con lo spazio vuoto per eliminare il segnale di sfondo.
- Effettuare una curva di calibrazione tracciando i valori di assorbimento (A860) rispetto alla quantità di standard Pi (nmol) ed eseguire una regressione lineare per ottenere la funzione di linea di tendenza utilizzando la seguente formula:
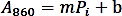
- Calcolare la quantità di fosfato (nmol) rilasciata dalla reazione ezimatica in base alla formula di regressione lineare di cui sopra.
- Calcolare l'attività specifica utilizzando la formula seguente:
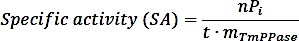
dove nPi è la quantità di fosfato rilasciato dalla reazione (nmol), t è il tempo di reazione (min), e mTmPPase è la quantità del MoPPase puro utilizzato nel saggio (mg). - Calcolare l'attività percentuale per ogni concentrazione di inibitore utilizzando la seguente formula:
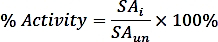
dove SAiis l'attività specifica di un campione con inibitore e SAun è l'attività specifica del campione disinibito. - Calcolare il logIC50 (stima) e IC50 (stima) con una regressione non lineare adatta dalla curva dose-risposta a quattro parametri utilizzando la seguente formula:
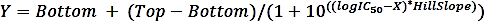
dove X è il registro di concentrazione (M), Y è l'attività (%), Top e Bottom sono altipiani nella stessa unità di Y (rispettivamente Y (100% e 0%)), logIC50 ha le stesse unità di log di X, e HillSlope - fattore di pendenza o pendenza collinare, che è senza unità.
NOTA: Per il raccordo viene utilizzato il software (Table of Materials). Utilizzare la concentrazione di 0,01 M (invece di 0,00 M) per il campione senza inibitore poiché il logaritmo di zero non è definito.
Risultati
In questo protocollo, sono stati testati otto composti (Figura 2A) insieme a IDP, un inibitore comune di pirofofosofasi, come controllo positivo. Ogni composto è stato testato in triplice copia a tre diverse concentrazioni (1 M, 5 M e 20 M) in triplice copia. Il flusso di lavoro dello screening è illustrato nella Figura 1, a partire dalla preparazione del campione e del reagente fino alla misurazione dell'assorbimento a 860 nm.
Alla fine di questo protocollo, dopo l'aggiunta della soluzione A e B e dell'arsenite-citrato, le soluzioni sviluppano un colore blu stabile con il massimo assorbimento a 709 nm e 860 nm14 a causa della complessa formazione di ioni fosfati con molybdate che può essere osservata e mostra il verificarsi della reazione enzimatica. Per questo esperimento, utilizziamo l'assorbimento a 860 nm per la misurazione della quantità di Pi rilasciata in quanto ha un migliore limite di rilevamento e sensibilità rispetto all'assorbimento a 709 nm15. Il colore blu è completamente sviluppato in 30 min di incubazione a temperatura ambiente e stabile per almeno 5 h14. Il saggio ha la sensibilità fino alla concentrazione di Pi di 10 M e l'assorbimento è lineare su un intervallo di concentrazione di 10-800 M14. Nel risultato rappresentativo qui, i pozzi E1-E3 (Figura 2C) contengono la miscela di reazione senza inibitore e la soluzione blu può essere osservata alla fine del saggio. Questo può essere osservato anche a basse concentrazioni composte in cui non è stata raggiunta l'inibizione completa, come nei pozzi F1-F3 per IDP e pozzi A4-A6 per il composto 1 (ATC, un inibitore non competitivo recente di TmPPase9) alla concentrazione di 2,5 e 1 M, rispettivamente. La maggiore concentrazione di IDP e composto 1, meno o meno il colore blu può essere osservata (G1-G3 e H1-H3 per IDP e B4B6 e C4-C6 per il composto 1) indicando l'inibizione dell'attività enzimatica. Tutte e tre le concentrazioni di composti non inibitori (2, 3e 8) mostravano la stessa intensità di colore blu, così come i pozzi E1-E3 senza alcun inibitore (Figura 2C).
Dopo la misurazione dell'assorbimento a 860 nm, i dati possono essere elaborati e analizzati (vedere la sezione protocollo 5). La figura 2D mostra il grafico di calibrazione dello standard Pi con il suo raccordo lineare (y - 0,0576x - 0,0019; r2 - 0,999). La figura 3 mostra la trama dell'attività ezimatica (%) contro la concentrazione di ogni composto testato. Per i composti con attività di inibizione, viene mostrato anche un raccordo di curva non lineare. L'IDP, utilizzato come controllo positivo, mostra chiaramente una diminuzione dell'attività a una maggiore concentrazione. L'IC50 (stima) calcolato sulla base di tre diverse concentrazioni è di 88,2 M (tabella 1), che è simile alla misura precedente (80,0 M) con otto punti di concentrazione14. Composti 1, 4, 5, 6, e 7 hanno mostrato una tendenza simile a quella dell'IDP poiché la concentrazione è aumentata con l'IC50 (stima) di circa 1,3 m, 7,4 m, 19,0 M, 37,4M e 156,1 M, rispettivamente(tabella 1). Per i composti 2, 3, e 8 nessuna riduzione dell'attività o dell'inibizione può essere osservata alle concentrazioni di analisi. Un ulteriore saggio con otto punti di concentrazione può essere fatto per generare preciso IC50. La figura 4 mostra la curva di inibizione per i composti 1, 5, 6, 7 e 8 con un IC50 di 1,7 M, 21,4 m, 58,8 M, 239,0 M e >500 M, rispettivamente9 .
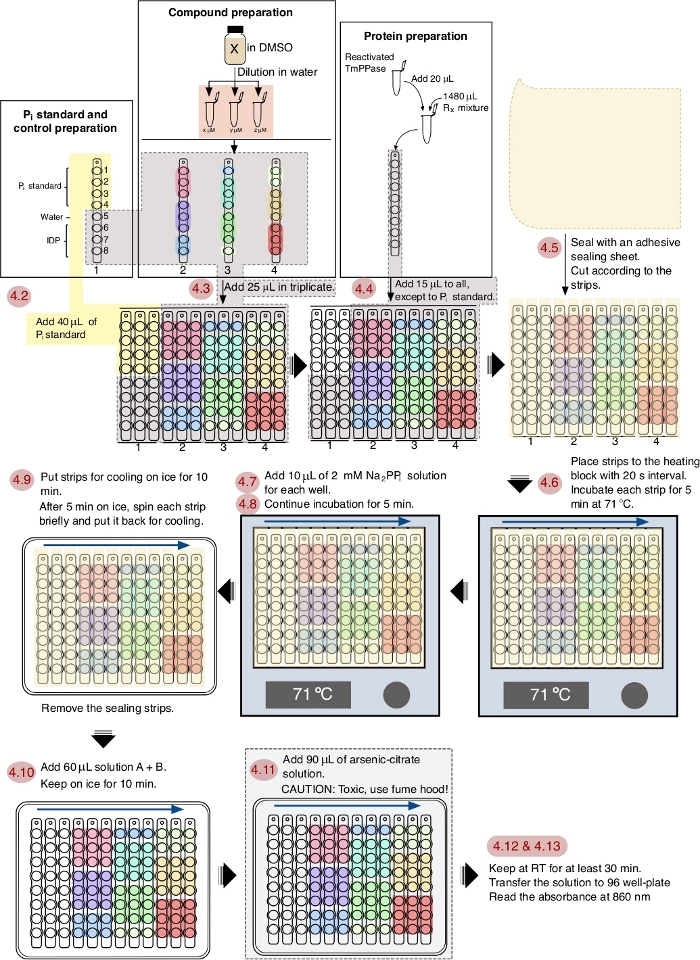
Figura 1: Un flusso di lavoro schematico del saggio di inibizione di TmPPase in un formato a 96 piastra. La numerazione rossa mostra i passi del saggio in base al protocollo e le frecce blu mostrano l'ordine degli intervalli. Fare clic qui per visualizzare una versione più grande di questa figura.
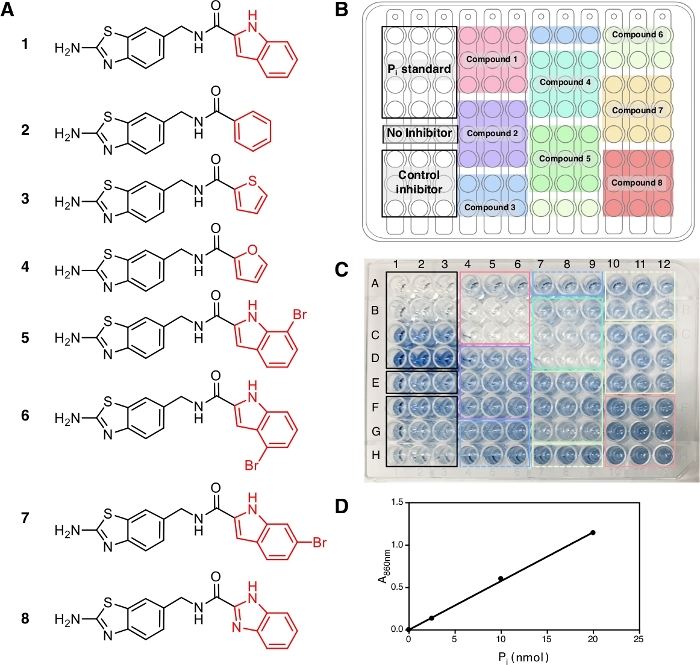
Figura 2: Campioni, la loro disposizione e lo sviluppo del colore in una piastra di 96 pozzi. (A) Le strutture dei composti 1e8 utilizzati per il saggio. L'attività di inibizione di questi composti è stata riportata in Vidilaseris et al.9. (B) Disposizione del campione. (C) Sviluppo del colore, 30 min dopo l'aggiunta della soluzione arsenite-citrato. Le concentrazioni di inibitore di controllo (IDP) e i campioni utilizzati, disposti dall'alto verso il basso, sono rispettivamente di 2,5 M, 25 m e di concentrazione di 250 M e di 1 concentrazione m, 5 M e 20 M. L'intensità del colore blu corrisponde alla quantità di Pi rilasciata a causa della reazione ezimatica e la mancanza di colore non corrisponde a nessuna reazione ezimatica. (D) Curva di calibrazione per lo standard Pi (nmol) rispetto a A860 con raccordo lineare (y - 0,0576x - 0,0019; r2 - 0,999). Fare clic qui per visualizzare una versione più grande di questa figura.
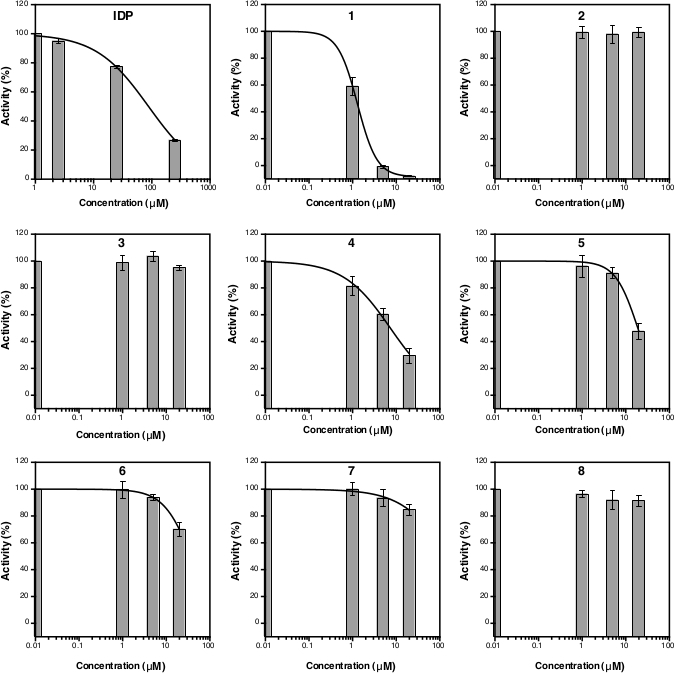
Figura 3: Curva dell'attività percentuale di TmPPase per tre diverse concentrazioni di inibitori. Le curve di regressione non lineari per calcolare l'IC50 (stima) sono indicate per idP così come per i composti 1, 4, 5 , 6e 7 ma non per i composti 2, 3e 8 in quanto non inibivano l'attività di TmPPase alle concentrazioni di analisi. Il logIC50 e IC50 (stima) di ogni composto è mostrato nella Tabella 1. Tutti i dati vengono visualizzati come media: SD con tre repliche. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 4: Curva di inibizione da otto punti di concentrazione di composti 1, 5, 6, 7 e 8. Questa cifra è tratta da Vidilaseris et al.9 con lievi modifiche. Tutti i dati vengono visualizzati come media: SD con tre repliche. Fare clic qui per visualizzare una versione più grande di questa figura.
| Esempio | LogIC50 (in modo | IC50 (stima) |
| Idp | 1,95 x 0,0142 | 87,9 x 2,46 |
| 1 | 0,112 - 0,0274 | 1,29 x 0,0816 |
| 2 | − | nessuna inibizione |
| 3 | − | nessuna inibizione |
| 4 | 0,870 - 0,0447 | 7,39 x 0,760 |
| 5 | 1.28 - 0,0296 | 19.0 - 1,29 |
| 6 | 1,57 - 0,0846 | 37,4 x 7,29 |
| 7 | 2.19 - 0,366 | 156 x 131 |
| 8 | − | nessuna inibizione |
Tabella 1: LogIC50 e IC50 (stima) di IDP e composti 1/8 in base ai dati della Figura 3.
Discussione
Qui riportiamo un protocollo dettagliato per lo screening semplice degli inibitori per la pirofosasi legata alla membrana da T. maritima in un formato 96 pozzo basato su Vidilaseris et al.14. Questo protocollo è poco costoso e basato su 12-acido fosforolismibdico, che è formato da ortopofosfato e molybdate in condizioni acide e ridotto a specie di fosfomilybdenum con un colore blu distinto12. Questo metodo è preferito rispetto ad altri protocolli, come il più sensibile asdetto verde malachite16, perché questo metodo non mostra interferenze in presenza di alta concentrazione di fosfolipidi che è necessaria per La riattivazione DiTmPPase14.
Il flusso di lavoro del protocollo di screening è illustrato nella Figura 1 e questo processo può essere completato in 1 h. Questo protocollo è ottimizzato per TmPPase con la temperatura di lavoro ottimale a 71 gradi centigradi e un tempo di reazione di 5 min. Man mano che l'acqua evapora a questa temperatura dalla miscela di reazione, viene applicato un foglio di tenuta adesivo (affettato per adattarsi e coprire le strisce) per evitare l'evaporazione14 e l'acqua evaporata viene semplicemente riraccoltata con centrifugazione. Il tempo di incubazione di 5 min è scelto in quanto è ancora nella gamma lineare del fosfato ezimaticamente rilasciato e sufficiente per uno screening affidabile14. In questo protocollo, i tempi e le abilità di pipettaggio sono fattori importanti per ottenere un risultato buono e affidabile. L'aggiunta di reagenti durante il saggio con intervallo di 20 s tra le strisce è un'opzione di temporizzazione ottimizzata per facilitare l'esecuzione dei passaggi successivi.
Per i diversi mPPases, la temperatura ottimale e il tempo di incubazione devono essere determinati separatamente prima dell'uso nell'anibizza. Il protocollo di riattivazione degli enzimi di cui sopra è ottimizzato per TmPPase e altri mPPases potrebbero aver bisogno di un protocollo di riattivazione diverso. Ad esempio, DDM non dovrebbe essere aggiunto per la riattivazione di mPPase da Pyrobaculum aerophilum in quanto diminuirà la sua attività ezimatica17. Poiché l'enzima diventerà meno attivo se preparato con largo anticipo, l'aggiunta dell'enzima riattivato deve essere aggiunta alla miscela di reazione poco prima dell'inizio del saggio. Dopo l'aggiunta della soluzione arsenite-citrato il prodotto di reazione è stabile per almeno 5 h14. Pertanto, il batch successivo del saggio può essere eseguito immediatamente e la misurazione dell'assorbimento può essere eseguita in un secondo momento per tutti i lotti contemporaneamente.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto dalle sovvenzioni della Jane and Aatos Erkko Foundation e della BBSRC (BB/M021610) ad Adrian Goldman, l'Accademia di Finlandia (n. 308105) a Keni Vidilaseris, (n. 310297) a Henri Xhaard, e (n. 265481) a Jari Yli-Kauhaluoma, e l'Università di Helsinki Fondi di Ricerca a Gustav Boije af Gennàs. Gli autori ringraziano Bernadette Gehl per il suo aiuto tecnico durante il progetto.
Materiali
| Name | Company | Catalog Number | Comments |
| Adhesive sealing sheet | Thermo Scientific | AB0558 | |
| Ammonium heptamolybdate tetrahydrate | Merck | F1412481 636 | |
| Ascorbic acid | Sigma-Aldrich | 95212-250G | |
| BioLite 96Well Multidish | Thermo Scientific | 130188 | |
| Dimethyl sulfoxide (DMSO) | Merck | 1167431000 | |
| 8-well PCR Tube Strips 0.2 ml without caps (120) | Nippon genetics | FG-028 | |
| Dodecyl maltoside (DDM) | Melford | B2010-100G | |
| Ethanol | Merck | 1009901001 | |
| Glacial acetic acid | Merck | 1000631011 | |
| Hydrochloric acid | Sigma-Aldrich | 258148-500ML | |
| Imidodiphosphate sodium salt | Sigma-Aldrich | I0631-1G | |
| L-α-Phosphatidyl choline from soybean lecithin | Sigma | 429415-100GM | |
| Magnesium chloride | Sigma-Aldrich | 8147330500 | |
| Multiplate 96-Well PCR Plates | Bio-Rad | MLL9651 | |
| MultiSkan Go | Thermo Scientific | 10680879 | |
| Nepheloskan Ascent (Type 750) | Labsystems | ||
| Polystyrene Petri dish (size 150 mm x 15 mm) | Sigma-Aldrich | P5981-100EA | |
| Potassium chloride | Merck | 104936 | |
| Prism 6 software | GraphPad | ||
| QBT2 Heating block | Grant Instruments | ||
| Sodium meta-arsenite | Fisher Chemical | 12897692 | |
| Sodium phosphate dibasic (Pi) | Sigma | S0876-1KG | |
| Sodium pyrophosphate dibasic | Fluka | 71501-100G | |
| Trisodium citrate dihydrate | Fluka | 71404-1KG |
Riferimenti
- Terstappen, G. C., Reggiani, A. In silico research in drug discovery. Trends in Pharmacological Sciences. 22 (1), 23-26 (2001).
- Rask-Andersen, M., Almen, M. S., Schioth, H. B. Trends in the exploitation of novel drug targets. Nature Reviews Drug Discovery. 10 (8), 579-590 (2011).
- Shah, N. R., Vidilaseris, K., Xhaard, H., Goldman, A. Integral membrane pyrophosphatases: a novel drug target for human pathogens. AIMS Biophysics. 3 (1), 171-194 (2016).
- Baykov, A. A., Malinen, A. M., Luoto, H. H., Lahti, R. Pyrophosphate-Fueled Na+ and H+ Transport in Prokaryotes. Microbiology and Molecular Biology Reviews. 77 (2), 267-276 (2013).
- Serrano, A., Perez-Castineira, J. R., Baltscheffsky, M., Baltscheffsky, H. H+-PPases: yesterday, today and tomorrow. IUBMB Life. 59 (2), 76-83 (2007).
- Liu, J., et al. A vacuolar-H+-pyrophosphatase (TgVP1) is required for microneme secretion, host cell invasion, and extracellular survival of Toxoplasma gondii. Molecular Microbiology. 93 (4), 698-712 (2014).
- Lemercier, G., et al. A pyrophosphatase regulating polyphosphate metabolism in acidocalcisomes is essential for Trypanosoma brucei virulence in mice. Journal of Biological Chemistry. 279 (5), 3420-3425 (2004).
- Belogurov, G. A., et al. Membrane-bound pyrophosphatase of Thermotoga maritima requires sodium for activity. Biochemistry. 44 (6), 2088-2096 (2005).
- Vidilaseris, K., et al. Asymmetry in catalysis by Thermotoga maritima membrane-bound pyrophosphatase demonstrated by a nonphosphorus allosteric inhibitor. Science Advances. 5 (5), (2019).
- Lin, S. M., et al. Crystal structure of a membrane-embedded H+-translocating pyrophosphatase. Nature. 484 (7394), 399-403 (2012).
- Fiske, C. H., Subbarow, Y. The colorimetric determination of phosphorus. Journal of Biological Chemistry. 66 (2), 375-400 (1925).
- Nagul, E. A., McKelvie, I. D., Worsfold, P., Kolev, S. D. The molybdenum blue reaction for the determination of orthophosphate revisited: Opening the black box. Analytica Chimica Acta. 890, 60-82 (2015).
- Kellosalo, J., Kajander, T., Palmgren, M. G., Lopez-Marques, R. L., Goldman, A. Heterologous expression and purification of membrane-bound pyrophosphatases. Protein Expression and Purification. 79 (1), 25-34 (2011).
- Vidilaseris, K., Kellosalo, J., Goldman, A. A high-throughput method for orthophosphate determination of thermostable membrane-bound pyrophosphatase activity. Analytical Methods. 10 (6), 646-651 (2018).
- He, Z. Q., Honeycutt, C. W. A modified molybdenum blue method for orthophosphate determination suitable for investigating enzymatic hydrolysis of organic phosphates. Communications in Soil Science and Plant Analysis. 36 (9-10), 1373-1383 (2005).
- Martin, B., Pallen, C. J., Wang, J. H., Graves, D. J. Use of fluorinated tyrosine phosphates to probe the substrate specificity of the low molecular weight phosphatase activity of calcineurin. Journal of Biological Chemistry. 260 (28), 14932-14937 (1985).
- Strauss, J., Wilkinson, C., Vidilaseris, K., Harborne, S. P. D., Goldman, A. A simple strategy to determine the dependence of membrane-bound pyrophosphatases on K+ as a cofactor. Methods in Enzymology. 607, 131-156 (2018).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon