Method Article
Uso de uma única molécula fluorescente hibridação In Situ (FISH-SM) para Quantify e Localize os mRNAs em oócitos murino
Neste Artigo
Resumo
Para reproducibly contar os números dos mRNAs em oócitos individuais, fluorescência única molécula de RNA in situ hibridação (RNA-FISH) foi otimizada para células não-aderentes. Oócitos foram coletados, hibridizados com sondas específicas transcrição e quantificados usando um software de quantificação de imagem.
Resumo
Métodos atuais rotineiramente usados para quantificar o mRNA de oócitos e embriões incluem digital polimerase transcrição reversa reação em cadeia (dPCR), quantitativo, real-time RT-PCR (RT-qPCR) e sequenciação do ARN. Quando estas técnicas são realizadas usando um único oócito ou embrião, baixo-cópia mRNAs confiável não são detectados. Para superar este problema, oócitos ou embriões podem ser agrupados para análise; no entanto, isso muitas vezes leva a alta variabilidade entre as amostras. Neste protocolo, descrevemos o uso da fluorescência hibridação in situ (FISH) usando química de DNA ramificada. Esta técnica identifica o padrão espacial dos mRNAs em células individuais. Quando a técnica está associada a encontrar o local e software de computador de rastreamento, a abundância de mRNAs na célula também pode ser quantificada. Usando esta técnica, há reduzida variabilidade dentro de um grupo experimental e menos oócitos e embriões são necessários para detectar diferenças significativas entre os grupos experimentais. Comercialmente disponível DNA ramificado SM-kits de peixes foram otimizados para detectar mRNAs em secionado tecidos ou células aderentes em slides. No entanto, oócitos efetivamente não adere aos slides e alguns reagentes do kit foram muito duras, resultando em lise do oócito. Para evitar este Lise, várias modificações foram feitas para o kit de peixe. Especificamente, buffers de permeabilização e lavagem de oócito projetados para a imunofluorescência de oócitos e embriões substituiu os buffers de proprietários. A permeabilização, lavagens e incubação com sondas e amplificador foram realizadas em placas de 6-poços e oócitos foram colocados em slides no final do protocolo utilizando meios de montagem. Essas modificações foram capazes de superar as limitações do kit comercialmente disponível, em particular, a lise do oócito. Para precisão e reproducibly contar o número dos mRNAs em oócitos individuais, utilizou-se o software de computador. Juntos, este protocolo representa uma alternativa para PCR e sequenciamento para comparar a expressão de transcritos específicos em células únicas.
Introdução
Transcriptase reversa cadeia da polimerase (PCR) tem sido o padrão-ouro para quantificação de mRNA. Dois ensaios, digital PCR (dPCR)1 e quantitativo, real time PCR (qPCR)2 são utilizados atualmente. Das duas técnicas de PCR, dPCR tem maior sensibilidade do que qPCR, sugerindo que poderia ser usado para medir a abundância de mRNA em células únicas. No entanto, em nossas mãos, dPCR análise de mRNAs baixa abundância nas piscinas de ovócitos de 5 a 10 por cada amostra experimental produziu dados com baixa reprodutibilidade e alta variação3. Isto é provavelmente devido ao erro experimental associado a extração do RNA e transcrição reversa eficiência. A sequenciação do ARN também foi executada usando um único rato e oócitos humanos4,5. Esta técnica requer etapas de amplificação do cDNA necessárias para a geração de biblioteca que provavelmente aumenta a variabilidade dentro de um grupo experimental. Além disso, transcrições de baixa abundância podem não ser detectáveis. Embora os preços de sequenciamento caíram nos últimos anos, ainda pode ser custo proibitivo devido ao alto custo das análises de Bioinformática. Finalmente, a localização de mRNA é um processo dinâmico, com alterações espaciais, contribuindo para a proteína função6. Portanto, decidimos para adotar uma técnica que produziria medidas quantitativas precisas e reprodutíveis e localização dos mRNAs individuais em oócitos único.
DNA em cadeia ramificada acoplado a fluorescência hibridação in situ amplifica o sinal de fluorescência em vez de amplificação do RNA/cDNA permita deteção dos mRNAs único em células individuais 7,8,9. O ensaio é realizado através de uma série de hibridização e amplificação (usando DNA em cadeia ramificada) fluorescência rotulagem passos a fim de amplificar o sinal de fluorescência7. A técnica começa com ligação de pares de sonda de 18 a 25-base do oligonucleotide que são complementares a uma específica do mRNA3,8,10. Quinze a vinte pares de sonda são projetados para cada especificidade de transcrição garantindo para a transcrição do alvo. A hibridação de mRNA específico é seguida por sondas pré-amplificador e amplificador que formam uma configuração ramificada. Aproximadamente, 400 rótulo fluorophores bind para cada amplificador, resultando em um 8000-fold aumento na fluorescência permitindo a deteção de mRNAs individuais (Figura 1)11.
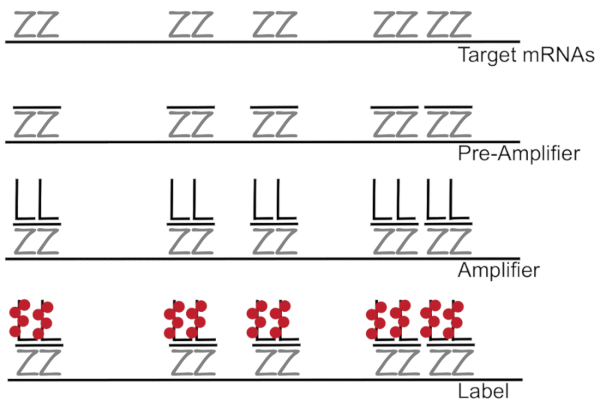
Figura 1: esquemático do protocolo SM-peixes. Sequencial hibridização da sonda específica de transcrição, ramificado DNA amplificador e fluoróforo para um destino de mRNA é mostrado. Clique aqui para ver uma versão maior desta figura.
Estudos anteriores usando fluorescência única molécula em situ da hibridação (SM-peixe) localizada β-actina mRNAs em neurônios individuais12 e DNA de papilomavírus humano em câncer de colo uterino de células linhas7. O software de computador, encontrar o local e o programa de rastreamento identifica o sinal fluorescente punctate individual e tem sido usada com sucesso para quantificar o número de mRNAs em cada célula3,13.
Baseado nos resultados da deteção do mRNA em neurônios12, formulamos a hipótese que SM-peixe provaria uma ferramenta útil para dosar os níveis de transcrição de murino oócitos e embriões, incluindo baixa abundância mRNAs. No entanto, a técnica é otimizada para uso com pilhas fixos aderentes e formaldeído fixada parafina incorporado cortes de tecido (FFPE). Oócitos não podem aderir a um slide, mesmo quando eles são revestidos com poli-L-lisina. Além disso, eles são mais frágeis do que as células somáticas e cortes de tecido, resultando em lise celular quando submetido a alguns dos buffers de proprietários em kits comercialmente disponíveis3. Para superar estes desafios, oócitos foram fixo e manualmente transferidos entre gotas dos buffers. Além disso, os buffers de permeabilização e lavagem nos kits foram substituídos para reduzir o lysis da pilha. Sondas preconcebidas são compradas juntamente com o kit de peixe ou transcrições específicas podem ser solicitadas. Cada conjunto de sonda proprietário está disponível em um dos três canais de fluorescência (C1, C2 e C3) para permitir a multiplexação. Na experiência atual, murino oócitos foram manchadas de dual e quantificados usando uma sonda de C2 Nanog e uma sonda de C3 Pou5f1 . Estas sondas foram selecionadas com base em expressão relatado de Nanog e Pou5f1 de oócitos e embriões. Na conclusão das etapas da hibridação, oócitos foram colocados em gotas de mídia anti-desvaneça-se montagem para aplicação de lâminas histológicas. Confocal imagens foram usadas para quantificar o número de sinais fluorescentes punctate que representam os mRNAs individuais. Além de quantificar os mRNAs, imagem também mostrou a distribuição espacial do mRNA específico na célula, quais outros métodos de quantificação de RNA são incapazes de alcançar. Esta técnica provou ter baixa variabilidade dentro de um grupo experimental, permitindo o uso de um menor número de oócitos em cada grupo experimental para identificar diferenças significativas entre os grupos experimentais3.
Protocolo
Animais procedimentos foram revisados e aprovados pelo Comitê de uso da Universidade de Nebraska-Lincoln e institucional Cuidado Animal e todos os métodos foram realizados em conformidade com os regulamentos e orientações pertinentes. Para este estudo, CD-1 consanguíneo ratos tem acesso ad libitum chow roedor normal e água; Eles foram mantidos em um 12:12 escuros: ciclo de luz.
1. preparação dos meios de comunicação necessários
- Para a mídia de base (OMM), adicione 100 mM NaCl, KCl de 5 mM, 0.5 mM KH2PO4e 1,7 mM CaCl2-2 H2O a 100 mL de água estéril.
Nota: Médio OMM pode ser armazenado por até 1 mês. - Para a mídia completa (OMOPS), adicionar 20 mM 3-morpholinopropane-1-sulfônico (MOPS), 1,2 mM de MgSO4-7 H2O, glicose de 0,5 mM, 6 mM L-lactato, 1mm ala-gln, taurina, 0,1 mM 1 x não aminoácidos essenciais (timina), 0,01 mM (de ácido etilenodiaminotetracético EDTA), o ácido alfa lipoico 10 µM, 10 gentamicina µ g/mL sem diluir, 21 mM 1 M NaOH, 5mm NaHCO3, 0,2 mM piruvato, 0.5 mM citrato, 4mg/mL FAF BSA para uma 01:10 diluição da OMM em água estéril para um volume total de 100 mL. Esterilize o meio com um filtro de 0,22 µM.
Nota: OMOPS pode ser armazenado por até 1 semana. - Por meio de exploração (HM) Adicione 5% de soro fetal bovino para OMOPS. Fazer 2 mL HM por rato.
- Para solução de hialuronidase Adicione 0,1 mg/mL de hialuronidase derivado de testículos de bovinos, a HM de 1 mL.
- Para o buffer de fixação combine paraformaldeído 4% em 10 mL de 1X PBS juntamente com 0,1% embrião grau polivinilpirrolidona (PVP)14.
- Para preparar 50 mL de tampão de lavagem (WB), adicionar 0,1% de tensoativo não-iônico e 0,1% PVP de 1X PBS14.
- Para preparar 10 mL de tampão de permeabilização, adicione 1% não - iónico de 1X PBS14.
Nota: Os buffers de lavagem e permeabilização descritos acima substituem os buffers proprietários nos kits comercialmente disponíveis.
2. recolha de ovócitos ovulados dos ratos fêmeas
- Preparação:
- Estimular os ratos fêmeas em 5-8 semanas de idade com uma injeção intraperitoneal (IP) de 5 IU equinos gonadotrofina coriônica (eCG) seguida de 5 IU gonadotrofina coriônica humana (hCG) 44-48 h mais tarde15,16.
- Manter pratos de Petri 35mm, contendo 2 mL de HM em uma placa de aquecimento de 37 ° C. Pipete uma, gota de 100 µ l de HM contendo hialuronidase diluída, seguido de três, gotas de 50 µ l de HM sem hialuronidase em um prato de petri de 60 mm. Coloque placas contendo gotas sobre a 37 ° C de aquecimento da placa antes da utilização.
Nota: As gotas de hialuronidase devem ser feitas antes da dissecação de cada par de oviduto para evitar a evaporação e concentração dos componentes da HM com ou sem hialuronidase.
- Eutanásia em ratos, 16 h após a injeção do IP do hCG, utilizar isoflurano sobredosagem seguido por deslocamento cervical.
- Limpe o mouse usando etanol a 70%. Expor na cavidade abdominal e visualizar o trato reprodutivo feminino. Segure o ovário com pinça e retire os ligamentos uterinos e o excesso de tecido adiposo ao redor do ovário. Corte do oviduto, do útero e coloque o par de ovário-oviduto no HM quente no prato de 35 mm.
- Remova o ovário e qualquer tecido adiposo circundante. Rasgo na ampola inchada de oviduto usando uma agulha de calibre 27 de ½ polegada. Empurre o oviduto no site do rasgo e os complexos de célula-oócito cumulus (COCs) serão expulso. Transferência de oócitos ovulated, que se presume ser em metáfase II (MII) da meiose, para a entrega de 100 μL contendo mídia HM com hialuronidase usando uma pipeta de boca (Figura 2).

Figura 2 : Peças da pipeta de boca usado para transferência de oócitos. (A) boca da parte (B) 0.22, 4mm filtro (C) tubulação (D) 1000 μL pipeta ponta aspiradora (E) 9" pipeta Pasteur. Clique aqui para ver uma versão maior desta figura.
- Pipete os complexos de célula cumulus-oócito MII acima e para baixo na hialuronidase contendo HM com a pipeta de boca para desalojar as células do cumulus. Cada oócito de transferência, uma vez que eles são desprovidos de células do cumulus para uma lavagem soltar HM contendo apenas usando a pipeta de boca. Repita isto para cada gota de lavagem. Não transferência de oócitos fragmentada ou transparente15.
Nota: É importante transferir os oócitos de cada gota no prato com 35mm como HM pouco quanto possível. Isto é verdadeiro para cada transferência no protocolo. Os oócitos MII não devem permanecer na hialuronidase contendo meio de HM para mais de um minuto.
3. SM-peixe de coloração de ovócitos
- Resolver os oócitos em um poço individual de uma placa de 6 contendo 500 µ l de tampão de fixação. Mergulhe 20 oócitos ou menos no poço. Incube durante 20 min à temperatura ambiente.
Nota: Cada passo de coloração SM-peixe ocorre dentro de um poço individual em uma placa 6-poço cónico. Certifique-se de que os oócitos são completamente submerso nos buffers e não flutuando em cima do buffer. Cada passo deve ser realizada com 20 oócitos ou menos em cada poço. - Transferência de ovócitos fixos para 500 μL de tampão de lavagem (WB descrito na etapa 1.6) por 10 min cada. Repita 2 vezes mais.
- Incube os oócitos no buffer de permeabilização por 30 min à temperatura ambiente.
Nota: O buffer de permeabilização descrito na etapa 1.7 substitui o buffer de permeabilização de decoro.- Reunir conjuntos de sonda e rapidamente girá-los para baixo em um microcentrifuge. Aquecer cada sonda definida por 10 min em um banho de água a 40 ° C ou incubadora. Esfriar até a temperatura de quarto.
Nota: Esta etapa deve ser executada durante a incubação de permeabilização
- Reunir conjuntos de sonda e rapidamente girá-los para baixo em um microcentrifuge. Aquecer cada sonda definida por 10 min em um banho de água a 40 ° C ou incubadora. Esfriar até a temperatura de quarto.
- Lave os oócitos em 500 µ l de WB por 10 min à temperatura ambiente.
- Transferência de ovócitos de 80 μL de Protease III Buffer (disponível a partir do kit), que é diluído 1:8 em 1X PBS, por 30 min à temperatura ambiente.
Nota: O volume de 80 µ l adequadamente cobre o fundo de um poço individual em uma placa de 6. - Lave os oócitos em 500 µ l de WB por 10 min à temperatura ambiente.
- Diluir os conjuntos de sonda aquecida para Nanog, Pou5f1 e DapB (um gene de controlo negativo), 01:50 no diluente de sonda. Incubar os oócitos em 80 μL da sonda específicos de transcrição por 2 horas a 40° C.
Nota: Cada conjunto de sonda proprietário está disponível em um dos três canais de fluorescência (C1, C2 e C3). As sondas Nanog e Pou5f1 foram marcadas com C2 e C3, respectivamente. - Aquecer o proprietário, 1 amplificador (1 AMP), amplificador 2 (AMP2), amplificador 3 (AMP3) e amplificador 4-fluorescência (AMP 4-FL) à temperatura ambiente.
Nota: Esta etapa deve ser executada durante a incubação sonda de transcrição específicos de 2 horas. - Transferência de oócitos para 500 μL de WB e incubar durante 10 minutos à temperatura ambiente.
- Incube os oócitos sequencialmente nos buffers de amplificação.
- Incube os oócitos em 80 µ l de AMP1 durante 30 min em oócitos de transferência de 40° c a 500 µ l de WB, durante 10 minutos à temperatura ambiente.
- Incubar os oócitos em 80 µ l de AMP2 por 15 min a 40 ° C. Transferência de ovócitos a 500 µ l de WB por 10 min à temperatura ambiente.
- Incubar os oócitos em 80 µ l de AMP3 por 30 min a 40 ° C. Transferência de ovócitos a 500 µ l de WB por 10 min à temperatura ambiente.
Nota: O restante do protocolo é realizado no escuro porque AMP-FL contém o fluoróforo. Quando se trabalha sob o microscópio de dissecação, reduza a luz, tanto quanto possível. - Adicionar oócitos de 80 μL de AMP4-FL 15 min a 40° C.
Nota: AMP4-FL é fornecido como alternativa de reserva-A (Alt-A), Alt-B ou Alt-C. Selecione o buffer de AMP4-FL dependente na qual emissão de comprimento de onda é desejado.
- Lave os oócitos em 500 µ l de WB por 10 min à temperatura ambiente. Incube os oócitos em 80 µ l de DAPI por 20 min em temperatura ambiente. Lave os oócitos em 500 µ l de WB por 5 min à temperatura ambiente.
- Pipete 12 µ l de mídia anti-fading montagem no centro de um slide sem adição de bolhas para o reagente. Transferir oócitos com como pouco WB quanto possível para a mídia de montagem e uma lamela.
- Lamela em um ângulo de inclinação e lenta e suavemente, coloque sobre o líquido no slide. Evite pressionar a lamela muito duro para impedir a distorção dos oócitos e introdução de bolhas.
- Armazene os slides em uma caixa escura para secar durante a noite em temperatura ambiente. Revesti as bordas dos slides em esmaltes claros para selar a lamela.
- Use um microscópio padrão para encontrar oócitos sobre o slide e o círculo com um marcador permanente.
Nota: Este passo não é obrigatório, mas melhora a localização oócitos no slide. Para melhores resultados, slides de imagem dentro de 1 a 5 dias como o sinal fluorescente começará a desvanecer-se.
4. processamento de imagem
- Imagem 3-dimensional oócitos, utilizando microscopia confocal de passo de z.
Nota: Para analisar com precisão as imagens, cada passo de z deve ser 1,0 µm/fatia. - Salve imagens confocal como um comprimido nd2 ou individuais. Arquivos TIFF para cada oócito. Ambos os tipos de imagem são compatíveis com o programa de processamento de imagem open source, Fiji.
- Baixe e instale o software de Fiji de acesso aberto (https://imagej.net/Fiji/Downloads).
- Arraste arquivos nd2 em Fiji e escolha hyperstack. Se imagens confocal foram salvos como. Arquivos TIFF pule para a etapa 4.4.
Nota: Quando o arquivo de nd2 é Descartado em Fiji o dropdown hyperstack deve aparecer automaticamente. - Clique na guia imagem , selecione a core clique em Canais de Split para separar os canais fluorescentes do arquivo nd2.
- Gere individuais. Arquivos TIFF para cada z-fatia do oócito em cada canal fluorescente. Clique na guia imagem , selecione Stackse clique em pilha de imagens. Clique na guia imagem , selecione o tipoe clique em Cor RGB para converter cada fatia de z para uma imagem de cor RGB individual.
Nota: A cor do RGB é artificial e pode ser escolhido como desejado para cada comprimento de onda de emissão. - Salve cada imagem convertida como. Arquivo TIFF. Coloque imagens de um oócito único para cada canal fluorescente em uma nova pasta para evitar confusão durante a costura (etapa 4.3).
- Arraste arquivos nd2 em Fiji e escolha hyperstack. Se imagens confocal foram salvos como. Arquivos TIFF pule para a etapa 4.4.
- Normalize a cada um. Imagem TIFF para Pou5f1 e Nanog usando imagens de controlo negativo (DapB).
Nota: A normalização é executada usando um programa de edição de fotos. Certifique-se de remover os mesmos níveis de fluorescência de fundo de cada imagem do controle. - Abra cada normalizado. Arquivo TIFF em Fiji para costurar todos os z-fatias para cada oócito em cada comprimento de onda.
- Clique na guia de Plugins , selecione costurae clique na Grade/coleção (Figura 3A). Selecione Imagens sequenciais do menu drop-down e clique Okey (Figura 3B).
- Procure o diretório e selecione a pasta que contém todas as imagens de z-fatia para um oócito individual em um comprimento de onda (ver passo 4.3.4). Clique Okey.
- Mova o controle deslizante na parte inferior da imagem costurada para o canal de cor apropriada para o comprimento de onda utilizado e criar a imagem final de costurados RGB clicando em imagem, selecionar o tipoe clique em Cor RGB.
Nota: Esta imagem será usada para quantificação de fluorescência descrita na etapa 4.6 abaixo.
- Converta a imagem costurada para 32-bit máxima imagens projetadas. Clique na imagem, selecione o tipoe clique em 32-bit (Figura 3). Salve esta imagem como um novo. Arquivo TIFF.
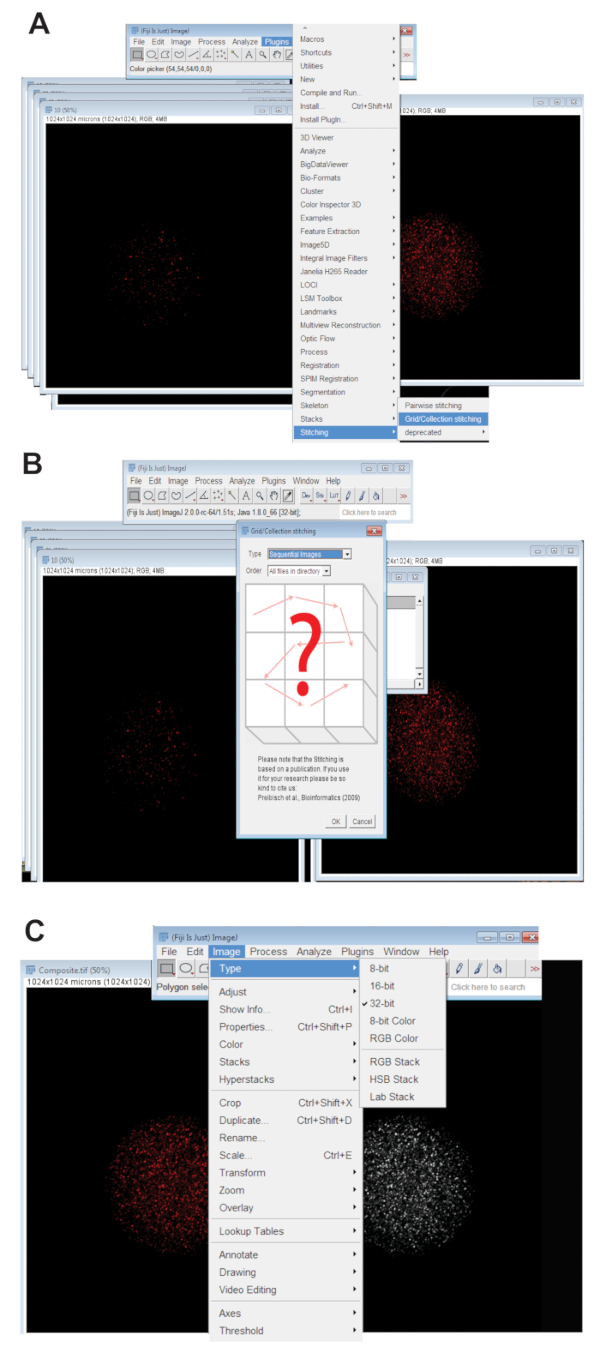
Figura 3 : Costura juntos de imagens confocal z-series de ovócitos. (A) Screenshot mostrando a ferramenta grade/coleção de plug-in em Fiji que foi usado para produzir imagens compostas do oócito. (B) imagens sequenciais usa sobreposição de fluorescência entre sequencial. Arquivos TIFF para gerar uma imagem composta. (C) a imagem composta foi salvo como um 32-bit. Arquivo TIFF. Clique aqui para ver uma versão maior desta figura.
- Baixe e instale o local encontrando e programa de rastreamento13, que está disponível no site para D.R. Larson, um investigador no nacional institutos de saúde Instituto Nacional de câncer (https://ccr.cancer.gov/Laboratory-of-Receptor-Biology-and-Gene-Expression/daniel-r-larson). Baixe e instale a máquina virtual de acesso livre para o sistema operacional no idioma (IDL) interativa de dados que é necessário para executar o programa de rastreamento (http://www.spacewx.com/pdf/idlvm.pdf) e encontrar o lugar.
- Abra a imagem de 32 bits, costurada, que foi gerada na etapa 4.6 (Figura 4A), no local encontrando e programa de rastreamento. Selecione o menu suspenso localizar e clique em Localizar (Figura 4B), que irá calcular as números manchas encontradas na imagem.
Nota: Cada ponto contado representa um mRNA individuais. Configurações de limite de banda pass e fóton são mostradas na captura de tela (Figura 4B). Para este protocolo, utilizou-se o padrão para cada configuração de limiar. Representante positivo e manchas de fundo são mostradas (Figura 4).

Figura 4 : Quantificação dos mRNAs utilizando Spot Finder e rastreamento. (A) z-série Individual imagens foram costuradas conforme descrito na Figura 3 e salvo como um máximo de 32-bit projectado. Arquivo TIFF. (B) imagem composta foi inaugurada em Spot Finder e rastreamento. Localizar foi usada para contar as manchas fluorescentes (caixa vermelha). Limite de banda pass e fóton são indicadas pela caixa azul. (C) a seta azul aponta para um sinal positivo (acima do limiar). A seta branca mostra uma fluorescente ponto abaixo do limiar e, portanto, não contados. Clique aqui para ver uma versão maior desta figura.
Resultados
Após a conclusão do protocolo, o resultado será imagens individuais de confocal z-series (Figura 4A e Figura 5), imagens costuradas (Figura 4), e contagens de mRNA (Figura 4B). Quando é executada a multiplexação, haverá também mescladas imagens mostrando o rótulo para dois diferentes mRNAs (Figura 5). As contagens de mRNA são geradas usando imagens costuradas geradas por Fiji (Figura 3) e as manchas de fluorescência punctate contadas usando o local encontrando e programa de rastreamento (Figura 4B).
As contagens de mRNA são posteriormente analisadas usando uma ferramenta de análise de dados padrão. Neste protocolo, que rotulado n = 12 ovócitos com Pou5f1 e Nanog. Os resultados para cada mRNA são médias e erro padrão da média calculada. Os dados coletados no presente protocolo mostrou 775 ± 26 SEM Pou5f1 transcrições e 113 ± 5 SEM Nanog transcrições em oócitos MII (Figura 5). Teste-t de student-uma determinada que diferença estatisticamente significativa no mRNA conta entre Pou5f1 e Nanog. Importante, não havia nenhuma mancha detectada em n = 5 oócitos que são rotulados com sonda DapB (ou seja, controle negativo). Observe o pequeno erro padrão usando apenas 12 ovócitos individuais. A sensibilidade do ensaio é também enfatizada por positivos podem ser reproduzidos na detecção de n(Figura 5) Nanog. Experiências anteriores de dPCR não detectar reproducibly Nanog indica que o número de mRNAs Nanog em um oócito individual é inferior a deteção de limite usando dPCR 3.
Em experiências piloto, detectamos a fluorescência reduzida se houvesse um atraso entre a fixação de ovócitos e iniciação do protocolo SM-peixe. Da mesma forma, se não forem utilizados os buffers de hibridização proprietárias, a sonda e amplificação de DNA em cadeia ramificada não entra na célula, resultando em fluorescência dos anéis ao redor da membrana plasmática do oócito. Isto é provavelmente devido à agregação de DNA em cadeia ramificada. Fluorescência com anéis ao redor da membrana do ovócito também resultará se houver pobre permeabilização da membrana plasmática. Degradação ideal de proteína vinculada a mRNAs também é necessária. O buffer de protease fornecido no kit de SM-peixe é uma concentração ideal para o tratamento de cortes de tecido e células aderentes. No entanto, quando usando células não-aderentes, é importante identificar empiricamente a melhor diluição de protease. Buffer muito pequeno de protease pode resultar em subcontagens de mRNAs devido a pouca acessibilidade da sonda para o mRNA. Da mesma forma, demasiado protease pode resultar em degradação de não só proteínas vinculado para o mRNA mas também desestabilização dos mRNAs. Neste protocolo, testamos a deteção do mRNA usando uma curva de titulação debuffer não diluído (1:1), 1:4 e 1:8 01:12 protease diluídos em PBS 1x (n = 2 a 3 oócitos por diluição). A média fluorescente expressão do mRNA da Pou5f1 em oócitos MII foi 169 ± 42 SEM (sem diluir) 176 ± 36 SEM (1:4), 308 ± 18 SEM (1:8) e 445 ± 24 SEM (01:12) (Figura 6). Diluição de protease usada neste protocolo foi 1:8 como ele mostrou a menor variação.

Figura 5 : Pou5f1 e Nanog mRNA no oócito MII. (A). imagens representativas da imagem meio z-series são mostradas à esquerda. Pou5f1 é detectado no vermelho (647 nm) comprimento de onda enquanto Nanog é detectado no verde (488 nm) comprimento de onda. DAPI coloração de cromossomos alinhados no eixo metáfase II, característico do oócito MII, é mostrado em branco. Não havia nenhuma mancha para DapB em também a 647 nm ou 488 comprimento de onda de emissão nm. (B) o número de Pou5f1 e Nanog mRNAs é mostrado como dizer SEM (n = 12 ovócitos); Não houve nenhuma detecção (N.D) de DapB. * indica P < 0.05, barra de escala é 10 µm. clique aqui para ver uma versão maior desta figura.

Figura 6 : Titulação empírica do buffer de protease para otimizar a contagem exata dos mRNAs. Imagens representativas de SM-peixe de Pou5f1 em oócitos MII. DAPI coloração mostra cromossomos alinhados no eixo de MII. Oócitos foram incubados com não diluído (1:1), 01:12, 1:8 ou 1:4 diluições do buffer de Protease III. O número de mRNAs Pou5f1 (n = 2-3 ovócitos) foram contados e a média SEM é mostrado. Barra de escala é 10 µm. clique aqui para ver uma versão maior desta figura.

Figura 7 : Transferência de oócitos MII através de buffers de fixação, protease e hibridização em poços de uma placa de 6. A forma de cada poço é mostrada. (A) células flutuam quando primeiro adicionados aos buffers (B) submersão de oócitos em buffers. (C) oócitos resolver para o fundo do poço, durante o período de incubação. Clique aqui para ver uma versão maior desta figura.
Discussão
Uma série de pequenas etapas durante o protocolo irá garantir sucesso fluorescência e contagens exatas dos mRNAs. Em primeiro lugar, o protocolo deve ser realizado imediatamente após a coleta e fixação de oócitos. Observe que o PVP é adicionado para o buffer de fixação paraformaldeído 4% para evitar oócitos grudem uns aos outros. Nós achamos que é necessário realizar o experimento imediatamente após a coleta e fixação de oócitos. Qualquer atraso resulta em um muito menor sinal de fluorescência que resultaria em subcontagens de transcrições. Isto é devido em parte à degradação de RNA. Não mais que 20 oócitos devem ser transferidos para um bem na placa de 6 ao mesmo tempo e cada um bem deve ser usado apenas uma vez. Tempos de incubação também devem ser seguidos com precisão sem encurtamento ou alongamento de cada etapa. A exceção é as etapas de tampão de lavagem; ovócitos podem ser deixados no tampão de lavagem durante um tempo prolongado sem alterar os resultados experimentais. As sondas de SM-peixe estão disponíveis em três canais de fluorescência C1, C2 e C3. Para multiplexação, não misture sondas que têm a mesma marca do canal. Isso resultará em ambas as sondas que fluoresce na mesma análise de processamento de emissão de onda impossível, como não haverá nenhuma maneira de distinguir entre os conjuntos de sonda. Sondas de controle positivo projetadas contra genes de limpeza estão disponíveis em cada um dos três canais. Sondas de controlo negativo (por exemplo, DapB) também estão disponíveis em conjuntos pré-misturados que contêm uma marca para todos os três canais. O experimento deve ser realizado na penumbra, como oócitos são sensíveis à luz após a remoção do oviduto17,18. Após a adição da fluorophores anexado ao AMP4, as etapas devem ser executadas com como pouca luz possível evitar o branqueamento do fluoróforo. Finalmente, ao montar os oócitos em lâminas histológicas, coloque cuidadosamente a lamela para evitar distorção do oócito e a formação de bolhas que podem interferir com a imagem. Se você achar difícil evitar a distorção da célula, espaçadores de slide devem ser usados para manter a forma esférica do oócito.
Uma modificação essencial do protocolo é a substituição de permeabilização e buffers de lavagem fornecidos no kit disponível comercialmente. Os buffers de protease e hibridação proprietários fornecidos são ambientes hostis para os oócitos, mas são necessários para o sucesso do protocolo. Se não utilizado, os amplificadores são incapazes de entrar na célula, que é provavelmente devido à agregação de DNA em cadeia ramificada. Movimentação de oócitos destes ambientes ásperos para a permeabilização relativamente suave e buffers de lavagem, projetados por imunofluorescência14, provou ser adequado para o sucesso do protocolo e ao mesmo tempo impediu lise de oócitos. Porque oócitos e embriões pré-implantação não aderem a uma lâmina histológica, outra modificação essencial foi colocando os oócitos para os buffers de dentro de um poço de um prato de cultura. Usamos uma placa de cultura de embrião 6-poços. Cada poço da placa tem cônicos e lados inclinados e 8 mm plana inferior (Figura 7), que melhora a recuperação do oócito. Isto é particularmente importante como oócitos perdem suas propriedades refratárias e tornar-se quase transparentes nos buffers de hibridização.
Quando a transferência de oócitos de bem de bem, é importante assegurar que os oócitos são totalmente submersa nas soluções em cada poço como células flutuará quando primeiro transferido para cada poço (Figura 7A). Uma vez que elas são submergidas mecanicamente para o buffer (Figura 7B), eles vão afundar no fundo do poço até ao final da incubação (Figura 7). A exceção é AMP 1 e 3 AMP; Quando mecanicamente submersed do oócitos não completamente resolver para o fundo do poço. Para encontrar esses ovócitos, você pode precisar alterar o plano de foco. Pipetar cuidadosamente e contar o número de células, sendo transferido para evitar a perda.
Várias técnicas de peixe única molécula, que amplificam o sinal fluorescente ao invés de cDNA, incluindo químicos de DNA ramificados, têm sido desenvolveram 9,19. Kits de comercialmente disponíveis otimizaram o método de DNA SM-peixe ramificada de detecção pode ser reproduzido de mRNAs individuais em cortes de tecido ou células aderentes em uma lâmina histológica. O protocolo descrito aqui foi modificado para uso com células não-aderentes únicas (por exemplo, oócitos e embriões pré-implantação)3. Isto permite não só a quantificação específica e reprodutível, mas também a localização de um mRNA dentro do oócito. Enquanto esta é uma vantagem do ensaio, claro existem limitações. Por exemplo, ao contrário a sequenciação do ARN, ele não pode identificar mRNAs romance. Uma limitação adicional do protocolo é a disponibilidade de sondas específicas de transcrição. Sondas proprietárias são comercialmente disponíveis a partir das empresas que vendem os kits de SM-peixe. Existem vários testes que são pre-feitos. Outros podem ser projetados pela companhia para qualquer mRNA anotada usando um algoritmo objetiva10. No entanto, se um mRNA é sequenciado mal seria difícil projeto sondas com alta especificidade. Para transcrições curtas também pode ser difícil identificar suficiente pares de sonda que não reagem de forma cruzada com outras transcrições, reduzindo a especificidade do ensaio. Da mesma forma, um número menor de sonda de moda pode ser insuficiente para produzir fluorescência sinal acima do limiar de detecção como positiva no local encontrando e programa de rastreamento. Nesta mesma veia, variantes de transcrição não podem ser detectadas com este método.
Apesar das limitações descritas acima, existem várias aplicações para SM-peixe. Por exemplo, dados de célula única-a sequenciação do ARN poderiam ser validados especialmente quando o número de células é pequenas e difíceis de obter (por exemplo, oócitos e embriões). Amplificação do cDNA para ensaios PCR apresenta um erro experimental, que normalmente é reduzido a um passo de normalização usando os dados de genes estàvel expressa das tarefas domésticas. No entanto, mudanças temporais no oócito através de pré-implantação de embriões também altera a expressão dos genes das tarefas domésticas. O protocolo de SM-peixe amplifica a fluorescência em vez de cDNA. Portanto, não há nenhuma exigência para normalização dos níveis de mRNA de transcrição específicos para obter resultados reprodutíveis com baixa variabilidade. Devido à variabilidade da eficiência de primeira demão do PCR, diferenças do número absoluto de espécies diferentes de mRNA não podem ser com precisão comparadas dentro ou entre tipos de células. SM-peixe localiza e quantifica o mRNA. Portanto, ele pode ser usado para identificar quais células expressam mRNA em uma população de células mistas. Por exemplo, quando oócitos estão crescendo dentro de folículos primários ou secundários, o folículo pode ser isolado e cultivado em esferas de alginato20 mas a separação do oócito de células somáticas é difícil. Portanto, estudos PCR e sequenciamento foram realizados usando populações de células mistas. O uso de SM-peixe pode determinar se os mRNAs são detectados em células somáticas ou o oócito do folículo. Finalmente, SM-peixe tem alta sensibilidade e especificidade, permitindo a deteção de transcrições de baixa abundância; por exemplo, detecção de Nanog em oócitos MII (Figura 5).
Armazenamento e degradação dos mRNAs são importantes mecanismos de regulação na expressão de proteínas. Regulação pós-transcricional de tradução, armazenamento e degradação são mediadas por proteínas que se ligam a mRNAs21. Atualmente, imunoprecipitação de RNA-proteína (RIP) pode ser rotineiramente executada quando um grande número de células está disponíveis22. Devido ao grande número de ovos de Xenopus que podem ser coletados a partir de um único animal, RIP foi executada com êxito neste modelo animal. No entanto, é difícil obter suficiente mamíferos oócitos e embriões pré-implantação para executar RIP. Acoplamento de SM-peixe e imunofluorescência (immunoFISH) 23 de cortes histologicos prendem o potencial para visualizar proteínas associadas mRNAs específicos incluindo translacional maquinaria24,25. Genómica mede variantes genéticas (por exemplo, polimorfismos de nucleotídeo pequeno, SNPs) associadas com saúde e doença,26. Phenomics identifica alterações em respostas celulares devido a pressões ambientais27,28. A pesquisa atual tem como objectivo encontrar o mecanismo que conecta as alterações no genoma com fenótipos específicos. O uso de immunoFISH tem o potencial de link SNP-dependente alterações na expressão do mRNA e a expressão de proteínas que contribuem para celulares fenótipos. Como a tecnologia evolui, provavelmente há outras aplicações de SM-peixes que irão identificar mecanismos importantes em vários sistemas biológicos.
Divulgações
Os autores não têm nada a declarar
Agradecimentos
Agradecemos o Dr. Daniel R. Larson por sua generosa ajuda com a instalação e utilização do local encontrando e programa de rastreamento 13 e o apoio técnico da Universidade de Nebraska Lincoln microscopia núcleo para a imagem de microscopia confocal. Este estudo representa uma contribuição da Universidade da divisão de pesquisa agrícola de Nebraska, Lincoln, Nebraska e foi apoiado por fundos de hachura UNL (NEB-26-206/adesão número-232435 e NEB-26-231/adesão número-1013511).
Materiais
| Name | Company | Catalog Number | Comments |
| (±)-α-Lipoic acid | Sigma-Aldrich | T1395 | Alpha Lipoic Acid |
| Albumin, Bovine Serum, Low Fatty Acid | MP Biomedicals, LLC | 199899 | FAF BSA |
| BD 10mL TB Syringe | Becton, Dickinson and Company | 309659 | 10 mL syringe |
| BD PrecisionGlide Needle | Becton, Dickinson and Company | 305109 | 27 1/2 gauge needle |
| Calcium chloride dihydrate | Sigma-Aldrich | C7902 | CaCl2-2H2O |
| Citric acid | Sigma-Aldrich | C2404 | Citrate |
| D-(+)-Glucose | Sigma-Aldrich | G6152 | Glucose |
| Disodium phosphate | Na2HPO4 | ||
| Easy Grip Petri Dish | Falcon Corning | 351008 | 35 mm dish |
| Edetate Disodium | Avantor | 8994-01 | EDTA |
| Extra Fine Bonn Scissors | Fine Science Tools | 14084-08 | Straight, Sharp/Sharp, non-serrated, 13mm cutting edge scissors |
| Fetal Bovine Serum | Atlanta biologicals | S10250 | FBS |
| Gentamicin Reagent Solution | gibco | 15710-064 | Gentamicin |
| GlutaMAX-I (100X) | gibco | 35050-061 | Glutamax |
| Gold Seal Micro Slides | Gold Seal | 3039 | 25 x 75mm slides |
| Gonadotropin, From Pregnant Mares' Serum | Sigma | G4877 | eCG |
| hCG recombinant | NHPP | AFP8456A | hCG |
| Hyaluronidase, Type IV-S: From Bovine Testes | Sigma-Aldrich | H3884 | Hyaluronidase |
| Jewelers Style Forceps | Integra | 17-305X | Forceps 4-3/8", Style 5F, Straight, Micro Fine Jaw |
| L-(+)-Lactic Acid, free acid | MP Biomedicals, LLC | 190228 | L-Lactate |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | MgSO4-7H2O |
| MEM Nonessential Amino Acids | Corning | 25-025-Cl | NEAA |
| Microscope Cover Glass | Fisher Scientific | 12-542-C | 25 x 25x 0.15 mm cover slips |
| Mm-Nanog-O2-C2 RNAscope Probe | Advanced Cell Diagnostics | 501891-C2 | Nanog Probe |
| Mm-Pou5f1-O1-C3 RNAscope Probe | Advanced Cell Diagnostics | 501611-C3 | Pou5f1 Probe |
| MOPS | Sigma-Aldrich | M3183 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Paraformaldehyde |
| PES 0.22 um Membrane -sterile | Millex-GP | SLGP033RS | 0.22 um filters |
| Polyvinylpyrrolidone | Sigma-Aldrich | P0930 | PVP |
| Potassium chloride | Sigma-Aldrich | 60128 | KCl |
| Potassium phosphate monobasic | Sigma-Aldrich | 60218 | KH2PO4 |
| Prolong Gold antifade reagent | invitrogen | P36934 | Antifade reagent without DAPI |
| RNAscope DAPI | Advanced Cell Diagnostics | 320858 | DAPI |
| RNAscope FL AMP 1 | Advanced Cell Diagnostics | 320852 | Amplifier 1 |
| RNAscope FL AMP 2 | Advanced Cell Diagnostics | 320853 | Amplifier 2 |
| RNAscope FL AMP 3 | Advanced Cell Diagnostics | 320854 | Amplifier 3 |
| RNAscope FL AMP 4 ALT A | Advanced Cell Diagnostics | 320855 | Amplifier 4 ALT A |
| RNAscope FL AMP 4 ALT B | Advanced Cell Diagnostics | 320856 | Amplifier 4 ALT B |
| RNAscope FL AMP 4 ALT C | Advanced Cell Diagnostics | 320857 | Amplifier 4 ALT C |
| RNAscope Fluorescent Multiplex Detection Reagents Kit | Advanced Cell Diagnostics | 320851 | FISH Reagent Kit |
| RNAscope Probe 3-plex Negative Control Probe | Advanced Cell Diagnostics | 320871 | Negative Control |
| RNAscope Probe 3-plex Positive Control | Advanced Cell Diagnostics | 320881 | Positive Control |
| RNAscope Probe Diluent | Advanced Cell Diagnostics | 300041 | Probe Diluent |
| RNAscope Protease III | Advanced Cell Diagnositics | 322337 | Protease III |
| RNAscope Protease III & IV Reagent Kit | Advanced Cell Diagnostics | 322340 | FISH Protease Kit |
| RNAscope Protease IV | Advanced Cell Diagnostics | 322336 | Protease IV |
| S/S Needle with Luer Hub 30G | Component Supply Co. | NE-301PL-50 | blunt 30 gauge needle |
| Sodium bicarbonate | Sigma-Aldrich | S6297 | NaHCO3 |
| Sodium chloride | Sigma-Aldrich | S6191 | NaCl |
| Sodium hydroxide | Sigma-Aldrich | 306576 | NaOH |
| Sodium pyruvate, >= 99% | Sigma-Aldrich | P5280 | Pyruvate |
| Solution 6 Well Dish | Agtechinc | D18 | 6 well dish |
| Taurine | Sigma-Aldrich | T8691 | Taurine |
| Tissue Culture Dish | Falcon Corning | 353002 | 60 mm dish |
| Triton X-100 | Sigma-Aldrich | X100 | Triton X-100 |
Referências
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proceedings of the National Academy of Sciences. 96 (16), 9236-9241 (1999).
- MacK, E. M., Smith, J. E., Kurz, S. G., Wood, J. R. CAMP-dependent regulation of ovulatory response genes is amplified by IGF1 due to synergistic effects on Akt phosphorylation and NF-kB transcription factors. Reproduction. 144 (5), 595-602 (2012).
- Xie, F., Timme, K. A., Wood, J. R. Using Single Molecule mRNA Fluorescent in Situ Hybridization (RNA-FISH) to Quantify mRNAs in Individual Murine Oocytes and Embryos. Scientific Reports. 8 (1), 7930 (2018).
- Ruebel, M. L., et al. Obesity modulates inflammation and lipidmetabolism oocyte gene expression: A single-cell transcriptome perspective. Journal of Clinical Endocrinology and Metabolism. 102 (6), 2029-2038 (2017).
- Borensztein, M., Syx, L., Servant, N., Heard, E. . Mouse Oocyte Development. 1818, 51-65 (2018).
- Jansova, D., Tetkova, A., Koncicka, M., Kubelka, M., Susor, A. Localization of RNA and translation in the mammalian oocyte and embryo. PLoS ONE. 13 (3), 1-25 (2018).
- Player, A. N., Shen, L. P., Kenny, D., Antao, V. P., Kolberg, J. A. Single-copy gene detection using branched DNA (bDNA) in situ hybridization. Journal of Histochemistry and Cytochemistry. 49 (5), 603-611 (2001).
- Wang, F., et al. RNAscope: A novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Itzkovitz, S., van Oudenaarden, A. Validating transcripts with probes and imaging technology. Nature Methods. 8 (4), S12-S19 (2011).
- Derti, A., et al. ProbeDesigner: for the design of probesets for branched DNA (bDNA) signal amplification assays. Bioinformatics. 15 (5), 348-355 (1999).
- Larson, B., Malayter, D., Shure, M. Multiplexed Detection of Cytokine Cancer Biomarkers using Fluorescence RNA In Situ Hybridization and Cellular Imaging. BioTek Application Notes. , 1-5 (2016).
- Buxbaum, A. R., Wu, B., Singer, R. H. Single β-Actin mRNA Detection in Neurons Reveals a Mechanism for Regulating Its Translatability. Science. 343 (6169), 419-422 (2014).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Herrick, J. R., Paik, T., Strauss, K. J., Schoolcraft, W. B., Krisher, R. L. Building a better mouse embryo assay: effects of mouse strain and in vitro maturation on sensitivity to contaminants of the culture environment. Journal of Assisted Reproduction and Genetics. 33 (2), 237-245 (2016).
- Pohlmeier, W. E., Xie, F., Kurz, S. G., Lu, N., Wood, J. R. Progressive obesity alters the steroidogenic response to ovulatory stimulation and increases the abundance of mRNAs stored in the ovulated oocyte. Molecular Reproduction and Development. 81 (8), 735-747 (2014).
- Xie, F., Anderson, C. L., Timme, K. R., Kurz, S. G., Fernando, S. C., Wood, J. R. Obesity-dependent increases in oocyte mRNAs are associated with increases in proinflammatory signaling and gut microbial abundance of lachnospiraceae in female mice. Endocrinology. 157 (4), 1630-1643 (2016).
- Hirao, Y., Yanagimachi, R. Detrimental effect of visible light on meiosis of mammalian eggs in vitro. Journal of Experimental Zoology. , (1978).
- Takenaka, M., Horiuchi, T., Yanagimachi, R. Effects of light on development of mammalian zygotes. Proceedings of the National Academy of Sciences. 104 (36), 14293-14293 (2007).
- Komminoth, P., Werner, M. Target and signal amplification: Approaches to increase the sensitivity of in situ hybridization. Histochemistry and Cell Biology. 108 (4-5), 325-333 (1997).
- Hornick, J. E., Duncan, F. E., Shea, L. D., Woodruff, T. K. Multiple follicle culture supports primary follicle growth through paracrine-acting signals. Reproduction. 145 (1), 19-32 (2013).
- Vlasova-St. Louis, I., Bohjanen, P. Feedback Regulation of Kinase Signaling Pathways by AREs and GREs. Cells. 5 (1), 4 (2016).
- Gilbert, C., Svejstrup, J. Q. RNA Immunoprecipitation for Determining RNA-Protein Associations In Vivo. Current Protocols in Molecular Biology. 75 (1), 27.4.1-27.4.11 (2006).
- Kwon, S., Chin, K., Nederlof, M., Gray, J. W. Quantitative, in situ analysis of mRNAs and proteins with subcellular resolution. Scientific Reports. 7 (1), 16459 (2017).
- Voigt, F., et al. Single-Molecule Quantification of Translation-Dependent Association of mRNAs with the Endoplasmic Reticulum. Cell Reports. 21 (13), 3740-3753 (2017).
- Halstead, J. M., Wilbertz, J. H., Wippich, F., Lionnet, T., Ephrussi, A., Chao, J. A. TRICK: A Single-Molecule Method for Imaging the First Round of Translation in Living Cells and Animals. Methods in Enzymology. 572, (2016).
- Cookson, W., Liang, L., Abecasis, G., Moffatt, M., Lathrop, M. Mapping complex disease traits with global gene expression. Nature Reviews Genetics. 10 (3), 184-194 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: the next challenge. Nature Reviews Genetics. 11, 855 (2010).
- Freimer, N., Sabatti, C. The Human Phenome Project. Nature Genetics. 34, 15 (2003).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados