Method Article
マウス卵母細胞における定量化するため、Localize Mrna に単一分子蛍光そのままの交配 (SM 魚) の使用
要約
再現性をもって RNA の単一分子蛍光 in-situ 個々 卵母細胞における Mrna の数字をカウントするには、交配 (RNA 魚) 非付着性のセルの最適化を行った。卵子採取、トラン スクリプトの特定プローブとハイブリダイズし、画像定量化ソフトウェアを用いて定量化します。
要約
デジタルの逆転写ポリメラーゼ連鎖反応 (dPCR)、量的、リアルタイム RT-PCR (RT qPCR) RNA シーケンスなど日常的に卵子および胚の mRNA を定量化するために使用する現在の方法。これらの技術を実行するには、単一の卵や胚を使用して、低コピー Mrna は確実に検出されません。この問題を克服するために卵子や胚をプールできる一緒に分析。しかし、これは多くの場合サンプルの中で高い可変性に します。このプロトコルでは、蛍光 in situ ハイブリダイゼーション (魚) 分岐 DNA 化学を使用しての使用を記述します。この手法は、個々 の細胞における Mrna の空間パターンを識別します。スポットを見つけると追跡コンピューター ソフトウェア技術を結合すると、細胞における Mrna の豊かさも定量化することができます。実験グループ内で減少の変動は, この手法を使用して、および少数の卵子および胚は実験群間に有意差を検出するために必要な。市販の分岐 DNA SM-魚キットは、断面化された組織またはスライド上の付着性のセルの Mrna を検出する最適化されています。しかし、卵子がスライドに効果的に準拠していない、いくつかの試薬キットであった卵母細胞の換散の結果があまりにも厳しかった。この溶解を防ぐためには、魚のキットにいくつかの変更が行われました。具体的には、卵子および胚の蛍光用に設計された卵母細胞透過および洗浄バッファーでは、独自のバッファーが置き換えられます。6 ウェル プレートで透過、洗浄、およびプローブ増幅器と孵化を行ったし、メディアをマウントを使用するプロトコルの最後にスライドに卵が置かれました。卵母細胞の換散特に、市販キットの限界を克服するためには、これらの変更ができた。正確かつ再現性をもって個々 の卵母細胞における Mrna の数をカウントする、コンピューターのソフトウェアを使用しました。一緒に、このプロトコルは PCR とシーケンスは、単一セルの特定のコピーの式を比較する方法を表します。
概要
逆転写酵素ポリメラーゼ連鎖反応 (PCR) mRNA 定量の金の標準となっています。2 つの試金、デジタル PCR (dPCR)1および定量的なリアル タイム PCR (qPCR)2現在使用されます。2 つの PCR 技術の dPCR は qPCR 示唆それは単一細胞における mRNA 量測定に使用することができるよりも高い感度です。しかし、私たちの手で各実験サンプルあたり 5 に 10 卵母細胞のプールで低豊富 dPCR 解析は低再現性と高いバリエーション3データを生産しています。これは RNA の抽出および逆のトランスクリプションの効率性に関連する実験的なエラー可能性があります。RNA シーケンスも単一のマウスと人間の卵子4,5を使用して行われています。この手法には、ライブラリを生成可能性が実験群内変動を増加させるために必要な cDNA 増幅手順が必要です。さらに、低豊富な成績証明書を検出できない場合があります。シーケンスの価格は、ここ数年ダウンしている、バイオインフォマティクス解析のコストが高いため法外なコストすることができます。最後に、mRNA の局在は、タンパク質機能6に貢献して空間的変化とダイナミックなプロセスです。そのため、正確かつ再現可能な量的および単一卵母細胞個々 の mRNAs のローカリゼーションを生成する手法を採用に着手しました。
蛍光 in situ ハイブリダイゼーションと結合した分岐 DNA 増幅蛍光信号ではなく、個々 のセル7,8,9の単一の Mrna の増幅の RNA/cDNA 有効に検出します。アッセイは、一連のハイブリダイゼーション、増幅 (分岐 DNA を使用)、および蛍光蛍光信号7を増幅するための手順によって行われます。技術は、特定の mRNA3,8,10を補足 18-25 ベース オリゴヌクレオチド プローブ ペアの結合に始まります。15 ~ 20 プローブ ペアはターゲット トラン スクリプトの各トラン スクリプトの特異性確保のために設計されています。MRNA 固有の交配には分岐の構成を形成する前置増幅器と増幅器のプローブが続きます。約、400 ラベル fluorophores が付いては、各増幅器、蛍光は、個々 の mRNAs (図 1)11の検出の 8000-fold 増加の結果にバインドします。
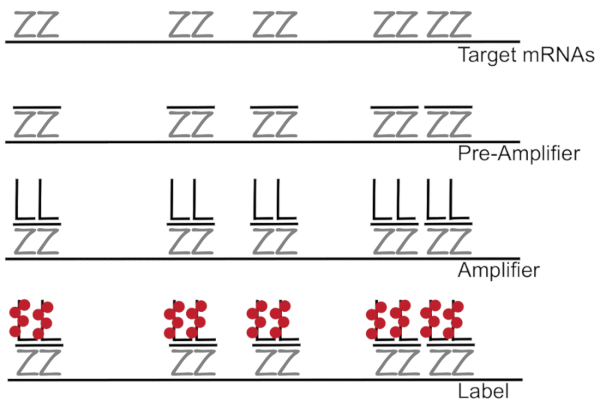
図 1: SM 魚のプロトコルの概略図。トラン スクリプト特定のプローブの連続交配が枝分かれしたは、DNA 増幅器、蛍光 mRNA を示すターゲットに。この図の拡大版を表示するのにはここをクリックしてください。
個々 のニューロン12及び子宮頚癌におけるひと乳頭腫ウイルス DNA にその場交配 (SM 魚) ローカライズされた β-アクチン Mrna の 1 分子蛍光を使った過去の研究は、ライン7 をセルします。ソフト スポットを見つけると追跡プログラム個々 の点状蛍光信号を識別し、正常に各セル3,13における Mrna の数を定量化するために使用されています。
ニューロン12の mRNA の検出の結果に基づき、我々 は SM 魚、マウス卵子および胚など低豊富な Mrna の転写レベルを量的に便利なツールを証明することを仮定しました。しかし、付着固定細胞用技術に最適化されて、ホルムアルデヒド固定パラフィン包が (FFPE) ティッシュ セクションを埋め込んだ。卵母細胞は、ポリ L リジン コーティングしている場合でも、スライドに従うことはできません。さらに、彼らは体細胞およびティッシュ セクションのセル換散市販キット3で独自のバッファーの一部を受けたときの結果より壊れやすい。これらの課題を克服するために卵母細胞は固定され手動でバッファーの滴間で転送されます。さらに、キットに透過し、洗浄バッファーは、セル換散を減らすために取り替えられました。あらかじめデザインされたプローブは魚キットと一緒にご購入または特定のコピーを要求することができます。独自のプローブ セットは、3 つの蛍光チャンネル (C1、C2、および C3) を可能にする多重化の一つで利用可能です。現在の実験では、マウス卵母細胞がデュアル染色と定量化された C2 Nanogプローブと C3 Pou5f1プローブを使います。これらのプローブは、卵子および胚のNanogおよびPou5f1の報告された式に基づいて選ばれました。交配の手順の最後に、卵母細胞は組織学的のスライド用耐フェード マウント メディアの滴に置かれました。共焦点画像は、個々 の mRNAs を表す点状蛍光信号の数を定量化するために使用されました。定量化、Mrna イメージングも、セルの特定の mRNA の分布を示した他の RNA 定量メソッドもない達成することができます。この手法は、実験群3の重要な違いを識別するために各実験群の卵母細胞の小さい数字の使用を許可する実験群内変動の少ないを持っている証明しました。
プロトコル
動物の手続き、審査し、承認機関動物ケアおよび使用委員会ネブラスカ大学リンカーン校と関連するガイドラインと規制に従ってすべての方法を行った。この研究のため CD 1 ザイモグラム マウス通常齧歯動物の食事、水にアドリブのアクセスしていた彼らは暗い、12:12 で維持された: 光のサイクル。
1. 必要なメディアの準備
- 基本メディア (OMM)、100 mL の滅菌水に 100 mM の NaCl、KCl 5 mM、0.5 mM KH2PO4、および 1.7 mM CaCl2-2 H2O を追加します。
注: OMM 媒体は 1 ヶ月保存できます。 - 完全なメディア (OMOPS)、追加 20 mM 3-morpholinopropane-1-スルホン酸 (モップ)、1.2 mM MgSO4-7 H2O、0.5 mM グルコース, L-乳酸 6 mM 1 mM, gln、タウリン、0.1 mM 1 x 非必須アミノ酸 (NEAA) 0.01 mM エチレンジアミン四酸 (EDTA)、10 μ M の α-リポ酸、10 μ g/mL 原液ゲンタマイシン、21 mM 1 M 水酸化ナトリウム、5 mM NaHCO3、0.2 mM ピルビン酸、クエン酸、0.5 mM を 1:10 に 4 mg/mL FAF BSA オム 100 mL の容量の滅菌水での希釈。0.22 μ M のフィルターで培地を滅菌します。
注: OMOPS は、1 週間まで保存できます。 - 開催中 (HM) 5% ウシ胎児血清を OMOPS に追加します。マウスあたり 2 mL HM を作る。
- ヒアルロニダーゼのソリューションは、0.1 mg/1 mL HM にウシ精巣から派生したヒアルロニダーゼの mL を追加します。
- 固定バッファーの 10 mL を 0.1% 胚グレード ポリビニルピロリドン (PVP)14と一緒に 1x PBS で 4% パラホルムアルデヒドを組み合わせます。
- 50 mL 洗浄バッファー (WB) を準備する追加 0.1% 非イオン性界面活性剤や 0.1 %1 × PBS14する PVP。
- 準備 10 mL 透過バッファーに 1 × PBS14に 1% 非イオン界面活性剤を追加します。
メモ: 上記洗浄と透過のバッファーは、市販のキットで独自のバッファーを交換します。
2. 雌マウス排卵卵子の収集
- 準備:
- 5 IU ひと絨毛性ゴナドトロピン (hCG) 44-48 h に続いて 5 IU 馬絨毛性ゴナドトロピン (eCG) 腹腔内 (IP) 投与による 5-8 週齢の雌マウスを刺激後15,16。
- 37 ° C の温暖化のプレートに HM の 2 mL を含む 35 mm のペトリ皿を維持します。1 つ、3 つ、60 mm のペトリ皿にヒアルロニダーゼなし HM 50 μ L 滴続いて希釈ヒアルロニダーゼを含む HM のドロップを 100 μ L のピペットします。37 ° C の温暖化を含む滴使用する前にプレート板を配置します。
注: ヒアルロニダーゼの滴は、蒸発とヒアルロニダーゼの有無に HM の成分濃度を防ぐために卵管の各ペアの郭清の直前に提示しなければなりません。
- マウス、hCG の IP 投与後 16 時間を安楽死させる、頚部転位続いてイソフルランの過剰摂取を使用します。
- 70% のエタノールを使用してマウスをクリーニングします。腹腔内を公開し、女性の生殖器官を視覚化します。鉗子で卵巣を押し子宮靭帯と卵巣から余分な脂肪を削除します。子宮から卵管をカットし、35 mm ディッシュで温かみのある HM で卵巣卵管のペアを配置します。
- 卵巣とその周囲の脂肪組織を削除します。1/2 インチ 27 ゲージ針を使用して卵管の腫れの乳頭を引き裂きます。涙のサイトで卵管を押し、卵丘細胞卵母細胞複合体 (Coc) が追放されます。中期口ピペット (図 2) を使用するヒアルロニダーゼと HM メディアを含む 100 μ L ドロップする減数分裂 II (MII) であると推定されている排卵卵子を転送します。

図 2: 卵の転送に使用口 pipettor の部分。(A)口ピース(B) 0.22 um、4 ミリメートル フィルター (C)吸引チューブ(D) 1000 μ L ピペット チップ(E) 9"パスツール ピペットで移しなさい。この図の拡大版を表示するのにはここをクリックしてください。
- 卵丘細胞を除去するため口ピペットと HM を含むヒアルロニダーゼで MII 卵子卵丘細胞複合体を上下にピペットします。各卵を転送、一度洗浄に卵丘細胞に欠けているドロップのみ口のピペットを使用して含んでいる HM。各洗浄液滴のこの手順を繰り返します。断片化または透明な卵15を転送しないでください。
注: として可能な限りほとんどの HM と 35 mm ディッシュの各ドロップから卵子を転送することが重要です。これはプロトコルに送金するたびに当てはまります。MII 卵子は、1 分以上の HM 媒体を含むヒアルロニダーゼで残す必要はありません。
3. SM 魚が卵母細胞の染色
- 500 μ L の固定バッファーを含む 6 ウェル プレートの個々 の井戸の卵母細胞を修正します。20 卵が水没または井戸の少ない。室温で 20 分間インキュベートします。
注: 各 SM 魚染色手順 6 も円錐形のプレートで個々 の井戸の内で発生します。卵母細胞がバッファーに完全に水没したと、バッファー上にない浮動でことを確認します。各ステップは、20 卵母細胞で行われた、または各ウェルで以下にする必要があります。 - 洗浄バッファー (WB 1.6 の手順で説明) の 500 μ L に固定の卵を転送で 10 分。2 回を繰り返します。
- 室温で 30 分間透過バッファーで卵を孵化させなさい。
注: 手順 1.7 で記述されている透過バッファー妥当性透過バッファーに置き換えられます。- プローブ セットを収集し、迅速にそれらを遠心機でスピンダウンします。各プローブの 40 ° C の水浴やインキュベーター 10 分セットを温めます。部屋の温度をクールします。
注: この手順は、透過潜伏中に実行する必要があります。
- プローブ セットを収集し、迅速にそれらを遠心機でスピンダウンします。各プローブの 40 ° C の水浴やインキュベーター 10 分セットを温めます。部屋の温度をクールします。
- 室温で 10 分間の WB の 500 μ L で卵母細胞を洗浄します。
- プロテアーゼ III のバッファー (キットから使用可能)、室温で 30 分間 1x PBS で希釈 1:8 の 80 μ L に卵子を転送します。
注: 80 μ L のボリュームは十分に 6 ウェル プレートで個々 の井戸の底をカバーしています。 - 室温で 10 分間の WB の 500 μ L で卵母細胞を洗浄します。
- Nanog、Pou5f1、 DapB (ネガティブ コントロール遺伝子) に温めたプローブ セットを希釈, 1:50 プローブ希釈液で。40 ° C で 2 時間インキュベートするトラン スクリプト固有のプローブの 80 μ L の卵母細胞
注: 独自のプローブ セット (C1、C2、および C3) 3 つの蛍光チャネルの 1 つで利用可能です。NanogおよびPou5f1プローブはそれぞれ C2 と C3 は、タグ付けされました。 - 独自のアンプ 1 (アンプ 1) アンプ 2 (AMP2) 増幅器 3 (AMP3) と常温 4-蛍光増幅器 (アンプ 4-フロリダ州) を温めます。
注: 2 時間トラン スクリプト固有プローブ潜伏中にこの手順を実行する必要があります。 - WB の 500 μ L に卵子を転送し、室温で 10 分間インキュベートします。
- 増幅バッファーの順番で卵を孵化させなさい。
- 30 分間の室温で 10 分間の WB の 500 μ L に 40 ° c. 転送卵子 AMP1 を 80 μ l 添加の卵を孵化させなさい。
- 40 ° C で 15 分間 AMP2 を 80 μ l 添加の卵を孵化させなさい卵を室温で 10 分間の WB の 500 μ L に転送します。
- 40 ° C で 30 分間 AMP3 を 80 μ l 添加の卵を孵化させなさい卵を室温で 10 分間の WB の 500 μ L に転送します。
メモ: アンプ FL 蛍光体が含まれるために、暗闇の中でプロトコルの残りの部分は実行されます。解剖顕微鏡の下で作業する場合は、可能な限り光を減らします。 - 40 ° C で 15 分間 AMP4 fl 80 μ L に卵を追加します。
注: AMP4 フロリダとして提供されます代替バッファー A (alt キー-A)、Alt B または Alt Cにおいて, 発光の波長が必要な依存 AMP4 フロリダ バッファーを選択します。
- 室温で 10 分間の WB の 500 μ L で卵母細胞を洗浄します。室温で 20 分間 DAPI の 80 μ L で卵を孵化させなさい。室温で 5 分間 WB の 500 μ L で卵母細胞を洗浄します。
- 試薬に泡を追加することがなくスライドの中央にフェージング マウント メディアの 12 μ L をピペットします。メディアをマウントにできるだけ小さな WB として卵を転送し、観察を適用します。
- 傾斜角度で coverslip とゆっくりと静かにスライドに液体かぶせます。卵母細胞と気泡の導入の歪みを防ぐためにあまりにもハード、coverslip を押すことは避けてください。
- 室温で一晩乾燥して暗いボックスで、スライドを保存します。Coverslip を密封するための明確なマニキュアのスライドのエッジをコートします。
- 標準的な顕微鏡を使用して、永続的なマーカー スライドやサークルの卵を見つけます。
注: この手順は必須ではありませんが、スライドの位置卵母細胞を改善します。最高の結果を得るには、衰退する蛍光信号として 1 〜 5 日内のイメージのスライドが始まります。
4. 画像処理
- Z ステップ共焦点顕微鏡を用いた 3 次元の卵を画像します。
注: イメージを的確に分析、各 z ステップは 1.0 μ m/スライスをする必要があります。 - 圧縮 nd2 または個々 の共焦点画像を保存します。各卵子の TIFF ファイルです。両方のイメージの種類は、オープン ソースの画像処理プログラム、フィジーと互換性が。
- ダウンロードし、オープン アクセス フィジー ソフトウェア (https://imagej.net/Fiji/Downloads) をインストールします。
- フィジーに nd2 ファイルをドラッグし、 hyperstackを選択します。場合は共焦点画像として保存されています。TIFF ファイルは、4.4 のステップに進みます。
注: フィジーに nd2 ファイルをドロップするとき hyperstack ドロップダウンが表示されます自動的に。 - [イメージ] タブをクリックして、色を選択し、nd2 ファイルの蛍光のチャンネルを分離するチャンネルの分割] をクリックします。
- 個々 を生成します。各蛍光チャネルにおける卵母細胞のそれぞれの z スライスの TIFF ファイルです。[イメージ] タブをクリックし、スタック画像にスタックをクリックします。[イメージ] タブをクリックして、種類を選択し、 Rgb各 z スライスを個々 の RGB カラー画像に変換する] をクリックします。
注: RGB カラーは人工に選ばれる各発光波長の希望することができます。 - 各変換された画像を保存します。TIFF ファイルです。ステッチ (ステップ 4.3) の間に混乱を避けるために新しいフォルダーに蛍光チャネルごとに 1 つの卵からイメージを配置します。
- フィジーに nd2 ファイルをドラッグし、 hyperstackを選択します。場合は共焦点画像として保存されています。TIFF ファイルは、4.4 のステップに進みます。
- それぞれを正規化します。TIFF イメージPou5f1のNanog陰性対照画像 (DapB) を用いたします。
メモ: 写真編集プログラムを使用して、正規化は実行されます。各コントロールのイメージから背景の蛍光性の同じレベルを削除することを確認します。 - 開いている各を正規化します。各波長における各卵子のすべての z スライスを一緒にステッチするフィジーの TIFF ファイルです。
- プラグインタブをクリックして、ステッチを選択し、グリッド/コレクション(図 3 a) をクリックします。ドロップダウン ・ メニューとOK をクリックして(図 3 b) から連続する画像を選択します。
- ディレクトリを参照し、すべての 1 つの波長で個々 の卵の z スライス画像を含むフォルダーを選択 (ステップ 4.3.4 参照)。[Ok]をクリックします。
- ステッチの画像の下にあるスライダーを使用する波長に対して適切な色チャネルに移動し画像の種類を選択をクリックして最終的な RGB ステッチ イメージを作成し、 RGB 色をクリックします。
注: このイメージは、蛍光定量ステップ 4.6 以下に記載されているため使用されます。
- ステッチの画像を 32 ビットの最大投影図に変換します。画像をクリックして、タイプを選択し、 32 ビット(図 3) をクリックします。新しいとしてこのイメージを保存します。TIFF ファイルです。
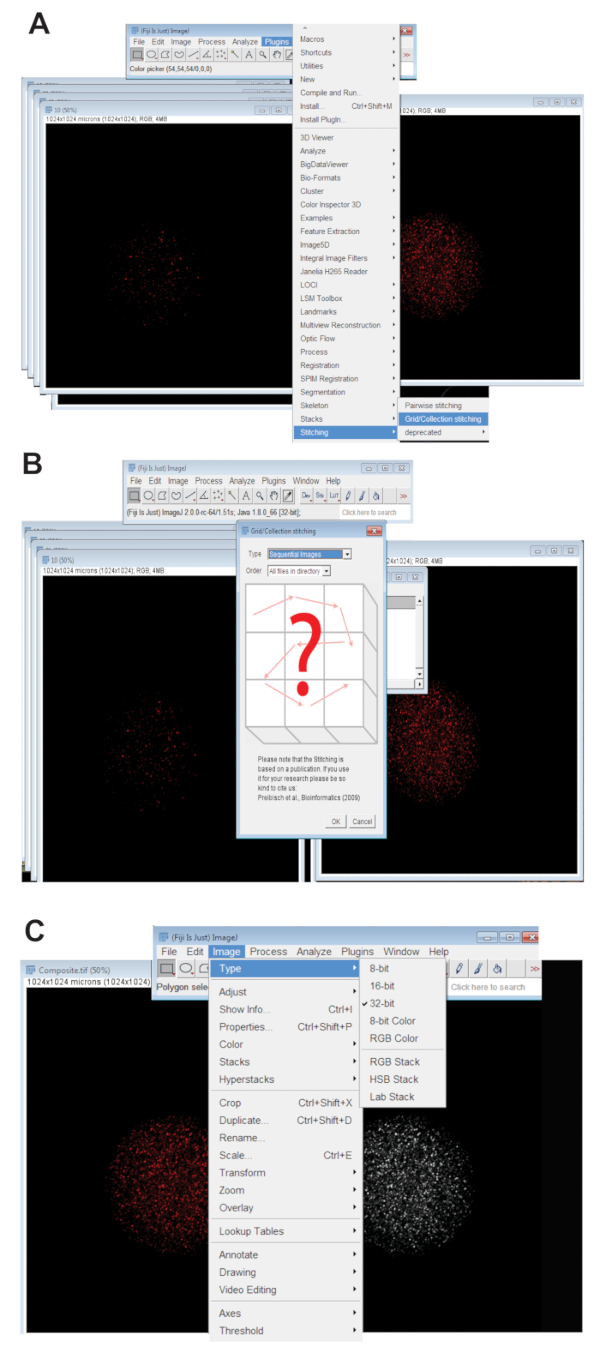
図 3: 卵母細胞の共焦点 z シリーズ画像のつなぎ合わせ。(A)卵母細胞の合成画像を生成する使用されたフィジーでプラグイン グリッド/コレクション ツールを示すスクリーン ショット。(B)連続画像蛍光重なり順序を使用します。TIFF ファイル合成画像を生成します。(C)合成画像は、32 ビットとして保存されました。TIFF ファイルです。この図の拡大版を表示するのにはここをクリックしてください。
- ダウンロードし、インストール、スポットを見つけると追跡プログラム13、d. r. ラーソン、国立機関の健康国立がん研究所 (https://ccr.cancer.gov/Laboratory-of-Receptor-Biology-and-Gene-Expression/daniel-r-larson) で探偵のウェブサイトから入手可能であります。ダウンロードして、スポットを見つけると追跡プログラム (http://www.spacewx.com/pdf/idlvm.pdf) を実行するために必要な対話型のデータ言語 (IDL) オペレーティング システムのオープン アクセスの仮想マシンをインストールします。
- スポットを見つけると追跡プログラムで 4.6 (図 4 a) の手順で生成された 32 ビット、ステッチの画像を開きます。Localize ドロップダウンを選択し、 Localize (図 4 b) イメージの数のスポットを計算するをクリックします。
注: カウント各スポットは、個々 の mRNA を表します。バンド パスと光子のしきい値の設定は、(図 4 b) のスクリーン ショットで表示されます。このプロトコルの各閾値設定の既定値が使用されました。肯定的な代表および背景スポット (図 4) を示しています。

図 4: スポット ・ ファインダーを用いた Mrna の定量化と追跡。(A)個々 の z シリーズ画像が図 3で説明したように一緒にステッチし、投影される 32 ビットの最大値として保存します。TIFF ファイルです。(B)合成画像をスポット検索と追跡に開設ローカライズする輝点 (赤い箱) をカウントする使用されました。バンドのパスと光子のしきい値は、青色のボックスで示されます。(C)青の矢印が指す (しきい値) 肯定的な信号。白い矢印は、しきい値を下回るスポット蛍光を示しています、したがって、カウントされません。この図の拡大版を表示するのにはここをクリックしてください。
結果
プロトコルの完了時に結果になります(図 4 aおよび図 5)、ステッチの画像(図 4)、共焦点の z シリーズから個々 の画像と mRNA カウント(図 4 b).多重化が実行されると、2 つの異なる Mrna (図 5)のラベルを示すイメージをマージがまたあるありますが。MRNA のカウントがフィジー (図 3)によって生成されたステッチの画像と点状蛍光スポット スポットを見つけると追跡プログラム(図 4 b)を使用してカウントを使用して生成されます。
MRNA カウントはその後、標準のデータ分析ツールを使用して分析します。このプロトコルでは n にレッテルを貼ったPou5f1とNanog12 卵母細胞を =。各 mRNA の結果の平均値し、平均値の標準誤差を計算します。MII 卵母細胞 (図 5) 775 ± 26 SEM Pou5f1成績証明書及び 113 ± 5 SEM Nanog成績を示したこのプロトコルでデータを収集しました。スチューデントの t 検定は、mRNA の統計的に有意な違いはPou5f1とNanogの間カウントを決定しました。重要なは、n で検出されたスポットはありませんでした DapB プローブ (すなわち、ネガティブ コントロール) で示されている 5 の卵を =。ちょうど 12 の個々 の卵子を使用して小さな標準エラーに注意してください。Nanog(図 5).の肯定的な再現性のある検出アッセイの感度を強調しても以前の dPCR 実験再現性をもって個々 の卵母細胞における Mrna はNanogの数が dPCR 3を使用してしきい値検出以下ことを示す Nanog が検出されませんでした。
卵母細胞の固定と SM 魚のプロトコルの開始の遅延があった場合パイロット実験では、減らされた蛍光を検出しました。同様に、独自の交配バッファーを使用しない分岐 DNA は、細胞内蛍光を入力しないプローブと増幅環状卵母細胞の細胞膜のまわり。これは、分岐 DNA の凝集による可能性が高いです。卵膜の周りの環状の蛍光は、形質膜の貧しい透過がある場合にもなります。Mrna に結合蛋白質の最適な劣化も必要です。SM 魚キットで提供されるプロテアーゼ バッファーは、切片と付着細胞の治療の最適な濃度です。ただし、非付着性のセルを使用している場合、経験的ベスト プロテアーゼ希釈を識別するために重要です。貧弱な mRNA へのプローブのための Mrna の社格プロテアーゼ バッファーが少なすぎる可能性があります。同様に、あまりにも多くのプロテアーゼのでだけでなくタンパク質 mRNA にバインドの劣化原因は、Mrna の不安定化も。このプロトコルでは、我々 は 1x PBS で原液 (1:1)、1:12、1:8、1:4 希釈プロテアーゼ バッファーの滴定曲線を用いた mRNA 検出テスト (n = 希釈あたり 2 に 3 の卵母細胞)。平均は、176 ± 36 SEM (1:4)、308 ± 18 SEM (1:8)、および 445 の ± 24 SEM (1:12) (図 6) を MII 卵子Pou5f1 mRNA の蛍光発現された 169 ± 42 SEM (原液) です。このプロトコルで使われるプロテアーゼ希釈だった最も低い変動を示したそれは 1:8。

図 5: Pou5f1 と MII 卵子における mRNA。(A).中間 z シリーズ画像の代表的なイメージが左側に表示されます。赤でPou5f1が検出された (647 nm) Nanogは緑検出波長 (488 nm) 波長。DAPI 染色染色体 MII 卵子の特徴的な中期 II のスピンドル上に配置が白で表示されます。DapBいずれかので、647 染色がなかった nm または発光波長 488 nm。(B) SEM を意味Pou5f1 、 Nanog Mrna の数が表示されます (n = 12 卵母細胞);DapBの検出 (N.D) はありませんでした。* P を示します < 0.05、スケール バーは 10 μ m ですこの図の拡大版を表示するにはここをクリックしてください。 。

図 6: Mrna の正確なカウントを最適化するためにプロテアーゼ バッファーの経験的な滴定します。MII 卵子Pou5f1の SM 魚の代表的なイメージ。DAPI 染色染色体 MII スピンドルに配置されますを示しています。卵母細胞プロテアーゼ III バッファーの原液 (1:1)、1:12、1:8、1:4 希釈で培養。Pou5f1 Mrna の数 (n = 2-3 卵母細胞) 数えられたと SEM を示す意味。スケール バーは、10 μ mこの図の拡大版を表示するにはここをクリックしてください。 。

図 7: MII 卵子 6 ウェル プレートの井戸で固定、プロテアーゼ、および交配バッファーを介して転送します。各ウェルの図形が表示されます。(A)セル フロートまずバッファーにバッファー内の卵母細胞(B)水没を追加。(C)卵母細胞は潜伏期間中に井戸の底に解決します。この図の拡大版を表示するのにはここをクリックしてください。
ディスカッション
一連のプロトコル間のマイナーなステップは、成功した蛍光・ Mrna の正確な数を確保します。まず、プロトコルはコレクションおよび卵子の固定後すぐに行う必要があります。PVP が卵母細胞が互いに付着するを防ぐために 4% パラホルムアルデヒド固定バッファーに追加されることに注意してください。コレクションおよび卵子の固定後すぐに実験を行うことが必要だとわかった。任意の遅延は、大いに低い蛍光シグナルの成績証明書の社格になるの結果します。これは RNA の劣化部分で予定です。以上 20 卵母細胞に転勤させる 6 ウェル プレートで 1 つも一度に、それぞれよくする必要があります一度だけ使用します。インキュベーション時間には、各ステップの延長または短縮することがなく正確に従ってください。例外は、洗浄バッファーの手順;卵母細胞は、実験の結果を変更することがなく長時間洗浄バッファーの左することができます。3 つの蛍光チャネル C1、C2、および C3 の SM 魚プローブがあります。多重化のためにチャネルの同じタグを持つプローブを混在させないでください。これは不可能な同じ発光波長のレンダリング解析に蛍光を発するプローブ セットを区別する方法はないだろう両方のプローブになります。ハウスキーピング遺伝子に対してポジティブ コントロール プローブは、3 つのチャネルのそれぞれにあります。ネガティブ コントロール プローブ (例えば、 DapB) は、タグに指定されたすべての 3 つのチャンネルが含まれる予混合のセットでご利用頂けます。実験は、卵母細胞は、光感受性の卵管17,18から削除された後に薄暗い照明で行われる必要があります。AMP4 に接続されている蛍光物質の添加後は、手順と蛍光体の白化を防ぐために可能な限り小さな光として実行必要があります。最後に、卵母細胞を病理組織学的のスライドをマウントするとき慎重にイメージングを妨げることができる泡の形成と卵母細胞の歪みを防ぐために coverslip を配置します。セル歪みを回避することは困難が見つかった場合は、卵母細胞の球形の形を維持するためにスライド スペーサーを使用する必要があります。
プロトコルの重要な変更の 1 つは、透過や市販のキットで提供される種類の洗浄バッファーの交換です。提供される独自のプロテアーゼおよび交配のバッファーは卵母細胞の過酷な環境ですが、プロトコルの成功に必要な。使用しない場合、アンプは分岐 DNA の凝集が原因と思われる細胞を入力することができます。卵母細胞の換散を防ぐこれらの過酷な環境から卵母細胞を比較的軽度の透過と蛍光抗体法14プロトコルのと同時に、成功のための十分な証明用の洗浄バッファーに移動します。卵子と初期胚組織学的のスライドに準拠していない、ために、別の重要な変更は培養皿のウェル内でバッファーに卵子を配置されました。6 ウェル胚培養プレートを使いました。プレートの各ウェルにテーパているが、側面の傾斜とフラット 8 mm 下(図 7)、卵母細胞の回復を改善します。これは、卵母細胞は、その耐火性の特性を失うし、交配バッファーでほぼ透明になると特に重要です。
井戸から井戸に卵子を転送するとき (図 7 a) 各ウェルに最初に転送されるときに細胞がフロートとして各ウェル内のソリューションに完全に、卵母細胞沈んでいることを確認することが重要です。彼らは機械的に (図 7 b) バッファーに沈水、一度彼らはインキュベーション (図 7) の終わりによって井戸の底にシンクされます。例外は、アンプ 1 とアンプ 3;機械的に水没して卵母細胞は完全ではないときは、井戸の底に解決します。これらの卵を見つけることの焦点面を変更する必要があります。ピペットの慎重に、損失を防ぐために転送されているセルの数をカウントします。
分岐 DNA 化学を含む cDNA がされているのではなく、蛍光信号を増幅する、複数の単一分子魚技術の開発9,19。市販のキットには、分岐 DNA SM 魚ティッシュ セクションまたは組織学的のスライドの付着性のセルの個々 の mRNAs の再現性のある検出法を最適化しています。ここで説明されているプロトコルは、単一の非付着性のセル (例えば、卵子と初期胚)3で使用するために変更されています。これにより、具体的かつ再現可能な数量だけでなく、卵母細胞内 mRNA の局在化。これは、アッセイの利点は、もちろん制限があります。たとえば、RNA 配列とは異なり新規 Mrna を識別できません。プロトコルの追加の制限は、トラン スクリプト固有のプローブの可用性です。独自のプローブが SM 魚キットを販売している会社から購入できます。あらかじめ作られていますいくつかのプローブがあります。他の人に客観的アルゴリズム10を使用して任意の注釈付きの mRNA の会社によって設計できます。ただし場合、mRNA はシーケンシャル悪い高い特異性を持つプローブをデザインすることは困難でしょう。短い転写産物のアッセイの特異性を減らす他の成績とは交差反応しない十分なプローブ ペアを識別することは困難できます。同様に、プローブ セットの小さい数のしきい値を超える陽性スポットを見つけると追跡プログラムで検出のための蛍光信号を生成するための十分なあります。これと同じ調子で転写バリアントはこのメソッドを検出できません。
上記の制限にもかかわらず SM 魚のいくつかのアプリケーションがあります。セル番号が小さく、入手が困難な場合は特にたとえば、単一細胞 RNA 塩基配列解読からデータが検証でした (例えば、卵子および胚)。PCR の試金のための cDNA の拡大は、通常安定発現ハウスキーピング遺伝子からのデータを使用して、正規化ステップによって減らされる実験的エラーを紹介します。ただし、着床前胚を介して卵母細胞の経時変化もハウスキーピング遺伝子の表現を変更します。SM 魚のプロトコルは cDNA ではなく蛍光を増幅します。したがって、変動の少ない再現性のある結果を得るにトラン スクリプト固有の mRNA のレベルの正規化の必要はありません。PCR プライマー効率の変動による mRNA 種の絶対数の違い正確に比較できません内または細胞のタイプ間。SM 魚をローカライズし、mRNA を定量化します。したがって、セル混合セル人口での mRNA の表現を識別するために使用することができます。たとえば、卵胞を分離およびアルギン酸ビーズ20で培養できるが、プライマリまたはセカンダリの卵胞内卵母細胞が成長しているときが体細胞から卵母細胞の分離は困難であります。したがって、シーケンスおよび PCR の研究は、混合セル人口を使用して行われています。SM 魚の使用は、体細胞や卵胞の卵母細胞で Mrna が検出されたかどうかを判断できます。最後に、SM 魚は高い感度と特異性の低い豊富なトラン スクリプトの検出を可能にします。たとえば、 Nanogの検出は、MII 卵母細胞(図 5)。
ストレージおよび Mrna の分解、蛋白質の表現の重要な調節機構。翻訳、ストレージ、および劣化の転写後調節は Mrna21に結合する蛋白質によって仲介されます。現在、多数のセルが利用可能な22RNA 蛋白質免疫沈降 (RIP) が日常的に実行します。1 つの動物から収集することができますアフリカツメガエル卵の数が多い、RIP はこの動物モデルで正常にされてきました。しかし、十分な哺乳類卵母細胞とリッピングを実行する着床前胚を得ることが困難です。SM 魚と螢光抗体 (immunoFISH) の結合組織切片の23は並進機械24,25を含む特定の Mrna に関連付けられている蛋白質を可視化する可能性を保持します。ゲノムは、健康と病気の26と関連する遺伝子変異 (例えば、小さなヌクレオチドの多形、SNPs) を測定します。Phenomics は、環境圧力27,28による細胞応答の変化を識別します。現在の研究は、特定の表現型を持つゲノムの変化を接続するメカニズムを探すものです。ImmunoFISH の使用には、SNP 依存的 mRNA 発現変化および貢献タンパク質発現細胞の表現型にリンクする可能性があります。技術が進化すると、可能性があります複数の生物学的システムの重要なメカニズムを識別する SM 魚の他のアプリケーションがあります。
開示事項
著者申告するものがあります。
謝辞
博士ダニエル ・ r ・ ラーソンは、インストールとスポットを見つけると追跡プログラム13の使用と共焦点顕微鏡イメージングのためのネブラスカ大学リンカーン顕微鏡コアの技術的なサポートの彼の寛大な助けを感謝いたします。本研究は、UNL ハッチ資金 (NEB-26-206/受入番号-232435 とくちばし-26-231/受入番号-1013511) によって支えられ、ネブラスカ州農業研究部、リンカーン、ネブラスカの大学の貢献を表します。
資料
| Name | Company | Catalog Number | Comments |
| (±)-α-Lipoic acid | Sigma-Aldrich | T1395 | Alpha Lipoic Acid |
| Albumin, Bovine Serum, Low Fatty Acid | MP Biomedicals, LLC | 199899 | FAF BSA |
| BD 10mL TB Syringe | Becton, Dickinson and Company | 309659 | 10 mL syringe |
| BD PrecisionGlide Needle | Becton, Dickinson and Company | 305109 | 27 1/2 gauge needle |
| Calcium chloride dihydrate | Sigma-Aldrich | C7902 | CaCl2-2H2O |
| Citric acid | Sigma-Aldrich | C2404 | Citrate |
| D-(+)-Glucose | Sigma-Aldrich | G6152 | Glucose |
| Disodium phosphate | Na2HPO4 | ||
| Easy Grip Petri Dish | Falcon Corning | 351008 | 35 mm dish |
| Edetate Disodium | Avantor | 8994-01 | EDTA |
| Extra Fine Bonn Scissors | Fine Science Tools | 14084-08 | Straight, Sharp/Sharp, non-serrated, 13mm cutting edge scissors |
| Fetal Bovine Serum | Atlanta biologicals | S10250 | FBS |
| Gentamicin Reagent Solution | gibco | 15710-064 | Gentamicin |
| GlutaMAX-I (100X) | gibco | 35050-061 | Glutamax |
| Gold Seal Micro Slides | Gold Seal | 3039 | 25 x 75mm slides |
| Gonadotropin, From Pregnant Mares' Serum | Sigma | G4877 | eCG |
| hCG recombinant | NHPP | AFP8456A | hCG |
| Hyaluronidase, Type IV-S: From Bovine Testes | Sigma-Aldrich | H3884 | Hyaluronidase |
| Jewelers Style Forceps | Integra | 17-305X | Forceps 4-3/8", Style 5F, Straight, Micro Fine Jaw |
| L-(+)-Lactic Acid, free acid | MP Biomedicals, LLC | 190228 | L-Lactate |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | MgSO4-7H2O |
| MEM Nonessential Amino Acids | Corning | 25-025-Cl | NEAA |
| Microscope Cover Glass | Fisher Scientific | 12-542-C | 25 x 25x 0.15 mm cover slips |
| Mm-Nanog-O2-C2 RNAscope Probe | Advanced Cell Diagnostics | 501891-C2 | Nanog Probe |
| Mm-Pou5f1-O1-C3 RNAscope Probe | Advanced Cell Diagnostics | 501611-C3 | Pou5f1 Probe |
| MOPS | Sigma-Aldrich | M3183 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Paraformaldehyde |
| PES 0.22 um Membrane -sterile | Millex-GP | SLGP033RS | 0.22 um filters |
| Polyvinylpyrrolidone | Sigma-Aldrich | P0930 | PVP |
| Potassium chloride | Sigma-Aldrich | 60128 | KCl |
| Potassium phosphate monobasic | Sigma-Aldrich | 60218 | KH2PO4 |
| Prolong Gold antifade reagent | invitrogen | P36934 | Antifade reagent without DAPI |
| RNAscope DAPI | Advanced Cell Diagnostics | 320858 | DAPI |
| RNAscope FL AMP 1 | Advanced Cell Diagnostics | 320852 | Amplifier 1 |
| RNAscope FL AMP 2 | Advanced Cell Diagnostics | 320853 | Amplifier 2 |
| RNAscope FL AMP 3 | Advanced Cell Diagnostics | 320854 | Amplifier 3 |
| RNAscope FL AMP 4 ALT A | Advanced Cell Diagnostics | 320855 | Amplifier 4 ALT A |
| RNAscope FL AMP 4 ALT B | Advanced Cell Diagnostics | 320856 | Amplifier 4 ALT B |
| RNAscope FL AMP 4 ALT C | Advanced Cell Diagnostics | 320857 | Amplifier 4 ALT C |
| RNAscope Fluorescent Multiplex Detection Reagents Kit | Advanced Cell Diagnostics | 320851 | FISH Reagent Kit |
| RNAscope Probe 3-plex Negative Control Probe | Advanced Cell Diagnostics | 320871 | Negative Control |
| RNAscope Probe 3-plex Positive Control | Advanced Cell Diagnostics | 320881 | Positive Control |
| RNAscope Probe Diluent | Advanced Cell Diagnostics | 300041 | Probe Diluent |
| RNAscope Protease III | Advanced Cell Diagnositics | 322337 | Protease III |
| RNAscope Protease III & IV Reagent Kit | Advanced Cell Diagnostics | 322340 | FISH Protease Kit |
| RNAscope Protease IV | Advanced Cell Diagnostics | 322336 | Protease IV |
| S/S Needle with Luer Hub 30G | Component Supply Co. | NE-301PL-50 | blunt 30 gauge needle |
| Sodium bicarbonate | Sigma-Aldrich | S6297 | NaHCO3 |
| Sodium chloride | Sigma-Aldrich | S6191 | NaCl |
| Sodium hydroxide | Sigma-Aldrich | 306576 | NaOH |
| Sodium pyruvate, >= 99% | Sigma-Aldrich | P5280 | Pyruvate |
| Solution 6 Well Dish | Agtechinc | D18 | 6 well dish |
| Taurine | Sigma-Aldrich | T8691 | Taurine |
| Tissue Culture Dish | Falcon Corning | 353002 | 60 mm dish |
| Triton X-100 | Sigma-Aldrich | X100 | Triton X-100 |
参考文献
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proceedings of the National Academy of Sciences. 96 (16), 9236-9241 (1999).
- MacK, E. M., Smith, J. E., Kurz, S. G., Wood, J. R. CAMP-dependent regulation of ovulatory response genes is amplified by IGF1 due to synergistic effects on Akt phosphorylation and NF-kB transcription factors. Reproduction. 144 (5), 595-602 (2012).
- Xie, F., Timme, K. A., Wood, J. R. Using Single Molecule mRNA Fluorescent in Situ Hybridization (RNA-FISH) to Quantify mRNAs in Individual Murine Oocytes and Embryos. Scientific Reports. 8 (1), 7930 (2018).
- Ruebel, M. L., et al. Obesity modulates inflammation and lipidmetabolism oocyte gene expression: A single-cell transcriptome perspective. Journal of Clinical Endocrinology and Metabolism. 102 (6), 2029-2038 (2017).
- Borensztein, M., Syx, L., Servant, N., Heard, E. . Mouse Oocyte Development. 1818, 51-65 (2018).
- Jansova, D., Tetkova, A., Koncicka, M., Kubelka, M., Susor, A. Localization of RNA and translation in the mammalian oocyte and embryo. PLoS ONE. 13 (3), 1-25 (2018).
- Player, A. N., Shen, L. P., Kenny, D., Antao, V. P., Kolberg, J. A. Single-copy gene detection using branched DNA (bDNA) in situ hybridization. Journal of Histochemistry and Cytochemistry. 49 (5), 603-611 (2001).
- Wang, F., et al. RNAscope: A novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Itzkovitz, S., van Oudenaarden, A. Validating transcripts with probes and imaging technology. Nature Methods. 8 (4), S12-S19 (2011).
- Derti, A., et al. ProbeDesigner: for the design of probesets for branched DNA (bDNA) signal amplification assays. Bioinformatics. 15 (5), 348-355 (1999).
- Larson, B., Malayter, D., Shure, M. Multiplexed Detection of Cytokine Cancer Biomarkers using Fluorescence RNA In Situ Hybridization and Cellular Imaging. BioTek Application Notes. , 1-5 (2016).
- Buxbaum, A. R., Wu, B., Singer, R. H. Single β-Actin mRNA Detection in Neurons Reveals a Mechanism for Regulating Its Translatability. Science. 343 (6169), 419-422 (2014).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Herrick, J. R., Paik, T., Strauss, K. J., Schoolcraft, W. B., Krisher, R. L. Building a better mouse embryo assay: effects of mouse strain and in vitro maturation on sensitivity to contaminants of the culture environment. Journal of Assisted Reproduction and Genetics. 33 (2), 237-245 (2016).
- Pohlmeier, W. E., Xie, F., Kurz, S. G., Lu, N., Wood, J. R. Progressive obesity alters the steroidogenic response to ovulatory stimulation and increases the abundance of mRNAs stored in the ovulated oocyte. Molecular Reproduction and Development. 81 (8), 735-747 (2014).
- Xie, F., Anderson, C. L., Timme, K. R., Kurz, S. G., Fernando, S. C., Wood, J. R. Obesity-dependent increases in oocyte mRNAs are associated with increases in proinflammatory signaling and gut microbial abundance of lachnospiraceae in female mice. Endocrinology. 157 (4), 1630-1643 (2016).
- Hirao, Y., Yanagimachi, R. Detrimental effect of visible light on meiosis of mammalian eggs in vitro. Journal of Experimental Zoology. , (1978).
- Takenaka, M., Horiuchi, T., Yanagimachi, R. Effects of light on development of mammalian zygotes. Proceedings of the National Academy of Sciences. 104 (36), 14293-14293 (2007).
- Komminoth, P., Werner, M. Target and signal amplification: Approaches to increase the sensitivity of in situ hybridization. Histochemistry and Cell Biology. 108 (4-5), 325-333 (1997).
- Hornick, J. E., Duncan, F. E., Shea, L. D., Woodruff, T. K. Multiple follicle culture supports primary follicle growth through paracrine-acting signals. Reproduction. 145 (1), 19-32 (2013).
- Vlasova-St. Louis, I., Bohjanen, P. Feedback Regulation of Kinase Signaling Pathways by AREs and GREs. Cells. 5 (1), 4 (2016).
- Gilbert, C., Svejstrup, J. Q. RNA Immunoprecipitation for Determining RNA-Protein Associations In Vivo. Current Protocols in Molecular Biology. 75 (1), 27.4.1-27.4.11 (2006).
- Kwon, S., Chin, K., Nederlof, M., Gray, J. W. Quantitative, in situ analysis of mRNAs and proteins with subcellular resolution. Scientific Reports. 7 (1), 16459 (2017).
- Voigt, F., et al. Single-Molecule Quantification of Translation-Dependent Association of mRNAs with the Endoplasmic Reticulum. Cell Reports. 21 (13), 3740-3753 (2017).
- Halstead, J. M., Wilbertz, J. H., Wippich, F., Lionnet, T., Ephrussi, A., Chao, J. A. TRICK: A Single-Molecule Method for Imaging the First Round of Translation in Living Cells and Animals. Methods in Enzymology. 572, (2016).
- Cookson, W., Liang, L., Abecasis, G., Moffatt, M., Lathrop, M. Mapping complex disease traits with global gene expression. Nature Reviews Genetics. 10 (3), 184-194 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: the next challenge. Nature Reviews Genetics. 11, 855 (2010).
- Freimer, N., Sabatti, C. The Human Phenome Project. Nature Genetics. 34, 15 (2003).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved