Method Article
Uso di singola molecola fluorescente ibridazione In Situ (SM-pesce) per quantificare e Localize mRNAs in ovociti murini
In questo articolo
Riepilogo
Per riproducibile contare i numeri dei mRNAs in singoli ovociti, in situ di fluorescenza di singola molecola del RNA (RNA-pesce) l'ibridazione è stato ottimizzato per cellule non aderenti. Gli ovociti sono stati raccolti, ibridata con le sonde specifiche di trascrizione e quantificati tramite un software di quantificazione di immagine.
Abstract
Attuali metodi abitualmente utilizzati per quantificare il mRNA di ovociti ed embrioni includono reazione a catena della polimerasi d'inversione-trascrizione digitale (dPCR), quantitativa, Real-Time RT-PCR (RT-qPCR) e sequenziamento di RNA. Quando queste tecniche vengono eseguite utilizzando un singolo oocita o embrione, basso-copia mRNA non vengono rilevati in modo affidabile. Per ovviare a questo problema, gli ovociti o embrioni possono essere riuniti insieme per analisi; Tuttavia, questo porta spesso a un'elevata variabilità tra i campioni. In questo protocollo, descriviamo l'uso di ibridazione fluorescente in situ (FISH) utilizzando la chimica del DNA ramificato. Questa tecnica identifica il pattern spazio dei mRNAs in singole celle. Quando la tecnica è accoppiata con ricerca di Spot e software di monitoraggio computer, l'abbondanza del mRNA nella cellula anche può essere quantificato. Utilizzando questa tecnica, c'è ridotta variabilità all'interno di un gruppo sperimentale e un minor numero di ovociti ed embrioni sono necessarie per rilevare differenze significative tra i gruppi sperimentali. Commercialmente disponibile DNA ramificato SM-Kit di pesce sono stati ottimizzati per rilevare mRNAs in sezionato tessuti o cellule aderenti sulle diapositive. Tuttavia, gli ovociti non aderiscono in modo efficace alle diapositive e alcuni reagenti contenuti nel kit erano troppo duri con conseguente lisi degli ovociti. Per evitare questo Lisi, diverse modifiche sono state fatte per il kit di pesce. In particolare, il buffer di permeabilizzazione e lavaggio di ovocita progettato per l'immunofluorescenza di ovociti ed embrioni sostituito i buffer proprietari. La permeabilizzazione, lavaggi e le incubazioni con sonde e amplificatore sono state eseguite in piastre da 6 pozzetti e ovociti sono stati collocati sulle diapositive alla fine del protocollo utilizzando mezzi di montaggio. Queste modifiche sono state in grado di superare le limitazioni del kit disponibili in commercio, in particolare, la lisi dell'ovocita. Per accurato e riproducibile contare il numero dei mRNAs in singoli ovociti, software per computer è stato utilizzato. Insieme, questo protocollo rappresenta un'alternativa alla PCR e sequenziamento per confrontare l'espressione di specifici trascritti in singole cellule.
Introduzione
Reazione a catena d'inversione-transcriptase della polimerasi (PCR) è stato il gold standard per la quantificazione di mRNA. Due saggi, digitale PCR (dPCR)1 e quantitativa in tempo reale PCR (qPCR)2 sono attualmente utilizzati. Le due tecniche di PCR, dPCR ha una maggiore sensibilità rispetto qPCR suggerendo che potrebbe essere utilizzato per misurare l'abbondanza del mRNA in singole cellule. Tuttavia, nelle nostre mani, dPCR analisi dei mRNAs abbondanza bassa nelle piscine di ovociti di 5 a 10 per ogni campione sperimentale ha prodotto dati con scarsa riproducibilità e variazione alta3. Ciò è probabilmente dovuto l'errore sperimentale associata con estrazione del RNA e l'efficienza di trascrizione inversa. Sequenziamento di RNA è stata eseguita anche utilizzando un unico mouse e ovociti umani4,5. Questa tecnica richiede passaggi di amplificazione del cDNA necessari per la generazione della libreria che probabilmente aumenta la variabilità all'interno di un gruppo sperimentale. Inoltre, trascrizioni di abbondanza bassa possono non essere rilevabile. Anche se i prezzi di sequenziamento sono scesi di ultimi anni, può ancora essere costi proibitivi a causa del costo elevato di analisi bioinformatica. Infine, la localizzazione di mRNA è un processo dinamico con cambiamenti spaziali contribuendo alla proteina funzione6. Pertanto, abbiamo deciso di adottare una tecnica che avrebbe prodotto accurate e riproducibile misure quantitative e localizzazione di singoli mRNA in singoli ovociti.
DNA ramificato accoppiato all'ibridazione fluorescente in situ amplifica il segnale di fluorescenza piuttosto che amplificazione RNA/cDNA abilitazione individuazione dei singoli mRNAs in singole celle 7,8,9. Il dosaggio è effettuato attraverso una serie di ibridazione, amplificazione (usando il DNA ramificato) e fluorescenza etichettatura passi al fine di amplificare il segnale di fluorescenza7. La tecnica comincia con l'associazione di coppie di sonda di 18 - 25-base del oligonucleotide che sono complementari a un specifico mRNA3,8,10. Quindici-venti coppie di sonda sono progettate per ogni specificità assicurando di trascrizione per la trascrizione di destinazione. L'ibridazione di specifici mRNA è seguita dalle sonde preamplificatore e amplificatore che formano una configurazione ramificata. Circa, 400 etichetta fluorofori associare a ciascun amplificatore, conseguente 8000-fold aumento in fluorescenza permettendo la rilevazione di singoli mRNA (Figura 1)11.
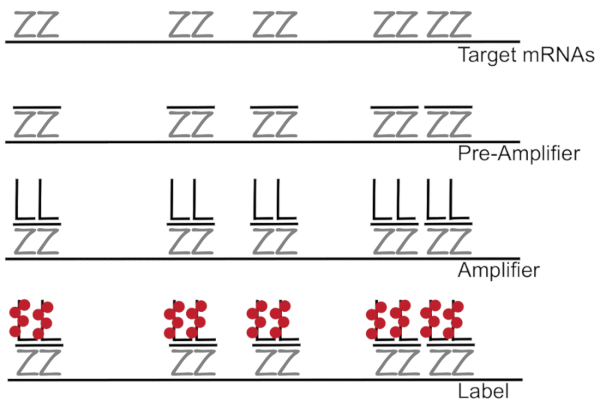
Figura 1: schema del protocollo SM-pesce. Sequenza ibridazione della sonda specifica trascrizione, ramificata amplificatore DNA e fluoroforo ad un target di mRNA viene mostrato. Clicca qui per visualizzare una versione più grande di questa figura.
Gli studi precedenti usando la singola molecola fluorescenza in situ di ibridazione (SM-pesce) localizzato β-actina mRNA in singoli neuroni12 e papillomavirus umano DNA nel cancro cervicale delle cellule linee7. Il software per computer trovare posto e programma di controllo identifica singolo segnale fluorescente punctato ed è stato usato con successo per quantificare il numero dei mRNAs in ogni cella3,13.
Sulla base dei risultati della rilevazione di mRNA in neuroni12, abbiamo supposto che SM-pesce si sarebbe rivelato un utile strumento per quantificare i livelli di trascrizione in murini ovociti ed embrioni tra cui abbondanza bassa mRNAs. Tuttavia, la tecnica è ottimizzata per l'utilizzo con celle fisse aderenti e formaldeide fissata paraffina embedded sezioni di tessuto (FFPE). Gli ovociti non possono aderire a una diapositiva, anche quando essi sono rivestiti con poli-L-lisina. Inoltre, essi sono più fragili di cellule somatiche e sezioni di tessuto con conseguente lisi cellulare quando sottoposto ad alcuni dei buffer proprietarie in kit commercialmente disponibili3. Per superare queste sfide, ovociti erano fissi e trasferiti manualmente tra gocce dei buffer. Inoltre, la permeabilizzazione e wash buffer nei kit sono stati sostituiti per ridurre la lisi delle cellule. Sonde predefiniti vengono acquistati a fianco il kit di pesce o possono essere richiesti specifici trascritti. Ogni set di sonde proprietarie è disponibile in uno dei tre canali di fluorescenza (C1, C2 e C3) per consentire per multiplexing. Nell'esperimento attuale, ovociti murini sono stati doppia colorazione e quantificati mediante una sonda di C2 Nanog e una sonda di C3 Pou5f1 . Queste sonde sono state selezionate basato sull'espressione di Nanog e Pou5f1 segnalato di ovociti ed embrioni. A conclusione delle operazioni di ibridazione, gli ovociti sono stati collocati in gocce di mezzi di montaggio anti-dissolvenza per vetrini istologici dell'applicazione. Immagini confocal sono stati utilizzati per quantificare il numero di segnali fluorescenti punctati che rappresentano singoli mRNA. Oltre a quantificare i mRNAs, formazione immagine ha mostrato anche la distribuzione spaziale di specifici mRNA nella cellula, quali altri metodi di quantificazione di RNA sono in grado di raggiungere. Questa tecnica ha dimostrato di avere bassa variabilità all'interno di un gruppo sperimentale che consente di utilizzare numeri più piccoli degli ovociti in ogni gruppo sperimentale per identificare differenze significative tra i gruppi sperimentali3.
Protocollo
Procedure di animali sono stata esaminate e approvate dal comitato di impiego presso l'Università di Nebraska-Lincoln e istituzionali Animal Care e tutti i metodi sono stati effettuati conformemente alle norme e linee guida pertinenti. Per questo studio, CD-1 esogami topi ha avuto accesso ad libitum di chow roditore normale e acqua; sono stati mantenuti in un 12:12 scuri: ciclo di luce.
1. preparazione del supporto richiesto
- Per i supporti di base (OMM), aggiungere 100 mM NaCl, 5 mM KCl, 0,5 mM KH2PO4e 1,7 mM CaCl2-2 H2O a 100 mL di acqua sterile.
Nota: Medio OMM possa essere memorizzato fino ad 1 mese. - Per i media completi (OMOPS), aggiungere 20 mM 3-morpholinopropane-1-solfonico acido (MOPS), 1,2 mM MgSO4-7 H2O, glucosio di 0,5 mM, 6 mM L-lattato, 1mm ala-gln, 0,1 mM della taurina, 1 x aminoacidi non essenziali (NEAA), (acido etilendiamminotetracetico di 0,01 mM EDTA), l'acido alfa lipoico 10 µM, gentamicina di 10 µ g/mL non diluito, 21 mM 1 M NaOH, 5mm NaHCO3, 0,2 mM piruvato, 0,5 millimetri di citrato, FAF 4 mg/mL BSA per un 01:10 diluizione di OMM in acqua sterile per un volume totale di 100 mL. Sterilizzare il mezzo con un filtro da 0,22 µM.
Nota: OMOPS possono essere memorizzati fino a 1 settimana. - Per il mezzo di supporto (HM) aggiungere 5% di siero fetale bovino OMOPS. Fare 2 mL HM per topo.
- Per la soluzione di ialuronidasi aggiungere 0,1 mg/mL di ialuronidasi derivato dai testicoli bovini, a 1 mL HM.
- Per il buffer di fissazione combinare paraformaldeide al 4% in 10 mL di 1X PBS con 0,1% embrione grado polivinilpirrolidone (PVP)14.
- Per preparare 50 mL di tampone di lavaggio (WB), aggiungere 0,1% tensioattivi non ionici e 0.1% PVP a 1X PBS14.
- Per preparare 10 mL di tampone di permeabilizzazione, aggiungere 1% tensioattivo non - ionico a 1X PBS14.
Nota: I buffer di lavaggio e permeabilizzazione descritti sopra sostituiscono i buffer proprietari nei kit disponibile in commercio.
2. raccolta di ovociti ovulati da topi femmine
- Preparazione:
- Stimolare i topi femmina a 5-8 settimane di età tramite l'iniezione intraperitoneale (IP) di 5 IU gonadotropina corionica equina (eCG) seguita da 5 UI gonadotropina corionica umana (hCG) 44-48 h successive15,16.
- Mantenere il 35mm di Petri contenente 2 mL di HM su una piastra riscaldante a 37 ° C. Pipettare uno, goccia 100-µ l di HM contenente ialuronidasi diluito seguita da tre, gocce di 50 µ l di HM senza ialuronidasi in una capsula Petri 60 mm. Posizionare le piastre contenenti gocce sul 37 ° C riscaldamento piastra prima dell'uso.
Nota: Le gocce di ialuronidasi devono essere effettuate appena prima la dissezione di ciascuna coppia di ovidotto per evitare l'evaporazione e concentrazione dei componenti del HM con o senza ialuronidasi.
- Eutanasia di topi, 16 h dopo l'iniezione di IP di hCG, utilizzando isoflurano sovradosaggio seguita da dislocazione cervicale.
- Pulire il mouse utilizzando etanolo al 70%. Esporre la cavità addominale e visualizzare il tratto riproduttivo femminile. Tenere l'ovaio con il forcipe e togliere i legamenti uterini e del tessuto adiposo in eccesso da intorno l'ovaia. Tagliare ovidotto dall'utero e la coppia di ovaia-ovidotto in HM caldo nel piatto di 35 mm.
- Rimuovere l'ovaio e qualsiasi tessuto adiposo circostante. Tear ampulla gonfio dell'ovidotto utilizzando un ago calibro 27 ½ pollice. Spingere l'ovidotto presso il sito dello strappo e i complessi di celle-ovocita cumulus (COC) saranno espulsi. Trasferire gli ovociti ovulated, che si presumono essere in metafase II (MII) di meiosi, per il calo di 100 μL contenente supporto HM con ialuronidasi usando una pipetta di bocca (Figura 2).

Figura 2 : Parti del pipettatore bocca utilizzato per trasferire gli ovociti. (A) bocca pezzo (B) 0.22 um, 4 mm (C) aspiratore tubo (D) 1000 μL dispensare punta del filtro (E) 9" pipetta Pasteur. Clicca qui per visualizzare una versione più grande di questa figura.
- Pipettare i complessi di cellula ovocita-cumulus MII su e giù nell'ialuronidasi contenente HM con la pipetta di bocca per sloggiare le cellule del cumulo. Trasferire ogni ovocita, una volta che essi sono privi di cellule del cumulo ad un lavaggio drop HM contenente solo usando la pipetta di bocca. Ripetere questa operazione per ogni goccia di lavaggio. Non trasferire gli ovociti frammentati o trasparente15.
Nota: È importante trasferire gli ovociti da ogni goccia nel piatto 35 mm con come piccolo HM come possibile. Questo è vero per ogni trasferimento nel protocollo. Gli ovociti MII non devono rimanere nell'ialuronidasi contenenti HM mezzo per più di un minuto.
3. SM-pesce colorazione degli ovociti
- Difficoltà ovociti in un singolo pozzo di una piastra a 6 pozzetti contenenti 500 µ l di tampone di fissazione. Immergere 20 ovociti o meno nel pozzo. Incubare per 20 minuti a temperatura ambiente.
Nota: Ogni passo colorazione SM-pesce si verifica all'interno di un singolo pozzo in un piatto conico 6 pozzetti. Garantire che gli ovociti sono completamente sommerse nei buffer e non galleggianti in cima il buffer. Ogni passaggio deve essere eseguita con 20 ovociti o meno in ciascun pozzetto. - Trasferire gli ovociti fissi a 500 μL di tampone di lavaggio (WB descritto al punto 1.6) per 10 minuti ciascuno. Ripetere 2 volte.
- Incubare gli ovociti nel buffer di permeabilizzazione per 30 min a temperatura ambiente.
Nota: Il buffer di permeabilizzazione descritto al punto 1.7 sostituisce il buffer di permeabilizzazione di correttezza.- Raccogliere set di sonda e ruota velocemente verso il basso in una microcentrifuga. Riscaldare ogni sonda impostata per 10 min in un bagno d'acqua 40 ° C o in un incubatore. Raffreddare a temperatura ambiente.
Nota: Questo passaggio deve essere eseguito durante l'incubazione di permeabilizzazione
- Raccogliere set di sonda e ruota velocemente verso il basso in una microcentrifuga. Riscaldare ogni sonda impostata per 10 min in un bagno d'acqua 40 ° C o in un incubatore. Raffreddare a temperatura ambiente.
- Lavare gli ovociti in 500 µ l di WB per 10 min a temperatura ambiente.
- Trasferire gli ovociti alle 80 μL di proteasi III Buffer (disponibile dal kit), che è diluito 1:8 in 1X PBS, per 30 min a temperatura ambiente.
Nota: Il volume di 80 µ l copre adeguatamente il fondo di un pozzo individuo in una piastra a 6 pozzetti. - Lavare gli ovociti in 500 µ l di WB per 10 min a temperatura ambiente.
- Diluire i set sonda riscaldata per Nanog, Pou5f1 e DapB (un gene di controllo negativo), 01:50 in diluente di sonda. Incubare gli ovociti in 80 μL della sonda trascrizione specifici per 2 ore a 40° C.
Nota: Ogni set di sonde proprietarie è disponibile in uno dei tre canali di fluorescenza (C1, C2 e C3). Le sonde Nanog e Pou5f1 erano etichettate come C2 e C3, rispettivamente. - Scaldare la proprietaria, 1 amplificatore (AMP 1), amplificatore 2 (AMP2), amplificatore 3 (AMP3) e amplificatore 4-fluorescenza (AMP 4-FL) a temperatura ambiente.
Nota: Questo passaggio deve essere eseguito durante l'incubazione di sonda di trascrizione-specific di 2 ore. - Trasferire gli ovociti a 500 μL di WB e incubare per 10 minuti a temperatura ambiente.
- Incubare gli ovociti in sequenza nei buffer di amplificazione.
- Incubare gli ovociti in 80 µ l di AMP1 per 30 min a ovociti di trasferimento di 40° c a 500 µ l di WB per 10 min a temperatura ambiente.
- Incubare gli ovociti in 80 µ l di AMP2 per 15 min a 40 ° C. Trasferire gli ovociti a 500 µ l di WB per 10 min a temperatura ambiente.
- Incubare gli ovociti in 80 µ l di AMP3 per 30 min a 40 ° C. Trasferire gli ovociti a 500 µ l di WB per 10 min a temperatura ambiente.
Nota: Il resto del protocollo viene eseguito al buio perché AMP-FL contiene il fluoroforo. Quando si lavora sotto il microscopio per dissezione, ridurre quanto più possibile la luce. - Aggiungere gli ovociti a 80 μL di AMP4-FL per 15 min a 40° C.
Nota: AMP4-FL viene fornito come alternativa tampone-A (Alt-A), Alt-B o Alt-C. Selezionare il dipendente buffer AMP4-FL su quale emissione lunghezza d'onda è desiderato.
- Lavare gli ovociti in 500 µ l di WB per 10 min a temperatura ambiente. Incubare gli ovociti in 80 µ l di DAPI per 20 min a temperatura ambiente. Lavare gli ovociti in 500 µ l di WB per 5 min a temperatura ambiente.
- Pipettare 12 µ l di mezzi di montaggio anti-affievolimento al centro di una diapositiva senza aggiunta di bolle per il reagente. Trasferire gli ovociti con WB piccolo come possibile nel supporto di montaggio e di applicare un vetrino coprioggetti.
- Il coprioggetto ad un angolo di inclinazione e lentamente e delicatamente posto sopra il liquido sulla diapositiva. Evitare di premere troppo duro per evitare la distorsione del ovociti e introduzione di bolle il vetrino coprioggetti.
- Conservare i vetrini in una scatola scura per asciugare durante la notte a temperatura ambiente. Rivestire i bordi delle diapositive in smalto trasparente per sigillare il vetrino coprioggetti.
- Utilizzare un microscopio standard per trovare ovociti sulla diapositiva e cerchio con un pennarello indelebile.
Nota: Questo passaggio non è obbligatorio ma migliora posizionamento ovociti sulla diapositiva. Per risultati ottimali, diapositive di immagini all'interno di 1 a 5 giorni come il segnale fluorescente inizierà a svanire.
4. image Processing
- Immagine 3-dimensionale ovociti, usando la microscopia confocal di z passo.
Nota: Per analizzare con precisione le immagini, ogni passaggio di z deve essere 1,0 µm/fetta. - Salvare immagini confocal come un nd2 compresso o individuali. File TIFF per ogni ovocita. Entrambi i tipi di immagine sono compatibili con il programma di elaborazione di immagine di open source, Fiji.
- Scaricare e installare il software di accesso aperto Fiji (https://imagej.net/Fiji/Downloads).
- Trascinare i file nd2 nelle Fiji e scegliere hyperstack. Se immagini confocal sono stati salvati come. I file TIFF e passare al punto 4.4.
Nota: Quando il file nd2 è caduto in Fiji l'elenco a discesa hyperstack dovrebbe apparire automaticamente. - Fare clic sulla scheda immagine , selezionare coloree fare clic su Canali Split per separare i canali fluorescenti del file nd2.
- Generare singoli. File TIFF per ogni fetta z dell'ovocita in ciascun canale fluorescente. Fare clic sulla scheda immagine , selezionare stacke fare clic su Stack di immagini. Fare clic sulla scheda immagine , selezionare tipoe fare clic su Colore RGB per convertire ogni fetta z in una singola immagine di colore RGB.
Nota: Il colore RGB è artificiale e può essere scelto come desiderato per ogni lunghezza d'onda di emissione. - Salvare ogni immagine convertita come. File TIFF. Inserire immagini da un singolo oocita per ogni canale fluorescente in una nuova cartella per evitare confusione durante la cucitura (punto 4.3).
- Trascinare i file nd2 nelle Fiji e scegliere hyperstack. Se immagini confocal sono stati salvati come. I file TIFF e passare al punto 4.4.
- Normalizzare ciascuno. Immagine TIFF per Pou5f1 e Nanog utilizzando immagini di controllo negativo (DapB).
Nota: La normalizzazione viene eseguita utilizzando un programma di fotoritocco. Assicurarsi di rimuovere gli stessi livelli di fluorescenza di fondo da ogni immagine di controllo. - Aperto ogni normalizzato. File TIFF in Fiji per cucire insieme tutte le z-fette per ogni ovocita in ogni lunghezza d'onda.
- Fare clic sulla scheda plugin , selezionare impunturee fare clic su Griglia/raccolta (Figura 3A). Selezionare Immagini sequenziali dal menu a discesa e fare clic su OK (Figura 3B).
- Individuare la directory e selezionare la cartella contenente tutte le immagini di fetta z per un ovocita individuale ad una lunghezza d'onda (Vedi punto 4.3.4). Fare clic su OK.
- Spostare il dispositivo di scorrimento nella parte inferiore dell'immagine cucita sul canale di colore appropriato per la lunghezza d'onda utilizzata e creare l'immagine finale di cucito RGB facendo clic su immagine, selezionando il tipoe fare clic su Colore RGB.
Nota: Questa immagine verrà essere utilizzata per la quantificazione di fluorescenza descritta al punto 4.6 qui sotto.
- Convertire l'immagine cucita a 32-bit massima immagine proiettata. Fare clic su immagine, selezionare tipoe scegliere a 32-bit (Figura 3). Salva l'immagine come nuova. File TIFF.
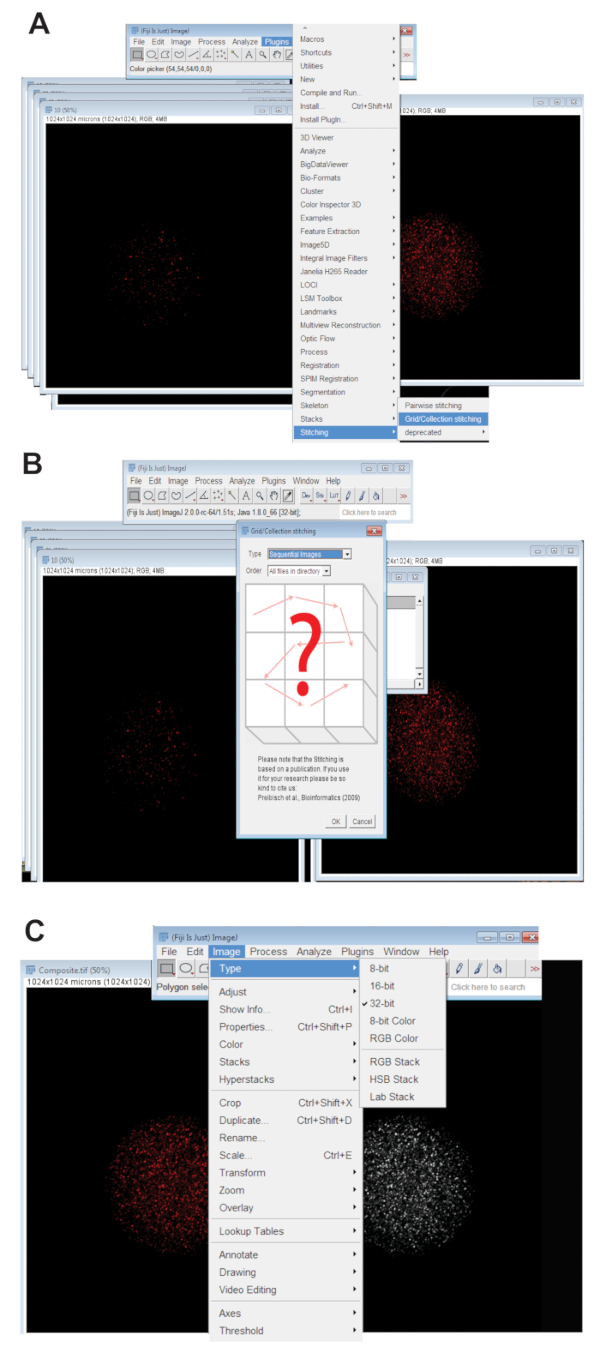
Figura 3 : Cucendo insieme di immagini confocal z-serie di ovociti. (A) Screenshot che mostra lo strumento griglia/raccolta di plug-in Fiji che fu usato per produrre immagini composite dell'ovocita. (B) immagini sequenziali utilizza fluorescenza sovrapposizione tra sequenziale. File TIFF per generare un'immagine composita. (C) l'immagine composita è stato salvato come un 32-bit. File TIFF. Clicca qui per visualizzare una versione più grande di questa figura.
- Scaricare e installare la ricerca di Spot e programma di controllo13, che è disponibile sul sito Web per D.R. Larson, un ricercatore presso il National Institutes di salute National Cancer Institute (https://ccr.cancer.gov/Laboratory-of-Receptor-Biology-and-Gene-Expression/daniel-r-larson). Scaricare e installare la macchina virtuale di accesso aperto per il sistema operativo in lingua (IDL) interattiva dei dati che è necessario per eseguire la ricerca di Spot e programma di controllo (http://www.spacewx.com/pdf/idlvm.pdf).
- Aprire l'immagine a 32-bit, cucita, che è stato generato nel passaggio 4.6 (Figura 4A), nella ricerca di Spot e programma di controllo. Selezionare l' elenco a discesa Localize e scegliere Localize (Figura 4B), che consente di calcolare il numero macchie presenti nell'immagine.
Nota: Ogni spot contato rappresenta un singolo mRNA. Le impostazioni di soglia band pass e fotone sono mostrate nella schermata (Figura 4B). Per questo protocollo, è stato utilizzato il valore predefinito per ogni impostazione di soglia. Rappresentante positivo e macchie di sfondo vengono visualizzate (Figura 4).

Figura 4 : Quantificazione del mRNA usando Spot Finder e Tracking. (A) immagini individuali z-serie sono stati cuciti insieme come descritto nella Figura 3 e salvati come un massimo di 32 bit proiettato. File TIFF. (B) immagine composita è stato aperto in Spot Finder e rilevamento. Localizzare è stato usato per contare le macchie fluorescenti (riquadro rosso). Soglia di banda pass e fotone sono indicati da blue box. (C) la freccia blu indica un segnale positivo (sopra soglia). La freccia bianca indica una fluorescente posto sotto la soglia e, pertanto, non conteggiati. Clicca qui per visualizzare una versione più grande di questa figura.
Risultati
A completamento del protocollo, il risultato sarà singole immagini da confocale z-series (Figura 4A e Figura 5), immagini unite (Figura 4), e conta di mRNA (Figura 4B). Quando viene eseguita la multiplazione, ci sarà anche Unite immagini che mostrano l'etichetta per due differenti mRNA (Figura 5). I conteggi di mRNA vengono generati utilizzando immagini unite generati da Fiji (Figura 3) e le macchie di fluorescenza punctate contati usando la ricerca di Spot e programma di controllo (Figura 4B).
I conteggi di mRNA sono successivamente analizzati utilizzando uno strumento di analisi di dati standard. In questo protocollo, abbiamo etichettato n = 12 ovociti con Pou5f1 e Nanog. I risultati per ogni mRNA vengono calcolata la media e l'errore standard della media calcolata. I dati raccolti in questo protocollo ha mostrato 775 ± 26 SEM Pou5f1 trascrizioni e 113 ± 5 SEM Nanog trascrizioni negli ovociti MII (Figura 5). Un test di student t-determinato che una differenza statisticamente significativa nel mRNA conta tra Pou5f1 e Nanog. Soprattutto, c'erano macchie rilevate in n = 5 ovociti che vengono etichettati con sonda DapB (cioè, controllo negativo). Si noti il piccolo errore standard utilizzando solo 12 ovociti individuali. La sensibilità del dosaggio è sottolineata anche dal riscontro positivo riproducibile di Nanog(Figura 5). Esperimenti di dPCR precedenti non hanno rilevato riproducibile Nanog che indica che il numero dei mRNAs Nanog in un ovocita individuo è sotto il rilevamento di soglia utilizzando dPCR 3.
In esperimenti pilota, abbiamo rilevato la fluorescenza ridotta se c'è stato un ritardo fra fissazione degli ovociti e l'inizio del protocollo SM-pesce. Allo stesso modo, se non vengono utilizzati i buffer proprietarie di ibridazione, la sonda e l'amplificazione di DNA ramificato non entrare nella cellula con conseguente fluorescenza inanellati intorno la membrana plasmatica dell'ovocita. Ciò è probabilmente dovuto l'aggregazione del DNA ramificato. Fluorescenza anellata intorno la membrana dell'ovocita si tradurrà anche se c'è scarsa permeabilizzazione della membrana plasmatica. È necessario anche ottimale degradazione della proteina associata a mRNA. Il buffer di proteasi fornito nel kit SM-pesce è a una concentrazione ottimale per il trattamento di sezioni di tessuto e cellule aderenti. Tuttavia, quando si utilizza cellule non aderenti, è importante individuare empiricamente la diluizione di proteasi migliore. Troppo poco buffer di proteasi potrebbe sottoconteggio di mRNA a causa della scarsa accessibilità della sonda e il mRNA. Allo stesso modo, troppa proteasi potrebbero causare riduzione delle non solo proteine associato a mRNA ma anche la destabilizzazione dei mRNAs. In questo protocollo, abbiamo testato il rilevamento di mRNA mediante una curva di titolazione di non diluito (1:1), 1:4, 1:8 e 01:12 buffer di proteasi diluito in PBS 1X (n = 2 o 3 ovociti per diluizione). La media fluorescente espressione del mRNA di Pou5f1 negli ovociti MII era 169 ± 42 SEM (non diluito), 176 ± 36 SEM (1:4), 308 ± 18 SEM (1:8) e 445 ± 24 SEM (01:12) (Figura 6). Diluizione di proteasi utilizzata nel presente protocollo era 1:8 come ha mostrato la minima variazione.

Figura 5 : Pou5f1 e Nanog mRNA nell'oocita MII. (A). immagini rappresentative dell'immagine centrale serie z sono indicati sulla sinistra. Pou5f1 viene rilevato in rosso (647 nm) lunghezza d'onda mentre Nanog è rilevato nel verde (488 nm) lunghezza d'onda. DAPI la macchiatura dei cromosomi allineati sul mandrino metafase II, caratteristico dell'ovocita MII, appare in nero. Non c'era nessuna macchiatura per DapB in entrambi il 647 nm o 488 nm emissione lunghezza d'onda. (B) il numero dei mRNAs Pou5f1 e Nanog è mostrato come media SEM (n = 12 ovociti); non c'era nessun rilevamento (n. d) di DapB. * indica P < 0,05, barra della scala è 10 µm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 6 : Titolazione empirica del buffer di proteasi per ottimizzare il conteggio accurato dei mRNAs. Immagini rappresentative di SM-pesci di Pou5f1 negli ovociti MII. DAPI macchiatura Mostra i cromosomi allineati sul mandrino MII. Gli ovociti sono stati incubati con non diluito (1:1), 01:12, 1:8 o 1:4 diluizioni del buffer di proteasi III. Il numero dei mRNAs Pou5f1 (n = 2-3 ovociti) sono stati contati e la media di SEM è mostrato. Barra della scala è 10 µm. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 7 : Trasferimento degli ovociti MII attraverso la fissazione, la proteasi e l'ibridazione buffer nei pozzetti di una piastra a 6 pozzetti. Viene visualizzata la forma di ciascun pozzetto. (A) cellule galleggiano quando prima aggiunto al buffer (B) sommersione degli ovociti nei buffer. (C) ovociti si depositano alla parte inferiore del pozzo durante il periodo di incubazione. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Una serie di passaggi minore durante il protocollo garantirà successo fluorescenza e conteggi precisi dei mRNAs. In primo luogo, il protocollo deve essere eseguito immediatamente dopo la raccolta e la fissazione degli ovociti. Si noti che il PVP viene aggiunto nel buffer di fissazione del paraformaldeide al 4% per impedire che gli ovociti attaccare a vicenda. Abbiamo trovato che è necessario eseguire l'esperimento immediatamente dopo la raccolta e la fissazione degli ovociti. Qualsiasi ritardo si traduce in un segnale di fluorescenza molto inferiore che si tradurrebbe in sottoconteggio delle trascrizioni. Ciò è dovuto in parte alla degradazione del RNA. Non più di 20 ovociti dovrebbero essere trasferiti a un pozzetto della piastra a 6 pozzetti in una sola volta e ogni bene deve essere utilizzato solo una volta. Tempi di incubazione dovrebbero anche essere seguiti con precisione senza accorciamento o allungamento di ogni passaggio. L'eccezione è le fasi di tampone di lavaggio; ovociti possono essere lasciati nel tampone di lavaggio per un tempo prolungato senza alterare i risultati sperimentali. Le sonde di SM-pesce sono disponibili in tre canali di fluorescenza C1, C2 e C3. Per multiplexing, non mescolare le sonde che hanno la stessa etichetta di canale. Questo si tradurrà in entrambe le sonde fluorescenti presso la stessa emissione lunghezza d'onda rendering analisi Impossibile come non ci sarà alcun modo per distinguere tra i set di sonda. Sonde di controllo positivo progettati contro geni housekeeping sono disponibili in ciascuno dei tre canali. Sonde di controllo negativo (ad esempio, DapB) inoltre sono disponibili in set premiscelato che contengono un tag per tutti i tre canali. L'esperimento deve essere condotto in condizioni di scarsa illuminazione come gli ovociti sono sensibili alla luce dopo la rimozione dal ovidotto17,18. Dopo l'aggiunta di fluorofori attaccato al AMP4, i passaggi devono essere eseguiti con come poca luce come possibile evitare lo sbiancamento del fluoroforo. Infine, quando gli ovociti su vetrini istologici di montaggio, posizionare delicatamente il vetrino coprioggetto per evitare la formazione di bolle che possono interferire con la formazione immagine e distorsione dell'ovocita. Se trovate difficile evitare le distorsioni di cella, distanziali diapositiva devono essere utilizzati al fine di mantenere la forma sferica dell'ovocita.
Una modifica essenziale del protocollo è la sostituzione di permeabilizzazione e wash buffer fornito nel kit disponibile in commercio. I buffer di proteasi e ibridazione proprietari forniti sono ambienti difficili per gli ovociti ma sono necessari per il successo del protocollo. Se non utilizzato, gli amplificatori sono in grado di entrare nella cellula che è probabilmente dovuto l'aggregazione del DNA ramificato. Gli ovociti trasferirsi da questi ambienti la permeabilizzazione relativamente mite e wash buffer, progettato per immunofluorescenza14, che si è rivelato adeguato per il successo del protocollo e allo stesso tempo ha impedito la lisi degli ovociti. Perché gli ovociti e gli embrioni preimpianto non aderiscono a una diapositiva istologica, un'altra modifica essenziale stava mettendo gli ovociti nei buffer all'interno di un pozzetto di una piastra di coltura. Abbiamo usato una piastra di coltura dell'embrione 6 pozzetti. Ciascun pozzetto della piastra è affusolato e lati inclinati e un 8 mm piatto inferiore (Figura 7), che migliora il recupero degli ovociti. Questo è particolarmente importante poiché gli ovociti perdono le loro proprietà refrattarie e diventano quasi trasparenti nei buffer di ibridazione.
Quando si trasferiscono gli ovociti da un pozzetto a altro, è importante verificare che gli ovociti sono completamente sommersa nelle soluzioni in ogni pozzetto come cellule galleggierà quando in primo luogo trasferito ad ogni pozzetto (figura 7A). Una volta che essi sono meccanicamente sommersi nel buffer (figura 7B), essi affonderanno alla parte inferiore del pozzo entro la fine dell'incubazione (Figura 7). L'eccezione è AMP 1 e 3 AMP; Quando meccanicamente sommersa la do ovociti non completamente depositano sul fondo del pozzo. Per trovare questi ovociti, potrebbe essere necessario cambiare il piano di messa a fuoco. Pipettare attentamente e contare il numero di celle viene trasferito per evitare la perdita.
Tecniche multiple di pesce di singola molecola, che amplificano il segnale fluorescente anziché cDNA, tra cui composizioni chimiche del DNA ramificati, è stati sviluppati 9,19. Kit disponibili in commercio hanno ottimizzato il metodo DNA SM-FISH ramificati per rilevamento riproducibile di singoli mRNA in sezioni di tessuto o cellule aderenti su un vetrino istologico. Il protocollo descritto qui è stato modificato per l'utilizzo con singole cellule non aderenti (ad es., ovociti ed embrioni preimpianto)3. In questo modo non solo quantificazione specifica e riproducibile, ma anche la localizzazione di un mRNA all'interno dell'ovocita. Mentre questo è un vantaggio del dosaggio, naturalmente non ci sono limitazioni. Ad esempio, a differenza di sequenza di RNA, non è possibile identificare romanzo mRNAs. Un'ulteriore limitazione del protocollo è la disponibilità di sonde di trascrizione-specific. Sono commercialmente disponibili dalle aziende che vendono il kit SM-pesce sonde proprietarie. Ci sono parecchie sonde che sono pre-fatte. Gli altri possono essere progettati dalla società per qualsiasi mRNA con annotazioni utilizzando un algoritmo obiettivo10. Tuttavia, se un mRNA è scarsamente sequenziale sarebbe difficile progettazione sonde con alta specificità. Per brevi trascrizioni può anche essere difficile identificare abbastanza paia di sonda che non cross-reagiscono con altre trascrizioni riducendo la specificità del dosaggio. Allo stesso modo, un minor numero di set di sonda potrebbe essere insufficiente per produrre il segnale di fluorescenza sopra la soglia per il rilevamento come positivo nella ricerca di Spot e programma di controllo. In questa stessa ottica, varianti di trascrizione non possono essere rilevati con questo metodo.
Nonostante le limitazioni descritte sopra, ci sono diverse applicazioni per SM-pesce. Ad esempio, i dati di cella singola sequenza di RNA potrebbero essere convalidati soprattutto quando i numeri di cellulare sono piccole e difficili da ottenere (ad es., ovociti ed embrioni). Amplificazione del cDNA per analisi di PCR introduce un errore sperimentale che è in genere ridotta da una fase di normalizzazione utilizzando dati da geni housekeeping stabilmente espressa. Tuttavia, cambiamenti temporali nell'oocita attraverso gli embrioni pre-impianto cambia anche l'espressione di geni housekeeping. Il protocollo SM-pesce amplifica fluorescenza invece di cDNA. Di conseguenza, non è necessario per la normalizzazione dei livelli di mRNA di trascrizione specifico per ottenere risultati riproducibili con bassa variabilità. A causa della variabilità dell'efficienza di primer PCR, differenze nei numeri assoluti di diverse specie di mRNA non possono essere paragonate con precisione all'interno o tra i tipi delle cellule. SM-pesce localizza e quantifica mRNA. Di conseguenza, può essere utilizzato per identificare le celle che esprimono mRNA in una popolazione mista delle cellule. Ad esempio, quando gli ovociti sono in crescita nei follicoli primari o secondari, il follicolo può essere isolato e coltivato in alginato perline20 ma la separazione dell'ovocita dalle cellule somatiche è difficile. Pertanto, sequenziamento e gli studi di PCR sono stati effettuati utilizzando le popolazioni delle cellule miste. L'uso di SM-pesce può determinare se mRNAs rilevati nelle cellule somatiche o l'oocita del follicolo. Infine, SM-pesce ha alta sensibilità e specificità che consente per la rilevazione delle trascrizioni di abbondanza bassa; ad esempio il rilevamento di Nanog in ovociti MII (Figura 5).
Conservazione e degradazione di mRNA sono importanti meccanismi di regolazione per l'espressione della proteina. Regolazione post-trascrizionale di traduzione, di deposito e di degradazione sono mediati da proteine che si legano a mRNAs21. Attualmente, immunoprecipitazione della RNA-proteina (RIP) può essere effettuata ordinariamente quando un numero elevato di celle è disponibili22. A causa del grande numero di uova di Xenopus che possono essere raccolti da un singolo animale, RIP è stato effettuato con successo in questo modello animale. Tuttavia, è difficile ottenere abbastanza dei mammiferi ovociti ed embrioni pre-impianto per eseguire RIP. Accoppiamento di SM-pesce e immunofluorescenza (immunoFISH) 23 delle sezioni del tessuto tenere il potenziale di visualizzare proteine associate con specifici mRNA compresi macchinari traslazionale24,25. Genomica misura varianti genetiche (ad es., polimorfismi di piccolo nucleotide, SNP) associati a salute e malattia26. Phenomics identifica le modifiche nelle risposte cellulari a causa di pressioni ambientali27,28. La ricerca si propone di trovare il meccanismo che collega le alterazioni del genoma con fenotipi specifici. L'uso di immunoFISH ha il potenziale per collegare SNP-dipendente cambiamenti nell'espressione di mRNA e l'espressione delle proteine che contribuiscono a fenotipi cellulari. Come la tecnologia si evolve, ci sono probabilmente altre applicazioni di SM-pesci che identificheranno meccanismi importanti nei sistemi biologici più.
Divulgazioni
Gli autori non hanno nulla da dichiarare
Riconoscimenti
Ringraziamo il Dr. Daniel R. Larson per il suo generoso aiuto con l'installazione e l'uso della ricerca di Spot e programma di controllo 13 ed il supporto tecnico di Università del Nebraska Lincoln microscopia Core per l'imaging di microscopia confocale. Questo studio rappresenta un contributo dell'Università di divisione di ricerca agricola di Nebraska, Lincoln, Nebraska ed è stato sostenuto da fondi di tratteggio UNL (NEB-26-206/adesione numero-232435 e NEB-26-231/adesione numero-1013511).
Materiali
| Name | Company | Catalog Number | Comments |
| (±)-α-Lipoic acid | Sigma-Aldrich | T1395 | Alpha Lipoic Acid |
| Albumin, Bovine Serum, Low Fatty Acid | MP Biomedicals, LLC | 199899 | FAF BSA |
| BD 10mL TB Syringe | Becton, Dickinson and Company | 309659 | 10 mL syringe |
| BD PrecisionGlide Needle | Becton, Dickinson and Company | 305109 | 27 1/2 gauge needle |
| Calcium chloride dihydrate | Sigma-Aldrich | C7902 | CaCl2-2H2O |
| Citric acid | Sigma-Aldrich | C2404 | Citrate |
| D-(+)-Glucose | Sigma-Aldrich | G6152 | Glucose |
| Disodium phosphate | Na2HPO4 | ||
| Easy Grip Petri Dish | Falcon Corning | 351008 | 35 mm dish |
| Edetate Disodium | Avantor | 8994-01 | EDTA |
| Extra Fine Bonn Scissors | Fine Science Tools | 14084-08 | Straight, Sharp/Sharp, non-serrated, 13mm cutting edge scissors |
| Fetal Bovine Serum | Atlanta biologicals | S10250 | FBS |
| Gentamicin Reagent Solution | gibco | 15710-064 | Gentamicin |
| GlutaMAX-I (100X) | gibco | 35050-061 | Glutamax |
| Gold Seal Micro Slides | Gold Seal | 3039 | 25 x 75mm slides |
| Gonadotropin, From Pregnant Mares' Serum | Sigma | G4877 | eCG |
| hCG recombinant | NHPP | AFP8456A | hCG |
| Hyaluronidase, Type IV-S: From Bovine Testes | Sigma-Aldrich | H3884 | Hyaluronidase |
| Jewelers Style Forceps | Integra | 17-305X | Forceps 4-3/8", Style 5F, Straight, Micro Fine Jaw |
| L-(+)-Lactic Acid, free acid | MP Biomedicals, LLC | 190228 | L-Lactate |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | MgSO4-7H2O |
| MEM Nonessential Amino Acids | Corning | 25-025-Cl | NEAA |
| Microscope Cover Glass | Fisher Scientific | 12-542-C | 25 x 25x 0.15 mm cover slips |
| Mm-Nanog-O2-C2 RNAscope Probe | Advanced Cell Diagnostics | 501891-C2 | Nanog Probe |
| Mm-Pou5f1-O1-C3 RNAscope Probe | Advanced Cell Diagnostics | 501611-C3 | Pou5f1 Probe |
| MOPS | Sigma-Aldrich | M3183 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Paraformaldehyde |
| PES 0.22 um Membrane -sterile | Millex-GP | SLGP033RS | 0.22 um filters |
| Polyvinylpyrrolidone | Sigma-Aldrich | P0930 | PVP |
| Potassium chloride | Sigma-Aldrich | 60128 | KCl |
| Potassium phosphate monobasic | Sigma-Aldrich | 60218 | KH2PO4 |
| Prolong Gold antifade reagent | invitrogen | P36934 | Antifade reagent without DAPI |
| RNAscope DAPI | Advanced Cell Diagnostics | 320858 | DAPI |
| RNAscope FL AMP 1 | Advanced Cell Diagnostics | 320852 | Amplifier 1 |
| RNAscope FL AMP 2 | Advanced Cell Diagnostics | 320853 | Amplifier 2 |
| RNAscope FL AMP 3 | Advanced Cell Diagnostics | 320854 | Amplifier 3 |
| RNAscope FL AMP 4 ALT A | Advanced Cell Diagnostics | 320855 | Amplifier 4 ALT A |
| RNAscope FL AMP 4 ALT B | Advanced Cell Diagnostics | 320856 | Amplifier 4 ALT B |
| RNAscope FL AMP 4 ALT C | Advanced Cell Diagnostics | 320857 | Amplifier 4 ALT C |
| RNAscope Fluorescent Multiplex Detection Reagents Kit | Advanced Cell Diagnostics | 320851 | FISH Reagent Kit |
| RNAscope Probe 3-plex Negative Control Probe | Advanced Cell Diagnostics | 320871 | Negative Control |
| RNAscope Probe 3-plex Positive Control | Advanced Cell Diagnostics | 320881 | Positive Control |
| RNAscope Probe Diluent | Advanced Cell Diagnostics | 300041 | Probe Diluent |
| RNAscope Protease III | Advanced Cell Diagnositics | 322337 | Protease III |
| RNAscope Protease III & IV Reagent Kit | Advanced Cell Diagnostics | 322340 | FISH Protease Kit |
| RNAscope Protease IV | Advanced Cell Diagnostics | 322336 | Protease IV |
| S/S Needle with Luer Hub 30G | Component Supply Co. | NE-301PL-50 | blunt 30 gauge needle |
| Sodium bicarbonate | Sigma-Aldrich | S6297 | NaHCO3 |
| Sodium chloride | Sigma-Aldrich | S6191 | NaCl |
| Sodium hydroxide | Sigma-Aldrich | 306576 | NaOH |
| Sodium pyruvate, >= 99% | Sigma-Aldrich | P5280 | Pyruvate |
| Solution 6 Well Dish | Agtechinc | D18 | 6 well dish |
| Taurine | Sigma-Aldrich | T8691 | Taurine |
| Tissue Culture Dish | Falcon Corning | 353002 | 60 mm dish |
| Triton X-100 | Sigma-Aldrich | X100 | Triton X-100 |
Riferimenti
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proceedings of the National Academy of Sciences. 96 (16), 9236-9241 (1999).
- MacK, E. M., Smith, J. E., Kurz, S. G., Wood, J. R. CAMP-dependent regulation of ovulatory response genes is amplified by IGF1 due to synergistic effects on Akt phosphorylation and NF-kB transcription factors. Reproduction. 144 (5), 595-602 (2012).
- Xie, F., Timme, K. A., Wood, J. R. Using Single Molecule mRNA Fluorescent in Situ Hybridization (RNA-FISH) to Quantify mRNAs in Individual Murine Oocytes and Embryos. Scientific Reports. 8 (1), 7930 (2018).
- Ruebel, M. L., et al. Obesity modulates inflammation and lipidmetabolism oocyte gene expression: A single-cell transcriptome perspective. Journal of Clinical Endocrinology and Metabolism. 102 (6), 2029-2038 (2017).
- Borensztein, M., Syx, L., Servant, N., Heard, E. . Mouse Oocyte Development. 1818, 51-65 (2018).
- Jansova, D., Tetkova, A., Koncicka, M., Kubelka, M., Susor, A. Localization of RNA and translation in the mammalian oocyte and embryo. PLoS ONE. 13 (3), 1-25 (2018).
- Player, A. N., Shen, L. P., Kenny, D., Antao, V. P., Kolberg, J. A. Single-copy gene detection using branched DNA (bDNA) in situ hybridization. Journal of Histochemistry and Cytochemistry. 49 (5), 603-611 (2001).
- Wang, F., et al. RNAscope: A novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Itzkovitz, S., van Oudenaarden, A. Validating transcripts with probes and imaging technology. Nature Methods. 8 (4), S12-S19 (2011).
- Derti, A., et al. ProbeDesigner: for the design of probesets for branched DNA (bDNA) signal amplification assays. Bioinformatics. 15 (5), 348-355 (1999).
- Larson, B., Malayter, D., Shure, M. Multiplexed Detection of Cytokine Cancer Biomarkers using Fluorescence RNA In Situ Hybridization and Cellular Imaging. BioTek Application Notes. , 1-5 (2016).
- Buxbaum, A. R., Wu, B., Singer, R. H. Single β-Actin mRNA Detection in Neurons Reveals a Mechanism for Regulating Its Translatability. Science. 343 (6169), 419-422 (2014).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Herrick, J. R., Paik, T., Strauss, K. J., Schoolcraft, W. B., Krisher, R. L. Building a better mouse embryo assay: effects of mouse strain and in vitro maturation on sensitivity to contaminants of the culture environment. Journal of Assisted Reproduction and Genetics. 33 (2), 237-245 (2016).
- Pohlmeier, W. E., Xie, F., Kurz, S. G., Lu, N., Wood, J. R. Progressive obesity alters the steroidogenic response to ovulatory stimulation and increases the abundance of mRNAs stored in the ovulated oocyte. Molecular Reproduction and Development. 81 (8), 735-747 (2014).
- Xie, F., Anderson, C. L., Timme, K. R., Kurz, S. G., Fernando, S. C., Wood, J. R. Obesity-dependent increases in oocyte mRNAs are associated with increases in proinflammatory signaling and gut microbial abundance of lachnospiraceae in female mice. Endocrinology. 157 (4), 1630-1643 (2016).
- Hirao, Y., Yanagimachi, R. Detrimental effect of visible light on meiosis of mammalian eggs in vitro. Journal of Experimental Zoology. , (1978).
- Takenaka, M., Horiuchi, T., Yanagimachi, R. Effects of light on development of mammalian zygotes. Proceedings of the National Academy of Sciences. 104 (36), 14293-14293 (2007).
- Komminoth, P., Werner, M. Target and signal amplification: Approaches to increase the sensitivity of in situ hybridization. Histochemistry and Cell Biology. 108 (4-5), 325-333 (1997).
- Hornick, J. E., Duncan, F. E., Shea, L. D., Woodruff, T. K. Multiple follicle culture supports primary follicle growth through paracrine-acting signals. Reproduction. 145 (1), 19-32 (2013).
- Vlasova-St. Louis, I., Bohjanen, P. Feedback Regulation of Kinase Signaling Pathways by AREs and GREs. Cells. 5 (1), 4 (2016).
- Gilbert, C., Svejstrup, J. Q. RNA Immunoprecipitation for Determining RNA-Protein Associations In Vivo. Current Protocols in Molecular Biology. 75 (1), 27.4.1-27.4.11 (2006).
- Kwon, S., Chin, K., Nederlof, M., Gray, J. W. Quantitative, in situ analysis of mRNAs and proteins with subcellular resolution. Scientific Reports. 7 (1), 16459 (2017).
- Voigt, F., et al. Single-Molecule Quantification of Translation-Dependent Association of mRNAs with the Endoplasmic Reticulum. Cell Reports. 21 (13), 3740-3753 (2017).
- Halstead, J. M., Wilbertz, J. H., Wippich, F., Lionnet, T., Ephrussi, A., Chao, J. A. TRICK: A Single-Molecule Method for Imaging the First Round of Translation in Living Cells and Animals. Methods in Enzymology. 572, (2016).
- Cookson, W., Liang, L., Abecasis, G., Moffatt, M., Lathrop, M. Mapping complex disease traits with global gene expression. Nature Reviews Genetics. 10 (3), 184-194 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: the next challenge. Nature Reviews Genetics. 11, 855 (2010).
- Freimer, N., Sabatti, C. The Human Phenome Project. Nature Genetics. 34, 15 (2003).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon