Method Article
השימוש יחיד מולקולה פלורסנט ב באתרו הכלאה (SM-דג) כדי mRNAs Quantify ו Localize ב Oocytes מאתר
In This Article
Summary
Reproducibly לספור את המספרים של mRNAs ב oocytes בודדים, מולקולה בודדת RNA פלורסצנטיות באתרו הכלאה (RNA-דג) היה אופטימיזציה עבור תאים שאינם מחסידי. Oocytes נאספו, hybridized עם הגששים ספציפית התעתיק, לכמת באמצעות תוכנה כימות של התמונה.
Abstract
שיטות בשימוש שגרתי לכמת mRNA oocytes, העוברים כוללים תגובת שרשרת של פולימראז הפוכה-תמלול דיגיטלי (dPCR), כמותיים, בזמן אמת RT-PCR (RT-qPCR), רצפי RNA. כאשר שיטות אלה נעשות באמצעות oocyte יחיד או העובר, נמוך-עותק mRNAs לא אמינה מזוהים. כדי להתגבר על בעיה זו, oocytes או עוברי יכול להיות איחדו יחד לניתוח; עם זאת, זה מוביל לעתים קרובות השתנות גבוהה בין הדגימות. ב פרוטוקול זה, אנו מתארים את השימוש זריחה בהכלאה באתרו (דגים) באמצעות הכימיה DNA מסועף. טכניקה זו מזהה התבנית המרחבי של mRNAs בתאים בודדים. כאשר הטכניקה זה משולב עם מציאת מקום ותוכנות מחשב מעקב, השפע של mRNAs בתא גם ניתן לכמת. בעזרת טכניקה זו, יש השתנות מופחתת בתוך קבוצת הניסוי, פחות oocytes, העוברים נדרשים לזהות הבדלים משמעותיים בין קבוצות הניסוי. SM-DNA זמינים מסחרית-דגים ערכות מוטבו לזהות mRNAs רקמות משרטוטי או תאים חסיד בשקופיות. עם זאת, oocytes אינן מתיישבות ביעילות על שקופיות, כמה ריאגנטים בערכה היו קשים מדי וכתוצאה מכך oocyte פירוק. כדי למנוע את פירוק, נעשו מספר שינויים לערכה דגים. באופן ספציפי, oocyte permeabilization ושטוף מאגרים המיועדים את immunofluorescence של oocytes, העוברים החליף את המאגרים קניינית. Permeabilization, שוטף, וכן incubations עם המחקרים וכל מגבר בוצעו ב- 6-ובכן צלחות, oocytes הונחו על שקופיות בסוף הפרוטוקול באמצעות מדיה הרכבה. שינויים אלה הצליחו להתגבר על המגבלות של ערכת זמינים מסחרית, בפרט, פירוק oocyte. כדי במדויק, reproducibly לספור את מספר mRNAs ב oocytes בודדים, שימש תוכנות מחשב. יחד, פרוטוקול זה מייצג חלופה PCR ורצף כדי להשוות את הביטוי של הפרוטוקולים ספציפי בתאים בודדים.
Introduction
רוורס טרנסקריפטאז תגובת שרשרת פולימראזית (PCR) כבר תקן זהב עבור כימות mRNA. שני מבחני, PCR (dPCR) דיגיטלי1 ו זמן אמיתי כמותיים PCR (qPCR)2 משמשים כיום. של שתי טכניקות PCR, dPCR יש רגישות רבה יותר מאשר qPCR רומז כי זה יכול לשמש כדי למדוד את שפע mRNA בתאים בודדים. עם זאת, בידיים שלנו, הפיק dPCR ניתוח של שפע נמוך mRNAs בבריכות של 5-10 oocytes לכל כל מדגם ניסיוני נתונים עם הפארמצבטית נמוך ווריאציה גבוהה3. . זה ככל הנראה בגלל השגיאה ניסיוני המשויך החילוץ-RNA ויעילות שעתוק במהופך. רצפי RNA גם בוצעה באמצעות עכבר אחת oocytes האנושי4,5. טכניקה זו דורשת cDNA הגברה השלבים הנדרשים עבור הדור ספריה אשר סביר להניח מגביר את השתנות בתוך קבוצת הניסוי. יתר על כן, שפע נמוך תעתיקים לא ניתן לזיהוי. למרות רצף המחירים ירדו בשנים האחרונות, הוא עדיין יכול להיות ושדורשת עקב העלות הגבוהה של ביואינפורמטיקה ניתוחים. לבסוף, mRNA לוקליזציה היא תהליך דינמי עם שינויים מרחביים התורמים חלבון פונקציה6. לכן, יצאנו לאמץ טכניקה היה לייצר אמצעי כמותית מדויקת לשחזור ולוקליזציה של הפרט mRNAs ב oocytes יחיד.
DNA מסועף מצמידים זריחה בהכלאה באתרו מגביר את האות זריחה במקום RNA/cDNA הגברה המאפשרת זיהוי mRNAs יחיד תאים בודדים- 7,-8,-9. הבדיקה מבוצעת דרך סדרה של הכלאה הגברה (באמצעות DNA קנים), קרינה פלואורסצנטית תיוג צעדים כדי להגביר את האות של קרינה פלואורסצנטית7. הטכניקה מתחילה עם קשירה של זוגות בדיקה oligonucleotide 18 - 25-לבסיס כי הם משלימים על ספציפיות mRNA3,8,10. 15-20 זוגות בדיקה מיועדים ירידה לפרטים הבטחת כל תורגם על-ידי היעד. הכלאה mRNA ספציפי ואחריו הגששים קדם מגבר, מגבר כי הטופס תצורה מסועף. כ, fluorophores תווית 400 לאגד כל מגבר, וכתוצאה מכך עלייה 8000-fold ב פלורסצנטיות המאפשר זיהוי של הפרט mRNAs (איור 1)11.
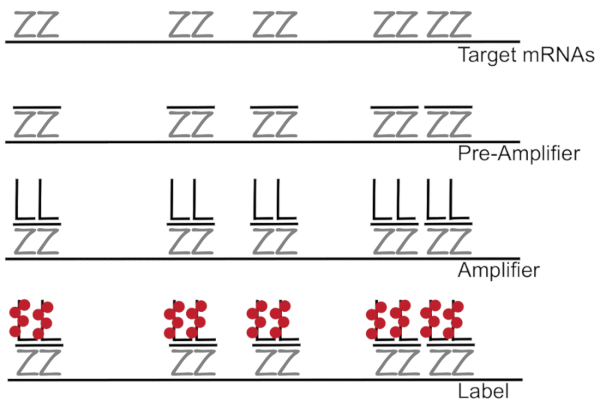
איור 1: סכימטי של פרוטוקול SM-דג- הכלאה רציפים של התעתיק בדיקה ספציפיים, כפועל DNA מגבר ו fluorophore ליעד mRNA מוצג. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
מחקרים קודמים באמצעות קרינה פלואורסצנטית מולקולה בודדת ב באתרו הכלאה (SM-דג) לשפות אחרות β-אקטין mRNAs נוירונים בודדים12 , וירוס הפפילומה האנושי DNA של סרטן צוואר הרחם תא קווים7. תוכנת מחשב במקום למצוא תוכנית מעקב אחר מזהה אות ניאון punctate בודדים, שימש בהצלחה כדי לכמת את מספר mRNAs כל3,תא13.
בהתבסס על התוצאות של גילוי mRNA נוירונים12, אנחנו שיערו כי SM-דג יוכיח כלי שימושי כדי quantitate רמות תורגם מאתר oocytes, העוברים כולל שפע נמוך mRNAs. עם זאת, הטכניקה ממוטבת לשימוש עם תאים קבוע חסיד, פורמלדהיד קבוע פרפין משובצים קטעי רקמה (FFPE). Oocytes לא יכולה לדבוק שקופית, גם כאשר הם מצופים פולי-L-ליזין. יתר על כן, הן שבירות יותר תאים סומטיים ומקטעים רקמות וכתוצאה מכך פירוק התא כאשר נתון של המאגרים קניינית ערכות זמינים מסחרית3. כדי להתגבר על האתגרים הללו, oocytes היו קבוע, מועברים באופן ידני בין טיפות של המאגרים. יתר על כן, מאגרי permeabilization ושטוף ערכות הוחלפו כדי להפחית את פירוק התא. שתגבש רגשים נרכשים לצד ערכת דגים או תעתיקים ספציפי ניתן לבקש. כל ערכת בדיקה קניינית זמין באחד בשלושה ערוצי קרינה פלואורסצנטית (C1, C2, C3) כדי לאפשר ריבוב. בניסוי הנוכחי, oocytes מאתר היו שהוכתמו כפול, כימות באמצעות בדיקה C2 Nanog ובדיקה C3 Pou5f1 . רגשים אלו נבחרו מבוסס על הביטוי המדווחת של Nanog , Pou5f1 oocytes, העוברים. בסיום השלבים הכלאה, oocytes היו ממוקמים בתוך טיפות אנטי-לדעוך הרכבה מדיה עבור יישום בשקופיות היסטולוגית. תמונות קונאפוקלית שימשו כדי לכמת את מספר punctate אותות פלואורסצנט אשר מייצגים mRNAs בודדים. בנוסף לכימות של mRNAs, ההדמיה הראתה גם את התפוצה המרחבית של ה-mRNA ספציפי בתא, אילו שיטות כימות אחרים של RNA הם הצליחו. להגיע להישגים. טכניקה זו הוכיח יש השתנות נמוך בתוך קבוצת הניסוי המאפשר שימוש מספר קטן יחסית של oocytes בכל קבוצה ניסיונית כדי לזהות הבדלים משמעותיים בין קבוצות הניסוי3.
Protocol
ההליכים בבעלי חיים היו נבדקו ואושר על ידי טיפול בעלי חיים מוסדיים ועל שימוש הוועדה-אוניברסיטת נברסקה, כל השיטות בוצעו בהתאם להנחיות הרלוונטיים ולתקנות. במחקר זה, outbred CD-1 עכברים הייתה גישה ad libitum צ'או מכרסמים רגיל ומים; הם היו מתוחזקים ביומן 12:12 כהה: אור מחזור.
1. הכנת מדיה הנדרשים
- על בסיס מדיה (אום), להוסיף 100 מ מ NaCl, 5 מ מ אשלגן כלורי, 0.5 מ מ ח'2PO4ו- 1.7 מ"מ CaCl2-2 H2O במים סטריליים 100 מ.
הערה: ניתן לאחסן OMM בינוני עד כחודש. - התקשורת מלאה (OMOPS), להוסיף 20 מ מ 3-morpholinopropane-1-sulfonic חומצה (MOPS), 1.2 מ מ MgSO4-7 H2O, גלוקוז 0.5 מ מ, 6 מ מ L-לקטט, 1 מ מ ala-אך זה לא נגמר, טאורין, 0.1 מ"מ x 1 שאינם חיוניים חומצות אמינו (NEAA), 0.01 מ מ ethylenediaminetetraacetic (חומצה EDTA), חומצה אלפא ליפואית מיקרומטר 10, 10 µg/mL מדולל גנטמיצין, 21 מ מ 1 מ' NaOH, 5 מ מ NaHCO3, 0.2 מ מ פירובט, 0.5 מ מ ציטראט, BSA 4 מ"ג/מ"ל FAF על 1:10 לדילול OMM במים סטריליים עבור הנפח הכולל של 100 מ ל. לעקר את המדיום עם מסנן 0.22 מיקרומטר.
הערה: OMOPS ניתן לאחסן עד 1 לשבוע. - עבור המדיום אחזקות (HM) להוסיף סרום שור העוברי 5% OMOPS. לעשות 2 מ ל HM לכל העכבר.
- פתרון hyaluronidase הוסיפו 0.1 מ"ג/מ"ל של hyaluronidase נגזר מן האשכים שור, 1 מ ל HM.
- המאגר פיקסציה לשלב 4% paraformaldehyde ב- 10 מ"ל של PBS x 1 יחד עם 0.1% העובר כיתה פוליוינילפירולידון (PVP)14.
- כדי להכין מאגר שטיפת 50 מ ל (WB), להוסיף חומרים פעילי שטח ללא יונית 0.1% ו- 0.1% PVP 1 x PBS14.
- כדי להכין 10 מ"ל permeabilization מאגר, להוסיף 1% שאינם - יונית חומרים פעילי שטח PBS 1 x14.
הערה: המאגרים רוחצים את permeabilization המתואר לעיל להחליף את המאגרים קניינית ערכות זמינים מסחרית.
2. אוסף של oocytes ovulated של עכברים הנשי
- הכנה:
- לעורר עכברים נקבה 5-8 שבועות של גיל על-ידי הזרקת בקרום הבטן (IP) של 5 IU סוסים שליה גונדוטרופין (א) ואחריו 5 IU גונדוטרופין כוריוני אנושי (hCG) 44-48 h15,מאוחר יותר16.
- לשמור על 35 מ מ פטרי המכילות 2 מ של HM על צלחת ההתחממות 37 º C. פיפטה אחת, 100-µL טיפה של HM המכיל מדולל hyaluronidase ואחריו שלוש, 50 µL טיפות של HM ללא hyaluronidase בצלחת פטרי 60 מ מ. במקום לוחות המכילים טיפות על התחממות 37 ° C צלחת לפני השימוש.
הערה: הטיפות hyaluronidase צריכה להיעשות רק לפני ניתוח כל זוג oviduct כדי למנוע אידוי את הריכוז של הרכיבים של HM עם או בלי hyaluronidase.
- המתת חסד עכברים, h 16 אחרי הזריקה IP של hCG, שימוש יתר איזופלוריין ואחריו נקע בצוואר הרחם.
- נקה את העכבר באמצעות אתנול 70%. לחשוף את חלל הבטן והמחש את מערכת הרבייה הנשית. מחזיקים את השחלה עם מלקחיים, להסיר את הרצועות הרחם ואת רקמת שומן עודף מ סביב השחלה. לחתוך את oviduct מן הרחם ולמקם את זוג שחלה-oviduct HM חם בצלחת 35 מ מ.
- להסיר את השחלה כל רקמת שומן שמסביב. דמעה נפוחות אמפולת oviduct באמצעות מחט 27-מד ½ אינץ '. לדחוף את oviduct באתר של הקרע, מתחמי תא-oocyte תלולית (COCs) יסולקו. להעביר את oocytes ovulated, אשר נמצאים בחזקת להיות מפה של השני (MII) של מיוזה, לירידה 100 μL המכיל HM מדיה עם hyaluronidase באמצעות פיפטה של הפה (איור 2).

איור 2 : חלקי pipettor הפה המשמש להעברת oocytes. (א) את הפה. חתיכת (B) 0.22, 4 מ מ (ג) ליניקת אבובים (D) 1000 μL pipet קצה עם פילטר (E) 9" פסטר pipet. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
- Pipette את מתחמי תא oocyte-תלולית MII למעלה ולמטה ב hyaluronidase המכיל HM עם פיפטה בפה מכך cumulus תאים. העברת כל oocyte, ברגע שהם נטולי cumulus לתאים שטיפה זרוק HM המכיל רק באמצעות פיפטה את הפה. חזור על הפעולה עבור כל droplet לשטוף. אל תעביר מפוצלים או שקוף oocytes15.
הערה: חשוב להעביר את oocytes מכל טיפה בצלחת 35 מ מ עם כמו HM קטן ככל האפשר. זה נכון לכל העברה בפרוטוקול. Oocytes MII לא להישאר ב- hyaluronidase המכיל HM בינונית במשך יותר מדקה.
3. SM-דגים צביעה של Oocytes
- לתקן את oocytes טוב בודדים של צלחת 6-ובכן המכיל 500 µL מאגר קיבעון. להטביע 20 oocytes או פחות בתוך הבאר. תקופת דגירה של 20 דקות בטמפרטורת החדר.
הערה: כל שלב צביעת SM-דג מתרחשת בתוך ובכן בודדים בצלחת חרוט 6-. טוב. ודא oocytes לחלוטין המשוקע במאגרי ואני לא צף על המאגר. בכל שלב צריך להיות שבוצעו עם 20 oocytes או פחות מכל קידוח. - להעביר oocytes קבוע μL 500 שטיפת מאגר (WB שמתואר בשלב 1.6) למשך 10 דקות. חזור על 2 פעמים נוספות.
- דגירה oocytes במאגר permeabilization למשך 30 דקות בטמפרטורת החדר.
הערה: המאגר permeabilization שמתואר בשלב 1.7 מחליף את המאגר permeabilization הנימוס.- איסוף ערכות בדיקה, במהירות לסובב אותם למטה ב microcentrifuge. חמים כל בדיקה להגדיר עבור 10 דקות 40 ° צלזיוס המים הרותחים או חממה. מגניב כדי טמפרטורת החדר.
הערה: שלב זה צריך להתבצע במשך הדגירה permeabilization
- איסוף ערכות בדיקה, במהירות לסובב אותם למטה ב microcentrifuge. חמים כל בדיקה להגדיר עבור 10 דקות 40 ° צלזיוס המים הרותחים או חממה. מגניב כדי טמפרטורת החדר.
- לשטוף oocytes ב µL 500 של WB 10 דקות בטמפרטורת החדר.
- העברת oocytes 80 μL פרוטאז השלישי מאגר (זמין הערכה), זה מדולל 1:8 ב- 1 X PBS, למשך 30 דקות בטמפרטורת החדר.
הערה: אמצעי האחסון 80 µL מכסה במידה מספקת הבאר בודדים בצלחת 6-. טוב. - לשטוף oocytes ב µL 500 של WB 10 דקות בטמפרטורת החדר.
- לדלל את מערכות החללית ומחוממת של Nanog, Pou5f1, ו DapB (גן שליטה שלילי), 1:50 ב בדיקה diluent. תקופת דגירה oocytes ב 80 μL של המכשיר הספציפי תעתיק של 2 שעות ב- 40 מעלות צלזיוס.
הערה: כל ערכת בדיקה קניינית זמין באחד בשלושה ערוצי קרינה פלואורסצנטית (C1, C2, C3). Nanog ואת Pou5f1 הגששים תויגו עם C2, C3, בהתאמה. - חמים קניינית, מגבר 1 (1 אמפר), מגבר 2 (AMP2), מגבר 3 (AMP3) ומגבר 4-קרינה פלואורסצנטית (מגבר 4-FL) בטמפרטורת החדר.
הערה: שלב זה צריכה להתבצע במהלך בדיקה ספציפית התעתיק 2 שעות. - להעביר את oocytes μL 500 של WB, תקופת דגירה של 10 דקות בטמפרטורת החדר.
- דגירה oocytes ברצף במאגרי הגברה.
- דגירה oocytes ב 80 µL של AMP1 במשך 30 דקות ב- 40 מעלות צלזיוס oocytes העברה כדי 500 µL של WB 10 דקות בטמפרטורת החדר.
- דגירה oocytes ב µL 80 של AMP2 למשך 15 דקות ב- 40 מעלות צלזיוס. העברת oocytes µL 500 של WB 10 דקות בטמפרטורת החדר.
- דגירה של oocytes ב 80 µL של AMP3 במשך 30 דקות ב- 40 מעלות צלזיוס. העברת oocytes µL 500 של WB 10 דקות בטמפרטורת החדר.
הערה: השארית של הפרוטוקול מבוצעת בחושך כי כח-FL מכיל את fluorophore. כאשר עובד תחת המיקרוסקופ ויבתר, להפחית כמה שיותר את האור. - להוסיף oocytes 80 μL של AMP4-FL למשך 15 דקות ב- 40 מעלות צלזיוס.
הערה: AMP4-FL מסופק כאלטרנטיבה מאגר-A Alt-B (Alt-A), או Alt-סי בחר AMP4-FL המאגר תלוי על איזו פליטה גל היא הרצויה.
- לשטוף oocytes ב µL 500 של WB 10 דקות בטמפרטורת החדר. דגירה oocytes ב µL 80 של דאפי במשך 20 דקות בטמפרטורת החדר. לשטוף oocytes ב µL 500 של WB למשך 5 דקות בטמפרטורת החדר.
- פיפטה 12 µL של התקשורת נגד דהייה הרכבה במרכז שקופית מבלי להוסיף בועות הכימית. להעביר את oocytes עם כמה WB קטן ככל האפשר לתוך כלי התקשורת הרכבה ולהחיל על coverslip.
- להטות את coverslip בזווית, לאט ובעדינות למקם מעל הנוזל בשקופית. הימנעו מלחיצה על coverslip קשה מדי למנוע עיוות של oocytes, הקדמה של בועות.
- אחסן את השקופיות תיבה כהה לילה יבש בטמפרטורת החדר. מעיל קצות השקופיות לק ברור לאטום coverslip.
- השתמש מיקרוסקופ רגיל כדי למצוא oocytes על שקופיות, מעגל עם סמן קבוע.
הערה: שלב זה אינו נדרש, אך משפר את oocytes איתור בשקופית. לקבלת תוצאות מיטביות, תמונות שקופיות בתוך 1-5 ימים כמו האות פלורסנט יתחיל לדעוך.
4. עיבוד תמונה
- תמונה של oocytes תלת-ממדי, באמצעות z שלב קונפוקלית.
הערה: כדי לנתח במדויק את התמונות, כל שלב z להיות מיקרומטר 1.0/פרוסה. - שמור תמונות קונאפוקלית של nd2 דחוס או בודדים. קובצי TIFF עבור כל oocyte. שני הסוגים תמונה תואמים התוכנית עיבוד התמונה קוד פתוח, פיג'י.
- הורד והתקן את התוכנה פיג'י גישה פתוחה (https://imagej.net/Fiji/Downloads).
- לגרור קבצים nd2 פיג'י, לבחור hyperstack. אם תמונות קונאפוקלית נשמרו בתור. קובצי TIFF דלג לשלב 4.4.
הערה: כאשר הקובץ nd2 הוא נפל לתוך פיג'י מהרשימה הנפתחת hyperstack אמורה להופיע באופן אוטומטי. - לחץ על הכרטיסיה תמונה לבחור צבע, לחץ על פצל ערוצים כדי להפריד בין הערוצים פלורסנט של הקובץ nd2.
- צור בודדים. קובצי TIFF לכל z-פרוסה של oocyte בכל אחד מהערוצים פלורסנט. לחץ על הכרטיסיה תמונה , בחר את סטאקסולאחר לחץ על אוסף תמונות. לחץ על הכרטיסיה תמונה , בחר סוג, לחץ על צבע RGB כדי להמיר כל פרוסה z תמונת צבע RGB בודדים.
הערה: צבע RGB הוא מלאכותי, יכול להיות נבחרת הרצוי עבור כל גל פליטה. - שמור כל תמונה המומר. קובץ TIFF. מקם תמונות oocyte יחיד לכל ערוץ פלורסנט בתיקייה חדשה כדי למנוע בלבול במהלך תפרים (שלב 4.3).
- לגרור קבצים nd2 פיג'י, לבחור hyperstack. אם תמונות קונאפוקלית נשמרו בתור. קובצי TIFF דלג לשלב 4.4.
- לנרמל את אחד. תמונות TIFF עבור Pou5f1 ו- Nanog שימוש בתמונות שליטה שלילי (DapB).
הערה: נורמליזציה מתבצעת באמצעות בתוכנית לעריכת תמונות. הקפד להסיר רמות זהות פלורסצנטיות רקע מתמונה כל שליטה. - פתוח אחד מנורמל. קובץ TIFF בפיג'י לתפור יחד כל z-פרוסות עבור כל oocyte ב כל אורך הגל.
- לחץ על הכרטיסיה תוספים , בחר Stitching, ולחץ על רשת/אוסף (איור 3 א). בחר תמונות רציפים בתפריט הנפתח, לחץ על אישור (איור 3B).
- ספריית עיון, בחר את התיקיה המכילה את כל התמונות פרוסות z עבור oocyte בודדים-גל אחד (ראה שלב 4.3.4). לחץ על אישור.
- הזז את המחוון בתחתית התמונה תפור לערוץ הצבע המתאים עבור הגל המשמש ו ליצור את התמונה הסופית תפור RGB על-ידי לחיצה על התמונה, בחירת סוג, לחץ על צבע RGB.
הערה: תמונה זו ישמש עבור כימות פלורסצנטיות שמתואר בשלב 4.6 להלן.
- המירו את התמונה תפור לתמונה המוקרנת מקסימום 32 סיביות. לחץ על התמונה, בחר סוג, ולחץ על ' 32-bit (איור 3C). שמור את התמונה הזו חדשה. קובץ TIFF.
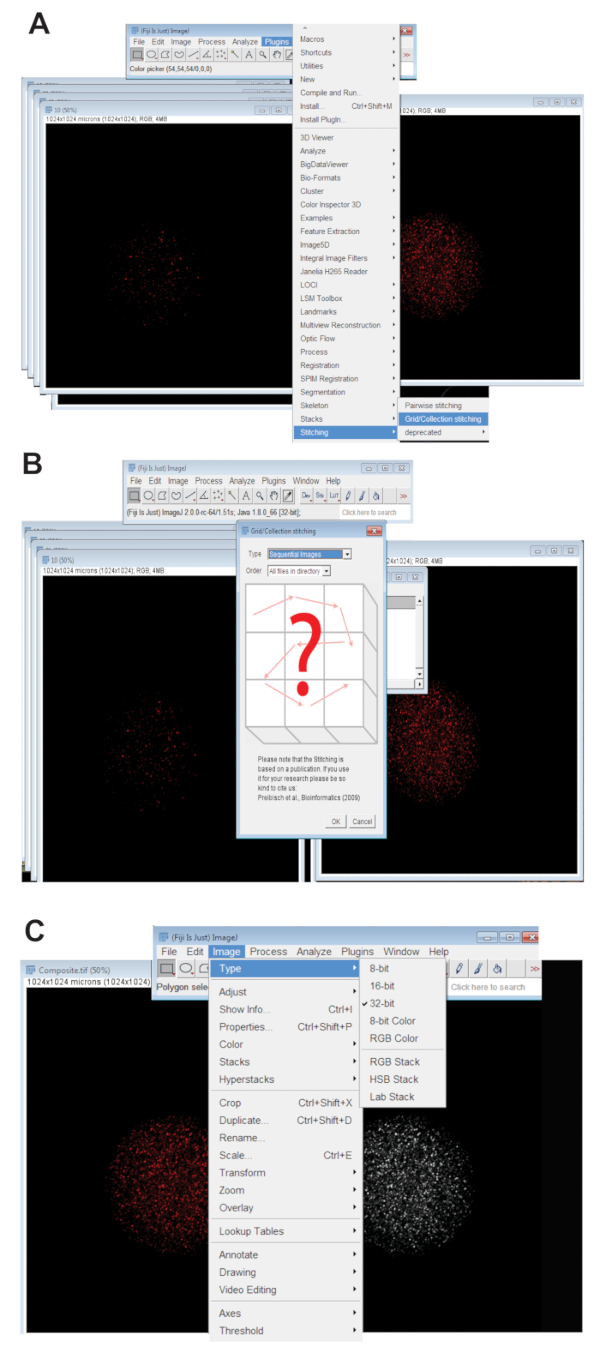
איור 3 : תפרים יחד של z קונאפוקלית-סדרת תמונות של oocytes. (א) המסך מציג את הכלי יישום plug-in רשת/אוסף בפיג'י שבה השתמשת כדי להפיק תמונות ללא הפרדות צבע של oocyte. (B) תמונות רציפים משתמש פלורסצנטיות חפיפה בין רציפים. קובצי TIFF כדי ליצור תמונה מורכבת. (ג) תמונה מורכבת נשמר כמו 32 סיביות. קובץ TIFF. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
- הורד והתקן את מציאת נקודה, מעקב אחר תוכנית13, אשר זמין מאתר האינטרנט של D.R. לארסון, חוקר במוסדות של הבריאות הלאומית המכון הלאומי לסרטן (https://ccr.cancer.gov/Laboratory-of-Receptor-Biology-and-Gene-Expression/daniel-r-larson). הורד והתקן את גישה פתוחה וירטואלי עבור מערכת ההפעלה של נתונים אינטראקטיביים שפה (מסוג IDL) אשר נדרש כדי להפעיל את המקום למצוא תוכנית מעקב (http://www.spacewx.com/pdf/idlvm.pdf).
- פתחו את התמונה 32 סיביות, תפור, אשר נוצר בשלב 4.6 (איור 4A), מציאת מקום, תוכנית מעקב. בחר מהרשימה הנפתחת Localize ולחץ Localize (איור 4B), אשר יחשב את מספר הנקודות נמצא בתמונה.
הערה: בכל מקום נספרים מייצג של mRNA בודדות. הגדרות הסף פוטון ותעודת הלהקה מוצגים צילום מסך (איור 4B). עבור פרוטוקול זה, שימש ברירת המחדל עבור כל הגדרה הסף. נציג חיובי ולנקודות רקע מוצגים (איור 4C).

איור 4 : כימות של mRNAs באמצעות נקודת מוצא ומעקב. (א) z בודדים-סדרת תמונות היו תפרו יחד כמתואר באיור 3 ונשמרו מקסימום 32 סיביות מוקרן. קובץ TIFF. (B) תמונה המשולב נפתח ב ספוט Finder ומעקב. בתרגום שימש כדי לספור את הנקודות פלורסנט (התיבה האדומה). סף ותעודת פוטון הלהקה מסומנים באמצעות התיבה הכחולה. (ג) החץ הכחול מצביע על איתות חיובי (מעל מפתן). החץ הלבן מראה של פלורסנט ספוט מתחת לסף וספרתי, לכן, לא. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
תוצאות
עם הסיום של הפרוטוקול, התוצאה תהיה תמונות בודדות מתוך קונאפוקלית z-סדרות (איור 4A ואיור 5), תפור תמונות (איור 4C), וסופרת mRNA (איור 4B). בעת ביצוע ריבוב, יהיו גם התמונות הממוזגות מציג את התווית עבור שני mRNAs שונים (איור 5). הסעיפים mRNA נוצרים באמצעות תמונות התפר שנוצר על ידי פיג'י (איור 3) המקומות זריחה punctate נספרים באמצעות מציאת מקום תוכנית מעקב (איור 4B).
הסעיפים mRNA מנותחים לאחר מכן באמצעות כלי ניתוח נתוני סטנדרטיים. ב פרוטוקול זה, סיווגנו את n = 12 oocytes עם Pou5f1 ו- Nanog. חישוב הממוצע התוצאות עבור כל ה-mRNA ומחושבים שגיאת התקן של הממוצע. הנתונים שנאספו ב פרוטוקול זה הראו 775 ± 26 SEM Pou5f1 תמלילים וגיליון הציונים SEM Nanog ± 5 113 MII oocytes (איור 5). מבחן t של סטודנט נקבע שהבדל משמעותי סטטיסטית ב- mRNA נחשב בין Pou5f1 לבין Nanog. חשוב לציין, היו. אין כתמים זוהה n = 5 oocytes המסומנות עם בדיקה DapB (קרי, בקרה שלילית). הערה שגיאת התקן קטן באמצעות רק 12 oocytes בודדים. הרגישות של וזמינותו מודגשת גם על ידי זיהוי לשחזור חיובי של Nanog(איור 5). ניסויים dPCR הקודם לא זיהה reproducibly Nanog המציין המספר של Nanog mRNAs ב oocyte בודדים הוא מתחת לסף גילוי באמצעות dPCR 3.
בניסויים פיילוט, הבחנו פלורסצנטיות מופחת אם היה עיכוב בין הקיבעון של oocytes, חניכה של פרוטוקול SM-דג. באופן דומה, אם לא נעשה שימוש המאגרים הכלאה קניינית, החללית ואת ההגברה DNA מסועף תזין את התא וכתוצאה מכך פלורסצנטיות מוקף סביב קרום הפלזמה של oocyte. . זה ככל הנראה בגלל צבירה של ה-DNA מסועף. קרינה פלואורסצנטית מחויג סביב קרום oocyte תגרום גם אם יש עניים permeabilization של קרום פלזמה. השפלה אופטימלית של החלבון קשור mRNAs נדרש גם. המאגר פרוטאז המסופק בערכה SM-דג נמצא ריכוז אופטימלי לטיפול של מקטעים רקמות, תאים חסיד. עם זאת, בעת שימוש שאינו דוגל תאים, חשוב לזיהוי מדעית דילול פרוטאז הטוב ביותר. מאגר פרוטאז מעט מדי יכול לגרום undercounting של mRNAs עקב הנגישות המסכן של המכשיר ל mRNA. באופן דומה, פרוטאז מדי עלול לגרום השפלה של לא רק חלבונים מאוגדים של mRNA אבל גם destabilization של mRNAs. ב פרוטוקול זה, בדקנו גילוי mRNA באמצעות עיקול טיטור מדולל (1:1), 1:4, 1:8 ו 1:12 פרוטאז מדולל מאגר ב- PBS 1 x (n = 2-3 oocytes לכל דילול). הממוצע פלורסנט ביטוי של mRNA Pou5f1 ב MII oocytes היה SEM ± 42 169 (מדולל), ± 36 176 SEM (1:4), ± 18 308 SEM (1:8) ו ± 24 445 SEM (1:12) (איור 6). דילול פרוטאז בשימוש פרוטוקול זה היה 1:8 כפי שהוא הראה את הווריאציה הנמוך.

איור 5 : Pou5f1 ו- Nanog mRNA ב MII oocyte. (א). להחליפן בתמונות של התמונה מסדרת z האמצעי מוצגים בצד השמאל. Pou5f1 מזוהה באדום (647 ננומטר) גל בזמן Nanog מזוהה בירוק (488 ננומטר) אורך הגל. דאפי מכתים של כרומוזומים מיושרים על מפה של השני בכישור, האופייניים oocyte MII, מוצג בלבן. היה לא מכתים עבור DapB גם את 647 ננומטר או 488 ננומטר פליטה אורך הגל. (ב') המספר של mRNAs Pou5f1 , Nanog מוצגות כפי מתכוון SEM (n = 12 oocytes); לא היה שום גילוי (N.D) של DapB. * מציין P < 0.05, סולם בר הוא 10 מיקרומטר. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 6 : טיטור אמפירי פרוטאז המאגר כדי למטב את ספירת מדויק mRNAs. תמונות נציג של SM-דג של Pou5f1 ב MII oocytes. דאפי מכתים מראה כרומוזומים מיושרים בכישור MII. Oocytes היו מודגרות עם מדולל (1:1), 1:4, 1:8 או 1:12 דילולים פרוטאז השלישי המאגר. המספר של Pou5f1 mRNAs (n = 2-3 oocytes) נספרו ו הממוצע SEM מוצג. סולם בר הוא 10 מיקרומטר. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.

איור 7 : העברה של MII oocytes דרך מאגרי קיבוע פרוטאז, הכלאה בבארות של צלחת 6-ובכן. צורת מכל קידוח מוצג. (א) תאים לצוף כאשר לראשונה נוספו מאגרי (B) ועלייתו של oocytes במאגרי. (ג) Oocytes להתיישב לתחתית הבאר במהלך תקופת הדגירה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של הדמות הזאת.
Discussion
סדרה של צעדים קלים במהלך הפרוטוקול יבטיח פלורסצנטיות מוצלחת וספירות מדויק של mRNAs. ראשית, הפרוטוקול חייב להתבצע מיד לאחר איסוף של קיבוע oocytes. שימו לב כי PVP נוסף למאגר קיבוע paraformaldehyde 4% כדי למנוע oocytes נצמדים אחד לשני. מצאנו כי יש צורך לבצע את הניסוי מיד אחרי אוסף קיבוע oocytes. כל עיכוב תוצאות הרבה נמוך יותר קרינה פלואורסצנטית האות, כי כתוצאה undercounting של הפרוטוקולים. . זה בחלקו בשל RNA השפלה. Oocytes לא יותר מ 20 יש להעביר טוב בצלחת. ובכן 6 בבת אחת, כל טוב להשתמש בהם רק פעם אחת. הדגירה פעמים צריכה גם להיות מדויק מלווה ללא קיצור או הארכה של כל שלב. החריג הוא השלבים מאגר שטיפת; oocytes יכול להשאיר במאגר שטיפת במשך זמן ממושך מבלי לשנות את תוצאות הניסוי. הגששים SM-דג זמינות בשלושה ערוצי פלורסצנטיות C1, C2, C3. עבור ריבוב, אל תערבב בין הגששים שיש לאותו ערוץ תג. לכך שני רגשים ואזוריט באותו פולטות גל עיבוד ניתוח בלתי אפשרי מכיוון שיהיה שום דרך להבדיל בין קבוצות בדיקה. הגששים בקרה חיובית מיועד נגד גנים משק בית זמינות כל שלושת הערוצים. הגששים בקרה שלילית (למשל, DapB) זמינים גם בערכות מעורבבים מראש להכיל תג עבור כל שלושת הערוצים. הניסוי צריך להתבצע בתאורה עמומה כמו oocytes גם רגיש אור לאחר הסרת מ17,oviduct18. לאחר התוספת של fluorophores המחוברים AMP4, הפעולות יש לבצע עם אור קטן ככל האפשר למנוע הלבנת של fluorophore. לבסוף, כאשר הרכבה oocytes על גבי שקופיות היסטולוגית, בזהירות למקם את coverslip כדי למנוע עיוות של oocyte היווצרות בועות אשר עלולים להפריע הדמיה. אם אתה מתקשה למנוע עיוות תא, מפרידי שקופית אמור לשמש על מנת לשמור על הצורה הכדורית של oocyte.
שינוי מהותי אחד של הפרוטוקול היא ההחלפה של permeabilization ולאחר שטיפת מאגרי המסופק בערכה זמינים מסחרית. קניינית פרוטאז, הכלאה המאגרים סיפק סביבות קשות עבור oocytes אך נדרשים להצלחת הפרוטוקול. אם לא נעשה שימוש, מגברים אינם יכולים להיכנס לתא אשר, ככל הנראה עקב צבירה של ה-DNA מסועף. העברת את oocytes מסביבות קשות אלה permeabilization מתון יחסית, מאגרי לשטוף, המיועדים immunofluorescence14, הוכיח נאותה ההצלחה של הפרוטוקול ו באותו הזמן למנוע פירוק של oocytes. כי oocytes, העוברים של אינן מתיישבות שקופית היסטולוגית, שינוי מהותי אחר היה הצבת את oocytes לתוך המאגרים בתוך טוב של צלחת תרבות. השתמשנו צלחת תרבות 6-ובכן העובר. כל טוב בצלחת יש צמצום והתחתונים משופע הצדדים ואת של 8 מ מ שטוח (איור 7), אשר משפרת את oocyte שחזור. הדבר חשוב במיוחד כפי oocytes לאבד את תכונותיהם עקשן ולהיות כמעט שקוף במאגרי הכלאה.
בעת העברת oocytes טוב טוב, חשוב לוודא כי oocytes באופן מלא טובע פתרונות לכל היטב כמו תאים יצוף כאשר הועבר קודם כל טוב (איור 7 א). ברגע שהם נמצאים מכנית submersed לתוך המאגר, (איור 7 ב), הם ישקעו לתחתית הבאר בסוף הדגירה (איור 7C). החריג הוא כח 1 ו- 3 אמפר; כאשר מכנית submersed לעשות oocytes לא לגמרי להתיישב בתחתית הבאר. כדי למצוא את oocytes האלה, ייתכן שיהיה עליך לשנות את המטוס של המוקד. פיפטה בקפידה, לספור את מספר התאים מועבר למניעת אובדן.
מספר טכניקות דגים מולקולה בודדת, המדגישים את האות פלורסנט מאשר cDNA, כולל מסועף DNA בדיקות, הביוכימיה, כבר פיתח 9,19. ערכות זמינים מסחרית יש אופטימיזציה בשיטת ה-DNA SM-דגים קנים לגילוי לשחזור של הפרט mRNAs רקמות מקטעים או תאים חסיד בשקופית היסטולוגית. הפרוטוקול המתואר כאן שונתה לשימוש עם תאים שאינם מחסידי בודדים (למשל, oocytes, של עוברי)3. פעולה זו מאפשרת לא רק כימות לשחזור וספציפית, אלא גם לוקליזציה של ה-mRNA בתוך oocyte. אמנם זה היתרון וזמינותו, ישנם כמובן מגבלות. לדוגמה, בניגוד RNA-רצף, זה לא יכול לזהות mRNAs רומן. מגבלה נוספת של הפרוטוקול הוא הזמינות של הגששים התעתיק הספציפי. הגששים קניינית זמינים מסחרית של החברות שמוכרות את ערכות SM-דג. ישנן מספר בדיקות שאינן מוכנות מראש. אחרים יכולים להיות מתוכנן על ידי החברה עבור כל mRNA המבואר באמצעות אלגוריתם אובייקטיבית של10. עם זאת, אם mRNA הוא לקוי וסודרו קשה כדי עיצוב רגשים עם ירידה לפרטים גבוה. תחפש ברישומים קצר זה יכול להיות גם קשה לזהות מספיק זוגות בדיקה לא cross-react עם אחרים תעתיקים הפחתת יחודיות של וזמינותו. באופן דומה, ייתכן מספר קטן יותר של ערכות בדיקה מספיקות לייצר אות זריחה מעל הסף לצורך זיהוי חיובי מציאת מקום, תוכנית מעקב. ברוח זו זהה, התעתיק גרסאות לא ניתן לאתרם באמצעות שיטה זו.
למרות המגבלות שתוארו לעיל, ישנם מספר יישומים עבור SM-דג. לדוגמה, יכול לאמת נתונים מתא יחיד RNA-רצף במיוחד כאשר מספרי הטלפון הנייד הם קטנים קשה להשיג (למשל, oocytes, העוברים). הגברה של cDNA עבור מבחני ה-PCR מציג שגיאה ניסיוני אשר בדרך כלל הוא מופחת על ידי צעד נורמליזציה באמצעות נתוני משק stably ביטוי גנים. עם זאת, טמפורלית לשינויים oocyte דרך טרום השרשה עוברי משנה גם את הביטוי של גנים משק בית. פרוטוקול SM-דג מגבירה קרינה פלואורסצנטית במקום cDNA. לכן, יש ללא דרישה נורמליזציה של רמות ה-mRNA ספציפי התעתיק כדי להשיג תוצאות לשחזור עם השתנות נמוכה. לאור ההשתנות של PCR פריימר יעילות, הבדלים מספרים מוחלטים של מינים שונים של mRNA לא מדויקת להשוות בתוך או בין סוגי תאים. SM-דג רגישה, מכמת mRNA. לכן, זה יכול לשמש כדי לזהות אילו תאים אקספרס mRNA באוכלוסיה לתא מעורב. לדוגמה, כאשר oocytes גדלים בתוך זקיקי ראשית או משנית, הזקיק יכול להיות מבודדת ותרבותית alginate חרוזים20 אך ההפרדה של oocyte תאים סומטיים קשה. לכן, רצף ולימודי PCR בוצעו באמצעות אוכלוסיות מעורבות לתאים. השימוש SM-דג יכול לקבוע אם mRNAs מזוהים תאים סומטיים או את oocyte של הזקיק. לבסוף, SM-דגים יש רגישות גבוהה וספציפיות המאפשרות זיהוי של הפרוטוקולים שפע נמוכה; כך למשל, גילוי של Nanog ב MII oocytes (איור 5).
אחסון השפלה של mRNAs חשוב מנגנוני הרגולציה ביטוי חלבון בקרב אנשי עסקים ותיירים כאחד. תקנה post-transcriptional של תרגום, אחסון, השפלה הם מתווך על ידי חלבונים לאגד mRNAs21. כיום, ה-RNA-חלבון immunoprecipitation (RIP) ניתן באופן שגרתי לבצע כאשר מספר גדול של תאים אינם זמינים22. עקב מספר רב של ביצים צפרדע רפואית הניתנים לאיסוף מחיה יחיד, RIP בוצעה בהצלחה במודל בעלי חיים זה. עם זאת, קשה להשיג מספיק בתרבית של oocytes, העוברים טרום השרשה לביצוע RIP. צימוד של SM-דגים, immunofluorescence (immunoFISH) 23 סעיפים רקמות צופנות בחובן פוטנציאל להמחיש חלבונים הקשורים ספציפית mRNAs כולל מכונות translational24,25. גנומיקה למדוד משתנים גנטיים (למשל, פולימורפיזמים נוקלאוטיד קטן, SNPs) המשויך מחלה ובריאות26. Phenomics מזהה שינויים בתגובות הסלולר עקב לחצים סביבתיים27,28. המחקר הנוכחי נועד למצוא מנגנון שמחבר שינויים בגנום עם פנוטיפים ספציפיים. השימוש immunoFISH יש הפוטנציאל לקשר הסנ פ תלוית שינויים בביטוי mRNA וביטוי חלבונים התורמים פנוטיפים הסלולר. ככל שהטכנולוגיה מתפתחת, יש כנראה יישומים אחרים של SM-דג יהיה לזהות מנגנונים חשובים במערכות ביולוגיות מרובים.
Disclosures
המחברים יש ריהצהל
Acknowledgements
אנו מודים ד ר דניאל ר לארסון לעזרתו נדיב עם ההתקנה ואת השימוש מציאת נקודה, מעקב אחר תוכנית 13 ותמיכה טכנית של אוניברסיטת נברסקה לינקולן מיקרוסקופ הליבה עבור ההדמיה מיקרוסקופיה קונפוקלית. מחקר זה מייצג תרומה של האוניברסיטה של אגף המחקר החקלאי נבראסקה, לינקולן, נברסקה, נתמך על ידי קרנות הפתח UNL (נב-26-206/ההצטרפות-232435 ומספר נאב-26-231/ההצטרפות-1013511).
Materials
| Name | Company | Catalog Number | Comments |
| (±)-α-Lipoic acid | Sigma-Aldrich | T1395 | Alpha Lipoic Acid |
| Albumin, Bovine Serum, Low Fatty Acid | MP Biomedicals, LLC | 199899 | FAF BSA |
| BD 10mL TB Syringe | Becton, Dickinson and Company | 309659 | 10 mL syringe |
| BD PrecisionGlide Needle | Becton, Dickinson and Company | 305109 | 27 1/2 gauge needle |
| Calcium chloride dihydrate | Sigma-Aldrich | C7902 | CaCl2-2H2O |
| Citric acid | Sigma-Aldrich | C2404 | Citrate |
| D-(+)-Glucose | Sigma-Aldrich | G6152 | Glucose |
| Disodium phosphate | Na2HPO4 | ||
| Easy Grip Petri Dish | Falcon Corning | 351008 | 35 mm dish |
| Edetate Disodium | Avantor | 8994-01 | EDTA |
| Extra Fine Bonn Scissors | Fine Science Tools | 14084-08 | Straight, Sharp/Sharp, non-serrated, 13mm cutting edge scissors |
| Fetal Bovine Serum | Atlanta biologicals | S10250 | FBS |
| Gentamicin Reagent Solution | gibco | 15710-064 | Gentamicin |
| GlutaMAX-I (100X) | gibco | 35050-061 | Glutamax |
| Gold Seal Micro Slides | Gold Seal | 3039 | 25 x 75mm slides |
| Gonadotropin, From Pregnant Mares' Serum | Sigma | G4877 | eCG |
| hCG recombinant | NHPP | AFP8456A | hCG |
| Hyaluronidase, Type IV-S: From Bovine Testes | Sigma-Aldrich | H3884 | Hyaluronidase |
| Jewelers Style Forceps | Integra | 17-305X | Forceps 4-3/8", Style 5F, Straight, Micro Fine Jaw |
| L-(+)-Lactic Acid, free acid | MP Biomedicals, LLC | 190228 | L-Lactate |
| Magnesium sulfate heptahydrate | Sigma-Aldrich | M2773 | MgSO4-7H2O |
| MEM Nonessential Amino Acids | Corning | 25-025-Cl | NEAA |
| Microscope Cover Glass | Fisher Scientific | 12-542-C | 25 x 25x 0.15 mm cover slips |
| Mm-Nanog-O2-C2 RNAscope Probe | Advanced Cell Diagnostics | 501891-C2 | Nanog Probe |
| Mm-Pou5f1-O1-C3 RNAscope Probe | Advanced Cell Diagnostics | 501611-C3 | Pou5f1 Probe |
| MOPS | Sigma-Aldrich | M3183 | |
| Paraformaldehyde | Sigma-Aldrich | P6148 | Paraformaldehyde |
| PES 0.22 um Membrane -sterile | Millex-GP | SLGP033RS | 0.22 um filters |
| Polyvinylpyrrolidone | Sigma-Aldrich | P0930 | PVP |
| Potassium chloride | Sigma-Aldrich | 60128 | KCl |
| Potassium phosphate monobasic | Sigma-Aldrich | 60218 | KH2PO4 |
| Prolong Gold antifade reagent | invitrogen | P36934 | Antifade reagent without DAPI |
| RNAscope DAPI | Advanced Cell Diagnostics | 320858 | DAPI |
| RNAscope FL AMP 1 | Advanced Cell Diagnostics | 320852 | Amplifier 1 |
| RNAscope FL AMP 2 | Advanced Cell Diagnostics | 320853 | Amplifier 2 |
| RNAscope FL AMP 3 | Advanced Cell Diagnostics | 320854 | Amplifier 3 |
| RNAscope FL AMP 4 ALT A | Advanced Cell Diagnostics | 320855 | Amplifier 4 ALT A |
| RNAscope FL AMP 4 ALT B | Advanced Cell Diagnostics | 320856 | Amplifier 4 ALT B |
| RNAscope FL AMP 4 ALT C | Advanced Cell Diagnostics | 320857 | Amplifier 4 ALT C |
| RNAscope Fluorescent Multiplex Detection Reagents Kit | Advanced Cell Diagnostics | 320851 | FISH Reagent Kit |
| RNAscope Probe 3-plex Negative Control Probe | Advanced Cell Diagnostics | 320871 | Negative Control |
| RNAscope Probe 3-plex Positive Control | Advanced Cell Diagnostics | 320881 | Positive Control |
| RNAscope Probe Diluent | Advanced Cell Diagnostics | 300041 | Probe Diluent |
| RNAscope Protease III | Advanced Cell Diagnositics | 322337 | Protease III |
| RNAscope Protease III & IV Reagent Kit | Advanced Cell Diagnostics | 322340 | FISH Protease Kit |
| RNAscope Protease IV | Advanced Cell Diagnostics | 322336 | Protease IV |
| S/S Needle with Luer Hub 30G | Component Supply Co. | NE-301PL-50 | blunt 30 gauge needle |
| Sodium bicarbonate | Sigma-Aldrich | S6297 | NaHCO3 |
| Sodium chloride | Sigma-Aldrich | S6191 | NaCl |
| Sodium hydroxide | Sigma-Aldrich | 306576 | NaOH |
| Sodium pyruvate, >= 99% | Sigma-Aldrich | P5280 | Pyruvate |
| Solution 6 Well Dish | Agtechinc | D18 | 6 well dish |
| Taurine | Sigma-Aldrich | T8691 | Taurine |
| Tissue Culture Dish | Falcon Corning | 353002 | 60 mm dish |
| Triton X-100 | Sigma-Aldrich | X100 | Triton X-100 |
References
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proceedings of the National Academy of Sciences. 96 (16), 9236-9241 (1999).
- MacK, E. M., Smith, J. E., Kurz, S. G., Wood, J. R. CAMP-dependent regulation of ovulatory response genes is amplified by IGF1 due to synergistic effects on Akt phosphorylation and NF-kB transcription factors. Reproduction. 144 (5), 595-602 (2012).
- Xie, F., Timme, K. A., Wood, J. R. Using Single Molecule mRNA Fluorescent in Situ Hybridization (RNA-FISH) to Quantify mRNAs in Individual Murine Oocytes and Embryos. Scientific Reports. 8 (1), 7930(2018).
- Ruebel, M. L., et al. Obesity modulates inflammation and lipidmetabolism oocyte gene expression: A single-cell transcriptome perspective. Journal of Clinical Endocrinology and Metabolism. 102 (6), 2029-2038 (2017).
- Borensztein, M., Syx, L., Servant, N., Heard, E. Mouse Oocyte Development. 1818, 51-65 (2018).
- Jansova, D., Tetkova, A., Koncicka, M., Kubelka, M., Susor, A. Localization of RNA and translation in the mammalian oocyte and embryo. PLoS ONE. 13 (3), 1-25 (2018).
- Player, A. N., Shen, L. P., Kenny, D., Antao, V. P., Kolberg, J. A. Single-copy gene detection using branched DNA (bDNA) in situ hybridization. Journal of Histochemistry and Cytochemistry. 49 (5), 603-611 (2001).
- Wang, F., et al. RNAscope: A novel in situ RNA analysis platform for formalin-fixed, paraffin-embedded tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Itzkovitz, S., van Oudenaarden, A. Validating transcripts with probes and imaging technology. Nature Methods. 8 (4), S12-S19 (2011).
- Derti, A., et al. ProbeDesigner: for the design of probesets for branched DNA (bDNA) signal amplification assays. Bioinformatics. 15 (5), 348-355 (1999).
- Larson, B., Malayter, D., Shure, M. Multiplexed Detection of Cytokine Cancer Biomarkers using Fluorescence RNA In Situ Hybridization and Cellular Imaging. BioTek Application Notes. , https://www.biotek.com/resources/application-notes/multiplexed-detection-of-cytokine-cancer-biomarkers-using-fluorescence-rna-in-situ-hybridization-and-cellular-imaging/ 1-5 (2016).
- Buxbaum, A. R., Wu, B., Singer, R. H. Single β-Actin mRNA Detection in Neurons Reveals a Mechanism for Regulating Its Translatability. Science. 343 (6169), 419-422 (2014).
- Thompson, R. E., Larson, D. R., Webb, W. W. Precise nanometer localization analysis for individual fluorescent probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Herrick, J. R., Paik, T., Strauss, K. J., Schoolcraft, W. B., Krisher, R. L. Building a better mouse embryo assay: effects of mouse strain and in vitro maturation on sensitivity to contaminants of the culture environment. Journal of Assisted Reproduction and Genetics. 33 (2), 237-245 (2016).
- Pohlmeier, W. E., Xie, F., Kurz, S. G., Lu, N., Wood, J. R. Progressive obesity alters the steroidogenic response to ovulatory stimulation and increases the abundance of mRNAs stored in the ovulated oocyte. Molecular Reproduction and Development. 81 (8), 735-747 (2014).
- Xie, F., Anderson, C. L., Timme, K. R., Kurz, S. G., Fernando, S. C., Wood, J. R. Obesity-dependent increases in oocyte mRNAs are associated with increases in proinflammatory signaling and gut microbial abundance of lachnospiraceae in female mice. Endocrinology. 157 (4), 1630-1643 (2016).
- Hirao, Y., Yanagimachi, R. Detrimental effect of visible light on meiosis of mammalian eggs in vitro. Journal of Experimental Zoology. , (1978).
- Takenaka, M., Horiuchi, T., Yanagimachi, R. Effects of light on development of mammalian zygotes. Proceedings of the National Academy of Sciences. 104 (36), 14293-14293 (2007).
- Komminoth, P., Werner, M. Target and signal amplification: Approaches to increase the sensitivity of in situ hybridization. Histochemistry and Cell Biology. 108 (4-5), 325-333 (1997).
- Hornick, J. E., Duncan, F. E., Shea, L. D., Woodruff, T. K. Multiple follicle culture supports primary follicle growth through paracrine-acting signals. Reproduction. 145 (1), 19-32 (2013).
- Vlasova-St. Louis, I., Bohjanen, P. Feedback Regulation of Kinase Signaling Pathways by AREs and GREs. Cells. 5 (1), 4(2016).
- Gilbert, C., Svejstrup, J. Q. RNA Immunoprecipitation for Determining RNA-Protein Associations In Vivo. Current Protocols in Molecular Biology. 75 (1), 27.4.1-27.4.11 (2006).
- Kwon, S., Chin, K., Nederlof, M., Gray, J. W. Quantitative, in situ analysis of mRNAs and proteins with subcellular resolution. Scientific Reports. 7 (1), 16459(2017).
- Voigt, F., et al. Single-Molecule Quantification of Translation-Dependent Association of mRNAs with the Endoplasmic Reticulum. Cell Reports. 21 (13), 3740-3753 (2017).
- Halstead, J. M., Wilbertz, J. H., Wippich, F., Lionnet, T., Ephrussi, A., Chao, J. A. TRICK: A Single-Molecule Method for Imaging the First Round of Translation in Living Cells and Animals. Methods in Enzymology. 572, Elsevier Inc. (2016).
- Cookson, W., Liang, L., Abecasis, G., Moffatt, M., Lathrop, M. Mapping complex disease traits with global gene expression. Nature Reviews Genetics. 10 (3), 184-194 (2009).
- Houle, D., Govindaraju, D. R., Omholt, S. Phenomics: the next challenge. Nature Reviews Genetics. 11, 855(2010).
- Freimer, N., Sabatti, C. The Human Phenome Project. Nature Genetics. 34, 15(2003).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved