Method Article
Identificação de eventos de recombinação homóloga em células-tronco embrionárias de camundongo usando Southern Blotting e reação em cadeia da polimerase
* Estes autores contribuíram igualmente
Neste Artigo
Resumo
Aqui, apresentamos um protocolo detalhado para identificar os eventos de recombinação homóloga que ocorreram em células-tronco embrionárias do mouse usando a mancha do Sul e/ou PCR. Este método é exemplificado pela geração de nonmuscle myosin II substituição genética rato modelos usando embrionária baseada em células-tronco homóloga mediada por recombinação segmentação tecnologia tradicional.
Resumo
Relativo a questões dos efeitos fora do alvo e a dificuldade de inserção de um fragmento de DNA a longa na aplicação do desenhador nucleases para tronco embrionárias, edição de genoma tecnologia de gene-alvo baseada em célula (ES) não tem estas deficiências e é amplamente usado para modificar o genoma do animal/rato que vão desde grandes exclusões/inserções para substituições de nucleotídeo único. Notavelmente, identificar os relativamente poucos eventos de recombinação homóloga (HR) necessários para obter desejado ES clones é um passo fundamental, que exige métodos precisos e confiáveis. Mancha do Sul e/ou PCR convencional é frequentemente utilizado para esta finalidade. Aqui, descrevemos os procedimentos detalhados de usar esses dois métodos para identificar o HR eventos que ocorreram em pilhas do ES do rato em que o gene Myh9 endógeno destina-se a ser interrompido e substituído por cDNAs codificação outras cadeias pesadas de miosina nonmuscle do IIs (IIs NMHC). Todo o processo de mancha do Sul inclui a construção de direcionamento vector(s), eletroporação, seleção de drogas, a expansão e armazenamento de células de ES/clones, a preparação, digestão e mancha de DNA genômico (gDNA), a hibridização e lavagem de ição e uma etapa final de autoradiografia sobre os filmes de raio-x. PCR pode ser realizado diretamente com gDNA preparado e diluído. Para obter resultados ideais, as sondas e a enzima de restrição (RE) sites de corte para a mancha do Sul e os primers para PCR devem ser cuidadosamente planejadas. Embora a execução de mancha do Sul é demorada e trabalhosa e resultados PCR tem falsos positivos, a correta identificação pela mancha do Sul e a triagem rápida pelo PCR permite que o aplicativo único ou combinado desses métodos descritos Neste trabalho a ser amplamente utilizado e consultado pela maioria dos laboratórios na identificação de genótipos de pilhas do ES e geneticamente modificado animais.
Introdução
A tecnologia do gene definião HR em pilhas do ES murino fornece uma ferramenta poderosa para dissecando as celulares consequências de mutações genéticas específicas1,2. A importância e o significado desta tecnologia são refletidas no seu reconhecimento por 2007 Prêmio Nobel de Fisiologia ou medicina de3,4; Enquanto isso, ele representa o advento da era moderna da engenharia do gene5. Gene-alvo através de HR pode ser utilizado para engenheiro praticamente qualquer alteração desde mutações pontuais até grandes rearranjos cromossômicos no genoma do rato ES células6,7. É sabido que, antes do surgimento das ferramentas de edição chamada genoma, a geração de um rato de nocaute do gene necessário a aplicação da tecnologia de gene-alvo no ES células8,9,10. Durante as últimas duas décadas, mais de 5.000 ratos gene-alvo foram produzidos por esta abordagem para a modelagem de doenças humanas ou estudar o gene funções11. Estabeleceu-se um esforço de todo o genoma nocaute para a distribuição de vetores do gene-alvo, alvo clones de células ES e ratos ao vivo para a comunidade científica2,12,13,14 , 15. sem dúvida, ES baseada em célula mediada por HR gene-alvo tecnologia avançou grandemente nossa compreensão das funções dos genes, jogados num contexto fisiológico ou patológico.
Porque o HR é um evento relativamente pouco frequente em mamíferos células16,17, o importante e o próximo passo a seguir direcionamento de gene em células de ES murino é analisar inúmeras colônias de ES para identificar alguns clones com mutações resultantes HR com o direcionamento de vetor18. Os métodos de ouro para a identificação de eventos HR incluem mancha do Sul e PCR19,20. As vantagens das abordagens incluem que mancha do Sul pode identificar corretamente direcionados ES clones e permite que os pesquisadores analisar a estrutura do evento gene-alvo, tais como a verificação de uma inserção de cópia única de construção, enquanto um Estratégia baseado em PCR permite mais rápida triagem para HR eventos21,22. Embora esses métodos têm desvantagens, tais como que eles são demorados e podem ter falsos positivos, o uso combinacional deles é amplamente aceites e aplicados pela maioria dos laboratórios em identificar eventos HR, particular em pilhas do ES, para gerar geneticamente modificado animais.
Três isoformas de miosina nonmuscle II (NM II) nos mamíferos, cada um composto por dois IIs NMHC idênticos que são codificadas por genes diferentes três (nomeado Myh9, Myh10 e Myh14) e dois pares de cadeias leves, são referidas como NM II-A, II-B e II-C23. Estudos anteriores indicaram que pelo menos as isoformas de NM II-A e II-B são essenciais para o desenvolvimento do mouse porque a ablação na vivo dessas isoformas resulta em embrionárias letalidade24,25,26. Para contornar este problema e obter novos insights sobre as funções específicas do isoform de NM II-A e II-B em fases posteriores do desenvolvimento do mouse, uma substituição genética foi adoptada a estratégia usando ES baseada em célula mediada por HR gene-alvo tecnologia para gere uma série de modelos de rato27. No curso de identificação de clones ES desejados, mancha do Sul e métodos PCR foram utilizados, e estas provaram para ser eficiente e confiável27,28.
Este trabalho pretende fornecer uma descrição detalhada do Sul da mancha e PCR, incluindo o projeto de segmentação vector(s) ição e as primeiras demão e a execução de experiências, bem como a análise dos resultados, exemplificado através da identificação de evento HR ocorrência em pilhas do ES para a criação do mouse NM II de substituição genética de modelos e dados representativos. Os protocolos destes dois métodos aqui apresentados também podem ser adoptados para identificar os genótipos de células geneticamente modificadas ou de animais.
Protocolo
1. projeto de direcionamento Construct(s), sondas para Southern Blot e Primers para PCR
- Selecione o primeiro exon codificação (exon 2) do gene Myh9 para interrupção ou inserção na aplicação do nocaute/TOC-no relatado aqui.
- Recupere as 5 kb upstream e 5 kb a jusante sequências de DNA cercam o Myh9 exon 2 do site genome.ucsc.edu .
- Analise padrões de digestão restrição de enzimas (REs) com 1 – 2 cortes nesta região de 10 kb usando software de pDRAW para determinar o RE(s) apropriado para digerir o DNA genômico de mancha do Sul.
Nota: Dra eu cumpre esta exigência e é selecionado para o efeito. - Selecione um fragmento 4KB imediato a montante do exon Myh9 2 como o braço esquerdo de homologia (LHA) e uma 1,7 kb fragmento imediata sequência a jusante como o braço direito de homologia (RHA); Escolha um fragmento de 1 kb 5' montante do LHA como a sonda esquerda (LP) para a mancha do Sul e um fragmento de 1,2 kb 3' a jusante do RHA como a sonda certo (RP), com base na análise acima.
- Use um programa de primer3 para projetar primers para amplificar os quatro fragmentos de DNA por PCR para diante e reversos. Desenha um par de primers com o primer frente residido perto do terminal 3' de um gene de resistência de neomyocin seleção marcador (P1) e o primer reverso localizado fora o RHA (P2).
Nota: Este par de primer será usado para identificar o alvo ES clones por PCR29. - Encontrar um clone de BAC 129Sv cobrindo o locus do gene do rato Myh9 visitando o site bacpac.chori.org (Nota: DNA isogénicas é preferencial). Isole DNA de BAC usando um kit adequado para peças grandes de ADN, seguindo as instruções fornecidas pelo fabricante de purificação.
Nota: DNA purificado de BAC será usado como modelo para amplificação por PCR. - Desenhe uma representação esquemática do direcionamento construções, sondas e primers para resumir esta informação.
2. geração de segmentação Construct(s) e sondas para borrão do Sul e na preparação de Primers para PCR
- Encomendar os primers PCR descritos acima e dissolvê-los em uma concentração de 20 µM.
- Amplificar os braços de homologia e sondas por PCR em uma solução de reação, incluindo 1 µ l µ l para a frente e 1 reversa as primeiras demão, 1 µ l de DNA BAC (50 ng) como o modelo, 5 µ l de tampão para Pfu e 1 µ l de Pfu ultra como a DNA polimerase , e H2O até 50 µ l. realizar a PCR em uma máquina PCR nas seguintes condições: 95 ° C por 3 min; 95 ° C por 30 s; 60 ° C por 30 s; 72 ° C e 1 kb/min; 30 ciclos; Finalmente, 72 ° C por 10 min.
- Purificar os produtos de PCR usando um kit de limpeza PCR de acordo com o protocolo fornecido pelo fabricante e eluir os fragmentos de DNA com 40 µ l de H2s Digest O. os produtos PCR purificados com REs predefinidos em uma solução de reação contendo 5 µ l de tampão de x 10 para REs , 2,5 µ l de RE1 e 2,5 µ l de RE2 e o DNA eluted, a 37 ° C por 2 h. executar um gel de DNA de 1% para separar as faixas de DNA alvo, o gel que contém ADN sob a luz UV, o alvo do imposto especial de consumo purificar os fragmentos de DNA alvo do gel usando um DNA gel de extração kit de acordo o protocolo fornecido pelo fabricante e eluir os fragmentos de DNA com 40 µ l de H2O.
- Clonar a homologia braços e cassette(s) expressão de substituição para o vetor de mpNTKV-LoxP segundo a ordem do amplificado e purificado RHA, LHA e substituição expressão cassette(s) lançado de outros vetores para obter o direcionamento final construct(s). Clone fragmentos de DNA das sondas amplificados e purificados para o vetor T-fácil.
- Confirme as sequências de nucleotídeos de todos os fragmentos de DNA clonados por sequenciamento27,28.
3. preparação de direcionamento Construct(s), a eletroporação de pilhas do ES e a amplificação de Clones de ES
- Prepare cada construção direcionamento usando um kit de maxiprep de plasmídeo de acordo com o protocolo fornecido pelo fabricante. Linearizar cada construto shRNA digerindo isso com não I em uma reação de 400 µ l, incluindo 40 µ l de tampão 10x para não eu, 10 µ l de não eu, 100 µ g de DNA e H2O até 400 µ l, a 37 ° C durante a noite.
- Purifica o linear construct(s) de direcionamento.
- Extrair a solução digerido reação 1 x com igual volume de álcool isoamílico: fenol: clorofórmio (25:24:1) e centrifugar uma força de 2.000 x g por 10 min.
- Transferir o sobrenadante para um novo tubo de 1,5 mL, precipitar o DNA usando 2.5 x etanol e 3 x 0,1 M de acetato de sódio (pH 5.2) (relação de volume) e centrifugar a uma força de 2.000 x g durante 10 minutos.
- Remova o sobrenadante, lave o DNA de pelotas 1 x 1 ml de etanol a 75% e centrifugar a uma força de 2.000 x g por 5 min.
- Remover o sobrenadante e secar o sedimento de DNA por 5 min.
- Dissolva o sedimento de DNA linear em estéril tampão Tris-EDTA (TE) em uma concentração final de 1 µ g / µ l.
- Misture 50 µ g de cada construção direcionamento linear com 0,5 x 107 ES células. Executar o electroporation a 320 V e 250 µF. placa as células electroporated ES em pratos com neo-resistente alimentadores MEF.
- Após 24h, alterne para meio de celular ES com 400 µ g/mL G418 e 200 µM ganciclovir e continuar a cultura para 4-5 dias com uma mudança de média diária. Pega clones resistentes de ES em placas boas 48.
Nota: Normalmente, quatro placas de 48-boas são usadas por construção. - Duplica as placas de 48-bem.
Nota: Um conjunto de placas é criopreservado, e o outro conjunto é usado para a preparação de DNA genômica.
4. preparação de DNAs Genomic e a digestão com Restriction Enzyme(s)
- Prepare gDNAs de células ES usando um kit comercial (kit de purificação de DNA genômico) com pequenas modificações.
- Remover a mídia da cultura de pilha ES e adicione 500 µ l de solução de lise de núcleos, incluindo RNaseA, diretamente aos poços para lisar as células.
Nota: O lisado celular pode ser armazenado a-80 ° C ou tratado imediatamente. - Pipetar subindo e descendo várias vezes para lisar as células totalmente e transfira para um tubo limpo 1,5 mL.
- Adicione um terço do volume da solução de precipitação de proteínas para o tubo de 1,5 mL, vórtice vigorosamente durante 20 s, relaxa as amostras no gelo por 5 min e em seguida, centrifugar a uma força de 2.000 x g durante 5 min. o sobrenadante de transferência para outro limpo 1,5 mL tubo contai Ning igual volume de isopropanol; Misture suavemente a solução. (Note que o brancos como fio de vertentes podem ser vistos neste momento). Centrifugar a uma força de 2.000 x g por 1 min; em seguida, descarte o sobrenadante.
- Lave o pellet gDNA com 1 mL de etanol a 70% à temperatura ambiente, centrifugar uma força de 2.000 x g por 1 min, aspirar o sobrenadante cuidadosamente e em seguida, secar o sedimento gDNA por 3 min.
- Dissolver o gDNAs com 100 µ l de solução de reidratação de DNA e, em seguida, incubar a 65 ° C, durante 1 h, ou a 4 ° C durante a noite.
- Loja da gDNAs 2 – 8 ° c.
- Remover a mídia da cultura de pilha ES e adicione 500 µ l de solução de lise de núcleos, incluindo RNaseA, diretamente aos poços para lisar as células.
- Digerir o gDNAs com pré-concebidos RE Dra I. definir a reacção de digestão de 30 µ l misturando 3 µ l de 10x buffer para Dra I, 3 µ l de Dra eu, 10 µ g de H2O e gDNAs/amostra até 30 µ l e incubar a 37 ° C durante a noite.
- Verificar a integridade da digestão pelo gel de DNA, análise de 5 µ l da reação digerida e em seguida, adicione a 3 µ l de buffer de DNA-carregamento para a etapa subsequente de 10x.
5. sul da mancha e a identificação da PCR
- Rastreio de mancha do Sul
- Separe o gDNAs digerido por eletroforese e transferência a uma membrana.
- Preparar um gel de agarose a 1% da electroforese com brometo de etídio (EB), carregar as amostras da etapa 4.3 e uma escada de 1 kb e funcione o gel com uma tensão baixa (30-40 V) durante a noite.
- Retire o gel e tirar uma foto com um sistema de geração de imagens de gel de DNA após eletroforese. Verifique se o digerido e separados gDNAs exibir uma imagem do esfregaço como.
- Mergulhe o gel em uma bandeja com 0,2 N HCl solução e agitar suavemente durante 20 min à temperatura ambiente.
- Transferir o gel para DNA-desnaturar a solução e agitar suavemente durante 20 min à temperatura ambiente.
- Alternar o gel em solução de DNA-neutralizando e agitar suavemente durante 20 min à temperatura ambiente.
Nota: O gel é propenso à quebra após esta etapa, assim que deve ser manuseado com cuidado. - Use o sistema de transferência rápida descendente para transferir os DNAs do gel para a membrana. Monte a TurboBlotter e a pilha mancha de acordo com as instruções fornecidas pelo fabricante.
Nota: x 10 ou 20 solução salina-citrato de sódio (CCD) de x é usado como um buffer de transferência. Em geral, 3 h de transferência é suficiente para a transferência de 95% de gDNAs de gel para a membrana; no entanto, um maior tempo de transferência é inócuo. - Tirar a membrana e lavá-lo com 2 x SSC por 1 min, absorver o líquido com tecidos e em seguida, cross-link o DNA com a membrana usando um agente reticulante UV.
Nota: A membrana pode ser armazenada a 4 ° C, durante uma semana.
- Rotule as sondas de DNA com radioactividade.
- Purifica os plasmideos sonda usando um kit miniprep de acordo com o protocolo fornecido pelo fabricante.
- Lançamento do DNA fragmentos das sondas do vetor do plasmídeo por EcoR digestão em uma solução de reação, incluindo 5 µ l de tampão para EcoR eu, 2 µ l de EcoR eu enzima, 20 µ g de DNA de plasmídeo e H2O até 50 µ l , por 2 h.
- Executar um 1% de DNA do gel para separar os fragmentos de DNA de sonda do vetor e purificar os fragmentos de DNA das sondas com um kit de extração de gel de DNA de acordo com o protocolo fornecido pelo fabricante.
- Usando 1 µ l da solução de DNA, medir a concentração de DNA, os fragmentos de DNA de sonda com um espectrofotômetro no comprimento de onda de 260/280 nm.
- Preparar 40-ng da sonda DNAs em um tubo de 1,5 mL com 45 µ l de tampão TE, ferva por 3 min, girar rapidamente e em seguida, coloque os tubos no gelo por 2 min.
- Adicionar os DNAs de calor desnaturado sonda ao tubo contendo grânulos de DNA-rotulagem de ready-to-go (-dCTP), pipetar acima e para baixo, para misturar, adicionar 5 µ l de dCTP [α32P] e em seguida, incubar a 37 ° C por 15 min.
- Purificar as sondas etiquetadas usando microcolumns de G-50 de acordo com as instruções fornecidas pelo fabricante e, em seguida, medir a radioatividade por um contador de cintilação (opcional).
- Cruzar o membrane(s) com as sondas rotuladas.
- Prehybridize da membrana.
- Escaldar a solução de hibridação a 42 ° C por 30 min. Mix 20 mL da solução de hibridação escaldadas com 200 µ g de esperma de salmão cozida DNA em um tubo de 50 mL.
- Coloca a membrana no tubo de hibridização. Adicione a solução de prehybridization mista no tubo de hibridização. Coloque-o no forno da hibridação (conjunto de rolamento e a temperatura de 42 ° C) e deixar o prehybridization prosseguir por 30 min.
- Cruzar a membrana com a rotulado ição.
- Retire o tubo de hibridização e despeje a solução prehybridization em um tubo de 50 mL; Adicione a sonda desnaturada (aquecida a 100 ° C por 3 min) da etapa 5.1.2.7 a este tubo e misture delicadamente.
Nota: Reduza quaisquer bolhas de indução. - Retornar a solução mista para o tubo de hibridização e realizar a hibridação a 42 ° C durante a noite.
- Retire o tubo de hibridização e despeje a solução prehybridization em um tubo de 50 mL; Adicione a sonda desnaturada (aquecida a 100 ° C por 3 min) da etapa 5.1.2.7 a este tubo e misture delicadamente.
- Prehybridize da membrana.
- Lave o membrane(s) para remover sondas nonhybridized.
- Coloque o membrane(s) em uma bandeja com 1x SSC + 0,1% SDS e agitar suavemente em 55 a 60 ° C por 10 min.
- Transferir o membrane(s) para uma bandeja com 0.5 x SSC + 0,1% SDS e agitar suavemente em 55 a 60 ° C por 10 min.
- Verificar a radioatividade sobre a membrane(s) usando um contador Geiger portátil para decidir se uma terceira lavagem é necessária.
- Expor a radioatividade na membrana para películas de raio x.
- Retire o líquido do lavado membrane(s).
- Abraçar o membrane(s) com filme plástico e consertar tudo na gaveta a exposição.
- Expor a membrana de duas folhas de filme de raio-x em um quarto escuro.
- Coloque a fita de exposição a-80 ° C durante a noite ou mais.
- Desenvolva os filmes para visualizar os resultados. Avalie se um clone de ES correspondente é o desejado com a recombinação direcionada ou não, de acordo com os tamanhos das bandas de DNA detectados por sondas.
- Rehybridize na mesma membrana por outra sonda após tirar a sonda utilizada de acordo com o seguinte procedimento: Retire a membrana usada, lavá-lo 1x com limpar H2O e então, incube-lo na solução de distribuição (formamida 55%, 2% SEPI, 1% SDS, H 2 O) a 65 ° C com agitação suave para 1 – 2 h.
- Separe o gDNAs digerido por eletroforese e transferência a uma membrana.
-
Identificação da PCR
- Realizar a identificação de PCR dos clones ES desejados em uma solução de reação de 50 µ l incluindo 5 µ l de tampão de x PCR 10, 2 µ l de 50 mM de MgSO4, 1 µ l de 10mm dNTP, 1 µ l de primer para a frente de 20 µM, 1 µ l de primer reverso de 20 µM , 1 µ l de alta-fidelidade platinum Taq, gDNAs (~ 100 ng) e H2O até 50 µ l.
- Use as seguintes condições de reação de PCR: uma desnaturação inicial a 94° C por 3 min, 30 ciclos de desnaturação a 94° C por 30 s, recozendo a 60° C por 30 s e uma extensão a 68° C para 132 s e uma etapa final de 68° C por 10 min.
- Analise os produtos PCR por eletroforese em agarose 1,0%.
- Clone os fragmentos PCR com seu tamanho esperado para o vetor T-fácil e sequência para confirmar a presença de uma sequência parcial do vetor alvo.
Resultados
Neste trabalho, um protocolo detalhado da mancha do Sul e do PCR é descrito, que é utilizado para identificar o HR eventos que ocorreram em pilhas do ES do rato para a geração de modelos de rato de substituição genética II NM, usando ES baseados em células mediada por HR segmentação tecnologia. Embora a mancha do Sul e PCR, bem como tecnologia de gene-alvo tradicional, têm sido amplamente utilizada por várias décadas, o sucesso da aplicação deles precisa ser cuidadosamente planejada. Pelo menos estes aspectos são necessários para ser considerado: o comprimento dos braços longos e curtos, as posições e o comprimento das sondas, os REs apropriados para cortar os DNAs genomic e os primers para PCR, como resumidos na Figura 1, que é útil para análise subsequente. Como um passo importante da mancha do Sul, os preparado e digeridos DNAs genomic são obrigados a ser separados em gel de DNA para a deteção pela sonda. Porque DNAs genomic são cortadas em um monte de fragmentos com comprimentos diferentes, eles exibem um status de esfregaço, como sobre o gel de DNA, sugerindo uma digestão completa dos DNAs genomic, conforme indicado na Figura 2. Como uma etapa final da mancha do Sul, os sinais de uma sonda de radioactividade-rotulado hybridizing com um fragmento de DNA alvo são mostrados no filme, que refletem a ocorrência de eventos de HR em clones do ES, indicando desse modo se um clone de ES é aquela desejada. De acordo com a predesign neste estudo, ES clones com o alelo mutante tem duas faixas de tamanho distinto, enquanto o selvagem-tipo ES clones só tem uma banda, sugerindo que os clones ES desejados são heterozigotos (Figura 3). Relativa ao procedimento e resultados da mancha do Sul, a operação e resultados de PCR são simples e direto. Após a reação de PCR, os produtos PCR podem ser analisados em gel do DNA. Se as bandas PCR são específicas e sequenciamento dos produtos PCR clonados confirma a presença de uma sequência parcial de vetor alvo como um gene de neo-resistência, bem como regiões genômicas estão apenas fora o braço de homologia, a ocorrência de eventos HR pode ser esperado e verificado (Figura 4).
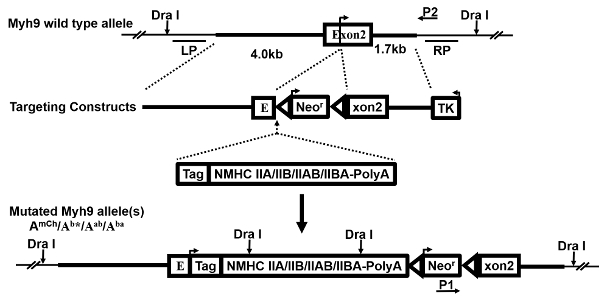
Figura 1 : Direcionamento construções. Isto é um esquema demonstrando a geração de várias construções de direcionamentos. O alelo selvagem-tipo com gene Myh9 (WT), vetor do gene-alvo, reposição exógena de expressão cassette(s) e o allele(s) mutante resultante, bem como as sondas (LP, RP) para Southern blot e os primers (P1, P2) para o PCR, são mostradas e descritas anteriormente 27. uma seta no exon 2 indica o local de iniciação translacional. Após a ocorrência de sucesso do HR, o gene de resistência de neomicina (Neor) e a gaveta de expressão de substituição são inseridos apenas 5' do códon ATG iniciador. Portanto, o alelo Myh9 endógeno é interrompido e o bateu-nos genes é/são expressos nas células mutantes e ratos. Clique aqui para ver uma versão maior desta figura.

Figura 2 : Digerido DNAs genomic com Dra I. DNAs genomic de clones ES voltados com a construção substituindo NMHC II-A, II-B são digeridos com Dra eu e, em seguida, separados em um gel de agarose por eletroforese. Observa-se um gDNA digerido como esfregaço. C1 - C8 retratam clones individuais do ES. Uma digestão completa de gDNA produz um monte de fragmentos de DNA com um comprimento diferente, assim, exibindo uma imagem de como esfregaço. Esse resultado também reflete a boa qualidade de gDNAs preparado e a completude da digestão. Clique aqui para ver uma versão maior desta figura.

Figura 3 : Resultados representativos da mancha do Sul. Estes painéis mostram uma seleção de mancha do Sul dos DNAs genomic de clones ES voltados com a construção de substituir NMHC II-A com II-AB, utilizando as sondas de esquerda e direita. O alelo mutante mostra uma banda 12,1 kb ou kb 6 quando a sonda esquerda ou direita sonda é usada, respectivamente, enquanto o WT mostra uma faixa de 9,7 kb. M: marcador; PC1-PC5: clones positivos; NC: clone negativo. Os tamanhos das bandas sul da mancha também são indicados. Todos os procedimentos da mancha do Sul são estritamente realizados e a especificidade das sondas é bom o suficiente; Não deve haver nenhum inespecíficos bandas esperam para as bandas esperadas. Clique aqui para ver uma versão maior desta figura.
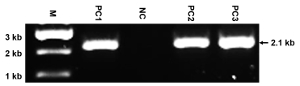
Figura 4 : Resultados representativos do PCR. Este painel mostra a identificação de PCR dos DNAs genomic de clones ES voltados com a construção de substituir NMHC II-A com II-BA usando o par de primer P1 + P2. O alelo mutante produz uma banda 2,1 kb, enquanto o alelo WT não produz nenhuma banda. M: marcador; PC1-PC3: clones positivos; NC: clone negativo. O tamanho da banda de PCR também é indicado. Desde as primeiras demão são projetadas para detectar somente o alelo mutante, o surgimento de uma banda única e esperada reflecte a especificidade dos primers e a alta qualidade do gDNAs preparado. Clique aqui para ver uma versão maior desta figura.
Discussão
Atualmente, desenhador nucleases para genoma edição ainda não podem substituir tecnologia gene-alvo baseada em célula ES devido a seus problemas de efeitos fora do alvo e a dificuldade em inserir uma longa DNA fragmento30,31. Como os métodos de ouro para a identificação de HR eventos que ocorreram em pilhas do rato ES, este relatório fornece um protocolo detalhado da mancha do Sul e PCR para o campo. Nós validado a confiabilidade desses métodos, analisando clones individuais de pilhas do ES do mouse voltadas com uma série de construções. Os clones de ES desejados identificados por esses métodos tinham sido usados com sucesso para gerar correspondente do mouse modelos27.
Embora outras técnicas para a seleção de clones de ES alvo têm sido descrito19,32, os métodos da mancha do Sul e PCR não pode ser completamente substituído por aqueles estabeleceram posteriormente32, porque estas iniciais técnicas têm uma história mais aplicada e são amplamente aceitos e confirmaram pela sociedade científica, realizada pela maioria dos laboratórios biológicos e são a origem de outras tecnologias. Importante, o bom desempenho da mancha do Sul e PCR na identificação de eventos HR é bem exemplificado no anterior trabalho29. Os resultados da mancha do Sul indicam várias características únicas: entre os clones selecionados aleatoriamente do ES, mais de 90% deles são desejados, sem bandas inespecíficas são detectadas e o HR ocorreu preferencialmente em um alelo do gene Myh9. Enquanto isso, os dados do PCR, juntamente com o sequenciamento, confirmam que a ocorrência de eventos HR é site-specific e combinar bem com os da mancha do Sul.
De acordo com nossa prática, vários fatores devem ser considerados quando mancha do Sul e PCR são usados para identificar os eventos de HR em pilhas do ES, obtendo assim bons e esperados resultados. O primeiro é o comprimento dos braços de homologia; em geral, aumentar o comprimento do braço de homologia irá aumentar a eficiência da HR33. No entanto, isto não é sempre o caso. Por um lado, braços mais longos aumentam a dificuldade de manipulação; por outro lado, o comprimento dos braços de homologia (4 kb para o braço esquerdo e 1,7 kb para o braço direito) relatado aqui resultou na mais alta frequência HR obtida até o momento entre experiências semelhantes. Além disso, um comprimento razoável de braços de homologia facilita a identificação pelo PCR. A segunda é a utilização de DNA isogénicas para preparar os braços de homologia e sul da mancha sondas34. Isto pode ser satisfeito por encomendar um clone BAC contendo a região do gene de interesse ou usando DNA genômico das células que se destina a ser alvo. A terceira é a seleção de REs apropriados para digerir DNAs genomic. Em geral, um RE ou a combinação de dois REs que cortam o alelo selvagem-tipo ou mutante apenas uma ou duas vezes ao redor da região de direcionamento são preferidos; Além disso, o fragmento de DNA resultante maior não deve exceder 15 kb, e a diferença de tamanho entre os diferentes fragmentos de DNA é mais de 2 kb. Esses requisitos podem facilitar a separação e identificação das bandas esperadas pela mancha do Sul. O quarto é o comprimento das sondas e a menor semelhança com outras sequências no genoma. Geralmente, o comprimento das sondas é 500-1.000 bp. A semelhança com outras sequências no genoma pode ser analisada com o programa de NCBI BLAST. Além disso, um software usado para desenho de sondas para a mancha do Sul tem sido descritos35. O quinto fator a ser considerado é usar os métodos convencionais para preparar DNA genômico para um melhoria do rendimento. DNAs genomic preparados a partir de um poço confluente de uma placa de 48 são geralmente suficiente pelo menos duas rodadas de análises mancha do Sul. Como desenhar os primers para PCR, a melhor estratégia é usar uma primeira demão presente sobre o marcador de seleção em conjunto com um primer fora dos braços de direcionamentos. Além disso, sequenciamento dos produtos PCR é importante para provar a HR eventos20,36. Notavelmente, triagem baseados em PCR não pode substituir completamente as informações obtidas através da mancha do Sul, enquanto pode eficazmente reduzir o número de clones a serem avaliadas.
Em conclusão, mancha do Sul e PCR são métodos bem demonstrados para a seleção de clones de ES para identificar eventos de gene-direcionamento mediada por HR em pilhas do ES. Embora o protocolo detalhado descrito aqui principalmente focado a seleção de clones de substituição genética ES NM II desejadas, pode ser usado para os ratos de genotipagem que posteriormente são gerados usando os clones ES positivos. Isso pode ser facilmente adaptado para a identificação de eventos de HR em outros tipos de células, como células iPS ou células somáticas.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho recebeu o apoio do programa geral da Fundação Nacional de ciências naturais da China (subvenções no. 31571432), humano Fundação Provincial de ciências naturais da China (Grant No. 2015JC3097) e a pesquisa Fundação de educação Bureau de Hunan Província, China (Grant No. 15 K 054).
Materiais
| Name | Company | Catalog Number | Comments |
| BAC CLONE | BACPAC Resources Center (BPRC) | bMQ-330E21 | |
| QIAGEN Large-Construct Kit | QIAGEN | 12462 | |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| QIAprep Spin Miniprep Kit | QIAGEN | 27104 | |
| QIAGEN Plasmid Plus Maxi Kit | QIAGEN | 12963 | |
| PfuUltra High-Fidelity DNA Polymerase | Agilent | 600382 | |
| T-easy vector | Promega | A1360 | |
| Nuclei Lysis Solution | Promega | A7941 | |
| Protein Precipitation Solution | Promega | A7951 | |
| DNA Denaturing Solution | VWR | 351-013-131 | |
| DNA Neutralizing Solution | VWR | 351-014-131 | |
| Ready-To-Go DNA Labeling Beads (-dCTP) | VWR | 27-9240-01 | |
| UltraPure SSC, 20x | Thermo Fisher | 15557036 | |
| UltraPur Phenol:Chloroform:Isoamyl Alcohol (25:24:1, v/v) | Thermo Fisher | 15593031 | |
| G418 | Thermo Fisher | 10131035 | |
| Salmon Sperm DNA Solution | Thermo Fisher | 15632011 | |
| Platinu Taq DNA Polymerase High Fidelity | Thermo Fisher | 11304029 | |
| Not I | Thermo Scientific | ER0592 | |
| Dra I | Thermo Scientific | ER0221 | |
| EcoR I | Thermo Scientific | ER0271 | |
| Ganciclovir | Sigma | G2536 | |
| Whatman TurboBlotter Transfer System, Large Kits | Fisher Scientific | 09-301-188 | |
| [α32P]dCTP | PerkinElmer | NEG013H100UC | |
| ProbeQuan G-50 Micro Columns | GE Healthcare | 28-9034-08 | |
| Hybrisol I Hybridization Solution | Millipore | S4040 | |
| Kodak X-Ray Film | Z&Z Medical | 844 5702 |
Referências
- Gao, G., McMahon, C., Chen, J., Rong, Y. A powerful method combining homologous recombination and site-specific recombination for targeted mutagenesis in Drosophila. Proceedings of the National Academy of Sciences of the United States of America. 105 (37), 13999-14004 (2008).
- Skarnes, W., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474 (7351), 337-342 (2011).
- Vogel, G. Nobel Prizes. A knockout award in medicine. Science. 318 (5848), 178-179 (2007).
- Salsman, J., Dellaire, G. Precision genome editing in the CRISPR era. Biochemistry. Cell Biology. 95 (2), 187-201 (2017).
- Capecchi, M. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nature Reviews Genetics. 6 (6), 507-512 (2005).
- Van, d. W. L., Adams, D. J., Bradley, A. Tools for targeted manipulation of the mouse genome. Physiological Genomics. 11 (3), 133-164 (2002).
- Glaser, S., Anastassiadis, K., Stewart, A. F. Current issues in mouse genome engineering. Nature Genetics. 37 (11), 1187 (2005).
- Bradley, A., Evans, M., Kaufman, M. H., Robertson, E. Formation of germ-line chimaeras from embryo-derived teratocarcinoma cell lines. Nature. 309 (5965), 255-256 (1984).
- Robertson, E., Bradley, A., Kuehn, M., Evans, M. Germ-line transmission of genes introduced into cultured pluripotential cells by retroviral vector. Nature. 323 (6087), 445-448 (1986).
- Thomas, K. R., Capecchi, M. R. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells. Cell. 51 (3), 503-512 (1987).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474 (7351), 337 (2011).
- Collins, F. S., Rossant, J., Wurst, W. A mouse for all reasons. Cell. 128 (1), 9-13 (2007).
- Poueymirou, W. T., et al. F0 generation mice fully derived from gene-targeted embryonic stem cells allowing immediate phenotypic analyses. Nature Biotechnology. 25 (1), 91-99 (2007).
- Pettitt, S. J., et al. Agouti C57BL/6N embryonic stem cells for mouse genetic resources. Nature Methods. 6 (7), 493-495 (2009).
- Gertsenstein, M., et al. Efficient Generation of Germ Line Transmitting Chimeras from C57BL/6N ES Cells by Aggregation with Outbred Host Embryos. PLoS One. 5 (6), 11260 (2012).
- Smithies, O., Gregg, R. G., Boggs, S. S., Koralewski, M. A., Kucherlapati, R. S. Insertion of DNA sequences into the human chromosomal |[beta]|-globin locus by homologous recombination. Nature. 317 (6034), 230-234 (1985).
- Deng, C., Capecchi, M. R. Reexamination of gene targeting frequency as a function of the extent of homology between the targeting vector and the target locus. Molecular & Cellular Biology. 12 (8), 3365 (1992).
- Lay, J. M., Friishansen, L., Gillespie, P. J., Samuelson, L. C. Rapid confirmation of gene targeting in embryonic stem cells using two long-range PCR techniques. Transgenic Research. 7 (2), 135-140 (1998).
- Langerak, P., Nygren, A. O. H., Schouten, J. P., Jacobs, H. Rapid and quantitative detection of homologous and non-homologous recombination events using three oligonucleotide MLPA. Nucleic Acids Research. 33 (22), 188 (2005).
- Gómezrodríguez, J., et al. Advantages of q-PCR as a method of screening for gene targeting in mammalian cells using conventional and whole BAC-based constructs. Nucleic Acids Research. 36 (18), 117 (2008).
- Kim, H. S., Smithies, O. Recombinant fragment assay for gene targeting based on the polymerase chain reaction. Nucleic Acids Research. 16 (18), 8887-8903 (1988).
- Joyner, A. L., Skarnes, W. C., Rossant, J. Production of a mutation in mouse En-2 gene by homologous recombination in embryonic stem cells. Nature. 338 (6211), 153-156 (1989).
- Ma, X., Adelstein, R. S. The role of vertebrate nonmuscle Myosin II in development and human disease. Bioarchitecture. 4 (3), 88-102 (2014).
- Malonek, D. Relationships between the dynamics of cortical blood flow, oxygenation, and volume changes following sensory stimulation. Proceedings of the National Academy of Sciences of the United States of America. 94, (1997).
- Takeda, K., Kishi, H., Ma, X., Yu, Z. X., Adelstein, R. S. Ablation and mutation of nonmuscle myosin heavy chain II-B results in a defect in cardiac myocyte cytokinesis. Circulation Research. 93 (4), 330-337 (2003).
- Conti, M. A., Evenram, S., Liu, C., Yamada, K. M., Adelstein, R. S. Defects in cell adhesion and the visceral endoderm following ablation of nonmuscle myosin heavy chain II-A in mice. Journal of Biological Chemistry. 279 (40), 41263-41266 (2004).
- Wang, A., et al. Nonmuscle myosin II isoform and domain specificity during early mouse development. Proceedings of the National Academy of Sciences of the United States of America. 107 (33), 14645-14650 (2010).
- Zhang, Y., et al. Mouse models of MYH9-related disease: mutations in nonmuscle myosin II-A. Blood. 119 (1), 238 (2012).
- Liu, T., et al. Identification and characterization of MYH9 locus for high efficient gene knock-in and stable expression in mouse embryonic stem cells. PLoS One. 13 (2), 0192641 (2018).
- Saito, S., Adachi, N. Advances in the Development of Gene-Targeting Vectors to Increase the Efficiency of Genetic Modification. Biological & Pharmaceutical Bulletin. 39 (1), 25-32 (2016).
- Langerak, P., Nygren, A., Schouten, J., Jacobs, H. Rapid and quantitative detection of homologous and non-homologous recombination events using three oligonucleotide MLPA. Nucleic Acids Research. 33 (22), 188 (2005).
- Martin, S. L., et al. A single amino acid substitution in ORF1 dramatically decreases L1 retrotransposition and provides insight into nucleic acid chaperone activity. Nucleic Acids Research. 36 (18), 5845-5854 (2008).
- Kamisugi, Y., Cuming, A. C., Cove, D. J. Parameters determining the efficiency of gene targeting in the moss Physcomitrella patens. Nucleic Acids Research. 33 (19), 173 (2005).
- Luo, Y., Bolund, L., Sørensen, C. B. Pig gene knockout by rAAV-mediated homologous recombination: comparison of BRCA1 gene knockout efficiency in Yucatan and Göttingen fibroblasts with slightly different target sequences. Transgenic Research. 21 (3), 671-676 (2012).
- Croning, M. D., Fricker, D. G., Komiyama, N. H., Grant, S. G. Automated design of genomic Southern blot probes. BMC Genomics. 11 (1), 74 (2010).
- Zimmer, A., Gruss, P. Production of chimaeric mice containing embryonic stem (ES) cells carrying a homoeobox Hox 1.1 allele mutated by homologous recombination. Nature. 338 (6211), 150-153 (1989).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados