Method Article
Identificación de eventos de recombinación homóloga en células madre embrionarias del ratón mediante Southern blot y reacción en cadena de polimerasa
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí, presentamos un protocolo detallado para la identificación de eventos de recombinación homóloga que se produjeron en células madre embrionarias del ratón mediante Southern blot o PCR. Este método es ejemplificado por la generación de modelos UROLOGICAL miosina II reemplazo genético ratón tecnología tradicional embrionario basada en células madre homólogo mediada por la recombinación dirigida a.
Resumen
Relativo a las cuestiones de efectos off-target y la dificultad de insertar un largo fragmento de ADN en la aplicación de diseño nucleasas para edición tallo genoma embrionario (ES) de la célula gene targeting tecnología no tiene estas deficiencias y es ampliamente utilizado para modificar el genoma del animal y el ratón que van desde grandes deleciones/inserciones a sustituciones de un solo nucleótido. En particular, identificando los relativamente pocos eventos de recombinación homóloga (HR) necesarios para obtener el deseado ES clones es un paso fundamental, que exige métodos precisos y fiables. Southern blot y/o PCR convencional se utiliza a menudo para este propósito. Aquí, describimos los procedimientos detallados de la utilización de estos dos métodos para identificar eventos de recursos humanos que ocurrieron en células ES de ratón que el gen Myh9 endógeno pretende ser interrumpida y reemplazada por cDNAs codifican otros cadena pesada del myosin UROLOGICAL IIs (IIs de NMHC). Todo el procedimiento de borrar meridional incluye la construcción de objetivos vector(s), electroporación, selección de drogas, la expansión y el almacenamiento de las células ES/clones, preparación, digestión y blot de DNA genómico (gDNA), la hibridación y lavado de sonda y un paso final de la autorradiografía en las películas de rayos x. PCR se puede realizar directamente con gDNA preparada y diluida. Para obtener resultados ideales, las sondas y la enzima de la restricción (RE) sitios de corte para borrar meridional y los cebadores para PCR deben ser cuidadosamente planificadas. Si la ejecución de borrar meridional es desperdiciador de tiempo y mano de obra intensiva y PCR resultados falsos positivos, la correcta identificación por borrar meridional y el cribado rápido por PCR permite la aplicación única o en combinación de estos métodos descritos en este documento para ser ampliamente utilizado y consultado por los laboratorios más en la identificación de genotipos de células madre embrionarias y genéticamente modificados animales.
Introducción
La tecnología del gene targeting por HR en células de madre embrionarias murinas proporciona una poderosa herramienta para disecar las consecuencias celulares de mutaciones genéticas específicas1,2. La importancia y el significado de esta tecnología se reflejan en su reconocimiento por el Premio 2007 Nobel en la fisiología o la medicina3,4; mientras tanto, representa el advenimiento de la era moderna de la ingeniería gen5. Gene targeting por hora puede ser utilizado para diseñar prácticamente cualquier alteración que van desde mutaciones puntuales a grandes cambios cromosómicos en el genoma del ratón ES células6,7. Es bien sabido que, antes de la aparición de herramientas de edición llamado genoma, la generación de un ratón knockout de genes requiere la aplicación de tecnología gene targeting en ES las células8,9,10. Durante las últimas dos décadas, más de 5.000 ratones dirigidos por gene fueron producidos por este enfoque para modelar enfermedades humanas o el estudio de las funciones del gen11. Un esfuerzo de nocaut en el genoma se ha establecido para la distribución de vectores gene targeting objetivos clones de células ES y ratones vivos a la comunidad científica2,12,13,14 , 15. sin duda, ES celular tecnología basada en HR-mediated gene targeting ha avanzado grandemente nuestra comprensión de las funciones de los genes en el contexto fisiológico o patológico.
Debido a que HR es un evento relativamente infrecuente en mamíferos células16,17, la importante y siguiente paso después de gene targeting en células de madre embrionarias murinas es analizar numerosas colonias ES para la identificación de algunos clones con mutaciones resultantes de Recursos humanos con el objetivo de vectores18. Los métodos del oro para la identificación de eventos de recursos humanos incluyen Southern blot y PCR19,20. Las ventajas de los enfoques incluyen que borrar meridional puede identificar clones ES correctamente dirigidas y permite a los investigadores a analizar la estructura del evento dirigido de genes, como una verificación de una inserción de copia única de la construcción, mientras que un Estrategia basada en la PCR permite la detección más rápida de HR eventos21,22. Aunque estos métodos tienen inconvenientes, como que son lentos y pueden tener falsos positivos, el uso de la combinatoria de ellos es ampliamente aceptados y aplicados por los laboratorios más en la identificación de eventos de recursos humanos, particularmente en células madre embrionarias, para generar genéticamente modificado animales.
Tres isoformas de la miosina UROLOGICAL II (NM II) en los mamíferos, cada uno con dos IIs NMHC idénticos que son codificadas por tres genes diferentes (llamado Myh9 Myh10 y Myh14) y dos pares de cadenas ligeras, se denominan NM II-A, II B y II-C23. Estudios previos han indicado que al menos las isoformas de NM II-A y II-B son esenciales para el desarrollo del ratón debido a la ablación en vivo de estas isoformas en mortalidad embrionaria24,25,26. Para evitar este problema y obtener nuevos conocimientos sobre las funciones específicas de la isoforma de NM II-A y II-B en las últimas etapas del desarrollo del ratón, un reemplazo genético estrategia tecnología ES celular HR-mediated gene targeting se adoptó para generar una serie de modelos de ratón27. En el curso de identificación de los clones ES deseados, borrar meridional y los métodos de PCR fueron utilizados, y estas demostraron para ser eficiente y confiable27,28.
Este documento pretende proporcionar una descripción detallada de Southern blotting y PCR, incluyendo el diseño de la focalización vector(s), sonda y cartillas y la ejecución de los experimentos, así como el análisis de resultados ejemplificados mediante la identificación de eventos de recursos humanos ocurrencia en células madre embrionarias para la creación de ratón de recambio genético NM II modelos y datos representativos. Los protocolos de estos dos métodos presentados aquí también pueden ser adoptados para la identificación de los genotipos de células genéticamente modificadas o los animales.
Protocolo
1. diseño de la focalización Construct(s), puntas de prueba de Southern Blot y cebadores para PCR
- Seleccione el primer exón (exón 2) codificación del gen Myh9 de inserción o interrupción en la aplicación del nocaut/knock-in divulgado aquí.
- Recuperar las 5 kb aguas arriba y 5 kb abajo secuencias de ADN que rodea el Myh9 exón 2 de la Página Web genome.ucsc.edu .
- Analizar patrones de digestión de la restricción de enzimas (REs) con cortes de 1-2 en esta región de 10 kb, utilizando el software de pDRAW para determinar RE(s) conveniente para digerir el ADN genómico para borrar meridional.
Nota: Dra cumple este requisito y se selecciona para el propósito. - Seleccione un fragmento de 4 kb inmediato aguas arriba del exón Myh9 2 como el brazo izquierdo de homología (LHA) y una secuencia descendente inmediata de 1,7 kb fragmento como el brazo derecho de homología (RHA); elegir un fragmento de 1 kb 5' aguas arriba de la LHA como la sonda de izquierda (LP) para borrar meridional y un fragmento de 1,2 kb 3' aguas abajo de la RHA como la punta de prueba derecho (RP), basado en el análisis anterior.
- Utilice un programa primer3 para diseñar los cebadores de avance y retroceso para amplificar los cuatro fragmentos de DNA por PCR. Diseño de un par de primers con el primer avance residido cerca de la terminal 3' de un gen de resistencia del neomyocin del marcador de la selección (P1) y la cartilla reversa ubicado a las afueras de la RHA (P2).
Nota: Este par de imprimación se utilizará para la identificación de clones ES dirigidas por PCR29. - Encontrar un clon de BAC 129Sv cubriendo el lugar geométrico del gene de ratón Myh9 visitando el sitio web de bacpac.chori.org (Nota: ADN isogénicas es preferido). Aislar ADN BAC utilizando un kit adecuado para purificar los pedazos grandes de ADN siguiendo las instrucciones proporcionadas por el fabricante.
Nota: Se utilizará como plantilla de ADN purificado de BAC para amplificación por PCR. - Dibujar una representación esquemática del targeting construcciones, sondas y primers para resumir esta información.
2. generación de apuntar Construct(s) y puntas de prueba de Southern Blot y la preparación de los Primers para PCR
- Ordenar los cebadores PCR descritos anteriormente y disolverlos en una concentración de 20 μm.
- Amplificar los brazos de homología y las puntas de prueba por PCR en una solución de reacción incluye 1 μl μl hacia adelante y 1 reversa imprimaciones, 1 μl de ADN BAC (50 ng) como la plantilla, 5 μl de tampón de Pfu y 1 μl de Pfu ultra como la ADN polimerasa , y H2O hasta 50 μl. llevar a cabo la polimerización en cadena en una máquina PCR bajo las siguientes condiciones: 95 ° C por 3 min; 95 ° C por 30 s; 60 ° C por 30 s; 72 ° C y a 1 kb/min; 30 ciclos; por último, 72 ° C 10 minutos.
- Purificar productos PCR utilizando un kit de limpieza PCR según el protocolo proporcionado por el fabricante y eluir los fragmentos de ADN con 40 μl de H2O. Resumen de los productos PCR purificados con REs prediseñados en una solución de reacción que contiene 5 μl de tampón de x 10 para la REs , 2.5 μl de RE1 y 2,5 μl de RE2 y el ADN eluído, a 37 ° C por 2 h. correr un gel de DNA 1% separar bandas de ADN blanco, suprimir el gel que contiene el ADN bajo luz UV, Diana purificar los fragmentos de DNA de la blanco del gel con una extracción de gel de DNA kit según en el protocolo proporcionado por el fabricante y eluir los fragmentos de ADN con 40 μl de H2O.
- Clonar los brazos de homología y casetes de expresión de reemplazo en el vector de mpNTKV-LoxP según el orden de los amplificados y purificados RHA, LHA y reemplazo expresión casetes de otros vectores para obtener el objetivo final Construct(s). Clonar fragmentos de ADN de las sondas amplificadas y purificadas en el vector T-fácil.
- Las secuencias de nucleótidos de todos los fragmentos de ADN clonados para confirmar la secuencia27,28.
3. preparación de apuntar el Construct(s), la electroporación de células madre embrionarias y la amplificación de los Clones ES
- Preparar cada dirigida a construir usando un plásmido maxiprep kit según el protocolo proporcionado por el fabricante. Alinear cada plásmido construcción por digerir no i en una reacción de 400 μL incluyendo 40 μl de buffer 10 x para no I, 10 μl de no, 100 μg de DNA y H2O hasta 400 μL, a 37 ° C durante la noche.
- Purificar el construct(s) segmentación lineal.
- Extraer la solución de reacción digerido 1 x con un volumen igual de Alcohol de fenol: cloroformo: isoamílico (25:24:1) y centrifugar a una fuerza de 2.000 x g por 10 min.
- Transferir el sobrenadante a un tubo nuevo de 1,5 mL, precipitar el ADN con 2.5 x etanol y 0.1 x 3M acetato de sodio (pH 5,2) (cociente del volumen) y centrifugar a una fuerza de 2.000 x g por 10 min.
- Eliminar el sobrenadante, lavado el ADN 1 x con 1 mL de etanol al 75% de pellets y centrifugar a una fuerza de 2.000 x g durante 5 minutos.
- Quite el sobrenadante y secar el pellet de ADN durante 5 minutos.
- Disolver el precipitado de DNA lineal en estéril buffer Tris-EDTA (TE) a una concentración final de 1 μg/μl.
- Mezclar 50 μg de cada construcción segmentación lineal con 0,5 x 107 ES células. Realizar la electroporación en 320 V y 250 μF. placa las células electroporated ES en platos con alimentadores MEF neo-resistente.
- Después de 24 h, cambie a medio de celular ES con 400 μg/mL G418 y 200 μm ganciclovir y continúan cultura de 4 – 5 días con un cambio medio diario. Recoger resistente a los medicamentos ES clones en placas de 48 pocillos.
Nota: Normalmente, se utilizan cuatro placas de 48 pocillos por construir. - Duplicar las placas 48-bien.
Nota: Un sistema de las placas es criopreservado, y el otro sistema se utiliza para la preparación de ADN genómico.
4. preparación de DNAs Genomic y la digestión con Restriction Enzyme(s)
- Preparar gDNAs de células madre embrionarias utilizando un kit comercial (kit de purificación de ADN genómico) con modificaciones menores.
- Quitar los medios de comunicación de la cultura de célula ES y agregar 500 μl de solución de lisis de núcleos, incluyendo RNaseA, directamente a los pozos para lisar las células.
Nota: El lisado celular puede ser almacenada a-80 ° C o tratada inmediatamente. - Pipeta de arriba y abajo varias veces para lisar las células completamente y transferirlos a un tubo limpio de 1,5 mL.
- Añadir un tercio del volumen de la solución de precipitación de la proteína en el tubo de 1,5 mL, vortex vigorosamente durante 20 s, enfriar las muestras en hielo durante 5 minutos y luego centrifugar a una fuerza de 2.000 x g durante 5 min transferencia el sobrenadante a otro limpio de 1,5 mL tubo contai Ning un volumen igual de isopropanol; mezclar suavemente la solución. (Nótese que blancas hebras de hilo-como pueden verse en este momento). Centrifugar a una fuerza de 2.000 x g durante 1 min; a continuación, descartar el sobrenadante.
- Lavar el sedimento gDNA con 1 mL de etanol al 70% a temperatura ambiente, centrifugar a una fuerza de 2.000 x g durante 1 min, aspirar el sobrenadante con cuidado y luego, secar el pellet gDNA por 3 min.
- Disolver el gDNAs con 100 μl de solución de rehidratación de ADN y, luego, incubar a 65 ° C por 1 h o a 4 ° C durante la noche.
- Tienda el gDNAs a 2 – 8 ° C.
- Quitar los medios de comunicación de la cultura de célula ES y agregar 500 μl de solución de lisis de núcleos, incluyendo RNaseA, directamente a los pozos para lisar las células.
- Resumen de la gDNAs con prediseñados RE Dra I. situado encima de una reacción de digestión 30 μL mezclando 3 μl de 10 x tampón para Dra I, 3 μl de Dra I, 10 μg de muestra de gDNAs y H2O hasta 30 μl e incubar a 37 ° C durante la noche.
- Comprobar la integridad de la digestión por el gel de ADN, análisis de 5 μl de la reacción digerida y luego agregar 3 μl de 10 x buffer de carga de ADN para el paso posterior.
5. Southern blot y PCR identificación
- Southern blot proyección
- Separar la gDNAs digerido por electroforesis y transferencia a una membrana.
- Preparar un gel de electroforesis de agarosa al 1% con bromuro de etidio (EB), cargar las muestras de paso 4.3 y una escalera de 1 kb y correr el gel con una tensión baja (30-40 V) durante la noche.
- Sacar el gel y tomar una foto con un sistema de proyección de imagen de gel de ADN después de la electroforesis. Compruebe si el digerido y separados gDNAs mostrar una borrón de transferencia de imagen.
- Remojo el gel en una bandeja con solución de ácido clorhídrico N 0.2 y agitar suavemente durante 20 min a temperatura ambiente.
- Transferir el gel a la solución de desnaturalización de DNA y agitar suavemente durante 20 min a temperatura ambiente.
- Cambiar el gel en la solución de ADN neutralizando y agitar suavemente durante 20 min a temperatura ambiente.
Nota: El gel es propenso a la rotura después de este paso, por lo que debe ser manipulado con cuidado. - Utilice el sistema de transferencia hacia abajo rápido para transferir el ADN del gel a la membrana. Monte la TurboBlotter y Blot pila según las instrucciones proporcionadas por el fabricante.
Nota: 10 x o 20 x solución de citrato de sodio solución salina (SSC) se utiliza como un buffer de transferencia. En general, 3 h de transferencia es bastante para transferir el 95% de gDNAs de gel a la membrana; sin embargo, un tiempo de transferencia es inofensivo. - Sacar la membrana y lavar con 2 x SSC de 1 minuto, absorber el líquido con los tejidos y luego reaccione el ADN con la membrana usando un crosslinker UV.
Nota: La membrana puede almacenarse a 4 ° C durante una semana.
- Etiqueta de las sondas de ADN con radiactividad.
- Purificar la plásmidos de la sonda utilizando un miniprep kit según el protocolo proporcionado por el fabricante.
- Liberación del ADN fragmentos de las sondas del vector plásmido por EcoR digestión en una solución de reacción incluyendo 5 μl de tampón de EcoR I, 2 μl de EcoR I enzima, 20 μg de plásmido ADN y H2O hasta 50 μl , por 2 h.
- Ejecutar un 1% gel de DNA para separar los fragmentos de ADN de la sonda desde el vector y purificar los fragmentos de ADN de las sondas con un kit de extracción de gel de ADN según el protocolo proporcionado por el fabricante.
- Utilizando 1 μl de solución de ADN, medir la concentración de ADN de los fragmentos de ADN de la sonda con un espectrofotómetro a una longitud de onda de 260/280 nm.
- Preparar 40 ng de sonda de ADN en un tubo de 1,5 mL con 45 μl de tampón TE, hervir durante 3 minutos, girar brevemente y luego, coloque los tubos en hielo durante 2 minutos.
- Añadir el ADN sonda desnaturalizada por el calor al tubo que contiene granos de etiquetado ADN listo a (-dCTP), pipeta hacia arriba y hacia abajo para mezclar, añadir 5 μl de [α32P] dCTP y luego, incubar a 37 ° C durante 15 minutos.
- Purificar las puntas de prueba etiquetadas usando microcolumns G-50 según las instrucciones proporcionadas por el fabricante y, luego, medir la radiactividad de un contador de centelleo (opcional).
- Hibridar el membrane(s) con las puntas de prueba etiquetadas.
- Prehybridize la membrana.
- Precaliente la solución de hibridación a 42 ° C para 30 min mezclar 20 mL de solución de hibridación precalentado con 200 μg de esperma de salmón hervida ADN en un tubo de 50 mL.
- Colocar la membrana en el tubo de hibridación. Agregar la solución prehybridization mezclada al tubo de hibridación. Coloque en el horno de hibridación (sistema del balanceo y la temperatura a 42 ° C) y dejar que la prehybridization durante 30 minutos.
- Hibridar la membrana con la sonda marcada.
- Sacar el tubo de hibridación y verter la solución prehybridization en un tubo de 50 mL; Añadir la sonda desnaturalizada (calentada a 100 ° C por 3 min) de paso 5.1.2.7 a este tubo y mezcla suavemente.
Nota: Reducir cualquier inducir burbujas. - Devolver la solución mezclada al tubo de hibridación y efectuar la hibridación a 42 ° C durante la noche.
- Sacar el tubo de hibridación y verter la solución prehybridization en un tubo de 50 mL; Añadir la sonda desnaturalizada (calentada a 100 ° C por 3 min) de paso 5.1.2.7 a este tubo y mezcla suavemente.
- Prehybridize la membrana.
- Lavar la membrane(s) para quitar las puntas de prueba nonhybridized.
- Coloque el membrane(s) en una bandeja con 1 x SSC + 0.1% SDS y agitar suavemente en el 55 – 60 ° C durante 10 minutos.
- Transferir el membrane(s) a una bandeja con 0,5 x SSC + SDS 0.1% y agitar suavemente en el 55 – 60 ° C durante 10 minutos.
- Comprobar la radiactividad en el membrane(s) mediante un contador Geiger portátil para decidir si un tercer lavado es necesario.
- Exponga la radiactividad en la membrana a películas de rayos x.
- Retire el líquido de los lavados membrane(s).
- Envuelven la membrane(s) con envoltura de plástico y fijar it/ellos en el cassette de la exposición.
- Exponer la membrana dos hojas de película de rayos x en un cuarto oscuro.
- Coloque el cartucho de la exposición a-80 ° C durante la noche o más.
- Desarrollar las películas para visualizar los resultados. Evaluar si un clon ES correspondiente es la deseada con la recombinación específica o no, según los tamaños de las bandas de ADN detectados por las sondas.
- Rehybridize la misma membrana por otra sonda después de la extracción de la sonda usada según el siguiente procedimiento: sacar la membrana utilizada, lavar 1 x con limpiar el H2O y luego, la incubar en la solución de bandas (55% de formamida, 2% PES, 1% SDS, H 2 O) a 65 ° C con agitación suave durante 1 – 2 h.
- Separar la gDNAs digerido por electroforesis y transferencia a una membrana.
-
Identificación de la PCR
- Realizar la identificación de la polimerización en cadena de los clones ES deseados en una solución de reacción de 50 μl incluyendo 5 μl de buffer de x PCR 10, 2 μl de 50 mM MgSO4, 1 μl de dNTP 10 mM, 1 μl de cebador forward 20 μm, 1 μl de cebador inverso de 20 μm , 1 μl de Taq platinum de alta fidelidad, gDNAs (~ 100 ng) y H2O hasta 50 μl.
- Utilice las siguientes condiciones de reacción de PCR: una desnaturalización inicial a 94° C por 3 min, 30 ciclos de desnaturalización a 94° C por 30 s, recocido a 60° C por 30 s y una extensión a 68° C 132 s y un paso final de 68° C durante 10 minutos.
- Analizar los productos PCR por electroforesis en agarosa al 1.0%.
- Clonar los fragmentos PCR con el tamaño esperado en el vector T-easy y la secuencia para confirmar la presencia de una secuencia parcial del vector objetivo.
Resultados
En este trabajo se describe un protocolo detallado de Southern blot y de PCR, que se utiliza para identificar eventos de HR que se produjeron en las células ES de ratón para la generación de modelos de ratón de recambio genético NM II, tecnología ES basada en células mediada por recursos humanos dirigidos a. Aunque borrar meridional y PCR, así como técnica de gene targeting tradicional, ha sido ampliamente utilizado desde hace varias décadas, la aplicación exitosa de ellos debe ser planeado cuidadosamente. Al menos estos aspectos deben considerarse: la longitud de los brazos largos y cortos, las posiciones y la longitud de las sondas, las REs adecuados para cortar el DNAs genómicos y los cebadores para PCR, como se resume en la figura 1, que es útil para análisis subsecuente. Como un paso importante de borrar meridional, están obligados a separar las DNAs genomic preparadas y digeridas en gel de la DNA para la detección por la sonda. Porque DNAs genomic se cortan en una gran cantidad de fragmentos con diferentes longitudes, indica que un borrón de transferencia-como en el gel de la DNA, sugiriendo una digestión completa de los DNAs genómicos, como se indica en la figura 2. Como paso final de borrar meridional, las señales de una sonda marcada con radiactividad cruzamiento por hibridación con un fragmento de DNA de la blanco aparecen en la película, que refleja la ocurrencia de eventos de recursos humanos en los clones ES, lo que indica si un clon ES es la deseada. Según el prediseño en este estudio, ES clones con el alelo mutado tienen dos bandas de distinto tamaño, mientras que los clones de tipo salvaje ES sólo tienen una banda, lo que sugiere los clones ES deseados son heterozigóticos (figura 3). Comparado con el procedimiento y los resultados de borrar meridional, la operación y resultados de PCR son simples y directos. Después de la reacción de PCR, se pueden analizar los productos PCR en el gel de DNA. Si son específicas de las bandas PCR y secuenciación de los productos PCR clonados confirma la presencia de una secuencia parcial del vector objetivo como un gen de resistencia a la neo, así como regiones genómicas que están a las afueras del brazo de la homología, la ocurrencia de eventos de recursos humanos puede ser espera y verificado (figura 4).
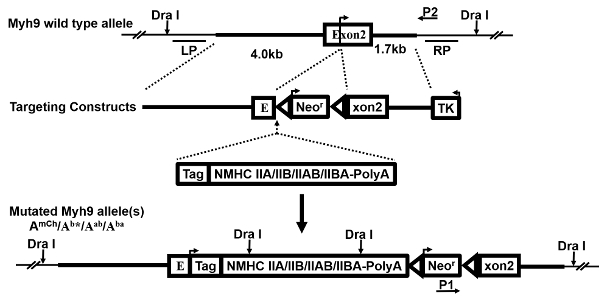
Figura 1 : Orientación construcciones. Este es un esquema que demuestra la generación de constructos objetivos múltiples. El alelo de tipo salvaje (WT) Myh9 gene, gene targeting vector, casetes de expresión exógena de recambio y los alelos mutados resultante, así como las sondas (LP, RP) de Southern blot y los cebadores (P1, P2) para PCR, se muestran y se ha descrito anteriormente 27. una flecha en el exón 2 indica el sitio de iniciación de traslación. Tras la exitosa aparición de HR, se insertaron el casete de expresión de reemplazo y el gen de resistencia a neomicina (Neor) a sólo 5' del iniciación codón ATG. Por lo tanto, el alelo de Myh9 endógeno se interrumpe y el golpeó en los genes es se expresan en las células mutantes y ratones. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2 : Digerir DNAs genómicos con Dra I. DNAs genomic de clones ES objeto de la construcción sustitución de NMHC II A II b son digeridos con la Dra y, a continuación, separados en un gel de agarosa por electroforesis. Se observa un gDNA digerido como borrón de transferencia. C1 - C8 representan clones ES individuales. Una digestión completa del gDNA produce gran cantidad de fragmentos de ADN con una longitud diferente, mostrando así una imagen de desprestigio. Este resultado refleja también la buena calidad de gDNAs preparado y lo completo de la digestión. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3 : Resultados representativos de borrar meridional. Estos paneles muestran una proyección Blot Southern de DNAs genomic de clones ES con el concepto de sustitución de NMHC II-A con II-AB, usando las sondas de izquierda y derecha. El alelo mutado muestra una banda 12,1 kb o 6 kb cuando la sonda izquierda o derecha punta de prueba se utiliza, respectivamente, mientras que el peso muestra una banda de 9,7 kb. M: marcador; PC1-PC5: clones positivos; NC: clon negativo. También se indican los tamaños de las bandas Southern blot. Se realizan estrictamente todos los procedimientos de borrar meridional y la especificidad de las sondas es suficiente; no debería haber específico bandas esperan para las bandas esperadas. Haga clic aquí para ver una versión más grande de esta figura.
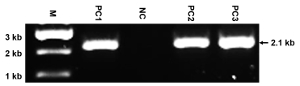
Figura 4 : Resultados representativos de PCR. Este panel muestra la identificación de la PCR de los ADNs genómicos de clones ES con el concepto de sustitución de NMHC II-A con II-BA con el primer par P1 + P2. El alelo mutado produce una banda de 2,1 kb, mientras que el alelo WT no produce ninguna banda. M: marcador; PC1-PC3: clones positivos; NC: clon negativo. También se indica el tamaño de la banda de la polimerización en cadena. Puesto que los iniciadores están diseñados para sólo detectar el alelo mutado, la aparición de una banda única y esperada refleja la especificidad de los cebadores y la alta calidad de los preparados gDNAs. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
En la actualidad, diseñador nucleasas para la edición del genoma todavía no pueden reemplazar ES célula gene targeting tecnología debido a sus problemas de efectos off-target y dificultad en la inserción de un ADN largo fragmento30,31. Como los métodos de oro para identificar eventos de HR que se producido en células de ratón ES, este informe proporciona un protocolo detallado de Southern blot y de PCR para el campo. Validamos la fiabilidad de estos métodos mediante el análisis de clones individuales de células ES de ratón con una serie de construcciones. Los clones ES deseados identificados por estos métodos se habían utilizados con éxito para generar los correspondientes modelos de ratón27.
Aunque otras técnicas para la selección de clones específicos de ES han descrito19,32, los métodos de Southern blot y PCR no puede ser reemplazado totalmente por ésos establecidos después32, porque estas iniciales técnicas tienen ya historia aplicada y son ampliamente aceptadas y confirmaron por la sociedad científica, realizada por los laboratorios biológicos la mayoría y son el origen de otras tecnologías. Lo importante, el buen desempeño de Southern blot y de PCR en la identificación de eventos de recursos humanos se ejemplifica bien en el anterior trabajo29. Los resultados de borrar meridional indican varias características únicas: entre los clones ES aleatoriamente seleccionados, sobre 90% de ellos son deseados, no vendas son detectadas, y la hora ocurrió preferentemente en un alelo del gen Myh9. Mientras tanto, los datos de PCR, junto con la secuencia, confirman que la ocurrencia de eventos de recursos humanos específica y coinciden bien con los de borrar meridional.
Según nuestra práctica, se deben considerar varios factores cuando Southern blot y PCR se utilizan para identificar eventos de recursos humanos en células madre embrionarias, obteniendo así buenos resultados esperados. La primera de ellas es la longitud de los brazos de la homología; en general, aumentando la longitud del brazo de homología mejorará la eficiencia de los recursos humanos33. Sin embargo, esto no es siempre el caso. Por un lado, los brazos más largos aumentan la dificultad de manipulación; por otro lado, la longitud de los brazos de homología (4 kb para el brazo izquierdo y 1,7 kb para el brazo derecho) divulgada aquí dio lugar a la frecuencia más alta de h obtenida hasta el momento entre experimentos similares. Adicionalmente, una longitud razonable de brazos de homología facilita la identificación por PCR. La segunda es la utilización de ADN isogénicas para la preparación de los brazos de homología y Southern blot sondas34. Esto puede ser satisfecho, ordenando un clon BAC que contiene la región del gen de interés o usando ADN genómico de las células que se pretende ser objetivo. La tercera es la selección de resolución adecuado para digerir DNAs genomic. En general, se prefieren; uno RE o la combinación de dos REs que el alelo de tipo salvaje o mutante sólo una vez o dos veces alrededor de la región dirigida a Además, el fragmento de ADN mayor resultante no debe exceder 15 kb y la diferencia de tamaño entre los distintos fragmentos de ADN es de más de 2 kb. Estos requisitos pueden facilitar la separación y la identificación de bandas esperadas por borrar meridional. La cuarta es la longitud de las sondas y la menos semejanza con otras secuencias en el genoma. Generalmente, la longitud de las sondas es de 500 – 1.000 bp. La similitud con otras secuencias en el genoma puede ser analizada con el programa BLAST del NCBI. Además, un software para el diseño de las sondas para borrar meridional ha sido descrito35. El quinto factor a considerar es usar los métodos convencionales para preparar la DNA genomic para una mejora de rendimiento. DNAs genomic de un confluente pozo de una placa de 48 pozos suelen ser suficiente para al menos dos rondas de análisis Blot Southern. En cuanto a diseño de los cebadores para PCR, la mejor estrategia es usar una cartilla presente en el marcador de selección junto con una cartilla fuera de los brazos dirigidos a. Además, los productos PCR de secuenciación es importante probar HR eventos20,36. En particular, basado en el PCR no puede sustituir totalmente la información obtenida a través de borrar meridional, mientras que puede reducir con eficacia el número de clones que se evaluará.
En conclusión, Southern blot y PCR son métodos bien demostrados para la detección de clones ES para identificar mediada por HR eventos gene targeting en células madre embrionarias. Aunque el protocolo detallado descrito aquí principalmente se centró en la proyección deseada NM II reemplazo genético ES clones, puede ser utilizado para los ratones de genotipificación que posteriormente se generan con los clones ES positivo. Puede ser adaptado fácilmente a la identificación de eventos de recursos humanos en otros tipos celulares, como células iPS o células somáticas.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo recibió apoyo del Programa General de la Fundación Nacional de Ciencias naturales de China (subvenciones no. 31571432), el humano Provincial Ciencias naturales Fundación de China (Grant No. 2015JC3097) y la investigación Fundación de Educación Oficina de Hunan Provincia, China (Grant no. 15 K 054).
Materiales
| Name | Company | Catalog Number | Comments |
| BAC CLONE | BACPAC Resources Center (BPRC) | bMQ-330E21 | |
| QIAGEN Large-Construct Kit | QIAGEN | 12462 | |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| QIAprep Spin Miniprep Kit | QIAGEN | 27104 | |
| QIAGEN Plasmid Plus Maxi Kit | QIAGEN | 12963 | |
| PfuUltra High-Fidelity DNA Polymerase | Agilent | 600382 | |
| T-easy vector | Promega | A1360 | |
| Nuclei Lysis Solution | Promega | A7941 | |
| Protein Precipitation Solution | Promega | A7951 | |
| DNA Denaturing Solution | VWR | 351-013-131 | |
| DNA Neutralizing Solution | VWR | 351-014-131 | |
| Ready-To-Go DNA Labeling Beads (-dCTP) | VWR | 27-9240-01 | |
| UltraPure SSC, 20x | Thermo Fisher | 15557036 | |
| UltraPur Phenol:Chloroform:Isoamyl Alcohol (25:24:1, v/v) | Thermo Fisher | 15593031 | |
| G418 | Thermo Fisher | 10131035 | |
| Salmon Sperm DNA Solution | Thermo Fisher | 15632011 | |
| Platinu Taq DNA Polymerase High Fidelity | Thermo Fisher | 11304029 | |
| Not I | Thermo Scientific | ER0592 | |
| Dra I | Thermo Scientific | ER0221 | |
| EcoR I | Thermo Scientific | ER0271 | |
| Ganciclovir | Sigma | G2536 | |
| Whatman TurboBlotter Transfer System, Large Kits | Fisher Scientific | 09-301-188 | |
| [α32P]dCTP | PerkinElmer | NEG013H100UC | |
| ProbeQuan G-50 Micro Columns | GE Healthcare | 28-9034-08 | |
| Hybrisol I Hybridization Solution | Millipore | S4040 | |
| Kodak X-Ray Film | Z&Z Medical | 844 5702 |
Referencias
- Gao, G., McMahon, C., Chen, J., Rong, Y. A powerful method combining homologous recombination and site-specific recombination for targeted mutagenesis in Drosophila. Proceedings of the National Academy of Sciences of the United States of America. 105 (37), 13999-14004 (2008).
- Skarnes, W., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474 (7351), 337-342 (2011).
- Vogel, G. Nobel Prizes. A knockout award in medicine. Science. 318 (5848), 178-179 (2007).
- Salsman, J., Dellaire, G. Precision genome editing in the CRISPR era. Biochemistry. Cell Biology. 95 (2), 187-201 (2017).
- Capecchi, M. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nature Reviews Genetics. 6 (6), 507-512 (2005).
- Van, d. W. L., Adams, D. J., Bradley, A. Tools for targeted manipulation of the mouse genome. Physiological Genomics. 11 (3), 133-164 (2002).
- Glaser, S., Anastassiadis, K., Stewart, A. F. Current issues in mouse genome engineering. Nature Genetics. 37 (11), 1187 (2005).
- Bradley, A., Evans, M., Kaufman, M. H., Robertson, E. Formation of germ-line chimaeras from embryo-derived teratocarcinoma cell lines. Nature. 309 (5965), 255-256 (1984).
- Robertson, E., Bradley, A., Kuehn, M., Evans, M. Germ-line transmission of genes introduced into cultured pluripotential cells by retroviral vector. Nature. 323 (6087), 445-448 (1986).
- Thomas, K. R., Capecchi, M. R. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells. Cell. 51 (3), 503-512 (1987).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474 (7351), 337 (2011).
- Collins, F. S., Rossant, J., Wurst, W. A mouse for all reasons. Cell. 128 (1), 9-13 (2007).
- Poueymirou, W. T., et al. F0 generation mice fully derived from gene-targeted embryonic stem cells allowing immediate phenotypic analyses. Nature Biotechnology. 25 (1), 91-99 (2007).
- Pettitt, S. J., et al. Agouti C57BL/6N embryonic stem cells for mouse genetic resources. Nature Methods. 6 (7), 493-495 (2009).
- Gertsenstein, M., et al. Efficient Generation of Germ Line Transmitting Chimeras from C57BL/6N ES Cells by Aggregation with Outbred Host Embryos. PLoS One. 5 (6), 11260 (2012).
- Smithies, O., Gregg, R. G., Boggs, S. S., Koralewski, M. A., Kucherlapati, R. S. Insertion of DNA sequences into the human chromosomal |[beta]|-globin locus by homologous recombination. Nature. 317 (6034), 230-234 (1985).
- Deng, C., Capecchi, M. R. Reexamination of gene targeting frequency as a function of the extent of homology between the targeting vector and the target locus. Molecular & Cellular Biology. 12 (8), 3365 (1992).
- Lay, J. M., Friishansen, L., Gillespie, P. J., Samuelson, L. C. Rapid confirmation of gene targeting in embryonic stem cells using two long-range PCR techniques. Transgenic Research. 7 (2), 135-140 (1998).
- Langerak, P., Nygren, A. O. H., Schouten, J. P., Jacobs, H. Rapid and quantitative detection of homologous and non-homologous recombination events using three oligonucleotide MLPA. Nucleic Acids Research. 33 (22), 188 (2005).
- Gómezrodríguez, J., et al. Advantages of q-PCR as a method of screening for gene targeting in mammalian cells using conventional and whole BAC-based constructs. Nucleic Acids Research. 36 (18), 117 (2008).
- Kim, H. S., Smithies, O. Recombinant fragment assay for gene targeting based on the polymerase chain reaction. Nucleic Acids Research. 16 (18), 8887-8903 (1988).
- Joyner, A. L., Skarnes, W. C., Rossant, J. Production of a mutation in mouse En-2 gene by homologous recombination in embryonic stem cells. Nature. 338 (6211), 153-156 (1989).
- Ma, X., Adelstein, R. S. The role of vertebrate nonmuscle Myosin II in development and human disease. Bioarchitecture. 4 (3), 88-102 (2014).
- Malonek, D. Relationships between the dynamics of cortical blood flow, oxygenation, and volume changes following sensory stimulation. Proceedings of the National Academy of Sciences of the United States of America. 94, (1997).
- Takeda, K., Kishi, H., Ma, X., Yu, Z. X., Adelstein, R. S. Ablation and mutation of nonmuscle myosin heavy chain II-B results in a defect in cardiac myocyte cytokinesis. Circulation Research. 93 (4), 330-337 (2003).
- Conti, M. A., Evenram, S., Liu, C., Yamada, K. M., Adelstein, R. S. Defects in cell adhesion and the visceral endoderm following ablation of nonmuscle myosin heavy chain II-A in mice. Journal of Biological Chemistry. 279 (40), 41263-41266 (2004).
- Wang, A., et al. Nonmuscle myosin II isoform and domain specificity during early mouse development. Proceedings of the National Academy of Sciences of the United States of America. 107 (33), 14645-14650 (2010).
- Zhang, Y., et al. Mouse models of MYH9-related disease: mutations in nonmuscle myosin II-A. Blood. 119 (1), 238 (2012).
- Liu, T., et al. Identification and characterization of MYH9 locus for high efficient gene knock-in and stable expression in mouse embryonic stem cells. PLoS One. 13 (2), 0192641 (2018).
- Saito, S., Adachi, N. Advances in the Development of Gene-Targeting Vectors to Increase the Efficiency of Genetic Modification. Biological & Pharmaceutical Bulletin. 39 (1), 25-32 (2016).
- Langerak, P., Nygren, A., Schouten, J., Jacobs, H. Rapid and quantitative detection of homologous and non-homologous recombination events using three oligonucleotide MLPA. Nucleic Acids Research. 33 (22), 188 (2005).
- Martin, S. L., et al. A single amino acid substitution in ORF1 dramatically decreases L1 retrotransposition and provides insight into nucleic acid chaperone activity. Nucleic Acids Research. 36 (18), 5845-5854 (2008).
- Kamisugi, Y., Cuming, A. C., Cove, D. J. Parameters determining the efficiency of gene targeting in the moss Physcomitrella patens. Nucleic Acids Research. 33 (19), 173 (2005).
- Luo, Y., Bolund, L., Sørensen, C. B. Pig gene knockout by rAAV-mediated homologous recombination: comparison of BRCA1 gene knockout efficiency in Yucatan and Göttingen fibroblasts with slightly different target sequences. Transgenic Research. 21 (3), 671-676 (2012).
- Croning, M. D., Fricker, D. G., Komiyama, N. H., Grant, S. G. Automated design of genomic Southern blot probes. BMC Genomics. 11 (1), 74 (2010).
- Zimmer, A., Gruss, P. Production of chimaeric mice containing embryonic stem (ES) cells carrying a homoeobox Hox 1.1 allele mutated by homologous recombination. Nature. 338 (6211), 150-153 (1989).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados