Method Article
Identifikation der homologen Rekombinationsereignisse in embryonalen Stammzellen der Maus mit Southern Blotting und Polymerase-Kettenreaktion
* Diese Autoren haben gleichermaßen beigetragen
In diesem Artikel
Zusammenfassung
Hier präsentieren wir Ihnen ein detailliertes Protokoll zur Identifizierung von homologen Rekombinationsereignisse in embryonalen Stammzellen der Maus mit Southern blotting und/oder PCR. Diese Methode ist durch die Generierung von Nonmuscle Myosin II genetische Ersatz Mausmodellen mit traditionellen embryonalen Stammzell-basierte homologe Rekombination-vermittelten targeting-Technologie veranschaulicht.
Zusammenfassung
Im Verhältnis zu den Fragen der Ziel-Effekte und die Schwierigkeit des Einsetzens einer langen DNA-Fragment in der Anwendung von Designer Nukleasen für Genom Bearbeitung, embryonale Stammzellen (ES) Zell-basierte gen-targeting Technologie verfügt nicht über diese Mängel und ist weithin verwendet, um Tier/Maus Genom von großen Deletionen/Insertionen bis hin zu Einzel-Nukleotid-Substitutionen ändern. Vor allem gewünscht Identifizierung von relativ wenigen homologe Rekombination (HR) Veranstaltungen notwendig ES-Klone ist ein wichtiger Schritt, der genaue und zuverlässige Methoden verlangt. Südliche beflecken und/oder konventionellen PCR werden häufig für diesen Zweck genutzt. Hier beschreiben wir die Modalitäten der Verwendung dieser beiden Verfahren HR Ereignisse zu identifizieren, die in ES-Zellen aufgetreten in denen endogene Myh9 gen gestört und durch cDNAs Codierung andere Nonmuscle Myosin schwere Kette IIs (NMHC IIs) ersetzt werden soll. Das gesamte Verfahren der Southern blotting umfasst den Bau von Vector(s), Elektroporation, Droge-Auswahl, Ausbau und Lagerung von ES-Zellen/Klone, die Vorbereitung, Verdauung-targeting und Beflecken der genomic DNA (gDNA), die Hybridisierung und Waschen der Wortanzahl und letzten Schritt der Autoradiographie auf die x-ray-Filme. PCR kann direkt mit vorbereitet und verdünnte gDNA durchgeführt werden. Um optimale Ergebnisse zu erzielen, sollten die Sonden und Restriktionsenzym (Re-) schneiden Websites für das südliche beflecken und die Primer für PCR sorgfältig geplant werden. Obwohl die Ausführung von Southern blotting ist zeitaufwendig und arbeitsintensiv und PCR-Ergebnisse sind Fehlalarme, die korrekte Identifizierung von Southern blotting und die schnelle Screening mittels PCR erlauben die alleinige oder kombinierte Anwendung dieser Methoden beschrieben in diesem Papier auf allgemein verwendet und Anhörung durch die meisten Labore bei der Identifizierung von Genotypen von ES-Zellen und genetisch veränderter Tiere.
Einleitung
Die Technologie der Gene targeting-HR in murinen embryonaler Stammzellen ist ein leistungsstarkes Tool für sezieren die zellulären Folgen von bestimmten Genmutationen1,2. Die Bedeutung und die Bedeutung dieser Technologie spiegeln sich in der Anerkennung von 2007 den Nobelpreis für Physiologie oder Medizin3,4; Unterdessen stellt es das Aufkommen der modernen Ära von gen-engineering-5. Gene targeting durch HR kann genutzt werden, um praktisch jede Änderung von Punktmutationen bis hin zu großen chromosomale Rearrangements im Genom der Maus ES Zellen6,7-Ingenieur. Es ist bekannt, dass vor der Entstehung der sogenannten Genome editing-Tools, die Generation von einer gen-Knockout-Maus die Anwendung der Gene-targeting Technologie ES Zellen8,9,10erforderlich. In den vergangenen zwei Jahrzehnten wurden mehr als 5.000 gen gezielt Mäuse durch diesen Ansatz für die Modellierung menschlicher Erkrankungen oder studieren gen Funktionen11produziert. Eine genomweite Ko Anstrengung hat sich etabliert für die Verteilung von gen-targeting Vektoren, gezielte ES Zellklonen und live Mäuse an die wissenschaftliche Gemeinschaft2,12,13,14 , 15. zweifellos, ES zellbasierte HR-mediated Gene-targeting-Technologie hat stark erweiterte unser Verständnis der Funktionen von Genen in physiologische oder pathologische Kontext gespielt.
Weil HR ist eine relativ seltene Ereignis in Säugetier-Zellen16,17, die wichtig und nächster Schritt nach soll gen gezielt in murinen Embryonische Stammzellen zahlreiche ES Kolonien zur Identifizierung von ein paar Klone mit Mutationen aus analysieren Mit dem targeting Vektor-18Std. Die gold Methoden zur Identifizierung von HR-Veranstaltungen gehören südliche beflecken und PCR19,20. Die Vorteile der Ansätze beinhalten, dass Southern blotting richtig gezielte ES-Klone zu identifizieren und erlaubt es den Forschern, die Struktur der gen ausgerichtete Veranstaltung, wie eine Überprüfung der eine einzelne Kopie Einfügung des Konstrukts, analysieren während einer PCR-basierte Strategie ermöglicht eine schnellere Screening für HR Veranstaltungen21,22. Obwohl diese Methoden Nachteile, wie z. B. haben, dass sie sind zeitaufwendig und können Fehlalarme, die kombinatorische Nutzung von ihnen ist weithin akzeptiert und angewendet, indem die meisten Labore bei der Identifizierung von HR-Veranstaltungen, insbesondere in ES-Zellen für Erzeugung genetisch geändert Tiere.
Drei Isoformen von Nonmuscle Myosin II (NM II) bei Säugetieren, jeweils bestehend aus zwei identischen NMHC IIs die durch drei verschiedene Gene (benannte Myh9, Myh10 und Myh14) und zwei Paare von Lichterketten, kodiert sind gekennzeichnet als NM II A, II B und II-C23. Frühere Studien haben gezeigt, dass mindestens der Isoformen von NM II A und II B sind wesentlich für Maus Entwicklung, da die in Vivo -Ablation von diese Isoformen embryonalen Tödlichkeit24,25,26führt. Um dieses Problem zu umgehen und erhalten neue Einblicke in die Isoform-spezifischen Funktionen der NM II A und II B in den späteren Stadien der Maus Entwicklung, ein genetischer Austausch war ES zellbasierte HR-mediated Gene-targeting Technologie, Strategie erzeugen Sie eine Reihe von Maus Modelle27. Im Zuge der Ermittlung der gewünschten ES-Klone, Southern blotting und PCR-Methoden wurden eingesetzt, und diese erwies sich als effiziente und zuverlässige27,28.
Dieses Papier soll eine detaillierte Beschreibung der südlichen Beflecken und PCR, einschließlich der Gestaltung von targeting, Vector(s), Wortanzahl, und Primer und die Durchführung von Experimenten sowie die Analyse der Ergebnisse durch die Identifizierung von HR-Ereignis veranschaulicht Vorkommen in ES-Zellen für die Erstellung von genetischen Austausch NM II Maus Modelle und repräsentative Daten. Die Protokolle der beiden hier vorgestellten Methoden können auch für die Ermittlung der Genotypen von genetisch veränderten Zellen oder Tieren angenommen werden.
Protokoll
1. Aufbau des Zielens Construct(s), Sonden für südlicher Fleck und Primer für PCR
- Wählen Sie das erste kodierende Exon (Exon 2) des Gens Myh9 für Störungen oder Einfügung in der Anwendung von Ko/Knock-in hier berichtet.
- Rufen Sie die 5-kb vor- und 5 kb nachgeschaltete DNA-Sequenzen rund um das Myh9 Exon 2 von der genome.ucsc.edu -Website ab.
- Analysieren Sie Einschränkung Verdauung Muster von Enzymen (REs) mit 1 – 2 Schnitten in dieser 10-kb-Region mithilfe von pDRAW Software geeignet RE(s) genomischen DNA für das südliche beflecken zu verdauen zu bestimmen.
Hinweis: Dra ich erfüllt diese Anforderung und ist für den Zweck ausgewählt. - Wählen Sie ein 4 kb-Fragment unmittelbar oberhalb des Myh9-Exon 2 als linken Homologie Arm (LHA) und eine 1,7 kb Fragment unmittelbar nachgeschalteten Sequenz als richtige Homologie Arm (RHA); Wählen Sie eine 1 kb-Fragment 5' stromaufwärts von der LHA als die linke Sonde (LP) für Southern blotting und ein 1,2 kb-Fragment 3' stromabwärts von der RHA als die richtige Sonde (RP), anhand der obigen Analyse.
- Verwenden Sie ein primer3 Programm, um die vorwärts und rückwärts Primer für die vier DNA-Fragmente mittels PCR Verstärkung zu entwerfen. Entwerfen Sie ein paar Zündkapseln mit der forward Primer wohnte in der Nähe von Terminal 3' eine Auswahl Neomyocin Widerstand Markergens (P1) und die rückwärts-Primer befindet sich etwas außerhalb der RHA (P2).
Hinweis: Diese Primerpaar wird verwendet zur Identifizierung von gezielten ES-Klone durch PCR-29. - Einen 129Sv-BAC-Klon für Maus Myh9 gen-Locus durch den Besuch der Website bacpac.chori.org zu finden (Anmerkung: isogenen DNA wird bevorzugt). BAC-DNA mit einem Kit geeignet zur Reinigung von großer Stücken von DNA nach den Anweisungen des Herstellers zu isolieren.
Hinweis: Gereinigtes BAC-DNA wird als Vorlage für PCR Verstärkung verwendet werden. - Zeichnen Sie eine schematische Darstellung der Ausrichtung Konstrukte, Sonden und Grundierungen, diese Informationen zusammenzufassen.
2. Generation des Zielens, Construct(s) und Sonden für südlicher Fleck und die Vorbereitung der Zündkapseln für PCR
- Bestellen Sie die oben beschriebenen PCR-Primer und lösen sie in einer Konzentration von 20 µM.
- Die Homologie Arme und Sonden durch PCR in eine Reaktionslösung einschließlich 1 µL vorwärts- und 1 µL reverse Primer, 1 µL BAC DNA zu verstärken (50 ng) als die Vorlage, 5 µL Puffer für Pfu und 1 µL der Pfu Ultra als die DNA-Polymerase , und H2O bis 50 µL. die PCR in einer PCR-Maschine unter folgenden Bedingungen durchzuführen: 95 ° C für 3 min; 95 ° C für 30 s; 60 ° C für 30 s; 72 ° C und 1 kb/min; 30 Zyklen; zu guter Letzt 72 ° C für 10 Minuten.
- PCR-Produkte mit einem PCR-Bereinigung-Kit nach dem Protokoll des Herstellers zu reinigen und die DNA-Fragmente mit 40 µL H2O. Digest die gereinigten PCR-Produkte mit vordefinierten REs in eine Reaktionslösung mit 5 µL 10 X Puffer für REs eluieren , 2,5 µL RE1 und 2,5 µL RE2 und der eluierten DNA, bei 37 ° C für 2 h laufen eine 1 % DNA-Gel zum Ziel DNA-Bänder zu trennen, das Gel mit dem Ziel DNA unter UV-Licht, Verbrauchsteuern zu reinigen die Ziel-DNA-Fragmente aus dem Gel verwenden, eine DNA-Gel Extraction Kit nach zum Protokoll des Herstellers und eluieren die DNA-Fragmente mit 40 µL H2O.
- Klonen der Homologie Arme und Ersatz Ausdruck Kassette(n) in den MpNTKV-LoxP-Vektor nach der Reihenfolge der die verstärkte und gereinigten RHA, LHA und Ersatz Ausdruck Kassette(n) befreit andere Vektoren, die endgültige Ausrichtung zu erhalten Construct(s). Klonen Sie DNA-Fragmente der verstärkten und gereinigten Sonden in den T-easy-Vektor.
- Bestätigen Sie die Nukleotidsequenzen der geklonte DNA-Fragmente durch Sequenzierung27,28.
3. Vorbereitung des Zielens, Construct(s), die Elektroporation von ES-Zellen und die Verstärkung der ES-Klone
- Bereiten Sie jedes targeting Konstrukt mit einem Plasmid Maxiprep Kit nach dem Protokoll des Herstellers. Linearisieren jedes Konstrukt Plasmid von mit ich nicht in einer 400 µL Reaktion einschließlich 40 µL 10-fach Puffer für nicht ich, nicht ich 10 µL, 100 µg DNA, zu verdauen und H2O bis 400 µL bei 37 ° C über Nacht.
- Reinigen Sie die linearisierte targeting Construct(s).
- Extrahieren die verdauten Reaktionslösung 1 X mit ein gleiches Volumen von Phenol: Chloroform: Isoamyl Alkohol (25:24:1) und Zentrifuge bei einer Kraft von 2.000 X g für 10 Minuten.
- Übertragen Sie den Überstand auf eine neue 1,5-mL-Tube, überstürzen Sie sich die DNA mit 2,5 x Ethanol und 0,1 x 3M Natriumacetat (pH 5,2) (Volumen-Verhältnis) und bei einer Kraft von 2.000 X g für 10 min zentrifugieren.
- Entfernen Sie das überstand, waschen die DNA pellet-1 X mit 1 mL 75 % Ethanol, und bei einer Kraft von 2.000 X g für 5 min zentrifugieren.
- Den Überstand zu entfernen und das DNA-Pellet für 5 min an der Luft trocknen.
- Lösen Sie die linearisierte DNA-Pellet in sterilen Tris-EDTA (TE)-Puffer auf eine Endkonzentration von 1 µg/µL.
- Mix 50 µg jedes linearisierten targeting Konstrukt mit 0,5 x 107 ES-Zellen. Führen Sie die Elektroporation bei 320 V und 250 µF.-Platte die elektroporiert ES-Zellen auf Gerichte mit Neo-resistente MEF-Feeder.
- Nach 24 h wechseln Sie zu ES Zelle Medium mit 400 µg/mL G418 und 200 µM Ganciclovir und Kultur für 4 – 5 Tage mit einer täglichen mittleren Änderung weiter. Medikamenten-resistenten ES-Klone in 48-Well Platten abholen.
Hinweis: Normalerweise werden vier 48-Well-Platten pro Konstrukt verwendet. - Duplizieren Sie die 48-Well-Platten.
Hinweis: Ein Satz der Platten ist konserviert, und der andere Satz dient zur genomischen DNA-Vorbereitung.
4. Vorbereitung der genomischen DNA und die Verdauung mit Restriction Enzyme(s)
- Bereiten Sie gDNAs von ES-Zellen mithilfe eines kommerziellen Kits (genomische DNA-Reinigung-Kit) mit geringfügigen Änderungen vor.
- Medien aus der Zellkultur ES entfernen und Hinzufügen von 500 µL Kerne Lyse-Lösung, einschließlich RNaseA, direkt in die Vertiefungen zu die Zellen zu lösen.
Hinweis: Die Zelle lysate kann bei-80 ° C gelagert oder sofort behandelt werden. - Pipettieren nach oben und unten mehrmals, um die Zellen vollständig lysiert und übertragen Sie sie auf eine saubere 1,5 mL-Tube.
- Fügen Sie ein Drittel des Volumens der Proteinlösung Niederschlag, der 1,5 mL Tube, kräftig vortexen 20 s, kühlen die Proben für 5 min auf Eis, und dann, bei einer Kraft von 2.000 X g für 5 min. Transfer der Überstand in ein anderes sauber 1,5 mL Tube Apart Zentrifugieren Ning ein gleiches Volumen von Isopropanol; Mischen Sie vorsichtig die Lösung. (Beachten Sie, dass weiße fadenförmige Stränge in diesem Moment gesehen werden können.) Bei einer Kraft von 2.000 X g für 1 min Zentrifugieren; verwerfen Sie den überstand.
- Waschen der gDNA Pellet mit 1 mL 70 % igem Ethanol bei Raumtemperatur, Zentrifuge bei einer Kraft von 2.000 X g für 1 min, Aspirieren des Überstands vorsichtig und dann an der Luft trocknen gDNA Pellet für 3 min.
- GDNAs mit 100 µL DNA Rehydratationslösung auflösen und dann brüten bei 65 ° C für 1 h oder bei 4 ° C über Nacht.
- Speichern der gDNAs bei 2 – 8 ° C.
- Medien aus der Zellkultur ES entfernen und Hinzufügen von 500 µL Kerne Lyse-Lösung, einschließlich RNaseA, direkt in die Vertiefungen zu die Zellen zu lösen.
- Digest gDNAs mit RE Dra I. eingestellt bis eine 30 µL Verdauung Reaktion predesigned, durch das Mischen von 3 µL 10-fach Puffer für Dra I, 3 µL Dra I, 10 µg gDNAs/Probe und H2O bis zu 30 µL, und über Nacht bei 37 ° C inkubieren.
- Überprüfen Sie die Vollständigkeit der Verdauung durch DNA-Gel, 5 µL der verdauten Reaktion, zu analysieren und fügen Sie dann die 3 µL 10 x DNA Loading Puffer für den nachfolgenden Schritt.
(5) Southern Blotting und PCR-Identifikation
- Southern Blot screening
- Die verdaute gDNAs durch Elektrophorese und Übertragung auf eine Membrane zu trennen.
- Bereiten Sie eine 1 % Agarose-Gelelektrophorese-Gel mit Interkalation Bromid (EB), laden Sie die Proben aus Schritt 4.3 und eine 1 kb-Leiter und laufen Sie das Gel über Nacht mit einer niedrigen Spannung (30 – 40 V).
- Nehmen Sie das Gel und machen Sie ein Foto mit einem DNA-Gel-Imaging-System nach der Elektrophorese. Überprüfen Sie, ob verdaut und getrennten gDNAs ein Abstrich-wie Bild anzeigen.
- Einweichen Sie das Gel in ein Tablett mit 0,2 N HCl-Lösung und schütteln Sie sie vorsichtig für 20 min bei Raumtemperatur.
- Übertragen Sie das Gel auf DNA Denaturierung Lösung und schütteln Sie sie vorsichtig für 20 min bei Raumtemperatur.
- Wechseln Sie das Gel in DNA-neutralisierende Lösung und schütteln Sie sie vorsichtig für 20 min bei Raumtemperatur.
Hinweis: Das Gel ist anfällig für Brüche nach diesem Schritt, so dass es vorsichtig umgegangen werden muss. - Verwenden Sie der schnellen abwärts Transfersystem, um die DNA aus dem Gel auf die Membran übertragen. Montieren der TurboBlotter und befleckende Stack entsprechend den Anweisungen des Herstellers.
Hinweis: 10 X oder 20 X Kochsalzlösung Natriumcitrat (SSC) Lösung dient als Puffer übertragen. 3 h Übertragung reicht in der Regel 95 % der gDNAs von Gel auf Membran übertragen; eine längere Zeit der Übertragung ist jedoch harmlos. - Die Membran herausnehmen und waschen Sie es mit 2 X SSC für 1 min, absorbieren die Flüssigkeit mit Gewebe, und dann die DNA mit der Membran mit einer UV-Vernetzer zu vernetzen.
Hinweis: Die Membran kann bei 4 ° C eine Woche aufbewahrt werden.
- Beschriften Sie die DNA-Sonden mit Radioaktivität.
- Reinigen Sie die Sonde Plasmide mit einem Miniprep Kit nach dem Protokoll des Herstellers.
- Freigabe der DNA Fragmente der Sonden aus dem Plasmid-Vektor von EcoR Verdauung in eine Reaktionslösung einschließlich 5 µL Puffer für EcoR I, 2 µL EcoR ich Enzym, 20 µg Plasmid DNA und H2O bis 50 µL , 2 h.
- Führen Sie eine 1 % DNA-gel für die Trennung von der Sonde DNA-Fragmente aus dem Vektor und reinigen Sie die DNA-Fragmente der Sonden mit den DNA-Gel Extraction Kit nach dem Protokoll des Herstellers zu.
- Messen Sie mit 1 µL DNA-Lösung, DNA-Konzentration der Probe DNA-Fragmente mit einem Spektrophotometer bei einer Wellenlänge von 260/280 nm.
- 40-ng der Sonde DNAs in der 1,5 mL Tube mit 45 µL TE-Puffer vorbereiten, Kochen für 3 min, kurz drehen und legen Sie dann die Tube(n) für 2 min auf Eis.
- Die Röhrchen mit Ready-to-Go DNA Kennzeichnung Perlen der Hitze denaturiert Sonde DNAs hinzufügen (-dCTP), Pipettieren rauf und runter zu mischen, 5 µL [α32P] dCTP hinzufügen und dann bei 37 ° C für 15 min inkubieren.
- Reinigen Sie die beschrifteten Prüfspitzen mit G50 Microcolumns gemäß den Anweisungen des Herstellers zu, und dann messen Sie die Radioaktivität von einem Szintillationszähler (optional).
- Hybridisieren Sie die Membrane(s) mit den markierten Sonden.
- Prehybridize der Membran.
- Prewarm die Hybridisierung Lösung bei 42 ° C für 30 min. Mix 20 mL vorgewärmte Hybridisierung Lösung mit 200 µg von gekochtem Lachs Samenzellen DNA in ein 50-mL-Tube.
- Legen Sie die Membran in der Hybridisierung-Röhre. Die Hybridisierung Röhre die gemischte prehybridization Lösung hinzufügen. Legen Sie sie in den Ofen Hybridisierung (Satz Rollen und die Temperatur bei 42 ° C) und lassen Sie die Prehybridization für 30 min gehen.
- Hybridisieren Sie die Membran mit den beschrifteten Wortanzahl.
- Nehmen Sie die Hybridisierung Röhre und gießen Sie die prehybridization Lösung in ein 50 mL-Tube; Hinzu kommen die denaturierten Sonde (beheizt bei 100 ° C für 3 min) ab Schritt 5.1.2.7 Rohr und Mischung sanft.
Hinweis: Reduzieren Sie induzierenden Bläschen. - Die gemischte Lösung zur Hybridisierung Röhre zurück und führen Sie die Hybridisierung bei 42 ° C über Nacht.
- Nehmen Sie die Hybridisierung Röhre und gießen Sie die prehybridization Lösung in ein 50 mL-Tube; Hinzu kommen die denaturierten Sonde (beheizt bei 100 ° C für 3 min) ab Schritt 5.1.2.7 Rohr und Mischung sanft.
- Prehybridize der Membran.
- Waschen Sie die Membrane(s) um nonhybridized Sonden zu entfernen.
- Legen Sie die Membrane(s) in ein Fach mit 1 x SSC + 0,1 % SDS und sanft bei 55-60 ° C für 10 Minuten schütteln.
- Übertragen Sie der Membrane(s) auf ein Tablett mit 0,5 x SSC + 0,1 % SDS und sanft bei 55-60 ° C für 10 Minuten schütteln.
- Überprüfen Sie die Radioaktivität auf die Membrane(s) mit ein tragbarer Geigerzähler um zu entscheiden, ob eine dritte Reinigung erforderlich ist.
- Setzen Sie die Radioaktivität auf der Membran zu Röntgenfilme.
- Entfernen Sie die Flüssigkeit aus der gewaschenen Membrane(s).
- Entfalten die Membrane(s) mit Plastikfolie und fix It/Sie in der Exposition-Kassette.
- Setzen Sie die Membran, zwei Blätter von Röntgenfilm in einem dunklen Raum.
- Legen Sie die Belichtung Kassette bei-80 ° C, Nacht oder länger.
- Entwickeln Sie die Filme um die Ergebnisse zu visualisieren. Prüfen Sie, ob ein entsprechende ES Klon der gewünschten mit der gezielten Rekombination oder nicht, nach den Größen der DNA-Bands durch die Sonden erkannt ist.
- Rehybridize die gleichen Membran von einer anderen Sonde nach Abstreifen der verwendeten Sonde nach folgendem Verfahren: die verwendeten Membran herausnehmen, waschen Sie es 1 X mit sauber H2O und inkubieren Sie es dann in Ringel-Lösung (55 % Formamid, 2 % SSPE, 1 % SDS, H 2 (O) bei 65 ° C mit sanft schütteln für 1 – 2 h.
- Die verdaute gDNAs durch Elektrophorese und Übertragung auf eine Membrane zu trennen.
-
PCR-Identifikation
- Durchführen Sie PCR-Kennung des gewünschten ES-Klone in einer 50-µL-Reaktion-Lösung einschließlich 5 µL 10 X PCR Puffer, 2 µL 50 mM MgSO4, 1 µL 10 mM dNTP, 1 µL vorwärts Grundierung 20 µM, 20 µM-rückwärts-Primer 1 µL , 1 µL der High-Fidelity-Platin Taq, gDNAs (~ 100 ng), und H2O bis 50 µL.
- Verwenden Sie die folgenden Reaktionsbedingungen PCR: eine erste Denaturierung bei 94° C für 3 min 30 Zyklen der Denaturierung bei 94° C für 30 s, Glühen bei 60° C für 30 s und eine Erweiterung bei 68° C 132 s und einer dritten Stufe 68° c für 10 min.
- Analysieren Sie die PCR-Produkte durch Elektrophorese in 1,0 % Agarose.
- Klonen der PCR-Fragmente mit ihrer erwarteten Größe in den T-easy Vektor und Sequenz, die das Vorhandensein einer Teilsequenz des Vektors Ziel zu bestätigen.
Ergebnisse
In diesem Papier wird ein detailliertes Protokoll der Southern blotting und PCR beschrieben, die genutzt wird, HR Ereignisse zu identifizieren, die in ES-Zellen für die Erzeugung von NM II genetischen Austausch Mausmodelle, verwenden ES Zellen-basierten HR-vermittelten targeting-Technologie aufgetreten. Obwohl Southern blotting PCR, sowie traditionelle Gene-targeting-Technologie seit mehreren Jahrzehnten verbreitet haben, muss die erfolgreiche Anwendung von ihnen sorgfältig geplant werden. Mindestens die folgenden Aspekte müssen berücksichtigt werden: die Länge der lange und kurze Arme, die Positionen und die Länge der Sonden, geeignet zum Schneiden von genomischer DNA und die Primer für PCR, wie in Abbildung 1zusammengefasst ist hilfreich für REs nachträgliche Analyse. Als ein wichtiger Schritt der Southern blotting, vorbereitet und verdauten genomischen DNA sind erforderlich, um getrennt werden auf DNA-Gel für die Erkennung von der Sonde. Weil genomische DNA in eine Menge von Fragmenten mit unterschiedlichen Längen geschnitten sind, zeigen sie einen Abstrich-Status wie auf dem DNA-Gel, eine komplette Verdauung der genomischen DNA, was darauf hindeutet, wie in Abbildung 2gezeigt. Als letzter Schritt der Southern blotting sind die Signale von Radioaktivität-markierten Sonden Hybridisierung mit einer Ziel-DNA-Fragment auf dem Film gezeigt die widerspiegeln, des Auftretens von HR-Veranstaltungen in die ES-Klone, damit angibt, ob ein ES-Klon der gewünschte ist. Laut der Vorbestimmung in dieser Studie haben ES-Klone mit mutierten Allel zwei unterschiedliche Größe Bands während Wildtyp ES-Klone nur ein Band haben, was darauf hindeutet, die gewünschte ES Klone sind heterozygot (Abbildung 3). Bezogen auf das Verfahren und die Ergebnisse der Southern blotting, den Betrieb und die Ergebnisse der PCR sind einfach und direkt. Nach der PCR-Reaktion, die PCR-Produkte analysiert werden können auf dem DNA-Gel. Wenn die PCR-Bands spezifisch sind und Sequenzierung der geklonten PCR-Produkte bestätigt die Anwesenheit von einer Teilsequenz Ziel Vektor wie ein Neo-Resistenz-Gen sowie genomische Regionen, die nur außerhalb der Homologie-Arm sind, kann das Auftreten von HR-Veranstaltungen erwartet und überprüft (Abbildung 4).
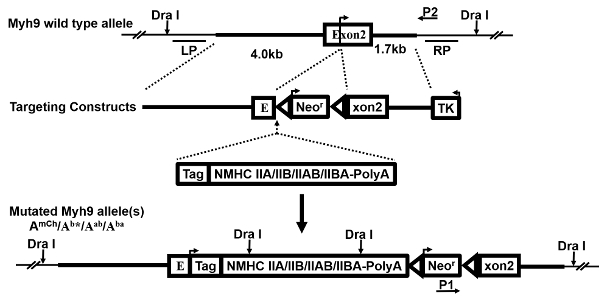
Abbildung 1 : Targeting Konstrukte. Dies ist eine schematische Darstellung zeigt die Erzeugung von mehreren Zielen Konstrukte. Wildtyp (WT) Myh9 gen Allel, gen-targeting Vektor, Ersatz exogene Ausdruck Kassette(n), sowie die daraus resultierenden mutierten Allele(s) und Sonden (LP, RP) für südlicher Fleck und die Primer (P1, P2) für PCR, werden gezeigt und beschrieben 27. ein Pfeil auf Exon 2 zeigt die translationale Einleitung Seite. Im Anschluss an das erfolgreiche Auftreten von HR, Ersatz-Expressionskassette und Neomycin-Resistenz-Gen (NeoR) eingefügt sind nur 5 Minuten von der initiierenden ATG-Codon. Daher das endogene Myh9 Allel ist gestört und klopfte in Wirtsgenom ist/sind in der mutierten Zellen und Mäuse zum Ausdruck gebracht. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 2 : Verdaut genomische DNA mit Dra I. Genomische DNA aus ES-Klone, die gezielt mit dem Konstrukt ersetzt NMHC II-A mit II-B sind mit Dra ich verdaut und dann auf einem Agarosegel durch Elektrophorese getrennt. Ein Abstrich-ähnliche verdaute gDNA wird beobachtet. C1 - C8 zeigen einzelne ES-Klone. Eine vollständige Verdauung des gDNA produziert eine Menge von DNA-Fragmenten mit eine andere Länge, damit eine Abstrich-wie Bild angezeigt wird. Dieses Ergebnis spiegelt auch die gute Qualität des vorbereiteten gDNAs und die Vollständigkeit der Verdauung. Bitte klicken Sie hier für eine größere Version dieser Figur.

Abbildung 3 : Repräsentative Ergebnisse der Southern blotting. Diese Tafeln zeigen eine Southern Blot Screening der genomische DNA aus ES-Klone, die gezielt mit dem Konstrukt des Ersetzens NMHC II-A mit II-AB, mit der linken und rechten Sonden. Das mutierte Allel zeigt eine 12,1 kb oder 6 kb Band, wenn die linke Sonde oder richtige Sonde bzw., verwendet wird während die WT ein 9,7 kb Band zeigt. M: Marker; PC1-PLZ5: positive Klone; NC: negative Klon. Die Größen der Southern blotting Bands werden ebenfalls angezeigt. Alle Verfahren der Southern blotting sind streng durchgeführt und die Spezifität der Sonden ist gut genug; Es darf keine unspezifische, die Bänder für die erwarteten Bands erwarten. Bitte klicken Sie hier für eine größere Version dieser Figur.
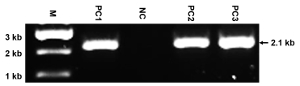
Abbildung 4 : Repräsentative Ergebnisse der PCR. Dieses Fenster zeigt die PCR Identifikation des genomische DNA aus ES-Klone, die gezielt mit dem Konstrukt des Ersetzens NMHC II-A mit II-BA mit dem Primerpaar P1 + P2. Das mutierte Allel ergibt eine 2,1 kb-Band, während das WT-Allel ergibt sich kein Band. M: Marker; PC1-PC3: positive Klone; NC: negative Klon. Die Größe der PCR-Band wird ebenfalls angezeigt. Da die Primer entworfen sind, um nur das mutierte Allel zu erkennen, spiegelt das Aussehen einer Einzel- und erwartete Band die Spezifität der Primer und die hohe Qualität der zubereiteten gDNAs. Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Derzeit können Designer Nukleasen für Genom-Bearbeitung noch nicht ES zellbasierte Gene-targeting Technologie aufgrund ihrer Probleme von Ziel-Effekten und Schwierigkeiten beim Einfügen einer langen DNA-Fragment30,31ersetzen. Dieser Bericht enthält als die goldenen Methoden zur Identifizierung von HR Ereignisse in Maus-ES-Zellen, ein detailliertes Protokoll der Southern blotting und PCR für das Feld. Wir validiert die Zuverlässigkeit dieser Methoden durch die Analyse von einzelnen Klone von ES-Zellen mit einer Reihe von Konstrukten ausgerichtet. Die gewünschte ES-Klone, die durch diese Methoden identifiziert hatte erfolgreich eingesetzt, um entsprechende Maus Modelle27zu generieren.
Obwohl andere Techniken für das Screening von gezielten ES-Klone beschrieben19,32, die Methoden der Southern blotting wurden und PCR nicht vollständig durch ersetzt werden hergestellt, die danach32, weil diese erste Techniken haben eine mehr angewandte Geschichte und werden fast überall akzeptiert und bestätigt durch die wissenschaftliche Gesellschaft, durch die meisten biologischen Labors durchgeführt und sind der Ursprung von anderen Technologien. Wichtig ist, ist die gute Performance der Southern blotting und PCR bei der Identifizierung von HR-Veranstaltungen auch in früheren Arbeiten29beispielhaft dargestellt. Die Ergebnisse von Southern blotting zeigen einige einzigartige Eigenschaften: die nach dem Zufallsprinzip überwachten ES-Klone, mehr als 90 % von ihnen sind gewünschte, keine unspezifische Bands werden erkannt und die HR ereignete sich bevorzugt am ein Allel des Gens Myh9. Inzwischen bestätigen die Daten von PCR, zusammen mit der Sequenzierung, dass das Auftreten von HR-Veranstaltungen ist standortspezifisch und gut mit jenen von Southern blotting überein.
Nach unserer Praxis sollten mehrere Faktoren bei Southern blotting und PCR zur Identifizierung werden HR-Veranstaltungen in ES-Zellen verwendet, gute und erwarteten Ergebnisse erhalten. Die erste ist die Länge der Homologie Arme; im Allgemeinen wird die Erhöhung der Homologie Armlänge die Effizienz der HR33verbessern. Dies ist jedoch nicht immer der Fall. Auf der einen Seite erhöhen längere Arme die Schwierigkeit der Manipulation; auf der anderen Seite führte die Länge der Arme Homologie (4 kb für den linken Arm und 1,7 kb für den rechten Arm) berichtet hier die höchste HR-Frequenz, die bisher erzielten unter ähnliche Experimente. Darüber hinaus erleichtert eine angemessene Länge der Homologie Arme die Identifizierung mittels PCR. Die zweite ist die Nutzung der isogenen DNA zur Vorbereitung der Homologie Arme und Southern Blot Sonden34. Dies kann durch die Bestellung eines BAC-Klons mit der Region des Gens von Interesse oder mithilfe von genomischer DNA aus den Zellen ausgerichtet sein sollen zufrieden sein. Die dritte ist die Auswahl der geeigneten REs für genomische DNA zu verdauen. In der Regel werden ein RE oder die Kombination von zwei REs, die das Wildtyp oder mutierte Allel nur ein-oder zweimal um das targeting Region geschnitten bevorzugt; Darüber hinaus das daraus resultierende größere DNA-Fragment 15 kb nicht überschreiten und der Größenunterschied zwischen den unterschiedlichen DNA-Fragmente ist mehr als 2 kb. Diese Anforderungen können die Trennung und Identifizierung von erwarteten Bands durch Southern blotting erleichtern. Die vierte ist die Länge der Sonden und die geringste Ähnlichkeit mit anderen Sequenzen im Genom. Im Allgemeinen ist die Länge der Sonden 500 – 1.000 bp. Die Ähnlichkeit mit anderen Sequenzen im Genom kann mit dem BLAST NCBI-Programm analysiert werden. Darüber hinaus beschrieb eine Software zur Gestaltung der Sonden für Southern blotting wurde35. Die fünfte Faktor zu berücksichtigen ist die herkömmlichen Methoden verwenden, um eine Verbesserung Ertrag genomischen DNA Vorbereitung. Genomische DNA aus einem konfluierende Brunnen eine 48-Well-Platte vorbereitet sind in der Regel genug für mindestens zwei Runden der Southern Blot Analysen. Hinsichtlich der Gestaltung der Zündkapseln für PCR, ist die beste Strategie vorhanden eine Grundierung auf die Auswahlmarkierung in Verbindung mit einer Grundierung außerhalb der targeting Arme verwenden. Darüber hinaus ist es wichtig für den Nachweis der HR Veranstaltungen20,36, Sequenzierung der PCR-Produkte. Insbesondere kann PCR-basierten Screening nicht vollständig ersetzen die Informationen über Southern blotting, es kann effektiv reduzieren die Anzahl der Klone ausgewertet werden.
Im Ergebnis sind Southern blotting und PCR gut nachgewiesene Methoden für das screening von ES-Klone um HR-mediated Gene-targeting Veranstaltungen in ES-Zellen zu identifizieren. Obwohl das ausführliche Protokoll beschrieben hier vor allem auf die Vorführung des gewünschten NM II genetischen Austausch ES Klone konzentriert, kann es für die Genotypisierung Mäuse verwendet werden, die anschließend generiert werden über die positiven ES-Klone. Es kann leicht zur Identifizierung der HR-Veranstaltungen in andere Zelltypen wie iPS-Zellen oder somatischen Zellen angepasst werden.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit erhielt Unterstützung durch das allgemeine Programm der National Natural Science Foundation of China (Grants Nr. 31571432), die menschlichen Provincial Natural Science Foundation of China (Grant No. 2015JC3097) und der Forschung Grundlage der Bildung Bureau Hunan Provinz, China (Grant Nr. 15 K 054).
Materialien
| Name | Company | Catalog Number | Comments |
| BAC CLONE | BACPAC Resources Center (BPRC) | bMQ-330E21 | |
| QIAGEN Large-Construct Kit | QIAGEN | 12462 | |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| QIAprep Spin Miniprep Kit | QIAGEN | 27104 | |
| QIAGEN Plasmid Plus Maxi Kit | QIAGEN | 12963 | |
| PfuUltra High-Fidelity DNA Polymerase | Agilent | 600382 | |
| T-easy vector | Promega | A1360 | |
| Nuclei Lysis Solution | Promega | A7941 | |
| Protein Precipitation Solution | Promega | A7951 | |
| DNA Denaturing Solution | VWR | 351-013-131 | |
| DNA Neutralizing Solution | VWR | 351-014-131 | |
| Ready-To-Go DNA Labeling Beads (-dCTP) | VWR | 27-9240-01 | |
| UltraPure SSC, 20x | Thermo Fisher | 15557036 | |
| UltraPur Phenol:Chloroform:Isoamyl Alcohol (25:24:1, v/v) | Thermo Fisher | 15593031 | |
| G418 | Thermo Fisher | 10131035 | |
| Salmon Sperm DNA Solution | Thermo Fisher | 15632011 | |
| Platinu Taq DNA Polymerase High Fidelity | Thermo Fisher | 11304029 | |
| Not I | Thermo Scientific | ER0592 | |
| Dra I | Thermo Scientific | ER0221 | |
| EcoR I | Thermo Scientific | ER0271 | |
| Ganciclovir | Sigma | G2536 | |
| Whatman TurboBlotter Transfer System, Large Kits | Fisher Scientific | 09-301-188 | |
| [α32P]dCTP | PerkinElmer | NEG013H100UC | |
| ProbeQuan G-50 Micro Columns | GE Healthcare | 28-9034-08 | |
| Hybrisol I Hybridization Solution | Millipore | S4040 | |
| Kodak X-Ray Film | Z&Z Medical | 844 5702 |
Referenzen
- Gao, G., McMahon, C., Chen, J., Rong, Y. A powerful method combining homologous recombination and site-specific recombination for targeted mutagenesis in Drosophila. Proceedings of the National Academy of Sciences of the United States of America. 105 (37), 13999-14004 (2008).
- Skarnes, W., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474 (7351), 337-342 (2011).
- Vogel, G. Nobel Prizes. A knockout award in medicine. Science. 318 (5848), 178-179 (2007).
- Salsman, J., Dellaire, G. Precision genome editing in the CRISPR era. Biochemistry. Cell Biology. 95 (2), 187-201 (2017).
- Capecchi, M. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nature Reviews Genetics. 6 (6), 507-512 (2005).
- Van, d. W. L., Adams, D. J., Bradley, A. Tools for targeted manipulation of the mouse genome. Physiological Genomics. 11 (3), 133-164 (2002).
- Glaser, S., Anastassiadis, K., Stewart, A. F. Current issues in mouse genome engineering. Nature Genetics. 37 (11), 1187 (2005).
- Bradley, A., Evans, M., Kaufman, M. H., Robertson, E. Formation of germ-line chimaeras from embryo-derived teratocarcinoma cell lines. Nature. 309 (5965), 255-256 (1984).
- Robertson, E., Bradley, A., Kuehn, M., Evans, M. Germ-line transmission of genes introduced into cultured pluripotential cells by retroviral vector. Nature. 323 (6087), 445-448 (1986).
- Thomas, K. R., Capecchi, M. R. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells. Cell. 51 (3), 503-512 (1987).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474 (7351), 337 (2011).
- Collins, F. S., Rossant, J., Wurst, W. A mouse for all reasons. Cell. 128 (1), 9-13 (2007).
- Poueymirou, W. T., et al. F0 generation mice fully derived from gene-targeted embryonic stem cells allowing immediate phenotypic analyses. Nature Biotechnology. 25 (1), 91-99 (2007).
- Pettitt, S. J., et al. Agouti C57BL/6N embryonic stem cells for mouse genetic resources. Nature Methods. 6 (7), 493-495 (2009).
- Gertsenstein, M., et al. Efficient Generation of Germ Line Transmitting Chimeras from C57BL/6N ES Cells by Aggregation with Outbred Host Embryos. PLoS One. 5 (6), 11260 (2012).
- Smithies, O., Gregg, R. G., Boggs, S. S., Koralewski, M. A., Kucherlapati, R. S. Insertion of DNA sequences into the human chromosomal |[beta]|-globin locus by homologous recombination. Nature. 317 (6034), 230-234 (1985).
- Deng, C., Capecchi, M. R. Reexamination of gene targeting frequency as a function of the extent of homology between the targeting vector and the target locus. Molecular & Cellular Biology. 12 (8), 3365 (1992).
- Lay, J. M., Friishansen, L., Gillespie, P. J., Samuelson, L. C. Rapid confirmation of gene targeting in embryonic stem cells using two long-range PCR techniques. Transgenic Research. 7 (2), 135-140 (1998).
- Langerak, P., Nygren, A. O. H., Schouten, J. P., Jacobs, H. Rapid and quantitative detection of homologous and non-homologous recombination events using three oligonucleotide MLPA. Nucleic Acids Research. 33 (22), 188 (2005).
- Gómezrodríguez, J., et al. Advantages of q-PCR as a method of screening for gene targeting in mammalian cells using conventional and whole BAC-based constructs. Nucleic Acids Research. 36 (18), 117 (2008).
- Kim, H. S., Smithies, O. Recombinant fragment assay for gene targeting based on the polymerase chain reaction. Nucleic Acids Research. 16 (18), 8887-8903 (1988).
- Joyner, A. L., Skarnes, W. C., Rossant, J. Production of a mutation in mouse En-2 gene by homologous recombination in embryonic stem cells. Nature. 338 (6211), 153-156 (1989).
- Ma, X., Adelstein, R. S. The role of vertebrate nonmuscle Myosin II in development and human disease. Bioarchitecture. 4 (3), 88-102 (2014).
- Malonek, D. Relationships between the dynamics of cortical blood flow, oxygenation, and volume changes following sensory stimulation. Proceedings of the National Academy of Sciences of the United States of America. 94, (1997).
- Takeda, K., Kishi, H., Ma, X., Yu, Z. X., Adelstein, R. S. Ablation and mutation of nonmuscle myosin heavy chain II-B results in a defect in cardiac myocyte cytokinesis. Circulation Research. 93 (4), 330-337 (2003).
- Conti, M. A., Evenram, S., Liu, C., Yamada, K. M., Adelstein, R. S. Defects in cell adhesion and the visceral endoderm following ablation of nonmuscle myosin heavy chain II-A in mice. Journal of Biological Chemistry. 279 (40), 41263-41266 (2004).
- Wang, A., et al. Nonmuscle myosin II isoform and domain specificity during early mouse development. Proceedings of the National Academy of Sciences of the United States of America. 107 (33), 14645-14650 (2010).
- Zhang, Y., et al. Mouse models of MYH9-related disease: mutations in nonmuscle myosin II-A. Blood. 119 (1), 238 (2012).
- Liu, T., et al. Identification and characterization of MYH9 locus for high efficient gene knock-in and stable expression in mouse embryonic stem cells. PLoS One. 13 (2), 0192641 (2018).
- Saito, S., Adachi, N. Advances in the Development of Gene-Targeting Vectors to Increase the Efficiency of Genetic Modification. Biological & Pharmaceutical Bulletin. 39 (1), 25-32 (2016).
- Langerak, P., Nygren, A., Schouten, J., Jacobs, H. Rapid and quantitative detection of homologous and non-homologous recombination events using three oligonucleotide MLPA. Nucleic Acids Research. 33 (22), 188 (2005).
- Martin, S. L., et al. A single amino acid substitution in ORF1 dramatically decreases L1 retrotransposition and provides insight into nucleic acid chaperone activity. Nucleic Acids Research. 36 (18), 5845-5854 (2008).
- Kamisugi, Y., Cuming, A. C., Cove, D. J. Parameters determining the efficiency of gene targeting in the moss Physcomitrella patens. Nucleic Acids Research. 33 (19), 173 (2005).
- Luo, Y., Bolund, L., Sørensen, C. B. Pig gene knockout by rAAV-mediated homologous recombination: comparison of BRCA1 gene knockout efficiency in Yucatan and Göttingen fibroblasts with slightly different target sequences. Transgenic Research. 21 (3), 671-676 (2012).
- Croning, M. D., Fricker, D. G., Komiyama, N. H., Grant, S. G. Automated design of genomic Southern blot probes. BMC Genomics. 11 (1), 74 (2010).
- Zimmer, A., Gruss, P. Production of chimaeric mice containing embryonic stem (ES) cells carrying a homoeobox Hox 1.1 allele mutated by homologous recombination. Nature. 338 (6211), 150-153 (1989).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten