Method Article
마우스 배아 줄기 세포 사용 하 여 남부 럽 연쇄 반응에서에서 동종 재결합 이벤트의 식별
* 이 저자들은 동등하게 기여했습니다
요약
여기, 우리 남부 럽 그리고/또한 PCR를 사용 하 여 마우스 배아 줄기 세포에서 발생 하는 동종 재결합 이벤트를 식별 하는 데 자세한 프로토콜 제시. 이 메서드는 nonmuscle myosin II 유전자 대체 마우스 모델 기술을 사용 하 여 전통적인 배아 줄기 세포 기반 동종 재결합 중재 대상의 세대에 의해 exemplified입니다.
초록
오프 대상 효과의 문제 및 디자이너 nucleases 게놈 편집, 배아 줄기에 대 한 응용 프로그램에서 긴 DNA 파편을 삽입의 어려움에 상대 (ES) 세포 기반 유전자를 대상으로 이러한 결점 없는 기술과 널리 큰 삭제/삽입에서 단일 염기 치환에 이르기까지 동물/마우스 게놈을 수정 하는 데 사용. 특히, 상대적으로 적은 동종 재결합 (HR) 이벤트를 얻기 위해 필요한 식별 원하는 ES 클론은 정확 하 고 안정적인 방법 요구 중요 한 단계. 남부 럽 또는 기존의 PCR 수시로이 목적을 위해 활용 됩니다. 여기, 우리는 그 두 가지 방법을 사용 하 여 마우스 ES 세포는 생 Myh9 유전자는 중단 하 고 다른 nonmuscle myosin 무 겁 사슬 IIs (NMHC IIs) 인코딩 cDNAs에 의해 대체 하기 위한 것에서 발생 하는 시간 이벤트를 식별 하는 자세한 절차를 설명 합니다. 남부 럽의 모든 절차는 vector(s), electroporation, 약물 선택, 확장 및 ES 세포/클론, 준비, 소화, 저장을 지정 하 고 게놈 DNA (gDNA)는 교 잡의 럽의 건설을 포함 하 고 probe(s), 및 x-선 필름에 autoradiography에의 마지막 단계 세척. PCR는 준비 하 고 희석 gDNA를 직접 수행할 수 있습니다. 이상적인 결과 얻기 위해 프로브 및 (재) 남부 럽에 대 한 절단 사이트 및 PCR 위한 뇌관 제한 효소 수 신중 하 게 계획 합니다. 비록 남부 럽의 실행 시간과 노동 집약적 이며 PCR 결과 판정, 남부 럽 및 PCR에 의해 신속한 검사 하 여 올바른 식별 설명 하는 이러한 방법을 단독 또는 결합 된 응용 프로그램 허용 널리 사용 하 고 대부분의 실험실 genotypes ES 세포 및 유전자의 식별에 의해 상담이 종이에서 동물 수정.
서문
기술 murine ES 세포에서 시간에 의해 대상으로 하는 유전자의 특정 유전자 변이1,2의 셀룰러 결과 해 부를 위한 강력한 도구를 제공 합니다. 중요성 및이 기술의 중요성 2007 노벨상 생리학 또는 약3,4;에 의해 그것의 인식에 반영 됩니다. 한편, 그것은 유전자 공학5의 현대 시대의 출현을 나타냅니다. 진 HR을 통해 대상 포인트 돌연변이에서 ES 세포6,7마우스의 게놈에 있는 큰 염색체 재배열에 이르기까지 거의 모든 변화를 활용할 수 있습니다. 그것은 잘 알려진 소위 게놈 편집 도구의 출현 하기 전에, 유전자 녹아웃 마우스의 세대 ES 세포8,9,10에 유전자를 대상으로 한 기술의 응용 프로그램 필요. 지난 2 년 동안 5000 개 이상의 유전자 타겟팅 마우스 모델링 인간의 질병 이나 유전자 기능11공부에 대 한이 접근 방식에 의해 생산 되었다. 유전자를 대상으로 벡터, 타겟된 ES 세포 클론, 및 사회 과학2,12,,1314 라이브 마우스를 배포 하기 위한 게놈 넓은 녹아웃 노력 설립 되었습니다. , 15. 의심할 여 지 없이, 셀 기반 HR 중재 유전자를 대상으로 기술에 크게 생리 적 또는 병 적인 맥락에서 유전자의 기능에 대 한 우리의 이해 고급.
HR은 포유류에는 상대적으로 드문 이벤트16,17, 중요 한 세포 및 다음 다음 단계 때문에 수많은 ES 식민지에서 발생 하는 돌연변이와 몇 클론을 식별을 위해 분석 하는 murine ES 세포에 유전자를 대상으로 대상 벡터18로 시간입니다. 시간 이벤트를 식별 하는 데 금 방법 등 남부 럽 PCR19,20. 접근의 이점 등는 남부 럽 올바르게 타겟된 ES 클론을 식별할 수 있는 유전자 타겟 이벤트 구문, 단일 복사본 삽입의 검증 등의 구조를 분석 하는 연구를 허용 하면서는 PCR 기반 전략 HR 이벤트21,22에 대 한 더 빠른 심사를 허용합니다. 하지만 이러한 방법은 단점, 같은 그들은 소모와 잘못 된 반응, 그들의 조합 사용은 널리 허용 하 고 가질 수 시간 이벤트 식별에 대부분의 실험실에 의해 적용, ES 세포에서 특정 유전자 생성에 대 한 수정 동물입니다.
포유류에서 각각의 3 개의 다른 유전자 (명명 된 Myh9, Myh10, 및 Myh14) 및 경 쇄의 두 쌍으로 인코딩됩니다 두 동일한 NMHC IIs 구성 된 nonmuscle myosin II (NM II)의 3 개의 isoforms II-A, ⅱ B, 및 II C23을 이라고 합니다. 이전 연구는 적어도 표시는 배아 치24,,2526이 isoforms의 비보에 절제 하기 때문에 II A 및 II B의 isoforms 마우스 개발에 대 한 필수적입니다. 이 문제를 회피 하 고 마우스 개발, 유전자 보충의 나중 단계에서 II A 및 II B의 isoform 특정 기능에 대 한 새로운 통찰력을 얻을 셀 기반 HR 중재 하는 유전자를 대상으로 기술 ES를 사용 하 여 전략을 채택 마우스 모델27의 일련을 생성 합니다. 원하는 ES 클론 식별, 동안 남부 럽 및 PCR 방법을 이용 하였다, 그리고 이러한 입증 효율적이 고 신뢰할 수 있는27,28.
이 종이 남부 럽 고 PCR, vector(s), probe(s), 및 뇌관, 및 실험의 실행 뿐만 아니라 시간 이벤트 식별 exemplified 결과의 분석 대상의 디자인을 포함 하 여에 대 한 자세한 설명을 제공 하고자 모델 및 대표 데이터 유전 교체 NM II 마우스를 만들기 위한 ES 세포에 발생. 여기에 제시 된 이러한 두 메서드의 프로토콜 식별 유전자 변형된 세포 또는 동물의 genotypes 또한 채택 수 있습니다.
프로토콜
1. 설계 대상으로의 Construct(s), 남쪽 오 점, 조사 및 PCR 위한 뇌관
- 녹아웃/노크-에서 보고 여기의 응용 프로그램에서 중단 또는 삽입에 대 한 Myh9 유전자의 첫 번째 코딩 exon (exon 2)를 선택 합니다.
- 5 kb 업스트림 및 5 kb 다운스트림 DNA 시퀀스 Myh9 exon 2 genome.ucsc.edu 웹사이트에서 주변을 검색 합니다.
- 남부 럽에 대 한 게놈 DNA를 소화 하기 위해 적당 한 RE(s)를 pDRAW 소프트웨어를 사용 하 여 제한 효소 (입술)이 10 kb 지역에서 1-2 컷으로의 소화 패턴 분석.
이 요구 사항을 충족 하 고 목적을 위해 선택에 Dra를 참고: - 즉각적인 4 조각을 선택 왼쪽된 상 동 팔 (LHA) 2와 1.7 kb 조각 즉각적인 다운스트림 시퀀스 오른쪽 상 동 팔 (RHA); Myh9 exon의 업스트림 1kb 단편 5' 상류 남부 럽 및 1.2 kb 조각 3' 하류 오른쪽 프로브 (RP)으로 나의 왼쪽된 프로브 (LP)으로 LHA의 위의 분석에 기반을 선택 합니다.
- Primer3 프로그램을 사용 하 여 PCR에 의해 그 4 개의 DNA 파편을 증폭에 대 한 정방향 및 역방향 뇌관 디자인. 앞으로 뇌관 선택 마커 neomyocin 저항 유전자 (P1)의 3' 터미널 근처 거주와 나 (P2) 바로 외곽에 위치한 역 뇌관 뇌관 쌍을 디자인 합니다.
참고:이 뇌관 쌍 사용 됩니다 식별 대상된 ES 클론 PCR29에 의해. - 마우스 Myh9 유전자 소재 시 bacpac.chori.org 웹사이트를 방문 하 여 취재 129Sv BAC 클론 찾기 (참고: isogenic DNA는 선호). BAC DNA 정화는 제조업체에서 제공 하는 지침에 따라 DNA의 큰 조각에 대 한 적합 한 키트를 사용 하 여 격리 합니다.
참고: 순화 된 BAC DNA 사용 됩니다 템플릿으로 PCR 확대를 위한. - 대상 구문, 프로브, 및이 정보를 요약 하는 뇌관의 도식 적인 표현을 그립니다.
2. 세대를 대상으로의 Construct(s) 및 남쪽 오 점, 그리고 PCR 위한 뇌관의 준비에 대 한 조사
- 위에서 설명한 PCR 뇌관을 주문 하 고 20 µ M의 농도에 그들을 분해.
- 상 동 팔 1 µ L 앞으로 1 µ L 역 뇌관, BAC DNA의 1 µ L를 포함 하 여 반응 솔루션에서 프로브 PCR에 의해 증폭 (50 기) 템플릿, Pfu에 대 한 버퍼의 5 µ L, Pfu DNA 중 합 효소로 울트라 1 µ L H2O 최대 50 µ L. PCR 기계는 다음과 같은 조건에서 PCR을 수행: 95 ° C 3 분;에 대 한 95 ° C 30에 대 한 s; 60 ° C 30에 대 한 s; 72 ° C와 1 킬로바이트/분; 30 주기; 마지막으로, 72 ° C 10 분입니다.
- 제조업체에서 제공 하는 프로토콜에 따라 PCR 대 청소 키트를 사용 하 여 PCR 제품을 정화 하 고 elute H2o. 다이제스트의 40 µ L로 DNA 파편 반응 솔루션 5 µ L 10 x 버퍼의 해상도 대 한 포함 된 미리 디자인 된 해상도 함께 정제 PCR 제품 2.5 µ L RE1 및 RE2, 그리고 실행 대상 DNA 밴드를 분리, 소비 세 대상 UV 빛에서 DNA 포함 하 젤 1 %DNA 젤 2 h. 위해 37 ° C에서 eluted DNA의 2.5 µ L의 정화는 DNA 젤 추출 키트 따라 사용 하 여 젤에서 대상 DNA 파편 프로토콜에는 제조업체에서 제공 하 고 H2o.의 40 µ L로 DNA 파편을 elute
- 최종 대상으로 얻으려면는 증폭 하 고 정화 나, LHA, 및 대체 식 cassette(s) 다른 벡터에서 발표의 순서에 따라 mpNTKV LoxP 벡터에 상 동 팔, 대체 식 cassette(s) 복제 construct(s). T-간편한 벡터에 DNA 파편 증폭 및 순화 된 프로브의 클론.
- 시퀀싱27,28여 모든 복제 된 DNA 파편의 뉴클레오티드 시퀀스를 확인 합니다.
3. Construct(s), ES 세포, Electroporation 그리고 ES 클론의 증폭을 대상으로의 준비
- 제조업체에서 제공 하는 프로토콜에 따라 플라스 미드 maxiprep 키트를 사용 하 여 각 대상 건설을 준비 합니다. 내가 하지 내가, 내가의 10 µ L, 100 µ g의 DNA, 10 x 버퍼의 40 µ L을 포함 하 여 400 µ L 반응에서와 그것을 소화 하 여 각 구문 플라스 미드를 선형화 및 H2O 37 ° C에 400 µ L까지 하룻밤.
- 선형화 대상 construct(s) 정화.
- 추출 소화 반응 솔루션 1 x 페 놀: 클로 프롬: Isoamyl 알콜 (25:24:1)와 10 분 2000 x g 의 힘에 원심 분리기 같은 볼륨.
- 새로운 1.5 mL 튜브에는 상쾌한 전송, 에탄올과 0.1 x 3 M 나트륨 아세테이트 (pH 5.2) x 2.5 (볼륨 비율)를 사용 하 여 DNA를 침전 하 고 10 분 2000 x g 의 힘에 원심.
- 표면에 뜨는, 세척 DNA 1 x 75% 에탄올의 1 mL와 함께 작은 고 2000 x g 5 분의 힘에 원심 분리 합니다.
- 상쾌한을 제거 하 고 5 분 동안 DNA 펠 릿 건조.
- 1 µ g / µ L의 최종 농도에서 살 균 트리 스-EDTA (테) 버퍼에서 선형화 DNA 펠 릿을 디졸브.
- 0.5 x 10 각 선형화 대상 구조물의 50 µ g7 ES 세포를 혼합. 320 V에서 electroporation와 250 µ F. 네오 저항 MEF 지류와 함께 요리에 electroporated ES 세포 접시.
- 24 시간 후 400 µ g/mL G418와 200 µ M ganciclovir ES 세포 매체 전환 하 고 4-5 일 매일 매체 변화에 대 한 문화를 계속 합니다. 48-잘 접시에 약물 내성 ES 클론을 선택 합니다.
참고: 일반적으로, 4 개의 48-잘 접시 구조 당 사용 됩니다. - 48-잘 접시를 복제.
참고: 격판덮개의 1 세트는 cryopreserved, 그리고 다른 집합 게놈 DNA 준비를 위해 사용 됩니다.
4. 게놈 DNAs 및 금지와 소화의 준비
- 사소한 수정 상업 키트 (genomic DNA 정제 키트)를 사용 하 여 ES 세포에서 gDNAs을 준비 합니다.
- ES 세포 배양에서 미디어를 제거 하 고 세포를 lyse를 웰 스에 직접 RNaseA를 포함 한 핵 세포 솔루션의 500 µ L를 추가 합니다.
참고: 세포 lysate-80 ° C에 저장 하거나 즉시 치료 될 수 있습니다. - 피펫으로 완전 세포를 lyse 깨끗 한 1.5 mL 튜브에 그들을 전송 하는 여러 번 왔다 갔다.
- 1.5 mL 튜브에 단백질 강 수 솔루션, 20에 대 한 적극적으로 소용돌이의 볼륨의 1 / 3 추가 s, 5 분 동안 얼음에 샘플을 진정 하 고 5 분 다른 깨끗 한 1.5 mL 튜브 contai에는 상쾌한 전송에 대 한 2000 x g 의 힘에 그 후, 원심 닝은 동등한 양의 소 프로 파 놀; 부드럽게 혼합 솔루션. (스레드-가닥 처럼 화이트는이 순간에 볼 수 있습니다.) 1 분 2000 x g 의 힘에 원심 그런 다음에 상쾌한을 삭제 합니다.
- 실내 온도, 1 분에 2000 x g 의 힘에 원심 분리기에서 gDNA 펠 릿의 70% 에탄올 1 mL와 함께 세척 하 고 발음은 상쾌한 신중 하 게, 다음, 건조 3 분 gDNA 펠 릿.
- DNA rehydration 솔루션의 100 µ L 함께 gDNAs를 녹이 고, 다음, 품 어 또는 4 ° C에서 65 ° C 1 h에서 하룻밤.
- 2-8 ° c.에 gDNAs 저장
- ES 세포 배양에서 미디어를 제거 하 고 세포를 lyse를 웰 스에 직접 RNaseA를 포함 한 핵 세포 솔루션의 500 µ L를 추가 합니다.
- 다이제스트와 gDNAs 10 x 3 µ L를 혼합 하 여 30 µ L 소화 반응을 Dra I. 설정 다시 미리 버퍼 Dra에 대 한 난, 난, gDNAs/샘플, 및 H2O 10 µ g 30 µ L, 고 37 ° C에서 밤새 품 어 Dra의 3 µ L.
- DNA 젤, 5 µ L 소화 반응의 분석에 의해 소화의 완전성을 확인 하 고, 후속 단계에 대 한 DNA-로딩 버퍼 x 10의 3 µ L을 추가.
5. 남부 럽 및 PCR 식별
- 남쪽 더 럽 히 심사
- 전기 및 막에 의해 소화 gDNAs을 구분 합니다.
- Ethidium 평범한 사람 (EB)와 1 %agarose 전기 영동 젤을 준비 하 고 단계 4.3 및 1 kb 사다리에서 샘플 로드 달린 젤 낮은 전압 (30 ~ 40 V) 하룻밤.
- 꺼내서 젤 전기 이동 법 후 DNA 젤 이미징 시스템 사진의 찍을. 소화와 분리 된 gDNAs 얼룩 같은 이미지를 표시 하는지 여부를 확인 합니다.
- 실 온에서 20 분 동안 부드럽게 흔들어와 0.2 N HCl 솔루션 트레이에 젤을 담근 다.
- DNA 변성 시키기 솔루션 젤을 전송 하 고 실 온에서 20 분 동안 부드럽게 흔들어.
- 젤 DNA 중화 솔루션으로 전환 하 고 실 온에서 20 분 동안 부드럽게 흔들어.
참고: 젤 이므로이 단계 후 파손 하는 경향이 그것을 신중 하 게 처리 해야 합니다. - 급속 한 하향 전송 시스템을 사용 하 여 막에 젤에서는 DNAs를 전송. TurboBlotter, 더 럽 히 스택 제조 업체에서 제공 하는 지침에 따라 조립.
참고: 10 배 또는 20 배 염 분-나트륨 시트르산 (SSC) 솔루션 전송 버퍼로 사용 됩니다. 일반적으로, 전송의 3 h는 젤에서 막; gDNAs의 95%를 전송 하기에 충분 한 그러나, 전송의 장시간 무해 하지 않습니다. - 막 밖으로가 고 2로 그것을 씻어 1 분, x SSC 조직, 액체를 흡수 하 고 다음 UV crosslinker를 사용 하 여 막과 DNA를 상호 링크.
참고: 막 저장할 수 있습니다 4 ° C에서 1 주일 동안.
- 방사능과 DNA 프로브를 레이블을 지정 합니다.
- 제조업체에서 제공 하는 프로토콜에 따라 miniprep 키트를 사용 하 여 프로브 플라스 미드 정화.
- 릴리스는 DNA 파편의 EcoR에 의해 플라스 미드 벡터에서 프로브 나 버퍼의 5 µ L를 포함 하 여 EcoR에 대 한 반응 솔루션에 소화 나, EcoR의 2 µ L 나 효소, 플라스 미드 DNA의 20 µ g 및 H2O 50 µ L까지 2 h.
- DNA는 벡터에서 프로브 DNA 파편 분리 젤 1%를 실행 하 고 제조업체에서 제공 하는 프로토콜에 따라 DNA 젤 추출 키트 프로브의 DNA 파편을 정화.
- 260/280의 파장에서 분 광 광도 계 프로브 DNA 파편의 DNA 농도 측정 DNA 솔루션의 1 µ L를 사용 하 여 nm.
- 준비 테 버퍼의 45 µ L와 1.5 mL 튜브에 프로브 DNAs의 40-ng, 3 분 동안 삶아, 짧게, 회전 하 고, 장소는 tube(s) 2 분 동안 얼음에.
- 준비-가 DNA 라벨 구슬 포함 된 튜브에 열 변성 프로브 DNAs를 추가 (-dCTP), 혼합, [α32P] dCTP의 5 µ L을 추가 하 고 다음, 15 분 동안 37 ° C에서 품 어를 아래로 피펫으로.
- 제조업체에서 제공 하는 지침에 따라 G-50 microcolumns를 사용 하 여 레이블이 지정 된 프로브를 정화 하 고, 다음, 섬광 카운터 (옵션)에서 방사능을 측정.
- 레이블이 지정 된 프로브는 membrane(s)를 교배.
- 멤브레인 prehybridize
- 30 분 믹스 20 mL prewarmed 교 잡 솔루션 50 mL 튜브에의 삶은 연어 정자 DNA의 200 µ g와 42 ° C에서 교 잡 솔루션 prewarm
- 교 잡 튜브에 막 장소입니다. 교 잡 튜브를 혼합된 prehybridization 솔루션을 추가 합니다. 교 잡 오븐 (롤링 세트와 42 ° C에 온도)에 그것을 배치 하 고 prehybridization 30 분 동안 진행 하자.
- 레이블이 지정 된 probe(s)와 막 교배.
- 교 잡 튜브 밖으로가 고; 50 mL 튜브에 prehybridization 솔루션을 부 어 부드럽게이 튜브를 혼합 단계 5.1.2.7에서에서 변성된 프로브 (3 분 동안 100 ° C에가 열)를 추가 합니다.
참고: 모든 유도 거품을 줄일 수 있습니다. - 교 잡 튜브를 혼합된 솔루션을 반환 하 고 42 ° C에서 교 잡 하룻밤을 수행 합니다.
- 교 잡 튜브 밖으로가 고; 50 mL 튜브에 prehybridization 솔루션을 부 어 부드럽게이 튜브를 혼합 단계 5.1.2.7에서에서 변성된 프로브 (3 분 동안 100 ° C에가 열)를 추가 합니다.
- 멤브레인 prehybridize
- 워시 nonhybridized 프로브를 제거 하는 membrane(s).
- SSC + 0.1% SDS x 1 트레이에 membrane(s)를 놓고 10 분 동안 55-60 ° C에서 부드럽게 흔들어.
- 0.5 x SSC + 0.1 %SDS 용지함에는 membrane(s)를 전송 하 고 10 분 동안 55-60 ° C에서 부드럽게 흔들어.
- 3 세척 필요 여부를 결정 하는 휴대용가 거 카운터를 사용 하 여는 membrane(s)에 방사능을 확인 합니다.
- X 선 필름 막에 방사능 노출.
- 씻어 membrane(s)에서 액체를 제거 합니다.
- 플라스틱 포장을 가진 membrane(s) 싸다 고 노출 카세트에 그것/그들이 수정.
- 어두운 방에 x 선 필름 2 매를 막 노출.
- 1 박 이상-80 ° C에서 노출 카세트를 놓습니다.
- 개발 결과 시각화 하는 영화. 해당 ES 복제 대상된 재결합 또는 조사에 의해 검출 DNA 밴드의 크기에 따라, 원하는 하나 인지를 평가 합니다.
- 다음 절차에 따라 사용 된 프로브 스트립 후 또 다른 조사에 의해 동일한 막 rehybridize: 사용된 막 밖으로, 그것을 씻어 1 x H2O, 깨끗 하 고, 스트라이핑 솔루션에서 품 어 (formamide 55%, 2 %SSPE, 1 %SDS, H 2 O)에 1-2 h에 대 한 부드러운 떨고와 65 ° C.
- 전기 및 막에 의해 소화 gDNAs을 구분 합니다.
-
PCR 식별
- 10 x PCR 버퍼, 10mm dNTP의 50mm MgSO4, 1 µ L의 2 µ L, 20 µ M 앞으로 뇌관의 1 µ L의 5 µ L, 20 µ M 역 뇌관의 1 µ L를 포함 하 여 50 µ L 반응 솔루션에서 원하는 ES 클론의 PCR 식별을 수행 높은 충실도 플래티넘 Taq, gDNAs의 1 µ L (~ 100 ng), 그리고 H2O 50 µ L까지.
- 다음 PCR 반응 조건 사용: 30 94 ° C에서 변성의 30 주기 3 분, 94 ° C에서는 초기 변성 30 60 ° C에서 어 닐 링 s s와 132 68 ° C에서 확장 s, 그리고 10 분 동안 68 ° C의 마지막 단계.
- 1.0 %agarose 전기 이동 법으로 PCR 제품을 분석.
- T-쉬운 벡터 및 대상 벡터의 부분 시퀀스의 존재를 확인 하는 순서에 그들의 예상된 크기와 PCR 파편을 복제할.
결과
이 논문에서는 남부 럽 PCR의 상세한 프로토콜 설명, 셀 기반 HR 중재 대상 기술 ES를 사용 하 여 NM II 유전자 대체 마우스 모델의 생성에 대 한 마우스 ES 세포에서 발생 하는 이벤트를 시간을 식별 하기 위해 이용 되는 합니다. 비록 남부 럽 고 PCR, 뿐만 아니라 전통적인 유전자를 대상으로 기술, 널리 사용 되었습니다 수 십년 동안, 그들의 성공적인 적용 신중 하 게 계획 해야 합니다. 적어도 이러한 측면은 고려 되어야 하는 데 필요한: 길고 짧은 팔, 위치 및 프로브, 게놈 DNAs 뇌관 도움이 그림 1에 요약 된 대로, PCR를 위한 절단 적당 한 해상도의 길이의 길이 이후 분석입니다. 남부 럽의 중요 한 단계, 준비 하 고 소화 게놈 DNAs는 구분 하는 데 필요한 조사에 의해 감지에 대 한 DNA 젤에. DNAs 게놈 많은 서로 다른 길이와 조각으로 잘라는, 때문에 그들은 DNA 젤, 그림 2에 표시 된 대로 게놈 DNAs의 완전 한 소화를 제안에 얼룩 같은 상태를 표시 합니다. 남부 럽의 마지막 단계로, 방사능 표시 조사 대상 DNA 파편으로 교배 시키기의 신호는 그로 인하여 ES 클론 원하는 하나 인지를 나타내는 ES 클론에서 HR 이벤트의 발생을 반영, 영화에 표시 됩니다. 이 연구에서 predesign에 따라 돌연변이 대립 유전자와 ES 클론 야생-타입 ES 클론 하나만 한 밴드, 원하는 ES 클론 heterozygous (그림 3)은 제안 하는 동안 두 개의 명백한 크기 밴드, 있다. 절차 및 결과 남부 럽의 상대, 운영 및 PCR의 결과 간단 하 고 직접적인. PCR 반응에 따라 PCR 제품 분석 될 수 있다 DNA 젤에. PCR 악대는 특정 대상 벡터 상 동 팔 밖에 있는 게놈 영역으로 신 저항 유전자의 부분 시퀀스의 존재를 확인 복제 PCR 제품을 시퀀싱 하는 경우 시간 이벤트의 발생 수 있습니다. 예상 하 고 확인 (그림 4).
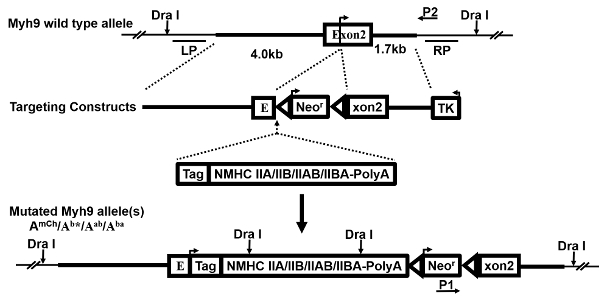
그림 1 : 구문을 타겟팅. 이것은 여러 대상 구문 생성을 보여주는 회로도 이다. 야생-타입 (WT) Myh9 유전자 대립 유전자, 유전자를 대상으로 벡터, 교체 외 인 식 cassette(s), 결과 돌연변이 allele(s) 뿐만 아니라 남쪽 오 점 및 PCR 위한 뇌관 (P1, P2) 프로브 (LP, RP) 표시 및 이전에 설명한 27. exon 2에는 화살표가 표시 변환 개시 사이트. HR의 성공적인 발생, 다음 대체 식 카세트와 네오 마이 신 저항 유전자 (네오r) 삽입은 그냥 5' 시작 ATG codon의. 따라서, 생 Myh9 대립 유전자는 중단 하 고 절에 gene(s)/돌연변이 세포와 생쥐에서 표현 됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 2 : Dra I.와 게놈 DNAs 소화 ES 클론 II B NMHC II-A 대체 구문을 함께 대상에서 게놈 DNAs는 내가 Dra로 소화 하 고, 다음, agarose 젤에 전기 이동 법으로 분리. 얼룩 같은 소화 gDNA 관찰 됩니다. C1-c 8 개별 ES 클론 묘사. GDNA의 완전 한 소화 얼룩 같은 이미지를 표시 함으로써 서로 다른 길이 가진 DNA 파편의 많은 생성 합니다. 이 결과 또한 준비 된 gDNAs의 좋은 품질 및 소화의 완전성을 반영 한다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.

그림 3 : 남부 럽의 대표적인 결과. 이러한 패널 왼쪽 및 오른쪽 프로브를 사용 하 여 ES 클론 NMHC II-A II-AB, 교체의 구조와 대상에서 게놈 DNAs의 남쪽 더 럽 히 심사를 보여줍니다. 돌연변이 allele 왼쪽된 프로브 또는 오른쪽 프로브를 사용 하면 각각에 WT 표시 9.7 kb 밴드 12.1 kb 또는 6 kb 밴드를 보여줍니다. M: 마커; P c 1-PC5: 긍정적인 복제품; NC: 부정적인 클론입니다. 남쪽 더 럽 히 밴드의 크기 또한 표시 됩니다. 남부 럽의 모든 절차는 엄격 하 게 실시 하 고 시험의 특이성은 충분; 밴드 예상된 밴드에 대 한 기대 없이 특이 현상 해야 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
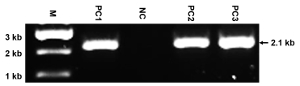
그림 4 : PCR의 대표적인 결과. 이 패널은 P1 + P2 뇌관 쌍을 사용 하 여 ES 클론 II-바 NMHC II-A 대체의 구조와 대상에서 게놈 DNAs의 PCR 식별을 보여 줍니다. 아무 밴드 WT 대립 유전자에 의하여 열매를 산출 하는 동안 돌연변이 대립 유전자 2.1 kb 밴드를 생성 합니다. M: 마커; P c 1-PC3: 긍정적인 복제품; NC: 부정적인 클론입니다. PCR 밴드의 크기 또한 표시 됩니다. 뇌관만 mutated 대립 유전자를 검색 하도록 설계 되었습니다, 이후 싱글과 예상 밴드의 모양을 뇌관 및 준비 gDNAs의 높은 품질의 특이성을 반영 합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭 하십시오.
토론
현재, 게놈 편집 디자이너 nucleases 여전히 ES 세포 기반 유전자를 대상으로 기술 대상에서 효과 및 긴 DNA 조각30,31삽입에 어려움의 그것의 문제로 바꿀 수 없습니다. 메서드로 골든 마우스 ES 세포에서 발생 하는 시간 이벤트를 식별 하는 데,이 보고서는 필드에 대 한 남부 럽 고 PCR의 상세한 프로토콜을 제공 합니다. 우리는 일련의 구문 대상으로 마우스 ES 세포에서 개별 클론을 분석 하 여 이러한 방법의 신뢰성을 검증. 이러한 방법으로 식별 원하는 ES 클론 생성 해당 마우스 모델27를 성공적으로 사용 되었습니다 했다.
타겟된 ES 클론의 심사에 대 한 다른 기술이 설명된19,32, 남쪽 더 럽 히기의 방법 그리고 PCR에 의해 완전히 대체 될 수 없다 비록 그 설립 이후32를 이러한 초기 때문에 기술을 더 이상 적용 된 역사를가지고 고 널리 사용할 수 있습니다 및 과학적인 사회, 대부분의 생물학 실험실에 의해 수행에 의해 확인 고 다른 기술의 근원. 중요 한 것은, 시간 이벤트의 식별에서 남부 럽 및 PCR의 좋은 성능은 잘 이전 작업29에서 궁 행. 남부 럽에서 결과 몇 가지 독특한 기능을 나타냅니다: 무작위로 상영된 ES 클론 중 이상 그들의 90%는 원하는 것 들, 아니 일반적인 밴드 감지 및 시간 Myh9 유전자의 1 개의 대립 유전자에 우선적으로 발생. 한편, 시퀀싱, PCR에서 데이터 확인 시간 이벤트의 발생 사이트 이며 남부 럽에서 그와 잘 일치 합니다.
우리의 연습에 따르면 남부 럽 고 PCR를 사용 하 ES 세포에서 시간 이벤트를 식별 함으로써 좋은 고 예상 결과 얻기 몇 가지 요소를 고려 한다. 첫 번째는; 상 동 팔의 길이 일반적으로, 상 동 팔 길이 증가 시간33의 효율성을 강화할 것 이다. 그러나, 이것은 아닙니다 항상 경우입니다. 한편으로, 더 이상 무기 조작;의 어려움 증가 다른 한편으로, 여기에서 보고 된 상 동 팔 (왼쪽된 팔과 오른쪽 팔에 대 한 1.7 kb 4 kb)의 길이 비슷한 실험 중 지금까지 얻은 최고의 시간 주파수 결과. 또한, 상 동 팔의 합리적인 길이 PCR에 의해 식별 용이. 두 번째 상 동 팔, 남쪽 더 럽 히 프로브34를 준비 하기 위한 isogenic DNA의 활용 이다. 이 유전자의 관심 영역을 포함 하는 BAC 클론을 주문 하거나 타겟팅 하기 위한 세포에서 게놈 DNA를 사용 하 여 만족하실 수 있습니다. 세 번째는 DNAs 게놈을 소화에 대 한 적당 한 해상도의 선택 이다. 일반적으로, 한 재 또는 대상 지역의 야생-타입 또는 돌연변이 체 대립 유전자만 번을 잘라 두 입술의 조합을 선호 하는; 또한, 결과 더 큰 DNA 파편 15 kb를 초과 하지 않아야 하 고 고유한 DNA 파편의 크기 차이 2 kb 이상. 이러한 요구 사항 수 용이 하 게 분리 및 예상된 밴드의 식별 남부 blotting에 의해. 4는 프로브 및 게놈에 다른 시퀀스와 최소 유사성의 길이입니다. 일반적으로, 프로브 길이 500-1000 혈압입니다. 게놈에 있는 다른 시퀀스와 유사성 NCBI 폭발 프로그램 분석할 수 있습니다. 또한, 남부 blotting에 대 한 프로브 되었습니다 디자인 하는 데 사용 하는 소프트웨어는35를 설명 합니다. 고려해 야 할 다섯 번째 요소 사용 하는 기존의 방법을 향상 수율에 대 한 게놈 DNA를 준비 하입니다. 48-잘 접시의 confluent 우물에서 준비 하는 게놈 DNAs는 일반적으로 충분히 남쪽 더 럽 히 분석의 적어도 2 개의 라운드. PCR를 위한 뇌관을 설계로 최고의 전략 대상 무기 이외의 입문서와 함께에서 선택 표식에 대 한 뇌관 존재를 사용 하는. 또한, PCR 제품을 시퀀싱은 시간 이벤트20,36을 증명 중요 하다입니다. 특히, PCR 기반 심사 완전히 그것은 효과적으로 평가 수 클론의 숫자를 줄일 수 있는 동안 남부 럽를 통해 얻은 정보를 바꿀 수 없습니다.
결론적으로, 남부 럽 고 PCR ES 세포에 유전자를 대상으로 이벤트 시간 중재를 식별 하기 위해 ES 클론 심사 잘 검증 된 방법이 있습니다. 여기서 주로 설명 하는 자세한 프로토콜 원하는 NM II 유전자 대체 ES 클론의 심사에 초점을 맞춘, 그것은 긍정적인 ES 클론을 사용 하 여 생성 된 이후에 유전형 마우스 사용할 수 있습니다. 그것은 다른 세포 유형, iPS 세포 등 체세포에에서 시간 이벤트의 식별에 쉽게 적응 될 수 있다.
공개
저자는 공개 없다.
감사의 말
이 작품의 국가 자연과학 기초의 중국 (보조금 번호 31571432), 인간의 지방 자연 과학 재단의 중국 (보조금 번호 2015JC3097), 그리고는 연구 재단의 교육 국의 호남 일반 프로그램에서 지원 받은 (보조금 번호 15 K 054) 지방, 중국.
자료
| Name | Company | Catalog Number | Comments |
| BAC CLONE | BACPAC Resources Center (BPRC) | bMQ-330E21 | |
| QIAGEN Large-Construct Kit | QIAGEN | 12462 | |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| QIAprep Spin Miniprep Kit | QIAGEN | 27104 | |
| QIAGEN Plasmid Plus Maxi Kit | QIAGEN | 12963 | |
| PfuUltra High-Fidelity DNA Polymerase | Agilent | 600382 | |
| T-easy vector | Promega | A1360 | |
| Nuclei Lysis Solution | Promega | A7941 | |
| Protein Precipitation Solution | Promega | A7951 | |
| DNA Denaturing Solution | VWR | 351-013-131 | |
| DNA Neutralizing Solution | VWR | 351-014-131 | |
| Ready-To-Go DNA Labeling Beads (-dCTP) | VWR | 27-9240-01 | |
| UltraPure SSC, 20x | Thermo Fisher | 15557036 | |
| UltraPur Phenol:Chloroform:Isoamyl Alcohol (25:24:1, v/v) | Thermo Fisher | 15593031 | |
| G418 | Thermo Fisher | 10131035 | |
| Salmon Sperm DNA Solution | Thermo Fisher | 15632011 | |
| Platinu Taq DNA Polymerase High Fidelity | Thermo Fisher | 11304029 | |
| Not I | Thermo Scientific | ER0592 | |
| Dra I | Thermo Scientific | ER0221 | |
| EcoR I | Thermo Scientific | ER0271 | |
| Ganciclovir | Sigma | G2536 | |
| Whatman TurboBlotter Transfer System, Large Kits | Fisher Scientific | 09-301-188 | |
| [α32P]dCTP | PerkinElmer | NEG013H100UC | |
| ProbeQuan G-50 Micro Columns | GE Healthcare | 28-9034-08 | |
| Hybrisol I Hybridization Solution | Millipore | S4040 | |
| Kodak X-Ray Film | Z&Z Medical | 844 5702 |
참고문헌
- Gao, G., McMahon, C., Chen, J., Rong, Y. A powerful method combining homologous recombination and site-specific recombination for targeted mutagenesis in Drosophila. Proceedings of the National Academy of Sciences of the United States of America. 105 (37), 13999-14004 (2008).
- Skarnes, W., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474 (7351), 337-342 (2011).
- Vogel, G. Nobel Prizes. A knockout award in medicine. Science. 318 (5848), 178-179 (2007).
- Salsman, J., Dellaire, G. Precision genome editing in the CRISPR era. Biochemistry. Cell Biology. 95 (2), 187-201 (2017).
- Capecchi, M. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nature Reviews Genetics. 6 (6), 507-512 (2005).
- Van, d. W. L., Adams, D. J., Bradley, A. Tools for targeted manipulation of the mouse genome. Physiological Genomics. 11 (3), 133-164 (2002).
- Glaser, S., Anastassiadis, K., Stewart, A. F. Current issues in mouse genome engineering. Nature Genetics. 37 (11), 1187 (2005).
- Bradley, A., Evans, M., Kaufman, M. H., Robertson, E. Formation of germ-line chimaeras from embryo-derived teratocarcinoma cell lines. Nature. 309 (5965), 255-256 (1984).
- Robertson, E., Bradley, A., Kuehn, M., Evans, M. Germ-line transmission of genes introduced into cultured pluripotential cells by retroviral vector. Nature. 323 (6087), 445-448 (1986).
- Thomas, K. R., Capecchi, M. R. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells. Cell. 51 (3), 503-512 (1987).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474 (7351), 337 (2011).
- Collins, F. S., Rossant, J., Wurst, W. A mouse for all reasons. Cell. 128 (1), 9-13 (2007).
- Poueymirou, W. T., et al. F0 generation mice fully derived from gene-targeted embryonic stem cells allowing immediate phenotypic analyses. Nature Biotechnology. 25 (1), 91-99 (2007).
- Pettitt, S. J., et al. Agouti C57BL/6N embryonic stem cells for mouse genetic resources. Nature Methods. 6 (7), 493-495 (2009).
- Gertsenstein, M., et al. Efficient Generation of Germ Line Transmitting Chimeras from C57BL/6N ES Cells by Aggregation with Outbred Host Embryos. PLoS One. 5 (6), 11260 (2012).
- Smithies, O., Gregg, R. G., Boggs, S. S., Koralewski, M. A., Kucherlapati, R. S. Insertion of DNA sequences into the human chromosomal |[beta]|-globin locus by homologous recombination. Nature. 317 (6034), 230-234 (1985).
- Deng, C., Capecchi, M. R. Reexamination of gene targeting frequency as a function of the extent of homology between the targeting vector and the target locus. Molecular & Cellular Biology. 12 (8), 3365 (1992).
- Lay, J. M., Friishansen, L., Gillespie, P. J., Samuelson, L. C. Rapid confirmation of gene targeting in embryonic stem cells using two long-range PCR techniques. Transgenic Research. 7 (2), 135-140 (1998).
- Langerak, P., Nygren, A. O. H., Schouten, J. P., Jacobs, H. Rapid and quantitative detection of homologous and non-homologous recombination events using three oligonucleotide MLPA. Nucleic Acids Research. 33 (22), 188 (2005).
- Gómezrodríguez, J., et al. Advantages of q-PCR as a method of screening for gene targeting in mammalian cells using conventional and whole BAC-based constructs. Nucleic Acids Research. 36 (18), 117 (2008).
- Kim, H. S., Smithies, O. Recombinant fragment assay for gene targeting based on the polymerase chain reaction. Nucleic Acids Research. 16 (18), 8887-8903 (1988).
- Joyner, A. L., Skarnes, W. C., Rossant, J. Production of a mutation in mouse En-2 gene by homologous recombination in embryonic stem cells. Nature. 338 (6211), 153-156 (1989).
- Ma, X., Adelstein, R. S. The role of vertebrate nonmuscle Myosin II in development and human disease. Bioarchitecture. 4 (3), 88-102 (2014).
- Malonek, D. Relationships between the dynamics of cortical blood flow, oxygenation, and volume changes following sensory stimulation. Proceedings of the National Academy of Sciences of the United States of America. 94, (1997).
- Takeda, K., Kishi, H., Ma, X., Yu, Z. X., Adelstein, R. S. Ablation and mutation of nonmuscle myosin heavy chain II-B results in a defect in cardiac myocyte cytokinesis. Circulation Research. 93 (4), 330-337 (2003).
- Conti, M. A., Evenram, S., Liu, C., Yamada, K. M., Adelstein, R. S. Defects in cell adhesion and the visceral endoderm following ablation of nonmuscle myosin heavy chain II-A in mice. Journal of Biological Chemistry. 279 (40), 41263-41266 (2004).
- Wang, A., et al. Nonmuscle myosin II isoform and domain specificity during early mouse development. Proceedings of the National Academy of Sciences of the United States of America. 107 (33), 14645-14650 (2010).
- Zhang, Y., et al. Mouse models of MYH9-related disease: mutations in nonmuscle myosin II-A. Blood. 119 (1), 238 (2012).
- Liu, T., et al. Identification and characterization of MYH9 locus for high efficient gene knock-in and stable expression in mouse embryonic stem cells. PLoS One. 13 (2), 0192641 (2018).
- Saito, S., Adachi, N. Advances in the Development of Gene-Targeting Vectors to Increase the Efficiency of Genetic Modification. Biological & Pharmaceutical Bulletin. 39 (1), 25-32 (2016).
- Langerak, P., Nygren, A., Schouten, J., Jacobs, H. Rapid and quantitative detection of homologous and non-homologous recombination events using three oligonucleotide MLPA. Nucleic Acids Research. 33 (22), 188 (2005).
- Martin, S. L., et al. A single amino acid substitution in ORF1 dramatically decreases L1 retrotransposition and provides insight into nucleic acid chaperone activity. Nucleic Acids Research. 36 (18), 5845-5854 (2008).
- Kamisugi, Y., Cuming, A. C., Cove, D. J. Parameters determining the efficiency of gene targeting in the moss Physcomitrella patens. Nucleic Acids Research. 33 (19), 173 (2005).
- Luo, Y., Bolund, L., Sørensen, C. B. Pig gene knockout by rAAV-mediated homologous recombination: comparison of BRCA1 gene knockout efficiency in Yucatan and Göttingen fibroblasts with slightly different target sequences. Transgenic Research. 21 (3), 671-676 (2012).
- Croning, M. D., Fricker, D. G., Komiyama, N. H., Grant, S. G. Automated design of genomic Southern blot probes. BMC Genomics. 11 (1), 74 (2010).
- Zimmer, A., Gruss, P. Production of chimaeric mice containing embryonic stem (ES) cells carrying a homoeobox Hox 1.1 allele mutated by homologous recombination. Nature. 338 (6211), 150-153 (1989).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유