Method Article
Identificazione di eventi di ricombinazione omologa in cellule staminali embrionali del Mouse mediante Southern Blotting e reazione a catena della polimerasi
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, presentiamo un protocollo dettagliato per identificare gli eventi di ricombinazione omologa che si è verificato nelle cellule staminali embrionali del mouse usando macchiare del sud e/o PCR. Questo metodo è esemplificato dalla generazione di modelli di non muscolare miosina II sostituzione genetica del mouse utilizzando tradizionale embrionale basati su cellule staminali omologa ricombinazione mediata tecnologia di targeting.
Abstract
Relativo alle edizioni degli effetti fuori bersaglio e la difficoltà di inserimento di un frammento di DNA lungo nell'applicazione del progettista nucleasi per staminali embrionali, modifica del genoma (ES) basati su cellule gene-targeting tecnologia non ha queste carenze ed è ampiamente utilizzato per modificare il genoma di animali/mouse che vanno da grandi delezioni/inserzioni a sostituzioni di singoli nucleotidi. In particolare, identificazione degli eventi di ricombinazione omologa (HR) relativamente pochi necessari ottenere desiderato ES cloni è un passo fondamentale, che richiede metodi accurati e affidabili. Macchiarsi del sud e/o PCR convenzionale sono spesso utilizzati per questo scopo. Qui, descriviamo le modalità di utilizzo di questi due metodi per identificare gli eventi HR che si sono verificati in cellule di topo ES in cui il gene Myh9 endogeno è destinato ad essere interrotto e sostituito da cDNA codificanti altre catena pesante di miosina non muscolare IIs (IIs NMHC). Tutta la procedura di macchiare del sud comprende la costruzione di targeting tutt', elettroporazione, selezione della droga, l'espansione e la conservazione di cellule ES/cloni, la preparazione, la digestione e macchiare del DNA genomico (gDNA), l'ibridazione e lavaggio della/delle sonde e una fase finale di autoradiografia sulle pellicole di raggi x. PCR può essere eseguita direttamente con gDNA preparato e diluito. Per ottenere risultati ideali, le sonde e l'enzima di restrizione (ri) siti di taglio per macchiare del sud e i primers per la PCR deve essere pianificati con attenzione. Anche se l'esecuzione di macchiare del sud è molto tempo e laborioso e risultati di PCR sono falsi positivi, la corretta identificazione di Southern blotting e screening rapido mediante PCR consentono l'applicazione di sola o in combinazione di questi metodi descritti in questa carta per essere ampiamente utilizzati e consultati dalla maggior parte dei laboratori nell'identificazione di genotipi di cellule ES e geneticamente animali modificati.
Introduzione
La tecnologia del gene targeting per HR in cellule ES murine fornisce un potente strumento per la dissezione delle conseguenze cellulari di specifiche mutazioni genetiche1,2. L'importanza e il significato di questa tecnologia sono riflessi nel suo riconoscimento dal 2007 Premio Nobel in fisiologia o medicina3,4; nel frattempo, rappresenta l'avvento dell'era moderna di ingegneria gene5. Gene targeting attraverso HR può essere utilizzato per progettare virtualmente qualsiasi alterazione che vanno da mutazioni puntiformi a grandi riorganizzazioni cromosomiche nel genoma del topo le cellule ES6,7. È noto che, prima della nascita di strumenti di editing del cosiddetto genoma, la generazione di un mouse di knockout del gene necessaria l'applicazione di tecnologia di targeting di gene in cellule ES8,9,10. Durante le due decadi scorse, più di 5.000 topi gene targeting sono state prodotte da questo approccio per malattie umane di modellazione o studiare funzioni gene11. Uno sforzo di genoma ad eliminazione diretta è stato stabilito per la distribuzione dei vettori gene-targeting, cloni di cellule ES mirati e topi dal vivo per la comunità scientifica2,12,13,14 , 15. indubbiamente, tecnologia da gene-targeting HR-mediata basati su cellule ES ha notevolmente migliorato la nostra comprensione delle funzioni dei geni giocati nel contesto fisiologico o patologico.
Perché HR è un evento relativamente raro nei mammiferi le cellule16,17, l'importante e il prossimo passo seguendo gene-targeting in cellule ES murine è quello di analizzare numerose colonie di ES per l'identificazione di alcuni cloni con mutazioni derivanti da HR con il targeting per vector18. I metodi d'oro per identificare gli eventi HR includono Southern blotting e PCR19,20. I vantaggi degli approcci comprendono che macchiarsi del sud può identificare correttamente mirati ES cloni e permette ai ricercatori di analizzare la struttura dell'evento gene targeting, come una verifica di un inserimento di copia singola del costrutto, mentre un Strategia basata su PCR permette più rapido di screening per HR eventi21,22. Anche se questi metodi sono inconvenienti, quali che essi sono che richiede tempo e possono avere falsi positivi, il loro utilizzo combinatorio è ampiamente accettati e applicati dalla maggior parte dei laboratori nell'identificare eventi HR, particolare in cellule ES, per generare geneticamente modificata animali.
Tre isoforme della miosina non muscolare II (NM II) nei mammiferi, ciascuno composto da due identici NMHC IIs che sono codificati da tre geni differenti (denominati Myh9, Myh10 e Myh14) e due coppie di catene leggere, sono denominate NM II A, II B e II-C23. Gli studi precedenti hanno indicato che almeno le isoforme di NM II A e II B sono essenziali per lo sviluppo del mouse perché l'ablazione in vivo di queste isoforme provoca mortalità embrionale24,25,26. Per aggirare questo problema e ottenere nuove conoscenze sulle funzioni specifiche di isoforma di NM II A e II B nelle fasi successive di sviluppo del topo, una sostituzione di genetica è stata adottata la strategia utilizzando tecnologia di gene-targeting HR-mediata basati su cellule ES a generare una serie di modelli di mouse27. Nel corso di identificare i cloni ES desiderati, Southern blotting e metodi di PCR sono stati utilizzati, e questi si è rivelata efficiente e affidabile27,28.
Questo libro intende fornire una descrizione dettagliata di Southern blotting e PCR, compresa la progettazione di targeting tutt', le sonde e gli iniettori e l'esecuzione di esperimenti, così come l'analisi dei risultati esemplificato identificando evento HR avvenimento in cellule ES per la creazione del mouse sostituzione genetica NM II modelli e dati rappresentativi. I protocolli di questi due metodi qui presentati possono essere adottati anche per l'identificazione dei genotipi di cellule geneticamente modificate o di animali.
Protocollo
1. design di Targeting Construct(s), sonde per sud della macchia e primer per PCR
- Selezionare il primo esone codifica (esone 2) del gene Myh9 per rottura o inserimento nell'applicazione di knockout/knock-in segnalato qui.
- Recuperare il 5-kb a Monte e 5-kb a valle sequenze di DNA che circondano l'esone Myh9 2 dal sito Web genome.ucsc.edu .
- Analizzare i modelli di digestione di limitazione degli enzimi (REs) con 1 – 2 tagli in questa regione di 10 kb utilizzando il software pDRAW per determinare RE(s) adatto per digerire il DNA genomic per macchiare del sud.
Nota: Dra soddisfa questo requisito e viene selezionato per lo scopo. - Selezionare un frammento di 4 kb immediatamente a monte dell'essone Myh9 2 come il braccio di sinistra omologia (LHA) e una sequenza di 1,7 kb frammento immediata a valle come il braccio destro di omologia (RHA); scegliere un frammento di 1 kb 5' a Monte di LHA come la sonda sinistra (LP) per macchiare del sud e un frammento di 1,2 kb 3' a valle di RHA come la sonda destra (RP), sulla base dell'analisi di cui sopra.
- Utilizzare un programma primer3 per progettare il primer forward e reverse per amplificare quei quattro frammenti di DNA mediante PCR. Progettare un paio degli iniettori con il primer forward riseduto vicino al terminal 3' di un gene di resistenza neomyocin per marcatore di selezione (P1) e il primer reverse situato appena fuori il RHA (P2).
Nota: Questa coppia di primer verrà utilizzata per l'identificazione dei cloni ES mirati da PCR29. - Trovare un clone BAC 129Sv che copre il luogo del gene del mouse Myh9 visitando il sito Web bacpac.chori.org (Nota: isogeniche DNA è preferito). Isolare il DNA di BAC utilizzando un kit adatto per purificare grandi pezzi di DNA seguendo le istruzioni fornite dal produttore.
Nota: DNA purificato BAC verrà utilizzato come modello per l'amplificazione di PCR. - Disegnare una rappresentazione schematica del targeting costrutti, sonde e primer per riepilogare le informazioni.
2. generazione di Targeting Construct(s) e sonde per sud della macchia e la preparazione degli iniettori per la PCR
- Ordinare gli iniettori PCR descritti sopra e scioglierli in una concentrazione di 20 µM.
- Amplificare le braccia di omologia e sonde di PCR in una soluzione di reazione tra cui iniettori d'inversione avanti e 1 µ l di 1 µ l, 1 µ l di DNA di BAC (50 ng) come il modello, 5 µ l di tampone per Pfu e 1 µ l di Pfu ultra come la polimerasi del DNA , e H2O fino a 50 µ l. realizzare la PCR in una macchina PCR nelle seguenti condizioni: 95 ° C per 3 min; 95 ° C per 30 s; 60 ° C per 30 s; 72 ° C e 1 kb/min; 30 cicli; Infine, 72 ° C per 10 min.
- Purificare i prodotti di PCR utilizzando un kit di pulizia PCR secondo il protocollo fornito dal produttore ed eluire i frammenti di DNA con 40 µ l di H2O. Digest i prodotti di PCR purificati con REs predefiniti in una soluzione di reazione contenente 5 µ l di tampone 10x 10 per REs , 2,5 µ l di RE1 e 2,5 µ l di RE2 ed il DNA eluito, a 37 ° C per 2 h. eseguire un gel del DNA di 1% per separare le bande di DNA bersaglio, asportare il gel contiene il target DNA sotto la luce UV, purificare i frammenti di DNA bersaglio dal gel utilizzando un gel DNA estrazione kit secondo il protocollo fornito dal produttore ed eluire i frammenti di DNA con 40 µ l di H2O.
- Clonare le braccia di omologia e sostituzione se espressione di utilizzare plafoniere nel vettore mpNTKV-LoxP secondo l'ordine dell'amplificato e purificata RHA, LHA e sostituzione espressione se utilizzare plafoniere rilasciato da altri vettori per ottenere la destinazione finale Construct(s). Clonare frammenti di DNA delle sonde amplificate e purificate nel vettore T-facile.
- Confermare le sequenze nucleotidiche di tutti i frammenti di DNA clonati mediante sequenziamento27,28.
3. preparazione di Targeting Construct(s), l'elettroporazione delle celle di ES e l'amplificazione di cloni ES
- Preparare ogni costrutto targeting utilizzando un plasmide maxiprep kit secondo il protocollo fornito dal produttore. Linearizzare ogni plasmide costrutto di digerirlo con non ho in una reazione di 400 µ l compreso 40 µ l di tampone 10x per non I, 10 µ l di non io, 100 µ g di DNA e H2O fino a 400 µ l, a 37 ° C durante la notte.
- Purificare il construct(s) targeting linearizzato.
- Estrarre la soluzione di reazione digerito 1 x con un volume uguale del fenolo: cloroformio: alcool isoamilico (25:24:1) e centrifugare a una forza di 2.000 x g per 10 min.
- Trasferire il surnatante in una provetta da 1,5 mL nuova, precipitare il DNA utilizzando 2,5 x etanolo e 0,1 x 3m acetato di sodio (pH 5.2) (rapporto di volume) e centrifugare a una forza di 2.000 x g per 10 min.
- Rimuovere il surnatante, lavare il DNA a pellet 1x con 1 mL di etanolo al 75% e centrifugare a una forza di 2.000 x g per 5 min.
- Rimuovere il supernatante e asciugare all'aria il pellet di DNA per 5 min.
- Dissolva la pallina del DNA linearizzata in tampone Tris-EDTA (TE) sterile ad una concentrazione finale di 1 µ g / µ l.
- Mescolare 50 µ g di ogni costrutto linearizzato targeting con 0,5 x 10 cellule7 ES. Eseguire l'elettroporazione a 320 V e 250 µF. piastra le cellule elettroporate ES su piatti con alimentatori MEF neo-resistente.
- Dopo 24 h, passare al medium di cellule ES con 400 µ g/mL G418 e 200 µM ganciclovir e continuare alla cultura per 4 – 5 giorni con un cambio medio giornaliero. Ritirare il farmaco-resistenti ES cloni in 48 pozzetti.
Nota: Normalmente, quattro piastre 48 pozzetti vengono utilizzate per costrutto. - Duplicare i 48 pozzetti.
Nota: Un insieme delle piastre è crioconservato, e l'altro set viene utilizzato per la preparazione di DNA genomico.
4. preparazione di DNAs Genomic e la digestione con Restriction Enzyme(s)
- Preparare gDNAs da cellule ES utilizzando un kit commerciale (kit di purificazione di DNA genomico) con modifiche minori.
- Rimuovere i supporti dalla coltura delle cellule di ES e aggiungere 500 µ l di soluzione di lisi di nuclei, tra cui RNaseA, direttamente ai pozzetti per lisare le cellule.
Nota: Il lysate delle cellule possono essere conservati a-80 ° C o trattata immediatamente. - Dispensare su e giù più volte per lisare le cellule completamente e trasferirli in una provetta pulita 1,5 mL.
- Aggiungere un terzo del volume della soluzione di precipitazione della proteina per la provetta da 1,5 mL, vortexare vigorosamente per 20 s, chill i campioni in ghiaccio per 5 minuti e poi, centrifugare a una forza di 2.000 x g per 5 min, il surnatante di trasferimento a un altro pulito 1,5 mL tubo contai Ning un uguale volume di isopropanolo; mescolare delicatamente la soluzione. (Si noti che i filamenti bianchi filiformi possono essere visto in questo momento). Centrifugare a una forza di 2.000 x g per 1 min; quindi, eliminare il supernatante.
- Lavare la pallina gDNA con 1 mL di etanolo al 70% a temperatura ambiente, centrifugare a una forza di 2.000 g per 1 min, aspirare il surnatante con cura e poi asciugare all'aria il pellet gDNA per 3 min.
- Sciogliere il gDNAs con 100 µ l di soluzione di reidratazione del DNA e, quindi, incubare a 65 ° C per 1 h o a 4 ° C durante la notte.
- Negozio gDNAs a 2 – 8 ° C.
- Rimuovere i supporti dalla coltura delle cellule di ES e aggiungere 500 µ l di soluzione di lisi di nuclei, tra cui RNaseA, direttamente ai pozzetti per lisare le cellule.
- Digest gDNAs con predefiniti RE Dra I. insieme a una reazione di digestione 30-µ l mescolando 3 µ l di 10X tampone per Dra I, 3 µ l di Dra I, 10 µ g di gDNAs/campione e H2O fino a 30 µ l e incubare a 37 ° C durante la notte.
- Verifica la completezza della digestione di gel del DNA, l'analisi di 5 µ l del digerito e poi, aggiungere i 3 µ l di tampone di DNA-caricamento per il passo successivo: 10x.
5. Southern Blotting e PCR identificazione
- Southern blotting screening
- Separare il gDNAs digeriti mediante elettroforesi e trasferimento in una membrana.
- Preparare un gel di agarosio all'1% elettroforesi con bromuro di etidio (EB), caricare i campioni da passo 4.3 e una scala di 1 kb ed eseguire il gel a bassa tensione (30 – 40 V) durante la notte.
- Prendere il gel e scattare una foto con un sistema di formazione immagine di gel del DNA dopo l'elettroforesi. Controllare se il digerito e separato gDNAs visualizzare un'immagine simile a striscio.
- Immergere il gel in un vassoio con 0,2 N HCl soluzione e scuoterla delicatamente per 20 min a temperatura ambiente.
- Trasferire il gel al DNA-denaturare la soluzione e scuoterla delicatamente per 20 min a temperatura ambiente.
- Passare il gel nella soluzione di DNA-neutralizzante e scuoterla delicatamente per 20 min a temperatura ambiente.
Nota: Il gel è incline alla rottura dopo questo passaggio, quindi deve essere maneggiato con cura. - Utilizzare il sistema di trasferimento rapido verso il basso per trasferire il DNA dal gel alla membrana. Assemblare il TurboBlotter e lo stack macchiante seguendo le istruzioni fornite dal produttore.
Nota: x 10 o 20 x salino-sodio citrato (SSC) soluzione viene utilizzato come un buffer di trasferimento. In generale, 3 h di trasferimento è sufficiente per trasferire il 95% dei gDNAs dal gel alla membrana; Tuttavia, un tempo più lungo di trasferimento è innocuo. - Togliere la membrana e lavarlo con 2 x SSC per 1 min, assorbire il liquido con i tessuti e quindi del legame incrociato del DNA con la membrana utilizzando un reticolante UV.
Nota: La membrana può essere memorizzata a 4 ° C per una settimana.
- Etichettare le sonde di DNA con la radioattività.
- Purificare i plasmidi della sonda utilizzando un kit miniprep secondo il protocollo fornito dal produttore.
- Rilascio il DNA frammenti delle sonde dal vettore plasmidico di EcoR ho digestione in una soluzione di reazione con 5 µ l di tampone per EcoR I, 2 µ l di EcoR io H2O fino a 50 µ l, 20 µ g di DNA plasmidico e degli enzimi , per 2 h.
- Eseguire un 1% del gel del DNA per separare i frammenti di DNA sonda dal vettore e purificare i frammenti di DNA delle sonde con un kit di estrazione del gel del DNA secondo il protocollo fornito dal produttore.
- Con 1 µ l di soluzione di DNA, misurare la concentrazione di DNA dei frammenti del DNA sonda con uno spettrofotometro alla lunghezza d'onda di 260/280 nm.
- Preparare 40-ng di sonda DNAs in una provetta da 1,5 mL con 45 µ l di tampone di TE, far bollire per 3 minuti, girare brevemente e quindi, posizionare le provette in ghiaccio per 2 min.
- Aggiungere il DNAs sonda denaturata al calore nella provetta contenente microsfere di DNA-etichettatura di ready-to-go (-dCTP), dispensare su e giù per mescolare, aggiungere 5 µ l di [α32P] dCTP e quindi, incubare a 37 ° C per 15 min.
- Purificare le sonde identificate utilizzando microcolonne di G-50 secondo le istruzioni fornite dal produttore e, quindi, misurare la radioattività di un contatore di scintillazione (opzionale).
- Ibridare il membrane(s) con la sonda marcata.
- Prehybridize la membrana.
- Preriscaldare la soluzione di ibridazione a 42 ° C per 30 min. Mix 20 mL di soluzione di ibridazione preriscaldati con 200 µ g di sperma di salmone bollito del DNA in un tubo da 50 mL.
- Inserire la membrana nella provetta di ibridazione. Aggiungere la soluzione mista di prehybridization per il tubo di ibridazione. Metterlo in forno ibridazione (set di rotolamento e la temperatura a 42 ° C) e lasciare che il prehybridization procedere per 30 min.
- Ibridare la membrana con il sonde con etichettate.
- Estrarre il tubo di ibridazione e versare la soluzione prehybridization in una provetta da 50 mL; aggiungere la sonda denaturata (riscaldata a 100 ° C per 3 min) dal passaggio 5.1.2.7 questo tubo e mescolare delicatamente.
Nota: Ridurre qualsiasi bolle d'induzione. - Restituire la soluzione mista al tubo di ibridazione ed eseguire l'ibridazione a 42 ° C durante la notte.
- Estrarre il tubo di ibridazione e versare la soluzione prehybridization in una provetta da 50 mL; aggiungere la sonda denaturata (riscaldata a 100 ° C per 3 min) dal passaggio 5.1.2.7 questo tubo e mescolare delicatamente.
- Prehybridize la membrana.
- Lavare il membrane(s) per rimuovere nonhybridized sonde.
- Inserire il membrane(s) in un vassoio con 1 x SSC + 0,1% SDS e agitare delicatamente a 55 – 60 ° C per 10 min.
- Trasferire la membrane(s) a un vassoio con 0,5 x SSC + 0,1% SDS e agitare delicatamente a 55 – 60 ° C per 10 min.
- Controllare la radioattività sul membrane(s) utilizzando un contatore Geiger portatile per decidere se un terzo lavaggio è necessario.
- Esporre la radioattività sulla membrana per pellicole radiografiche.
- Rimuovere il liquido dal membrane(s) lavato.
- Avvolgono il membrane(s) con involucro di plastica e difficoltà/li nel cassetto dell'esposizione.
- Esporre la membrana a due fogli di pellicola di raggi x in una stanza buia.
- Posizionare la cassetta di esposizione a-80 ° C in una notte o più.
- Sviluppare le pellicole per visualizzare i risultati. Valutare se un clone ES corrispondente è quella desiderata con la ricombinazione mirata o non, secondo le dimensioni delle bande di DNA rilevate dalle sonde.
- Rehybridize la membrana stessa di un'altra sonda dopo stripping off la sonda utilizzata secondo la seguente procedura: estrarre la membrana usata, lavarlo 1 x con H2O di pulire e incubare in soluzione lo striping (55% formammide, 2% SSPE, 1% SDS, H 2 O) a 65 ° C con agitazione delicata per 1 – 2 h.
- Separare il gDNAs digeriti mediante elettroforesi e trasferimento in una membrana.
-
Identificazione di PCR
- Eseguire l'identificazione di PCR dei cloni ES desiderati in un 50-µ l di soluzione di reazione tra cui 5 µ l di buffer di 10 x PCR, 2 µ l di 50mm MgSO4, 1 µ l di 10 mM dNTP, 1 µ l di primer avanti 20 µM, 1 µ l di primer inverso di 20 µM , 1 µ l di Taq platino ad alta fedeltà, gDNAs (~ 100 ng) e H2O fino a 50 µ l.
- Utilizzare le seguenti condizioni di reazione di PCR: una denaturazione iniziale a 94° C per 3 min, 30 cicli di denaturazione a 94° C per 30 s, ricottura a 60° C per 30 s e un'estensione a 68° C per 132 s e un passaggio finale di 68° C per 10 min.
- Analizzare i prodotti PCR mediante elettroforesi in agarosio 1.0%.
- Duplicare i frammenti PCR con loro dimensione prevista nel vettore T-facile e la sequenza per confermare la presenza di una sequenza parziale del vettore di destinazione.
Risultati
In questa carta, un protocollo dettagliato di Southern blotting e PCR è descritto, che viene utilizzato per identificare gli eventi HR che si sono verificati in cellule di topo ES per la generazione di modelli murini di NM II sostituzione genetica, utilizzando ES basati su cellule HR-mediato tecnologia di targeting. Anche se Southern blotting e PCR, nonché tradizionale tecnologia gene-targeting, è stati ampiamente utilizzata per diversi decenni, la riuscita applicazione di loro deve essere pianificato con cura. Almeno questi aspetti sono necessari per essere considerato: la lunghezza delle braccia lunghe e Corte, le posizioni e la lunghezza delle sonde, la REs adatta per il taglio di DNAs genomic e i primers per PCR, come riassunto nella Figura 1, che è utile per analisi successiva. Come un passo importante di Southern blotting, il preparato e digerito DNAs genomic sono tenuti a essere separati su gel del DNA per la rilevazione della sonda. Perché DNAs genomic sono tagliate in un sacco di frammenti con lunghezze diverse, mostrano uno status di striscio-come sul gel DNA, suggerendo una digestione completa di DNAs genomic, come indicato nella Figura 2. Come passo finale del Southern blotting, i segnali di una sonda marcata con radioattività ibridare con un frammento di DNA bersaglio sono mostrati sulla pellicola, che riflettono il verificarsi di eventi HR in cloni ES, indicando così sia un clone di ES è quello desiderato. Secondo il predesign in questo studio, ES cloni con allele mutato hanno due dimensioni distinte bande, mentre selvaggio-tipo ES cloni hanno solo una banda, suggerendo i cloni ES desiderati sono eterozigoti (Figura 3). Relativo alla procedura e risultati di Southern blotting, il funzionamento e i risultati della PCR sono semplici e diretti. A seguito della reazione di PCR, i prodotti PCR possono essere analizzati su gel del DNA. Se le bande PCR specifiche e sequenziamento dei prodotti PCR clonati conferma la presenza di una sequenza parziale di vettore di destinazione come un gene neo-resistenza, come pure le regioni genomiche che sono appena fuori il braccio di omologia, il verificarsi di eventi HR può essere previsto e verificato (Figura 4).
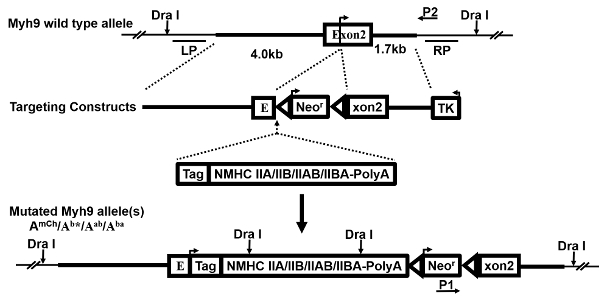
Figura 1 : Targeting costrutti. Questo è lo schema dimostrando la generazione di costrutti di targeting multiple. L'allele del gene wild type (WT) Myh9, gene-targeting vettoriale, se utilizzare plafoniere espressione esogena di sostituzione e il risultante alleli mutati, come pure le sonde (LP, RP) del sud della macchia per il primer (P1, P2) per PCR, sono mostrate e descritti in precedenza 27. una freccia sull'esone 2 indica il sito di iniziazione traduzionale. In seguito il successo di HR, la cassetta di espressione di sostituzione e il gene di resistenza di neomicina (Neor) vengono inseriti solo 5' del codone d'inizio ATG. Di conseguenza, l'allele di Myh9 endogeno è interrotta e bussò-in geni sono/sono espressi nelle cellule mutanti e topi. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2 : Digerito DNAs genomic con Dra I. DNAs genomic da cloni ES mirati con il costrutto sostituendo NMHC II-A con II-B sono digeriti con Dra io e, quindi, separate su un gel di agarosio mediante elettroforesi. Un gDNA digerito di striscio-come è stato osservato. C1 - C8 raffigurano singoli cloni ES. Una digestione completa di gDNA produce un sacco di frammenti di DNA con una lunghezza diversa, quindi visualizzazione di un'immagine simile a striscio. Questo risultato riflette anche la buona qualità dei preparati gDNAs e la completezza della digestione. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3 : Risultati rappresentativi di Southern blotting. Questi pannelli mostrano uno screening Southern blotting di DNAs genomic da cloni ES mirati con il costrutto della sostituzione NMHC II-A con II-AB, utilizzando le sonde di sinistra e destra. L'allele mutato Mostra una band 12,1 kb o 6 kb quando la sonda sinistra o destra sonda viene utilizzato, rispettivamente, mentre il WT Mostra una banda di 9,7 kb. M: marcatore; PC1-PC5: cloni positivi; NC: clone negativo. Sono anche indicate le dimensioni delle bande Southern blotting. Tutte le procedure di Southern blotting sono rigorosamente effettuate e la specificità delle sonde è abbastanza buona; non ci dovrebbe essere nessun aspecifici bande si aspettano per le bande previste. Clicca qui per visualizzare una versione più grande di questa figura.
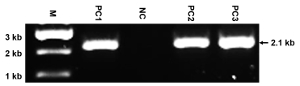
Figura 4 : Risultati rappresentativi della PCR. Questo pannello mostra l'identificazione di PCR di DNAs genomic da cloni ES mirati con il costrutto della sostituzione NMHC II-A con II-BA utilizzando la coppia di primer P1 + P2. L'allele mutato produce una banda di 2,1 kb, mentre l'allele WT non produce nessuna banda. M: marcatore; PC1-PC3: cloni positivi; NC: clone negativo. La dimensione della banda PCR è anche indicata. Poiché i primer sono progettati per rilevare solo l'allele mutato, la comparsa di una banda singola e attesa riflette la specificità dei primers e l'alta qualità del gDNAs preparato. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Attualmente, progettista nucleasi per editing genomico ancora non possono sostituire la tecnologia di gene-targeting basati su cellule ES a causa di suoi problemi di effetti fuori bersaglio e la difficoltà nell'inserimento di un lungo di30,di DNA frammento31. Come i metodi d'oro per identificare gli eventi HR che si sono verificati in cellule di topo ES, questo rapporto fornisce un protocollo dettagliato di Southern blotting e PCR per il campo. Abbiamo validato l'affidabilità di questi metodi analizzando singoli cloni da cellule di topo ES mirati con una serie di costrutti. I cloni ES desiderati identificati da questi metodi erano stati utilizzati con successo per generare il corrispondente del mouse modelli27.
Anche se altre tecniche per lo screening di cloni ES mirati sono stati descritti19,32, i metodi di Southern blotting e PCR non possono essere completamente sostituiti da quelli stabiliti da allora in poi32, perché questi iniziale tecniche hanno una storia più applicata e sono ampiamente accettati e confermati dalla società scientifica, eseguita dalla maggior parte dei laboratori biologici e sono all'origine di altre tecnologie. D'importanza, la buona prestazione di Southern blotting e PCR nell'identificazione di eventi HR è ben esemplificata nel precedente lavoro29. I risultati da macchiare del sud indicano diverse caratteristiche uniche: tra i cloni ES casualmente schermati, oltre il 90% di loro sono quelli desiderati, fasce non specifiche non sono rilevate e l'alto rappresentante si è verificato preferenzialmente su un allele del gene Myh9. Nel frattempo, i dati da PCR, sequenziamento, insieme a confermano che il verificarsi di eventi HR è site-specific e abbinare bene con quelli da macchiare del sud.
Secondo la nostra pratica, diversi fattori da considerare quando Southern blotting e PCR sono utilizzati per identificare gli eventi HR in cellule ES, ottenendo così risultati buoni e previsti. Il primo è la lunghezza delle braccia omologia; in generale, aumentando la lunghezza del braccio di omologia migliorerà l'efficienza di HR33. Tuttavia, questo non è sempre il caso. Da un lato, braccia più lunghe aumentano la difficoltà di manipolazione; d'altra parte, la lunghezza dei bracci omologia (4 kb per il braccio sinistro e 1,7 kb per il braccio destro) segnalata qui ha provocato la più alta frequenza di HR finora ottenuta tra esperimenti simili. Inoltre, una lunghezza ragionevole delle braccia di omologia facilita l'identificazione mediante PCR. Il secondo è l'utilizzo di DNA isogeniche per preparare le braccia di omologia e Southern blotting sonde34. Questo può essere soddisfatto ordinando un clone BAC contenente la regione del gene di interesse o usando il DNA genomic dalle cellule destinate ad essere bersaglio. Il terzo è la selezione di REs adatto per digerire DNAs genomic. In generale, un RE o la combinazione di due REs che tagliato l'allele wild-type o mutante solo una volta o due volte intorno alla regione di destinazione sono preferiti; Inoltre, il frammento di DNA più grande risultante non deve superare i 15 kb e la differenza di dimensioni tra i frammenti di DNA distinti è superiore a 2 kb. Questi requisiti possono facilitare la separazione e l'identificazione di bande previsti dal macchiare del sud. Il quarto è la lunghezza delle sonde e la minima somiglianza con altre sequenze nel genoma. Generalmente, la lunghezza delle sonde è 500 – 1.000 bp. La somiglianza con altre sequenze nel genoma possa essere analizzata con il programma di NCBI BLAST. Inoltre, un software utilizzato per la progettazione delle sonde per macchiare del sud è stato descritto35. Il quinto fattore da considerare è quello di utilizzare i metodi convenzionali per preparare il DNA genomico per una resa d'aumento. DNAs genomic preparato da un pozzo confluente di una piastra a 48 pozzetti sono generalmente abbastanza per almeno due turni di Southern blotting analisi. Quanto a progettare il primer per PCR, la strategia migliore consiste nell'utilizzare un primer presenti sul marcatore di selezione in combinazione con un primer fuori le braccia targeting. Inoltre, i prodotti PCR di sequenziamento è importante per dimostrare HR eventi20,36. In particolare, screening basato su PCR non può sostituire completamente le informazioni ottenute attraverso Southern blotting, mentre può efficacemente ridurre i numeri dei cloni da valutare.
In conclusione, Southern blotting e PCR sono ben dimostrate metodi per lo screening di cloni ES per identificare eventi di gene-targeting HR-mediati in cellule ES. Se il protocollo dettagliato descritto qui principalmente incentrato sullo screening di cloni di sostituzione genetica ES II NM desiderati, può essere utilizzato per topi di genotipizzazione che successivamente vengono generati utilizzando i cloni positivi ES. Può essere facilmente adattato per l'individuazione degli eventi HR in altri tipi cellulari, quali le cellule iPS o cellule somatiche.
Divulgazioni
Gli autori non hanno nulla a rivelare.
Riconoscimenti
Questo lavoro ha ricevuto il supporto dal programma generale del National Natural Science Foundation of China (sovvenzioni No. 31571432), l'umano provinciale Natural Science Foundation della Cina (Grant No. 2015JC3097) e la Fondazione ricerca di formazione Bureau di Hunan Provincia, Cina (Grant n ° 15 K 054).
Materiali
| Name | Company | Catalog Number | Comments |
| BAC CLONE | BACPAC Resources Center (BPRC) | bMQ-330E21 | |
| QIAGEN Large-Construct Kit | QIAGEN | 12462 | |
| QIAquick Gel Extraction Kit | QIAGEN | 28704 | |
| QIAquick PCR Purification Kit | QIAGEN | 28104 | |
| QIAprep Spin Miniprep Kit | QIAGEN | 27104 | |
| QIAGEN Plasmid Plus Maxi Kit | QIAGEN | 12963 | |
| PfuUltra High-Fidelity DNA Polymerase | Agilent | 600382 | |
| T-easy vector | Promega | A1360 | |
| Nuclei Lysis Solution | Promega | A7941 | |
| Protein Precipitation Solution | Promega | A7951 | |
| DNA Denaturing Solution | VWR | 351-013-131 | |
| DNA Neutralizing Solution | VWR | 351-014-131 | |
| Ready-To-Go DNA Labeling Beads (-dCTP) | VWR | 27-9240-01 | |
| UltraPure SSC, 20x | Thermo Fisher | 15557036 | |
| UltraPur Phenol:Chloroform:Isoamyl Alcohol (25:24:1, v/v) | Thermo Fisher | 15593031 | |
| G418 | Thermo Fisher | 10131035 | |
| Salmon Sperm DNA Solution | Thermo Fisher | 15632011 | |
| Platinu Taq DNA Polymerase High Fidelity | Thermo Fisher | 11304029 | |
| Not I | Thermo Scientific | ER0592 | |
| Dra I | Thermo Scientific | ER0221 | |
| EcoR I | Thermo Scientific | ER0271 | |
| Ganciclovir | Sigma | G2536 | |
| Whatman TurboBlotter Transfer System, Large Kits | Fisher Scientific | 09-301-188 | |
| [α32P]dCTP | PerkinElmer | NEG013H100UC | |
| ProbeQuan G-50 Micro Columns | GE Healthcare | 28-9034-08 | |
| Hybrisol I Hybridization Solution | Millipore | S4040 | |
| Kodak X-Ray Film | Z&Z Medical | 844 5702 |
Riferimenti
- Gao, G., McMahon, C., Chen, J., Rong, Y. A powerful method combining homologous recombination and site-specific recombination for targeted mutagenesis in Drosophila. Proceedings of the National Academy of Sciences of the United States of America. 105 (37), 13999-14004 (2008).
- Skarnes, W., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474 (7351), 337-342 (2011).
- Vogel, G. Nobel Prizes. A knockout award in medicine. Science. 318 (5848), 178-179 (2007).
- Salsman, J., Dellaire, G. Precision genome editing in the CRISPR era. Biochemistry. Cell Biology. 95 (2), 187-201 (2017).
- Capecchi, M. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nature Reviews Genetics. 6 (6), 507-512 (2005).
- Van, dW. L., Adams, D. J., Bradley, A. Tools for targeted manipulation of the mouse genome. Physiological Genomics. 11 (3), 133-164 (2002).
- Glaser, S., Anastassiadis, K., Stewart, A. F. Current issues in mouse genome engineering. Nature Genetics. 37 (11), 1187(2005).
- Bradley, A., Evans, M., Kaufman, M. H., Robertson, E. Formation of germ-line chimaeras from embryo-derived teratocarcinoma cell lines. Nature. 309 (5965), 255-256 (1984).
- Robertson, E., Bradley, A., Kuehn, M., Evans, M. Germ-line transmission of genes introduced into cultured pluripotential cells by retroviral vector. Nature. 323 (6087), 445-448 (1986).
- Thomas, K. R., Capecchi, M. R. Site-directed mutagenesis by gene targeting in mouse embryo-derived stem cells. Cell. 51 (3), 503-512 (1987).
- Skarnes, W. C., et al. A conditional knockout resource for the genome-wide study of mouse gene function. Nature. 474 (7351), 337(2011).
- Collins, F. S., Rossant, J., Wurst, W. A mouse for all reasons. Cell. 128 (1), 9-13 (2007).
- Poueymirou, W. T., et al. F0 generation mice fully derived from gene-targeted embryonic stem cells allowing immediate phenotypic analyses. Nature Biotechnology. 25 (1), 91-99 (2007).
- Pettitt, S. J., et al. Agouti C57BL/6N embryonic stem cells for mouse genetic resources. Nature Methods. 6 (7), 493-495 (2009).
- Gertsenstein, M., et al. Efficient Generation of Germ Line Transmitting Chimeras from C57BL/6N ES Cells by Aggregation with Outbred Host Embryos. PLoS One. 5 (6), 11260(2012).
- Smithies, O., Gregg, R. G., Boggs, S. S., Koralewski, M. A., Kucherlapati, R. S. Insertion of DNA sequences into the human chromosomal |[beta]|-globin locus by homologous recombination. Nature. 317 (6034), 230-234 (1985).
- Deng, C., Capecchi, M. R. Reexamination of gene targeting frequency as a function of the extent of homology between the targeting vector and the target locus. Molecular & Cellular Biology. 12 (8), 3365(1992).
- Lay, J. M., Friishansen, L., Gillespie, P. J., Samuelson, L. C. Rapid confirmation of gene targeting in embryonic stem cells using two long-range PCR techniques. Transgenic Research. 7 (2), 135-140 (1998).
- Langerak, P., Nygren, A. O. H., Schouten, J. P., Jacobs, H. Rapid and quantitative detection of homologous and non-homologous recombination events using three oligonucleotide MLPA. Nucleic Acids Research. 33 (22), 188(2005).
- Gómezrodríguez, J., et al. Advantages of q-PCR as a method of screening for gene targeting in mammalian cells using conventional and whole BAC-based constructs. Nucleic Acids Research. 36 (18), 117(2008).
- Kim, H. S., Smithies, O. Recombinant fragment assay for gene targeting based on the polymerase chain reaction. Nucleic Acids Research. 16 (18), 8887-8903 (1988).
- Joyner, A. L., Skarnes, W. C., Rossant, J. Production of a mutation in mouse En-2 gene by homologous recombination in embryonic stem cells. Nature. 338 (6211), 153-156 (1989).
- Ma, X., Adelstein, R. S. The role of vertebrate nonmuscle Myosin II in development and human disease. Bioarchitecture. 4 (3), 88-102 (2014).
- Malonek, D. Relationships between the dynamics of cortical blood flow, oxygenation, and volume changes following sensory stimulation. Proceedings of the National Academy of Sciences of the United States of America. 94, (1997).
- Takeda, K., Kishi, H., Ma, X., Yu, Z. X., Adelstein, R. S. Ablation and mutation of nonmuscle myosin heavy chain II-B results in a defect in cardiac myocyte cytokinesis. Circulation Research. 93 (4), 330-337 (2003).
- Conti, M. A., Evenram, S., Liu, C., Yamada, K. M., Adelstein, R. S. Defects in cell adhesion and the visceral endoderm following ablation of nonmuscle myosin heavy chain II-A in mice. Journal of Biological Chemistry. 279 (40), 41263-41266 (2004).
- Wang, A., et al. Nonmuscle myosin II isoform and domain specificity during early mouse development. Proceedings of the National Academy of Sciences of the United States of America. 107 (33), 14645-14650 (2010).
- Zhang, Y., et al. Mouse models of MYH9-related disease: mutations in nonmuscle myosin II-A. Blood. 119 (1), 238(2012).
- Liu, T., et al. Identification and characterization of MYH9 locus for high efficient gene knock-in and stable expression in mouse embryonic stem cells. PLoS One. 13 (2), 0192641(2018).
- Saito, S., Adachi, N. Advances in the Development of Gene-Targeting Vectors to Increase the Efficiency of Genetic Modification. Biological & Pharmaceutical Bulletin. 39 (1), 25-32 (2016).
- Langerak, P., Nygren, A., Schouten, J., Jacobs, H. Rapid and quantitative detection of homologous and non-homologous recombination events using three oligonucleotide MLPA. Nucleic Acids Research. 33 (22), 188(2005).
- Martin, S. L., et al. A single amino acid substitution in ORF1 dramatically decreases L1 retrotransposition and provides insight into nucleic acid chaperone activity. Nucleic Acids Research. 36 (18), 5845-5854 (2008).
- Kamisugi, Y., Cuming, A. C., Cove, D. J. Parameters determining the efficiency of gene targeting in the moss Physcomitrella patens. Nucleic Acids Research. 33 (19), 173(2005).
- Luo, Y., Bolund, L., Sørensen, C. B. Pig gene knockout by rAAV-mediated homologous recombination: comparison of BRCA1 gene knockout efficiency in Yucatan and Göttingen fibroblasts with slightly different target sequences. Transgenic Research. 21 (3), 671-676 (2012).
- Croning, M. D., Fricker, D. G., Komiyama, N. H., Grant, S. G. Automated design of genomic Southern blot probes. BMC Genomics. 11 (1), 74(2010).
- Zimmer, A., Gruss, P. Production of chimaeric mice containing embryonic stem (ES) cells carrying a homoeobox Hox 1.1 allele mutated by homologous recombination. Nature. 338 (6211), 150-153 (1989).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon