Method Article
Testando o papel de plasmídeos multicópia na evolução da resistência aos antibióticos
Neste Artigo
Resumo
Aqui nós apresentamos um método experimental para testar o papel de plasmídeos multicópia na evolução da resistência aos antibióticos.
Resumo
Multicópia plasmídeos são extremamente abundantes em procariontes, mas seu papel na evolução bacteriana continua a ser mal compreendido. Recentemente mostramos que o aumento no número de cópia do gene por célula fornecido por plasmídeos multicópia poderia acelerar a evolução dos genes codificados do plasmídeo. Neste trabalho, apresentamos um sistema experimental para testar a capacidade de plasmídeos multicópia para promover a evolução do gene. Utilizando métodos simples de biologia molecular, construímos um sistema de modelo onde um gene de resistência a antibióticos pode ser inserido em MG1655 de Escherichia coli , o cromossomo ou em um plasmídeo multicópia. Nós usamos uma abordagem de evolução experimental para propagar as diferentes estirpes sob concentrações crescentes de antibióticos e medimos a sobrevivência das populações bacterianas ao longo do tempo. A escolha da molécula do antibiótica e o gene de resistência é para que o gene só pode conferir resistência através da aquisição de mutações. Esta abordagem de "resgate evolutivo" fornece um método simples para testar o potencial de plasmídeos multicópia para promover a aquisição de resistência aos antibióticos. Na próxima etapa do sistema experimental, caracterizam-se as bases moleculares da resistência aos antibióticos. Para identificar as mutações responsável pela aquisição da resistência aos antibióticos, nós usamos a profundo sequenciamento de DNA das amostras obtidas a partir de clones e populações inteiras. Finalmente, para confirmar o papel das mutações no gene em estudo, podemos reconstruí-los em segundo plano parental e teste o fenótipo de resistência das cepas resultantes.
Introdução
Resistência aos antibióticos em bactérias é um problema de saúde1. Em um nível fundamental, a propagação da resistência aos antibióticos em bactérias patogênicas é um exemplo simples da evolução por seleção natural2,3. Simplificando, o uso de antibióticos gera a seleção de cepas resistentes. Dos principais problemas em biologia evolutiva, portanto, é compreender os fatores que influenciam a capacidade das populações bacterianas de desenvolver resistência aos antibióticos. Experimentos de seleção surgiram como uma ferramenta muito poderosa para investigar a biologia evolutiva das bactérias, e este campo produziu incríveis insights sobre uma vasta gama de problemas evolutivos4,5,6. Na evolução experimental, populações bacterianas, iniciadas a partir de uma única estirpe parental em série são passadas sob condições definidas e rigidamente controladas. Algumas das mutações que ocorrem durante o crescimento destas culturas aumentam a aptidão bacteriana, e estas se propagam através das culturas pela seleção natural. Durante o experimento, as amostras das populações são periodicamente criogenicamente preservadas para criar um registro fóssil congelado não evoluindo. Um grande número de abordagens pode ser usado para caracterizar a evolução de populações bacterianas, mas os dois métodos mais comuns são ensaios de aptidão, que medem a capacidade da bactéria evoluiu para competir contra seus antepassados distantes e o sequenciamento do genoma inteiro, que é usado para identificar as alterações genéticas que a adaptação de carro. Na sequência de um trabalho pioneiro por Richard Lenski e colegas7,8, a abordagem padrão na evolução experimental tem sido a desafiar um número relativamente pequeno de populações replicar (tipicamente < 10) com a adaptação a um novo desafio ambiental, tais como novas fontes de carbono, temperatura ou um fago predatório.
Infecções causadas por bactérias resistentes aos antibióticos tornam-se um grande problema quando a resistência é alta o suficiente para que não é possível aumentar as concentrações de antibiótico para níveis letais nos tecidos do pacientes. Os clínicos são, portanto, interessados no que permite que as bactérias desenvolver resistência a altas doses de antibióticos que estão acima desta concentração de antibiótico limiar, o ponto de interrupção clínica. Como estudar isso experimentalmente? Se um pequeno número de populações bacterianas é desafiado com uma alta dose de antibiótico, como em estilo Lenski experimento e, em seguida, o resultado mais provável é que o antibiótico conduzam todas as populações à extinção. Ao mesmo tempo, se a dose de antibiótico que é usado é baixa, abaixo da concentração inibitória mínima (MIC) da estirpe parental, então é improvável que as populações bacterianas irão evoluir clinicamente relevantes níveis de resistência, especialmente se resistência carrega um grande custo. Um compromisso entre estes dois cenários é a utilização de um "resgate evolutivo" experimento9,10,11. Nesta abordagem, um número muito grande de culturas (normalmente > 40) é desafiado com doses de antibióticos que aumentam ao longo do tempo, normalmente, duplicando a concentração de antibiótica cada dia12. A marca registrada desta experiência é que qualquer população que não evolui maior resistência será conduzida à extinção. A maioria das populações que são desafiadas desta forma serão conduzidas extintas, mas uma pequena minoria persistirá evoluindo a altos níveis de resistência. Neste trabalho, mostramos como esse delineamento pode ser usado para investigar a contribuição do plasmídeo para a evolução da resistência.
As bactérias adquirem resistência a antibióticos através de duas rotas principais, mutações cromossômicas e aquisição de elementos genéticos móveis, principalmente de plasmídeos de13. Plasmídeos desempenham um papel chave na evolução da resistência aos antibióticos, porque eles são capazes de transferir genes de resistência entre as bactérias por conjugação14,15. Plasmídeos podem ser divididos em dois grupos de acordo com seu tamanho e Biologia: "pequeno", com o número elevado de cópia por célula bacteriana e "grandes", com baixa copiar número16,17. O papel de grandes plasmídeos na evolução da resistência aos antibióticos tem sido extensivamente documentado porque eles incluem conjugativo plasmídeos, que são os impulsores-chave de divulgação da resistência e multi resistência entre as bactérias15. Pequenos multicópia plasmídeos também são extremamente comuns em bactérias17,18, e eles muitas vezes codificam para genes de resistência aos antibióticos19. No entanto, o papel de plasmídeos multicópia pequenos na evolução da resistência aos antibióticos tem sido estudado em menor grau.
Em um trabalho recente, propusemos que plasmídeos multicópia poderiam acelerar a evolução dos genes que carregam pelo aumento das taxas de mutação do gene devido ao maior número de cópia gene por célula12. Usando um modelo experimental com uma cepa de Escherichia coli MG1655 e o gene de β-lactamase blaTEM-1 foi demonstrado que os plasmídeos multicópia aceleraram a taxa de aparecimento de mutações de TEM-1 confere resistência para a terceira geração Ceftazidima cefalosporina. Estes resultados indicaram que multicópia plasmídeos podem desempenhar um papel importante na evolução da resistência aos antibióticos.
Aqui, apresentamos uma descrição detalhada do método que desenvolvemos para investigar a evolução multicópia mediada por plasmídeo da resistência aos antibióticos. Este método tem três etapas diferentes: primeira, inserção do gene em estudo em um plasmídeo multicópia ou o cromossomo da bactéria anfitrião. Em segundo lugar, use da evolução experimental (resgate evolutiva) para avaliar o potencial das estirpes diferentes para se adaptar à pressão seletiva. E terceiro, determinando a base molecular subjacente a evolução mediada por plasmídeo usando DNA, sequenciamento e reconstruir as mutações suspeitas individualmente no genótipo parental.
Finalmente, embora o protocolo descrito aqui foi projetado para investigar a evolução da resistência aos antibióticos, pode-se argumentar que este método poderia ser geralmente útil para analisar a evolução das inovações adquirida por mutações em qualquer multicopy gene codificado do plasmídeo.
Protocolo
1. construção do sistema Experimental de codificação de Gene de resistência aos antibióticos
Nota: Aqui MG1655 de Escherichia coli foi usado como a destinatário estirpe do gene de resistência aos antibióticos plasmídeo ou cromossomo-codificado. O gene de resistência aos antibióticos é codificado no cromossomo ou de um plasmídeo multicópia em uma cepa isogénicas caso contrário (Figura 1).
-
Inserção do gene da resistência aos antibióticos no λ do fago integração local (attB)20 do cromossomo de MG1655.
- Amplifica 500 bp-long regiões em ambos os lados do site cromossômica attB pelo PCR. Utilizam oligonucleotídeos YbhC-F (5'-CCTGTACCGTACAGAGTAAT-3') e attB-R (5'-GCCCGCCACCCTCCGGGCCGGTATAAAAAAGCAGGCTTCA-3') para a região de homologia esquerdo e attB-F (5'-AGCGCCCTAGCGCCCGCTCCTTATACTAACTTGAGCGAAA-3') e YbhB/R (5'- TGGCGATAATATTTCACCGC-3') para a pessoa certa. Amplificar o gene de resistência blaTEM-1 usando as primeiras demão Tem1-pBAD-F (5'-TGAAGCCTGCTTTTTTATACCGGCCCGGAGGGTGGCGGGC-3') e Tem1-pBAD-R (5'-TTTCGCTCAAGTTAGTATAAGGAGCGGGCGCTAGGGCGCT-3'). Projetar as primeiras demão com aproximadamente 20 bp (na extremidade 5') de sequência mostra a complementaridade com o fragmento que vai ser fundida a.
- Fusível de regiões de homologia para o produto PCR do gene de resistência a antibióticos (incluindo a sua região promotora) usando isotérmico conjunto21, a 50 ° C por 30 min.
- Electroporate MG1655 células contendo plasmídeo pKOBEG.
Nota: pKOBEG é um vetor de termossensível que contém o λ máquinas vermelha, promovendo trocas alélicas baseadas em homologia entre os cromossomos e de produtos PCR22.- Use 1 µ l do produto da etapa 1.1.223 em 40 µ l de células electrocompetent em cubetas de 2mm a 4 ° C e 2,5 kV. Ressuspender as células em 1 mL de caldo LB + arabinose 0,2% para manter a expressão de λ a maquinaria vermelha.
- Transferir o volume total de um tubo de microcentrífuga e incubar h 2 a 30 ° C (temperatura permissiva para pKOBEG) com intensa agitação (200 rpm em um agitador orbital) para permitir a expressão fenotípica dos marcadores de resistência inserido no cromossomo.
- As células em LB ágar contendo o antibiótico adequado para selecionar para o gene de resistência a antibióticos (ampicilina ou carbenicilina 100 mg/l para blaTEM-1) da placa.
- Placa 100 µ l sobre uma placa de Petri e girar para baixo o resto do volume, resuspenda isso em 100 µ l de caldo LB fresco e placa-lo em outra placa de Petri. Incubar durante uma noite a 42 ° C.
Nota: Esta temperatura é não-permissivo para a replicação de pKOBEG. As colônias capazes de crescer nestas condições terá perdido pKOBEG e apresentará o gene de resistência integrado no site attB (para simplificar esta tensão será chamada MG1655::resA daqui em diante). - Teste para a perda de pKOBEG, replicando as colônias de interesse em placas com cloranfenicol (antibiótico contra qual pKOBEG contém um marcador de resistência).
Nota: A falta de crescimento nas placas de cloranfenicol na temperatura permissiva de 30 ° c é indicativa da ausência do plasmídeo. - A fim de verificar a clones, PCR amplificar a construção usando fixadores externos YbhC-externo (5'-TTTGTGACCAGAAGACCGCA-3') e YbhB-externo (5'-CTCATCAGTAACGATCTGCG-3'), verificar através de eletroforese em gel o tamanho correto do amplicon e sequência a purificado de produto para garantir que a sequência do gene inserido é correta.
-
Inserir o gene de resistência aos antibióticos em um plasmídeo multicópia.
Nota: Porque plasmídeos com número de cópia muito alta tendem a impor uma grande redução na aptidão de anfitrião bacteriana, recomendamos a utilização de plasmídeos multicópia com uma origem natural de replicação, como p3655 (pSU18T-pBADgfp2, ColE1-tipo de origem de replicação24). Estes plasmídeos geralmente apresentam um número moderado de cópia de cerca 15-20 cópias por célula.- PCR amplificar o gene de resistência aos antibióticos de escolha (incluindo a região promotora), fosforilar o produto do PCR e ligate-lo ao backbone do plasmídeo PCR amplificados:
- PCR amplificar o gene de resistência aos antibióticos; para blaTEM-1 usar primers Tem1-pBAD-F (5'-TGAAGCCTGCTTTTTTATACCGGCCCGGAGGGTGGCGGGC-3') e Tem1-pBAD-R (5'-TTTCGCTCAAGTTAGTATAAGGAGCGGGCGCTAGGGCGCT-3').
- Fosforilar o produto purificado usando quinase de polinucleotido T4, seguindo as instruções do fabricante.
- PCR amplificar a espinha dorsal do plasmídeo usando uma alta fidelidade polimerase e oligonucleotides pBAD-F (5'-CGTTGATCGGCACGTAAGAG-3') e pBAD-R (5'-AAACGACGGCCAGTGCCAAG-3').
- Ligar os dois fragmentos PCR usando T4 ligase seguindo as orientações do fabricante.
- Electroporate, conforme descrito anteriormente, 40 µ l de células de electrocompetent Escherichia coli DH5-α com um máximo de 1 µ l do produto da ligadura e selecione os antibióticos apropriados para o gene de resistência a antibióticos (ampicilina ou carbenicilina 100 mg/l para blaTEM-1) e a espinha dorsal do plasmídeo.
Nota: Completando LB com arabinosa aqui é desnecessário. - Extraia o plasmídeo utilizando o kit comercial mini-preparação de escolha do leitor:
- Colha 5 mL de uma cultura durante a noite por centrifugação a 12.000 x g, à temperatura ambiente. Ressuspender as células em 250 µ l de solução de ressuspensão e misture bem. Adicionar 250 µ l de solução de Lise e misture bem. Adicione 350 µ l de neutralização e misture bem.
- Transferir o sobrenadante para a coluna de ligação de DNA fornecida com o kit, centrifugar por 1 min e descartar a fluir. Lave duas vezes a coluna com 500 µ l de solução de lavagem, centrifugação durante um minuto e descartando o flowthrough em cada lavagem. Centrifugue a coluna vazia para 1 min adicional remover o tampão de lavagem residual.
- Coloque a coluna em um tubo limpo e adicionar 50 µ l de água ultra-pura para a membrana para Eluir o DNA purificado. Centrífuga para 1-2 min e coletar o fluxo através do que contém o plasmídeo purificado. Sequência de Sanger o gene de interesse no plasmídeo usando primeiras demão de fora a sequência inserida para confirmar que a sequência está correta e que existem sem mutações no gene da resistência antes das experiências de evolução.
- Uma vez que a sequência é verificada, electroporate o plasmídeo na cepa de Escherichia coli MG1655, conforme descrito acima.
Nota: Este produz tensão pRESA/MG1655.
- PCR amplificar o gene de resistência aos antibióticos de escolha (incluindo a região promotora), fosforilar o produto do PCR e ligate-lo ao backbone do plasmídeo PCR amplificados:
2. abordagem de resgate evolutiva para experimentalmente desenvolver resistência aos antibióticos (Figura 1)
- Raia para fora as tensões sob estudo em placas LB: MG1655::resA e MG1655/pRESA além da pressão parental suscetível, MG1655.
Nota: A pRESA do plasmídeo deve ser estável em MG1655, então não há nenhuma necessidade de adicionar antibióticos para as placas LB. Taxas de mutação em e. coli são suficientemente baixas para que uma vez que a construção é verificada não é necessário verificar a sequência do plasmídeo em cada etapa. - Preparar as placas de 96 poços com 200 µ l de LB em cada poço e inocular 48 colónias isoladas de cada estirpe em poços independentes (um prato por estirpe). Incube as placas durante a noite a 37 ° C e 200 rpm. Manter um estoque congelado dessas populações do fundador.
Nota: Para prevenir e controlar para cultura contaminação cruzada nas placas de 96 poços, use um projeto de placa de xadrez pelo intercalante poços inoculados com bactérias livre médio em toda a placa de 96 poços. Use este design da placa durante a toda evolução experimental. - Comece o experimento de resgate evolutivo inoculando µ l 2 de cada poço das placas com as populações do fundador em novas placas de 96 poços, com 198 µ l de LB em cada poço com uma sub-inibitória concentração do antibiótico sob estudo. Para a abordagem evolutiva de resgate começam com ¼ ou 1/8 da concentração inibitória mínima (MIC, anteriormente determinado25) do antibiótico em LB para cada uma das três estirpes e dobrar a concentração do antibiótico diariamente. Use esta abordagem para maximizar as chances de populações para adquirir resistência mutações26. Incube as placas a 37 ° C e 200 rpm por 20 h.
Nota: Medir o OD da noite das populações fundador. Se existem diferenças significativas no OD entre cepas corrigi o inóculo inicial para iniciar o experimento com o mesmo número de células por poço. - Todos os dias, medir o OD de cada população após a cultura da noite e realizar uma transferência das culturas, conforme descrito no ponto 2.3, para novas placas de 96 poços, com o dobro da concentração do antibiótico no dia anterior. Incube as placas 20 h a 37 ° C e 200 rpm.
Nota: O fator de diluição de 1: 100 produz aproximadamente 6-7 gerações por dia. - Em paralelo, propaga o controle das populações de cada estirpe nas mesmas condições, conforme descrito em 2.3 e 2.4, mas na ausência de antibióticos.
Nota: As populações de controle ajudará a discriminar entre mutações decorrentes devido à presença dos antibióticos e mutações gerais ajudando as bactérias para se adaptar às condições experimentais. Paralelas mutações decorrentes na ausência de antibióticos são susceptíveis de estar ajudando as bactérias para se adaptar às condições experimentais e não relacionada com a resistência aos antibióticos. - Controlar o número de populações sobreviventes diariamente através da medição da absorvância no comprimento de onda de 600 nm (OD) das culturas usando um leitor de placa.
Nota: Valores de densidade óptica inferiores 0.1 indicam a extinção da população. Consulte a Figura 2 para obter um exemplo de curvas de sobrevivência. - Manter um estoque congelado de todas as populações periodicamente (a cada 3-5 dias).
- Use testes de log-rank [pacote de "sobrevivência" em RStudio (versão 0.99.486)] para determinar diferenças estatísticas na sobrevivência das populações das estirpes diferentes ao longo do tempo sob crescente concentração de antibióticos12.
Nota: Esta experiência irá determinar se Plasmideos multicópia potenciam a evolução da resistência aos antibióticos para o antibiótico específico e gene em estudo.
3. molecular base da evolução da resistência a antibióticos (Figura 1)
- Realize extrações de DNA totais de um número representativo de populações e clones em cepas e tratamentos. Incluem as linhagens parentais (MG1655, MG1655::resA e MG1655/pRESA) para ser capaz de detectar as mutações acumulando durante o experimento.
Nota: Para obter exemplos de kits de extração de DNA, consulte a tabela de materiais. Cada kit tem diferentes protocolos; Siga as instruções do fabricante. - Quantificar a concentração e qualidade do DNA. Existem diferentes métodos para determinar a concentração e qualidade do DNA. Determine a qualidade medindo os rácios de absorvância 260 nm/280 nm e 260 nm/230 nm. Use uma tintura fluorescente, de ligação a DNA para medir a concentração de DNA, seguindo as instruções do fabricante, e eletroforese em gel de agarose para confirmar isso, lá não é nenhuma degradação de DNA ou RNA contaminação.
- Sequenciamento de DNA profundo utilização de amostras de populações inteiras e individuais de clones para investigar as bases genéticas da resistência aos antibióticos.
Nota: Nós conduzimos todos sequenciamento no centro Wellcome Trust de genética humana, Oxford, UK. Para mais detalhes ver San Millan et al 201612. - Use o gasoduto de breseq 0.26.127,28 para detectar as mutações, usando o modo de polimorfismo para estimar a frequência das mutações nas populações. Compare os diferentes genomas evoluiu para o genoma dos pais para detectar mutações que se acumularam durante o experimento.
- Comparar as mutações acumuladas em paralelo nas populações com aqueles das populações controle evoluiu sob o aumento da concentração de antibióticos para diferenciar entre mutações ajudando as bactérias para se adaptar a condições experimentais ( aqueles encontrados nas populações de controle) e os envolvidos na resistência aos antibióticos (aqueles encontrados exclusivamente em populações submetidas à pressão antibiótica).
Nota: O número de mutações paralelos é geralmente baixo, então diferentes mutações paralelas entre tratamentos podem ser facilmente avaliadas. Aparte as mutações no gene da resistência aos antibióticos sob estudo, é bastante provável que encontre outras mutações associado com resistência aos antibióticos no cromossomo, tais mutações em porinas ou efluxo sistemas12. - Reconstrua as mutações no gene da resistência aos antibióticos na estirpe parental usando a mesma abordagem, como na seção 1 do presente protocolo. Siga os pontos 1.1 e 1.2 do protocolo para introduzir as novas mutações de fundo parental (usando os genes evoluiu como um modelo PCR). Analise os fenótipos de resistência aos antibióticos destas novas construções para confirmar o papel das mutações.
Resultados
Em nosso trabalho anterior, a evolução do β-lactamase gene blaTEM-1 para conferir resistência para a terceira geração cefalosporina Ceftazidima12 foi investigada. Este gene foi selecionado porque, apesar de TEM-1 não confere resistência à Ceftazidima, mutações no blaTEM-1 podem ampliar o leque de atividade de TEM-1 para hidrolisar cefalosporinas como Ceftazidima29. As mutações em enzimas de resistência aos antibióticos, como a β-lactamases ou aminoglicosídeo modificando levando a alterações na oferta de atividade de enzimas são comuns29,30. Este sistema experimental é ideal para explorar a evolução deste tipo de enzimas. Para um relatório detalhado de uma experiência bem sucedida seguindo este protocolo, consulte San Millan et al. 201612.
Aqui, um exemplo dos resultados possíveis deste sistema experimental é apresentado para ilustrar o protocolo (Observe que os dados utilizados para este exemplo não são reais). Para investigar o papel potencial de multicopy plasmídeos na evolução do gene da resistência aos antibióticos sob estudam neste exemplo (chamemos-lhe resA), desenvolvemos o sistema experimental, seção 1 do protocolo acima descrito a seguir. Os experimentos produzem três estirpes: MG1655, MG1655::resA e MG1655/pRESA. A evolução da resistência a duas diferentes ß-lactâmicos (Ceftazidima e meropenem) foi testada seguindo os passos descritos na secção 2 do protocolo. A Figura 2 mostra as curvas de sobrevivência das populações em estudo. Neste exemplo, há um aumento significativo na sobrevivência das populações pertencentes a MG1655/pRESA evoluindo em Ceftazidima comparada de MG1655 ou MG1655::resA (teste log-rank, P< 0,05). Por outro lado, no caso de meropenem, não há nenhuma diferença significativa na sobrevivência das populações pertencentes as diferentes cepas (teste log-rank, P> 0,05). Portanto, estes resultados sugerem que a presença do gene resA em um plasmídeo multicópia potencializa a evolução da resistência a Ceftazidima mas não a meropenem.
Na etapa final do experimento, a base molecular da resistência aos antibióticos é investigada, conforme explicado na seção 3 do protocolo. Primeiro, o sequenciamento de DNA revelará as mutações em resA que poderia ser responsável para o fenótipo de resistência. E em segundo lugar, reconstrução de resA mutações no MG1655 parental (tanto no cromossomo e plasmídeo) irá confirmar ou descartar o seu papel no fenótipo de resistência aos antibióticos.
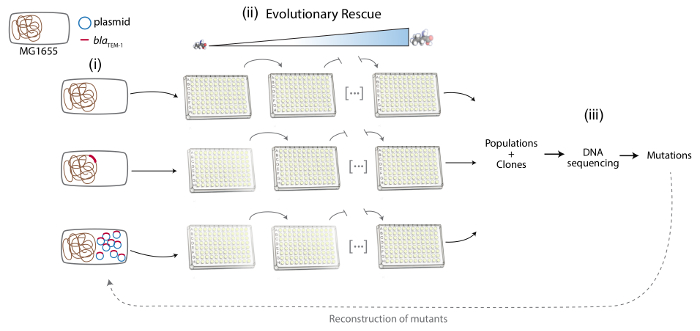
Figura 1 . Representação esquemática das diferentes fases do protocolo. Da esquerda para a direita: (i) a construção do sistema experimental: MG1655, MG1655::resA e MG1655/pRESA. Cromossomo bacteriano é representado em castanho, o plasmídeo em azul e o gene resA em vermelho. (ii) abordagem de resgate evolutiva para experimentalmente desenvolver resistência aos antibióticos: várias populações das diferentes variedades são propagadas sob aumento da concentração do antibiótico. (iii) análise da base molecular da resistência aos antibióticos: sequenciamento de DNA amostras da população evoluída e clones, detecção das mutações de resistência aos antibióticos e reconstrução destas mutações na estirpe parental. Clique aqui para ver uma versão maior desta figura.
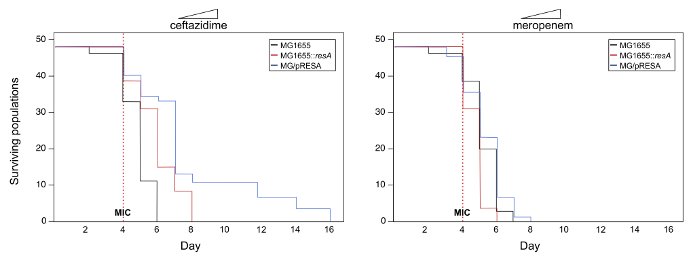
Figura 2. Curvas de sobrevivência com concentrações crescentes de antibióticos. Representação do número de populações viáveis pertencentes a variedades MG1655, MG1655::resAe MG1655/pRESA ao longo do tempo. 48 populações de todas as estirpes foram propagadas sob concentrações crescentes de antibióticos Ceftazidima e meropenem, começando com 1/8 do MIC no dia 1 e duplicação da concentração do antibiótico todos os dias. A linha vertical tracejada vermelha representa o MIC dos antibióticos sob estudo. Observe que, no caso de ceftazidima, existem diferenças significativas na sobrevivência das populações pertencentes a diferentes cepas ao longo do tempo (teste log-rank, P< 0,05). Crucialmente, apenas populações carregando o plasmídeo são capazes de sobreviver até concentrações de alto nível de antibiótico. Por outro lado, no caso de meropenem, não há nenhuma diferença significativa na sobrevivência das populações diferentes ao longo do tempo (teste log-rank, P> 0,05). Clique aqui para ver uma versão maior desta figura.
Discussão
Apresentamos um novo protocolo combinando a biologia molecular, evolução experimental e sequenciamento de DNA profundo visa investigar o papel de plasmídeos multicópia na evolução da resistência aos antibióticos em bactérias. Embora este protocolo combina técnicas de diferentes áreas, todos os métodos necessários para desenvolvê-la são simples e podem ser realizados em um laboratório de microbiologia regular. As etapas mais críticas no protocolo são provavelmente a construção de estirpes de sistema o modelo e a reconstrução das mutações observadas após a evolução experimental (que são executadas usando o mesmo método exato). No entanto, o sistema isotérmico conjunto21, simplifica significativamente este protocolo para que qualquer usuário com um nível intermediário de experiência em biologia molecular pode implementá-lo.
Outro passo crítico do protocolo é a evolução experimental sob concentrações crescentes de antibióticos. Como exemplo, este protocolo começa o experimento com ¼-1/8 do MIC de cepas e então duplicando a concentração de antibiótico todos os dias. No entanto, uma menor taxa de mudança de antibiótica pode aumentar a chance de resgate evolutivo de extinção26. Portanto, a taxa de variação das concentrações é um dos parâmetros que podem ser modificados para promover a evolução da resistência aos antibióticos.
Análise e sequenciamento de DNA também são aspectos-chave do projeto experimental. Os resultados são mais simples quando sequenciamento é realizado em amostras de DNA de populações inteiras e de clones individuais, nos pontos de tempo diferente no experimento. Resultados de populações de sequenciamento revelará gerais diferenças nos perfis de mutação entre tratamentos, bem como varreduras seletivas de mutações benéficas ao longo do tempo e eventos potenciais de interferência clonal. Ao analisar sequências de populações, é melhor filtrar as mutações que nunca ultrapassaram os 10% de frequência em qualquer população. Sequências de clones individuais ajudam a confirmar os resultados obtidos com as populações e, sobretudo, revelam as combinações específicas entre as diferentes mutações observadas a nível da população. Estas associações específicas podem ajudar a descobrir epistatic interações entre mutações, que desempenham um papel crítico na adaptação bacteriana31.
Usando esse método, recentemente mostramos que plasmídeos multicópia aceleram a evolução da resistência aos antibióticos, primeiro aumentando a taxa de aparecimento de novas mutações e, em seguida, amplificando o efeito das mutações devido a dosagem aumentada do gene12 . Portanto, nós desenvolvemos o método como uma ferramenta para investigar a evolução da resistência aos antibióticos, mas pode ter uma gama muito ampla de aplicações. Ou seja, este sistema poderia ser usado para investigar a capacidade de qualquer gene bacteriano para evoluir para uma função nova ou melhorada de forma mais geral. Este sistema poderia ser usado, por exemplo, para testar a capacidade de uma enzima/via metabólica para uso de substratos de carbono novo32. Além disso, poderia ser usado em vez de hypermutators (bactérias com um defeito nos sistemas celulares envolvidos no reparo de ADN incompatibilidade) para investigar a evolução do gene adaptável em bactérias, evitando o preconceito mutacional, introduzido por hypermutators.
Divulgações
Os autores não têm nada para divulgar.
Agradecimentos
Este trabalho foi financiado pelo Instituto de Salud Carlos III (plano Estatal de eu + D + eu 2013-2016): concede CP15-00012, PI16-00860 e CIBER (0053/CB06/02), co-financiado pelo Fundo Europeu Desenvolvimento Regional ' uma maneira de alcançar a Europa ' (FEDER). JAE é suportado pelo programa de atração de talento do governo da região de Madrid (2016-T1/BIO-1105) e a I + D de Excelencia de espanhol Ministerio de Economía, Industria y competitividade (BIO2017-85056-P). ASM é apoiado por uma bolsa de Servet Miguel do Instituto de Salud Carlos III (MS15/00012), co-financiado pelo Fundo Social Europeu "Investir no seu futuro" (FSE) e do FEDER.
Materiais
| Name | Company | Catalog Number | Comments |
| Thermocycler | BioRad | C1000 | |
| Electroporator | BiorRad | 1652660 | |
| Electroporation cuvettes | Sigma-Aldrich | Z706078 | |
| NanoDrop 2000/2000c | Thermo Fisher Scientific | ND-2000 | Determine DNA quality measuring the ratios of absorbance 260nm/280nm and 260nm/230nm |
| Incubator | Memmert | UF1060 | |
| Incubator (shaker) | Cole-Parmer Ltd | SI500 | |
| Electrophoresis power supply | BioRad | 1645070 | Agarose gel electrophoresis |
| Electrophoresis chamber | BioRad | 1704405 | Agarose gel electrophoresis |
| Pippettes | Biohit | 725020, 725050, 725060, 725070 | |
| Multi-channel pippetes | Biohit | 728220, 728230, 728240 | |
| Plate reader Synergy HTX | BioTek | BTS1LF | |
| Inoculating loops | Sigma-Aldrich | I8388 | |
| 96-well plates | Falcon | 351172 | |
| LB | BD Difco | DF0446-17-3 | |
| LB agar | Fisher scientific | BP1425-500 | |
| Phusion Polymerase | Thermo Fisher Scientific | F533S | |
| Gibson Assembly | New England Biolabs | E2611S | |
| Resctriction enzymes | Fermentas FastDigest | ||
| Antibiotics | Sigma-Aldrich | ||
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | Plasmid extraction kit |
| Wizard Genomic DNA Purification Kit | Promega | A1120 | gDNA extraction kit |
| DNeasy Blood & Tissue Kits | Qiagen | 69506 | gDNA extraction kit |
| Electroporation cuvettes | Sigma-Aldrich | Z706078 | |
| Petri dishes | Sigma-Aldrich | D9054 | |
| Cryotubes | ClearLine | 390701 | |
| 96-well plates (-80ºC storage) | Thermo Fisher Scientific | 249945 | |
| QuantiFluor dsDNA System | Promega | E2670 | Quantification of DNA concentartion |
| Agarose | BioRad | 1613100 | Agarose gel electrophoresis |
| 50x TAE buffer | BioRad | 1610743 | Agarose gel electrophoresis |
| T4 Polynucleotide Kinase | Thermo Fisher Scientific | EK0031 | |
| T4 DNA Ligase | Thermo Fisher Scientific | EL0014 |
Referências
- Neill, J. TACKLING DRUG-RESISTANT INFECTIONS GLOBALLY: FINAL REPORT AND RECOMMENDATIONS. Review on Antimicrobal Resistance. , (2016).
- Palmer, A. C., Kishony, R. Understanding, predicting and manipulating the genotypic evolution of antibiotic resistance. Nat Rev Genet. 14 (4), 243-248 (2013).
- MacLean, R. C., Hall, A. R., Perron, G. G., Buckling, A. The population genetics of antibiotic resistance: integrating molecular mechanisms and treatment contexts. Nat Rev Genet. 11 (6), 405-414 (2010).
- Buckling, A., Maclean, R. C., Brockhurst, M. A., Colegrave, N. The Beagle in a bottle. Nature. 457 (7231), 824-829 (2009).
- Barrick, J. E., Lenski, R. E. Genome dynamics during experimental evolution. Nature Reviews Genetics. 14 (12), 827-839 (2013).
- Elena, S. F., Lenski, R. E. Evolution experiments with microorganisms: The dynamics and genetic bases of adaptation. Nature Reviews Genetics. 4 (6), 457-469 (2003).
- Lenski, R. E., Rose, M. R., Simpson, S. C., Tadler, S. C. Long-Term Experimental Evolution in Escherichia coli. I. Adaptation and Divergence During 2,000 Generations. The American Naturalist. 138 (6), 1315-1341 (1991).
- Bennett, A. F., Dao, K. M., Lenski, R. E. Rapid evolution in response to high-temperature selection. Nature. 346 (6279), 79-81 (1990).
- Bell, G. Evolutionary rescue and the limits of adaptation. Philosophical Transactions of the Royal Society B-Biological Sciences. 368 (1610), (2013).
- Bell, G., Gonzalez, A. Adaptation and Evolutionary Rescue in Metapopulations Experiencing Environmental Deterioration. Science. 332 (6035), 1327-1330 (2011).
- Bell, G., Gonzalez, A. Evolutionary rescue can prevent extinction following environmental change. Ecology Letters. 12 (9), 942-948 (2009).
- San Millan, A., Escudero, J. A., Gifford, D. R., Mazel, D., MacLean, R. C. Multicopy plasmids potentiate the evolution of antibiotic resistance in bacteria. Nature Ecology & Evolution. 1, 0010 (2016).
- Alekshun, M. N., Levy, S. B. Molecular mechanisms of antibacterial multidrug resistance. Cell. 128 (6), 1037-1050 (2007).
- Ochman, H., Lawrence, J. G., Groisman, E. A. Lateral gene transfer and the nature of bacterial innovation. Nature. 405 (6784), 299-304 (2000).
- Carattoli, A. Plasmids and the spread of resistance. Int J Med Microbiol. 303 (6-7), 298-304 (2013).
- Smillie, C., Garcillán-Barcia, M. P., Francia, M. V., Rocha, E. P., de la Cruz, F. Mobility of plasmids. Microbiol Mol Biol Rev. 74 (3), 434-452 (2010).
- San Millan, A., Heilbron, K., Maclean, R. C. Positive epistasis between co-infecting plasmids promotes plasmid survival in bacterial populations. ISME J. , (2013).
- Stoesser, N., et al. Evolutionary History of the Global Emergence of the Escherichia coli Epidemic Clone ST131. MBio. 7 (2), (2016).
- San Millan, A., et al. Multiresistance in Pasteurella multocida is mediated by coexistence of small plasmids. Antimicrob Agents Chemother. 53 (8), 3399-3404 (2009).
- Escudero, J. A., et al. Unmasking the ancestral activity of integron integrases reveals a smooth evolutionary transition during functional innovation. Nat Commun. 7, 10937 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc Natl Acad Sci U S A. 97 (12), 6640-6645 (2000).
- Chaveroche, M. K., Ghigo, J. M., d'Enfert, C. A rapid method for efficient gene replacement in the filamentous fungus Aspergillus nidulans. Nucleic Acids Res. 28 (22), 97 (2000).
- Le Roux, F., Binesse, J., Saulnier, D., Mazel, D. Construction of a Vibrio splendidus mutant lacking the metalloprotease gene vsm by use of a novel counterselectable suicide vector. Appl Environ Microbiol. 73 (3), 777-784 (2007).
- . . Performance standards for antimicrobial susceptibility testing; 19th ed. Approved standard M100-S19. , (2009).
- Lindsey, H. A., Gallie, J., Taylor, S., Kerr, B. Evolutionary rescue from extinction is contingent on a lower rate of environmental change. Nature. 494 (7438), 463-467 (2013).
- Deatherage, D. E., Barrick, J. E. Identification of mutations in laboratory-evolved microbes from next-generation sequencing data using breseq. Methods Mol Biol. 1151, 165-188 (2014).
- Barrick, J. E., et al. Identifying structural variation in haploid microbial genomes from short-read resequencing data using breseq. BMC Genomics. 15, 1039 (2014).
- Salverda, M. L., De Visser, J. A., Barlow, M. Natural evolution of TEM-1 β-lactamase: experimental reconstruction and clinical relevance. FEMS Microbiol Rev. 34 (6), 1015-1036 (2010).
- Ramirez, M. S., Tolmasky, M. E. Aminoglycoside modifying enzymes. Drug Resist Updat. 13 (6), 151-171 (2010).
- Jerison, E. R., Desai, M. M. Genomic investigations of evolutionary dynamics and epistasis in microbial evolution experiments. Current Opinion in Genetics & Development. 35, 33-39 (2015).
- Toll-Riera, M., San Millan, A., Wagner, A., MacLean, R. C. The Genomic Basis of Evolutionary Innovation in Pseudomonas aeruginosa. PLoS Genet. 12 (5), 1006005 (2016).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados