Method Article
Prueba el papel de Multicopy plásmidos en la evolución de resistencia a los antibióticos
En este artículo
Resumen
Aquí presentamos un método experimental para probar el papel de multicopy plásmidos en la evolución de resistencia a los antibióticos.
Resumen
Multicopy plásmidos son extremadamente abundantes en procariotas pero su papel en la evolución bacteriana sigue siendo mal entendida. Recientemente demostró que el aumento en el número de copias del gen por celular proporcionado por plásmidos multicopy podría acelerar la evolución de los genes codificados por el plásmido. En este trabajo, presentamos un sistema experimental para poner a prueba la capacidad de multicopy plásmidos para promover la evolución del gene. Utilizando métodos de biología molecular simple, construimos un modelo de sistema donde puede insertarse un gen de resistencia a los antibióticos en MG1655 de Escherichia coli , en el cromosoma o en un plásmido de multicopy. Utilizamos un enfoque experimental de la evolución para propagar variedades cada vez más las concentraciones de antibióticos y medimos la supervivencia de las poblaciones bacterianas en el tiempo. La elección de la molécula del antibiótica y el gen de resistencia es para que el gen sólo puede conferir resistencia a través de la adquisición de mutaciones. Este enfoque de "rescate evolutivo" proporciona un método simple para probar el potencial de multicopy plásmidos para promover la adquisición de resistencia a los antibióticos. En el paso siguiente del sistema experimental, se caracterizan las bases moleculares de resistencia a los antibióticos. Para identificar las mutaciones responsables de la adquisición de resistencia a los antibióticos utilizamos profundo secuenciación del ADN de las muestras obtenidas de clones y poblaciones enteras. Finalmente, para confirmar el papel de las mutaciones en el gen objeto de estudio, reconstruir en el fondo los padres y el fenotipo de resistencia de las cepas resultantes de la prueba.
Introducción
Resistencia a los antibióticos en las bacterias es un problema de salud importante1. A un nivel fundamental, la propagación de la resistencia a los antibióticos en bacterias patógenas es un simple ejemplo de la evolución por selección natural2,3. En pocas palabras, el uso de antibióticos genera selección de cepas resistentes. Un problema fundamental en biología evolutiva, por lo tanto, es entender los factores que influyen en la capacidad de las poblaciones bacterianas para evolucionar la resistencia a los antibióticos. Experimentos de selección se han convertido en una herramienta muy poderosa para investigar la biología evolutiva de bacterias, y este campo ha producido penetraciones increíbles en una amplia gama de problemas evolutivos4,5,6. En la evolución experimental, poblaciones bacterianas desde una única cepa parental son pasadas en serie bajo condiciones definidas y muy controladas. Algunas de las mutaciones que se producen durante el crecimiento de estas culturas aumentan fitness bacteriano, y éstos se propagación a través de las culturas por la selección natural. Durante el experimento, las muestras de las poblaciones se conservan criogénicamente periódicamente para crear un registro fósil congelado no evoluciona. Un gran número de métodos puede utilizarse para caracterizar la evolución de las poblaciones bacterianas, pero los dos métodos más comunes son los ensayos de aptitud, que miden la capacidad de las bacterias evolucionadas para competir contra sus antepasados lejanos y secuenciación del genoma entero, que es utiliza para identificar los cambios genéticos adaptación de la unidad. Siguiendo el trabajo pionero de Richard Lenski y colegas7,8, el enfoque estándar en evolución experimental ha sido desafiar un número relativamente pequeño de las poblaciones de replicar (típicamente < 10) con adaptación a un nuevo desafío ambiental, tales como nuevas fuentes de carbono, temperatura o un depredador fago.
Infecciones causadas por bacterias resistentes a los antibióticos se convierten en un gran problema cuando la resistencia es lo suficientemente alta como para que no es posible aumentar las concentraciones de antibióticos a niveles letales en los tejidos del paciente. Los médicos por lo tanto están interesados en lo que permite que las bacterias desarrollar resistencia a las altas dosis de antibióticos que están por encima de esta concentración antibiótico de umbral, el punto de corte clínico. ¿Cómo estudiar esto experimentalmente? Si un pequeño número de las poblaciones bacterianas se desafió con una alta dosis de antibiótico, como en un estilo de Lenski experimento, el resultado más probable es que el antibiótico todas las poblaciones conduce a la extinción. Al mismo tiempo, si la dosis de antibiótico que se utiliza es baja, por debajo de la concentración inhibitoria mínima (CIM) de la cepa parental, entonces es poco probable que las poblaciones bacterianas evolucionará clínicamente relevantes niveles de resistencia, especialmente si resistencia lleva un costo grande. Un compromiso entre estos dos escenarios es utilizar un "rescate evolutivo" experimento9,10,11. En este enfoque, un gran número de culturas (por lo general > 40) es desafiado con dosis de antibióticos, que aumentan con el tiempo, por lo general duplicando la concentración de antibiótico cada día12. El sello de este experimento es que cualquier población que desarrollan mayor resistencia será conducido a la extinción. Mayoría de las poblaciones que se enfrentan de esta manera se impulsará extinto, pero una pequeña minoría persistirá por altos niveles de resistencia en evolución. En este artículo mostramos cómo este diseño experimental se puede utilizar para investigar la contribución de multicopy plásmido a la evolución de la resistencia.
Las bacterias adquieren resistencia a los antibióticos a través de dos rutas principales, las mutaciones cromosómicas y adquisición de elementos genéticos móviles, mayormente plásmidos13. Plásmidos desempeñan un papel clave en la evolución de resistencia a los antibióticos porque son capaces de transferir genes de resistencia entre las bacterias por conjugación14,15. Plásmidos pueden dividirse en dos grupos según su tamaño y Biología: "pequeño", con número de alta copia por célula bacteriana y "grande", con baja copia número16,17. El papel de grandes plásmidos en la evolución de resistencia a los antibióticos ha sido ampliamente documentado porque incluyen plásmidos conjugativos, que son motores principales de la difusión de la resistencia y la multi resistencia entre bacterias15. Los plásmidos multicopy pequeños también son muy comunes en las bacterias17,18, y a menudo codifican genes de resistencia a los antibióticos19. Sin embargo, se ha estudiado el papel de los plásmidos multicopy pequeños en la evolución de resistencia a antibióticos en menor medida.
En un trabajo reciente, hemos propuesto que los plásmidos multicopy podrían acelerar la evolución de los genes que llevan por aumento de las tasas de mutación de gen debido al número más alto de la copia del gene por célula de12. Utilizando un modelo experimental con e. coli tensión MG1655 y la β-lactamasa gen blaTEM-1 se ha demostrado que plásmidos multicopy aceleraron la tasa de aparición de mutaciones de TEM-1 que confiere resistencia a la tercera generación cefalosporinas ceftazidima. Estos resultados indicaron que los plásmidos multicopy podrían desempeñar un papel importante en la evolución de resistencia a los antibióticos.
Aquí, presentamos una descripción detallada del método que hemos desarrollado para investigar la evolución multicopy mediada por plásmidos de resistencia a los antibióticos. Este método tiene tres etapas: primera, inserción del gen en estudio o en un plásmido de multicopy o en el cromosoma de la bacteria huésped. En segundo lugar, el uso de evolución experimental (rescate evolutivo) para evaluar el potencial de las cepas diferentes para adaptarse a la presión selectiva. Y en tercer lugar, determinar la base molecular subyacente evolución mediada por plásmido con ADN secuenciación y reconstruyendo las mutaciones sospecha individualmente en el genotipo de los padres.
Por último, aunque el protocolo descrito aquí fue diseñado para investigar la evolución de la resistencia a los antibióticos, se puede argumentar que este método podría ser generalmente útil analizar la evolución de las innovaciones adquiridas por las mutaciones en cualquier MultiCopia genes codificados por el plásmido.
Protocolo
1. construcción del sistema Experimental de codificación de genes de resistencia a los antibióticos
Nota: Aquí MG1655 de e. coli fue utilizado como la cepa receptora del gen de resistencia a los antibióticos codificadas por plásmidos o cromosoma. El gen de resistencia a los antibióticos está codificado en el cromosoma o en un plásmido de multicopy en una cepa lo contrario isogénicas (figura 1).
-
Inserción del gen de resistencia a los antibióticos en el λ phage integración sitio (attB)20 del cromosoma de MG1655.
- Amplificar las 500 regiones de bp de largo a ambos lados del sitio cromosómico attB por PCR. Uso de oligonucleótidos YbhC-F (5'-CCTGTACCGTACAGAGTAAT-3') y attB-R (5'-GCCCGCCACCCTCCGGGCCGGTATAAAAAAGCAGGCTTCA-3') de la región de homología izquierda y attB-F (5'-AGCGCCCTAGCGCCCGCTCCTTATACTAACTTGAGCGAAA-3') y YbhB/R (5'- TGGCGATAATATTTCACCGC-3') para el derecho. Amplificar el gen de resistencia blaTEM-1 usando las cartillas Tem1-pBAD-F (5'-TGAAGCCTGCTTTTTTATACCGGCCCGGAGGGTGGCGGGC-3') y Tem1-pBAD-R (5'-TTTCGCTCAAGTTAGTATAAGGAGCGGGCGCTAGGGCGCT-3'). Diseño de primers con aproximadamente 20 bp (en el extremo 5') de la secuencia que muestra la complementariedad para el fragmento que va a ser fundido a.
- Fusible de regiones de homología en el producto de PCR de gen de resistencia a los antibióticos (incluyendo su región promotora) mediante Asamblea isotermo21, a 50 ° C durante 30 minutos.
- Electroporate MG1655 células que contienen plásmido pKOBEG.
Nota: pKOBEG es un vector termosensible que contiene el λ maquinaria roja, promover intercambios alélicos basadas en la homología entre el cromosoma y de los productos PCR22.- Use 1 μl del producto de paso 1.1.223 en 40 μl de células de espectro en cubetas de 2 mm a 4 ° C y 2,5 kV. Resuspender las células en 1 mL de caldo LB + arabinosa 0.2% para mantener la expresión de la λ maquinaria roja.
- Transferir el volumen a un tubo de microcentrífuga e incubar 2 h a 30 ° C (temperatura permisiva para pKOBEG) con intensa agitación (200 rpm en un agitador orbital) para permitir la expresión fenotípica de los marcadores de resistencia insertado en el cromosoma.
- Las células en LB agar que contienen el antibiótico apropiado para seleccionar para el gen de resistencia a los antibióticos (ampicilina o carbenicilina 100 mg/l para blaTEM-1) de la placa.
- Placa de 100 μL en una placa de Petri, girar por el resto del volumen, resuspender en 100 μl de caldo LB fresco y placa sobre otra placa de Petri. Incubar durante una noche a 42 ° C.
Nota: Esta temperatura es no-permisivo para la replicación de pKOBEG. Las colonias capaces de crecer en estas condiciones habrá perdido pKOBEG y presentará el gen de resistencia integrado en el sitio de attB (para simplificar se llamará esta cepa MG1655::resA de aquí en adelante). - Prueba para la pérdida de pKOBEG al replicar las colonias de interés en las placas con cloranfenicol (antibiótico contra que pKOBEG contiene un marcador de resistencia).
Nota: Falta de crecimiento en placas de cloranfenicol a la temperatura permisiva de 30 º c es indicativo de la ausencia del plásmido. - Para verificar clones, PCR amplifican la construcción utilizando cebadores externos YbhC-externo (5'-TTTGTGACCAGAAGACCGCA-3') y YbhB externo (5'-CTCATCAGTAACGATCTGCG-3'), verifique a través de electroforesis en gel del tamaño correcto de los amplicones y la secuencia de la producto purificado para que la secuencia del gen insertado sea correcta.
-
Insertar el gen de resistencia a los antibióticos en un plásmido de multicopy.
Nota: Como plásmidos con número de copias de muy alta tienden a imponer una reducción grande en hospedero bacteriano, recomendamos el uso de multicopy plásmidos con origen de replicación, como p3655 natural (pSU18T-pBADgfp2, ColE1 tipo origen replicación24). Estos plásmidos suelen presentan un número de copia moderada de aproximadamente 15-20 copias por célula.- PCR amplificar el gen de resistencia a los antibióticos de elección (incluyendo la región del promotor), fosforilan el producto PCR y ligar a la espina dorsal del polimerización en cadena-amplificada del plásmido:
- PCR amplifica el gen de resistencia a los antibióticos; para blaTEM-1 utilizar primers Tem1-pBAD-F (5'-TGAAGCCTGCTTTTTTATACCGGCCCGGAGGGTGGCGGGC-3') y Tem1-pBAD-R (5'-TTTCGCTCAAGTTAGTATAAGGAGCGGGCGCTAGGGCGCT-3').
- Fosforilan el producto purificado con la cinasa T4 Polinucleótido, siguiendo las instrucciones del fabricante.
- PCR amplifica la columna vertebral de plásmido con una alta fidelidad de polimerasa y oligonucleótidos pBAD-F (5'-CGTTGATCGGCACGTAAGAG-3') y pBAD-R (5'-AAACGACGGCCAGTGCCAAG-3').
- Ligar ambos fragmentos PCR utilizando T4 ligasa siguiendo las instrucciones del fabricante.
- Electroporate, como se describe anteriormente, 40 μl de células de e. coli DH5-α del espectro con un máximo de 1 μl del producto de la ligadura y selección de los antibióticos apropiados para el gen de resistencia a los antibióticos (ampicilina o carbenicilina 100 mg/l para blaTEM-1) y la columna vertebral del plásmido.
Nota: Complementar LB con arabinosa aquí es innecesario. - Extraiga el plásmido con el kit comercial Mini-prep de la elección:
- Recoger 5 mL de un cultivo durante la noche por centrifugación a 12.000 x g a temperatura ambiente. Resuspender las células en 250 μl de solución de resuspensión y mezclar bien. Añadir 250 μl de solución de lisis y mezclar bien. Añadir 350 μl de la neutralización y mezclar bien.
- Transferir el sobrenadante a la columna de enlace de ADN suministrada con el kit, centrifugue durante 1 min y descartar el flujo a través. Lavar dos veces la columna con 500 μl de solución de lavado, centrifugado durante un minuto y descartar el flujo en cada lavado. Centrifugar 1 min adicional retirar el tampón de lavado residual en la columna vacía.
- Coloque la columna en un tubo limpio y añadir 50 μl de agua ultra pura a la membrana a fin de eluir el ADN purificado. Centrifugar durante 1-2 min y recoge el flujo a través de la contiene el plásmido purificado. Secuencia de Sanger el gen de interés en el plásmido usando las cartillas de fuera de la secuencia insertada para confirmar que la secuencia es correcta y que ningunas mutaciones en el gen de resistencia antes de los experimentos de evolución.
- Una vez verificada la secuencia, electroporate el plásmido en la cepa de e. coli MG1655, como se describe anteriormente.
Nota: Esto produce tensión MG1655/pRESA.
- PCR amplificar el gen de resistencia a los antibióticos de elección (incluyendo la región del promotor), fosforilan el producto PCR y ligar a la espina dorsal del polimerización en cadena-amplificada del plásmido:
2. evolutivo rescate enfoque experimental evolucionar resistencia a los antibióticos (figura 1)
- Raya a las cepas en estudio en placas de LB: MG1655::resA y MG1655/pRESA además de la cepa parental susceptible, MG1655.
Nota: Debe ser estable en MG1655, plásmido pRESA por lo que no es necesario añadir antibióticos a las placas LB. Tasas de mutación en e. coli son lo suficientemente bajas como para que una vez verificada la construcción no es necesario verificar la secuencia del plásmido en cada paso. - Preparar placas de 96 pocillos con 200 μL de LB en cada pozo e inocular 48 colonias aisladas de cada cepa en pozos independientes (una placa por cepa). Incubar las placas durante la noche a 37 ° C y 200 rpm. Mantener un stock congelado de estas poblaciones del fundador.
Nota: Para prevenir y controlar la contaminación del cultivo en placas de 96 pocillos, utilizan un diseño de placa de tablero de ajedrez por intercalando pozos inoculados con medio libre de bacterias a lo largo de la placa de 96 pocillos. Utilizar este diseño de la placa durante la entera evolución experimental. - Comience el rescate evolutivo experimento inoculando 2 μl de cada pocillo de las placas con las poblaciones del fundador en nuevas placas de 96 pocillos con 198 μl de LB en cada pocillo con una concentración subinhibitorias del antibiótico en estudio. Para el enfoque evolutivo rescate comience con 1/4 o 1/8 de la concentración inhibitoria mínima (MIC, determinada previamente25) del antibiótico en libras para cada una de las tres cepas y doble de la concentración del antibiótico diario. Utilizar este enfoque para maximizar las posibilidades de la población para adquirir resistencia mutaciones26. Incubar las placas a 37 ° C y 200 rpm durante 20 h.
Nota: Medir la do durante la noche de las poblaciones del fundador. Si existen diferencias significativas en el OD entre cepas corregir el inóculo inicial para comenzar el experimento con el mismo número de células por pocillo. - Cada día, medir la do de cada población después de la cultura durante la noche y realizar a una transferencia de las culturas, como se describe en 2.3, nuevas placas de 96 pocillos con doble concentración de antibiótico que el día antes. Incubar las placas de 20 h a 37 ° C y 200 rpm.
Nota: El factor de dilución 1: 100 produce aproximadamente 6-7 generaciones por día. - En paralelo, propagar controlar las poblaciones de cada cepa en las mismas condiciones como se describe en 2.3 y 2.4, pero en ausencia de antibióticos.
Nota: Las poblaciones de Control ayudará a discriminar entre las mutaciones que se presentan debido a la presencia de los antibióticos y las mutaciones generales ayudando a las bacterias adaptarse a las condiciones experimentales. Mutaciones paralelas que se presentan en la ausencia de antibióticos son propensos a las bacterias adaptarse a las condiciones experimentales y no relacionados con resistencia a los antibióticos. - Seguimiento del número de las poblaciones sobrevivientes diariamente midiendo la absorbancia a una longitud de onda de 600 nm (OD) de las culturas usando un lector de placas.
Nota: Valores de densidad óptica menor que 0.1 indica la extinción de la población. Vea la figura 2 un ejemplo de curvas de supervivencia. - Mantener un stock congelado de todas las poblaciones periódicamente (cada 3-5 días).
- Utilizar pruebas de log-rank [paquete "supervivencia" en RStudio (versión 0.99.486)] para determinar diferencias estadísticas en la supervivencia de las poblaciones de las diferentes cepas en el tiempo bajo aumento de la concentración de antibióticos12.
Nota: Este experimento determinará si los plásmidos multicopy potencian la evolución de la resistencia a los antibióticos para el antibiótico particular y bajo estudio.
3. molecular base de la evolución de resistencia a los antibióticos (figura 1)
- Realizar extracciones de ADN total de un número representativo de clones y poblaciones a través de tratamientos y variedades. Incluyen las cepas parentales (MG1655, MG1655::resA y MG1655/pRESA) para poder detectar las mutaciones que se acumulan durante el experimento.
Nota: Ejemplos de kits de extracción de ADN consulte la tabla de materiales. Cada kit tiene diferentes protocolos; Siga las instrucciones del fabricante. - Cuantificar la concentración y calidad de ADN. Existen diferentes métodos para determinar la concentración y calidad de ADN. Determinar la calidad de medición de la relación de absorbancia 260/280 nm nm y 260 nm/230 nm. Utilizar un colorante fluorescente, de ADN para medir la concentración de ADN siguiendo las instrucciones del fabricante, y electroforesis del gel de agarosa para confirmar que allí no es ninguna degradación del ADN o RNA contaminación.
- Secuencia de la DNA profunda uso de muestras de poblaciones enteras e individuales clones para investigar la base genética de resistencia a los antibióticos.
Nota: Llevamos a cabo toda la secuencia en el Wellcome Trust Centre for Human Genetics, Oxford, Reino Unido. Para más detalles véase San Millán et al. 201612. - Utilice el breseq 0.26.1 tubería27,28 para detectar las mutaciones, usando el modo de polimorfismo para estimar la frecuencia de mutaciones en las poblaciones. Comparar los diferentes genomas evolucionados al genoma parental para detectar las mutaciones que se han acumulado durante el experimento.
- Comparar las mutaciones acumuladas en paralelo en las poblaciones de control con los de las poblaciones evolucionaron bajo aumento de la concentración de antibióticos para distinguir entre mutaciones ayudando a las bacterias adaptarse a las condiciones experimentales ( ésos encontrados en las poblaciones de control) y los involucrados en la resistencia a los antibióticos (las que se encuentran exclusivamente en las poblaciones sometidas a la presión antibiótica).
Nota: El número de mutaciones paralelas es generalmente bajo, por lo que diferentes mutaciones paralelas entre tratamientos pueden ser fácilmente evaluadas. Aparte de las mutaciones en el gen de resistencia a los antibióticos en estudio es bastante probable encontrar otras mutaciones asociadas con resistencia a los antibióticos en el cromosoma, tales mutaciones en porinas o emanación de sistemas12. - Reconstruir las mutaciones en el gene de resistencia a los antibióticos en la cepa parental utilizando el mismo enfoque que en el apartado 1 del presente Protocolo. Siga los puntos 1.1 y 1.2 del protocolo para introducir las mutaciones nuevas en el fondo los padres (con los genes evolucionados como una plantilla de la polimerización en cadena). Análisis de los fenotipos de resistencia a los antibióticos de estas nuevas construcciones para confirmar el papel de las mutaciones.
Resultados
En nuestro trabajo anterior, se investigó la evolución de las β-lactamasas gen blaTEM-1 hacia que confiere resistencia a la tercera generación cefalosporina ceftazidima12 . Este gen fue seleccionado porque, aunque TEM-1 no confiere resistencia a ceftazidima, mutaciones en blaTEM-1 pueden ampliar el rango de actividad de TEM-1 para hidrolizar las cefalosporinas como ceftazidima29. Las mutaciones en las enzimas de resistencia a los antibióticos tales como β-lactamasas o conduce a cambios en su gama de la actividad de enzimas modificadoras de aminoglucósidos son comunes29,30. Este sistema experimental es ideal para explorar la evolución de este tipo de enzimas. Para un informe detallado de un exitoso experimento siguiendo este protocolo véase San Millán et al. 201612.
Aquí, un ejemplo de los posibles resultados de este sistema experimental se presenta para ilustrar el protocolo (nota que los datos utilizados en este ejemplo no están reales). Para investigar el papel potencial de MultiCopia plásmidos en la evolución del gen de resistencia a los antibióticos en estudian en este ejemplo (vamos a llamarlo resA), desarrollamos el sistema experimental siguiente sección 1 del protocolo descrito anteriormente. Los experimentos producen tres cepas: MG1655, MG1655::resA y MG1655/pRESA. La evolución de la resistencia a dos diferentes ß-lactámicos (ceftazidima y meropenem) fue probada siguiendo los pasos descritos en el artículo 2 del protocolo. La figura 2 muestra las curvas de supervivencia de las poblaciones bajo estudio. En este ejemplo, hay un aumento significativo en la supervivencia de las poblaciones pertenecientes a la evolución de MG1655/pRESA de ceftazidima en comparación con los de MG1655 o MG1655::resA (log-rank test, P< 0.05). Por otra parte, en el caso de meropenem, no hay hubo diferencias significativas en la supervivencia de las poblaciones pertenecientes a las diferentes cepas (log-rank test, P> 0.05). Por lo tanto, estos resultados sugieren que la presencia de gene resA en un plásmido de multicopy potencializa la evolución de la resistencia a ceftazidima pero no a meropenem.
En el paso final del experimento, se investigan las bases moleculares de resistencia a los antibióticos, como se explica en la sección 3 del protocolo. En primer lugar, secuencia de la DNA revelará las mutaciones en resA que podrían ser responsables del fenotipo de resistencia. Y en segundo lugar, reconstrucción de mutaciones de resA en el parental MG1655 (ambos en el cromosoma y plásmido) será confirmar o descartar su papel en el fenotipo de resistencia a los antibióticos.
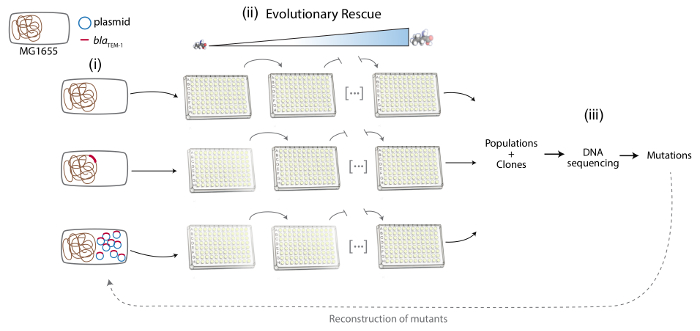
Figura 1 . Representación esquemática de las diferentes fases del Protocolo de. De izquierda a derecha: (i) construcción del sistema experimental: MG1655, MG1655::resA y MG1655/pRESA. Cromosoma bacteriano está representada en marrón, el plásmido en azul y el gene de resA en rojo. (ii) enfoque de rescate evolución evolucionar experimentalmente la resistencia a los antibióticos: varias poblaciones de las diferentes cepas se propagan bajo aumento de la concentración del antibiótico. (iii) análisis de las bases moleculares de resistencia a los antibióticos: secuenciación del ADN de muestras de poblaciones evolucionadas y clones, detección de las mutaciones de resistencia a los antibióticos y la reconstrucción de estas mutaciones en la cepa parental. Haga clic aquí para ver una versión más grande de esta figura.
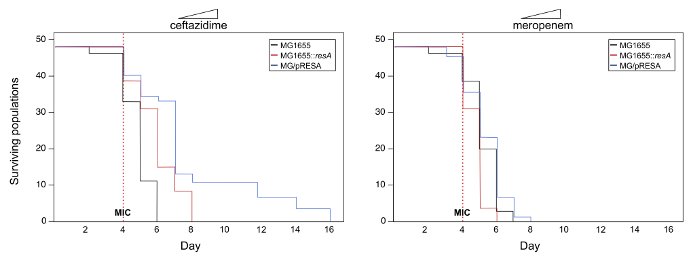
Figura 2. Las curvas de supervivencia con el aumento de las concentraciones de antibióticos. Representación del número de poblaciones viables pertenecientes a cepas MG1655, MG1655::resAy MG1655/pRESA con el tiempo. 48 poblaciones de cada cepa se propagaban cada vez más las concentraciones de antibióticos ceftazidima y meropenem, a partir de 1/8 de la MIC en el día 1 y duplicando la concentración de antibiótica todos los días. La línea vertical discontinua roja representa el MIC de los antibióticos en estudio. Tenga en cuenta que en el caso de ceftazidima existen diferencias significativas en la supervivencia de las poblaciones pertenecientes a las diferentes cepas en el tiempo (log-rank test, P< 0.05). Crucial, sólo las poblaciones llevan el plásmido son capaces de sobrevivir hasta concentraciones de alto nivel de antibiótico. Por otra parte, en el caso de meropenem, no hay hubo diferencias significativas en la supervivencia de las diferentes poblaciones en el tiempo (log-rank test, P> 0.05). Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Presentamos un nuevo protocolo que combina la biología molecular, evolución experimental y profundo secuenciación de ADN diseñado para investigar el papel de multicopy plásmidos en la evolución de la resistencia antibiótica en bacterias. Aunque este protocolo combina técnicas de diferentes ámbitos, todos los métodos necesarios para su desarrollo son simples y pueden ser realizados en un laboratorio de Microbiología regular. Los pasos más críticos en el protocolo son probablemente la construcción de las cepas de sistema del modelo y la reconstrucción de las mutaciones observadas después de la evolución experimental (que se realizan utilizando el mismo método). Sin embargo, el sistema conjunto isotermo21, simplifica significativamente este protocolo para que cualquier usuario con un nivel intermedio de experiencia en biología molecular puede implementarlo.
Otro paso fundamental del protocolo es la evolución experimental bajo aumento de las concentraciones de antibióticos. Por ejemplo, este protocolo comienza el experimento con ¼-1/8 de la CMI de las cepas y luego duplicando la concentración de antibiótico todos los días. Sin embargo, una menor tasa de cambio de antibiótico podría aumentar las posibilidades de rescate evolutivo de extinción26. Por lo tanto, la tasa de cambio de las concentraciones de antibióticos es uno de los parámetros que pueden ser modificados para promover la evolución de resistencia a los antibióticos.
Análisis y secuenciación de ADN son también aspectos clave del diseño experimental. Los resultados son más simples cuando la secuencia se realiza en muestras de ADN de poblaciones enteras y de clones individuales, en momentos diferentes en el experimento. Secuencia resultados de poblaciones revelará diferencias generales en los perfiles de la mutación entre los tratamientos, así como barridos selectivos de mutaciones beneficiosas en el tiempo y posibles eventos de interferencia clonal. Al analizar las secuencias de las poblaciones, es mejor filtrar las mutaciones que nunca superaron el 10% de frecuencia en cualquier población. Secuencias de clones individuales ayudan a confirmar los resultados obtenidos de las poblaciones y, lo más importante, revelan las combinaciones específicas entre las diferentes mutaciones observadas a nivel de población. Estas asociaciones específicas pueden ayudar a descubrir las interacciones epistáticas entre mutaciones, que juegan un papel fundamental en la adaptación bacteriana31.
Usando este método, hemos demostrado recientemente que plásmidos multicopy aceleran la evolución de la resistencia a los antibióticos, primero mediante el aumento de la tasa de aparición de mutaciones y amplificando el efecto de las mutaciones debido a la dosis de gen mayor12 . Por lo tanto, hemos desarrollado el método como una herramienta para investigar la evolución de la resistencia a los antibióticos, pero puede tener una gran variedad de aplicaciones. Es decir, este sistema podría ser utilizado para investigar la capacidad de cualquier gen bacteriano para evolucionar hacia una función nueva o mejorada de una manera más general. Este sistema podría ser utilizado, por ejemplo, para poner a prueba la capacidad de una vía de enzima metabólica de uso de nuevos sustratos de carbono32. También, podría ser utilizado en lugar de hypermutators (bacterias con un defecto en los sistemas celulares implicados en la reparación del ADN coincidencia) para investigar la evolución adaptativa gen en las bacterias, evitando el sesgo mutacional introducido por hypermutators.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue financiado por el Instituto de Salud Carlos III (Plan Estatal de I + D + i 2013-2016): otorga CP15-00012, PI16-00860 y CIBER (CB06/02/0053), cofinanciado por el Fondo Europeo de Desarrollo Regional '' una forma de alcanzar Europa '' (FEDER). JAE es apoyado por el programa de Atracción de talento del gobierno de la región de Madrid (2016-T1/BIO-1105) y el I + D de Excelencia de la Española Ministerio de Economía, Industria y Competitividad (BIO2017-85056-P). ASM es apoyado por una beca de investigación Miguel Servet desde el Instituto de Salud Carlos III (MS15/00012) cofinanciado por el Fondo Social Europeo "Invertir en tu futuro" (FSE) y el FEDER.
Materiales
| Name | Company | Catalog Number | Comments |
| Thermocycler | BioRad | C1000 | |
| Electroporator | BiorRad | 1652660 | |
| Electroporation cuvettes | Sigma-Aldrich | Z706078 | |
| NanoDrop 2000/2000c | Thermo Fisher Scientific | ND-2000 | Determine DNA quality measuring the ratios of absorbance 260nm/280nm and 260nm/230nm |
| Incubator | Memmert | UF1060 | |
| Incubator (shaker) | Cole-Parmer Ltd | SI500 | |
| Electrophoresis power supply | BioRad | 1645070 | Agarose gel electrophoresis |
| Electrophoresis chamber | BioRad | 1704405 | Agarose gel electrophoresis |
| Pippettes | Biohit | 725020, 725050, 725060, 725070 | |
| Multi-channel pippetes | Biohit | 728220, 728230, 728240 | |
| Plate reader Synergy HTX | BioTek | BTS1LF | |
| Inoculating loops | Sigma-Aldrich | I8388 | |
| 96-well plates | Falcon | 351172 | |
| LB | BD Difco | DF0446-17-3 | |
| LB agar | Fisher scientific | BP1425-500 | |
| Phusion Polymerase | Thermo Fisher Scientific | F533S | |
| Gibson Assembly | New England Biolabs | E2611S | |
| Resctriction enzymes | Fermentas FastDigest | ||
| Antibiotics | Sigma-Aldrich | ||
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | Plasmid extraction kit |
| Wizard Genomic DNA Purification Kit | Promega | A1120 | gDNA extraction kit |
| DNeasy Blood & Tissue Kits | Qiagen | 69506 | gDNA extraction kit |
| Electroporation cuvettes | Sigma-Aldrich | Z706078 | |
| Petri dishes | Sigma-Aldrich | D9054 | |
| Cryotubes | ClearLine | 390701 | |
| 96-well plates (-80ºC storage) | Thermo Fisher Scientific | 249945 | |
| QuantiFluor dsDNA System | Promega | E2670 | Quantification of DNA concentartion |
| Agarose | BioRad | 1613100 | Agarose gel electrophoresis |
| 50x TAE buffer | BioRad | 1610743 | Agarose gel electrophoresis |
| T4 Polynucleotide Kinase | Thermo Fisher Scientific | EK0031 | |
| T4 DNA Ligase | Thermo Fisher Scientific | EL0014 |
Referencias
- Neill, J. TACKLING DRUG-RESISTANT INFECTIONS GLOBALLY: FINAL REPORT AND RECOMMENDATIONS. Review on Antimicrobal Resistance. , (2016).
- Palmer, A. C., Kishony, R. Understanding, predicting and manipulating the genotypic evolution of antibiotic resistance. Nat Rev Genet. 14 (4), 243-248 (2013).
- MacLean, R. C., Hall, A. R., Perron, G. G., Buckling, A. The population genetics of antibiotic resistance: integrating molecular mechanisms and treatment contexts. Nat Rev Genet. 11 (6), 405-414 (2010).
- Buckling, A., Maclean, R. C., Brockhurst, M. A., Colegrave, N. The Beagle in a bottle. Nature. 457 (7231), 824-829 (2009).
- Barrick, J. E., Lenski, R. E. Genome dynamics during experimental evolution. Nature Reviews Genetics. 14 (12), 827-839 (2013).
- Elena, S. F., Lenski, R. E. Evolution experiments with microorganisms: The dynamics and genetic bases of adaptation. Nature Reviews Genetics. 4 (6), 457-469 (2003).
- Lenski, R. E., Rose, M. R., Simpson, S. C., Tadler, S. C. Long-Term Experimental Evolution in Escherichia coli. I. Adaptation and Divergence During 2,000 Generations. The American Naturalist. 138 (6), 1315-1341 (1991).
- Bennett, A. F., Dao, K. M., Lenski, R. E. Rapid evolution in response to high-temperature selection. Nature. 346 (6279), 79-81 (1990).
- Bell, G. Evolutionary rescue and the limits of adaptation. Philosophical Transactions of the Royal Society B-Biological Sciences. 368 (1610), (2013).
- Bell, G., Gonzalez, A. Adaptation and Evolutionary Rescue in Metapopulations Experiencing Environmental Deterioration. Science. 332 (6035), 1327-1330 (2011).
- Bell, G., Gonzalez, A. Evolutionary rescue can prevent extinction following environmental change. Ecology Letters. 12 (9), 942-948 (2009).
- San Millan, A., Escudero, J. A., Gifford, D. R., Mazel, D., MacLean, R. C. Multicopy plasmids potentiate the evolution of antibiotic resistance in bacteria. Nature Ecology & Evolution. 1, 0010 (2016).
- Alekshun, M. N., Levy, S. B. Molecular mechanisms of antibacterial multidrug resistance. Cell. 128 (6), 1037-1050 (2007).
- Ochman, H., Lawrence, J. G., Groisman, E. A. Lateral gene transfer and the nature of bacterial innovation. Nature. 405 (6784), 299-304 (2000).
- Carattoli, A. Plasmids and the spread of resistance. Int J Med Microbiol. 303 (6-7), 298-304 (2013).
- Smillie, C., Garcillán-Barcia, M. P., Francia, M. V., Rocha, E. P., de la Cruz, F. Mobility of plasmids. Microbiol Mol Biol Rev. 74 (3), 434-452 (2010).
- San Millan, A., Heilbron, K., Maclean, R. C. Positive epistasis between co-infecting plasmids promotes plasmid survival in bacterial populations. ISME J. , (2013).
- Stoesser, N., et al. Evolutionary History of the Global Emergence of the Escherichia coli Epidemic Clone ST131. MBio. 7 (2), (2016).
- San Millan, A., et al. Multiresistance in Pasteurella multocida is mediated by coexistence of small plasmids. Antimicrob Agents Chemother. 53 (8), 3399-3404 (2009).
- Escudero, J. A., et al. Unmasking the ancestral activity of integron integrases reveals a smooth evolutionary transition during functional innovation. Nat Commun. 7, 10937 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc Natl Acad Sci U S A. 97 (12), 6640-6645 (2000).
- Chaveroche, M. K., Ghigo, J. M., d'Enfert, C. A rapid method for efficient gene replacement in the filamentous fungus Aspergillus nidulans. Nucleic Acids Res. 28 (22), 97 (2000).
- Le Roux, F., Binesse, J., Saulnier, D., Mazel, D. Construction of a Vibrio splendidus mutant lacking the metalloprotease gene vsm by use of a novel counterselectable suicide vector. Appl Environ Microbiol. 73 (3), 777-784 (2007).
- . . Performance standards for antimicrobial susceptibility testing; 19th ed. Approved standard M100-S19. , (2009).
- Lindsey, H. A., Gallie, J., Taylor, S., Kerr, B. Evolutionary rescue from extinction is contingent on a lower rate of environmental change. Nature. 494 (7438), 463-467 (2013).
- Deatherage, D. E., Barrick, J. E. Identification of mutations in laboratory-evolved microbes from next-generation sequencing data using breseq. Methods Mol Biol. 1151, 165-188 (2014).
- Barrick, J. E., et al. Identifying structural variation in haploid microbial genomes from short-read resequencing data using breseq. BMC Genomics. 15, 1039 (2014).
- Salverda, M. L., De Visser, J. A., Barlow, M. Natural evolution of TEM-1 β-lactamase: experimental reconstruction and clinical relevance. FEMS Microbiol Rev. 34 (6), 1015-1036 (2010).
- Ramirez, M. S., Tolmasky, M. E. Aminoglycoside modifying enzymes. Drug Resist Updat. 13 (6), 151-171 (2010).
- Jerison, E. R., Desai, M. M. Genomic investigations of evolutionary dynamics and epistasis in microbial evolution experiments. Current Opinion in Genetics & Development. 35, 33-39 (2015).
- Toll-Riera, M., San Millan, A., Wagner, A., MacLean, R. C. The Genomic Basis of Evolutionary Innovation in Pseudomonas aeruginosa. PLoS Genet. 12 (5), 1006005 (2016).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados