Method Article
Analyse le rôle des plasmides multi-copies dans l’évolution de la résistance aux antibiotique
Dans cet article
Résumé
Nous présentons ici une méthode expérimentale pour évaluer le rôle des plasmides multi-copies dans l’évolution de la résistance aux antibiotique.
Résumé
Multicopie plasmides sont extrêmement abondants chez les procaryotes, mais leur rôle dans l’évolution bactérienne reste mal comprise. Récemment, nous avons montré que l’augmentation en nombre de copies du gène par cellule fournie par plasmides multi-copies pourrait accélérer l’évolution des gènes codés plasmide. Dans ce travail, nous présentons un système expérimental pour tester la capacité de Multicopie plasmides à promouvoir l’évolution des gènes. À l’aide de méthodes simples de biologie moléculaire, nous avons construit un modèle de système où un gène de résistance aux antibiotiques peut être inséré dans Escherichia coli MG1655, dans le chromosome ou sur un plasmide Multicopie. Nous utilisons une approche de l’évolution expérimentale pour propager les différentes souches au titre de l’augmentation des concentrations d’antibiotiques et nous mesurons la survie des populations bactériennes au fil du temps. Le choix de la molécule antibiotique et le gène de résistance est donc que le gène peut seulement conférer résistance grâce à l’acquisition de mutations. Cette approche « évolutionniste al Rescate » fournit une méthode simple pour tester le potentiel des plasmides multi-copies pour favoriser l’acquisition de la résistance aux antibiotique. Dans l’étape suivante du système expérimental, les bases moléculaires de résistance aux antibiotique sont caractérisés. Pour identifier les mutations responsables de l’acquisition de la résistance aux antibiotique nous utilisons profond séquençage de l’ADN des échantillons obtenus à partir de clones et de populations entières. Enfin, pour confirmer le rôle des mutations du gène à l’étude, les reconstituer en arrière-plan parental et tester le phénotype de résistance des souches qui en résulte.
Introduction
La résistance aux antibiotique chez les bactéries est un problème majeur de santé1. À un niveau fondamental, la propagation de la résistance aux antibiotique chez les bactéries pathogènes est un exemple simple de l’évolution par sélection naturelle2,3. Autrement dit, l’utilisation d’antibiotiques génère de sélection de souches résistantes. Un problème majeur en biologie de l’évolution, est donc de comprendre les facteurs qui influencent la capacité des populations bactériennes à évoluer de la résistance aux antibiotiques. Expériences de sélection ont émergé comme un outil très puissant pour étudier la biologie de l’évolution des bactéries, et ce domaine a produit un aperçu incroyable un large éventail de problèmes évolutifs4,5,6. En évolution expérimentale, les populations bactériennes effectuées à partir d’une seule souche parentale sont en série repiquées dans des conditions définies et étroitement contrôlées. Certaines mutations qui se produisent au cours de la croissance de ces cultures augmentent bactérienne remise en forme, et ceux-ci se propager à travers les cultures par la sélection naturelle. Pendant l’expérience, les échantillons des populations sont conservés périodiquement cryogénie pour créer un enregistrement fossile congelé non en évolution. Un grand nombre d’approches peut être utilisé pour caractériser l’évolution des populations bactériennes, mais les deux méthodes les plus courantes sont les essais de remise en forme, qui mesurent la capacité des bactéries évolués de rivaliser contre leurs lointains ancêtres et le séquençage du génome entier, qui est permet d’identifier les changements génétiques que l’adaptation en voiture. Après les travaux pionniers de Richard Lenski et collègues7,8, l’approche standard en évolution expérimentale a été de contester un nombre relativement faible des populations répétées (typiquement < 10) à s’adapter à un nouveau défi environnemental, telles que de nouvelles sources de carbone, la température ou un phage prédatrice.
Les infections causées par des bactéries résistantes aux antibiotiques deviennent un gros problème quand la résistance est suffisamment élevée pour qu’il n’est pas possible d’augmenter les concentrations d’antibiotique à des niveaux mortels dans les tissus du patient. Les cliniciens sont donc intéressés par ce qui permet aux bactéries d’évoluer résistance à de fortes doses d’antibiotiques qui sont au-dessus de ce seuil de concentration antibiotique, le point d’arrêt clinique. Comment étudier ceci expérimentalement ? Si un petit nombre de populations bactériennes est mis au défi avec une forte dose d’antibiotique, comme dans un style Lenski experiment, alors le résultat le plus probable est que l’antibiotique conduira toutes les populations à l’extinction. Dans le même temps, si la dose d’antibiotique qui est utilisé est basse, inférieure à la concentration minimale inhibitrice (CMI) de la souche parentale, puis il est peu probable que les populations bactériennes évoluera cliniquement pertinents niveaux de résistance, surtout si résistance entraînera un coût important. Un compromis entre ces deux scénarios consiste à utiliser un « sauvetage évolutive » expérience9,10,11. Dans cette approche, un très grand nombre de cultures (typiquement > 40) doit relever le défi avec des doses d’antibiotiques qui augmentent au fil du temps, généralement en doublant la concentration d’antibiotique chaque jour12. La principale caractéristique de cette expérience est que toute la population qui n’évolue pas de résistance accrue est conduite à l’extinction. La plupart des populations qui sont mis au défi de cette manière seront pilotées disparues, mais une petite minorité sera présent en évoluant à des niveaux élevés de résistance. Dans cet article, nous montrons comment cette conception expérimentale peut être utilisée pour étudier la contribution de plasmide Multicopie à l’évolution de la résistance.
Les bactéries acquièrent la résistance aux antibiotiques par le biais de deux voies principales, mutations chromosomiques et d’acquisition d’éléments génétiques mobiles, pour la plupart des plasmides13. Plasmides jouent un rôle clé dans l’évolution de la résistance aux antibiotique, parce qu’ils sont en mesure de transférer des gènes de résistance entre bactéries par conjugaison14,15. Plasmides peuvent être divisés en deux groupes selon leur taille et de la biologie : « petit », avec le nombre élevé de copies par cellule bactérienne et les « grands », avec faible copie numéro16,17. Le rôle des grands plasmides dans l’évolution de la résistance aux antibiotique a été largement documenté parce qu’elles incluent les plasmides conjugatifs, qui sont les principaux moteurs de la diffusion de la résistance et la résistance chez les bactéries15multi. Petits plasmides multi-copies sont également extrêmement fréquents chez les bactéries17,18, et ils codent souvent pour de gènes de résistance aux antibiotiques19. Cependant, le rôle des petits plasmides multi-copies dans l’évolution de la résistance aux antibiotique a été étudié dans une moindre mesure.
Dans un ouvrage récent, nous avons proposé que les plasmides multi-copies pourraient accélérer l’évolution des gènes qu’ils transportent en augmentant le taux de mutation de gène en raison du nombre de copie de gène supérieur par cellule12. En utilisant un modèle expérimental avec la souche e. coli MG1655 et la β-lactamase gène blaTEM-1 , il a été démontré que les plasmides multi-copies accélèrent le taux d’apparition de TEM-1 mutations conférant la résistance à la troisième génération céphalosporines ceftazidime. Ces résultats indiquent que les plasmides multi-copies pourraient jouer un rôle important dans l’évolution de la résistance aux antibiotique.
Nous présentons ici une description détaillée de la méthode que nous avons mis au point pour analyser l’évolution de la médiation plasmidique multicopie de résistance aux antibiotique. Cette méthode a trois différentes étapes : tout d’abord, insertion du gène à l’étude dans un plasmide Multicopie ou le chromosome de la bactérie hôte. En second lieu, utiliser des évolution expérimentale (sauvetage évolutive) pour évaluer le potentiel des souches différentes pour s’adapter à la pression de sélection. Et Troisièmement, déterminer les bases moléculaires qui sous-tendent la médiation plasmidique evolution en utilisant l’ADN, séquençage et la reconstruction de ces mutations présumées individuellement dans le génotype parental.
Enfin, bien que le protocole décrit ici a été conçu pour analyser l’évolution de la résistance aux antibiotique, on peut affirmer que cette méthode pourrait être généralement utile pour analyser l’évolution des innovations acquis par des mutations dans n’importe quel multicopies gène plasmidique codée.
Protocole
1. construction du système expérimental codant le gène de résistance aux antibiotiques
Remarque : Ici MG1655 d’e. coli a été utilisé comme la souche réceptrice du gène plasmidique ou chromosomique-codé la résistance aux antibiotiques. Le gène de résistance aux antibiotiques est encodé dans le chromosome ou un plasmide Multicopie dans une souche sinon isogénique (Figure 1).
-
Insertion du gène de résistance aux antibiotiques dans le λ phage intégration site (attB)20 du chromosome de MG1655.
- Amplifier 500 PB régions aux deux extrémités du site chromosomique attB par PCR. Utilisation des oligonucléotides YbhC-F (5'-CCTGTACCGTACAGAGTAAT-3') et attB-R (5'-GCCCGCCACCCTCCGGGCCGGTATAAAAAAGCAGGCTTCA-3') pour la région d’homologie gauche et attB-F (5'-AGCGCCCTAGCGCCCGCTCCTTATACTAACTTGAGCGAAA-3') et YbhB/R (5'- TGGCGATAATATTTCACCGC-3') pour celui de droite. Amplifier le gène de résistance blaTEM-1 en utilisant des amorces Tem1-pBAD-F (5'-TGAAGCCTGCTTTTTTATACCGGCCCGGAGGGTGGCGGGC-3') et Tem1-pBAD-R (5'-TTTCGCTCAAGTTAGTATAAGGAGCGGGCGCTAGGGCGCT-3'). Concevoir des amorces avec environ 20 bp (à l’extrémité 5') de la séquence montrant la complémentarité au fragment qui va être fusionné à.
- Fusible de régions d’homologie pour le produit de PCR de gène de résistance aux antibiotiques (y compris la région du promoteur) à l’aide de montage isotherme21, à 50 ° C pendant 30 min.
- Electroporate MG1655 pKOBEG de plasmide contenant les cellules.
Remarque : pKOBEG est un vecteur de thermosensible qui contient le λ machines rouges, favorisant les échanges alléliques axée sur l’homologie entre les chromosomes et de produits PCR22.- Utiliser 1 µL du produit de l’étape 1.1.223 40 µL de cellules electrocompetent dans des cuvettes de 2 mm à 4 ° C et 2,5 kV. Remettre les cellules dans 1 mL de bouillon LB + 0,2 % arabinose pour maintenir l’expression de la λ machines rouges.
- Transférer le volume total d’un tube à centrifuger et incuber 2 h à 30 ° C (température permissive pour pKOBEG) avec intense agitation (200 tr/min dans un agitateur orbital) afin de permettre l’expression phénotypique des marqueurs résistance insérée dans le chromosome.
- Plaque de cellules de LB agar contenant l’antibiotique approprié pour sélectionner pour le gène de résistance aux antibiotiques (ampicilline ou carbénicilline à 100 mg/l pour blaTEM-1).
- Plaque 100 µL sur une boîte de Pétri et filer vers le bas le reste du volume, il resuspendre dans 100 µL de frais bouillon LB et il la plaque sur une autre boîte de Pétri. Incuber une nuit à 42 ° C.
Remarque : Cette température est non permissif pour la réplication de pKOBEG. Les colonies capables de se développer dans ces conditions auront perdu pKOBEG et présentera le gène de résistance intégré dans le site attB (pour plus de simplicité, cette souche s’appellera MG1655 ::resA partir d’ici). - Test pour la perte de pKOBEG en multipliant les colonies d’intérêt dans des boîtes avec chloramphénicol (l’antibiotique contre laquelle pKOBEG contient un marqueur de résistance).
Remarque : Manque de croissance dans des plats de chloramphénicol à la température permissive de 30 ºC est révélateur de l’absence du plasmide. - Afin de vérifier les clones, PCR amplifier la construction utilisant les amorces externes YbhC-Extern (5'-TTTGTGACCAGAAGACCGCA-3') et YbhB-Extern (5'-CTCATCAGTAACGATCTGCG-3'), vérifier par électrophorèse sur gel de la taille correcte de l’amplicon et la séquence des produit purifié pour s’assurer que la séquence du gène inséré est correcte.
-
Insérer le gène de résistance aux antibiotiques dans un plasmide Multicopie.
Remarque : Parce que les plasmides avec nombre de copies de très haute tendent à imposer une forte réduction du fitness hôte bactérien, nous recommandons l’utilisation des plasmides multi-copies avec une origine naturelle de la réplication, comme p3655 (pSU18T-pBADgfp2, ColE1 type origine de réplication24). Ces plasmides présentent généralement un nombre de copies modérée d’environ 15-20 copies par cellule.- PCR amplifier le gène de résistance aux antibiotiques de choix (y compris la région du promoteur), phosphoryler le produit PCR et il ligaturer au backbone amplifié par PCR du plasmide :
- PCR amplification du gène de résistance aux antibiotiques ; pour blaTEM-1 utiliser les amorces Tem1-pBAD-F (5'-TGAAGCCTGCTTTTTTATACCGGCCCGGAGGGTGGCGGGC-3') et Tem1-pBAD-R (5'-TTTCGCTCAAGTTAGTATAAGGAGCGGGCGCTAGGGCGCT-3').
- Phosphoryler le produit purifié à l’aide de T4 polynucléotide kinase, suivant les instructions du fabricant.
- PCR amplification le squelette plasmidique utilisant une haute fidélité polymérase et oligonucléotides pBAD-F (5'-CGTTGATCGGCACGTAAGAG-3') et pBAD-R (5'-AAACGACGGCCAGTGCCAAG-3').
- Ligaturer les deux fragments PCR utilisant T4-ligase suite aux directives du fabricant.
- Electroporate, comme décrit précédemment, 40 µL de cellules electrocompetent e. coli DH5-α, avec un maximum de 1 µL du produit de ligation et select sur les antibiotiques appropriés pour le gène de résistance aux antibiotiques (ampicilline ou carbénicilline à 100 mg/L pour blaTEM-1) et le squelette plasmidique.
Remarque : Complétant LB avec arabinose ici n’est pas nécessaire. - Extraire le plasmide grâce au kit commercial mini-préparent des choix du lecteur :
- Récolter 5 mL d’une culture au jour le jour par centrifugation à 12 000 x g à la température ambiante. Remettre en suspension les cellules 250 µL de solution de resuspension et bien mélanger. Ajouter 250 µL de solution de lyse et bien mélanger. Ajouter 350 µL de neutralisation et bien mélanger.
- Transvaser le surnageant dans la colonne de liaison de l’ADN fournie avec le kit, centrifuger pendant 1 min et jeter le débit à travers. Laver deux fois la colonne avec 500 µL de solution de lavage, centrifugation pendant une minute et en jetant la redistribution à chaque lavage. Centrifuger la colonne vide pendant 1 minute supplémentaire retirer le tampon de lavage résiduelle.
- Placer la colonne dans un tube propre et ajouter 50 µL d’eau ultra pure à la membrane pour éluer l’ADN purifié. Centrifuger pendant 1-2 min et recueillir le débit à travers qui contient le plasmide purifié. Séquence du gène d’intérêt dans le plasmide en utilisant des amorces d’en dehors de la séquence insérée pour confirmer que la séquence est correcte et qu’il existe aucune mutation dans le gène de résistance avant les expériences de l’évolution de Sanger.
- Une fois que la séquence est vérifiée, electroporate le plasmide dans la souche e. coli MG1655, tel que décrit ci-dessus.
Remarque : Ceci donne souche MG1655/pRESA.
- PCR amplifier le gène de résistance aux antibiotiques de choix (y compris la région du promoteur), phosphoryler le produit PCR et il ligaturer au backbone amplifié par PCR du plasmide :
2. évolution de sauvetage approche d’évoluer expérimentalement la résistance aux antibiotique (Figure 1)
- Rayure sur les souches étudiées sur des plaques LB : MG1655 ::resA et MG1655/pRESA ainsi que la souche parentale sensible, MG1655.
Remarque : PRESA de plasmide devrait être stable en MG1655, il n’est pas nécessaire d’ajouter des antibiotiques aux plaques LB. Taux de mutation chez e. coli sont assez bas, une fois vérifiée la construction n’est pas nécessaire de vérifier la séquence plasmide à chaque étape. - Préparer les plaques à 96 puits avec 200 µL de LB à chaque puits et inoculer 48 colonies isolées de chaque souche dans les puits indépendants (une plaque par souche). Incuber les boîtes pendant une nuit à 37 ° C et 200 tr/min. Maintenir un stock congelé de ces populations de fondateur.
Remarque : Pour prévenir et contrôler de culture contamination croisée dans les plaques de 96 puits, utilisent une conception de plaque de damier en intercalant des puits inoculés avec un milieu sans bactéries tout au long de la plaque à 96 puits. Utilisez cette conception de la plaque au cours de l’évolution expérimentale ensemble. - Commencez l’expérience évolutionnaire sauvetage par inoculation 2 µL de chaque puits des plaques avec les populations de fondateur de nouvelles plaques de 96 puits avec 198 µL de LB à chaque puits avec une Sub-inhibitrice concentration de l’antibiotique à l’étude. Pour l’approche évolutionniste de sauvetage commencez avec ¼ ou 1/8 de la concentration minimale inhibitrice (CMI, déterminé précédemment25) de cet antibiotique dans LB pour chacune des trois souches et doubler la concentration de l’antibiotique tous les jours. Cette approche permet de maximiser les chances des populations en vue d’acquérir des mutations de résistance26. Incuber les plaques à 37 ° C et 200 tr/min pendant 20 h.
Remarque : Mesurer la nuit OD les populations de fondateur. S’il y a des différences significatives dans l’OD parmi les souches de corrigent l’inoculum initial pour démarrer l’expérience avec le même nombre de cellules par puits. - Chaque jour, mesurez le diamètre extérieur de chaque population après une culture et effectuez un transfert des cultures, tel que décrit au point 2.3, de nouvelles plaques de 96 puits avec double de la concentration de l’antibiotique que la veille. Incuber les boîtes de 20 h à 37 ° C et 200 tr/min.
Remarque : Le facteur de dilution au 1/100 produit environ 6-7 générations par jour. - En parallèle, propager le contrôle des populations de chaque souche dans les mêmes conditions comme décrit dans la 2.3 et 2.4, mais en l’absence d’antibiotiques.
Remarque : Contrôle des populations permettra de distinguer les mutations résultant de la présence des antibiotiques et des mutations générales aident les bactéries de s’adapter à des conditions expérimentales. Mutations parallèles résultant en l’absence d’antibiotiques sont susceptibles de pouvoir aider les bactéries de s’adapter à des conditions expérimentales et non liés à la résistance aux antibiotique. - Suivre le nombre de populations survivantes chaque jour en mesurant l’absorbance à une longueur d’onde de 600 nm (OD) des cultures à l’aide d’un lecteur.
Remarque : Des valeurs de densité optique inférieures 0,1 indiquent l’extinction de la population. Voir la Figure 2 pour obtenir un exemple de courbes de survie. - Maintenir un stock congelé de toutes les populations périodiquement (tous les 3-5 jours).
- Utilisez les tests de Mantel-Haenzel [paquet « survie » dans RStudio (Version 0.99.486)] pour déterminer des différences significatives dans la survie des populations de différentes souches au fil du temps au titre de l’augmentation de la concentration d’antibiotiques12.
Remarque : Cette expérience permettra de déterminer si les plasmides multi-copies potentialisent l’évolution de la résistance aux antibiotique pour l’antibiotique particulier et le gène à l’étude.
3. base moléculaire de l’évolution de la résistance aux antibiotique (Figure 1)
- Effectuer les extractions d’ADN totales d’un nombre représentatif des populations et des clones à travers les souches et les traitements. Inclure les souches parentales (MG1655, MG1655 ::resA et MG1655/pRESA) pour pouvoir détecter les mutations s’accumulent durant l’expérience.
Remarque : Pour obtenir des exemples de kits d’extraction de l’ADN, voir la table des matières. Chaque kit comporte différents protocoles ; Suivez les instructions du fabricant. - Quantifier la concentration et la qualité de l’ADN. Il existe différentes méthodes pour déterminer la concentration et la qualité de l’ADN. Déterminer la qualité mesurant les ratios d’absorbance 260 nm nm/280 et 260 nm/230 nm. Utiliser un colorant fluorescent, de la liaison à l’ADN pour mesurer la concentration d’ADN suivant instructions du fabricant, et sur gel d’agarose pour confirmer qu’il n’est aucune dégradation de l’ADN ou de contamination de la RNA.
- Séquençage de l’ADN profond utilisation des échantillons de populations entières et les clones d’enquêter sur les bases génétiques de la résistance aux antibiotique.
Remarque : Nous avons effectué toutes les séquençage au Wellcome Trust Centre for Human Genetics, Oxford, UK. Pour plus de détails, voir San Millan et al. 12de 2016. - Le breseq 0.26.1 pipeline27,28 permet de détecter les mutations, en utilisant le mode de polymorphisme pour estimer la fréquence des mutations chez les populations. Comparer différents génomes évolués au génome parental pour détecter les mutations qui se sont accumulés au cours de l’expérience.
- Comparez les mutations accumulées en parallèle dans les populations de contrôle avec ceux des populations a évolué sous l’augmentation de la concentration d’antibiotiques pour faire la différence entre les mutations aident les bactéries de s’adapter à la conjoncture expérimentale ( ceux trouvés dans les contrôle des populations) et ceux impliqués dans la résistance aux antibiotique (ceux trouvés exclusivement dans les populations soumises à la pression antibiotique).
Remarque : Le nombre de mutations parallèles est généralement faible, donc différentes mutations parallèles entre les traitements peuvent être facilement évaluées. Mis à part les mutations dans le gène de résistance aux antibiotiques à l’étude, qu'il est fort probable de trouver d’autres mutations associée à la résistance aux antibiotique dans le chromosome, ces mutations porines ou l’efflux des systèmes12. - Reconstruire les mutations dans le gène de résistance aux antibiotiques chez la souche parentale en utilisant la même approche qu’à l’article 1 du présent protocole. Suivez les points 1.1 et 1.2 du Protocole visant à introduire les nouvelles mutations en arrière-plan parental (en utilisant les gènes évolués comme modèle PCR). Analyser les phénotypes de résistance aux antibiotiques de ces nouvelles constructions pour confirmer le rôle des mutations.
Résultats
Dans nos travaux précédents, on a étudié l’évolution le gène les β-lactamase blaTEM-1 vers conférant la résistance à la troisième génération de céphalosporines ceftazidime12 . Ce gène a été choisi parce que, bien que TEM-1 ne confère pas de résistance à la ceftazidime, mutations blaTEM-1 peuvent élargir l’éventail des activités de TEM-1 d’hydrolyser les céphalosporines comme ceftazidime29. Enzymes de résistance aux antibiotiques comme β-lactamases ou aminoglycoside modification des enzymes conduisant à des changements dans leur aire de répartition de l’activité des mutations sont communs29,30. Ce système expérimental est idéal pour explorer l’évolution de ce type d’enzymes. Pour un rapport détaillé d’une expérience réussie en suivant ce protocole s’il vous plaît voir San Millan et al. 12de 2016.
Ici, un exemple des résultats possibles de ce système expérimental est présenté pour illustrer le protocole (Notez que les données utilisées dans cet exemple ne soient pas réelles). D’enquêter sur le rôle potentiel de multicopies plasmides dans l’évolution de la gène de résistance aux antibiotiques sous étudient dans cet exemple (appelons-le resA), nous développons le système expérimental suivant l’article 1 du protocole décrit ci-dessus. Les expériences de produisent trois lignées : MG1655, MG1655 ::resA et MG1655/pRESA. L’évolution des résistances aux deux différentes ß-lactamines (ceftazidime et Méropénem) a été testée en suivant les étapes décrites à l’article 2 du protocole. La figure 2 montre les courbes de survie des populations à l’étude. Dans cet exemple, il y a une augmentation significative de la survie des populations appartenant à MG1655/pRESA évoluant dans la ceftazidime comparée à ceux de MG1655 ou MG1655 ::resA (log-rank test, P< 0,05). En revanche, dans le cas de meropenem, il n’y a pas de différences significatives dans la survie des populations appartenant à différentes souches (log-rank test, P> 0,05). Par conséquent, ces résultats suggèrent que la présence du gène resA dans un plasmide Multicopie potentialise l’évolution de la résistance à la ceftazidime, mais pas à meropenem.
Dans la dernière étape de l’expérience, les bases moléculaires de résistance aux antibiotique sont étudiée, comme expliqué dans l’article 3 du protocole. Tout d’abord, le séquençage de l’ADN révélera les mutations resA qui pourraient être responsables du phénotype de résistance. Et Deuxièmement, reconstruction des mutations de resA dans le MG1655 parental (à la fois dans le chromosome et les plasmides) confirmera ou jeter leur rôle sur le phénotype de résistance aux antibiotiques.
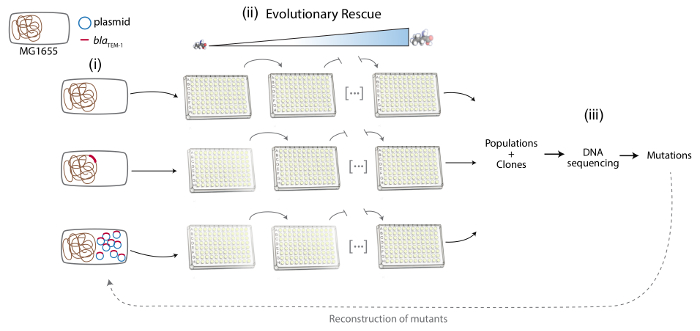
Figure 1 . Représentation schématique des différentes phases du protocole. De gauche à droite : (i) la Construction du système expérimental : MG1655, MG1655 ::resA et MG1655/pRESA. Chromosome bactérien est représenté en brun, le plasmide en bleu et le gène de la resA en rouge. (ii) approche de sauvetage évolutive d’évoluer expérimentalement la résistance aux antibiotique : plusieurs populations de différentes souches sont propagées au titre de l’augmentation de la concentration de l’antibiotique. (iii) l’analyse de la base moléculaire de la résistance aux antibiotique : séquençage de l’ADN d’échantillons de populations évoluées et de clones, de détection des mutations de résistance aux antibiotiques et à la reconstruction de ces mutations chez la souche parentale. S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
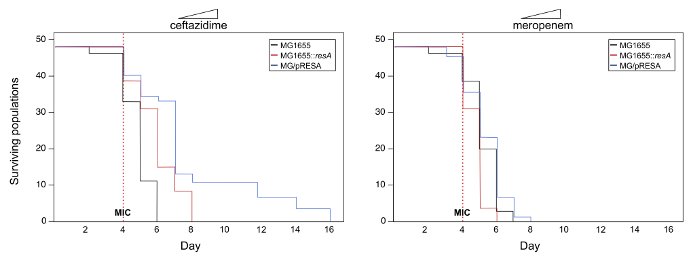
La figure 2. Des courbes de survie avec augmentation des concentrations d’antibiotiques. Représentation du nombre de populations viables appartenant aux souches MG1655, MG1655 ::resAet MG1655/pRESA au fil du temps. 48 les populations de chaque souche ont été propagées au titre de l’augmentation des concentrations d’antibiotiques ceftazidime et méropénem, commençant par 1/8 de la MIC le jour 1 et doublement de la concentration d’antibiotique tous les jours. La ligne verticale en pointillés rouge représente le MIC des antibiotiques à l’étude. Notez qu’il y a des différences significatives dans la survie des populations appartenant à différentes souches au fil du temps dans le cas de la ceftazidime (log-rank test, P< 0,05). C’est essentiel, que les populations portant le plasmide sont capables de survivre jusqu'à des concentrations de haut niveau d’antibiotique. En revanche, dans le cas de meropenem, il n’y a pas de différences significatives dans la survie des populations différentes au fil du temps (log-rank test, P> 0,05). S’il vous plaît cliquez ici pour visionner une version agrandie de cette figure.
Discussion
Nous présentons un nouveau protocole combinant biologie moléculaire, évolution expérimentale et profond séquençage de l’ADN visant à examiner le rôle des plasmides multi-copies dans l’évolution de la résistance aux antibiotique chez les bactéries. Bien que ce protocole combine les techniques de différentes disciplines, toutes les méthodes nécessaires pour le développer sont simples et peuvent être effectuées dans un laboratoire de microbiologie ordinaire. Les étapes plus critiques dans le protocole sont probablement la construction des souches modèle système et la reconstruction des mutations observées après l’évolution expérimentale (qui sont fait en utilisant la même méthode). Toutefois, le système de montage isotherme21, simplifie considérablement ce protocole afin que tout utilisateur disposant d’un niveau intermédiaire d’expérience en biologie moléculaire peut mettre en œuvre.
Une autre étape critique du protocole est l’évolution expérimentale au titre de l’augmentation des concentrations d’antibiotiques. À titre d’exemple, ce protocole commence l’expérience avec ¼-1/8 de la CMI de souches et ensuite doubler la concentration d’antibiotique tous les jours. Toutefois, un taux inférieur de changement aux antibiotiques pourrait augmenter les chances de sauvetage évolutionnaire d’extinction26. Par conséquent, le taux de variation des concentrations d’antibiotique est l’un des paramètres qui peuvent être modifiés pour promouvoir l’évolution de la résistance aux antibiotique.
L’analyse et le séquençage de l’ADN sont également des aspects essentiels du plan expérimental. Les résultats sont plus simples lorsque séquençage est effectué sur des échantillons d’ADN de populations entières et de clones individuels, à des moments différents dans l’expérience. Séquençage des résultats provenant de populations révélera les différences générales dans les profils de mutation entre les traitements, ainsi que des balayages sélectives des mutations bénéfiques dans le temps et les événements possibles d’interférence clonale. Lors de l’analyse des séquences des populations, il est préférable de filtrer les mutations qui jamais dépassé 10 % fréquence dans une population. Séquences de clones individuels viennent confirmer les résultats obtenus par les populations et, surtout, de révéler les combinaisons spécifiques entre les différentes mutations observées au niveau des populations. Ces associations spécifiques peuvent aider à découvrir les interactions épistatiques entre mutations, qui jouent un rôle essentiel dans l’adaptation bactérienne31.
En utilisant cette méthode, nous avons récemment démontré que les plasmides multi-copies accélèrent l’évolution de la résistance aux antibiotique, tout d’abord en augmentant le taux d’apparition de nouvelles mutations, puis en amplifiant l’effet de mutations en raison du dosage des gènes accrue12 . Par conséquent, nous avons développé la méthode comme un outil pour analyser l’évolution de la résistance aux antibiotique, mais il peut avoir un plus large éventail d’applications. À savoir, ce système pourrait être utilisé pour étudier la possibilité d’un gène bactérien d’évoluer vers une fonction nouvelle ou améliorée d’une manière plus générale. Ce système pourrait être utilisé, par exemple, pour tester la capacité d’une enzyme/voie métabolique d’utiliser les nouveaux carbone substrats32. En outre, il pourrait être utilisé au lieu de hypermutators (bactéries présentant un défaut dans les systèmes cellulaires impliqués dans des mésappariements de l’ADN) pour étudier l’évolution des gènes adaptatif chez les bactéries, en évitant le biais mutationnel introduit par hypermutators.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par l’Instituto de Salud Carlos III (Plan Estatal de I + D + je 2013-2016) : subventions CP15-00012, PI16-00860 et CIBER (CB06/02/0053), cofinancé par le Fonds européen de développement régional '' une façon d’atteindre l’Europe '' (FEDER). JAE est soutenue par le programme Atracción de talento du gouvernement de la région de Madrid (2016-T1/BIO-1105) et la j’ai + D Excelencia de l’espagnol Ministerio de Economía, Industria y Competitividad (BIO2017-85056-P). ASM est soutenu par une bourse de recherche Miguel Servet de l’Instituto de Salud Carlos III (MS15/00012) cofinancés par le Fonds Social européen « Investir dans votre avenir » (FSE) et le FEDER.
matériels
| Name | Company | Catalog Number | Comments |
| Thermocycler | BioRad | C1000 | |
| Electroporator | BiorRad | 1652660 | |
| Electroporation cuvettes | Sigma-Aldrich | Z706078 | |
| NanoDrop 2000/2000c | Thermo Fisher Scientific | ND-2000 | Determine DNA quality measuring the ratios of absorbance 260nm/280nm and 260nm/230nm |
| Incubator | Memmert | UF1060 | |
| Incubator (shaker) | Cole-Parmer Ltd | SI500 | |
| Electrophoresis power supply | BioRad | 1645070 | Agarose gel electrophoresis |
| Electrophoresis chamber | BioRad | 1704405 | Agarose gel electrophoresis |
| Pippettes | Biohit | 725020, 725050, 725060, 725070 | |
| Multi-channel pippetes | Biohit | 728220, 728230, 728240 | |
| Plate reader Synergy HTX | BioTek | BTS1LF | |
| Inoculating loops | Sigma-Aldrich | I8388 | |
| 96-well plates | Falcon | 351172 | |
| LB | BD Difco | DF0446-17-3 | |
| LB agar | Fisher scientific | BP1425-500 | |
| Phusion Polymerase | Thermo Fisher Scientific | F533S | |
| Gibson Assembly | New England Biolabs | E2611S | |
| Resctriction enzymes | Fermentas FastDigest | ||
| Antibiotics | Sigma-Aldrich | ||
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | Plasmid extraction kit |
| Wizard Genomic DNA Purification Kit | Promega | A1120 | gDNA extraction kit |
| DNeasy Blood & Tissue Kits | Qiagen | 69506 | gDNA extraction kit |
| Electroporation cuvettes | Sigma-Aldrich | Z706078 | |
| Petri dishes | Sigma-Aldrich | D9054 | |
| Cryotubes | ClearLine | 390701 | |
| 96-well plates (-80ºC storage) | Thermo Fisher Scientific | 249945 | |
| QuantiFluor dsDNA System | Promega | E2670 | Quantification of DNA concentartion |
| Agarose | BioRad | 1613100 | Agarose gel electrophoresis |
| 50x TAE buffer | BioRad | 1610743 | Agarose gel electrophoresis |
| T4 Polynucleotide Kinase | Thermo Fisher Scientific | EK0031 | |
| T4 DNA Ligase | Thermo Fisher Scientific | EL0014 |
Références
- Neill, J. TACKLING DRUG-RESISTANT INFECTIONS GLOBALLY: FINAL REPORT AND RECOMMENDATIONS. Review on Antimicrobal Resistance. , (2016).
- Palmer, A. C., Kishony, R. Understanding, predicting and manipulating the genotypic evolution of antibiotic resistance. Nat Rev Genet. 14 (4), 243-248 (2013).
- MacLean, R. C., Hall, A. R., Perron, G. G., Buckling, A. The population genetics of antibiotic resistance: integrating molecular mechanisms and treatment contexts. Nat Rev Genet. 11 (6), 405-414 (2010).
- Buckling, A., Maclean, R. C., Brockhurst, M. A., Colegrave, N. The Beagle in a bottle. Nature. 457 (7231), 824-829 (2009).
- Barrick, J. E., Lenski, R. E. Genome dynamics during experimental evolution. Nature Reviews Genetics. 14 (12), 827-839 (2013).
- Elena, S. F., Lenski, R. E. Evolution experiments with microorganisms: The dynamics and genetic bases of adaptation. Nature Reviews Genetics. 4 (6), 457-469 (2003).
- Lenski, R. E., Rose, M. R., Simpson, S. C., Tadler, S. C. Long-Term Experimental Evolution in Escherichia coli. I. Adaptation and Divergence During 2,000 Generations. The American Naturalist. 138 (6), 1315-1341 (1991).
- Bennett, A. F., Dao, K. M., Lenski, R. E. Rapid evolution in response to high-temperature selection. Nature. 346 (6279), 79-81 (1990).
- Bell, G. Evolutionary rescue and the limits of adaptation. Philosophical Transactions of the Royal Society B-Biological Sciences. 368 (1610), (2013).
- Bell, G., Gonzalez, A. Adaptation and Evolutionary Rescue in Metapopulations Experiencing Environmental Deterioration. Science. 332 (6035), 1327-1330 (2011).
- Bell, G., Gonzalez, A. Evolutionary rescue can prevent extinction following environmental change. Ecology Letters. 12 (9), 942-948 (2009).
- San Millan, A., Escudero, J. A., Gifford, D. R., Mazel, D., MacLean, R. C. Multicopy plasmids potentiate the evolution of antibiotic resistance in bacteria. Nature Ecology & Evolution. 1, 0010 (2016).
- Alekshun, M. N., Levy, S. B. Molecular mechanisms of antibacterial multidrug resistance. Cell. 128 (6), 1037-1050 (2007).
- Ochman, H., Lawrence, J. G., Groisman, E. A. Lateral gene transfer and the nature of bacterial innovation. Nature. 405 (6784), 299-304 (2000).
- Carattoli, A. Plasmids and the spread of resistance. Int J Med Microbiol. 303 (6-7), 298-304 (2013).
- Smillie, C., Garcillán-Barcia, M. P., Francia, M. V., Rocha, E. P., de la Cruz, F. Mobility of plasmids. Microbiol Mol Biol Rev. 74 (3), 434-452 (2010).
- San Millan, A., Heilbron, K., Maclean, R. C. Positive epistasis between co-infecting plasmids promotes plasmid survival in bacterial populations. ISME J. , (2013).
- Stoesser, N., et al. Evolutionary History of the Global Emergence of the Escherichia coli Epidemic Clone ST131. MBio. 7 (2), (2016).
- San Millan, A., et al. Multiresistance in Pasteurella multocida is mediated by coexistence of small plasmids. Antimicrob Agents Chemother. 53 (8), 3399-3404 (2009).
- Escudero, J. A., et al. Unmasking the ancestral activity of integron integrases reveals a smooth evolutionary transition during functional innovation. Nat Commun. 7, 10937 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc Natl Acad Sci U S A. 97 (12), 6640-6645 (2000).
- Chaveroche, M. K., Ghigo, J. M., d'Enfert, C. A rapid method for efficient gene replacement in the filamentous fungus Aspergillus nidulans. Nucleic Acids Res. 28 (22), 97 (2000).
- Le Roux, F., Binesse, J., Saulnier, D., Mazel, D. Construction of a Vibrio splendidus mutant lacking the metalloprotease gene vsm by use of a novel counterselectable suicide vector. Appl Environ Microbiol. 73 (3), 777-784 (2007).
- . . Performance standards for antimicrobial susceptibility testing; 19th ed. Approved standard M100-S19. , (2009).
- Lindsey, H. A., Gallie, J., Taylor, S., Kerr, B. Evolutionary rescue from extinction is contingent on a lower rate of environmental change. Nature. 494 (7438), 463-467 (2013).
- Deatherage, D. E., Barrick, J. E. Identification of mutations in laboratory-evolved microbes from next-generation sequencing data using breseq. Methods Mol Biol. 1151, 165-188 (2014).
- Barrick, J. E., et al. Identifying structural variation in haploid microbial genomes from short-read resequencing data using breseq. BMC Genomics. 15, 1039 (2014).
- Salverda, M. L., De Visser, J. A., Barlow, M. Natural evolution of TEM-1 β-lactamase: experimental reconstruction and clinical relevance. FEMS Microbiol Rev. 34 (6), 1015-1036 (2010).
- Ramirez, M. S., Tolmasky, M. E. Aminoglycoside modifying enzymes. Drug Resist Updat. 13 (6), 151-171 (2010).
- Jerison, E. R., Desai, M. M. Genomic investigations of evolutionary dynamics and epistasis in microbial evolution experiments. Current Opinion in Genetics & Development. 35, 33-39 (2015).
- Toll-Riera, M., San Millan, A., Wagner, A., MacLean, R. C. The Genomic Basis of Evolutionary Innovation in Pseudomonas aeruginosa. PLoS Genet. 12 (5), 1006005 (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon