Method Article
抗生物質耐性の進化のマルチコピー プラスミドのロールのテスト
要約
抗生物質耐性の進化でマルチコピー プラスミドの役割をテストするための実験方法をご紹介します。
要約
マルチコピー プラスミドは原核生物に非常に多いが、細菌の進化におけるそれらの役割のままかり。我々 は最近、マルチコピー プラスミッドによって指定されたセルあたり遺伝子コピー数の増加がプラスミド エンコード遺伝子の進化を加速させる可能性を示した。この作品は、マルチコピー プラスミドの遺伝子の進化を促進するための能力をテストする実験システムを提案する.分子生物学の単純なメソッドを使用して、抗生物質耐性遺伝子を大腸菌MG1655、染色体またはマルチコピー プラスミド上に挿入できるモデル システムを構築しました。抗生物質の濃度の増加の下で異なる系統を反映する実験的進化のアプローチを使用して、我々 は時間をかけて細菌集団の生存率を測定します。抗生物質分子と抵抗性遺伝子の選択は、遺伝子突然変異の買収を通じて抵抗を与えることができるだけので、です。この「進化レスキュー」アプローチは、マルチコピー プラスミドの抗生物質抵抗性の獲得を促進するための可能性をテストする簡単な方法を提供します。実験システムの次のステップでは、抗生物質耐性の分子基盤が特徴です。抗生物質耐性の獲得のための責任の突然変異を識別するために、我々 は全体の人口とクローンから得られた試料のディープの DNA シーケンス使用します。最後に、調査の下で遺伝子の変異の役割を確認する我々 は親の背景にそれらを再構築し、結果系統の抵抗の表現型をテストします。
概要
細菌の抗生の抵抗は重大な健康問題1です。基本的なレベルで病原細菌の抗生物質耐性の普及、自然淘汰の2,の3による進化の単純な例です。簡単に言えば、抗生物質の使用は、耐性菌の選択を生成します。進化生物学の重要な問題は、したがって、抗生物質への耐性を進化する細菌集団の能力に影響を与える要因を理解します。選択の実験は、細菌の進化生物学を調査するための非常に強力なツールとして浮上しているし、このフィールドは幅広い進化問題4,5,6に信じられないほどの洞察力を生産しています。実験的進化の 1 つの親株から開始された細菌集団が定義され、厳しく管理された条件の下で連続継代します。これらの文化の成長中に発生する突然変異のいくつかの増加細菌のフィットネス、これらは自然淘汰によって文化を介して 。実験では、人口のサンプルは非進化する冷凍化石レコードを作成する定期的に低温保持されます。細菌集団を進化を特徴付けるためのアプローチの広い数を使用できますが、2 つの最も一般的な方法は、フィットネスの試金、遠い祖先と全ゲノム配列が進化した細菌の競争力を測定ドライブの適応の遺伝の変更を識別するために使用されます。リチャード Lenski および同僚7,8で先駆的な仕事は、次の実験進化の標準的なアプローチはレプリケート人口の比較的小さい数に挑戦するされている (通常 < 10) 新しいへの適応に環境チャレンジ、新しい炭素源、温度、捕食性ファージなど。
抗生物質耐性菌によって引き起こされる感染症抵抗は患者の体内を致死量に抗生物質の濃度を高めることが可能だが十分に高いとき大きな問題になります。臨床医は細菌がこのしきい値抗生物質濃度臨床ブレークポイント上抗生物質の高用量への抵抗を進化することができる内容に興味があるため。これを実験的に研究する方法?細菌集団の数が少ないが、高用量の抗生物質の挑戦レンスキー スタイルのように実験、その後、最も可能性の高い結果は、抗生物質が絶滅の危機に個体群のすべてを駆動すること。同時に使用される抗生物質の投与量は、親株の最小発育阻止濃度 (MIC) を下回る場合、ない細菌の集団は、抵抗性の臨床的に関連するレベルを進化していく可能性が高い場合は特に抵抗には、大きな代償が伴います。これらの 2 つのシナリオ間の 1 つの妥協は、「進化レスキュー」実験9,10,11を使用します。このアプローチは文化の非常に大きい数で (通常 > 40) が時間をかけて増加、通常抗生物質の濃度を倍にして毎日12抗生物質の用量で挑戦します。この実験の特徴は、高められた抵抗を進化していない人口が絶滅に追い込まれることです。この方法でほとんどの個体群は絶滅し、駆動されるが、少数は、高レベルの耐性を進化させることによって保持されます。本稿では、この実験的なデザインを使用して抵抗性の進化にマルチコピー プラスミドの貢献を調査する方法を示す.
細菌は、2 つの主要ルート、染色体突然変異およびモバイル遺伝的要素、主プラスミド13の取得を通じた抗生物質に耐性を獲得します。プラスミドは、活用14,15による細菌の抵抗性遺伝子を転送することができるので抗生物質耐性の進化で重要な役割を再生します。プラスミドは彼らのサイズおよび生物学によると 2 つのグループに分けることができます:「小」、高コピー数細菌の細胞と「大」と低コピー数の16,17。抵抗と細菌15マルチ抵抗の普及の主要なドライバーである接合性プラスミドが含まれるために、抗生物質耐性の進化での大型プラスミドの役割を広く記載されています。小さなマルチコピー プラスミドも細菌17,18, 非常に一般的な彼らはしばしば抗生物質耐性遺伝子19のコードします。ただし、抗生物質耐性の進化で小さなマルチコピー プラスミドの役割より少ない程度に研究されています。
最近の作品ではマルチコピー プラスミドが高い遺伝子コピー セル12数による遺伝子突然変異率を増やすことによって彼らを運ぶ遺伝子の進化を加速させる可能性を提案します。大腸菌の菌株 MG1655 と β-ラクタマーゼ遺伝子blaTEM 1実験モデルを用いたマルチコピー プラスミド加速の第三世代に耐性 TEM 1 突然変異の出現率であることが示されました。セファロスポリン セフタジジム。マルチコピーのプラスミドがあります抗生物質耐性の進化で重要な役割を果たすことが示唆されました。
今回開発した手法の詳細な説明、マルチコピー プラスミド性抗生物質耐性の進化を調査します。このメソッドは 3 つの異なる手順: 調査マルチコピー プラスミッドまたはホストの細菌の染色体のどちらかの下で遺伝子の最初に挿入します。第二に、実験的進化 (進化救助) の選択的な圧力に適応するため別系統の可能性を評価するために使用します。そして第三に、DNA シークエンシングと親の遺伝子型で個別に疑いのある突然変異を再構成を用いたプラスミド性の進化の分子基盤を決定します。
最後に、ここで説明されているプロトコルは、抗生物質耐性の進化を調査するため設計されました、1 つ主張することがこのメソッドは一般に任意の複数部数の突然変異によって取得した技術革新の進化を分析し便利かもしれないプラスミド エンコード遺伝子。
プロトコル
1. 抗生物質耐性遺伝子を符号化実験システムの構築
注: ここで大腸菌MG1655 プラスミッドまたは染色体符号化された抗生物質耐性遺伝子の受信者のひずみとして使用されていた。抗生物質耐性遺伝子は、染色体またはマルチコピー プラスミッドそれ以外同質遺伝子系統 (図 1) にエンコードされます。
-
MG1655 の染色体の λ のバクテリオファージの統合サイト (attB)20の抗生物質耐性遺伝子の挿入。
- Pcr 法による染色体attBサイトの両側に 500 bp 長い領域を増幅します。YbhC F のオリゴヌクレオチドを使用して (5'-CCTGTACCGTACAGAGTAAT-3')、attB R (5'-GCCCGCCACCCTCCGGGCCGGTATAAAAAAGCAGGCTTCA-3') 左相同領域、attB F (5'-AGCGCCCTAGCGCCCGCTCCTTATACTAACTTGAGCGAAA-3') と YbhB/R (5'-TGGCGATAATATTTCACCGC-3') 右の 1 つ。プライマー Tem1-pBAD-F を使用してblaTEM 1抵抗性遺伝子の増幅 (5'-TGAAGCCTGCTTTTTTATACCGGCCCGGAGGGTGGCGGGC-3') と Tem1-pBAD-R (5'-TTTCGCTCAAGTTAGTATAAGGAGCGGGCGCTAGGGCGCT-3')。約 20 のプライマーを設計の相補性に融合するのに起こっているフラグメントを示すシーケンス (5' 末端) の bp。
- 抗生物質耐性遺伝子 PCR 製品 (そのプロモーター領域を含む) に相同領域をヒューズ 30 分 50 ° C で等温アセンブリ21を使用して。
- Electroporate MG1655 細胞含むプラスミド pKOBEG の。
注: pKOBEG は、λ を含む感温ベクター赤い機械、相同性に基づく対立遺伝子を染色体と PCR 製品22間交流します。- 1.1.2 手順23から製品の 1 μ L を使用して、4 ° C で 2 mm キュヴェット electrocompetent 細胞の 40 μ L と 2.5 kV。赤い機械を 1 mL の流体培養基 LB + 0.2% アラビノース、λ の式を維持するために細胞を再懸濁します。
- および microfuge の管に総量を転送し、染色体に挿入した抵抗のマーカーの形質発現を許可する強烈な揺れ (軌道シェーカーで 200 rpm) と 30 ° C (pKOBEG の許容温度) で 2 h を孵化させなさい。
- (アンピシリンまたはblaTEM 1100 mg/l で carbenicillin) の抗生物質耐性遺伝子の選択に適切な抗生物質を含んでいる LB 寒天培地で細胞をプレートします。
- 1 つのシャーレに 100 μ L をプレートしボリュームの残りの部分をスピン、新鮮な流体培養基 LB の 100 μ L でそれを再懸濁します、もう一つのシャーレにプレートします。42 ° C で一晩インキュベートします。
注: この温度は pKOBEG を複製するための寛容です。これらの条件で成長することができる植民地 pKOBEG を失っている、 attBサイトに統合された抵抗性遺伝子を提示 (便宜上この株が呼び出されますMG1655::レザここから)。 - クロラムフェニ コールを板の興味のコロニーをレプリケートすることによって pKOBEG の損失のためのテスト (どの pKOBEG に対する抗生物質には抵抗性マーカーが含まれています)。
注: 30 º C の許容温クロラムフェニ コール プレートの成長の欠如はプラスミッドの不在を示すです。 - クローンを確認するために PCR 増幅外部プライマー YbhC Extern を使用して建設 (5'-TTTGTGACCAGAAGACCGCA-3') と YbhB Extern (5'-CTCATCAGTAACGATCTGCG-3')、ゲル電気泳動による増幅とシーケンスの正しいサイズを確認、挿入された遺伝子のシーケンスが正しいことを確認する精製した製品です。
-
マルチコピー プラスミドに抗生物質耐性遺伝子を挿入します。
注: 非常に高コピー数とプラスミドは、細菌のホストのフィットネスで大規模な削減を課す傾向がある、ためにをお勧めします p3655 など、レプリケーションの天然起源のマルチコピー プラスミドの使用 (pSU18T pBADgfp2、 ColE1 タイプの起源のレプリケーション24)。これらのプラスミッドは通常程度の適度なコピー数を提示細胞 1 個あたり 15 ~ 20 枚。- PCR (プロモーター領域を含む) 選択の抗生物質耐性遺伝子を増幅する PCR の製品に対する、プラスミッドの PCR 増幅バックボーンに縛る。
- PCR 増幅抗生物質耐性遺伝子;blaTEM 1使用プライマー Tem1-pBAD-F (5'-TGAAGCCTGCTTTTTTATACCGGCCCGGAGGGTGGCGGGC-3') と Tem1-pBAD-R (5'-TTTCGCTCAAGTTAGTATAAGGAGCGGGCGCTAGGGCGCT-3')。
- 製造元の指示に従って、T4 ポリヌクレオチド キナーゼを使用して精製した製品をリン酸化します。
- PCR 増幅を使用して高品質なポリメラーゼおよびオリゴヌクレオチド pBAD F プラスミド バックボーン (5'-CGTTGATCGGCACGTAAGAG-3') pBAD r (5'-AAACGACGGCCAGTGCCAAG-3')。
- T4 リガーゼを使って両方の PCR のフラグメントを縛る製造元のガイドラインに従います。
- Electroporate、electrocompetent大腸菌dh 5 位 α 細胞の結紮製品と適切な抗生物質 (アンピシリンまたは 100 mg/L で carbenicillin の抗生物質耐性遺伝子の選択の 1 μ L の最大 40 μ L を前に説明しました。blaTEM 1) 用とプラスミドのバックボーン。
注: ここでアラビノースを LB で補うことは必要ではありません。 - 読者の好みの市販の小型準備キットを使用してプラスミッドを抽出します。
- 常温 12,000 × g で遠心分離で一晩かけて培養 5 mL を収穫します。再懸濁液の 250 μ L で細胞を再懸濁します、よく混ぜます。溶解液の 250 μ L を加え、よく混ぜます。中和の 350 μ L を加え、よく混ぜます。
- 1 分間遠心するキットで提供される DNA 結合列に上清を転送し、流れを破棄します。500 μ L の洗浄液、1 分間遠心分離し、洗うたびに流れる通路を破棄 2 回を洗い流します。残留洗浄バッファーを削除する 1 の追加分の空の列を遠心します。
- クリーン チューブに列を配置し、浄化された DNA を溶出する膜に超純水の 50 μ L を追加します。1-2 分間遠心し、収集する流れは、精製プラスミドを含んでいます。サンガー シーケンスのシーケンスが正しいことと突然変異が進化の実験の前に抵抗性遺伝子の存在しないことを確認する挿入されたシーケンス外からプライマーを使用してプラスミッドの興味の遺伝子。
- シーケンスを確認すると、electroporate プラスミド大腸菌MG1655 ひずみ、前述のように。
注: これにより、ひずみMG1655/プレサ。
- PCR (プロモーター領域を含む) 選択の抗生物質耐性遺伝子を増幅する PCR の製品に対する、プラスミッドの PCR 増幅バックボーンに縛る。
2. 進化救助抗生の抵抗 (図 1) を実験的進化方法
- LB の版の調査の下で系統を連勝: MG1655:: MG1655 の影響を受けやすい親株と MG1655/プレサレザ。
LB の版に抗生物質を追加する必要はありませんので注意:、プラスミド プレサは MG1655 で安定しているはずです。エシェリヒア属大腸菌の突然変異率は、建設が確認される各ステップでプラスミッド シーケンスを確認する必要はありませんように十分に低い。 - 各ウェルに LB の 200 μ L で 96 ウェル プレートを準備し、独立した井戸 (ひずみあたり 1 プレート) の各ひずみ 48 隔離されたコロニーを接種します。一晩で、37 ° C、200 rpm 版を孵化させなさい。これらの創始者集団の冷凍ストックを維持します。
注: 96 ウェル プレートで文化の交差汚染の制御を防ぎ、96 ウェル プレートで無菌培地で接種の井戸の間をインターカ レートによってプレート、市松模様を使用します。全体の実験的進化の過程でこのプレートのデザインを使用します。 - 抗生物質研究中の抗生物質の濃度を各ウェルに LB の 198 μ L で新しい 96 ウェル プレートで創設者の集団とプレートの各ウェルから接種した 2 μ L で進化レスキュー実験を開始します。進化の救助のアプローチのためには、各 3 系統の LB の抗生物質の 1/4 または 1/8 (マイク、決定以前25) 最小発育阻止濃度を起動し、毎日の抗生物質の濃度を 2 倍します。抵抗の突然変異26を取得するのに人口のチャンスを最大化するためにこのアプローチを使用します。37 ° C で 20 時間の 200 の rpm 版を孵化させなさい。
注: は、創設者の人口の一晩の外径を測定します。OD に系統間の差がある場合もあたりのセルの同じ番号を持つ実験を開始する初期の接種を修正します。 - 毎日、一晩培養後各人口の外径を測定、前日よりも抗生物質の濃度が 2 倍の 96 ウェル プレートに新しい 2.3 で説明されているように、文化の転送を実行します。プレートは、37 ° C、200 rpm 20 h を孵化させなさい。
注: 1: 100 希釈倍率は、1 日あたり約 6-7 世代を生成します。 - 並行して、抗生物質の不在の 2.3 と 2.4 で説明同じ条件でそれぞれのひずみ制御集団を反映します。
注: コントロール集団を助ける抗生物質や実験条件に適応する細菌を助ける一般的な突然変異の存在のために生じた突然変異を区別します。抗生物質の不在で生じた平行変異は実験条件に適応する細菌を支援する可能性が高いと抗生物質耐性に関連していません。 - 600 の波長で吸光度を測定することによって存続の人口数を毎日追跡プレート リーダーを使用して文化の nm (OD)。
注: 光学濃度値は、0.1 の人口の絶滅を示すより低い。生存曲線の例については図 2を参照してください。 - (すべての 3-5 日) 定期的に人口のすべての冷凍ストックを維持します。
- ログランク テスト [パッケージ RStudio (バージョン 0.99.486) の「サバイバル」] を使用して、抗生物質12の濃度の増加の下で時間をかけて異なる系統の個体群の生存率に有意差を確認します。
注: この実験は、マルチコピー プラスミドが特定の抗生物質や遺伝子研究中の抗生物質耐性の進化を増強するかどうかは決定されます。
3. 抗生物質抵抗 (図 1) の進化の解明
- 系統、治療集団とクローンの代表的な数から総 DNA の抽出を実行します。親系統が含まれます (MG1655、MG1655::レザと MG1655/プレサ) 実験中に蓄積変異を検出できるようにします。
注: DNA 抽出キットの例は、材料の表を参照してください。各キットには別のプロトコルメーカーの指示に従います。 - DNA の品質と濃度を定量化します。DNA の品質と濃度を決定するさまざまな方法があります。260 nm/230 nm の吸光度 260 nm/280 nm 比の測定品質を決定します。製造元の指示に従って DNA 濃度を測定する蛍光、DNA 結合色素を使用し、アガロース電気泳動することを確認しますは劣化 DNA または RNA 汚染します。
- 抗生物質耐性の遺伝的基礎を調査する全体集団と個々 のサンプルの使用ディープ DNA シーケンスを複製します。
注: 人類遺伝学、オックスフォード、イギリスのウェルカム ・ トラスト ・ センターですべてのシーケンス処理を実施しました。詳細は、サン ミジャンらを参照してください。201612。 - Breseq 0.26.1 パイプライン27,28を使用すると、個体群の変異の頻度を推定する多態性モードを使用して突然変異を検出できます。実験中に蓄積している突然変異を検出する親のゲノムに異なる進化ゲノムを比較します。
- 一般の実験条件 (に適応する細菌の突然変異を区別するための抗生物質の濃度を増加させるの進化は突然変異蓄積と人口のコントロール集団で並列比較それらは制御集団で発見) と抗生物質耐性 (抗生物質の圧力を受ける集団の排他的に見られるもの) に携わる。
注: 平行変異の数は通常低ので、治療の間の異なる平行変異を容易に評価できます。見つけることはかなりそうだ検討中の抗生物質耐性遺伝子の変異から離れて他の突然変異は染色体における抗生物質耐性、ポリン ・流出のシステムのための12のような突然変異に関連付けられています。 - このプロトコルのセクション 1 と同じアプローチを使用して親の緊張で抗生物質耐性遺伝子の変異を再構築します。1.1 と 1.2 (進化した遺伝子を PCR のテンプレートとして使用) 親の背景で新しい突然変異を導入するプロトコルの点に従ってください。突然変異の役割を確認するこれらの新建設の抗生の抵抗の表現型を分析します。
結果
筆者らは, β-ラクタマーゼ遺伝子blaTEM 1第 3 世代セファロスポリン セフタジジム12耐性に向けて進化を調べた。この遺伝子は、 blaTEM 1の変異がセフタジジム29などのセファロスポリンを分解する TEM 1 の活動の範囲を拡大することができます TEM 1 セフタジジムに抵抗を与えないがので選ばれました。Β-ラクタマーゼなど修正の酵素の活動の彼らの範囲の変化につながるアミノ配糖体抗生物質耐性酵素の変異は、一般的な29,30です。本実験系は酵素のこのタイプの進化の観光に便利です。このプロトコルを次の巧妙な実験の詳細なレポート サン ミジャンらを参照してください。201612。
ここでは、この実験システムの可能な結果の例は、プロトコル (この例で使用されるデータが本物ではないことに注意してください) を説明するためにされます。複数部数の潜在的な役割を調べるための下で抗生物質耐性遺伝子の進化でプラスミド研究 (いわばレザ) この例では、上記で説明したプロトコルのセクション 1 に続く実験システムを開発します。実験生産 3 系統: MG1655、MG1655::レザと MG1655/プレサ。(セフタジジムとメロペネム) 2 つの異なる β-ラクタム系抗生物質に耐性の進化は、プロトコルのセクション 2 で説明されている手順に従ってテストされました。図 2は、調査の下で個体群の生存曲線を示します。この例では、MG1655/プレサのセフタジジム MG1655 または MG1655 からのそれらと比較して進化してに属する個体群の生存率が大幅に増加::レザ(ログランク検定、 P< 0.05)。その一方で、メロペネム、場合は異なる系統に属する個体群の生存率に有意差 (ログランク検定、 P> 0.05)。したがって、マルチコピー プラスミドに遺伝子レザの存在がセフタジジム、メロペネム耐性の進化を促進することが示唆されました。
実験の最後のステップでプロトコルのセクション 3 で説明したように、抗生物質耐性の分子基盤は.最初に、DNA の配列での抵抗の表現型を担当できるレザで突然変異を明らかにすることが。第二に、レザ変異親 MG1655 (両方の染色体やプラスミド) の再建に抗生物質耐性表現型の役割を破棄します。
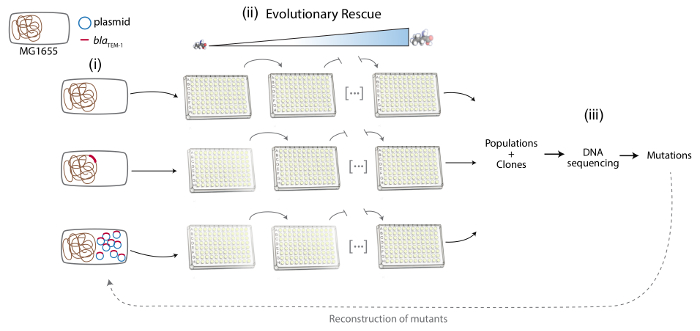
図 1.プロトコルの異なるフェーズの略図。左から右へ: (i) 実験システムの構築: MG1655、MG1655::レザと MG1655/プレサ。細菌の染色体は、ブラウン、ブルーのプラスミッド、赤レザ遺伝子で表されます。(抗生物質耐性の進化実験 ii) 進化救助アプローチ: 抗生物質の濃度の増加の下で異なった緊張のいくつかの集団が反映されます。(抗生物質耐性の分子基盤の解析 iii): DNA の塩基配列決定に進化した集団とクローン、抗生物質耐性遺伝子変異の検出と、親株のこれらの突然変異の復興からのサンプルします。この図の拡大版を表示するのにはここをクリックしてください。
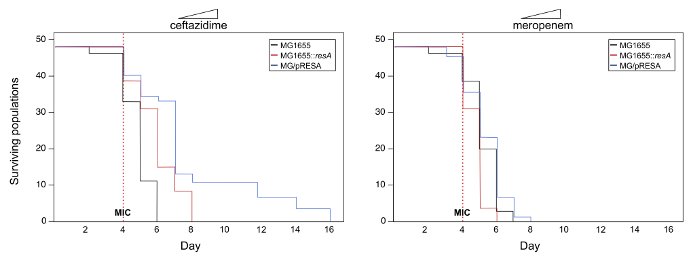
図 2。抗生物質の濃度の増加との生存曲線。系統 MG1655、MG1655 に属する実行可能な人口数の表現::レザと時間をかけて MG1655/プレサ。抗生物質セフタジジムとメロペネム、濃度の増加の下で, 各菌株 48 集団が伝達された日 1 1/8 マイクの始まると毎日の抗生の集中を倍増します。赤破線の縦線は、調査の下で抗生物質の MIC を表します。セフタジジムの場合時間の経過とともに異なる系統に属する個体群の生存率に有意差があることに注意してください (ログランク検定、 P< 0.05)。重大に、プラスミッドを運ぶ集団のみが抗生物質の高レベルの濃度まで生き残ることができます。その一方で、メロペネム、場合がない時間をかけて別の集団の生存率に有意差 (ログランク検定、 P> 0.05)。この図の拡大版を表示するのにはここをクリックしてください。
ディスカッション
分子生物学、実験進化マルチコピー プラスミド細菌の抗生物質耐性の進化の役割を調べるために考案ディープの DNA シーケンスを組み合わせた新しいプロトコルを提案します。このプロトコルは、さまざまな分野からの技術を結合がそれを開発するために必要なすべての方法は単純であり通常の微生物学実験室で実行することができます。プロトコルの最も重要な手順は、モデル システム系統の建設および突然変異の実験進化後に観察された (これは、正確な同じ方法を使用して実行されます) の改造におそらくです。ただし、等温アセンブリ システム21、大幅簡素化されこのプロトコル分子生物学の経験の中間レベルを持つユーザーが実装できるようにします。
プロトコルの別の重要なステップは、抗生物質の濃度の増加の下で実験的進化です。例として、このプロトコルは ¼-1/8 系統と毎日抗生物質濃度倍増のマイクの実験を開始します。ただし、抗生物質の変更率が低いは、絶滅26から進化の救助のチャンスを高めることができます。したがって、抗生物質の濃度の変化の割合は、抗生物質耐性の進化を促進するために変更することができますパラメーターの 1 つです。
DNA 塩基配列と解析は、実験的なデザインの重要な側面です。結果は、全体の人口と実験で異なる時点で、個々 のクローンからの DNA のサンプルでシーケンスが実行されるより簡単です。母集団から結果のシーケンス処理すると、時間とクローン干渉の潜在的なイベントのトリートメント、有益な突然変異の選択的なスイープ間変異プロファイルの一般的な違いが明らかになります。集団からシーケンスを解析するとき、任意の人口の 10% 周波数を上回ったことがない変異をフィルタ リングすることをお勧めします。個々 のクローンからシーケンスは集団から得られた結果を確認に役立つし、最も重要なは、人口レベルで観測された異なる突然変異間の特定の組み合わせを明らかにします。明らかに優性突然変異は、重要な役割を果たす細菌の適応31相互これらの特定の関連付けが役立つことがあります。
このメソッドを使用すると、我々、このところ増加遺伝子投与量12 による突然変異の効果を増幅して、新規変異の出現率を増やすことによって最初にマルチコピーのプラスミドが抗生物質耐性の進化を加速します。.したがって、抗生物質耐性の進化を調査するためのツールとして開発したメソッドがアプリケーションのより広い範囲があります。すなわち、このシステムより一般的な方法で新しいまたは改良された機能に向かって進化するあらゆる細菌の遺伝子の能力を調査するため使用できます。たとえば、このシステムを使用する新しい炭素基板32を使用する酵素・代謝の能力をテストします。また、それは hypermutators (細胞における DNA ミスマッチ修復に関与する欠陥を持つ細菌) の代わりにされる可能性があります細菌、hypermutators によって導入された変異のバイアスを回避する適応型遺伝子進化を調査します。
開示事項
著者が明らかに何もありません。
謝辞
この作品はセルバンテス デによって支えられたサラッド カルロス III (計画アジア デ私 + D + 私 2013-2016): CIBER (CB06/02/0053)、PI16-00860 CP15 00012 協調融資欧州開発地域基金 'ヨーロッパを達成する方法' を付与 (ERDF)。在マドリード (2016-T1/バイオ-1105) と私の地域の政府のAtracción ・ デ ・ タレントプログラムに支えられて + スペイン Ministerio デ Economía、インドゥストリア y Competitividad (BIO2017-85056-P) の D Excelencia です。ASM セルバンテス ・ デ ・からミゲル サーブレット交わりに支えられてサラッド カルロス 3 世 (MS15/00012) の欧州社会基金の協調融資「あなたの未来への投資」(ESF) と ERDF。
資料
| Name | Company | Catalog Number | Comments |
| Thermocycler | BioRad | C1000 | |
| Electroporator | BiorRad | 1652660 | |
| Electroporation cuvettes | Sigma-Aldrich | Z706078 | |
| NanoDrop 2000/2000c | Thermo Fisher Scientific | ND-2000 | Determine DNA quality measuring the ratios of absorbance 260nm/280nm and 260nm/230nm |
| Incubator | Memmert | UF1060 | |
| Incubator (shaker) | Cole-Parmer Ltd | SI500 | |
| Electrophoresis power supply | BioRad | 1645070 | Agarose gel electrophoresis |
| Electrophoresis chamber | BioRad | 1704405 | Agarose gel electrophoresis |
| Pippettes | Biohit | 725020, 725050, 725060, 725070 | |
| Multi-channel pippetes | Biohit | 728220, 728230, 728240 | |
| Plate reader Synergy HTX | BioTek | BTS1LF | |
| Inoculating loops | Sigma-Aldrich | I8388 | |
| 96-well plates | Falcon | 351172 | |
| LB | BD Difco | DF0446-17-3 | |
| LB agar | Fisher scientific | BP1425-500 | |
| Phusion Polymerase | Thermo Fisher Scientific | F533S | |
| Gibson Assembly | New England Biolabs | E2611S | |
| Resctriction enzymes | Fermentas FastDigest | ||
| Antibiotics | Sigma-Aldrich | ||
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | Plasmid extraction kit |
| Wizard Genomic DNA Purification Kit | Promega | A1120 | gDNA extraction kit |
| DNeasy Blood & Tissue Kits | Qiagen | 69506 | gDNA extraction kit |
| Electroporation cuvettes | Sigma-Aldrich | Z706078 | |
| Petri dishes | Sigma-Aldrich | D9054 | |
| Cryotubes | ClearLine | 390701 | |
| 96-well plates (-80ºC storage) | Thermo Fisher Scientific | 249945 | |
| QuantiFluor dsDNA System | Promega | E2670 | Quantification of DNA concentartion |
| Agarose | BioRad | 1613100 | Agarose gel electrophoresis |
| 50x TAE buffer | BioRad | 1610743 | Agarose gel electrophoresis |
| T4 Polynucleotide Kinase | Thermo Fisher Scientific | EK0031 | |
| T4 DNA Ligase | Thermo Fisher Scientific | EL0014 |
参考文献
- Neill, J. TACKLING DRUG-RESISTANT INFECTIONS GLOBALLY: FINAL REPORT AND RECOMMENDATIONS. Review on Antimicrobal Resistance. , (2016).
- Palmer, A. C., Kishony, R. Understanding, predicting and manipulating the genotypic evolution of antibiotic resistance. Nat Rev Genet. 14 (4), 243-248 (2013).
- MacLean, R. C., Hall, A. R., Perron, G. G., Buckling, A. The population genetics of antibiotic resistance: integrating molecular mechanisms and treatment contexts. Nat Rev Genet. 11 (6), 405-414 (2010).
- Buckling, A., Maclean, R. C., Brockhurst, M. A., Colegrave, N. The Beagle in a bottle. Nature. 457 (7231), 824-829 (2009).
- Barrick, J. E., Lenski, R. E. Genome dynamics during experimental evolution. Nature Reviews Genetics. 14 (12), 827-839 (2013).
- Elena, S. F., Lenski, R. E. Evolution experiments with microorganisms: The dynamics and genetic bases of adaptation. Nature Reviews Genetics. 4 (6), 457-469 (2003).
- Lenski, R. E., Rose, M. R., Simpson, S. C., Tadler, S. C. Long-Term Experimental Evolution in Escherichia coli. I. Adaptation and Divergence During 2,000 Generations. The American Naturalist. 138 (6), 1315-1341 (1991).
- Bennett, A. F., Dao, K. M., Lenski, R. E. Rapid evolution in response to high-temperature selection. Nature. 346 (6279), 79-81 (1990).
- Bell, G. Evolutionary rescue and the limits of adaptation. Philosophical Transactions of the Royal Society B-Biological Sciences. 368 (1610), (2013).
- Bell, G., Gonzalez, A. Adaptation and Evolutionary Rescue in Metapopulations Experiencing Environmental Deterioration. Science. 332 (6035), 1327-1330 (2011).
- Bell, G., Gonzalez, A. Evolutionary rescue can prevent extinction following environmental change. Ecology Letters. 12 (9), 942-948 (2009).
- San Millan, A., Escudero, J. A., Gifford, D. R., Mazel, D., MacLean, R. C. Multicopy plasmids potentiate the evolution of antibiotic resistance in bacteria. Nature Ecology & Evolution. 1, 0010 (2016).
- Alekshun, M. N., Levy, S. B. Molecular mechanisms of antibacterial multidrug resistance. Cell. 128 (6), 1037-1050 (2007).
- Ochman, H., Lawrence, J. G., Groisman, E. A. Lateral gene transfer and the nature of bacterial innovation. Nature. 405 (6784), 299-304 (2000).
- Carattoli, A. Plasmids and the spread of resistance. Int J Med Microbiol. 303 (6-7), 298-304 (2013).
- Smillie, C., Garcillán-Barcia, M. P., Francia, M. V., Rocha, E. P., de la Cruz, F. Mobility of plasmids. Microbiol Mol Biol Rev. 74 (3), 434-452 (2010).
- San Millan, A., Heilbron, K., Maclean, R. C. Positive epistasis between co-infecting plasmids promotes plasmid survival in bacterial populations. ISME J. , (2013).
- Stoesser, N., et al. Evolutionary History of the Global Emergence of the Escherichia coli Epidemic Clone ST131. MBio. 7 (2), (2016).
- San Millan, A., et al. Multiresistance in Pasteurella multocida is mediated by coexistence of small plasmids. Antimicrob Agents Chemother. 53 (8), 3399-3404 (2009).
- Escudero, J. A., et al. Unmasking the ancestral activity of integron integrases reveals a smooth evolutionary transition during functional innovation. Nat Commun. 7, 10937 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc Natl Acad Sci U S A. 97 (12), 6640-6645 (2000).
- Chaveroche, M. K., Ghigo, J. M., d'Enfert, C. A rapid method for efficient gene replacement in the filamentous fungus Aspergillus nidulans. Nucleic Acids Res. 28 (22), 97 (2000).
- Le Roux, F., Binesse, J., Saulnier, D., Mazel, D. Construction of a Vibrio splendidus mutant lacking the metalloprotease gene vsm by use of a novel counterselectable suicide vector. Appl Environ Microbiol. 73 (3), 777-784 (2007).
- . . Performance standards for antimicrobial susceptibility testing; 19th ed. Approved standard M100-S19. , (2009).
- Lindsey, H. A., Gallie, J., Taylor, S., Kerr, B. Evolutionary rescue from extinction is contingent on a lower rate of environmental change. Nature. 494 (7438), 463-467 (2013).
- Deatherage, D. E., Barrick, J. E. Identification of mutations in laboratory-evolved microbes from next-generation sequencing data using breseq. Methods Mol Biol. 1151, 165-188 (2014).
- Barrick, J. E., et al. Identifying structural variation in haploid microbial genomes from short-read resequencing data using breseq. BMC Genomics. 15, 1039 (2014).
- Salverda, M. L., De Visser, J. A., Barlow, M. Natural evolution of TEM-1 β-lactamase: experimental reconstruction and clinical relevance. FEMS Microbiol Rev. 34 (6), 1015-1036 (2010).
- Ramirez, M. S., Tolmasky, M. E. Aminoglycoside modifying enzymes. Drug Resist Updat. 13 (6), 151-171 (2010).
- Jerison, E. R., Desai, M. M. Genomic investigations of evolutionary dynamics and epistasis in microbial evolution experiments. Current Opinion in Genetics & Development. 35, 33-39 (2015).
- Toll-Riera, M., San Millan, A., Wagner, A., MacLean, R. C. The Genomic Basis of Evolutionary Innovation in Pseudomonas aeruginosa. PLoS Genet. 12 (5), 1006005 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved