Method Article
Testen die Rolle des Multicopy Plasmide in der Entwicklung von Antibiotika-Resistenzen
In diesem Artikel
Zusammenfassung
Hier präsentieren wir eine experimentelle Methode, um die Rolle des multicopy Plasmide in der Entwicklung von Antibiotika-Resistenzen zu testen.
Zusammenfassung
Multicopy Plasmide sind sehr reichlich in Prokaryoten, aber ihre Rolle in der bakteriellen Evolution bleibt schlecht verstanden. Vor kurzem zeigten wir, dass der Anstieg gen Kopienzahl pro Zelle von multicopy Plasmide zur Verfügung gestellt die Entwicklung der Plasmid-kodierte Gene beschleunigen könnte. In dieser Arbeit präsentieren wir ein experimentelles System um zu testen, die Fähigkeit des multicopy Plasmide gen Entwicklung zu fördern. Mit einfachen molekularbiologische Methoden, errichteten wir ein Modellsystem, wo eine Antibiotika-Resistenz-Gen in Escherichia coli MG1655, in das Chromosom oder auf ein multicopy Plasmid eingefügt werden können. Wir verwenden einen experimentelle Evolution Ansatz, um die verschiedenen Stämme unter steigenden Konzentrationen von Antibiotika zu propagieren und Überleben der Bakterienpopulationen im Laufe der Zeit messen. Die Wahl der Antibiotika Molekül und die Resistenz-Gen ist, dass das Gen nur Widerstand durch den Erwerb von Mutationen verleihen kann. Dieser "evolutionären Rettung" Ansatz bietet eine einfache Methode um zu testen, das Potenzial des multicopy Plasmide, die Übernahme von Antibiotika-Resistenzen zu fördern. Im nächsten Schritt des experimentellen Systems zeichnen sich die molekularen Grundlagen der Antibiotika-Resistenz. Um Mutationen zu identifizieren verantwortlich für den Erwerb von Antibiotika-Resistenzen verwenden wir Tiefe DNA-Sequenzierung von Proben aus ganzer Bevölkerungen und Klone. Schließlich, um die Rolle der Mutationen im Gen unter Studie zu bestätigen, wir im elterlichen Hintergrund zu rekonstruieren und Widerstand Phänotyp der daraus resultierenden Belastungen zu testen.
Einleitung
Antibiotika-Resistenz bei Bakterien ist ein großes gesundheitliches Problem1. Auf einer grundlegenden Ebene ist die Ausbreitung von Antibiotikaresistenzen bei pathogenen Bakterien ein einfaches Beispiel der Evolution durch natürliche Selektion2,3. Einfach ausgedrückt, generiert die Anwendung von Antibiotika Auswahl für resistente Stämme. Ein zentrales Problem in der Evolutionsbiologie, deshalb, um die Faktoren zu verstehen, die Einfluss auf die Fähigkeit der Bakterienpopulationen zu Resistenzen gegen Antibiotika entwickeln. Auswahl Experimente entstanden als ein sehr mächtiges Werkzeug um die Evolutionsbiologie von Bakterien zu untersuchen hat, und dieses Feld unglaubliche Einblicke in eine Vielzahl von evolutionären Probleme4,5,6. In experimentelle Evolution sind seriell Bakterienpopulationen initiiert von einem einzigen elterlichen Belastung unter definierten und streng kontrollierten Bedingungen passagiert. Einige Mutationen, die während des Wachstums dieser Kulturen auftreten bakterieller Fitness steigern und diese zu verbreiten durch die Kulturen durch natürliche Selektion. Während des Experiments sind Proben der Bevölkerungen in regelmäßigen Abständen tiefkalt beibehalten, um nicht weiterentwickelnden gefrorenen fossilen aufzuzeichnen. Eine Vielzahl von Ansätzen lässt sich entwickelnden Bakterienpopulationen zu charakterisieren, aber die zwei am häufigsten verwendeten Methoden sind Fitness-Assays, die messen die Fähigkeit der weiterentwickelten Bakterien konkurrieren gegen ihre Vorfahren und kompletten Genoms, das ist verwendet, um den genetischen Veränderungen, dass Laufwerk Anpassung identifizieren. Nach Pionierarbeit von Richard Lenski und Kollegen7,8, der Standardansatz in experimentelle Evolution wurde gegen eine relativ kleine Anzahl von replizieren Bevölkerungen (in der Regel < 10) mit der Anpassung an eine neue umweltpolitische Herausforderung, wie neue Kohlenstoffquellen, Temperatur oder eine räuberische Phagen.
Infektionen durch Antibiotika-resistente Bakterien werden ein großes Problem, wenn Widerstand hoch genug ist, dass es nicht möglich, Antibiotika-Konzentrationen zu tödlichen Niveaus in Patienten Gewebe zu erhöhen. Ärzte sind daher interessiert was ermöglicht Bakterien Resistenz gegen hohe Dosen von Antibiotika zu entwickeln, die über diese Antibiotika Grenzkonzentration, der klinischen Haltepunkt liegen. Wie um dies experimentell zu studieren? Wenn eine kleine Anzahl von Bakterienpopulationen mit einer hohen Dosis des Antibiotikums herausgefordert werden, wie in einem Lenski-Stil Experiment, dann das wahrscheinlichste Ergebnis ist, dass das Antibiotikum alle Bevölkerungen zum Aussterben führen wird. Zur gleichen Zeit wenn die Dosis des Antibiotikums, die verwendet wird niedrig, unterhalb der minimalen inhibitorischen Konzentration (MIC) des elterlichen Stammes ist ist dann es unwahrscheinlich, dass sich die Bakterienpopulationen klinisch relevante Ebenen des Widerstandes, vor allem, wenn Widerstand trägt eine große Kosten. Ein Kompromiss zwischen diesen beiden Szenarien ist eine "evolutionäre Rettung" Experiment9,10,11zu verwenden. Dieser Ansatz bietet eine sehr große Anzahl von Kulturen (in der Regel > 40) ist mit Dosen von Antibiotika, die im Laufe der Zeit in der Regel erhöhen durch eine Verdoppelung der Antibiotika Konzentration jeden Tag12gefordert. Das Markenzeichen dieses Experiments ist, dass eine Bevölkerung, die nicht erhöhten Resistenz entwickelt zum Aussterben getrieben wird. Die meisten Populationen, die auf diese Weise in Frage gestellt werden werden ausgestorben getrieben, aber eine kleine Minderheit bleibt bestehen, durch die sich verändernden hohes Maß an Widerstand. In diesem Beitrag zeigen wir Verwendung von dieser Versuchsanordnung multicopy Plasmid Beitrag zur Evolution des Widerstands zu untersuchen.
Bakterien resistent gegen Antibiotika durch zwei Hauptrouten, chromosomalen Mutationen und Akquisition von mobile genetische Elemente, vor allem Plasmide13. Plasmide spielen eine Schlüsselrolle in der Entwicklung von Antibiotika-Resistenzen, weil sie sich Resistenzgene zwischen Bakterien durch Konjugation14,15übertragen können. Plasmide können in zwei Gruppen entsprechend ihrer Größe und Biologie unterteilt werden: "klein", mit hohen Kopienzahl pro Bakterienzelle und "large", mit niedrigen kopieren Nummer16,17. Die Rolle der großen Plasmide in der Entwicklung von Antibiotika-Resistenzen ist ausführlich dokumentiert worden, weil sie konjugative Plasmide enthalten die wichtigsten Triebkräfte für die Verbreitung der Resistenz und Multi-Resistenz Bakterien15sind. Kleinen multicopy Plasmide sind ebenfalls sehr häufig in Bakterien17,18, und sie oft code für Antibiotika-Resistenz-Gene-19. Allerdings ist die Rolle des kleinen multicopy Plasmide in der Entwicklung von Antibiotika-Resistenzen in geringerem Umfang untersucht worden.
In einer neueren Arbeit haben wir vorgeschlagen, multicopy Plasmide die Entwicklung der Gene tragen sie durch die Erhöhung gen Mutationsraten aufgrund der höheren gen Kopienzahl pro Zelle12beschleunigen könnte. Mit einem experimentellen Modell mit E. Coli -Stamm MG1655 und β-Lactamase-gen BlaTEM-1 zeigte sich, dass multicopy Plasmide beschleunigt die Rate des Auftretens von TEM-1 Mutationen verleihen Resistenz gegen die dritte generation Cephalosporin Ceftazidim. Diese Ergebnisse zeigten, dass multicopy Plasmide eine wichtige Rolle bei der Entwicklung von Antibiotika-Resistenzen spielen könnte.
Hier präsentieren wir eine detaillierte Beschreibung der Methode wir entwickelt haben, um die multicopy Plasmid-vermittelten Entwicklung von Antibiotika-Resistenzen zu untersuchen. Diese Methode hat drei Stufen: Erstens Einfügen des Gens untersuchten in einem multicopy Plasmid oder das Chromosom des Bakteriums Host. Zweitens der experimentelle Evolution (evolutionäre Rettung) verwenden Sie, um das Potenzial der verschiedenen Stämme zur Anpassung an den Selektionsdruck zu beurteilen. Und drittens, Bestimmung der molekularen Grundlagen Plasmid-vermittelten Evolution mit der DNA Sequenzierung und die Rekonstruktion der vermuteten Mutationen einzeln in das elterliche Erbgut zugrunde.
Schließlich, obwohl das hier beschriebene Protokoll entwickelt wurde, um die Entwicklung von Antibiotika-Resistenzen zu untersuchen, kann man argumentieren, dass diese Methode in der Regel sinnvoll, die Entwicklung von Innovationen, die durch Mutationen in jedem Multicopy erworben zu analysieren sein könnte Plasmid-kodierte gen.
Protokoll
1. Aufbau des experimentellen Systems Codierung Antibiotikaresistenz-gen
Hinweis: Hier E. Coli MG1655 diente als der Empfänger Belastung der Antibiotikaresistenz-gen Plasmid oder Chromosom kodiert. Antibiotikaresistenz-gen ist das Chromosom oder ein multicopy Plasmid in eine ansonsten isogene Sorte (Abbildung 1) kodiert.
-
Einfügen von Antibiotikaresistenz-gen in der λ Phage Integration Website (AttB)20 des Chromosoms MG1655.
- 500 bp-lang-Regionen auf beiden Seiten des Standortes chromosomalen AttB mittels PCR zu verstärken. Verwenden Sie Oligonukleotide YbhC-F (5'-CCTGTACCGTACAGAGTAAT-3 ") und AttB-R (5'-GCCCGCCACCCTCCGGGCCGGTATAAAAAAGCAGGCTTCA-3") für die linke Homologie Region und AttB-F (5'-AGCGCCCTAGCGCCCGCTCCTTATACTAACTTGAGCGAAA-3 ") und YbhB/R (5'- TGGCGATAATATTTCACCGC-3 ") für die richtige ist. Verstärken die Bla-TEM-1 -Resistenz-Gen mit Primer Tem1-Madothertrucker-F (5'-TGAAGCCTGCTTTTTTATACCGGCCCGGAGGGTGGCGGGC-3 ") und Tem1-Madothertrucker-R (5'-TTTCGCTCAAGTTAGTATAAGGAGCGGGCGCTAGGGCGCT-3"). Design-Primer mit ca. 20 bp (in 5'-Ende) der Sequenz zeigt Komplementarität auf das Fragment, das mit verschmolzen werden soll.
- Sicherung der Homologie Regionen das Antibiotikaresistenz-gen-PCR-Produkt (einschließlich der Promotor-Region) mit Isothermen Montage21, bei 50 ° C für 30 Minuten.
- Electroporate MG1655 Zellen enthaltenden Plasmid pKOBEG.
Hinweis: pKOBEG ist ein wärmeempfindliches Vektor, der die λ enthält rote Maschinen, Homologie-basierte allelische Austausch zwischen dem Chromosom und PCR Produkte22zu fördern.- Verwenden Sie 1 µL des Produkts von Schritt 1.1.223 in 40 µL der Electrocompetent Zellen in 2 mm-Küvetten bei 4 ° C und 2,5 kV. Aufschwemmen Zellen in 1 mL LB Brühe + 0,2 % Arabinose weiterhin Ausdruck der λ roten Maschinen.
- Übertragen Sie das Gesamtvolumen auf einem Microfuge Rohr und inkubieren Sie 2 h bei 30 ° C (permissive Temperatur für pKOBEG) mit intensivem schütteln (200 u/min in einem Orbitalschüttler) um für die phänotypische Ausprägung der Antibiotikaresistenz-Markern eingefügt in das Chromosom zu ermöglichen.
- Platte die Zellen auf LB-Agar, enthält das entsprechende Antibiotikum für die Antibiotika-Resistenz-Gen (Ampicillin oder Carbenicillin bei 100 mg/l für BlaTEM-1) auswählen.
- Platte 100 µL auf eine Petrischale und spin-down der Rest des Bandes, in 100 µL frisches LB Brühe aufschwemmen und auf einem anderen Petrischale Platte. Inkubation über Nacht bei 42 ° C.
Hinweis: Diese Temperatur ist für die Replikation von pKOBEG nicht freizügig. Unter diesen Bedingungen wachsen die Kolonien wird pKOBEG verloren haben und präsentieren das Resistenzgen AttB Website integriert (der Einfachheit halber wird diese Sorte genannt werden MG1655::ResA ab hier). - Test für den Verlust von pKOBEG durch die Replikation der Kolonien des Interesses an Platten mit Chloramphenicol (Antibiotikum gegen die pKOBEG enthält einen Widerstand-Marker).
Hinweis: Fehlende Wachstum in Chloramphenicol Platten bei der freizügigen Temperatur von 30 ° c ist bezeichnend für das Fehlen des Plasmids. - Um zu überprüfen, Klone, PCR verstärken den Bau mit externen Primer YbhC-Extern (5'-TTTGTGACCAGAAGACCGCA-3 ") und YbhB-Extern (5'-CTCATCAGTAACGATCTGCG-3"), überprüfen durch Gelelektrophorese die richtige Größe der Amplifikate und Sequenz der gereinigtes Produkt um sicherzustellen, dass die Sequenz des eingefügten Gens korrekt ist.
-
Legen Sie das Antibiotikaresistenz-gen in ein multicopy Plasmid.
Hinweis: Da Plasmide mit sehr hohen Kopienzahl eine weitgehende Reduzierung in bakteriellen Host Fitness zu verhängen tendenziell, empfehlen wir die Verwendung von multicopy Plasmide mit natürlichen Ursprungs der Replikation, z. B. p3655 (pSU18T-Madothertruckergfp2, ColE1-Typ Ursprung der Replikation-24). Diese Plasmide präsentieren in der Regel eine moderate Exemplarzahl von ca. 15-20 Kopien pro Zelle.- PCR verstärken das Antibiotikaresistenz-gen der Wahl (einschließlich der Promotor-Region), das PCR-Produkt phosphorylieren und verbinden es mit dem PCR verstärkt Backbone des Plasmids:
- PCR verstärken das Antibiotikaresistenz-gen; für BlaverwendenTEM-1 Primer Tem1-Madothertrucker-F (5'-TGAAGCCTGCTTTTTTATACCGGCCCGGAGGGTGGCGGGC-3 ") und Tem1-Madothertrucker-R (5'-TTTCGCTCAAGTTAGTATAAGGAGCGGGCGCTAGGGCGCT-3").
- Phosphorylieren Sie das gereinigte Produkt mit T4-Polynukleotid-Kinase, nach Anweisungen des Herstellers.
- PCR verstärken das Plasmid Rückgrat mit einem High Fidelity Polymerase und Oligonukleotide Madothertrucker-F (5'-CGTTGATCGGCACGTAAGAG-3 ") und Madothertrucker-R (5'-AAACGACGGCCAGTGCCAAG-3").
- Verbinden Sie beide PCR-Fragmente mit T4-Ligase nach Richtlinien des Herstellers.
- Electroporate, wie zuvor beschrieben 40 µL Electrocompetent E. Coli DH5-α-Zellen mit einem Maximum von 1 µL der Ligatur Produkt und wählen Sie auf der entsprechenden Antibiotika für die Antibiotika-Resistenz-Gen (Ampicillin oder Carbenicillin bei 100 mg/L für BlaTEM-1) und das Plasmid Rückgrat.
Hinweis: Ergänzung LB mit Arabinose hier ist nicht erforderlich. - Extrahieren Sie das Plasmid mit dem kommerziellen Mini-Prep Kit von der Leserwahl:
- 5 mL einer Übernacht-Kultur durch Zentrifugieren bei 12.000 x g bei Raumtemperatur zu ernten. Zellen in 250 µL Wiederfreisetzung Lösung aufschwemmen und gut mischen. 250 µL Lyse-Lösung hinzufügen und gut verrühren. 350 µL der Neutralisierung hinzufügen und gut verrühren.
- Den überstand an der DNA-Bindung Spalte zur Verfügung gestellt mit dem Kit, Zentrifuge für 1 min übertragen und entsorgen der Durchströmung. Waschen Sie zweimal die Spalte mit 500 µL der Waschlösung, eine Minute lang Zentrifugieren und abwerfen der Bestrahlungskammer bei jedem Waschgang. Zentrifugieren der leeren Spalte 1 zusätzliche min., restliche Wäsche Puffer zu entfernen.
- Setzen Sie die Säule in ein sauberes Röhrchen und die Membran auf die gereinigte DNA eluieren fügen Sie 50 µL Ultrareines Wasser hinzu. Zentrifuge für 1 – 2 min. und sammeln die Durchströmung, gereinigte Plasmid enthält. Sanger Sequenz das Gen des Interesses in das Plasmid mit Primer von außerhalb der eingefügte Sequenz um zu bestätigen, dass die Reihenfolge richtig ist und dass keine Mutationen in der Resistenz-Gen vor der Evolution Experimente vorhanden sind.
- Sobald die Sequenz verifiziert ist, Electroporate das Plasmid in den MG1655 E. Coli -Stamm, wie oben beschrieben.
Hinweis: Dies ergibt Stamm MG1655/pRESA.
- PCR verstärken das Antibiotikaresistenz-gen der Wahl (einschließlich der Promotor-Region), das PCR-Produkt phosphorylieren und verbinden es mit dem PCR verstärkt Backbone des Plasmids:
(2) evolutionären Rettung Ansatz zu experimentell entwickeln Resistenz gegen Antibiotika (Abbildung 1)
- Streifen Sie die Stämme unter Studie auf LB-Platten: MG1655::ResA und MG1655/pRESA plus anfällig elterlichen Belastung, MG1655.
Hinweis: Plasmid pRESA sollte in MG1655, stabil sein, so gibt es keine Notwendigkeit, Antibiotika, die LB-Platten hinzuzufügen. Mutationsraten in E. Coli sind niedrig genug, so sobald die Konstruktion verifiziert ist es nicht notwendig, Plasmid-Reihenfolge bei jedem Schritt zu überprüfen. - 96-Well-Platten mit 200 µL LB in jedes gut vorbereiten und 48 isolierte Kolonien von jedem Stamm in unabhängige Brunnen (eins pro Stamm) zu impfen. Inkubieren Sie die Platten über Nacht bei 37 ° C und 200 u/min. Halten Sie einen gefrorenen Vorrat dieser Gründer-Populationen.
Hinweis: Zur Vorbeugung und Kontrolle für Kultur Cross-Kontamination in 96-Well-Platten, verwenden Sie ein Schachbrettmuster Platte von verschuppen beimpfte Brunnen mit bakterienfreies Medium während der 96-Well-Platte. Verwenden Sie diese Plattendesign während der gesamten experimentelle Evolution. - Zunächst das evolutionäre Rettung Experiment Impfkeimen 2 µL aus jedem Brunnen der Platten mit der Gründer-Populationen in neue 96-Well-Platten mit 198 µL LB in jede Vertiefung mit einem sub-inhibitory Konzentration des Antibiotikums unter Studie. Für den evolutionären Rettung Ansatz beginnen mit ¼ oder 1/8 der minimalen inhibitorischen Konzentration (MIC, bestimmt vorher25) des Antibiotikums in LB für jede der drei Stämme und die Konzentration der Antibiotika täglich verdoppeln. Verwenden Sie dieses Ansatz, um die Chancen der Bevölkerung Widerstand Mutationen26erwerben zu maximieren. Inkubieren Sie die Platten bei 37 ° C und 200 u/min für 20 h.
Hinweis: Messen der Nacht OD der Gründer Bevölkerungen. Wenn es erhebliche Unterschiede im OD unter Stämmen gibt korrigieren Sie das erste Inokulum um das Experiment mit der gleichen Anzahl der Zellen pro Bohrloch zu beginnen. - Jeden Tag Messen Sie OD jeder Bevölkerung nach Übernachtung Kultur und führen Sie eine Übertragung der Kulturen, wie unter 2.3, um neue 96-Well-Platten mit doppelt die Konzentration des Antibiotikums als am Vortag. Inkubieren Sie die Platten 20 h bei 37 ° C und 200 u/min.
Hinweis: 1: 100 Verdünnungsfaktor produziert ca. 6-7 Generationen pro Tag. - Gleichzeitig verbreiten Sie Kontrolle Populationen von jedem Stamm unter den gleichen Bedingungen wie beschrieben in 2.3 und 2.4, aber in Ermangelung von Antibiotika.
Hinweis: Kontrolle Bevölkerung hilft Mutationen entstehen aufgrund der Anwesenheit von Antibiotika und allgemeine Mutationen helfen Bakterien zur Anpassung an die experimentellen Bedingungen unterscheiden. Parallele Mutationen entstehen in der Abwesenheit von Antibiotika sind wahrscheinlich zu sein hilft Bakterien zur Anpassung an die experimentellen Bedingungen und nicht im Zusammenhang mit Antibiotika-Resistenz. - Verfolgen Sie die Anzahl der Überlebenden Bevölkerung jeden Tag durch die Messung der Extinktion bei einer Wellenlänge von 600 nm (OD) der Kulturen mit einem Teller-Reader.
Hinweis: Optische Dichtewerte niedriger als 0,1 das Aussterben der Bevölkerung zeigen. Siehe Abbildung 2 ein Beispiel für Überleben Kurven. - Halten Sie einen gefrorenen Vorrat aller Bevölkerungsgruppen in regelmäßigen Abständen (alle 3 bis 5 Tage).
- Nutzen Sie Log-Rank Tests [Paket "Überleben" in RStudio (Version 0.99.486)], um statistische Unterschiede für das Überleben der Bevölkerung der verschiedenen Stämme im Laufe der Zeit unter zunehmender Konzentration der Antibiotika-12zu bestimmen.
Hinweis: Dieses Experiment wird entscheiden, ob multicopy Plasmide die Entwicklung von Antibiotika-Resistenzen für bestimmtes Antibiotikum und gen unter Studie potenzieren.
3. Molekulare Grundlagen der Entwicklung von Antibiotika-Resistenzen (Abbildung 1)
- Durchführen Sie Gesamt DNA-Extraktion aus einer repräsentativen Anzahl von Populationen und Klone über Dehnungen und Behandlungen. Gehören die elterliche Stämme (MG1655, MG1655::ResA und MG1655/pRESA), die Mutationen ansammeln während des Experiments erkennen zu können.
Hinweis: Beispiele für DNA-Extraktion-Kits finden Sie in Tabelle von Materialien. Jedes Set hat verschiedene Protokolle; Befolgen Sie die Anleitungen des Herstellers. - Quantifizierung von DNA-Qualität und Konzentration. Es gibt verschiedene Methoden, um DNA-Qualität und Konzentration bestimmen. Bestimmen Sie die Qualität, die die Verhältnisse der Absorption 260 nm/280 nm und 260 nm/230 nm zu messen. Verwenden Sie einen Fluoreszenzfarbstoff DNA-Bindung, um DNA-Konzentration nach Anweisungen des Herstellers zu messen, und Agarose-Gelelektrophorese zu bestätigen, dass es ist keine Schädigung der DNA oder RNA Verschmutzung.
- Verwendung Tiefe DNA-Sequenzierung von Proben der gesamten Bevölkerung und individuelle Klone, um die genetische Basis der Antibiotikaresistenz zu untersuchen.
Hinweis: Wir haben alle Sequenzierung am Wellcome Trust Centre for Human Genetics, Oxford, UK. Für weitere Details siehe San Millan Et al. 2016-12. - Verwenden Sie die Breseq 0.26.1 Pipeline27,28 , um Mutationen, mit Polymorphismus-Modus, um die Häufigkeit von Mutationen in der Bevölkerung zu schätzen erkennen. Vergleichen Sie die verschiedenen weiterentwickelten Genome auf der elterlichen Genom Mutationen zu erkennen, die während des Experiments angesammelt haben.
- Vergleichen Sie die Mutationen parallel in der Steuerelement-Populationen mit denen der Bevölkerung gesammelt entwickelt unter zunehmender Konzentration der Antibiotika zu unterscheiden helfen Bakterien zur Anpassung an die allgemeine Versuchsbedingungen (Mutationen diejenigen in der Kontrolle Populationen gefunden) und Akteure der Antibiotika-Resistenz (die ausschließlich in Populationen Antibiotika Druck ausgesetzt).
Hinweis: Die Anzahl der parallelen Mutationen ist in der Regel gering, verschiedene parallele Mutationen zwischen den Behandlungen ganz einfach bewerten zu können. Abgesehen von Mutationen in der Antibiotikaresistenz-gen unter Studie, ist es sehr wahrscheinlich zu finden andere Mutationen im Zusammenhang mit Antibiotika-Resistenz in das Chromosom, solche Mutationen in porinen oder Ausfluss Systeme12. - Die Mutationen im Antibiotika-Resistenz-Gen in der elterlichen Belastung mit dem gleichen Ansatz wie in Abschnitt 1 dieses Protokolls zu rekonstruieren. Befolgen Sie die Punkte 1.1 und 1.2 des Protokolls, die neuen Mutationen im elterlichen Hintergrund (mit der weiterentwickelten Gene als Kopiervorlage PCR) einzuführen. Analysieren Sie die Antibiotika-Resistenz-Phänotypen dieser neue Konstruktionen, die Rolle der Mutationen zu bestätigen.
Ergebnisse
In unserer bisherigen Arbeit wurde die Entwicklung der β-Lactamase-gen BlaTEM-1 in Richtung verleiht Resistenz gegen die dritte Generation Cephalosporin Ceftazidim12 untersucht. Dieses Gen wurde ausgewählt, weil obwohl TEM-1 keine Resistenz gegenüber Ceftazidim verleihen, Mutationen im BlaTEM-1 Aktivität von TEM-1, Cephalosporine wie Ceftazidim29hydrolyseneigung erweitern können. Mutationen im Antibiotika-Resistenz-Enzyme wie β-Lactamases oder aminoglykosid Änderung Enzyme führt zu Veränderungen in ihren Tätigkeitsbereich sind gemeinsame29,30. Dieses experimentelle System ist ideal, um die Entwicklung dieser Art von Enzymen zu erkunden. Finden Sie für einen detaillierten Bericht über ein erfolgreiches Experiment nach diesem Protokoll San Millan Et Al. 2016-12.
Hier ist ein Beispiel für die möglichen Ergebnisse des experimentellen Systems präsentiert zur Veranschaulichung des Protokolls (Beachten Sie, dass die Daten für dieses Beispiel verwendet nicht real). Um zu untersuchen, die potenzielle Rolle von Mehrfachkopie Plasmide in der Entwicklung von Antibiotika-Resistenz-Gen unter studieren in diesem Beispiel (nennen wir es ResA), entwickeln wir das experimentelle System nach Abschnitt 1 des genannten Protokolls. Die Experimente produzieren drei Stämme: MG1655, MG1655::ResA und MG1655/pRESA. Die Entwicklung von Resistenzen zu zwei verschiedenen ß-Lactam-Antibiotika (Ceftazidim und Meropenem) wurde getestet, die beschriebenen Schritte in Abschnitt 2 des Protokolls. Abbildung 2 zeigt die überleben Kurven der Bevölkerung untersucht. In diesem Beispiel gibt es ein deutlichen Anstieg das Überleben der Bevölkerung zu MG1655/pRESA entwickelt sich in Ceftazidim im Vergleich zu denen von MG1655 oder MG1655 gehören::ResA (Log-Rank-Test, P< 0,05). Auf der anderen Seite, im Falle von Meropenem, gibt es keine signifikanten Unterschiede für das Überleben der Bevölkerung gehören zu den verschiedenen Klängen (Log-Rank-Test, P> 0.05). Daher diese Ergebnisse deuten darauf hin, dass das Vorhandensein des Gens ResA in ein multicopy Plasmid die Entwicklung von Resistenzen Ceftazidim aber nicht Meropenem potenziert.
Im letzten Schritt des Experiments die Molekulare Grundlagen der Antibiotika-Resistenz untersucht wird, wie in Abschnitt 3 des Protokolls erklärt. Erstens wird DNA-Sequenzierung die Mutationen in ResA offenbaren, die für den Widerstand Phänotyp verantwortlich sein könnte. Und zweitens: Wiederaufbau von ResA Mutationen in den elterlichen MG1655 (sowohl im Chromosom und Plasmid) bestätigen oder verwerfen Sie ihre Rolle in der Antibiotika-Resistenz-Phänotyp.
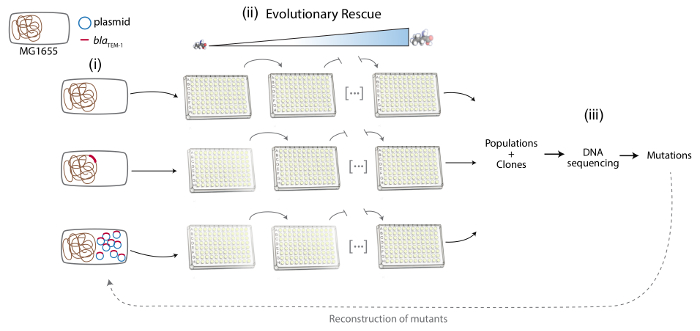
Abbildung 1 . Schematische Darstellung der verschiedenen Phasen des Protokolls. Von links nach rechts: (i) Aufbau des experimentellen Systems: MG1655, MG1655::ResA und MG1655/pRESA. Bakterielle Chromosom ist in braun, das Plasmid in blau und das ResA -gen in rot dargestellt. (Ii) evolutionären Rettung Ansatz zu experimentell Antibiotikaresistenz entwickeln: mehrere Populationen der verschiedenen Sorten sind unter zunehmender Konzentration des Antibiotikums propagiert. (Iii) die Analyse der molekularen Grundlagen von Antibiotika-Resistenzen: Sequenzierung von DNA-Proben aus dem weiterentwickelten Populationen und Klone, Entdeckung der Antibiotika-Resistenz-Mutationen und Rekonstruktion dieser Mutationen in der elterlichen Belastung. Bitte klicken Sie hier für eine größere Version dieser Figur.
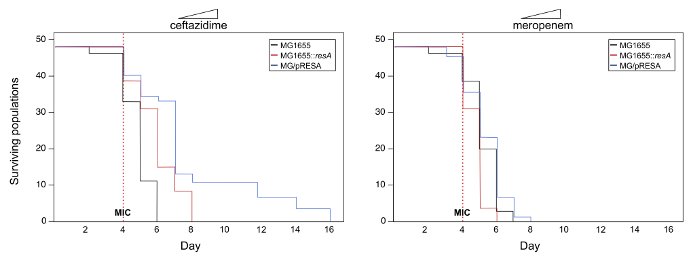
Abbildung 2. Überleben Kurven mit steigenden Konzentrationen von Antibiotika. Darstellung der Anzahl der lebensfähige Populationen gehören Stämme MG1655, MG1655::ResAund MG1655/pRESA im Laufe der Zeit. 48 Populationen von jedem Stamm wurden verbreitet unter steigenden Konzentrationen von Antibiotika Ceftazidim und Meropenem, beginnend mit 1/8 des MIC am Tag 1 und Verdoppelung der Antibiotika Konzentration täglich. Die rote gestrichelte senkrechte Linie repräsentiert das MIC von Antibiotika untersucht. Beachten Sie, dass im Fall von Ceftazidim gibt es signifikante Unterschiede für das Überleben der Bevölkerung im Laufe der Zeit zu verschiedenen Stämmen gehören (Log-Rank-Test, P< 0,05). Nur Populationen tragen das Plasmid sind in der Lage, bis zu hohe Konzentrationen des Antibiotikums zu überleben. Auf der anderen Seite, im Falle von Meropenem, gibt es keine signifikanten Unterschiede für das Überleben der verschiedenen Populationen im Laufe der Zeit (Log-Rank-Test, P> 0.05). Bitte klicken Sie hier für eine größere Version dieser Figur.
Diskussion
Wir präsentieren ein neues Protokoll, die Kombination von Molekulare Biologie, experimentelle Evolution und tiefen DNA-Sequenzierung, die entworfen, um die Rolle des multicopy Plasmide in der Entwicklung von Antibiotika-Resistenzen bei Bakterien zu untersuchen. Obwohl dieses Protokoll Techniken aus verschiedenen Bereichen verbindet, alle Methoden zu entwickeln sind einfach und in einem regelmäßigen Mikrobiologie-Labor durchgeführt werden können. Die wichtigsten Schritte im Protokoll sind wahrscheinlich den Bau von Modell-System-Stämmen und den Wiederaufbau der Mutationen nach der experimentellen Entwicklung beobachtet (die mit der exakt gleichen Methode durchgeführt werden). Jedoch vereinfacht die Isothermen Montage System21, erheblich dieses Protokoll so dass jeder Benutzer mit einer Zwischenebene Erfahrung im Bereich der Molekularbiologie es umsetzen kann.
Ein weiterer wichtiger Schritt des Protokolls ist die experimentelle Evolution unter steigenden Konzentrationen von Antibiotika. Als Beispiel beginnt dieses Protokoll das Experiment mit ¼-1/8 des MIC der Sorten und dann die Konzentration des Antibiotikums jeden Tag verdoppelt. Allerdings könnte eine niedrigere Rate von Antibiotika Veränderung die Chance auf evolutionäre Rettung vom Aussterben26erhöhen. Daher ist die Änderungsrate der Antibiotika-Konzentrationen einer der Parameter, die geändert werden können, um die Entwicklung von Antibiotika-Resistenzen zu fördern.
DNA-Sequenzierung und Analyse sind ebenfalls wichtige Aspekte des experimentellen Designs. Ergebnisse sind einfacher, wenn Sequenzierung auf DNA-Proben von ganzer Bevölkerungen und von einzelnen Klone zu unterschiedlichen Zeitpunkten im Experiment durchgeführt wird. Sequenzierung Ergebnisse aus Populationen zeigen generelle Unterschiede in der Mutation Profile unter Behandlungen sowie selektive fegt der vorteilhafte Mutationen über Zeit und mögliche Ereignisse klonalen Störquellen. Bei der Analyse der Sequenzen aus Populationen ist es besser, Mutationen zu filtern, die 10 % Frequenz in jeder Bevölkerung nie übertroffen. Sequenzen aus einzelnen Klone helfen, bestätigen die Ergebnisse von Populationen und am wichtigsten ist, zeigen die spezifischen Kombinationen zwischen den verschiedenen Mutationen auf Bevölkerungsebene beobachtet. Diese spezielle Verbände können helfen, epistatic Interaktionen zwischen Mutationen, entdecken Sie die bakterielle Anpassung31eine entscheidende Rolle spielen.
Mit dieser Methode haben wir vor kurzem gezeigt, dass multicopy Plasmide die Entwicklung von Antibiotika-Resistenzen, beschleunigen zunächst, indem Sie die Rate des Auftretens von neuartigen Mutationen und dann verstärken die Wirkung von Mutationen durch erhöhte gen Dosierung12 . Daher kann wir entwickelte die Methode als ein Werkzeug, um die Entwicklung von Antibiotika-Resistenzen zu untersuchen, aber es eine viel breitere Palette von Anwendungen. Nämlich, könnte dieses System verwendet werden, zu untersuchen, die Fähigkeit jedes bakterielle gen in Richtung eine neue oder verbesserte Funktion in allgemeinerer Form entwickeln. Dieses System könnte verwendet werden, um die Fähigkeit des Enzyms/Stoffwechselweg, neue Carbon Substrate32verwenden zu testen. Auch, es könnte verwendet werden, anstelle von Hypermutators (Bakterien mit einem Defekt in der zellulären DNA-Mismatch-Reparatur beteiligten Systemen) zu untersuchen, adaptive gen Entwicklung in Bakterien, vermeiden die Mutationszucht Vorspannung von Hypermutators eingeführt.
Offenlegungen
Die Autoren haben nichts preisgeben.
Danksagungen
Diese Arbeit wurde unterstützt durch das Instituto de Salud Carlos III (Plan Estatal de ich + D + ich 2013-2016): gewährt CP15-00012, PI16-00860 und CIBER (CB06/02/0053), kofinanziert durch den Europäischen Fonds für regionale Entwicklung '' einen Weg, um Europa zu erreichen"(EFRE). JAE wird unterstützt durch das Atracción de Talento -Programm der Regierung der Region Madrid (2016-T1/BIO-1105) und die I + D Shmeissani von das spanische Ministerio de Economía, Industria y Competitividad (BIO2017-85056-P). ASM wird unterstützt durch ein Miguel Servet Stipendium vom Instituto de Salud Carlos III (MS15/00012) kofinanziert durch den Europäischen Sozialfonds "Investition in Ihre Zukunft" (ESF) und EFRE.
Materialien
| Name | Company | Catalog Number | Comments |
| Thermocycler | BioRad | C1000 | |
| Electroporator | BiorRad | 1652660 | |
| Electroporation cuvettes | Sigma-Aldrich | Z706078 | |
| NanoDrop 2000/2000c | Thermo Fisher Scientific | ND-2000 | Determine DNA quality measuring the ratios of absorbance 260nm/280nm and 260nm/230nm |
| Incubator | Memmert | UF1060 | |
| Incubator (shaker) | Cole-Parmer Ltd | SI500 | |
| Electrophoresis power supply | BioRad | 1645070 | Agarose gel electrophoresis |
| Electrophoresis chamber | BioRad | 1704405 | Agarose gel electrophoresis |
| Pippettes | Biohit | 725020, 725050, 725060, 725070 | |
| Multi-channel pippetes | Biohit | 728220, 728230, 728240 | |
| Plate reader Synergy HTX | BioTek | BTS1LF | |
| Inoculating loops | Sigma-Aldrich | I8388 | |
| 96-well plates | Falcon | 351172 | |
| LB | BD Difco | DF0446-17-3 | |
| LB agar | Fisher scientific | BP1425-500 | |
| Phusion Polymerase | Thermo Fisher Scientific | F533S | |
| Gibson Assembly | New England Biolabs | E2611S | |
| Resctriction enzymes | Fermentas FastDigest | ||
| Antibiotics | Sigma-Aldrich | ||
| QIAprep Spin Miniprep Kit | Qiagen | 27104 | Plasmid extraction kit |
| Wizard Genomic DNA Purification Kit | Promega | A1120 | gDNA extraction kit |
| DNeasy Blood & Tissue Kits | Qiagen | 69506 | gDNA extraction kit |
| Electroporation cuvettes | Sigma-Aldrich | Z706078 | |
| Petri dishes | Sigma-Aldrich | D9054 | |
| Cryotubes | ClearLine | 390701 | |
| 96-well plates (-80ºC storage) | Thermo Fisher Scientific | 249945 | |
| QuantiFluor dsDNA System | Promega | E2670 | Quantification of DNA concentartion |
| Agarose | BioRad | 1613100 | Agarose gel electrophoresis |
| 50x TAE buffer | BioRad | 1610743 | Agarose gel electrophoresis |
| T4 Polynucleotide Kinase | Thermo Fisher Scientific | EK0031 | |
| T4 DNA Ligase | Thermo Fisher Scientific | EL0014 |
Referenzen
- Neill, J. TACKLING DRUG-RESISTANT INFECTIONS GLOBALLY: FINAL REPORT AND RECOMMENDATIONS. Review on Antimicrobal Resistance. , (2016).
- Palmer, A. C., Kishony, R. Understanding, predicting and manipulating the genotypic evolution of antibiotic resistance. Nat Rev Genet. 14 (4), 243-248 (2013).
- MacLean, R. C., Hall, A. R., Perron, G. G., Buckling, A. The population genetics of antibiotic resistance: integrating molecular mechanisms and treatment contexts. Nat Rev Genet. 11 (6), 405-414 (2010).
- Buckling, A., Maclean, R. C., Brockhurst, M. A., Colegrave, N. The Beagle in a bottle. Nature. 457 (7231), 824-829 (2009).
- Barrick, J. E., Lenski, R. E. Genome dynamics during experimental evolution. Nature Reviews Genetics. 14 (12), 827-839 (2013).
- Elena, S. F., Lenski, R. E. Evolution experiments with microorganisms: The dynamics and genetic bases of adaptation. Nature Reviews Genetics. 4 (6), 457-469 (2003).
- Lenski, R. E., Rose, M. R., Simpson, S. C., Tadler, S. C. Long-Term Experimental Evolution in Escherichia coli. I. Adaptation and Divergence During 2,000 Generations. The American Naturalist. 138 (6), 1315-1341 (1991).
- Bennett, A. F., Dao, K. M., Lenski, R. E. Rapid evolution in response to high-temperature selection. Nature. 346 (6279), 79-81 (1990).
- Bell, G. Evolutionary rescue and the limits of adaptation. Philosophical Transactions of the Royal Society B-Biological Sciences. 368 (1610), (2013).
- Bell, G., Gonzalez, A. Adaptation and Evolutionary Rescue in Metapopulations Experiencing Environmental Deterioration. Science. 332 (6035), 1327-1330 (2011).
- Bell, G., Gonzalez, A. Evolutionary rescue can prevent extinction following environmental change. Ecology Letters. 12 (9), 942-948 (2009).
- San Millan, A., Escudero, J. A., Gifford, D. R., Mazel, D., MacLean, R. C. Multicopy plasmids potentiate the evolution of antibiotic resistance in bacteria. Nature Ecology & Evolution. 1, 0010 (2016).
- Alekshun, M. N., Levy, S. B. Molecular mechanisms of antibacterial multidrug resistance. Cell. 128 (6), 1037-1050 (2007).
- Ochman, H., Lawrence, J. G., Groisman, E. A. Lateral gene transfer and the nature of bacterial innovation. Nature. 405 (6784), 299-304 (2000).
- Carattoli, A. Plasmids and the spread of resistance. Int J Med Microbiol. 303 (6-7), 298-304 (2013).
- Smillie, C., Garcillán-Barcia, M. P., Francia, M. V., Rocha, E. P., de la Cruz, F. Mobility of plasmids. Microbiol Mol Biol Rev. 74 (3), 434-452 (2010).
- San Millan, A., Heilbron, K., Maclean, R. C. Positive epistasis between co-infecting plasmids promotes plasmid survival in bacterial populations. ISME J. , (2013).
- Stoesser, N., et al. Evolutionary History of the Global Emergence of the Escherichia coli Epidemic Clone ST131. MBio. 7 (2), (2016).
- San Millan, A., et al. Multiresistance in Pasteurella multocida is mediated by coexistence of small plasmids. Antimicrob Agents Chemother. 53 (8), 3399-3404 (2009).
- Escudero, J. A., et al. Unmasking the ancestral activity of integron integrases reveals a smooth evolutionary transition during functional innovation. Nat Commun. 7, 10937 (2016).
- Gibson, D. G., et al. Enzymatic assembly of DNA molecules up to several hundred kilobases. Nat Methods. 6 (5), 343-345 (2009).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc Natl Acad Sci U S A. 97 (12), 6640-6645 (2000).
- Chaveroche, M. K., Ghigo, J. M., d'Enfert, C. A rapid method for efficient gene replacement in the filamentous fungus Aspergillus nidulans. Nucleic Acids Res. 28 (22), 97 (2000).
- Le Roux, F., Binesse, J., Saulnier, D., Mazel, D. Construction of a Vibrio splendidus mutant lacking the metalloprotease gene vsm by use of a novel counterselectable suicide vector. Appl Environ Microbiol. 73 (3), 777-784 (2007).
- . . Performance standards for antimicrobial susceptibility testing; 19th ed. Approved standard M100-S19. , (2009).
- Lindsey, H. A., Gallie, J., Taylor, S., Kerr, B. Evolutionary rescue from extinction is contingent on a lower rate of environmental change. Nature. 494 (7438), 463-467 (2013).
- Deatherage, D. E., Barrick, J. E. Identification of mutations in laboratory-evolved microbes from next-generation sequencing data using breseq. Methods Mol Biol. 1151, 165-188 (2014).
- Barrick, J. E., et al. Identifying structural variation in haploid microbial genomes from short-read resequencing data using breseq. BMC Genomics. 15, 1039 (2014).
- Salverda, M. L., De Visser, J. A., Barlow, M. Natural evolution of TEM-1 β-lactamase: experimental reconstruction and clinical relevance. FEMS Microbiol Rev. 34 (6), 1015-1036 (2010).
- Ramirez, M. S., Tolmasky, M. E. Aminoglycoside modifying enzymes. Drug Resist Updat. 13 (6), 151-171 (2010).
- Jerison, E. R., Desai, M. M. Genomic investigations of evolutionary dynamics and epistasis in microbial evolution experiments. Current Opinion in Genetics & Development. 35, 33-39 (2015).
- Toll-Riera, M., San Millan, A., Wagner, A., MacLean, R. C. The Genomic Basis of Evolutionary Innovation in Pseudomonas aeruginosa. PLoS Genet. 12 (5), 1006005 (2016).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten