Method Article
Usando CRISPR/Cas9 Gene edição para investigar a atividade oncogênica do mutante Calreticulin em células hematopoiéticas dependentes de Cytokine
Neste Artigo
Resumo
Gene alvo edição usando CRISPR/Cas9 facilitou muito a compreensão das funções biológicas dos genes. Aqui, nós utilizamos a metodologia CRISPR/Cas9 a mutações de calreticulin modelo em células hematopoiéticas dependentes de citocina para estudar sua atividade oncogênica.
Resumo
Clusterizado regularmente intercaladas curtas palíndromos repetições (CRISPR) é um sistema de imunidade adaptativa em procariontes que tem sido reaproveitado pelos cientistas para gerar nucleases RNA-guiada, tais como CRISPR-associado (Cas) 9 para site-specific do genoma eucariótico edição. Engenharia de genoma por Cas9 é usada eficientemente, facilmente e robustamente modificar genes endógenos em muitas linhas de células de mamíferos biomedically relevantes e organismos. Aqui mostramos um exemplo de como utilizar a metodologia CRISPR/Cas9 para entender a função biológica de mutações genéticas específicas. Usamos modelos calreticulin (CALR) mutações em murino interleucina-3 (3 mIL) dependentes pro-B (Ba/F3) células pela entrega de RNAs de guia única (sgRNAs) visando o locus Calr endógeno na região específica onde inserção e/ou exclusão (indel ) CALR mutações ocorrem em pacientes com Neoplasias mieloproliferativas (MPN), um tipo de câncer de sangue. Os sgRNAs criar quebras da cadeia dupla (DSBs) na região alvo que são reparadas por fim não-homóloga ingressar (NHEJ) para dar puntuais de vários tamanhos. Então, nós empregamos o ensaio de transformação celular Ba/F3 padrão para entender o efeito da expressão de nível fisiológico de Calr mutações na transformação celular hematopoiética. Esta abordagem pode ser aplicada a outros genes para estudar sua função biológica em várias linhas de células de mamíferos.
Introdução
CRISPR/Cas9 tecnologia recentemente revolucionou o genoma alvo edição nos organismos e células vivas. Tornou-se uma ferramenta extremamente poderosa para a investigação biomédica e atualmente está sendo utilizado como uma avenida de potencial para a terapia de doenças genéticas1. A base para todas as ferramentas de edição de genoma baseia-se na criação de uma induzida por nuclease DNA encalhada interrupção dupla (DSB) no locus genômico a ser modificado. Os DSBs podem ser reparados pelo não-homóloga final-adesão (NHEJ) ou reparação de homologia-dirigido (HDR)2,3. A vantagem da nuclease Cas9 sobre outro genoma engenharia nucleases, tais como as nucleases de dedo de zinco (ZFNs) e nucleases efetoras como ativador de transcrição (TALENs) é sua dependência de RNA para o direcionamento da nuclease para uma sequência de DNA desejada, em comparação para as interações proteína-ADN encontradas em ZFNs e TALENs2,3.
Após a descoberta da via de nuclease CRISPR/Cas9 como um sistema imune adaptativo em células procarióticas4,5, muito esforço tem ido para adaptar o caminho para o uso em linhas de células de mamíferos e de organismos modelo2, 3. como uma ferramenta para edição de gene, via CRISPR/Cas9 utiliza dois componentes principais: o Streptococcus pyogenes (Sp) derivado Cas9 nuclease e sgRNAs como alvo o gene de interesse,2,3. O sgRNA consiste de 20 nucleotídeos que Cas9 direto para um site específico sobre o genoma através de pares de base de RNA-DNA a complementaridade2,3. O site de destino do sgRNA devera ser adjacente a um site de motivo adjacentes (PAM) protospacer sob a forma de 5' NGG, que é reconhecido pela nuclease SpCas9. Com estas ferramentas, Cas9 pode ser direcionado para qualquer sequência de DNA através da concepção de sgRNAs destino a região de interesse. Além da Sp derivada Cas9, existem variantes adicionais para Cas9 com características diferentes, dependendo da aplicação específica. Por exemplo, existem variantes de Cas9 com maior especificidade para edição no alvo ou capacidade de clivagem de single-strand DNA roubando6,7. Além disso, recentemente foi desenvolvido Cas9 cataliticamente inactiva por regulamento transcriptional8. Cientistas agora têm usado o sistema CRISPR/Cas9 para uma variedade de aplicações, tais como gene batendo e nocaute para estudar as funções biológicas de genes9, da função de perda e ganho-de-função biblioteca telas10 e genética Engenharia de organismos modelo11.
Neste protocolo, aliamos a metodologia CRISPR/Cas9 com o ensaio de transformação celular Ba/F3 para entender a função biológica do CALR mutações. BA/F3 células são uma linha de células hematopoiéticas dependentes murino IL-3 que pode ser processada IL-3 independente sobre expressão de determinados oncogenes, como BCR-ABL12. A fim de compreender se calreticulin mutante pode transformar células Ba/F3 para crescimento independente de citocina, nós alvo exon 9 do locus endógeno de Calr usando CRISPR/Cas9 para introduzir mutações indel e então IL-3 retirou as células para aplicar um pressão de seleção positiva, com o objetivo de recapitulando mutações de ganho-de-função CALR encontradas em pacientes do MPN. O protocolo inclui o projeto, a clonagem e a entrega de sgRNAs, o desenvolvimento de células expressando de estável Cas9 e triagem para CRISPR gene edição no alvo. Este protocolo pode ser aplicado a diferentes genes e várias linhas de célula dependente de citocinas de interesse e é especialmente valioso em modelagem e estudar a função biológica de genes envolvidos no câncer.
Protocolo
1. sgRNA projeto usando ferramentas on-line13
-
Projeto sgRNAs como alvo o gene de interesse usando ferramentas disponíveis gratuitamente on-line.
- Copie e cole a sequência de referência NCBI do gene de interesse para a ferramenta de web designer do Broad Institute sgRNA: (http://portals.broadinstitute.org/gpp/public/analysis-tools/sgrna-design).
Nota: Esta ferramenta identifica sequências sgRNA com sítios de clivagem dentro exões e aqueles que abrangem a fronteira intron/exon mas ainda cleave dentro o exon. - Download e abrir o arquivo de saída de texto no excel.
- Concentre-se nas colunas preenchidas com as sequências de sgRNA e as sequências de contexto sgRNA. Observe que as sequências de sgRNA não contêm o motivo de protospacer adjacentes (PAM), mas fazer as sequências de contexto.
- Observe que o resultado de eficácia no alvo listas o escore de eficiência clivagem previsto numa escala de 0 a 1, onde uma pontuação de 1 denota uma maior eficiência de clivagem.
- Uso a função de 'tipo' do excel também encomendar os alvos com o placar de eficácia ou pela localização dentro do gene do alvo através da coluna '% corte de destino'.
Nota: Classificação por localização dentro do gene é útil para identificar sgRNAs que se destinam a um exão de interesse ou domínio específico. - Selecione sgRNAs de 3-6 que se destinam a área de interesse com alta (> 0,6) golo de eficácia no alvo. Pode ser útil saber quais os tipos de mutações podem ser responsáveis para o fenótipo que é desejado (por favor consulte a Figura 1 explicando a estratégia de segmentação utilizada para calreticulin).
Nota: sgRNAs abaixo do limiar de Pontuação sugerida 0,6 eficácia deve ser considerada quando existe uma falta de outros bons candidatos. - Use a ferramenta de web análise de gRNA MIT a tela para fora do alvo os efeitos potenciais (http://crispr.mit.edu/). Para cada sgRNA selecionado, execute a sequência de destino (incluindo o PAM).
Nota: A ferramenta de web designer do Broad Institute sgRNA também pode ser usada para a tela para fora do alvo efeitos (http://portals.broadinstitute.org/gpp/public/analysis-tools/sgrna-design).- Observe que a ferramenta de análise de sgRNA MIT pontua cada sgRNA em uma escala de 1-100 onde uma pontuação de 100 denota maior especificidade. Uma nota superior a 70 é ideal e representa um sgRNA com mínimos efeitos fora do alvo. Este site também gera uma lista de hits fora do alvo com sua localização dentro do genoma e quantos mismatches cada um deles tem com o guia do candidato.
Nota: Sucessos fora do alvo com incompatibilidades de quatro ou mais são considerados seguros14. O alvo hits com incompatibilidades de menos de quatro podem ser problemáticos se eles caem dentro exões14.
- Observe que a ferramenta de análise de sgRNA MIT pontua cada sgRNA em uma escala de 1-100 onde uma pontuação de 100 denota maior especificidade. Uma nota superior a 70 é ideal e representa um sgRNA com mínimos efeitos fora do alvo. Este site também gera uma lista de hits fora do alvo com sua localização dentro do genoma e quantos mismatches cada um deles tem com o guia do candidato.
- Escolha sgRNAs de 2-3 que visem locais distintos dentro da região de interesse para o gene alvo, e que têm as maiores pontuações de eficiência e o alvo de clivagem emparelhados (ver tabela 1 para sgRNA sequências alvo exon 9 de Calr).
Nota: Dependendo das metas experimentais, maior ênfase pode ser colocada sobre os diferentes valores obtidos a partir das ferramentas mencionadas.
- Copie e cole a sequência de referência NCBI do gene de interesse para a ferramenta de web designer do Broad Institute sgRNA: (http://portals.broadinstitute.org/gpp/public/analysis-tools/sgrna-design).
2. clonagem de Oligos sgRNA13
-
Gerar o oligo frente
- Crie uma lista de sequências de sgRNA sem o site de PAM.
- Adicionar um G à extremidade 5' da sequência se a extremidade 5' da sequência de sgRNA não é um G.
Nota: Este G é necessário para maximizar o nível de transcrição sgRNA U6-conduzido. Adição de um G é necessária para sgRNA m1 (tabela 1). - Adicionar a sequência "CACC" à extremidade 5' da sequência guia G-otimizado (sem PAM) (tabela 2), a fim de gerar as saliências necessárias para ligadura dos oligos recozidos em BsmBI digerido lentiGuide vetor (ver passo 3).
-
Gerar o oligo reverso
- Reversa complementar a sequência guia G-otimizado (sem PAM).
- Adicione a sequência "AAAC" à extremidade 5' da sequência inversa complementado guia G-otimizado (tabela 2).
- Ordem do oligos projetado de uma companhia de síntese da primeira demão.
-
Recoze e fosforilar oligos
- Adicionar 1 µ l de frente oligo (100 µM), 1 µ l de oligo reverso (100 µM), 1 µ l de T4 ligadura de 10x buffer, 0,5 µ l da quinase de polinucleotido T4 (PNK) e 6,5 µ l de H2O para um tubo PCR (Total = 10 µ l).
- Execute o programa a seguir na thermocycler: 37 ˚ c por 30 min, 95 ˚ c por 5 min, rampa de 95 a 25 a 0.1 ° C/s.
- Diluir o produto de reação em 1: 250 em H2O.
3. digestão de lentiGuide-Puro vetor13
-
Vetor de lentiGuide-Puro Digest
- Montar uma reação de digestão de 50 µ l com os seguintes componentes: 5 µ g de plasmídeo circular, 2 µ l (30 unidades) da enzima de restrição BsmBI, 5 µ l de tampão de enzima de restrição e até 50 µ l de H2O.
- Executar a reação para h 2 a 55 ° C. Após a primeira hora, adicione mais 1 μL de enzima de restrição BsmBI.
-
Dephosphorylate a espinha dorsal corte
- Adicione 7 µ l de tampão de reação da fosfatase e 2 µ l da enzima fosfatase para o vetor digerido.
- Incubar durante 30 min a 37 ° C.
- PCR purify o corte e a espinha dorsal dephosphorylated usando um kit comercial.
Nota: Substituto as colunas coloridas rosa fornecidas com o kit miniprep azul colunas coloridas desde que estas melhor podem acomodar tamanhos grandes do plasmídeo. O rendimento pode ser aumentado aquecendo o tampão de eluição em um bloco de calor de 90 ˚ c antes da eluição e por eluição do DNA da coluna duas vezes no final (executar o DNA eluted da primeira eluição volta através de uma segunda vez).
4. ligadura ofAnnealed Oligos em Backbone digerido13
- Adicionar 50 ng do backbone digerido, 1 µ l de recozido oligo diluição, 1 µ l de T4 ligadura de 10x buffer, 1 µ l de T4 ligase e até 10 µ l de H2O para um tubo PCR. Incubar durante 1 h em temperatura ambiente
- Transforme células de Stbl3 com 2 µ l de reação da ligadura. Placa em uma placa de ágar de caldo (LB) lisogenia com ampicilina 100 de µ g/mL e incubar durante uma noite a 37 ° C.
- Escolha 3 colônias e inocular em uma cultura de mini-preparação.
-
Validar a inserção correta oligo
- Execute um miniprep para cada cultura e sequência através do local de inserção do oligo a partir de um primer no promotor U6 usando um kit comercial.
- Execute um midi-prep ou uma maxipreparação da cultura sequência verificada.
5. geração de linhas de célula estàvel expressando SpCas9
Nota: Este protocolo envolve a entrega de pLX_TRC311-Cas9 plasmídeo por infecção Lentivirus. Este protocolo é descrito em detalhes para murino interleucina-3 (3 mIL) dependentes pro-B (Ba/F3) células, uma linhagem de células de suspensão e pode ser adaptado para outros tipos de célula usando as condições de cultura preferencial para cada tipo de célula. O meio de cultura para células F3/Ba consiste em RPMI suplementado com 10% de soro bovino fetal, 1% penicilina/estreptomicina/L-glutamina e 10 ng/mL de murino interleucina 3.
-
Transfect células HEK-293T
- Sementes de 3 x 106 HEK-293T células por placa de cultura de tecido de 10 cm. Manter células semeadas durante a noite antes do transfection.
Nota: As células HEK-293T são uma linhagem de células aderentes e são rotineiramente utilizadas para a produção de vírus. As células são mantidas em DMEM suplementado com 10% de soro bovino fetal, 1% penicilina/estreptomicina/L-glutamina. Células devem ser 80% confluente ao tempo do transfection. - Pré-aquecimento reduzido soro media (Opti-MEM) e meio de crescimento.
- Adicionar 500 µ l de mídia soro reduzida, 7 µ g de pLX_TRC311-Cas9 construção, 4 µ g de shRNA particulas de Lentivirus embalagens pCMV-VSV-G e 4 µ g de plasmídeo de Lentivirus de empacotamento de psPAX2 a um tubo
Nota: O DNA Total por prato de 10cm de células 293T é 15 µ g. - Misture o DNA bem e adicionar 45 µ l de reagente de transfeccao. Misture delicadamente e deixe por 20 min.
Nota: É importante usar mídia de soro reduzida porque o soro vai inibir a formação de complexa entre o reagente de transfeccao e plasmídeo. - Aspire o meio velho na placa 293T e adicionar 5 mL de meio de crescimento novo.
- Adicione a mistura de reagente DNA e transfecção de forma sábia de soltar a placa 293T. Agitar suavemente o frasco e incubar a 37 ° C e 5% CO2 por 24 h.
- Colheita sobrenadantes virais em 24 e 48 h post do transfection. Passe por um 0,22 µm filtro e alíquota em um tubo de cryovial (1,6 mL/tubo e 1,5 mL vai ser usado para infecção).
- Loja de sobrenadante viral a-80 ° C.
Nota: Lentivirus sobrenadante pode ser usado dentro de 6 meses. Se possível, transductions de spinfection devem ser realizadas com vírus fresco).
- Sementes de 3 x 106 HEK-293T células por placa de cultura de tecido de 10 cm. Manter células semeadas durante a noite antes do transfection.
-
Particulas de Lentivirus infecção das células F3/Ba
- Descongele o sobrenadante viral no gelo, se congelado.
- Centrifugar as células em 300 x g, durante 4 min.
- Aspirar o sobrenadante e ressuspender as células em uma concentração de 3 x 106 células/mL.
- Adicionar 500 µ l de células ressuspensa, 1,5 mL de sobrenadante viral, 4 µ l de polybrene (estoque: 2 mg/mL) e 10 ng/mL de m-IL3 em uma placa de cultura de tecidos de 6-poços. Certifique-se de incluir um não infectados com 1,5 mL de mídia em vez de sobrenadante viral como um controle.
Nota: A quantidade de sobrenadante viral adicionado deve corresponder a uma multiplicidade de infecção (MOI) < 1. - Centrifugue a placa a 440 x g para 120 min a 37 ° C.
- Levar o prato fora o centrifugador e coloque em 37 ° C e 5% CO2 incubadora e permitir a crescer durante a noite.
- Gire para baixo as células após 24 h e ressuspender com 5 mL de mídia quente fresca em uma placa de 6.
-
Seleção de células Cas9 infectado
- Gire para baixo as células e ressuspender com fresca mídia quente suplementada com 5 µ g/mL de blasticidin, 48 h post spinfection.
- Selecione as células por 9 dias com blasticidin ou até células não infectadas controle (negativo) estão mortas.
- Uma vez concluída a seleção, transferi as células resistentes ao meio com menor concentração de blasticidin.
6. repórter ensaio para Cas9 atividade15
- Transduce células parentais e celulas que expressam estàvel Cas9 com pXPR-011 por infecção Lentivirus (ver etapas 5.1-5.2).
Nota: Este vetor contém GFP e um guia direcionamento de GFP. Células contendo Cas9 ativo irão resultar em uma redução de GFP (Figura 2). - Selecione as células para 3 dias com 2 µ g/mL de puromicina, 48 h post spinfection.
- Analise as amostras para expressão de GFP por citometria de fluxo.
Nota: Redução de 50% ou mais GFP representa ideal Cas9 atividade. Uma maior concentração de blasticidin seleção poderia ser usada para aumentar a atividade Cas9 se menos do que uma redução de 50% é observada. Nem todas as linhagens celulares podem tolerar blasticidin seleção. Neste caso, o uso de vetores Cas9 com outras fitas de seleção pode ser necessário. Além disso, nem todas as linhagens celulares podem tolerar a expressao de Cas9. Neste caso, transfection transiente de Cas9 pode ser usado.
7. Spinfection de sgRNAs em células expressando-Cas9
- Siga os passos 5.1-5.2 para infecção Lentivirus de sgRNAs em células de interesse.
Nota: No passo 5.1.3, substitua pLX_TRC311-Cas9 construção construção de lentiGuide-Puro validada da seção 4. - 48 h post spinfection, selecione as células para 3 dias com 2 µ g/mL de puromicina.
- Transferir as células resistentes ao meio com menor concentração de antibiótico e continuar a seleção por mais 4 dias permitir a edição suficientes.
8. Ba/F3 transformação celular e selecção positiva usando m-IL3 retirada
Nota: Este ensaio é descrito para as células dependentes de mIL-3 Ba/F3, mas poderia ser aplicado a qualquer linha de célula dependente de citocinas.
- Spin para baixo crescimento exponencial Ba/F3 células expressando ectopically Cas9 e o sgRNA de interesse.
- Aspire o sobrenadante e lavar as células com 5 mL de fosfato tamponado salino (PBS).
- Gire para baixo as células e repetir a etapa de lavagem em 8.2 quatro vezes (esta etapa garante que a mídia está livre de IL-3).
- Aspirar a PBS e ressuspender as células em 5 mL de meio fresco sem IL-3.
- Conte as células usando um hemocytometer ou um analisador de viabilidade celular.
- As células em triplicado em uma placa de cultura de tecidos de 6-poços em uma concentração de 1 x 105 células/mL em um volume total de 2 mL de meio fresco sem IL-3 de sementes.
- Monitorar e contar as células para um total de 8 dias a cada 2 dias.
Nota: Células de Ba/F3 falta IL-3 normalmente morrem 2 dias post fome IL-3. É importante incluir um controle negativo no ensaio (tipicamente um direcionamento-guia é usado como um controle).
9. triagem para CRISPR no alvo edição
- Projeto CRISPR triagem primeiras demão a montante e a jusante do local de clivagem da sgRNA
Nota: use primers pelo menos 100 bp do site clivagem previsto para assegurar a deteção não iria ser impactada por uma grande inserção e/ou exclusão (indel) no local de destino de sgRNA. - Isole o DNA genômico (gDNA) de células transformadas (no final da curva de crescimento) e da retirada de células pre-citocinas.
Nota: Sempre incluem células superexpressão o guia não-alvo como um controle de edição de gene. Antes e depois transformação irá identificar clones que foram selecionadas positivamente para e ter uma vantagem proliferativa, isolando gDNA de células. - Montar um 50 µ l do PCR com os seguintes componentes: 25 µ l de mistura de x PCR 2 de 1 µ l de cartilha para a frente (10 µM), 1 µ l de primer reverso (10 µM), 50-100 ng de gDNA e H2O até 50 µ l.
Nota: Numerosos polimerases de alta-fidelidade podem ser usadas no passo 9.3. - Executar amostras em um thermocycler usando os seguintes parâmetros: 95° C por 1 min, 30 ciclos de (94 ° C por 1 min, 52 ° C por 30 s, 72 ° C por 30 s) e 72 ° C por 10 min. Este PCR é otimizado para as primeiras demão Calr listadas na tabela 3. Otimize as condições PCR para o par de primer desenhado na etapa 9.1 baseado em testá-lo em gDNA.
- Execute 5 µ l das amostras em um gel de agarose 2% a 10 V/cm usando 1 tampão Tris-Acetato-EDTA (TAE) de x. Examinar as amostras para a presença / ausência de uma banda amplificada correspondente em tamanho para o gene de interesse.
- Use o resto da reação de PCR (45 µ l) para realizar uma purificação de PCR.
- Clone o amplicons com um PCR kit de clonagem em um vetor do plasmídeo. Por exemplo, vetores pGEM T-easy são normalmente utilizados.
- Transforme o plasmídeo em células Stbl3 ou outras células compatíveis e placa em placas de ágar LB com o antibiótico relevante. Selecionar colônias de 10-20, mini-preparação cada um e submeter cada clone a Sanger sequenciamento para caracterizar o puntuais criado a partir de CRISPR/Cas9 edição.
Nota: Se desejar, única célula célula de fluorescência ativada que classificação (FACS) pode ser executada na maior edição população para isolar um clone com uma específica indel. Além disso, a próxima geração (NGS) métodos de sequenciamento pode ser usada para quantificar mais robustamente no alvo de edição usando o sequenciamento profundo de PCR amplicons, abrangendo a região de destino sgRNA. Por exemplo, rastreamento de puntuais por decomposição ou maré pode ser usado em vez de subcloning para determinar precisamente o espectro e a frequência de mutações específicas geradas em um pool de células (https://tide.nki.nl/#about)16.
Resultados
Usando o método descrito aqui, o objetivo deste experimento é estudar os efeitos funcionais da introduzir mutações indel para o locus Calr endógena na transformação de células hematopoiéticas. O sistema CRISPR/Cas9 é usado como uma ferramenta para criar endógena Calr mutações em células F3/Ba. Dois sgRNAs foram escolhidos para alvo exon 9 de Calr (Figura 1), na região onde inserções e/ou mutações de exclusão (indel) normalmente ocorrem em CALR-mutante MPN pacientes17,18. O primeiro sgRNA (m1) foi escolhido com base na sua clivagem alta eficiência e favorável golo fora do alvo (tabela 1). A segunda sgRNA (m2) foi escolhido principalmente pela sua localização dentro exon 9 e por falta de sgRNAs adicionais na região para editar com clivagem alta eficiência e favorável o alvo pontuações (tabela 1). Duas sgRNAs distintas (m1 ou m2) foram usados em infecções separadas para assegurar que os efeitos observados foram devido à edição de gene no alvo. Efeitos fora do alvo são susceptíveis de ser compartilhado por vários sgRNAs independente. O controle não-alvo (scramble) também foi usado como um controle negativo. Recrutamento da endonuclease Cas9 para Calr exon 9 prevê criar DSBs neste locus. Os DSBs então iria ser reparados pelo NHEJ, que pode gerar puntuais de tamanhos variáveis (Figura 1).
Para desenvolver o em vitro sistema CRISPR/Cas9 em Ba/F3 células, células estàvel expressando a proteína Cas9 foram feitas por transdução mediada por Lentivirus conforme o protocolo descrito acima. Expressao Cas9 resulta em atividade de Cas9 robusta. Cas9 atividade nas células Ba/F3-Cas9 foi medida por pXPR-011, uma construção de repórter que contém um guia direcionamento GFP15e GFP. Células contendo Cas9 ativo irão resultar em uma redução de GFP15. GFP foi medido nas células usando citometria de fluxo. BA/F3-Cas9 células exibido uma redução de cerca de 76% no GFP, correspondente à atividade de Cas9 robusta (Figura 2).
Digite 1 receptores de citocinas, tais como receptor de Trombopoietina (MPL), o receptor de eritropoetina (EPOR) e o granulócitos colônia-estimulando do fator do receptor (G-RFCE) foram cada um individualmente, expressa estàvel em Ba/F3-Cas9 células para determinar sua cooperatividade com mutante calreticulin na indução de transformação de células F3/Ba. Transdução das construções sgRNA (m1, m2 ou um scramble (direcionamento-guia)) Então foi realizada em cada uma das linhas celulares Ba/F3 estàvel expressando Cas9 e o receptor de interesse. Células foram então Selecionadodas por 7 dias com puromicina para permitir tempo suficiente para a edição de gene CRISPR/Cas9. Uma pressão de seleção positiva foi então aplicada por morrer de fome as células de citocina (mIL-3). O objetivo dessa pressão de fome é identificar se puntuais em Calr exon 9, semelhantes às observadas em pacientes MPN, são selecionados, resultando em crescimento independente de citocinas e transformação de células F3/Ba. A curva de crescimento foi realizada para um total de 8 dias, e as células foram contadas a cada 2 dias medir sua transformação (Figura 3)19. Célula de pelotas para extração de DNA genômica foram coletadas antes do início da curva de crescimento e no final da curva de crescimento para verificar para edição no alvo e monitorar o puntuais que expandiu post cytokine fome (Figura 3)19.
Observou-se crescimento independente de citocinas em Calr-alvo Ba/F3-MPL-Cas9 células (Figura 4B), mas não Calr-alvo parental Ba/F3-Cas9 células (Figura 4A), ou Ba/F3-Cas9 células expressando ectopically EPOR (Figura 4 ) ou G-RFCE (Figura 4)19. Para confirmar que o mIL-3 crescimento independente nas células Calr-alvo Ba/F3-MPL-Cas9 foi resultado do gene alvo na edição, as células foram colhidos 8 dias borne mIL-3 retirada e o DNA genômico foi extraído19. Uma região de bp 422 abrangendo o local de destino foi amplificado utilizando os primers listados na tabela 3. Sub clonagem de PCR amplicons foi realizada e 30 clones individuais foram enviados para Sanger sequenciamento. Para m1 ou m2 Calr-células alvo Ba/F3-MPL-Cas9, todos os clones sub contidos puntuais de tamanhos variados para o local pretendido de corte (Figura 5)19. 11 de 13 m1 sub clones foram encontrados conter puntuais que levou a + 1 bp frameshift mutações (Figura 5), semelhantes aos encontrados em pacientes. Para m2 Calr-alvo células, clones sub 10 de 17 foram encontrados para conter puntuais que levou a + 1 bp frameshifts (Figura 5)19. Estes dados confirmam que CRISPR/Cas9-mediada por introdução de + 1bp frameshift mutações para o endógeno Calr locus exon 9 é suficiente para conferir atividade oncogênica de Calr19. Os dados de sequenciamento na Figura 5 também sugerem que a introdução de heterozigotos + 1bp frameshift mutações para o locus Calr endógena é suficiente para transformar células Ba/F3-MPL, consistentes com a observação que CALR as mutações são normalmente heterozigotas em MPN pacientes17,18. Desde NHEJ reparar resulta em alta heterogeneidade entre o puntuais, única célula classificação para isolar um clone de interesse pôde ser executada. Isso permitiria que o pesquisador estudar os efeitos de mutações específicas criados por CRISPR/Cas9.
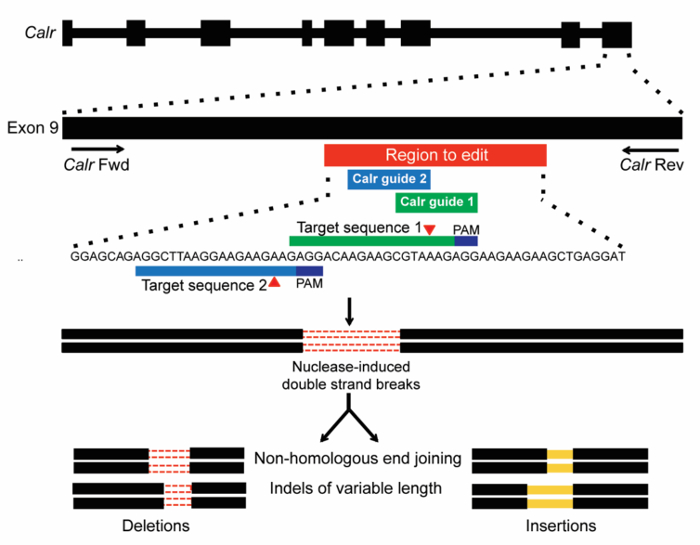
Figura 1: Esquema de CRISPR/Cas9 edição de gene alvejar de exon 9 de Calr. Dois sgRNAs foram projetados para atingir um local dentro da região de exon 9 rato Calr correspondente direcionado pelo + mutações frameshift 1bp humana MPN (barra vermelha). Sequências de destino com PAMs (azul escuro) são mostradas, e esperados sítios de clivagem por Cas9 são indicados por triângulos vermelhos. DSBs gerados pelo CRISPR/Cas9 então são reparados por NHEJ, que deverá gerar puntuais de comprimento variável19. Clique aqui para ver uma versão maior desta figura.
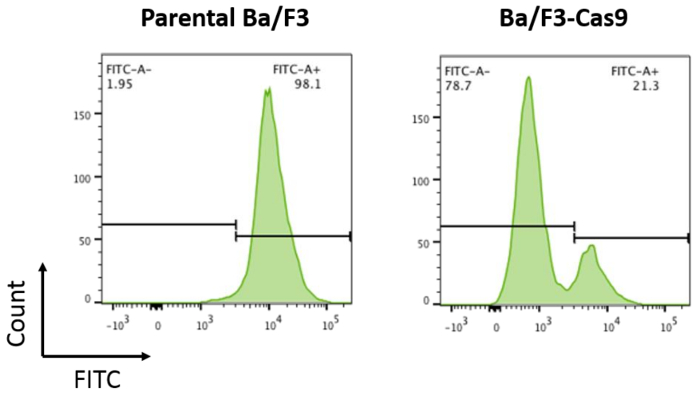
Figura 2: ensaio da atividade Cas9. Parental Ba/F3 células e células Ba/F3 superexpressão Cas9 foram transfectadas com pXPR-011 para medir a atividade Cas9. Uma redução de GFP correlaciona-se com o aumento da atividade Cas9. Células de Ba/F3 parentais são ~ 100% FITC + comparado com células Ba/F3-Cas9 onde o FITC é reduzido por ~ 76%. Clique aqui para ver uma versão maior desta figura.

Figura 3: cronograma para infecção e seleção de sgRNAs em células Ba/F3. Clique aqui para ver uma versão maior desta figura.

Figura 4: Introdução de + 1 bp frameshift mutações o locus Calr endógena é suficiente para conferir atividade oncogênica de Calr. Curvas de crescimento (A-D) em Ba/F3-Cas9 parental células (A) Ba/F3-MPL-Cas9 (B), Ba/F3-EPOR-Cas9 células (C), e Ba/F3-G-RFCE-Cas9 células (D) demonstra o crescimento independente de IL-3 em Ba Calr-alvo / F3-MPL-Cas9 células apenas19. Clique aqui para ver uma versão maior desta figura.

Figura 5: verificação de sequenciamento Sanger. Confirmação no alvo edição de endógena Calr (exon 9) nas células Ba/F3-MPL. + 1 bp frameshift mutações são indicadas em preto19. Clique aqui para ver uma versão maior desta figura.
| Gene alvo | sgRNA ID | Sequência de destino (5' para 3') | Strand | Eficiência de clivagem | O alvo Pontuação | ||
| Calr | M1 | AGAGGACAAGAAGCGTAAAGAGG | + | 0.7 | 70 | ||
| Calr | m2 | GAGGCTTAAGGAAGAAGAAGAGG | + | 0.3 | 28 | ||
Tabela 01:20-mer protospacer sequências incluindo o PAM do site (em negrito) para dois sgRNAs segmentação exon 9 de Calr 19.
| ID da primeira demão | Sequência (5' para 3') |
| M1-F | CACCGAGAGGACAAGAAGCGTAAAG |
| M1-R | AAACCTTTACGCTTCTTGTCCTCTC |
| m2-F | CACCGAGGCTTAAGGAAGAAGAAG |
| m2-R | AAACCTTCTTCTTCCTTAACCTC |
Tabela 2: sequências de Protospacer e sua inversa complementa com "CACC" e "AAAC" adicionado para clonagem em vetor de pLenti-guia usando a enzima de restrição BsmBI 19.
| ID da primeira demão | Sequência (5' para 3') |
| Calr_Fwd | ACCACCTGTCTTTCCGTTCT |
| Calr_Rev | GGCCTCTACAGCTCATCCTT |
Tabela 3: CRISPR triagem iniciadores de PCR a montante e a jusante do local de clivagem da sgRNA.
Discussão
Aqui vamos mostrar o uso de gene de CRISPR/Cas9 edição para estudar a função biológica de CALR mutações em células hematopoiéticas. O sucesso do presente protocolo é altamente dependente de múltiplos fatores. Em primeiro lugar, é importante saber quais os tipos de mutações podem ser responsáveis para o fenótipo desejado. Neste protocolo, a leitura é a transformação de células Ba/F3 para mIL-3 independência e os tipos de mutações são puntuais no exon 9 de CALR. No entanto, se a mutação desejada é uma substituição de pares de base única, então reparo HDR-mediada é o método preferido porque ele pode introduzir precisas mutações pontuais ou inserções de um single-strand ou double-stranded DNA doador modelo2, 3. se se deseja o nocaute do gene e, em seguida, a criação de em grande escala genômicas exclusões direcionamento precoce exões codificação é preferido20.
Em segundo lugar, a linha celular deve ser dependente de citocinas para permitir a retirada de citocina selecção positiva post de pressão. É importante notar que nem todas as linhagens celulares podem tolerar a expressao de Cas9 (consulte a etapa 6.3). Isto pode ser devido a seleção de blasticidin, que não é tolerada por todas as linhas de célula. Neste caso, a escolha de outras construções Cas9 com fitas de seleção diferente pode ser necessária. Uma ressalva de usar linhas de célula dependente de citocinas, como as células F3/Ba é que eles são suscetíveis ao crescimento independente de cytokine espontânea, às vezes em resultado da aquisição de mutações no gene ectopically expressa de interesse seguindo citocinas retirada21. Nesse sentido, o uso de controles apropriados é necessário para estar confiante de que a transformação celular observada é devido à edição de gene CRISPR no alvo. Neste protocolo, não observamos transformação celular em células-Ba/F3-MPL transfectadas com um controle de sgRNA não-direcionamento ou em Ba/F3-EPOR e Ba/F3-GCSFR células transfectadas com Calr-alvo sgRNAs. Isto proporciona mais confiança que transformação celular em células Ba/F3-MPL é um resultado de no alvo edição do locus Calr endógeno.
Neste protocolo, reparo de ADN por NHEJ introduz puntuais de tamanhos variáveis, como visto na Figura 5. Análise por Sanger sequenciamento foi realizado em uma subamostra da população em massa de células editadas a fim de procurar a expansão de puntuais que levam a + 1 bp mutações frameshift post cytokine retirada. Próxima geração de sequenciamento mencionado na etapa 9,9 é uma maneira mais robusta para quantificar no alvo de edição para populações de células em massa. Se a análise em um clone específico é desejado, então única célula classificação da população em massa é necessário. Única célula classificação é vantajosa em compreender o efeito biológico resultante de um tipo específico de mutação e é útil quando o objetivo é compreender as diferenças entre dois tipos diferentes de mutações.
Uma das preocupações com o sistema CRISPR/Cas9 é efeitos fora do alvo, que são eventos de clivagem do lado de fora da região de destino. Para avaliar a especificidade do sistema CRISPR/Cas9 e certifique-se de que as populações transformadas não contêm quaisquer efeitos fora do alvo, levando à transformação observada, nós teríamos que examinar se genoma edição ocorreu fora da região de interesse. Isto poderia ser feito por procurando por evidências de Cas9/NHEJ-driven genoma edição em potencial alvo genomic loci com similaridade da sequência significativa para o alvo pretendido. Para isso, próxima geração sequenciamento também pode ser usado para avaliar quantitativamente a frequência de efeitos fora do alvo em locais especificados ao longo do genoma. Para reduzir o potencial de efeitos fora do alvo, fatores diferentes poderiam ser incorporadas o protocolo. Como mencionado nos resultados, estudar vários sgRNAs como alvo a região de interesse garante que o fenótipo é o resultado de um evento no alvo. Além disso, estudos recentes têm sugerido que a sgRNAs truncado com 17 nucleotídeos pode reduzir a frequência de clivagem fora do alvo eventos22. Além disso, a exposição prolongada ao Cas9 de expressao pode resultar em uma maior frequência de efeitos fora do alvo. É importante notar que um sistema de duplo vetor contendo Cas9 e o sgRNA pode ser usado no protocolo em vez de expressao de Cas9; no entanto, estes vetores tendem a ser muito grande e ocasionar um baixo título Lentivirus e baixa eficiência de infecção. Como resultado, transfecção transiente da construção de Cas9 nas células por nucleofection poderia ser usada. Relatórios recentes também desenvolveram Cas9 construções com maior especificidade para edição no alvo, tais como o eSpCas9 construir6.
Em resumo, CRISPR/Cas9 representa um genoma eficiente, barato e confiável ferramenta de engenharia, permitindo o estudo de mutações genéticas em níveis fisiológicos de expressão. Aqui nós empregamos CRISPR/Cas9 gene edição como um método robusto e eficiente para avançar a compreensão dos efeitos funcionais de CALR mutações em células hematopoiéticas.
Divulgações
Temos sem conflitos de interesse relacionados a este relatório.
Agradecimentos
Este trabalho foi financiado pelo NIH (R01HL131835), um prêmio de investigador clínico de Damon Runyon e o consórcio de câncer Starr.
Materiais
| Name | Company | Catalog Number | Comments |
| BsmBI | New England Biolabs | R0580L | |
| Blasticidin | Sigma Aldrich | 15025 | |
| Puromycin | Life Technologies | A1113803 | |
| Stbl3 cells | Life Technologies | C737303 | |
| TransIT-LT1 | Thermo Fisher Scientific | MIR2300 | |
| Opti-MEM | Life Technologies | 51985034 | |
| RPMI | Thermo Fisher Scientific | MT10040CV | |
| DMEM | Thermo Fisher Scientific | 10-017-CV | |
| Fetal bovine serum (FBS) | Omega Scientific | FB-11 | |
| Penicillin/streptomycin/L-glutamine | Life Technologies | 10378016 | |
| mIL-3 | Peprotech | 213-13 | |
| psPAX2 | Addgene | N/A | |
| pCMV-VSV-G | Addgene | N/A | |
| pLX_TRC311-Cas9 | Addgene | N/A | |
| polybrene | Sigma Aldrich | H9268 | |
| pXPR-011 | Addgene | N/A | |
| Phosphate Buffered Saline (PBS) | Genessee Scientific | 25-507 | |
| TAE buffer | Thermo Fisher Scientific | FERB49 | |
| LentiGuide-Puro | Addgene | Plasmid #52963 | |
| PNK | New England Biolabs | M0201S | |
| T4 ligase | New England Biolabs | M0202S | |
| PCR purification kit | Qiagen | 28104 | |
| Miniprep kit | Qiagen | 27104 |
Referências
- DeWitt, M. A., et al. Selection-free genome editing of the sickle mutation in human adult hematopoietic stem/progenitor cells. Sci Trans Med. 8, 360(2016).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nat Biotechnol. 4, 347-355 (2014).
- Gaj, T., Gerbach, C. A., Barbas, C. F. ZFN, TALEN, CRISPR/Cas-based methods for genome engineering. Trends Biotechnol. 31 (7), 397-405 (2013).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Wiedenheft, B., Samuel, S. H., Doudna, J. A. RNA-guided genetic silencing systems in bacteria and archea. Nature. 482 (7385), 331-338 (2012).
- Slaymaker, I. M., Gao, L., Zetsche, B., Scott, D. A., Yan, W. X., Zhang, F. Rationally engineered Cas9 nucleases with improved specificity. Science. 351 (6268), 84-88 (2016).
- Ran, F. A., et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell. 154 (6), 1380-1389 (2013).
- Larson, M. H., et al. CRISPR interference (CRISPRi) for sequence-specific control of gene expression. Nat Protoc. 8 (11), 2180-2196 (2013).
- Wang, H., et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell. 153 (4), 910-918 (2013).
- Kim, H. S., et al. CRISPR/Cas9-mediated Gene-knockout screens and target identification via Whole genome sequencing uncover host genes required for picornavirus infection. J Biol Chem. 10, 1074(2017).
- Heckl, D., et al. Generation of mouse models of myeloid malignancies with combinatorial genetic lesions using CRISPR-Cas9 genome editing. Nat Biotechnol. 32 (9), 941-946 (2014).
- Daley, G. Q., Baltimore, D. Transformation of an interleukin 3-dependent hematopoietic cell line by the chronic myelogenous leukemia-specific bcr/abl protein. Proc Natl Acad Sci USA. 85, 9312-9316 (1988).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biol. 17, 148(2016).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation. Nat Biotechnol. 32, 1262-1267 (2014).
- Brinkman, E. K., et al. Easy quantitative assessment of genome editing by sequence trace decomposition. Nucl Acid Res. 42, e168(2014).
- Klampfl, T., et al. Somatic mutations of calreticulin in myeloproliferative neoplasms. N Engl J Med. 369, 2379-2390 (2013).
- Nangalia, J., et al. Somatic CALR mutations in myeloproliferative neoplasms with nonmutated JAK2. N Engl J Med. 369, 2391-2405 (2013).
- Elf, S., et al. Mutant Calreticulin requires both its mutant C-terminus and the thrombopoietin receptor for oncogenic transformation. Cancer Discovery. 6 (4), 368-381 (2016).
- Bauer, D. E., Canver, M. C., Orkin, S. H. Generation of Genomic Deletions in Mammalian Cell lines via CRISPR/Cas9. J. Vis. Exp. (95), e52118(2015).
- Watanabe-Smith, K., et al. Analysis of acquired mutations in transgenes arising in Ba/F3 transformation assays: findings and recommendations. Oncotarget. 8, 12596-12606 (2017).
- Fu, Y., et al. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. NatBiotechnol. 32, 279-284 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados