Method Article
Utilizzando CRISPR/Cas9 Gene Editing per studiare l'attività oncogena di calreticulina mutante in cellule ematopoietiche Cytokine dipendente
In questo articolo
Riepilogo
Genica mirata di editing con CRISPR/Cas9 ha notevolmente facilitato la comprensione delle funzioni biologiche dei geni. Qui, utilizziamo la metodologia CRISPR/Cas9 a modello calreticulina mutazioni in cellule ematopoietiche citochina-dipendente al fine di studiare la loro attività oncogena.
Abstract
Cluster regolarmente intercapedine ripetizioni brevi palindromi (CRISPR) è un sistema di immunità adattativa nei procarioti che è stato reimpiegato dagli scienziati per generare nucleasi RNA-guidato, come ad esempio CRISPR-collegata (Cas) 9 per site-specific genoma eucariotico di editing. Ingegneria del genoma da Cas9 viene utilizzato per in modo efficiente, robusta e facilmente modificare geni endogeni in molte linee cellulari di mammifero biomediche pertinenti e organismi. Qui vi mostriamo un esempio di come utilizzare la metodologia CRISPR/Cas9 per comprendere la funzione biologica di mutazioni genetiche specifiche. Abbiamo il modello calreticulina (CALR) mutazioni in murino interleukin-3 (mIL-3) pro-B (Ba/F3) le celle dipendenti dalla consegna di guida singola RNAs (sgRNAs) targeting per il locus Calr endogeno nella regione specifica dove inserimento e/o cancellazione (indel ) CALR mutazioni si verificano in pazienti con neoplasie mieloproliferative (MPN), un tipo di tumore del sangue. La sgRNAs creare doppio filo (DSBs) nella regione mirata che sono riparati da non-omologo fine unirsi (NHEJ) per dare indels di varie dimensioni. Utilizziamo quindi il test di trasformazione cellulare Ba/F3 standard per comprendere l'effetto dell'espressione di livello fisiologico di Calr mutazioni sulla trasformazione cellulare ematopoietico. Questo approccio può essere applicato ad altri geni di studiare la loro funzione biologica in varie linee cellulari di mammifero.
Introduzione
CRISPR/Cas9 tecnologia recentemente ha rivoluzionato l'editing genomico mirato negli organismi e cellule viventi. È divenuto uno strumento estremamente potente per la ricerca biomedica e attualmente viene utilizzato come un viale potenziale per la terapia di malattie genetiche1. La base per tutti gli strumenti di editing del genoma si basa sulla creazione di una nucleasi-indotta rottura del DNA doppia filamento (DSB) presso il locus genomico per essere modificato. Il DSBs può essere riparato da fine-unirsi non-omologo (NHEJ) o riparazione omologia-diretto (HDR)2,3. Il vantaggio della nucleasi Cas9 sopra altri genoma ingegneria nucleasi, quali nucleasi a dita di zinco (ZFNs) e nucleasi di trascrizione attivatore-come effettore (TALENs) è la dipendenza dal RNA per il targeting della nucleasi a una sequenza di DNA desiderata, rispetto le interazioni proteina-DNA trovato in ZFNs e TALENs2,3.
Dopo la scoperta del pathway di nucleasi CRISPR/Cas9 come un sistema immunitario adattativo in cellule procariotiche4,5, molto sforzo è andato in adattando la via per l'uso in linee cellulari di mammifero e modello organismi2, 3. come strumento per l'editing di gene, il pathway CRISPR/Cas9 utilizza due componenti principali: Streptococcus pyogenes (Sp) derivato Cas9 nucleasi e sgRNAs del gene di interesse2,3di targeting. Il sgRNA è costituito da 20 nucleotidi che Cas9 diretto a un sito specifico sul genoma a RNA-DNA base pair complementarità2,3. Il sito di destinazione del sgRNA deve trovarsi adiacente a un sito di motivi adiacenti (PAM) protospacer sotto forma di 5' NGG, che è riconosciuto dalla nucleasi SpCas9. Con questi strumenti, Cas9 può essere diretto a qualsiasi sequenza di DNA con la progettazione di sgRNAs che hanno come destinazione la regione di interesse. Inoltre, Sp derivato Cas9, esistono varianti supplementari per Cas9 con caratteristiche diverse a seconda dell'applicazione specifica. Ad esempio, ci sono varianti di Cas9 con maggiore specificità per l'editing on target o capacità di single-strand fenditura per DNA intaccare6,7. Inoltre, cataliticamente inattivo Cas9 recentemente è stata sviluppata per regolazione trascrizionale8. Gli scienziati ora hanno utilizzato il sistema CRISPR/Cas9 per una varietà di applicazioni, ad esempio Knockin ' genica e knockout per studiare le funzioni biologiche di geni9, perdita di funzione e guadagno di funzione libreria schermi10 e genetica Ingegneria di organismi modello11.
In questo protocollo, uniamo la metodologia CRISPR/Cas9 con il saggio di trasformazione cellulare Ba/F3 a capire la funzione biologica di CALR mutazioni. Le cellule Ba/F3 sono una linea di cellule ematopoietiche dipendente dal-3 murina che può essere reso IL-3 indipendente sull'espressione di alcuni oncogeni come BCR-ABL12. Al fine di comprendere se calreticulina mutante può trasformare le cellule Ba/F3 alla citochina crescita indipendente, abbiamo mirato dell'essone 9 del locus endogeno Calr usando CRISPR/Cas9 per introdurre le mutazioni indel e quindi si ritirò IL-3 dalle cellule per applicare un pressione di selezione positiva, con l'obiettivo di guadagno di funzione CALR le mutazioni trovate in pazienti MPN di ricapitolare. Il protocollo comprende la progettazione, la clonazione e la consegna di sgRNAs, lo sviluppo di cellule esprimenti Cas9 stabili e screening per CRISPR gene editing on target. Questo protocollo può essere applicato a diversi geni e varie linee di citochina-dipendente delle cellule di interesse ed è particolarmente prezioso nella modellazione e studiando la funzione biologica di geni coinvolti nel cancro.
Protocollo
1. sgRNA Design utilizzando strumenti Online13
-
Progettazione sgRNAs targeting per il gene di interesse utilizzando gli strumenti disponibili gratuitamente online.
- Copiare e incollare la sequenza di Riferimento NCBI del gene di interesse lo strumento di progettazione web sgRNA Broad Institute: (http://portals.broadinstitute.org/gpp/public/analysis-tools/sgrna-design).
Nota: Questo strumento identifica sgRNA sequenze con siti di taglio all'interno degli esoni e coloro che abbracciano il limite di essone / dell'introne ma ancora unirà all'interno dell'esone. - Scarica e Apri il file di output di testo in excel.
- Concentrarsi sulle colonne popolate con le sequenze di sgRNA e il contesto sgRNA. Si noti che le sequenze di sgRNA non contengono il motivo adiacente protospacer (PAM) ma fanno le sequenze di contesto.
- Si noti che il Punteggio di efficacia sul bersaglio elenca il Punteggio di efficienza di clivaggio previsto su una scala da 0 a 1, dove un punteggio pari a 1 indica una maggiore efficienza di clivaggio.
- Utilizzare la funzione 'Ordina' di excel per ordinare i bersagli con il Punteggio di efficacia o di posizione all'interno del gene dell'obiettivo attraverso la colonna '% Target Cut'.
Nota: L'ordinamento di posizione all'interno del gene è utile per identificare sgRNAs destinati a un dominio specifico o esone di interesse. - Selezionare sgRNAs di 3-6 che colpiscono l'area di interesse con alta (> 0,6) gol di efficacia sul bersaglio. Può essere utile sapere quali tipi di mutazioni possono essere responsabili del fenotipo che è desiderato (vedere Figura 1 che spiega la strategia di targeting utilizzata per calreticulina).
Nota: sgRNAs sotto la soglia del Punteggio di efficacia 0,6 suggerito dovrebbe essere considerato quando c'è una mancanza di altri buoni candidati. - Utilizzare lo strumento di web MIT gRNA analisi alla schermata per i potenziali effetti di fuori bersaglio (http://crispr.mit.edu/). Per ogni sgRNA selezionato, eseguire la sequenza di destinazione (tra cui il PAM).
Nota: Lo strumento di progettazione web di Broad Institute sgRNA poteva anche essere utilizzato per schermare per effetti fuori bersaglio (http://portals.broadinstitute.org/gpp/public/analysis-tools/sgrna-design).- Si noti che lo strumento di analisi sgRNA MIT ogni sgRNA i punteggi su una scala da 1 a 100 dove un punteggio di 100 denota maggiore specificità. Un punteggio superiore a 70 è ideale e rappresenta un sgRNA con minimi effetti fuori bersaglio. Questo sito genera inoltre un elenco di colpi fuori bersaglio con la loro posizione all'interno del genoma e mismatches quanti ognuno di essi hanno con la guida del candidato.
Nota: Colpi fuori bersaglio con quattro mismatch o superiore sono considerati sicuro14. Colpi fuori bersaglio con meno di quattro mismatch possono essere problematici se essi rientrano esoni14.
- Si noti che lo strumento di analisi sgRNA MIT ogni sgRNA i punteggi su una scala da 1 a 100 dove un punteggio di 100 denota maggiore specificità. Un punteggio superiore a 70 è ideale e rappresenta un sgRNA con minimi effetti fuori bersaglio. Questo sito genera inoltre un elenco di colpi fuori bersaglio con la loro posizione all'interno del genoma e mismatches quanti ognuno di essi hanno con la guida del candidato.
- Scegliere 2-3 sgRNAs che target località distinte all'interno della regione di interesse per il gene dell'obiettivo, e che hanno i punteggi più alti di efficienza e fuori bersaglio di clivaggio accoppiati (Vedi tabella 1 per le sequenze di sgRNA essone 9 di Calrdi targeting).
Nota: A seconda degli obiettivi sperimentali, maggiore enfasi può essere data ai diversi valori ottenuti dagli strumenti menzionati.
- Copiare e incollare la sequenza di Riferimento NCBI del gene di interesse lo strumento di progettazione web sgRNA Broad Institute: (http://portals.broadinstitute.org/gpp/public/analysis-tools/sgrna-design).
2. clonazione sgRNA Oligos13
-
Generare l'oligo avanti
- Creare un elenco di sequenze sgRNA senza il sito di PAM.
- Aggiungere un G all'estremità 5' della sequenza se 5' conclusione della sequenza sgRNA non è un G.
Nota: Questa G è necessario per massimizzare il livello di trascrizione sgRNA U6-driven. Aggiunta di un G è necessario per m1 sgRNA (tabella 1). - Aggiungere la sequenza "CACC" all'estremità 5' della sequenza ottimizzata G guida (no PAM) (tabella 2) al fine di generare gli strapiombi richiesti per la legatura dei oligos ricotto in BsmBI digerito lentiGuide vettoriale (vedi passo 3).
-
Generare l'oligo inversa
- Complemento inverso la sequenza di guida G-ottimizzato (nessun PAM).
- Aggiungere la sequenza "AAAC" all'estremità 5' della sequenza inversa integrata guida G-ottimizzato (tabella 2).
- Ordinare i oligos progettato da una società di sintesi di primer.
-
Tempri e fosforilano i oligos
- Aggiungere 1 µ l di oligo avanti (100 µM), 1 µ l di oligo inversa (100 µM), 1 µ l di 10 x T4 legatura tampone, 0,5 µ l della chinasi di polinucleotide T4 (PNK) e 6,5 µ l di H2O a un tubo PCR (Totale = 10 µ l).
- Eseguire il seguente programma nel termociclatore: 37 ˚ c per 30 min, 95 ˚ c per 5 min, rampa da 95 a 25 a 0.1 ° C/s.
- Diluire il prodotto di reazione alle 1: 250 in H2O.
3. la digestione dei lentiGuide-Puro vettoriale13
-
Vector lentiGuide-Puro digest
- Montare una reazione di digestione di 50 µ l con i seguenti componenti: 5 µ g di plasmide circolare, 2 µ l (30 unità) di enzima di restrizione BsmBI, 5 µ l di tampone di enzima di restrizione e fino a 50 µ l di H2O.
- Eseguire la reazione per 2 h a 55 ° C. Dopo la prima ora, aggiungere 1 µ l più di enzima di restrizione BsmBI.
-
Dephosphorylate la spina dorsale di taglio
- Aggiungere 7 µ l di tampone di reazione della fosfatasi e 2 µ l di enzima fosfatasi al vettore digerito.
- Incubare per 30 min a 37 ° C.
- PCR purificare il taglio e la spina dorsale defosforilata utilizzando un kit commerciale.
Nota: Sostituto colonne colorate rosa fornite con il kit miniprep blu colonne colorate poiché questi in grado di ospitare meglio plasmide di grandi dimensioni. Il rendimento può essere aumentato riscaldando il tampone di eluizione in un blocco di calore 90 ˚ c prima di eluizione e di eluizione del DNA dalla colonna due volte alla fine (eseguita il DNA eluito dall'eluizione prima indietro attraverso una seconda volta).
4. legatura ofAnnealed Oligos nella spina dorsale digerito13
- Aggiungere 50 ng di backbone digerito, 1 µ l di ricotto oligo diluizione, 1 µ l di 10 x T4 legatura tampone, 1 µ l di ligasi T4 e fino a 10 µ l di H2O a un tubo PCR. Incubare per 1 h a temperatura ambiente
- Trasformare le cellule Stbl3 con 2 µ l di reazione di legatura. Piastra su una piastra di agar Lisogenesi brodo (LB) con ampicillina di 100 µ g/mL e incubare per una notte a 37 ° C.
- Scegli 3 colonie e inoculare in una cultura di mini-prep.
-
Convalidare l'inserimento corretto oligo
- Eseguire un miniprep per ogni cultura e sequenza attraverso il sito di inserimento oligo a partire da un primer nel promotore U6 utilizzando un kit commerciale.
- Eseguire un midi-prep o un maxi-prep della cultura verificata in sequenza.
5. generazione di Cell lLines stabilmente esprimendo SpCas9
Nota: Questo protocollo comporta la consegna del plasmide pLX_TRC311-Cas9 da infezione lentivirale. Questo protocollo è descritto in dettaglio per interleukin-3 (mIL-3) dipendente pro-B (Ba/F3) cellule murine, una linea cellulare di sospensione e potrebbe essere adattato ad altri tipi di cella utilizzando le condizioni di cultura comodo per ogni tipo di cellula. Il terreno di coltura per le cellule Ba/F3 consiste di RPMI completato con 10% siero bovino fetale, 1% di penicillina/streptomicina/L-Glutammina e 10 ng/mL di murino interleukin 3.
-
Transfect HEK 293T cellule
- 3 x 106 HEK 293T cellule per piastra di coltura di tessuto di 10 cm del seme. Mantenere le cellule seminate una notte prima di transfezione.
Nota: Le cellule HEK 293T sono una linea cellulare aderente e sono abitualmente utilizzate per la produzione di virus. Le cellule sono mantenute in DMEM completati con 10% siero bovino fetale, 1% di penicillina/streptomicina/L-glutammina. Cellule dovrebbero essere 80% confluenti al momento della trasfezione. - Pre-caldo ridotta media del siero (Opti-MEM) e terreno di coltura.
- Aggiungere 500 µ l di media riduttori del siero, 7 µ g di pLX_TRC311-Cas9 costrutto, 4 µ g del plasmide pCMV-VSV-G lentivirali imballaggio e 4 µ g del plasmide lentivirali imballaggio psPAX2 in una provetta
Nota: DNA totale per ogni piatto di 10 cm di cellule 293T è 15 µ g. - Mescolare bene il DNA e 45 µ l di reagente di transfezione. Mescolare delicatamente e lasciare riposare per 20 min.
Nota: È importante usare i riduttori del siero media perché siero inibisce la formazione di complessa tra il reagente di transfezione e il DNA del plasmide. - Aspirare il vecchio mezzo nella piastra 293T e aggiungere 5 mL di nuovo mezzo di crescita.
- Aggiungere il mix di reagente DNA e transfezione in maniera saggia goccia alla piastra 293T. Miscelare delicatamente ed incubare a 37 ° C e 5% di CO2 per 24 h.
- Raccogliere i surnatanti virali a 24 e 48 h post transfezione. Passare attraverso un 0,22 µm filtro e aliquota in una provetta di cryovial (1,6 tubo/mL e 1,5 mL verrà utilizzato per l'infezione).
- Conservare il surnatante virale a-80 ° C.
Nota: lentivirali supernatante può essere utilizzato entro 6 mesi. Se possibile, spinfection trasduzioni devono essere eseguite con nuovi virus).
- 3 x 106 HEK 293T cellule per piastra di coltura di tessuto di 10 cm del seme. Mantenere le cellule seminate una notte prima di transfezione.
-
Lentivirale infezione delle cellule Ba/F3
- Scongelare il surnatante virale sul ghiaccio, se congelato.
- Centrifugare le cellule a 300 x g per 4 min.
- Aspirare il supernatante e risospendere le cellule ad una concentrazione di 3 x 106 cellule/mL.
- Aggiungere 500 µ l di sedimento cellule, 1,5 mL di surnatante virale, 4 µ l di polybrene (stock: 2 mg/mL) e 10 ng/mL di m-IL3 in una piastra di coltura del tessuto 6 pozzetti. Assicurati di includere un non infetti bene con 1,5 mL di media invece di surnatante virale come controllo.
Nota: La quantità di surnatante virale aggiunto dovrebbe corrispondere ad una molteplicità di infezione (MOI) < 1. - Centrifugare la piastra a 440 x g per 120 min a 37 ° C.
- Prendere la piastra fuori la centrifuga e posto in incubatore CO2 37 ° C e 5% e consentire a crescere durante la notte.
- Rotazione verso il basso le cellule dopo 24 h e risospendere con 5 mL di terreno fresco caldo in una piastra a 6 pozzetti.
-
Selezione di celle Cas9 infettata
- Rotazione verso il basso le cellule e risospendere con fresco caldo media completati con 5 µ g/mL di Dyfed, 48h post spinfection.
- Selezionare le celle per 9 giorni con Consciousness o fino a quando le cellule non infette (negativo) sono morti.
- Una volta completata la selezione, è possibile trasferire le cellule resistenti a mezzo con concentrazione più bassa di Dyfed.
6. reporter Assay per Cas9 attività15
- Trasducono cellule parentali e le cellule che esprimono Cas9 con pXPR-011 di infezione lentivirale (Vedi punti 5.1-5.2).
Nota: Questo vettore contiene sia GFP e una guida targeting GFP. Celle contenenti Cas9 attivo si tradurrà in una riduzione di GFP (Figura 2). - Selezionare le celle per 3 giorni con 2 µ g/mL di con puromicina, 48h post spinfection.
- Analizzare i campioni per l'espressione della GFP mediante citometria a flusso.
Nota: Una riduzione di GFP di 50% o più rappresenta attività Cas9 ottimale. Una maggiore concentrazione di Dyfed selezione potrebbe essere utilizzata per aumentare l'attività Cas9 se meno è osservata una riduzione del 50%. Non tutte le linee cellulari possono tollerare Consciousness selezione. In questo caso, l'uso di vettori Cas9 con altre cassette di selezione eventualmente. Inoltre, non tutte le linee cellulari possono tollerare espressione stabile di Cas9. In questo caso, trasfezione transiente di Cas9 può essere utilizzato.
7. Spinfection di sgRNAs in cellule che esprimono Cas9
- Seguire passaggi 5.1-5.2 per infezione lentivirale di sgRNAs nelle cellule di interesse.
Nota: Nel passaggio 5.1.3, sostituire pLX_TRC311-Cas9 costrutto con il costrutto lentiGuide-Puro convalidato dalla sezione 4. - 48h post spinfection, selezionare le celle per 3 giorni con 2 µ g/mL di con puromicina.
- Trasferire le cellule resistenti a mezzo con concentrazione più bassa di antibiotici e continua selezione per altri 4 giorni consentire sufficiente di editing.
8. Ba/F3 trasformazione cellulare e selezione positiva utilizzando m-IL3 ritiro
Nota: Questo test è descritto per le celle dipendenti di Ba/F3 mIL-3 ma potrebbe essere applicato a qualsiasi linea di cella dipendente di citochina.
- Rotazione verso il basso le cellule Ba/F3 ectopically che esprimono Cas9 e il sgRNA di interesse in crescita esponenziale.
- Aspirare il surnatante e lavare le cellule con 5 mL di Phosphate Buffered Saline (PBS).
- Rotazione verso il basso le cellule e ripetere la fase di lavaggio in 8.2 quattro volte (questo passaggio garantisce che il supporto sia libero di IL-3).
- Aspirare il PBS e risospendere le cellule in 5 mL di fresco media senza IL-3.
- Contare le celle utilizzando un emocitometro o un analizzatore di attuabilità delle cellule.
- Seme le cellule in triplice copia in una piastra di coltura del tessuto 6 pozzetti ad una concentrazione di 1 x 105 cellule/mL in un volume totale di 2 mL di fresco media senza IL-3.
- Monitorare e contare le celle ogni 2 giorni per un totale di 8 giorni.
Nota: Le cellule Ba/F3 manca IL-3 in genere muoiono 2 giorni dopo IL-3 inedia. È importante includere un controllo negativo nel dosaggio (in genere una guida come destinazione non viene utilizzata come controllo).
9. lo screening per l'Editing di CRISPR On target
- Progettare CRISPR primer a Monte e a valle del sito di clivaggio sgRNA di screening
Nota: utilizzare primer almeno 100 bp dal sito di clivaggio previsto per garantire la rilevazione non sarebbe influenzato da un inserimento di grandi dimensioni e/o la cancellazione (indel) presso il sito di destinazione sgRNA. - Isolare il DNA genomico (gDNA) da cellule trasformate (all'estremità della curva di crescita) e dal prelievo di cellule pre-citochina.
Nota: Includere sempre cellule che overexpressing la guida non-targeting come un controllo per la modifica del gene. Isolando gDNA dalle cellule prima e dopo la trasformazione sarà identificare cloni che sono stati selezionati positivamente per e hanno un vantaggio proliferativo. - Assemblare un 50 µ l PCR con i seguenti componenti: 25 µ l di 2 x PCR mix, 1 µ l di primer forward (10 µM), 1 µ l di primer reverse (10 µM), 50-100 ng di gDNA e H2O fino a 50 µ l.
Nota: Numerose le polimerasi ad alta fedeltà possono essere utilizzate nel passaggio 9.3. - Eseguire i campioni in un termociclatore utilizzando i seguenti parametri: 95° C per 1 min, 30 cicli di (94 ° C per 1 min, 52 ° C per 30 s, 72 ° C per 30 s) e 72 ° C per 10 min. La PCR è ottimizzato per i primers Calr riportati nella tabella 3. Ottimizzare le condizioni di PCR per la coppia di primer progettata nel passaggio 9,1 Basato su test su gDNA.
- Eseguire 5 µ l di campioni su un gel di agarosio 2% a 10 V/cm utilizzando tampone Tris-acetato-EDTA (TAE) 1x. Esaminare i campioni per la presenza / assenza di una fascia amplificata corrispondente nel formato al gene di interesse.
- Utilizzare il resto della reazione PCR (45 µ l) per eseguire una purificazione di PCR.
- Clonare gli ampliconi con una PCR kit di clonazione in un vettore plasmidico. Ad esempio, in genere vengono utilizzati vettori pGEM T-facile.
- Trasformare il plasmide in cellule Stbl3 o altre cellule compatibili e piastra su piastre di agar LB con l'antibiotico pertinente. Selezionare colonie di 10-20, mini-prep ciascuno e fatti salvi ogni clone Sanger sequenziamento per caratterizzare i indels creato dall'editing CRISPR/Cas9.
Nota: Se lo si desidera, delle cellule attivate la fluorescenza di singola cella che ordinamento (FACS) può essere eseguita sul grosso modificato popolazione per isolare un clone con un indel specifici. Inoltre, (NGS) metodi di sequenziamento di nuova generazione può essere utilizzato per quantificare più robustamente on target modifica mediante sequenziamento profondo degli ampliconi PCR che attraversa la regione di destinazione sgRNA. Ad esempio, Tracking di Indels di decomposizione o marea utilizzabile invece subcloning per determinare con precisione lo spettro e la frequenza delle mutazioni mirate generati in un pool di cellule (https://tide.nki.nl/#about)16.
Risultati
Utilizzando il metodo descritto qui, l'obiettivo di questo esperimento è quello di studiare gli effetti funzionali dell'introduzione di mutazioni indel al locus Calr endogeno sulla trasformazione delle cellule ematopoietiche. Il sistema CRISPR/Cas9 è usato come uno strumento per creare endogeno Calr mutazioni nelle cellule Ba/F3. Due sgRNAs sono stati scelti per indirizzare essone 9 di Calr (Figura 1), nella regione dove gli inserimenti e/o mutazioni di omissione (indel) si verificano in genere in CALR-mutante MPN pazienti17,18. Il primo sgRNA (m1) è stato scelto sulla sua scissione ad alta efficienza e favorevole fuori bersaglio punteggi base (tabella 1). Il secondo sgRNA (m2) è stato scelto principalmente per la sua posizione all'interno dell'essone 9 e per mancanza di ulteriori sgRNAs nella regione di modificare con fenditura ad alta efficienza e favorevole fuori bersaglio punteggi (tabella 1). Due distinti sgRNAs (m1 o m2) sono stati utilizzati nelle infezioni separate per garantire che gli effetti osservati erano dovuti a gene su target di editing. Fuori bersaglio effetti sono improbabili da essere condiviso da più indipendente sgRNAs. Il controllo non-targeting (scramble) è stato anche utilizzato come controllo negativo. Reclutamento del endonucleasi Cas9 a Calr essone 9 è preveduta per creare DSBs a questo luogo. Il DSBs quindi sarebbe stato riparato da NHEJ, che può generare indels di dimensioni variabili (Figura 1).
Per sviluppare l' in vitro CRISPR/Cas9 sistema in cellule Ba/F3, le cellule che esprimono la proteina Cas9 sono state fatte attraverso la trasduzione lentivirale-mediata secondo il protocollo descritto in precedenza. Cas9 espressione risultati stabili in robusto Cas9 attività. Attività di Cas9 in cellule Ba/F3-Cas9 è stato misurato da pXPR-011, una costruzione del reporter che contiene sia GFP e una guida GFP15di targeting. Celle contenenti Cas9 attivo si tradurrà in una riduzione di GFP15. GFP è stata misurata nelle cellule mediante citometria a flusso. Ba/F3-Cas9 cellule hanno visualizzato una riduzione di circa il 76% a GFP, corrispondenti a robusto Cas9 attività (Figura 2).
Digitare 1 recettori delle citochine, come il recettore della trombopoietina (MPL), il recettore dell'eritropoietina (EPOR) e del granulocyte distimolazione di fattore del ricevitore (G-CSFR) erano ciascuno individualmente, stabilmente espressa in cellule Ba/F3-Cas9 per determinare loro cooperativity con mutante calreticulina nell'induzione della trasformazione delle cellule Ba/F3. Trasduzione dei costrutti sgRNA (m1, m2 o un scramble (non-targeting Guida)) è stata quindi effettuata in ciascuna delle Ba/F3 linee cellulari che esprimono stabilmente Cas9 e il recettore di interesse. Cellule sono stati poi selezionate per 7 giorni con con puromicina per consentire tempo sufficiente per CRISPR/Cas9 gene editing. Una pressione di selezione positiva è stata poi applicata da morire di fame le cellule di citochina (mIL-3). L'obiettivo di questa pressione di inedia è identificare se indels in Calr essone 9, simili a quelli osservati in pazienti MPN, sono selezionati per, con conseguente crescita di citochina-indipendente e trasformazione delle cellule Ba/F3. La curva di crescita è stata effettuata per un totale di 8 giorni e le cellule sono state contate ogni 2 giorni per misurare la loro trasformazione (Figura 3)19. Pellet di cella per estrazione del DNA genomico sono stati raccolti prima dell'inizio della curva di crescita e alla fine della curva di crescita per verificare per l'editing on target e per monitorare i indels che espanso post citochina inedia (Figura 3)19.
Citochina crescita indipendente è stata osservata in Calr-mirato cellule Ba/F3-MPL-Cas9 (Figura 4B), ma non Calr-mirati cellule parentali di Ba/F3-Cas9 (Figura 4A), o Ba/F3-Cas9 cellule esprimendo ectopically EPOR (Figura 4 ) o G-CSFR (Figura 4)19. Per confermare che il crescita indipendente mIL-3 in cellule di targeting Calr Ba/F3-MPL-Cas9 era un risultato della modifica del gene il-obiettivo, le cellule erano raccolti 8 giorni post mIL-3 ritiro e DNA genomico è stato estratto19. Una regione di bp 422 che attraversa il sito di destinazione è stato amplificato utilizzando i primers riportati nella tabella 3. Sub-clonazione degli ampliconi PCR è stata effettuata e 30 singoli cloni sono stati inviati per Sanger sequenziamento. Per m1 o m2 Calr-mirati Ba/F3-MPL-Cas9 cellule, tutti i sub-cloni contenuti indels di varie dimensioni al sito taglio desiderato (Figura 5)19. 11 su 13 m1 Sub-cloni sono stati trovati per contenere indels che hanno portato a + 1 bp mutazioni frameshift (Figura 5), simili a quelli trovati in pazienti. Per m2 Calr-mirato cellule, Sub-cloni 10 su 17 sono stati trovati per contenere indels che hanno portato a + 1 bp frameshift (Figura 5)19. Questi dati confermano che CRISPR/Cas9-mediata introduzione di + mutazioni frameshift 1bp per l' endogeno Calr essone 9 locus è sufficiente a conferire attività oncogena a Calr19. I dati di sequenziamento nella Figura 5 suggeriscono anche che l'introduzione di eterozigoti + mutazioni frameshift 1bp al locus Calr endogena è sufficiente a trasformare cellule Ba/F3-MPL, coerente con l'osservazione che CALR le mutazioni sono in genere eterozigoti in MPN pazienti17,18. Dato NHEJ riparare risultati in alta eterogeneità tra i indels, singola cella ordinamento per isolare un clone di interesse potrebbe essere eseguita. In questo modo il ricercatore studiare gli effetti di mutazioni specifiche create da CRISPR/Cas9.
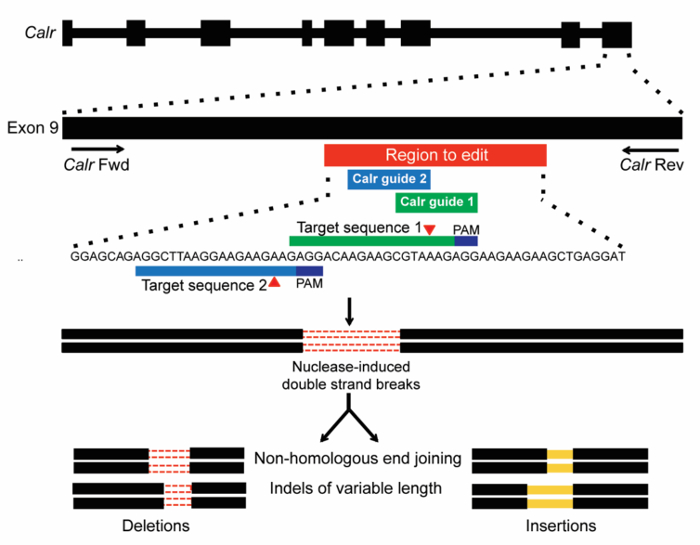
Figura 1: Schema di CRISPR/Cas9 modifica del gene targeting dell'essone 9 di Calr. Due sgRNAs sono stati progettati per destinare un sito all'interno della regione di essone 9 Calr del mouse corrispondente che la destinazione dei + 1bp mutazioni frameshift umano MPN (barra rossa). Sequenze bersaglio con PAMs (blu scuro) sono mostrati, e attesi siti di fenditura di Cas9 sono indicati da un triangolo. DSBs generati da CRISPR/Cas9 quindi sono riparati da NHEJ, che dovrebbe generare indels di lunghezza variabile19. Clicca qui per visualizzare una versione più grande di questa figura.
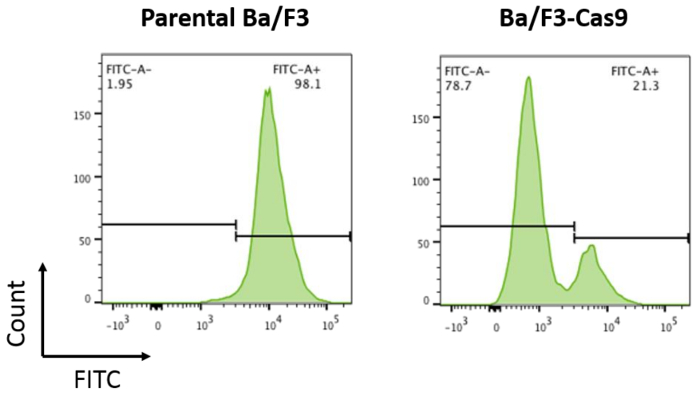
Figura 2: analisi di attività Cas9. Cellule parentali di Ba/F3 e Ba/F3 cellule che overexpressing Cas9 sono state trasdotte con pXPR-011 per misurare l'attività Cas9. Una riduzione di GFP correla con aumento dell'attività Cas9. Cellule Ba/F3 parentali sono ~ 100% FITC + rispetto a cellule Ba/F3-Cas9 dove il CIC è ridotto del ~ 76%. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Timeline per l'infezione e la selezione di sgRNAs in cellule Ba/F3. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Introduzione del + 1 bp frameshift mutazioni nel locus Calr endogeno è sufficiente a conferire attività oncogena di Calr. Curve di crescita (A-D) in parentale Ba/F3-Cas9 (A) Ba/F3-MPL-Cas9 cells (B), cellule Ba/F3-EPOR-Cas9 (C), e cellule Ba/F3-G-CSFR-Cas9 (D) dimostra IL-3 crescita indipendente nel targeting Calr Ba / F3-MPL-Cas9 cellule solo19. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 5: verifica di sequenziamento Sanger . Conferma di on target editing di endogeno Calr (esone 9) nelle cellule Ba/F3-MPL. + 1 bp frameshift mutazioni sono indicate in nero19. Clicca qui per visualizzare una versione più grande di questa figura.
| Gene target | sgRNA ID | Sequenza di destinazione (da 5' a 3') | Strand | Efficienza di scissione | Punteggio ottenuto fuori bersaglio | ||
| Calr | M1 | AGAGGACAAGAAGCGTAAAGAGG | + | 0,7 | 70 | ||
| Calr | m2 | GAGGCTTAAGGAAGAAGAAGAGG | + | 0.3 | 28 | ||
Tabella 01:20-mer protospacer sequenze tra cui il PAM sito (in grassetto) per le due sgRNAs targeting essone 9 di Calr 19.
| Primer ID | Sequenza (5' a 3') |
| M1-F | CACCGAGAGGACAAGAAGCGTAAAG |
| M1-R | AAACCTTTACGCTTCTTGTCCTCTC |
| m2-F | CACCGAGGCTTAAGGAAGAAGAAG |
| m2-R | AAACCTTCTTCTTCCTTAACCTC |
Tabella 2: sequenze di Protospacer e loro complementi inversione con "CACC" e "AAAC" aggiunto per clonazione in vettoriale pLenti-guida usando enzima di restrizione BsmBI 19.
| Primer ID | Sequenza (5' a 3') |
| Calr_Fwd | ACCACCTGTCTTTCCGTTCT |
| Calr_Rev | GGCCTCTACAGCTCATCCTT |
Tabella 3: CRISPR iniettori di PCR a Monte e a valle del sito di clivaggio sgRNA di screening.
Discussione
Qui dimostriamo l'uso di CRISPR/Cas9 gene editing per studiare la funzione biologica di CALR mutazioni nelle cellule ematopoietiche. Il successo di questo protocollo è altamente dipendente da molteplici fattori. In primo luogo, è importante sapere quali tipi di mutazioni possono essere responsabili del fenotipo che è desiderato. In questo protocollo, la lettura è la trasformazione delle cellule Ba/F3 per mIL-3 indipendenza e i tipi di mutazioni sono indels in essone 9 di CALR. Tuttavia, se la mutazione desiderata è una sostituzione di singola coppia di basi, quindi riparazione HDR-mediata è il metodo preferito perché si può introdurre le mutazioni di punto precise o inserimenti da un singolo filamento o double-stranded DNA donatore modello2, 3. Se si desidera knockout del gene, quindi la creazione di delezioni genomiche su larga scala primi essoni di codificazione di targeting è preferito20.
In secondo luogo, la linea cellulare deve essere citochina-dipendente al fine di consentire il ritiro di citochina di selezione positiva pressione post. È importante notare che non tutte le linee cellulari possono tollerare espressione stabile di Cas9 (Vedi punto 6.3). Questo potrebbe essere dovuto alla selezione di Dyfed, che non è tollerata da tutte le linee cellulari. In questo caso, scelta di altri costrutti Cas9 con cassette di selezione diversi può essere necessario. Un avvertimento di utilizzare linee di citochina-dipendente delle cellule, quali le cellule Ba/F3 è che essi sono suscettibili di crescita indipendente citochina spontanea, a volte come conseguenza dell'acquisizione di mutazioni nel gene ectopically espresso interesse seguendo citochina ritiro21. Pertanto, l'uso di adeguati controlli è tenuto per essere sicuri che la trasformazione cellulare osservata è dovuto on target CRISPR gene editing. In questo protocollo, non abbiamo osservato trasformazione cellulare in cellule Ba/F3-MPL trasformate con un controllo di sgRNA non-targeting o in Ba/F3-EPOR e Ba/F3-GCSFR cellule trasformate con Calr-mirati sgRNAs. Questo fornisce ulteriore sicurezza che trasformazione cellulare in cellule Ba/F3-MPL è un risultato di editing on target del locus Calr endogeno.
In questo protocollo, riparazione del DNA di NHEJ introduce indels di dimensioni variabili, come si vede nella Figura 5. Analisi di Sanger sequenziamento è stato effettuato su un sub-campione della popolazione di massa di celle modificate al fine di cercare l'espansione di indels che portano a + 1 bp mutazioni frameshift post citochina ritiro. Prossima generazione sequenziamento indicato nel passaggio 9,9 è un modo più robusto per quantificare il bersaglio editing per popolazioni cellulari all'ingrosso. Se si desidera l'analisi su un clone specifico, quindi singola cella ordinamento della popolazione alla rinfusa è necessario. Singola cella ordinamento è vantaggioso nel comprendere l'effetto biologico derivante da un tipo specifico di mutazione ed è utile quando l'obiettivo è comprendere le differenze tra due diversi tipi di mutazioni.
Una preoccupazione con il sistema CRISPR/Cas9 è fuori bersaglio effetti, che sono eventi di clivaggio fuori dalla regione di destinazione. Per valutare la specificità del sistema CRISPR/Cas9 e assicurarsi che le popolazioni trasformate non contengono effetti fuori bersaglio che conduce alla trasformazione osservata, avremmo dovuto esaminare se l'editing genomico si è verificato di fuori della regione di interesse. Questo potrebbe essere fatto cercando prove di Cas9/NHEJ-driven editing genomico alle potenziali loci genomici fuori bersaglio con somiglianza di sequenza significativa per la destinazione desiderata. Per questo, prossima generazione sequenziamento potrebbe essere utilizzato anche per valutare quantitativamente la frequenza degli effetti fuori bersaglio nei percorsi specificati lungo il genoma. Per ridurre il potenziale per gli effetti fuori bersaglio, diversi fattori potrebbero essere inseriti nel protocollo. Come accennato nei risultati, studiando più sgRNAs la regione di interesse di targeting assicura che il fenotipo è il risultato di un evento sulla destinazione. Inoltre, studi recenti hanno suggerito che sgRNAs troncato con 17 nucleotidi può ridurre la frequenza di clivaggio fuori bersaglio eventi22. Inoltre, l'esposizione prolungata a Cas9 da espressione stabile può provocare una maggiore frequenza di effetti fuori bersaglio. È importante notare che un sistema di doppio vettore contenente sia Cas9 e il sgRNA potrebbe essere utilizzato nel protocollo invece espressione stabile di Cas9; Tuttavia, questi vettori tendono ad essere molto grandi e provocare un titolo basso lentivirale ed efficienza di infezione inferiore. Di conseguenza, potrebbe essere utilizzato trasfezione transiente del costrutto Cas9 nelle cellule di nucleofection. I rapporti recenti hanno sviluppato Cas9 costrutti con maggiore specificità per l'editing on target, come il eSpCas9 costruire6.
In sintesi, CRISPR/Cas9 rappresenta un genoma efficiente, poco costoso ed affidabile strumento di ingegneria, consentendo lo studio delle mutazioni genetiche a livelli di espressione fisiologica. Qui ci avvaliamo di CRISPR/Cas9 gene editing come una tecnica robusta ed efficiente per far avanzare la comprensione degli effetti funzionali delle mutazioni di CALR in cellule ematopoietiche.
Divulgazioni
Non abbiamo conflitti di interesse relativi alla presente relazione.
Riconoscimenti
Questo lavoro è stato supportato da NIH (R01HL131835), un premio di ricercatore clinico di Damon Runyon e il Consorzio di cancro Starr.
Materiali
| Name | Company | Catalog Number | Comments |
| BsmBI | New England Biolabs | R0580L | |
| Blasticidin | Sigma Aldrich | 15025 | |
| Puromycin | Life Technologies | A1113803 | |
| Stbl3 cells | Life Technologies | C737303 | |
| TransIT-LT1 | Thermo Fisher Scientific | MIR2300 | |
| Opti-MEM | Life Technologies | 51985034 | |
| RPMI | Thermo Fisher Scientific | MT10040CV | |
| DMEM | Thermo Fisher Scientific | 10-017-CV | |
| Fetal bovine serum (FBS) | Omega Scientific | FB-11 | |
| Penicillin/streptomycin/L-glutamine | Life Technologies | 10378016 | |
| mIL-3 | Peprotech | 213-13 | |
| psPAX2 | Addgene | N/A | |
| pCMV-VSV-G | Addgene | N/A | |
| pLX_TRC311-Cas9 | Addgene | N/A | |
| polybrene | Sigma Aldrich | H9268 | |
| pXPR-011 | Addgene | N/A | |
| Phosphate Buffered Saline (PBS) | Genessee Scientific | 25-507 | |
| TAE buffer | Thermo Fisher Scientific | FERB49 | |
| LentiGuide-Puro | Addgene | Plasmid #52963 | |
| PNK | New England Biolabs | M0201S | |
| T4 ligase | New England Biolabs | M0202S | |
| PCR purification kit | Qiagen | 28104 | |
| Miniprep kit | Qiagen | 27104 |
Riferimenti
- DeWitt, M. A., et al. Selection-free genome editing of the sickle mutation in human adult hematopoietic stem/progenitor cells. Sci Trans Med. 8, 360(2016).
- Sander, J. D., Joung, J. K. CRISPR-Cas systems for editing, regulating and targeting genomes. Nat Biotechnol. 4, 347-355 (2014).
- Gaj, T., Gerbach, C. A., Barbas, C. F. ZFN, TALEN, CRISPR/Cas-based methods for genome engineering. Trends Biotechnol. 31 (7), 397-405 (2013).
- Jinek, M., et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Wiedenheft, B., Samuel, S. H., Doudna, J. A. RNA-guided genetic silencing systems in bacteria and archea. Nature. 482 (7385), 331-338 (2012).
- Slaymaker, I. M., Gao, L., Zetsche, B., Scott, D. A., Yan, W. X., Zhang, F. Rationally engineered Cas9 nucleases with improved specificity. Science. 351 (6268), 84-88 (2016).
- Ran, F. A., et al. Double nicking by RNA-guided CRISPR Cas9 for enhanced genome editing specificity. Cell. 154 (6), 1380-1389 (2013).
- Larson, M. H., et al. CRISPR interference (CRISPRi) for sequence-specific control of gene expression. Nat Protoc. 8 (11), 2180-2196 (2013).
- Wang, H., et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell. 153 (4), 910-918 (2013).
- Kim, H. S., et al. CRISPR/Cas9-mediated Gene-knockout screens and target identification via Whole genome sequencing uncover host genes required for picornavirus infection. J Biol Chem. 10, 1074(2017).
- Heckl, D., et al. Generation of mouse models of myeloid malignancies with combinatorial genetic lesions using CRISPR-Cas9 genome editing. Nat Biotechnol. 32 (9), 941-946 (2014).
- Daley, G. Q., Baltimore, D. Transformation of an interleukin 3-dependent hematopoietic cell line by the chronic myelogenous leukemia-specific bcr/abl protein. Proc Natl Acad Sci USA. 85, 9312-9316 (1988).
- Ran, F. A., et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc. 8 (11), 2281-2308 (2013).
- Haeussler, M., et al. Evaluation of off-target and on-target scoring algorithms and integration into the guide RNA selection tool CRISPOR. Genome Biol. 17, 148(2016).
- Doench, J. G., et al. Rational design of highly active sgRNAs for CRISPR-Cas9-mediated gene inactivation. Nat Biotechnol. 32, 1262-1267 (2014).
- Brinkman, E. K., et al. Easy quantitative assessment of genome editing by sequence trace decomposition. Nucl Acid Res. 42, e168(2014).
- Klampfl, T., et al. Somatic mutations of calreticulin in myeloproliferative neoplasms. N Engl J Med. 369, 2379-2390 (2013).
- Nangalia, J., et al. Somatic CALR mutations in myeloproliferative neoplasms with nonmutated JAK2. N Engl J Med. 369, 2391-2405 (2013).
- Elf, S., et al. Mutant Calreticulin requires both its mutant C-terminus and the thrombopoietin receptor for oncogenic transformation. Cancer Discovery. 6 (4), 368-381 (2016).
- Bauer, D. E., Canver, M. C., Orkin, S. H. Generation of Genomic Deletions in Mammalian Cell lines via CRISPR/Cas9. J. Vis. Exp. (95), e52118(2015).
- Watanabe-Smith, K., et al. Analysis of acquired mutations in transgenes arising in Ba/F3 transformation assays: findings and recommendations. Oncotarget. 8, 12596-12606 (2017).
- Fu, Y., et al. Improving CRISPR-Cas nuclease specificity using truncated guide RNAs. NatBiotechnol. 32, 279-284 (2014).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon