Method Article
Preparação de Mica e silício substratos para análise do DNA Origami e Experimentação
Neste Artigo
Resumo
Reproducible cleaning processes for substrates used in DNA origami research are described, including bench-top RCA cleaning and derivatization of silicon oxide. Protocols for surface preparation, DNA origami deposition, drying parameters, and simple experimental set-ups are illustrated.
Resumo
The designed nature and controlled, one-pot synthesis of DNA origami provides exciting opportunities in many fields, particularly nanoelectronics. Many of these applications require interaction with and adhesion of DNA nanostructures to a substrate. Due to its atomically flat and easily cleaned nature, mica has been the substrate of choice for DNA origami experiments. However, the practical applications of mica are relatively limited compared to those of semiconductor substrates. For this reason, a straightforward, stable, and repeatable process for DNA origami adhesion on derivatized silicon oxide is presented here. To promote the adhesion of DNA nanostructures to silicon oxide surface, a self-assembled monolayer of 3-aminopropyltriethoxysilane (APTES) is deposited from an aqueous solution that is compatible with many photoresists. The substrate must be cleaned of all organic and metal contaminants using Radio Corporation of America (RCA) cleaning processes and the native oxide layer must be etched to ensure a flat, functionalizable surface. Cleanrooms are equipped with facilities for silicon cleaning, however many components of DNA origami buffers and solutions are often not allowed in them due to contamination concerns. This manuscript describes the set-up and protocol for in-lab, small-scale silicon cleaning for researchers who do not have access to a cleanroom or would like to incorporate processes that could cause contamination of a cleanroom CMOS clean bench. Additionally, variables for regulating coverage are discussed and how to recognize and avoid common sample preparation problems is described.
Introdução
Introduzido pela primeira vez em 2006, origami de DNA utiliza a natureza de auto-montagem de oligonucleótidos de DNA para produzir nanoestruturas designable e altamente ordenados. 1 Uma miríade de estruturas têm sido relatados, que vão desde as caras do smiley travada para caixas em 3 dimensões. 2 origami de DNA pode ser funcionalizada com várias biomoléculas e nanoestruturas, dando origem a aplicações de investigação no domínio da nanoelectrónica, medicina e computação quântica. 3 No entanto, a análise e muitas aplicações futuras não são apenas dependente de concepção estrutural, mas também com a adesão das nanoestruturas origami de DNA em superfícies. Os métodos descritos neste manuscrito dizem respeito à preparação de amostras de ADN origami em dois tipos de substratos: mica e óxido de silício funcionalizado.
A mica é o substrato de escolha para estudos origami ADN porque é atomicamente plana, com uma altura de camada de 0,37 nm ± 0,02 nm. 4 É também easily limpo, tornando a preparação de amostras e microscopia de força atômica (AFM) estudos simples. Mica moscovite contém uma alta densidade de potássio em cada plano de clivagem, mas estes iões difundem para longe da superfície da mica quando em água. Para mediar a ligação de origami de DNA para o substrato de mica, Mg 2+ é usado para inverter a carga negativa da mica e se ligar electrostaticamente a espinha dorsal de fosfato do ADN para o substrato (Figura 1A). 5 As misturas de DNA emparelhado na presença de grande excessos de fios de grampos dar cobertura elevada e boas imagens em mica, porque a adesão de origami de DNA para Mg 2+ superfície do terminadas em é muito mais forte do que a adesão de oligonucleotídeos de cadeia simples (vertentes descontínuas). Outros iões carregados positivamente, incluindo Ni e Co 2+ 2+ pode ser utilizada para controlar a aderência de ADN em mica. 6,7 Alterar a concentração de catiões monovalentes e divalentes em solução pode mediar Adhetaxas de Sion e difusão superfície de origami de DNA. 8 No entanto, o protocolo para a preparação de substratos de mica e depositando e lavar o origami é muitas vezes não explicitamente descritos nos manuscritos publicados 9. Sem um protocolo claro, boa reprodutibilidade dos resultados pode ser difícil de obter.
A mica é um isolador, para que ele não é adequado como um substrato para algumas aplicações em nanoelectrónica. Silicon passivado com um óxido nativo fina tem propriedades eletrônicas desejáveis, incluindo a compatibilidade com o processamento antes de cortesia semicondutor de óxido metálico (CMOS) para criar estruturas de entrada / saída e características topográficas. Bolachas de silício são armazenados ao ar com passivado ou um óxido térmico de espessura ou película fina de óxido nativo que é relativamente sujo, com um elevado número de partículas. Óxido de silício tem uma muito mais baixa densidade de carga de superfície do que a mica, e a densidade de carga é altamente dependente de óxido de preparação e da história. Em concentrações de íons de magnésio above 150 mM, boas coberturas (até 4 / uM 2) de origami de DNA rectangular pode ser alcançado em substratos de silício de plasma tratado de oxigénio; no entanto, esta concentração e a cobertura pode mudar dependendo do tamanho e design das nanoestruturas a ser utilizado. 10 Um protocolo alternativo para ajustar a carga de superfície é para anexar uma monocamada auto-montada catiónico de 3-aminopropiltrietoxissilano (APTES) (Figura 1B) para o óxido. A amina primária na APTES pode ser protonado a valores de pH inferiores a 9, modificando a carga e hidrofobicidade do substrato. 11 Para uma monocamada completa de APTES a ser depositada com sucesso, o silício deve ser adequadamente limpo usando Radio Corporation of America (RCA) protocolos . Esses protocolos incluem tratamentos em hidróxido de amónio e soluções de peróxido de hidrogênio (RCA1) para remover resíduos orgânicos e contaminantes de partículas. Um curto etch em solução aquosa de ácido fluorídrico remove a camada de óxido nativo juntamente comquaisquer contaminantes iónicos que aderem ao óxido. Finalmente, as amostras são expostas a um ácido clorídrico e solução de peróxido de hidrogénio (RCA2) para remover o metal e contaminantes iónicos e formar uma camada fina de óxido, uniforme. 12 A maioria das salas limpas, designaram capuzes de protocolos de limpeza CMOS, com regras estritas sobre o que pode ser usada nestas áreas. Um problema comum vem sob a forma de íons tais como o sódio, o que pode perturbar as propriedades eletrônicas de estruturas CMOS criando armadilhas midbandgap. 13 íons comumente usado em origami de DNA de preparação e de deposição buffers poderia contaminar os banhos de CMOS e causar problemas para outros pesquisadores que utilizam o quarto limpo. Por esta razão, o nosso grupo utiliza um CMOS 'sujos' limpeza banco organizados especificamente para as pequenas amostras utilizadas para a investigação origami de DNA. Este processo é uma boa alternativa para a sala limpa tradicional set-up e pode ser adequado para laboratórios que não têm acesso a um banco de CMOS de sala limpa.
Protocolo
1. Experiência Planejamento e preparação do material
- Determinar o desenho, a concentração, ea funcionalidade do origami de DNA que vai ser usado nas experiências 14-16. Aqui, utilizamos um design ADN origami rectângulo preparado em 1x TAE / Mg 2+ (40 mM de base Tris, 20 mM ácido acético, EDTA 2 mM e acetato de magnésio 12 mM, pH 8,0). 17
- Autoclave todas as dicas, tubos e recipientes para ser usado. Estes materiais devem ser todos autoclave compatível.
- Prepare um suprimento de água esterilizada para a lavagem. Encha uma jarra estéril com cerca de 500 ml de 18 mohms x cm de água, coloque em uma chapa quente, deixe ferver por 5 minutos com a tampa fora, e tomar o pote fora do fogão e deixe esfriar com a tampa colocada no recipiente, mas não apertado . Guarde na geladeira e preparar uma nova oferta a cada mês ou quando necessário.
2. Preparar o Mica Substrato
- Cut substratos para o tamanho adequado (1 cm x 1 centímetro quadrados) com uma tesoura. A mica é fina e frágil. Alternativamente, compra de mica em forma de disco que não exige o corte.
- Clivar o mica usando fita dupla face. Mica é composta por camadas de minerais separados por intercalando íons, cada camada pode ser retirada quando adere a fita dupla face. 18
- Coloque os quadrados mica na fita dupla face ainda no dispensador de fita, certificando-se que é aderida firmemente à fita. Deslize cuidadosamente a pinça entre o mica e da fita, o mais alto camada serão removidos e permanecem na fita. Imediatamente após a remoção, o quadrado de mica terá o seu lado limpo virada para baixo. Certifique-se de virar a mica sobre antes de armazenar em um recipiente.
- Repetir esta três a quatro vezes para assegurar a remoção completa da camada mais superior e a limpeza adequada.
- Alternativamente, a mica aderir a um recipiente ou de mesa com um pedaço de fita adesiva de dupla face e utilizar uma segunda peça de sassinale a e retire a camada mais superior mica. A mica serão adequadamente feita em ambos os casos, embora o método alternativo do depósito faz com que o ADN e lavagem e secagem da amostra difícil devido à segunda adesão ao suporte.
3. Depositar DNA Origami em Mica
- Resumidamente, misture o frasco de origami de DNA usando um misturador de vórtice para assegurar uma dispersão de nanoestruturas em solução.
- Pipeta 4 ul de solução sobre a mica, assegurando que a ponta da pipeta não toque no substrato. Deixe o origami de DNA no mica por cerca de 10 min para garantir uma cobertura adequada. O tempo de deposição pode variar, dependendo da concentração de origami de DNA (Figuras 2 e 3) usado, bem como a cobertura desejada.
- Lavar a solução de origami de DNA fora do substrato mica utilizando 100 ml de água estéril sobre uma pia ou outro recipiente líquido. Pegue o mica, usando uma pinça. Pipeta da água nosubstrato, com o fluxo da gota para a ponta da pinça. Agitar a mica com um movimento para baixo afiadas para remover o excesso de água. Segurar a pinça na vertical de modo que a água vai fluir para as pinças para evitar a contaminação da amostra.
- Seca-se o substrato com um fluxo constante de azoto (N 2) durante 1 min. Certifique-se que o excesso de água é removido. Repetir a lavagem com um adicional de 100 ul de água estéril. Seca-se o substrato com N2 durante mais 3 minutos. Um substrato completamente seca é necessário para análise bem sucedida de microscopia de força atómica (AFM) (Figura 4).
- Analisar o substrato utilizando AFM ou armazene em um recipiente fechado.
4. CMOS / Silicon Cleaning Set-up
- CUIDADO: Ao usar o CMOS set-up, use equipamentos de proteção individual em todos os momentos. Os reagentes incluem ácidos fortes, bases fortes, ácido fluorídrico (HF), e agentes oxidantes fortes, que cAn reagir com solventes residuais se reagentes não são devidamente eliminados. Observar as seguintes precauções de segurança:
- Casa CMOS banco em um capuz químico com nenhum outro processo ou set-ups.
- Use luvas de borracha nitrílica, revestimento do laboratório, óculos de segurança, luvas de borracha nitrílica grandes industriais, um avental derramamento, e uma viseira em todos os momentos ao usar o banco CMOS.
- Use banheiras de plástico como contenção secundária quando as soluções são preparadas.
- Use um copo graduado fluorado polímero inerte para a manipulação do HF concentrado.
- Faça cálcio gluconato de pomada disponível como primeiros socorros para qualquer exposição da pele.
- Apenas permitir que o pessoal devidamente treinado para realizar o processo.
- Sempre garantir um outro membro do laboratório está presente em caso de emergência.
- Mantenha as informações MSDS para todos os produtos químicos perto do capô.
- Estar familiarizado com derramamento de produtos químicos e de exposição das políticas da empresa ou da instituição de.
Nota: HF prontamente permeia a pelee é um limpador de cálcio, afetando ossos e nervos prejudiciais se ocorrer exposição. Exposição cutânea a alguns mililitros de ácido fluorídrico concentrado pode ser perigoso e até fatal. Estabelecer as precauções necessárias para assegurar que a exposição não ocorre.
- Execute RCA1 e RCA2 em um separado 250 ml copos de vidro em placas separadas. Cada copo deve conter uma barra de agitação. Monitorar a temperatura da solução usando termómetros apertadas de modo a barra de agitação não bater no bulbo. Cobrir as provetas, utilizando um vidro de relógio para diminuir os efeitos de evaporação.
- RCA1 Preparação
- Coloque 50 ml de 18 mohms x cm de água no copo RCA1 designado utilizando um copo de medição.
- Adicionar 15 ml de hidróxido de amónio concentrado (NH4OH) para o copo. Lavar o copo de medição com 25 ml de água e adicionar a água da lavagem para o copo RCA1.
- Ligar o agitador e calor numa placa de aquecimento e trazer o banho RCA1 a 70 ° C.
- Adicionar 15 ml de 30% de peróxido de hidrogénio (H 2 O 2) para o copo de RCA1. Usar a solução RCA1 dentro de 1 hora após a H 2 O 2 foi adicionado. O banho pode ser utilizado várias vezes dentro do intervalo de três dias, se 15 ml de peróxido é adicionado ao banho de cada vez.
- Lavar o copo de medição cuidadosamente com água e descartar a lavagem em um frasco de resíduos RCA1 apropriado.
- RCA2 Preparação
- Adicionar 70 ml de 18 mohms x cm de água para o copo RCA2 designado utilizando o copo de medição cuidadosamente lavados.
- Adicionar 15 ml de ácido clorídrico concentrado (HCl). Lavar o copo de medição com 20 ml de água e adicioná-lo ao copo RCA2.
- Aumentar o aquecimento e agita-se a velocidade da placa de aquecimento até a solução atingir 70 ° C.
- Adicionar 15 ml de 30% de H 2 O 2. Como o banho RCA1, usar esta solução dentro de uma hora a partir de quando o H 2 O 2 é adicionado; Além disso, o banhopode ser reutilizado várias vezes dentro do intervalo de três dias, se 15 ml de H 2 O 2 é adicionada antes de cada utilização.
- IC Preparação de Solução
- Colocar 50 ml de água numa proveta de polímero fluorado inerte.
- Medida 4 ml de ácido fluorídrico concentrado (49%) na proveta de plástico de medição e adicioná-lo no copo de polímero fluorado inerte.
- Lave o plástico medindo copo com um total de 50 ml de água, adicionando a água da lavagem para o copo HF. Lave o copo de medição cuidadosamente com água e descartar as lavagens em um recipiente de descarte designado HF.
5. Preparação e limpeza do Silício Substrato
- Corte de wafers de silício em chips
- Identificar as direcções perpendiculares e paralelas treliça sobre a superfície polida plana da bolacha de silício. Estas instruções são usadas para ajudar a tornar mais fácil clivagem quadrados. As instruções a seguir referem-se a cleaving silício <110> e podem não ser adequados para outras orientações cristalinas.
- Coloque o wafer de silício polido-side-up em uma superfície macia, como um guardanapo. Usando a caneta com ponta de diamante escriba, suavemente do entalhe da parte inferior do disco de contacto ao longo da borda plana primário. Coloque um fio pequeno, como um clipe de papel, abaixo do nick e aplique suavemente pressão para a bolacha, colocando os dedos ou com uma pinça de cada lado do nick e empurrando para baixo. Fazendo isso irá separar o wafer em duas metades ao longo da linha de rede cristalina na direção decompor natural.
- Por outro guardanapo, com um lápis e régua, medir a largura desejada dos pontos quadrados por marcação sobre a parte superior e a parte inferior do penso. Conectar esses pontos com linhas retas. Isto irá servir como um guia para formas mesmo quadrados.
- Coloque uma das metades wafer ponta-primeiro entre as linhas medidas no guardanapo lavada contra a linha e repita o passo em 5.1.2 O recém quebrado perpendicular pieces deve agora ser a largura dos quadrados clivados. Rode o wafer horizontalmente no guardanapo. Coloque-o entre as linhas perpendiculares e repita o processo na etapa 5.1.2.
- Armazenar os chips da bolacha recém clivada num frasco limpo cheio de água DI para evitar arranhões. Os chips de silício pode ser armazenado indefinidamente, mas deve ser limpa antes de iniciar as experiências.
- Limpeza de CMOS Silicon
- Quando a solução RCA1 atingiu a temperatura adequada, submergir oito a dez de 1 cm x 1 centímetro chips de silício na solução usando um cesto de polímero fluorado inerte com um "diâmetro 2. As bolhas de oxigénio se formam sobre as fichas e as paredes da taça. Se nenhum borbulhante ocorre, o H 2 O 2 é degradado. Deixe os chips na solução por 10 a 20 min, agitando o cesto cima e para baixo a cada poucos minutos para manter os chips grudem umas nas outras.
- Levante a cesta contendo os chips de silício para cima e escorra bem. Mova a cesta sobre to do copo de resíduos e enxaguar bem com 18 mohms x cm de água. Mergulhe no copo de lavagem e sacudir para cima e para baixo por 20 s. Escorra o cesto e lavar abundantemente com água sobre o copo de resíduos. Esvazie o copo de resíduos em um frasco de resíduos RCA1 designado e encher com água.
- Após a limpeza RCA1 é completa, colocar o cesto na proveta 01:50 HF para de 10 a 20 seg. Use uma suave para cima e para baixo movimento de misturar as batatas fritas e HF. Levante o balde enterrada para permitir que o HF para drenar completamente.
Nota: As superfícies de fichas deve ser hidrofóbico; a água não vai molhar o chip, mas em vez disso formar gotas com ângulos de contacto elevadas. Isto indica que o óxido de silício tem sido gravado distância e o chip está agora terminada por ligações Si-H. - Colocar o copo de lavagem e enxágüe copo em uma banheira de plástico, mova a cesta sobre o copo de lavagem e enxágüe com 18 mohms x cm de água. Mergulhe a cesta no copo de lavagem e agitar por 20 s.
- Conclua uma segunda drenagem e rinsciclo e com 18 cm x mohms água. Despejar a água de lavagem para o copo de lavagem e encher o copo de lavagem com água. Despeje todos os resíduos em um frasco de resíduos de plástico HF designado.
- Quando a solução RCA2 atingiu a temperatura adequada, submergir os chips de silício nas soluções que utilizam o cesto. Deixar na solução durante 10 a 20 min. O chip vai agora ser hidrofílico, devido ao crescimento de uma (1-2 nm) óxido de película fina.
- Remover após a quantidade adequada de tempo e siga os mesmos procedimentos de lavagem como para RCA1. Eliminar os resíduos no recipiente de resíduos RCA2 apropriado. Remova cada chip da cesta com uma pinça de plástico, lave com água e seque com nitrogênio.
- Armazene batatas fritas em uma caixa de plástico ou bolacha em um frasco de 18 mohms x cm de água. O silício permanecerá limpo quando armazenados em água por aproximadamente três dias após a limpeza. Certifique-se de que a área de trabalho está devidamente limpo eo exterior das luvas CMOS foram lavados. Deixe a CMLuvas OS na cobertura para secar.
6. Depositar DNA Origami em Silicon APTES-funcionalizada
- Formação de auto-montados Monolayer em Silicon
- APTES aquecer à RT antes da abertura. Se a garrafa é muito frio, poderá ocorrer condensação, causando hidrólise dos APTES durante o armazenamento. Adicionar 1980 mL de 18 mohms x cm de água e 20 mL de APTES para um frasco de cintilação limpa e agite para misturar. Use esta solução imediatamente.
- Coloque um chip de silício limpos reflexivo-side-up no frasco de cintilação, tampá-lo, e deixe descansar por 20 min. Remover o chip utilizando pinças e lavar com 200 mL de água e seco durante 1 minuto com uma corrente de N 2.
- Depositar origami de DNA em APTES funcionalizados Silicon
Nota: Os passos para depositar origami de DNA em silício funcionalizado são análogas àquelas para depositar sobre a mica.- Resumidamente misturar o frasco origami de DNA e pipeta de 4 ml de soluçãosobre o substrato de silício. Se necessário, aumentar o volume de solução origami de DNA usadas para cobrir todo o substrato como o silicone funcionalizado é mais hidrofóbica do que o substrato de mica. Use uma lamela de vidro para pressionar a solução de deposição para baixo e evitar a evaporação durante longos depoimentos.
- Deixar a solução repousar durante a quantidade de tempo necessário para a concentração utilizada e a cobertura desejada (ver Figuras 2 e 3 para o efeito do tempo e da concentração na cobertura da superfície). Lavar o substrato com 100 ul de estéril 18 mohms x cm de água e seca com N 2 durante 1 min.
- Repetir a lavagem com um adicional de 100 ul de água estéril e secar o substrato com N 2 durante 3 min.
- Conservar a amostra em um recipiente limpo até que novos experimentos ou imagem pode ser realizada. Amostras começam a mostrar acumulação de partículas após cerca de uma a duas semanas de armazenamento de acordo com a forma como muCH eles são tratados.
Imagem 7. AFM e Análise de Imagem de Amostras de DNA de Origami
- Use AFM no modo de Tocar no ar para fins de imagem. Modo de batimento que garante um mínimo de força será aplicada às nanoestruturas frágeis, em comparação com o modo de contacto.
Nota: Os parâmetros de imagem será dependente do instrumento. Todas as imagens apresentadas foram capturados usando um MultiMode Nanoscope IIIa. - Seleccione sondas AFM para não-contato / modo no ar batendo, com revestimento reflector de ouro, uma freqüência de ressonância (nominal) de ~ 300 kHz, forçar constante de 40 raio N / m, e de ponta <10 nm.
- Processar e analisar as imagens de AFM, utilizando Nanoscope Analysis Software. Realizar cálculos de cobertura usando ImageJ. 19
Resultados
Duas variáveis ditar a cobertura de origami de DNA sobre o substrato: concentração da solução e do tempo de exposição. As características de adsorção de origami ADN sobre mica e APTES óxido de silício funcionalizados foram anteriormente relatados. 13 A relação entre a concentração de origami de DNA na solução de deposição e as coberturas finais sobre mica estão resumidos na Tabela 1 e Figura 2, que mostra os resultados de concentração crescentes num aumento da cobertura. O tempo-dependência da ligação é visto na cobertura da superfície Figura 3. Foi previamente estudado para quantificar o comportamento de ligação de origami de DNA em superfícies mica e óxido de silício modificados. Origami de DNA em 12 mM de magnésio em tampão 1 x TAE tem 83,3% ± 3,1% de cobertura em mica após 30 min de tempo de absorção na superfície. A cobertura máxima de óxido de silício modificado com APTES SAM é observada após 60 min, o que é menos do que a cobertura máxima em mica. UMAé necessário mais tempo de deposição se uma cobertura de superfície elevada é requerida no APTES funcionalizados de óxido de silício.
Há diversas variáveis que podem causar má preparação da amostra. O mais problemático é lavagem inadequada e secagem. Se a solução tampão não é enxaguada adequadamente, formar grandes agregados sobre o substrato (Figura 4A). são observados 'ilhas de ADN origami' quando as nanoestruturas aderir a manchas de sais de magnésio na superfície (Figura 4B). Finalmente, as amostras com elevado de cobertura, é possível ter os componentes do tampão em excesso em ponte entre origami de DNA individuais (Figura 4C), o que o torna difícil de diferenciar nanoestruturas utilizando AFM. Estes resultados podem ser evitados, seguindo uma lavagem completa e protocolo de secagem para ambos os substratos de mica e de silício.
A formação de filmes de óxido de APTES em substratos de silício pode levantar problemas bem. Wafers de silício têm umcamada de óxido de silício em bruto que tem de ser removido e uma camada mais fina suave, óxido de silício reformada antes que possa ser funcionalizada. Um substrato de silício devidamente limpo é ilustrada na Figura 5A. Durante a limpeza da bolacha de silício, é importante para garantir que o número de chips de silício não é demasiado elevado, uma vez que é possível para dois chips para ficar preso ao outro, impedindo a exposição dos reagentes (Figura 5B). Se silício limpos foi armazenada em 18 mohms x cm de água para mais de uma semana, a camada contaminante vai reformar e recleaning é necessário. O fornecimento APTES também pode causar problemas com a preparação da amostra. APTES polimeriza prontamente por hidrólise, que é a base para a formação de monocamada. 20 A extensão da polimerização esta é dependente da concentração de água que o APTES está exposta. Ao longo do tempo e com o uso repetido, é possível que a água condensar-se dentro do frasco e contaminar o APTESfornecem. A polimerização resultante produz grandes agregados que aderem ao substrato (Figura 5C). O aumento da rugosidade e presença de agregados torna a identificação de DNA nanoestruturas usando AFM difícil. É uma boa prática para armazenar a garrafa APTES em um saco plástico na geladeira, e deixar o APTES garrafa quente para RT antes de abrir para evitar a condensação.
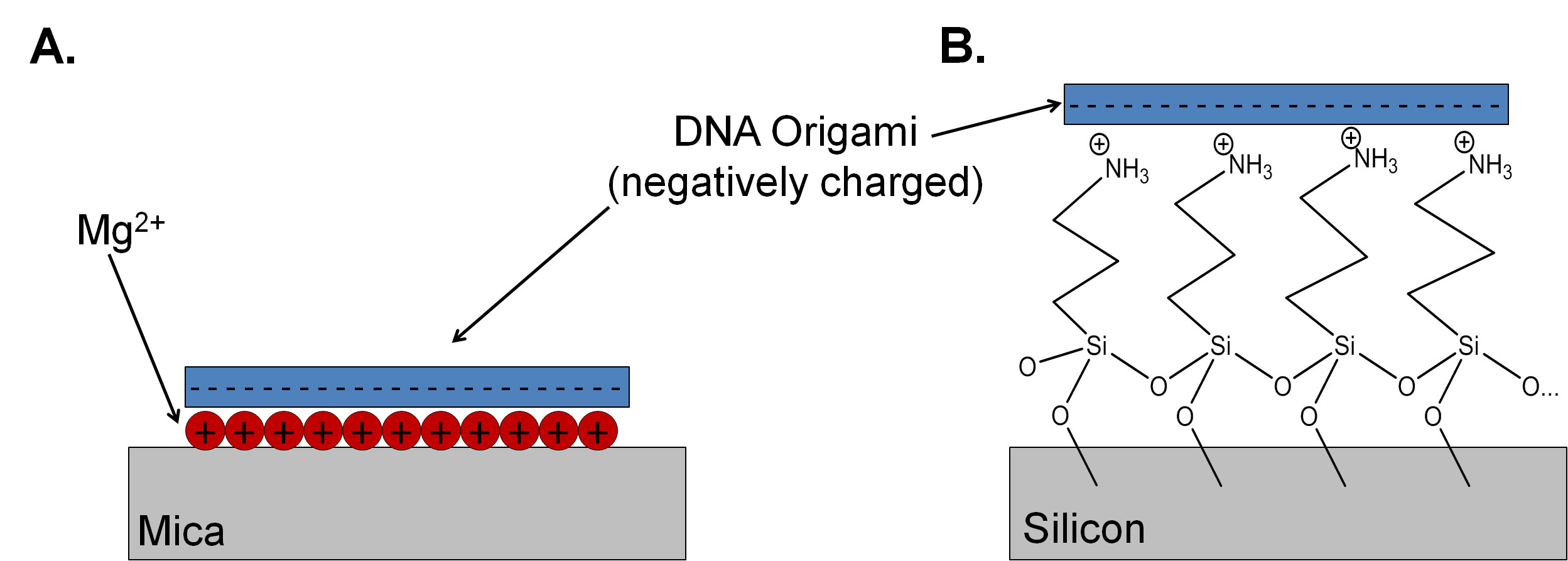
Figura 1. Um óxido de silício comparando o mecanismo de ligação para origami de DNA de (a) a mica e (B) APTES funcionalizado esquemático (sem escala). A ligação com a mica é mediada pela presença de catiões divalentes, geralmente Mg 2+. Uma monocamada da amina protonada terminada 3-aminopropiltrietoxissilano é usado para promover a adesão sobre o substrato de óxido de silício.

Figura 2. imagens AFM ilustram cobertura variável após 10 min de deposição (A) 2 nM, (B), 4 nM, e (C) 6 nM em mica. A escala da altura de todas as imagens é de 5 nm.

Figura 3. Tendências na cobertura da superfície por 2 nM origami de ADN em tampão 1x TAE, Mg 2+ 12 mM. MICA = linha de roxo e do círculo marcadores, APTES = linhas amarelas e marcadores triângulo. N = 3 na determinação do erro padrão.

Figura 4. Excelente lavagem e secagem podem causar (A) agregação solução sobre o substrato, (B) ilhas origami de ADN, e (C) de ponte em nanoestruturasamostras alta cobertura na presença de sais do tampão em excesso. A escala da altura de todas as imagens é de 5 nm.

Figura 5. (A) de silício Limpo deve ter RMS rugosidade inferior a 0,5 Å mais de 1 micron quadrado. Um completo APTES SAM depositado em um bom óxido nativo deve ter uma rugosidade RMS menos de 1 Å mais de 1 micron quadrado. (B) APTES filme formado em um óxido de silício de forma incompleta limpo com RMS aspereza de 2,29 nm ao longo de 1 micron quadrado. . Observe as lacunas e rugosidade do filme APTES (C) APTES superfície formada a partir de uma amostra de APTES contaminados com resíduos condensado; hidrólise nas formas de garrafa APTES partículas grandes. A escala de altura para todas as imagens é de 5 nm.
Tabela 1. medições de cobertura por cento para substratos de mica com varyinDNA g concentrações de solução origami. Todos os tempos de deposição são 10 min.
| Solução ADN Origami | % De cobertura na Mica Substrato |
| 2 nM | 8,49 ± 2,67 (n = 5) |
| 4 nM | 55,89 ± 5,65 (n = 3) |
| 6 nM | 77,44 ± 1,89 (n = 4) |
Discussão
Há vários passos que precisam ser enfatizados para atingir resultados consistentes e ideais. Para amostras de mica, após uma lavagem rigorosa e minuciosa e que secam regime, como nos passos 3.3 e 3.4, vai assegurar que as imagens de origami de DNA indivíduo de alta qualidade pode ser alcançada usando AFM sem os vários problemas descritos na seção Resultados Representante. De primordial importância para as amostras de silício é a limpeza do substrato. Seguindo os procedimentos de limpeza descritas na etapa 5.2 cuidadosamente e meticulosamente vai garantir que uma superfície de óxido de silício apropriadamente limpa irá ser alcançado. Além disso, o monitoramento da qualidade e eficácia dos produtos químicos, como o peróxido de hidrogênio, ácido fluorídrico, e APTES, irá garantir que o processo corra bem.
As técnicas descritas não estão limitadas a soluções aquosas de APTES. A monocamada misto de APTES e cloreto trimethylaminopropyltrimethoxysilyl (TMAC) pode ser-nosEd para sintonizar a carga de superfície de silício e promover a adesão variável origami de DNA. 11 O TMAC contém uma quaternário terminal carregado permanentemente amina -N (CH3) 3 +, em comparação com o pH de carga dependente APTES. Uma vez que o ambiente solução não afecta o estado de protonação ou a carga de TMAC, variando a concentração da solução de TMAC pode sintonizar a carga superficial da monocamada mista e afectar a interacção entre o substrato e o origami de DNA. Origami de DNA óptima foi observada ligação para monocamadas possuindo uma carga de superfície de cargas 0,75-1,5 / 2 nm, o que corresponde a SAM contendo 100% a 40% de concentração de TMAC. Esta carga de superfície ideal solicitado coberturas origami de DNA de aproximadamente 110 origami / mm 2 no APTES SAMs e 120 origami / mm 2 na TMAC SAMs.
Uma vantagem de silício é a sua compatibilidade com processos de padronização litográfica. Alta de magnésioconcentrações pode ser utilizado com os óxidos de silício tratado com plasma para promover a ligação selectiva de origami de DNA para o silício. Cuidados devem ser tomados quando retirar a amostra da solução de deposição para evitar lavagem dos íons de magnésio distância e deformando o origami de DNA. 21,22 As formas de processo APTES covalentemente cátions na superfície do óxido de silício, por isso lavagem ou lavagem com tampão ou água faz não danificar o origami de DNA ligado. O método 'decolagem molecular "é outra rota possível para padronização substratos de silício e promover a adesão origami de DNA. O substrato de silício é modelado utilizando litografia por feixe de electrões e APTES é depositado no substrato exposta. Seguindo decolagem do fotorresistente, origami de DNA pode ser depositada na superfície modelada, preferencialmente de ligação para os APTES. 23
A interação de origami de DNA com um substrato muda a sua estabilidade, abrindo novos caminhos para pesquisas e aplicações. Origam DNAi adere a mica pode ser aquecida até 150 ° C sem alteração visível de dimensões de nanoestrutura e das modificações químicas mínimas. 24 Isto está em contraste com a fragilidade do origami de DNA em solução, onde os nanoestruturas são dehybridized completa acima de 70 ° C. 25, 26 Esta estabilidade é mantida sobre suportes de óxido de silício aquecidos. 27 Mesmo em diversos sistemas solventes, tais como hexano, tolueno, etanol e, a forma e a cobertura das nanoestruturas são mantidas. A estabilidade surpreendente de origami de DNA indica que as aplicações que foram anteriormente consideradas incompatíveis, tais como deposição de vapor por plasma, utilização de fotorresistentes e solventes comuns, e ambientes de deposição química única, pode ser usada em conjunto com origami de DNA. No entanto, se o origami de DNA manter a sua funcionalidade ainda é desconhecida e pode limitar possíveis aplicações.
Embora a estabilidade de origami de DNA a uma temperatura elevadas e num número limitado de sistemas solventes tenha sido determinada, a estabilidade a longo prazo de origami de DNA em substratos ainda é desconhecida. A utilização de técnicas de esterilidade é necessário para evitar a possibilidade de contaminação, mas isto não pode ser evitada depois da deposição superficial. Imagem e análise de amostras deve ser concluída quase que imediatamente após a preparação da amostra; Se a amostra é armazenada durante muito tempo (mais de uma semana) vários exemplos de degradação da amostra são frequentemente identificados, incluindo a acumulação de partículas e nano-estruturas de ADN quebradas. Possíveis vias de investigação em nanoelectrónica, biosensoriamento, e outras aplicações origami de ADN substrato base pode ser limitado por dependente do tempo de desestabilização do ADN. Identificar as limitações dessas técnicas para aplicações que exigem estabilidade para longos períodos de tempo requer uma investigação mais aprofundada.
Divulgações
The authors have nothing to disclose.
Agradecimentos
The authors thank Dr. Gary Bernstein for use of the AFM.
Materiais
| Name | Company | Catalog Number | Comments |
| Eppendorf epT.I.P.S. Reloads, capacity 2-200 μl | VWR International, LLC | 22491733 | 10 reload tray of 96 tips |
| Microcentrifuge Tubes, Polypropylene | VWR International, LLC | 87003-290 | 0.65 ml, natural |
| Research Plus Pippete - Single Channel - 20-200 μl | A. Daigger & Company, Inc. | EF8960F-3120000054 EACH | Adjustable Volume |
| Research Plus Pippete - Single Channel - 2-20 μl | A. Daigger & Company, Inc. | EF8960D-3120000038 EACH | Adjustable Volume |
| Scotch 237 Permanent Double-Sided Tape | Office Depot, Inc. | 602710 | 3/4" x 300", Pack of 2 |
| Vortex Mixer | Thermo Scientific | M37610-33Q | |
| Wafer container single, 2" (50 mm), 60 mm x 11 mm | Electron Microscopy Sciences | 64917-2 | 6 per pack |
| 6" Wafer, P-type, <100> orientation, w/ primary flat | Nova Electronic Materials, Ltd. | GC49266 | |
| Powder-Free Nitrile Examination Gloves | VWR International, LLC | 82062-428 | Catalog number is for size large |
| High Accuracy Noncontact probes with Au reflective coating | K-Tek Nanotechnology, Inc. | HA_NC/15 | |
| Autoclave Pan | A. Daigger & Company, Inc. | NAL692-5000 EF25341C | |
| Sol-Vex II Aggressive Gloves, Size: 9-9.5; 15 mil, 13 inch - 1 dz | Spectrum Chemical Mfg. Corp. | 106-15055 | Before use, rinse with water and scrub together until no bubbles form on the gloves. |
| Tweezers PTFE 200 mm Square | Dynalon Corp. | 316504-0002 | |
| Muscovite Mica Sheets V-5 Quality | Electron Microscopy Sciences | 71850-01 | 10 per pack |
| Mica Disc, 10 mm | Ted Pella, Inc | 50 | Mica discs are optional |
| Scriber Diamon Pen for Glassware | VWR International, LLC | 52865-005 | |
| Scintillation Vials, Borosilicate Glass, with Screw Cap - 20 ml | VWR International, LLC | 66022-060 | Case of 500, with attached polypropylene cap and pulp foil liner |
| 4 x 5 Inch Top PC-200 Hot Plate, 120 V/60 Hz | Dot Scientific, Inc. | 6759-200 | |
| Straight-Sided Glass Jars, Wide Mouth | VWR International, LLC | 89043-554 | Case of 254, caps with pulp/vinyl liner attached |
| Standar-Grade Glass Beaker, 250 ml Capacity | VWR International, LLC | 173506 | |
| Beakers, PTFE | VWR International, LLC | 89026-022 | For use with HF |
| Shallow form watch glass, 3" | VWR International, LLC | 66112-107 | Case of 12 |
| Plastic Storage Container | VWR International, LLC | 470195-354 | For secondary container |
| General-Purpose Liquid-In-Glass Thermometers | VWR International, LLC | 89095-564 | |
| High precision and ultra fine tweezers | Electron Microscopy Sciences | 78310-0 | |
| Polycarbonate Faceshield | Fisher Scientific, Inc. | 18-999-4542 | |
| Neoprene Apron | Fisher Scientific, Inc. | 19-810-609 | |
| Calcium Gluconate, Calgonate | W.W Grainger, Inc. | 13W861 | Tube, 25 g |
| Hydrogen Peroxide 30% CR ACS 500 ml | Fisher Scientific, Inc. | H325 500 | HARMFUL, TOXIC |
| 3-Aminopropyltriethoxysilane | Gelest Inc. | SIA0610.0-25GM | Let warm to room temperature before use. |
| Ammonium hydroxide, 2.5 L | Fisher Scientific, Inc. | A669-212 | HARMFUL, TOXIC |
| Hydrochloric acid | Fisher Scientific, Inc. | A144-212 | HARMFUL, TOXIC |
| Hydrofluoric acid | Fisher Scientific, Inc. | A147-1LB | HARMFUL, TOXIC |
| MultiMode Nanoscope IIIa | Veeco Instruments, Inc. | Any AFM capable of tapping mode is suitable for analysis | |
| Dunk basket | Made in lab | Made in lab | The dunk basket was made using the bottom of a PTFE bottle with holes drilled in, PTFE handle, and all PTFE screws. |
Referências
- Rothemund, P. W. K. Folding DNA to create nanoscale shapes and patterns. Nature. 440, 297-302 (2006).
- Anderson, E. S., et al. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 459, 73-77 (2009).
- Wang, Z., Ding, B. Engineering DNA Self-Assemblies as Templates for Functional Nanostructures. Acc. Chem. Res. 47, 1654-1662 (2014).
- Xu, K., et al. Graphene Visualizes the First Water Adlayers on Mica at Ambient Conditions. Science. 329, 1188-1191 (2010).
- Bustamante, C., et al. Circular DNA-molecules imaged in air by scanning force microscopy. Biochemistry. 31, 22-26 (1992).
- Hsueh, C., et al. Localized Nanoscopic Surface Measurements of Nickel-Modified Mica for Single-Molecule DNA Sequence Sampling. ACS Appl Mater. Interfaces. 2, 3249-3256 (2010).
- Pastre, D., et al. Anionic polyelectrolyte adsorption on mica mediated by multivalent cations: A solution to DNA imaging by atomic force microscopy under high ionic strengths. Langmuir. 22, 6651-6660 (2006).
- Woo, S., et al. Self-assembly of two-dimensional DNA origami lattices using cation-controlled surface diffusion. Nature Communications. 5, 4889(2014).
- Vesenka, J., et al. Substrate preparation for reliable imaging of DNA molecules with the scanning force microscope. Ultramicroscopy. 42-44, 1243-1249 (1992).
- Adsorption studies of DNA origami on silicon dioxide. Albrechts, B., et al. 21st Micromechanics and Micro Systems Europe Workshop 2010, , (2010).
- Sarveswaran, K., et al. Adhesion of DNA Nanostructures and DNA Origami to lithographically patterned self-assembled monolayers in Si[100]. Proc. of SPIE-Soc. Opt. Eng. 7637, 76370M-1(2010).
- Kern, W., Puotien, D. A. Cleaning solutions based on hydrogen peroxide for use in silicon semiconductor technology. RCA Rev. 31, 187-206 (1970).
- Pillers, M., Goss, V., Lieberman, M. Electron-Beam Lithography and Molecular Liftoff for Directed Attachment of DNA Nanostructures on Silicon: Top-down Meets Bottom-up. Acc. Chem. Res. 47, 1759-1767 (2014).
- Saccá, B., Niemery, C. M. DNA Origami: The Art of Folding DNA. Angew. Chem. Int. Ed. 51, 58-66 (2012).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Res. 37, 5001-5006 (2009).
- Ben-Ishay, E., et al. Designing a Bio-responsive Robot from DNA. Origami. J. Vis. Exp. (77), e50268(2013).
- Woo, S., et al. Programmable molecular recognition based on the geometry of DNA nanostructures. Nature Chemistry. 3, 620-627 (2011).
- Schlegel, M. L., et al. Cation sorption on the muscovite (001) surface in chloride solutions using high-resolution X-ray reflectivity. Geochim. Cosmochim. Acta. 70, 3549-3565 (2006).
- Rasband, W. S., Howarter, J. A., et al. National Institutes of Health. Langmuir. 22, Bethesda, Maryland, USA. 11142-11147 (2006).
- Kershner, R. J., et al. Placement and orientation of individual DNA shapes on lithographically patterned surfaces. Nature Nanotechnology. 4, 557-561 (2009).
- Hung, A. M., et al. Large-area spatially ordered arrays of gold nanoparticles directed by lithographically confined DNA origami. Nature Nanotechnology. 5, 121-126 (2010).
- Sarveswaran, K., et al. et al.Adhesion of DNA nanostructure and DNA origami to lithographically patterned self-assembled monolayers on Si[100. Proc. SPIE-Int. Soc. Opt. Eng. 7637, 76370M(2010).
- Pillers, M. A., Lieberman, M. Thermal stability of DNA origami on mica. J. Vac. Sci. Technol. B. 32, 040602(2014).
- Song, J., et al. Direct Visualization of Transient Thermal Response of a DNA. Origami. J. Am. Chem. Soc. 134, 9844(2012).
- Wei, X., et al. Mapping the thermal behavior of DNA origami nanostructures. J. Am. Chem. Soc. 135 (16), 6165-6176 (2013).
- Hyojeong Kim,, et al. Stability of DNA Origami Nanostructures under Diverse Chemical Environments. Chem. Mater. 26, 5265-5273 (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados