Method Article
Herstellung von Mica und Siliziumsubstraten für die DNA-Origami-Analyse und Experimentieren
In diesem Artikel
Zusammenfassung
Reproducible cleaning processes for substrates used in DNA origami research are described, including bench-top RCA cleaning and derivatization of silicon oxide. Protocols for surface preparation, DNA origami deposition, drying parameters, and simple experimental set-ups are illustrated.
Zusammenfassung
The designed nature and controlled, one-pot synthesis of DNA origami provides exciting opportunities in many fields, particularly nanoelectronics. Many of these applications require interaction with and adhesion of DNA nanostructures to a substrate. Due to its atomically flat and easily cleaned nature, mica has been the substrate of choice for DNA origami experiments. However, the practical applications of mica are relatively limited compared to those of semiconductor substrates. For this reason, a straightforward, stable, and repeatable process for DNA origami adhesion on derivatized silicon oxide is presented here. To promote the adhesion of DNA nanostructures to silicon oxide surface, a self-assembled monolayer of 3-aminopropyltriethoxysilane (APTES) is deposited from an aqueous solution that is compatible with many photoresists. The substrate must be cleaned of all organic and metal contaminants using Radio Corporation of America (RCA) cleaning processes and the native oxide layer must be etched to ensure a flat, functionalizable surface. Cleanrooms are equipped with facilities for silicon cleaning, however many components of DNA origami buffers and solutions are often not allowed in them due to contamination concerns. This manuscript describes the set-up and protocol for in-lab, small-scale silicon cleaning for researchers who do not have access to a cleanroom or would like to incorporate processes that could cause contamination of a cleanroom CMOS clean bench. Additionally, variables for regulating coverage are discussed and how to recognize and avoid common sample preparation problems is described.
Einleitung
Zuerst im Jahr 2006 eingeführt wurde, nutzt DNA-Origami die selbstorganisierende Natur von DNA-Oligonukleotiden auf gestaltbar und hochgeordnete Nanostrukturen zu erzeugen. 1 Eine Vielzahl von Strukturen berichtet, von Smiley-Gesichter auf 3-dimensionale-Boxen verriegelt. 2 DNA-Origami kann funktionalisiert werden mit verschiedenen Biomolekülen und Nanostrukturen, was zu Forschungsanwendungen in der Nanoelektronik, Medizin und Quantencomputing. 3, die Analyse und viele zukünftige Anwendungen sind jedoch nicht nur abhängig von Konstruktion, sondern auch auf die Haftung der DNA-Origami-Nanostrukturen auf Oberflächen. Die in diesem Manuskript beschriebenen Verfahren betreffen die Herstellung von DNA origami Proben auf zwei Arten von Substraten: Glimmer und funktionalisierten Siliziumoxid.
Mica ist das Substrat der Wahl für die DNA-Origami-Studien, da es atomar flach, mit einer Schichthöhe von 0,37 nm 0,02 nm ±. 4 Es ist auch easily gereinigt, so dass die Probenvorbereitung und Rasterkraftmikroskopie (AFM) Untersuchungen unkompliziert. Muskovit-Glimmer enthält, die eine hohe Dichte von Kalium in jedem Spaltebene, aber diese Ionen diffundieren weg von der Glimmer-Oberfläche, wenn sie in Wasser. Um die Bindung von DNA an die origami Glimmersubstrat vermitteln, Mg 2+ in der Gegenwart von großen verwendet, um die negative Ladung des Glimmer umkehren und elektrostatisch zu binden, die DNA-Phosphat-Rückgrat mit dem Substrat (1A). 5 Mischungen von DNA anneliert Exzesse der Klammerstränge geben hohe Reichweite und gute Bilder von Glimmer, weil die Haftung der DNA-Origami auf die Mg 2+ -terminierten Oberfläche ist viel stärker als die Haftung der einzelsträngige Oligonukleotide (Stapelstränge). Andere positiv geladene Ionen, einschließlich Ni 2+ und Co 2+ kann die Adhäsion von DNA auf Glimmer steuern. 6,7 Veränderung der Konzentration der ein- und zweiwertigen Kationen in Lösung kann adhe vermittelnsion und Oberflächendiffusionsraten von DNA-Origami. 8 jedoch das Protokoll zur Herstellung von Glimmersubstraten und Abscheiden und Spülen des Origami ist häufig nicht explizit in veröffentlichten Manuskripten. 9 Ohne ein klares Protokoll beschrieben, können reproduzierbare Ergebnisse nur schwer zu erhalten.
Glimmer ist ein Isolator, so dass es nicht als Substrat für einige Anwendungen in der Nanoelektronik geeignet. Silicium mit einer dünnen native Oxid passiwünschenswerte elektronische Eigenschaften, einschließlich der Kompatibilität mit früheren Komplementär-Metalloxid-Halbleiter (CMOS) Verarbeitung zur Eingabe / Ausgabe-Strukturen und topographische Merkmale zu schaffen. In Luft gelagert Siliziumscheiben sind entweder mit einem dicken thermischen Oxid oder dünne natürliche Oxidschicht, die relativ schmutzig ist, mit einer hohen Partikelzahl passiviert. Siliciumoxid hat einen viel niedrigeren Oberflächenladungsdichte als Glimmer und die Ladungsdichte ist stark abhängig von Oxid Herstellung und Geschichte. Bei Magnesiumionenkonzentrationen above 150 mM, gute Beschichtungsstärken (bis zu 4 / & mgr; m 2) mit rechteckigem Origami auf Sauerstoffplasma behandelte Siliziumsubstraten erreicht werden kann; aber diese Konzentration und Abdeckung kann in Abhängigkeit von der Größe und Gestaltung der Nanostrukturen verwendet ein alternatives Protokoll für die Abstimmung der Oberflächenladung zu ändern. 10 ist, um eine kationische selbstorganisierte Monoschicht von 3-Aminopropyltriethoxysilan (APTES) (1B) anhängen das Oxid. Das primäre Amin auf APTES kann bei pH-Werten unter 9 protoniert werden, Modifizieren der Ladung und Hydrophobizität des Substrats. 11 für eine vollständige Monoschicht von APTES erfolgreich aufgebracht werden, muss das in geeigneter Weise unter Verwendung von Silizium-Radio Corporation of America (RCA) Protokollen gereinigt werden . Diese Protokolle sind Behandlungen in Ammoniumhydroxid und Wasserstoffperoxid-Lösungen (RCA1) um organische Rückstände und Partikel-Verunreinigungen zu entfernen. Eine kurze Ätzung in wäßriger Fluorwasserstoffsäurelösung entfernt den nativen Oxidschicht zusammen mitbeliebige ionische Verunreinigungen, die in das Oxid zu haften. Schließlich werden die Proben auf eine Chlorwasserstoffsäure und Wasserstoffperoxid-Lösung (RCA2) ausgesetzt, um Metall und ionischen Verunreinigungen zu entfernen und eine dünne, gleichmäßige Oxidschicht. 12 Die meisten Reinräume haben Hauben für CMOS Reinigungsprotokolle mit strengen Regeln darüber, was verwendet werden, bezeichnet, in diesen Bereichen. Ein häufiges Problem ist in der Form von Ionen, wie Natrium, die die elektronischen Eigenschaften des CMOS-Strukturen durch die Schaffung midbandgap Fallen stören können. 13 Ionen üblicherweise in DNA origami Herstellung und Ablagerung Puffer verwendet könnten die CMOS Bäder Probleme für andere Forscher, die verschmutzen und zu der Reinraum. Aus diesem Grund nutzt unsere Gruppe eine "schmutzige" CMOS Reinigung Bank für kleine Proben für DNA-Origami-Forschung speziell angeordnet. Dieser Prozess ist eine gute Alternative zu den traditionellen Reinraum Einrichtung und kann für Labors, die keinen Zugang zu einem Reinraum CMOS Bank sein.
Protokoll
1. Experiment Planung und Materialvorbereitung
- Bestimmen das Design, Konzentration und Funktionalität der DNA-Origami, die in den Experimenten verwendet wird. 14-16 Hier verwenden wir eine DNA-Origami-Design-Rechteck in 1x TAE zubereitet / Mg 2+ Lösung (40 mM Tris-Base, 20 mM Essigsäure, 2 mM EDTA und 12 mM Magnesium-Acetat, pH 8,0). 17
- Autoklaven Alle Tipps, Röhren und Behältern verwendet werden. Diese Materialien müssen alle Autoklaven kompatibel sein.
- Bereiten Sie eine Versorgung mit sterilem Wasser zum Spülen. Füllen Sie eine sterile Glas mit etwa 500 ml von 18 MOhm x cm Wasser, Platz auf einer Heizplatte, kochen für 5 Minuten mit der Kappe ab, und nehmen Sie das Glas aus der Kochstelle nehmen und abkühlen lassen mit dem Deckel auf den Behälter gelegt, aber nicht angezogen . Im Kühlschrank aufbewahren und bereiten eine neue Lieferung jeden Monat oder, wenn notwendig.
2. Vorbereiten der Glimmer-Substrat
- Cut Substrate auf die entsprechende Größe (1 cm x 1 cm große Quadrate) mit einer Schere. Mica ist dünn und brüchig. Alternativ kaufen Glimmer in Scheibenform, die nicht schneiden erfordert.
- Spalten die Glimmer mit doppelseitigem Klebeband. Glimmer ist aus Schichten von Mineralien durch interkalierenden Ionen getrennt sind, kann jede Schicht abgeschält, wenn ein zweiseitiges Klebeband verklebt werden. 18
- Platzieren der Glimmer Quadrate auf dem doppelseitigen Klebeband noch in der Bandausgabevorrichtung ein, dass sie fest mit dem Band hält. Der Pinzette zwischen dem Glimmer und dem Band Schieben wird die oberste Schicht entfernt und bleiben auf dem Band. Unmittelbar nach der Entnahme wird das Glimmer quadratisch seiner sauberen Seite nach unten zeigt. Achten Sie auf die Glimmer umdrehen vor der Lagerung in einem Behälter.
- Wiederholen Sie diesen Vorgang drei bis vier Mal, um die vollständige Entfernung von der obersten Schicht und eine angemessene Reinigung zu gewährleisten.
- Alternativ halten Sie die Glimmer zu einem Behälter oder Tisch mit einem Stück doppelseitigem Klebeband und verwenden Sie ein zweites Stück zu stick zu und ziehen Sie die oberste Glimmerschicht. Der Glimmer wird in beiden Fällen richtig gereinigt werden, wobei die alternative Methode macht Abscheiden DNA und dem Spülen und Trocknen der Probe schwierig, aufgrund der zweiten Haftung auf dem Träger.
3. Einzahlen DNA Origami auf Mica
- Kurz gesagt, mischen Sie das Fläschchen mit DNA-Origami mit Hilfe eines Vortex-Mischers, um sicherzustellen, gleichmäßige Verteilung von Nanostrukturen in Lösung.
- Pipette 4 ul der Lösung auf den Glimmer, gewährleisten, dass die Pipettenspitze das Substrat nicht berühren. Verlassen Sie die DNA-Origami auf dem Glimmer ca. 10 min, um eine ausreichende Abdeckung zu gewährleisten. Die Abscheidungsdauer richtet sich nach der Konzentration der DNA origami verwendet sowie die gewünschte Abdeckung (2 und 3) zu variieren.
- Spülen Sie die DNA-Origami-Lösung aus der Glimmer-Substrat unter Verwendung von 100 & mgr; l sterilem Wasser über ein Waschbecken oder andere Flüssigkeitsbehälter. Nehmen Sie den Glimmer mit einer Pinzette. Pipettieren Sie das Wasser auf dieSubstrat, mit dem Fluss des Tropfens in Richtung der Spitze der Pinzette. Schütteln Sie den Glimmer mit einer scharfen Bewegung nach unten, um das überschüssige Wasser zu entfernen. Halten Sie die Pinzette aufrecht, so dass das Wasser in Richtung der Pinzette zu fließen, um eine Kontamination der Probe zu vermeiden.
- Trocknen, das Substrat mit einem stetigen Strom von Stickstoff (N 2) für 1 min. Stellen Sie sicher, dass überschüssiges Wasser entfernt wird. Wiederholen Sie die Spülung mit weiteren 100 & mgr; l sterilem Wasser. Trocknen des Substrats mit N 2 für eine weitere 3 min. Ein vollständig trocken Substrat für eine erfolgreiche Rasterkraftmikroskopie (AFM) Analyse (Abbildung 4) notwendig.
- Analysieren Sie den Substrat mit AFM oder lagern in einem geschlossenen Behälter.
4. CMOS / Silicon-Reinigungs-Set-up
- ACHTUNG: Bei Verwendung der CMOS-Setup, Persönliche Schutzausrüstung verwenden zu allen Zeiten. Die Reagenzien sind starke Säuren, starke Basen, Fluorwasserstoffsäure (HF) und starken Oxidationsmitteln, welche can mit Abfällen Lösungsmittel reagieren, wenn Reagenzien werden nicht ordnungsgemäß entsorgt werden. Beachten Sie die folgenden Vorsichtsmaßnahmen:
- Haus der CMOS Bank in einem Chemieabzug ohne andere Prozesse oder Aufbauten.
- Tragen Sie Handschuhe aus Nitril, Laborkittel, Schutzbrille, große Industriehandschuhe aus Nitril, ein Überlaufblech und ein Gesichtsschutz zu jeder Zeit, wenn Sie den CMOS Bank.
- Verwenden Sie Plastikwannen als Auffang wenn Lösungen hergestellt.
- Verwenden eines inerten Fluorpolymer Messbecher für die Handhabung des konzentrierten HF.
- Machen Calciumgluconat Salbe erhältlich als Erste-Hilfe für jeden Hautexposition.
- Erlauben nur qualifiziertes Personal, um den Vorgang durchzuführen.
- Immer sicherstellen, ein weiteres Mitglied der Labor im Notfall vorliegt.
- Halten Sie MSDS Informationen für alle Chemikalien in der Nähe der Haube.
- Machen Sie sich mit der Institution oder Chemieunfall und Expositionsrichtlinien des Unternehmens vertraut.
Hinweis: HF leicht durchdringt die Hautund ist ein Kalzium-Scavenger, beeinflussen Knochen und Nerven schädlich, wenn die Exposition auftritt. Dermale Exposition auf wenige Milliliter der konzentrierten Fluorwasserstoffsäure kann gefährlich und sogar tödlich sein. Etablieren notwendigen Vorkehrungen, um die Exposition tritt nicht gewährleisten.
- Führen RCA1 und RCA2 in einem separaten 250-ml-Bechergläser auf separaten Kochplatten. Jedes Becherglas sollte einen Rührstab enthält. Überwachen Sie die Temperatur der Lösung unter Verwendung von Thermometern eingespannt, so der Rührstab nicht in den Kolben schlagen. Decke das Becherglas mit einem Uhrglas, die Auswirkungen der Verdunstung zu verringern.
- RCA1 Vorbereitung
- Zeigen 50 ml 18 M & Omega; x cm Wasser in den bezeichneten RCA1 Becherglas mit einem Messbecher.
- 15 ml konzentriertes Ammoniumhydroxid (NH 4 OH) in das Becherglas. Spülen Sie den Messbecher mit 25 ml Wasser und fügen Sie das Spülwasser in die RCA1 Becher.
- Schalten die Hitze und Rührer auf der Heizplatte und bringen die RCA1-Bad auf 70 ° C.
- 15 ml 30% Wasserstoffperoxid (H 2 O 2) zu dem Becherglas RCA1. Verwenden Sie die RCA1 Lösung innerhalb 1 Stunde nach der H 2 O 2 wurde hinzugefügt. Das Bad kann mehrmals innerhalb der Zeitspanne von drei Tagen verwendet werden, wenn 15 ml Peroxid wird dem Bad jedes Mal zugesetzt.
- Mit Wasser spülen Messbecher gründlich und entsorgen Sie die Spülung in einem geeigneten RCA1 Abfallflasche.
- RCA2 Vorbereitung
- In 70 ml 18 M & Omega; x cm Wasser an die angegebene RCA2 Becher mit dem Messbecher gründlich gespült.
- 15 ml konzentrierter Salzsäure (HCl). Spülen Sie den Messbecher mit 20 ml Wasser und es an die RCA2 Becher hinzuzufügen.
- Erhöhen die Hitze und Rührgeschwindigkeit der Kochplatte, bis die Lösung 70 ° C erreicht.
- 15 ml 30% H 2 O 2. Wie die RCA1 Bad, benutzen Sie diese Lösung innerhalb 1 Stunde von, wenn die H 2 O 2 zugegeben wird; Zusätzlich kann die Badewannekann mehrmals innerhalb der Spanne von drei Tagen wieder verwendet werden, wenn 15 ml H 2 O 2 wird vor jeder Verwendung zugegeben.
- HF Herstellung der Lösung
- Man gibt 50 ml Wasser in einem inerten fluorierten Polymers Becher.
- Maßnahme 4 ml konzentrierter Fluorwasserstoffsäure (49%) in der Kunststoff-Messbecher und es an den inerten Fluorpolymer-Becherglas.
- Spülen Sie den Kunststoff-Messbecher mit insgesamt 50 ml Wasser, das Hinzufügen des Spülwassers in die HF-Becherglas. Waschen Sie den Messbecher gründlich mit Wasser und entsorgen Sie die Waschungen in einem bestimmten HF Abfallbehälter.
5. Vorbereitung und Reinigung des Siliziumsubstrats
- Schneiden Siliziumwafer in Chips
- Identifizieren die senkrecht und parallel Gitterrichtungen auf der ebenen polierten Oberfläche des Siliziumwafers. Diese Richtungen werden verwendet, um zu helfen, zu spalten Plätzen einfacher. Die folgenden Anweisungen gelten für cleaving Silizium <110> und ist möglicherweise nicht für andere Kristallorientierungen werden.
- Setzen Sie den Silizium-Wafer poliert Seite nach oben auf eine weiche Oberfläche, wie eine Serviette. Unter Verwendung der Diamant Schreiber-Feder, sanft nick die Unterseite des Wafers entlang der primären flachen Rand. Setzen Sie einen kleinen Draht, wie einer Büroklammer, unter dem nick und Druck sanft gelten für die Wafer, indem Fingern oder Pinzette auf beiden Seiten des nick und nach unten drücken. Dadurch wird der Wafer in zwei Hälften entlang der Kristallgitterlinie im natürlichen spalten Richtung zu trennen.
- Auf einem anderen Serviette, mit einem Bleistift und Lineal, messen Sie die gewünschte Breite der Quadrate durch Markierungspunkte sowohl auf der Ober- und Unterseite der Binde. Verbinden Sie diese Punkte mit geraden Linien. Dies wird als Richtlinie für sogar quadratische Formen zu dienen.
- Legen Sie eine der Waferkante-first-Hälften zwischen den Messleitungen auf der Serviette gegen die Leitung gespült, und wiederholen Sie die in Schritt 5.1.2 Die frisch senkrecht piec gebrochenes sollte nun die Breite der Quadrate geschnitten sein. Drehen Sie den Wafer horizontal auf der Serviette. Legen Sie sie zwischen den senkrechten Linien und wiederholen Sie den Vorgang in Schritt 5.1.2.
- Bewahren Sie die frisch gespaltenen Wafer-Chips in einem sauberen Fläschchen mit DI-Wasser um Kratzer zu vermeiden gefüllt. Die Silizium-Chips können unbegrenzt gelagert werden, sollte aber vor Beginn der Versuche zu reinigen.
- CMOS Reinigung von Silicon
- Wenn die RCA1 Lösung die angemessene Temperatur erreicht hat, tauchen acht bis zehn 1 cm x 1 cm Silizium-Chips in der Lösung unter Verwendung eines inerten fluorierten Polymers Korb mit einem "Durchmesser. 2 Sauerstoffblasen auf die Chips und Becherwänden zu bilden. Wenn kein Blasenbildung auftritt, wird die H 2 O 2 abgebaut wird. Lassen Sie die Späne in der Lösung für 10 bis 20 min, Rühren der Korb nach oben und unten alle paar Minuten, um die Chips nicht aneinander haften.
- Heben Sie den Korb mit den Silizium-Chips und gut abtropfen lassen. Bewegen Sie den Korb über to Abfallbecher und gründlich mit 18 MOhm x cm Wasser. Tauchen Sie ein in die Waschbecher und wackeln auf und ab 20 Sek. Leeren Sie den Korb und gründlich mit Wasser über die Abfallbecher. Leeren Sie den Auffangbecherglas in einem bestimmten RCA1 Abfallflasche und füllen Sie mit Wasser.
- Nach RCA1 Reinigung abgeschlossen ist, setzen Sie den Korb in den 1.50 HF-Becher für 10 bis 20 sec. Verwenden Sie einen sanften Auf- und Abbewegung, die Chips und HF mischen. Heben Sie den dunk eimer die HF vollständig abfliessen kann.
Hinweis: Die Chipoberflächen hydrophob sein sollten; Wasser wird nicht nass den Chip, sondern bilden Tröpfchen mit hoher Kontaktwinkel. Dies zeigt, daß das Siliciumoxid weggeätzt worden ist, und der Chip wird nun von Si-H-Bindungen beendet. - Setzen Sie den Waschbecher und spülen Becherglas in einem Kunststoffwanne, bewegen Sie den Korb über dem Becherglas spülen und spülen Sie mit 18 MOhm x cm Wasser. Tauchen Sie den Korb in den Waschbecher und rühren für 20 Sekunden.
- Füllen Sie einen zweiten Drain und rinse-Zyklus mit 18 MOhm x cm Wasser. Dump das Waschwasser in das Becherglas spülen und füllen Sie den Waschbecher mit Wasser. Gießen Sie alle Abfälle in einem bestimmten Kunststoff-HF Abfallflasche.
- Wenn die RCA2 Lösung die entsprechende Temperatur erreicht, tauchen die Silizium-Chips in den Lösungen mit dem Korb. Lassen in der Lösung für 10 bis 20 min. Der Chip wird nunmehr hydrophil sein, aufgrund des Wachstums einer dünnen (1-2 nm) Oxidschicht.
- Nach der entsprechenden Menge Zeit zu entfernen und nach den gleichen Verfahren wie Spülen RCA1. Entsorgen Sie den Abfall in der entsprechenden RCA2 Abfallbehälter. Nehmen Sie jedes Chip aus dem Korb mit Plastikpinzette, mit Wasser abspülen und trocken blasen mit Stickstoff.
- Shop-Chips in einem Kunststoff-Wafer-Box oder in einer Ampulle von 18 MOhm x cm Wasser. Das Silizium wird sauber bleiben, wenn sie in Wasser für etwa drei Tage nach der Reinigung gespeichert. Stellen Sie sicher, dass der Arbeitsbereich richtig aufgeräumt und die Außenseite der CMOS-Handschuhe wurden gewaschen. Lassen Sie das CMOS Handschuhe in der Haube, um zu trocknen.
6. Hinterlegung DNA Origami auf APTES funktionalisierten Silicon
- Selbstorganisierten Monoschicht Formation auf Silicon
- Warm APTES auf RT vor dem Öffnen. Wenn die Flasche zu kalt ist, kann es zu Kondensation kommen, wodurch die Hydrolyse der APTES während der Lagerung. In 1980 & mgr; l von 18 MOhm x cm Wasser und 20 ul APTES auf eine saubere Szintillationsfläschchen und Schwenken vermischen. Verwenden Sie diese Lösung sofort.
- Legen Sie eine gereinigte Silizium-Chip reflektierenden Seite nach oben in den Szintillationsfläschchen, Kappe, und lassen Sie sich für 20 min. Entfernen Sie den Chip mit einer Pinzette und spülen mit 200 ul Wasser und trocken für 1 Minute mit einem Strom von N 2.
- Abscheiden von DNA-Origami auf APTES funktionalisierten Silicon
Anmerkung: Die Schritte zum Abscheiden Origami funktionalisierter Silizium sind analog zu denen für die Abscheidung auf Glimmer.- Kurz mischen die DNA-Origami-Fläschchen und Pipette 4 ul Lösungauf das Siliziumsubstrat. Erhöhen Sie gegebenenfalls die Lautstärke der verwendet wird, um das gesamte Substrat abzudecken, wie das funktionalisierte Silizium ist hydrophober als die Glimmer-Substrat DNA-Origami-Lösung. Verwenden Sie ein Deckglas, um die Abscheidungslösung nach unten drücken und Verdunstung während der langen Ablagerungen zu verhindern.
- Ließ die Lösung stehen für die Menge an Zeit, die für den verwendeten Konzentrationen und der gewünschten Abdeckung (siehe Figuren 2 und 3 für die Wirkung von Zeit und Konzentration auf der Fläche bewachsen). Mit 100 ul sterile 18 MOhm x cm Wasser abspülen und trocken das Substrat mit N 2 für 1 min.
- Wiederholen Sie das Spülen mit weiteren 100 ul sterilem Wasser und trocknen Sie das Substrat mit N 2 für 3 min.
- Die Probe ist in einem sauberen Behälter, bis weitere Experimente oder Bildgebung durchgeführt werden. Proben beginnen Teilchenspeichermenge nach ca. 1-2 Wochen Lagerung zeigen, je nachdem, wie much sie behandelt werden.
7. AFM Imaging und Bildanalyse von DNA-Origami Samples
- Verwenden AFM im Tapping-Modus in Luft zur Bildgebung. Tapping-Modus sorgt dafür, dass eine minimale Kraft auf die fragile Nanostrukturen angewendet werden, im Vergleich zu Kontaktmodus.
Anmerkung: Die Bildaufnahmeparameter werden abhängig von dem Instrument. Alle Bilder vorgestellt wurden mit einem Nanoscope IIIa Multimode erfasst. - Auswahl AFM-Sonden zur berührungs / Zapfmodus in Luft, mit reflektierenden Goldbeschichtung, eine Resonanzfrequenz (nominal) von ~ 300 kHz, Kraftkonstante von 40 N / m und einem Spitzenradius <10 nm.
- Prozess und die AFM-Bilder mit Nanoscope-Analyse-Software zu analysieren. Führen Sie Deckung Berechnungen mit ImageJ. 19
Ergebnisse
Zwei Variablen bestimmen die Berichterstattung über DNA-Origami auf dem Substrat: Konzentration der Lösung und Belichtungszeit. Die Absorptionseigenschaften der DNA Origami auf Glimmer und APTES funktionalisiertes Siliciumoxid sind zuvor berichtet worden. 13 die Beziehung zwischen der Konzentration der DNA origami in der Abscheidungslösung und die endgültige Beschichtungsstärken auf Glimmer sind in Tabelle 1 und 2 zusammengefaßt, zeigen zunehmende Konzentration Ergebnisse in die größere Flächendeckung. Die Zeitabhängigkeit der Bindung ist in Fig. 3 Abdeckfläche gesehen wurde zuvor untersucht, um das Bindungsverhalten von DNA origami auf Glimmer und modifizierte Siliciumoxidoberflächen quantifizieren. DNA-Origami in 12 mM Magnesium in 1x TAE Puffer 83,3% ± 3,1% Deckung auf Glimmer nach 30 min Absorptionszeit auf der Oberfläche. Maximale Abdeckung auf Siliciumoxid mit APTES SAMs modifiziert ist nach 60 min, die kleiner als die maximale Abdeckung auf Glimmer beobachtet. EINlängere Abscheidungszeit wird benötigt, wenn ein hoher Flächendeckung auf APTES funktionalisierten Siliziumoxid erforderlich.
Es gibt mehrere Variablen, die schlechte Probenvorbereitung verursachen können. Der schwierigste unzureichend Spülen und Trocknen. Wenn die Pufferlösung nicht ausreichend gespült, bilden große Aggregate auf dem Substrat (4A). "DNA-Origami-Inseln beobachtet werden, wenn die Nanostrukturen, um Flecken von Magnesiumsalze auf der Oberfläche (4B) zu halten. Schließlich mit hoher Deck Proben ist es möglich, überschüssiges Pufferkomponenten Brückenbildung zwischen einzelnen DNA origami (4C), was es schwierig macht, Nanostrukturen mit AFM unterscheiden macht. Diese Ergebnisse können durch Anschluss an eine gründliche Spülung und Trocknung Protokoll für beide Glimmer und Siliziumsubstraten vermieden werden.
Bildung APTES Filme auf Siliziumoxid Substrate können Probleme sowie posieren. Silizium-Wafer haben einenGrob Siliziumoxidschicht, die entfernt werden müssen und eine glattere, Verdünner Siliziumoxidschicht reformiert, bevor es funktionalisiert werden können. Ein richtig gereinigten Siliziumsubstrat ist in 5A dargestellt. Während der Reinigung der Siliciumscheibe ist es wichtig, sicherzustellen, dass die Anzahl der Si-Chips ist nicht zu hoch, da es möglich ist, zwei Chips stecken bleibt zueinander Sperr Exposition gegenüber den Reagenzien (5B). Wenn gereinigte Silizium in 18 M & Omega; x cm Wasser für mehr als eine Woche gelagert wurde, wird der Schmutzstoffschicht zu reformieren und Nachreinigung erforderlich ist. Die APTES Versorgung kann auch Probleme mit der Probenvorbereitung führen. APTES polymerisiert leicht durch Hydrolyse, die die Grundlage für die Bildung einer Monoschicht ist. 20. Das Ausmaß der Polymerisation von der Konzentration an Wasser, das APTES ausgesetzt ist, ist. Im Laufe der Zeit und mit wiederholtem Gebrauch ist es möglich, dass Wasser in das Innere der Flasche APTES kondensieren und verunreinigenVersorgung. Die resultierende Polymerisation produziert große Aggregate, die an dem Substrat (5C) zu haften. Die erhöhte Rauheit und Anwesenheit von Aggregaten macht Identifizierung von DNA-Nanostrukturen mit AFM schwierig. Es hat sich bewährt, um die APTES Flasche in einem Plastikbeutel im Kühlschrank zu lagern, und lassen Sie die Flasche APTES auf RT erwärmen vor dem Öffnen, um Kondensation zu vermeiden.
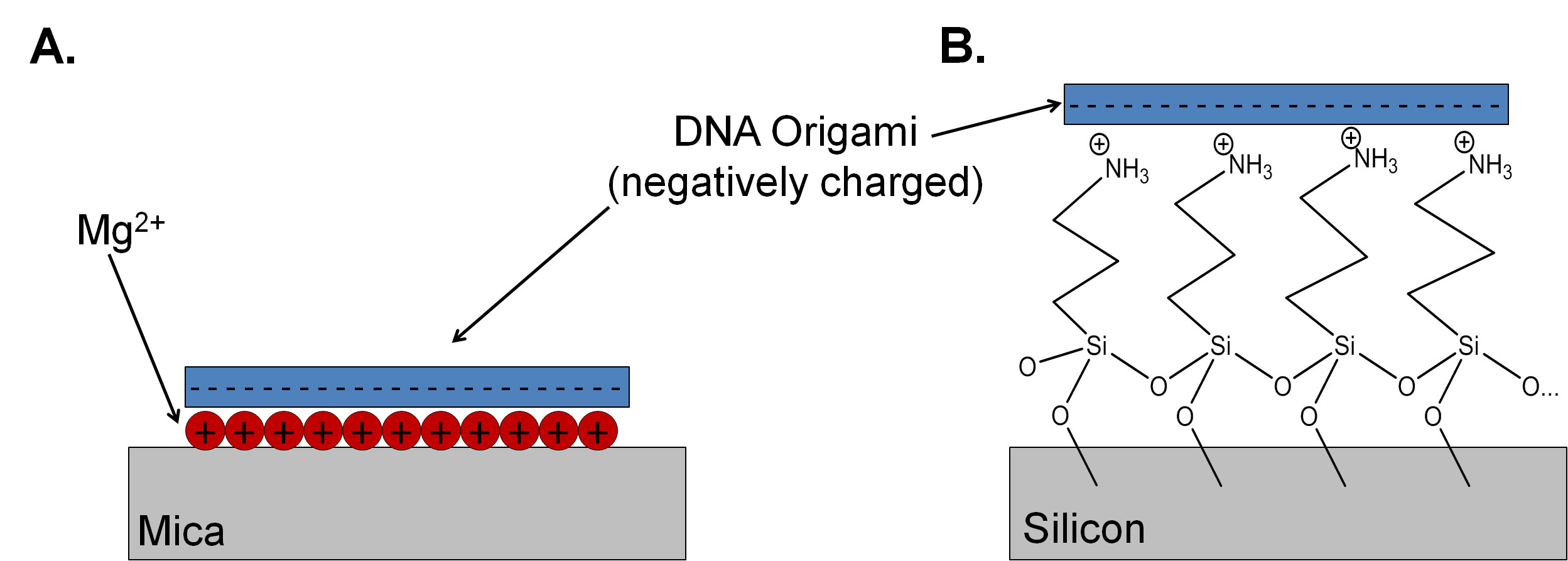
Abbildung 1 ist eine schematische Vergleichen des Bindungsmechanismus für die DNA Origami auf (A) Glimmer und (B) APTES funktionalisierten Siliziumoxid (nicht maßstabsgetreu). Die Bindung mit Glimmer wird durch die Anwesenheit von zweiwertigen Kationen, gewöhnlich Mg 2+, vermittelt. Eine Monoschicht des protonierten Amin-Endgruppen 3-Aminopropyltriethoxysilan verwendet wird, um die Haftung auf dem Silizium-Substrat zu fördern.

Abbildung 2. AFM-Aufnahmen nach 10 min Ablagerungen von (A) 2 nM, (B) 4 nM, und (C) 6 nM auf Glimmer. Die Höhenskala für alle Bilder ist 5 nm veranschau variable Abdeckung.

Abbildung 3. Trends in Oberflächenabdeckung für 2 nM DNA-Origami in 1x TAE-Puffer, 12 mM Mg 2+. MICA = lila Linie und Kreis Marker, APTES = gelbe Linien und Dreieck-Marker. N = 3 bei der Bestimmung der Standardfehler.

Abbildung 4. Schlechte Spülen und Trocknen kann (A) Lösung Aggregation auf dem Substrat, (B) DNA-Origami-Inseln, und (C) Überbrückung von Nanostrukturen auf verursachenhohe Abdeckung Proben in der Gegenwart von überschüssigen Puffersalze. Die Höhenskala für alle Bilder ist 5 nm.

Abbildung 5 (A) reinigen Silizium sollte RMS-Rauhigkeit von weniger als 0,5 Å haben mehr als 1 Quadratmikrometer. Eine vollständige APTES SAM abgeschieden auf einem guten native Oxid sollte eine RMS-Rauhigkeit von weniger als 1 Å haben mehr als 1 Quadratmikrometer. (B) APTES Film auf einem unvollständig gereinigt Siliziumoxid mit RMS-Rauhigkeit von 2,29 nm mehr als 1 Quadratmikrometer gebildet. . Beachten Sie die Lücken und Rauheit der APTES Film (C) APTES Oberfläche aus einer Stichprobe von APTES mit Kondensmilch Abfälle zu vermeiden gebildet; Hydrolyse in den APTES Flaschenformen große Partikel. Die Höhenskala für alle Bilder ist 5 nm.
Tabelle 1 Percent Versorgungsmessungen für Glimmersubstraten mit varying DNA-Origami-Lösungskonzentrationen. Alle Zeiten sind Abscheidung 10 min.
| DNA-Origami-Lösung | % Coverage auf Glimmer-Substrat |
| 2 nM | 8,49 ± 2,67 (n = 5) |
| 4 nM | 55,89 ± 5,65 (n = 3) |
| 6 nM | 77,44 ± 1,89 (n = 4) |
Diskussion
Es gibt mehrere Schritte, die betont, um konsistente und optimale Ergebnisse zu erzielen werden müssen. Für Glimmer Proben, die nach einem strengen und gründlichen Spülen und Trocknen Regime, wie in den Schritten 3.3 und 3.4, wird sicherstellen, dass qualitativ hochwertige Bilder von einzelnen DNA-Origami kann mit AFM, ohne die verschiedenen Probleme im Repräsentative Ergebnisse Abschnitt beschrieben erreicht werden. Von primärer Bedeutung für Siliziumproben ist die Sauberkeit des Substrats. Im Anschluss an die in Schritt 5.2 gründlich und sorgfältig skizzierten Reinigungsverfahren stellt sicher, dass ein entsprechend gereinigt Siliziumoxid Oberfläche erreicht werden. Zusätzlich die Überwachung der Qualität und Wirksamkeit von Chemikalien, wie Wasserstoffperoxid, Fluorwasserstoffsäure und APTES, wird sichergestellt, dass das Verfahren läuft.
Die beschriebenen Techniken sind nicht nur auf wässrige Lösungen von APTES beschränkt. Eine gemischte Monoschicht von APTES und trimethylaminopropyltrimethoxysilyl Chlorid (TMAC) kann unsed zum Abstimmen der Siliziumoberflächenladung und zu fördern variable DNA-Origami Haftung. 11 Die TMAC enthält eine permanent geladen Terminal quaternäres Amin-N (CH 3) 3 +, verglichen mit dem pH-abhängige Ladung auf APTES. Da die Lösung Umfeld beeinflusst nicht die Protonierungszustand oder die Ladung des TMAC, Variation der Lösungskonzentration von TMAC einzustellen, können die Oberflächenladung der gemischten Monoschicht und Einfluss auf die Wechselwirkung zwischen dem Substrat und der DNA-Origami. Optimale DNA origami Bindung wurde für Monoschichten mit einer Oberflächenladung von 0,75-1,5 Ladungen / nm 2, die SAMs, die 100% bis 40% TMAC Konzentration entspricht beobachtet. Diese optimale Oberflächenladung aufgefordert DNA-Origami-Bedeckungen von ca. 110 Origami / & mgr; m 2 auf APTES SAMs und 120 Origami / & mgr; m 2 auf TMAC SAMs.
Ein Vorteil von Silizium ist die Kompatibilität mit lithographischen Strukturierungsprozesse. Hohem MagnesiumKonzentrationen können mit plasmabehandelten Siliciumoxiden verwendet werden, um zu Silizium fördern selektive Bindung von DNA-Origami werden. Sorgfalt muss bei der Entnahme der Probe aus der Abscheidungslösung zu vermeiden Spülen der Magnesium-Ionen entfernt und Verformen der DNA origami genommen werden. Die 21,22 APTES Verlaufsformen kovalent gebundenen Kationen auf der Siliziumoxidoberfläche, so Waschen oder Spülen mit Puffer oder Wasser tut das beigefügte DNA-Origami nicht beschädigen. Die 'molekularen liftoff' Methode ist ein weiterer möglicher Weg zur Strukturierung Siliziumsubstraten und Förderung DNA-Origami Haftung. Das Siliziumsubstrat mittels Elektronenstrahllithographie strukturiert und APTES auf der freigelegten Substrat abgeschieden. Folgende Abheben des Photoresists können DNA Origami auf der strukturierten Oberfläche abgeschieden werden, vorzugsweise die Bindung an die APTES. 23
Das Zusammenspiel von DNA-Origami mit einem Substrat ändert seine Stabilität, die Eröffnung neuer Wege für die Forschung und Anwendung. DNA origami klebt Glimmer auf 150 ° C erhitzt werden, ohne sichtbare Veränderung der Nanoabmessungen und minimalem chemischen Veränderungen. 24. Dies steht in krassem Gegensatz zu der Brüchigkeit Origami in Lösung, wobei die Nanostrukturen vollständig oberhalb von 70 ° C. 25 dehybridisiert, 26 Diese Stabilität wird auf beheizten Siliziumoxid Substraten gehalten. 27. Auch in verschiedenen Lösungsmittelsystemen, wie Hexan, Toluol und Ethanol, die Form und die Reichweite der Nanostrukturen erhalten bleiben. Die überraschende Stabilität der DNA origami anzeigt, dass Anwendungen, die bisher unvereinbar galten, wie etwa plasmaunterstützte Dampfabscheidung Verwendung gemeinsamer Fotolacke und Lösungsmittel und einzigartige chemische Abscheidung Umgebungen kann in Verbindung mit DNA origami verwendet werden. , Ob die DNA-Origami behalten ihre Funktionalität ist jedoch noch nicht bekannt und können mögliche Anwendungen zu begrenzen.
Obwohl die Stabilität der DNA-Origami bei erhöhter Temperaturs und in eine begrenzte Anzahl von Lösungsmittelsystemen ermittelt wurde, ist die Langzeitstabilität der DNA origami auf Substraten noch unbekannt. Die Verwendung von sterilen Techniken notwendig ist, um eine mögliche Kontamination zu vermeiden, aber dies kann nicht nach der Oberflächenabscheidung vermieden werden. Imaging und Analyse von Proben muss fast unmittelbar nach der Probenvorbereitung durchgeführt werden; Wenn die Probe zu lange (mehr als eine Woche) verschiedene Beispiele von Probenabbau werden häufig festgestellt, die Ansammlung von Partikeln und Bruch DNA-Nanostrukturen gespeichert. Mögliche Forschungswege in der Nanoelektronik, Biosensorik und andere Substrat auf Basis DNA-Origami-Anwendungen können durch zeitabhängige Destabilisierung der DNA begrenzt. Identifizieren der Grenzen dieser Methoden für Anwendungen Stabilität erfordern längere Zeiträume erfordert weitere Untersuchungen.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
The authors thank Dr. Gary Bernstein for use of the AFM.
Materialien
| Name | Company | Catalog Number | Comments |
| Eppendorf epT.I.P.S. Reloads, capacity 2-200 μl | VWR International, LLC | 22491733 | 10 reload tray of 96 tips |
| Microcentrifuge Tubes, Polypropylene | VWR International, LLC | 87003-290 | 0.65 ml, natural |
| Research Plus Pippete - Single Channel - 20-200 μl | A. Daigger & Company, Inc. | EF8960F-3120000054 EACH | Adjustable Volume |
| Research Plus Pippete - Single Channel - 2-20 μl | A. Daigger & Company, Inc. | EF8960D-3120000038 EACH | Adjustable Volume |
| Scotch 237 Permanent Double-Sided Tape | Office Depot, Inc. | 602710 | 3/4" x 300", Pack of 2 |
| Vortex Mixer | Thermo Scientific | M37610-33Q | |
| Wafer container single, 2" (50 mm), 60 mm x 11 mm | Electron Microscopy Sciences | 64917-2 | 6 per pack |
| 6" Wafer, P-type, <100> orientation, w/ primary flat | Nova Electronic Materials, Ltd. | GC49266 | |
| Powder-Free Nitrile Examination Gloves | VWR International, LLC | 82062-428 | Catalog number is for size large |
| High Accuracy Noncontact probes with Au reflective coating | K-Tek Nanotechnology, Inc. | HA_NC/15 | |
| Autoclave Pan | A. Daigger & Company, Inc. | NAL692-5000 EF25341C | |
| Sol-Vex II Aggressive Gloves, Size: 9-9.5; 15 mil, 13 inch - 1 dz | Spectrum Chemical Mfg. Corp. | 106-15055 | Before use, rinse with water and scrub together until no bubbles form on the gloves. |
| Tweezers PTFE 200 mm Square | Dynalon Corp. | 316504-0002 | |
| Muscovite Mica Sheets V-5 Quality | Electron Microscopy Sciences | 71850-01 | 10 per pack |
| Mica Disc, 10 mm | Ted Pella, Inc | 50 | Mica discs are optional |
| Scriber Diamon Pen for Glassware | VWR International, LLC | 52865-005 | |
| Scintillation Vials, Borosilicate Glass, with Screw Cap - 20 ml | VWR International, LLC | 66022-060 | Case of 500, with attached polypropylene cap and pulp foil liner |
| 4 x 5 Inch Top PC-200 Hot Plate, 120 V/60 Hz | Dot Scientific, Inc. | 6759-200 | |
| Straight-Sided Glass Jars, Wide Mouth | VWR International, LLC | 89043-554 | Case of 254, caps with pulp/vinyl liner attached |
| Standar-Grade Glass Beaker, 250 ml Capacity | VWR International, LLC | 173506 | |
| Beakers, PTFE | VWR International, LLC | 89026-022 | For use with HF |
| Shallow form watch glass, 3" | VWR International, LLC | 66112-107 | Case of 12 |
| Plastic Storage Container | VWR International, LLC | 470195-354 | For secondary container |
| General-Purpose Liquid-In-Glass Thermometers | VWR International, LLC | 89095-564 | |
| High precision and ultra fine tweezers | Electron Microscopy Sciences | 78310-0 | |
| Polycarbonate Faceshield | Fisher Scientific, Inc. | 18-999-4542 | |
| Neoprene Apron | Fisher Scientific, Inc. | 19-810-609 | |
| Calcium Gluconate, Calgonate | W.W Grainger, Inc. | 13W861 | Tube, 25 g |
| Hydrogen Peroxide 30% CR ACS 500 ml | Fisher Scientific, Inc. | H325 500 | HARMFUL, TOXIC |
| 3-Aminopropyltriethoxysilane | Gelest Inc. | SIA0610.0-25GM | Let warm to room temperature before use. |
| Ammonium hydroxide, 2.5 L | Fisher Scientific, Inc. | A669-212 | HARMFUL, TOXIC |
| Hydrochloric acid | Fisher Scientific, Inc. | A144-212 | HARMFUL, TOXIC |
| Hydrofluoric acid | Fisher Scientific, Inc. | A147-1LB | HARMFUL, TOXIC |
| MultiMode Nanoscope IIIa | Veeco Instruments, Inc. | Any AFM capable of tapping mode is suitable for analysis | |
| Dunk basket | Made in lab | Made in lab | The dunk basket was made using the bottom of a PTFE bottle with holes drilled in, PTFE handle, and all PTFE screws. |
Referenzen
- Rothemund, P. W. K. Folding DNA to create nanoscale shapes and patterns. Nature. 440, 297-302 (2006).
- Anderson, E. S., et al. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 459, 73-77 (2009).
- Wang, Z., Ding, B. Engineering DNA Self-Assemblies as Templates for Functional Nanostructures. Acc. Chem. Res. 47, 1654-1662 (2014).
- Xu, K., et al. Graphene Visualizes the First Water Adlayers on Mica at Ambient Conditions. Science. 329, 1188-1191 (2010).
- Bustamante, C., et al. Circular DNA-molecules imaged in air by scanning force microscopy. Biochemistry. 31, 22-26 (1992).
- Hsueh, C., et al. Localized Nanoscopic Surface Measurements of Nickel-Modified Mica for Single-Molecule DNA Sequence Sampling. ACS Appl Mater. Interfaces. 2, 3249-3256 (2010).
- Pastre, D., et al. Anionic polyelectrolyte adsorption on mica mediated by multivalent cations: A solution to DNA imaging by atomic force microscopy under high ionic strengths. Langmuir. 22, 6651-6660 (2006).
- Woo, S., et al. Self-assembly of two-dimensional DNA origami lattices using cation-controlled surface diffusion. Nature Communications. 5, 4889(2014).
- Vesenka, J., et al. Substrate preparation for reliable imaging of DNA molecules with the scanning force microscope. Ultramicroscopy. 42-44, 1243-1249 (1992).
- Adsorption studies of DNA origami on silicon dioxide. Albrechts, B., et al. 21st Micromechanics and Micro Systems Europe Workshop 2010, , (2010).
- Sarveswaran, K., et al. Adhesion of DNA Nanostructures and DNA Origami to lithographically patterned self-assembled monolayers in Si[100]. Proc. of SPIE-Soc. Opt. Eng. 7637, 76370M-1(2010).
- Kern, W., Puotien, D. A. Cleaning solutions based on hydrogen peroxide for use in silicon semiconductor technology. RCA Rev. 31, 187-206 (1970).
- Pillers, M., Goss, V., Lieberman, M. Electron-Beam Lithography and Molecular Liftoff for Directed Attachment of DNA Nanostructures on Silicon: Top-down Meets Bottom-up. Acc. Chem. Res. 47, 1759-1767 (2014).
- Saccá, B., Niemery, C. M. DNA Origami: The Art of Folding DNA. Angew. Chem. Int. Ed. 51, 58-66 (2012).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Res. 37, 5001-5006 (2009).
- Ben-Ishay, E., et al. Designing a Bio-responsive Robot from DNA. Origami. J. Vis. Exp. (77), e50268(2013).
- Woo, S., et al. Programmable molecular recognition based on the geometry of DNA nanostructures. Nature Chemistry. 3, 620-627 (2011).
- Schlegel, M. L., et al. Cation sorption on the muscovite (001) surface in chloride solutions using high-resolution X-ray reflectivity. Geochim. Cosmochim. Acta. 70, 3549-3565 (2006).
- Rasband, W. S., Howarter, J. A., et al. National Institutes of Health. Langmuir. 22, Bethesda, Maryland, USA. 11142-11147 (2006).
- Kershner, R. J., et al. Placement and orientation of individual DNA shapes on lithographically patterned surfaces. Nature Nanotechnology. 4, 557-561 (2009).
- Hung, A. M., et al. Large-area spatially ordered arrays of gold nanoparticles directed by lithographically confined DNA origami. Nature Nanotechnology. 5, 121-126 (2010).
- Sarveswaran, K., et al. et al.Adhesion of DNA nanostructure and DNA origami to lithographically patterned self-assembled monolayers on Si[100. Proc. SPIE-Int. Soc. Opt. Eng. 7637, 76370M(2010).
- Pillers, M. A., Lieberman, M. Thermal stability of DNA origami on mica. J. Vac. Sci. Technol. B. 32, 040602(2014).
- Song, J., et al. Direct Visualization of Transient Thermal Response of a DNA. Origami. J. Am. Chem. Soc. 134, 9844(2012).
- Wei, X., et al. Mapping the thermal behavior of DNA origami nanostructures. J. Am. Chem. Soc. 135 (16), 6165-6176 (2013).
- Hyojeong Kim,, et al. Stability of DNA Origami Nanostructures under Diverse Chemical Environments. Chem. Mater. 26, 5265-5273 (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten