Method Article
Préparation de Mica et Silicon substrats pour l'ADN Origami Analyse et Expérimentation
Dans cet article
Résumé
Reproducible cleaning processes for substrates used in DNA origami research are described, including bench-top RCA cleaning and derivatization of silicon oxide. Protocols for surface preparation, DNA origami deposition, drying parameters, and simple experimental set-ups are illustrated.
Résumé
The designed nature and controlled, one-pot synthesis of DNA origami provides exciting opportunities in many fields, particularly nanoelectronics. Many of these applications require interaction with and adhesion of DNA nanostructures to a substrate. Due to its atomically flat and easily cleaned nature, mica has been the substrate of choice for DNA origami experiments. However, the practical applications of mica are relatively limited compared to those of semiconductor substrates. For this reason, a straightforward, stable, and repeatable process for DNA origami adhesion on derivatized silicon oxide is presented here. To promote the adhesion of DNA nanostructures to silicon oxide surface, a self-assembled monolayer of 3-aminopropyltriethoxysilane (APTES) is deposited from an aqueous solution that is compatible with many photoresists. The substrate must be cleaned of all organic and metal contaminants using Radio Corporation of America (RCA) cleaning processes and the native oxide layer must be etched to ensure a flat, functionalizable surface. Cleanrooms are equipped with facilities for silicon cleaning, however many components of DNA origami buffers and solutions are often not allowed in them due to contamination concerns. This manuscript describes the set-up and protocol for in-lab, small-scale silicon cleaning for researchers who do not have access to a cleanroom or would like to incorporate processes that could cause contamination of a cleanroom CMOS clean bench. Additionally, variables for regulating coverage are discussed and how to recognize and avoid common sample preparation problems is described.
Introduction
D'abord présenté en 2006, l'ADN origami utilise la nature d'auto-assemblage d'oligonucléotides d'ADN pour produire des nanostructures concevables et très ordonnées. 1 Une myriade de structures ont été signalés, allant de smiley face à verrouillée boîtes 3 dimensions. 2 ADN origami peut être fonctionnalisés avec diverses biomolécules et nanostructures, donnant lieu à des applications de recherche en nanoélectronique, la médecine et l'informatique quantique. 3 Cependant, l'analyse et de nombreuses applications futures ne sont pas seulement tributaire de la conception structurelle, mais aussi sur l'adhésion des nanostructures d'ADN origami à des surfaces. Les méthodes décrites dans ce manuscrit ont trait à la préparation des échantillons d'origami d'ADN sur deux types de supports: mica et d'oxyde de silicium fonctionnalisés.
Mica est le substrat de choix pour les études de l'ADN origami parce qu'il est atomiquement plat, avec une hauteur de couche de 0,37 nm ± 0,02 nm. 4 Il est également EASily nettoyé, ce qui rend la préparation des échantillons et de la microscopie à force atomique (AFM) des études simple. Mica moscovite contient une densité élevée de potassium dans chaque plan de clivage, mais ces ions diffuse loin de la surface du mica lorsque dans l'eau. Pour la médiation de la liaison de l'ADN origami pour le substrat de mica, Mg 2+ est utilisée pour inverser la charge négative du mica et de lier électrostatique du phosphate d'ADN épine dorsale du substrat (figure 1A). 5 mélanges d'ADN recuit en présence d'un grand excès de brins de base donnent une couverture élevée et de bonnes images de mica, car l'adhérence de l'ADN origami à la surface Mg 2+ à terminaison est beaucoup plus forte que l'adhérence d'oligonucléotides simple brin (brins de base). D'autres ions chargés positivement, y compris Ni 2+ et Co 2+ peuvent être utilisés pour contrôler l'adhérence de l'ADN sur du mica. 6,7 Modification de la concentration de cations monovalents et divalents dans la solution peut servir de médiateur adhésion et de diffusion de surface taux d'ADN origami. 8 Cependant, le protocole de préparation des substrats de mica et de dépôt et rinçage de la origami est souvent pas explicitement décrits dans les manuscrits publiés. 9 Sans un protocole clair, des résultats reproductibles peuvent être difficiles à obtenir.
Mica est un isolant, de sorte qu'il ne convient pas en tant que substrat pour certaines applications en nanoélectronique. Silicon passive avec un oxyde natif mince possède des propriétés électroniques souhaitables, y compris la compatibilité avec le traitement préalable gratuit semi-conducteurs à oxyde métallique (CMOS) pour créer / structures d'entrée-sortie et les caractéristiques topographiques. Les plaquettes de silicium stockées dans l'air sont passivées avec soit un oxyde thermique d'épaisseur ou d'un film d'oxyde natif mince qui est relativement sale, avec un nombre élevé de particules. L'oxyde de silicium a une densité de charge de surface beaucoup plus faible que le mica, et la densité de charge est très dépendante de la préparation de l'oxyde et de l'histoire. À des concentrations en ions magnésium above 150 mM, de bonnes couvertures (jusqu'à 4 / um 2) de l'origami d'ADN rectangulaire peut être réalisé sur l'oxygène plasma traité substrats de silicium; Cependant, cette concentration et la couverture peuvent varier selon la taille et la conception des nanostructures utilisé. 10 Un protocole alternatif pour accorder la charge de surface est d'attacher une monocouche auto-assemblée cationique de 3-aminopropyltriéthoxysilane (APTES) (figure 1B) à l'oxyde. L'aminé primaire sur APTES peut être protoné à un pH inférieur à 9, en modifiant la charge et le caractère hydrophobe du substrat. 11 Pour une monocouche complète de APTES être déposé avec succès, le silicium doit être nettoyé de manière appropriée en utilisant Radio Corporation of America (RCA) protocoles . Ces protocoles comprennent des traitements de l'hydroxyde d'ammonium et des solutions de peroxyde d'hydrogène (RCA1) pour éliminer les résidus organiques et les contaminants particulaires. Un court gravure dans une solution aqueuse d'acide fluorhydrique enlève la couche d'oxyde natif avectous les contaminants ioniques qui adhèrent à l'oxyde. Enfin, les échantillons sont exposés à de l'acide chlorhydrique et de solution de peroxyde d'hydrogène (RCA2) pour éliminer les métaux et les contaminants ioniques et former une couche mince et uniforme d'oxyde. 12 La plupart des salles blanches ont désigné les hottes de protocoles de nettoyage CMOS, avec des règles strictes sur ce qui peut être utilisé dans ces domaines. Un problème commun est livré sous la forme d'ions tels que le sodium, qui peuvent perturber les propriétés électroniques des structures CMOS en créant des pièges midbandgap. 13 Ions couramment utilisé dans l'ADN origami préparation et de dépôt des tampons pourrait contaminer les bains CMOS et causer des problèmes pour d'autres chercheurs utilisant la chambre propre. Pour cette raison, notre groupe utilise un CMOS «sales» banc de nettoyage disposés spécifiquement pour les petits échantillons utilisés pour la recherche de l'ADN origami. Ce processus est une bonne alternative à la salle blanche traditionnelle set-up et peut convenir pour les laboratoires qui ne disposent pas de l'accès à un banc CMOS de salle blanche.
Protocole
1. Expérience Planification et préparation Matériel
- Déterminer la conception, de la concentration, et la fonctionnalité de l'origami d'ADN qui sera utilisé dans les expériences. 14-16 Ici, nous utilisons un design ADN origami rectangle préparés 1x TAE / Mg 2+ solution (40 mM Tris-base 20 mM l'acide acétique, 2 mM d'EDTA et 12 mM d'acétate de magnésium, pH 8,0). 17
- Autoclave tous les bons plans, des tubes et des récipients pour être utilisé. Ces matériaux doivent tous être compatibles autoclave.
- Préparer un approvisionnement d'eau stérile pour le rinçage. Remplir un bocal stérile avec environ 500 ml de 18 MQ x cm d'eau, place sur une plaque de cuisson, faire bouillir pendant 5 min avec le bouchon, et de prendre le pot hors de la plaque chauffante et laisser refroidir avec le couvercle placé sur le conteneur, mais pas serré . Conserver au réfrigérateur et de préparer une nouvelle offre chaque mois ou lorsque nécessaire.
2. Préparation de la Mica Substrat
- substrats de couper à la taille appropriée (1 cm x 1 cm carrés) avec des ciseaux. Mica est mince et fragile. Alternativement, acheter mica sous forme de disque qui ne nécessite pas de coupe.
- Cliver le mica en utilisant du ruban adhésif double-face. Mica est composé de couches de minéraux séparés en intercalant ions, chaque couche peut être décollée quand adhéré à ruban adhésif double face. 18
- Placez les carrés de mica sur le ruban adhésif double face encore dans le dévidoir de ruban, en vous assurant qu'il adhère fermement à la bande. Faites glisser délicatement les pinces entre le mica et la bande, le haut-couche la plus sera supprimé et restent sur la bande. Immédiatement après l'enlèvement, la place de mica aura son côté propre vers le bas. Assurez-vous de retourner le mica plus avant de la ranger dans un conteneur.
- Répétez cette opération trois ou quatre fois pour assurer l'élimination complète de la couche la plus supérieure et un nettoyage adéquats.
- Alternativement, adhérer le mica à un conteneur ou une table-top avec un morceau de ruban adhésif double face et utiliser une deuxième pièce à scochez la case pour et de décoller la couche supérieure plus de mica. Le mica est correctement nettoyé dans les deux cas, bien que la méthode alternative et permet le dépôt d'ADN rinçage et séchage de l'échantillon difficile en raison de la deuxième adhérence sur le support.
3. Dépôt ADN Origami sur Mica
- Brièvement, mélangez le flacon de l'ADN origami utilisant un mélangeur vortex pour assurer une dispersion des nanostructures en solution.
- Pipette 4 pi de solution sur le mica, assurant que la pointe de la pipette ne touche pas le substrat. Laissez l'origami d'ADN sur le mica pour environ 10 min pour assurer une couverture adéquate. Le temps de dépôt varie en fonction de la concentration de l'ADN origami utilisé ainsi que la couverture souhaitée (figures 2 et 3).
- Rincer la solution ADN origami hors du substrat de mica en utilisant 100 pi d'eau stérile dessus d'un évier ou autre récipient de liquide. Ramassez le mica avec des pincettes. Pipeter l'eau sur lasubstrat, avec le flux de la chute vers la pointe de la pince à épiler. Agiter le mica avec une forte baisse de mouvement pour éliminer l'excès d'eau. Tenir la pince à épiler debout afin que l'eau coulera vers les pincettes pour éviter la contamination de l'échantillon.
- Sécher le substrat avec un flux régulier d'azote (N 2) pendant 1 min. Assurez-vous que toute l'eau en excès est éliminé. Répétez le rinçage avec un 100 pi supplémentaires de l'eau stérile. Sécher le substrat avec N2 pendant 3 minutes supplémentaires. Un substrat complètement sec est nécessaire pour la réussite de la microscopie à force atomique (AFM) analyse (figure 4).
- Analyser le substrat en utilisant l'AFM ou un magasin dans un récipient fermé.
4. CMOS / Silicon nettoyage Set-up
- ATTENTION: Lorsque vous utilisez les CMOS SET-UP, utiliser l'équipement de protection personnelle à tout moment. Les réactifs comprennent les acides forts, bases fortes, l'acide fluorhydrique (HF), et des agents oxydants puissants qui can réagir avec des solvants de déchets si les réactifs ne sont pas éliminés correctement. Respecter les consignes de sécurité suivantes:
- Maison CMOS banc dans une hotte chimique avec aucun autre processus ou set-ups.
- Porter des gants en nitrile, blouse de laboratoire, lunettes de sécurité, gants en nitrile grands industriels, un tablier en cas de déversement, et un écran facial en tout temps lorsque vous utilisez le banc CMOS.
- Utilisez bacs en plastique comme confinement secondaire lorsque les solutions sont préparées.
- Utilisez un fluoré gobelet gradué de polymère inerte pour la manipulation du HF concentré.
- Assurez-gluconate de calcium pommade disponible en premiers soins pour toute exposition de la peau.
- Seul le personnel correctement formé pour exécuter le processus.
- Toujours veiller à un autre membre du laboratoire est présent en cas d'urgence.
- Gardez les fiches signalétiques pour tous produits chimiques près de la hotte.
- Se familiariser avec l'institution de ou déversement de produits chimiques et d'exposition les politiques de l'entreprise.
Remarque: HF imprègne facilement la peauet est un trésor de calcium, affectant les os et les nerfs préjudiciables si l'exposition se produit. L'exposition cutanée à quelques millilitres d'acide fluorhydrique concentré peut être dangereux et même mortel. Établir les précautions nécessaires pour assurer l'exposition ne se produit pas.
- Effectuer RCA1 et RCA2 dans un document distinct béchers de 250 ml en verre sur plaques séparées. Chaque bécher doit contenir une barre d'agitation. Surveiller la température de la solution au moyen de thermomètres serrées de sorte que la barre d'agitation ne bang dans le bulbe. Couvrir les verres en utilisant un verre de montre pour diminuer les effets de l'évaporation.
- RCA1 Préparation
- Placer 50 ml de 18 MQ x cm d'eau dans le bécher de RCA1 désigné l'aide d'un gobelet gradué.
- Ajouter 15 ml d'hydroxyde d'ammonium concentré (NH 4 OH) dans le bêcher. Rincer le bécher de mesure avec 25 ml d'eau et ajouter l'eau de rinçage dans le bécher de RCA1.
- Mettez sur le feu et agitateur sur la plaque chauffante et porter le bain de RCA1 à 70 ° C.
- Ajouter 15 ml de 30% de peroxyde d'hydrogène (H 2 O 2) dans le bécher de RCA1. Utiliser la solution à l'intérieur de RCA1 1 h après le H 2 O 2 a été ajouté. Le bain peut être utilisé plusieurs fois en l'espace de trois jours si 15 ml de peroxyde est ajouté au bain à chaque fois.
- Rincer le bécher de mesure soigneusement à l'eau et jetez le liquide de rinçage dans un flacon de récupération de RCA1 appropriée.
- RCA2 Préparation
- Ajouter 70 ml de 18 MQ x cm d'eau dans le bécher de RCA2 désigné en utilisant le gobelet gradué et rincé.
- Ajouter 15 ml d'acide chlorhydrique concentré (HCl). Rincer le bécher de mesure avec 20 ml d'eau et ajoutez-le dans le bécher de RCA2.
- Augmentez la chaleur et la vitesse de la plaque chauffante remuer jusqu'à ce que la solution atteigne 70 ° C.
- Ajouter 15 ml de 30% de H 2 O 2. Comme le bain de RCA1, utiliser cette solution à moins de 1 heure entre le moment où le H 2 O 2 est ajouté; en outre, le bainpeut être réutilisé plusieurs fois en l'espace de trois jours si 15 ml de H 2 O 2 est ajouté avant chaque utilisation.
- Préparation de la solution de HF
- Placer 50 ml d'eau dans un bêcher en polymère fluoré inerte.
- Mesure 4 ml d'acide fluorhydrique concentré (49%) dans le bécher de mesure en plastique et l'ajouter dans le bécher de polymère fluoré inerte.
- Rincez le bécher en plastique mesurant avec un total de 50 ml d'eau, ajouter l'eau de rinçage dans le bécher de HF. Laver le bécher de mesure soigneusement à l'eau et jetez les eaux de lavage dans un conteneur à déchets HF désigné.
5. Préparation et Nettoyage du substrat de silicium
- De coupe des plaquettes de silicium en puces
- Identifier les directions de réseau perpendiculaire et parallèle à la surface plane polie de la plaquette de silicium. Ces directions sont utilisés pour aider à faire des carrés de clivage plus facile. Les instructions suivantes concernent cleaving silicium <110> et peut ne pas convenir pour d'autres orientations cristallines.
- Placez la plaquette de silicium polie côté-up sur une surface molle, comme une serviette. Avec le stylet scribe diamanté, doucement nick le fond de la plaquette le long du bord plat principal. Placez un petit fil, comme un trombone, en dessous de la nick et appliquer doucement la pression sur la tranche en plaçant les doigts ou une pince à épiler de chaque côté de l'entaille et en poussant vers le bas. Ceci vous permettra de séparer la plaquette en deux moitiés le long de la ligne de réseau cristallin dans la direction de clivage naturel.
- Sur un autre serviette, avec un crayon et une règle, mesurer la largeur souhaitée des carrés en marquant des points à la fois sur le haut et le bas de la serviette. Reliez ces points avec des lignes droites. Cela servira de ligne directrice pour les formes, même carrés.
- Placez l'une des moitiés de la plaquette bord abord entre les lignes mesurées sur la serviette rincé contre la ligne et répétez l'étape 5.1.2 Le fraîchement brisée perpendiculaire pieces doivent maintenant être la largeur des places clivés. Tourner la plaquette horizontalement sur la serviette. Placez-le entre les lignes perpendiculaires et répéter le processus à l'étape 5.1.2.
- Stocker les puces de la plaquette fraîchement clivé dans un flacon propre rempli d'eau déminéralisée pour éviter les rayures. Les puces de silicium peuvent être stockées indéfiniment, mais doivent être nettoyés avant de commencer les expériences.
- CMOS nettoyage de la Silicon
- Lorsque la solution de RCA1 a atteint la température appropriée, submerger huit à dix 1 cm x 1 cm puces de silicium dans la solution en utilisant un panier en polymère fluoré inerte avec un "diamètre 2. Des bulles d'oxygène se forment sur les puces et les parois du bêcher. Si aucun bouillonnement se produit, le H 2 O 2 est dégradée. Laisser les puces dans la solution pendant 10 à 20 min, en agitant le panier de haut en bas toutes les quelques minutes pour garder les puces de coller ensemble.
- Soulevez le panier contenant les puces de silicium et bien les égoutter. Déplacez le panier plus to le bécher des déchets et rincer abondamment à 18 MQ x cm d'eau. Plongez dans le bécher de lavage et se trémousser de haut en bas pendant 20 sec. Égoutter le panier et rincer abondamment à l'eau sur le bécher de déchets. Vider le bécher de déchets dans un flacon de récupération de RCA1 désigné et remplir avec de l'eau.
- Après le nettoyage de RCA1 est terminée, placer le panier dans le bécher 01h50 HF pour 10 à 20 sec. Utilisez un mouvement ascendant et descendant doucement pour mélanger les puces et HF. Soulevez le seau de dunk pour permettre à la HF se vider complètement l'écart.
Remarque: Les surfaces de puces devraient être hydrophobe; l'eau ne sera pas mouiller la puce, mais au lieu de former des gouttelettes avec des angles de contact élevées. Ceci indique que l'oxyde de silicium a été gravée et à une distance de la puce est maintenant terminé par liaisons Si-H. - Placer le bécher de lavage et de rinçage bêcher dans un bac en plastique, déplacer le panier sur le bécher de rinçage et rincer à 18 MQ x cm d'eau. Immerger le panier dans le bécher de lavage et agiter pendant 20 secondes.
- Procéder à un second drain et Lérinscycle de e avec 18 MQ x cm d'eau. Dump de l'eau de lavage dans le bécher de rinçage et remplissez le bécher de lavage avec de l'eau. Verser tous les déchets dans un flacon de récupération de HF plastique désigné.
- Lorsque la solution de RCA2 a atteint la température appropriée, immerger les morceaux de silicium dans les solutions en utilisant le panier. Laisser la solution pendant 10 à 20 min. La puce sera désormais hydrophile en raison de la croissance d'une mince (1-2 nm) film d'oxyde.
- Enlever après la quantité appropriée de temps et suivre les mêmes procédures de rinçage que pour RCA1. Éliminer les déchets dans le conteneur à déchets RCA2 appropriée. Retirer chaque puce du panier avec des pincettes en plastique, rincer avec de l'eau, et sécher avec de l'azote.
- copeaux de magasin dans une boîte de plaquette en plastique ou dans un flacon de 18 MQ x cm d'eau. Le silicium restera propre lorsqu'il est stocké dans l'eau pendant environ trois jours après le nettoyage. Assurez-vous que la zone de travail est correctement nettoyé et l'extérieur des gants CMOS ont été lavés. Laissez le CMgants de OS dans le capot pour sécher.
6. Dépôt ADN Origami sur silicium APTES fonctionnalisés
- Auto-assemblée Formation monocouche sur silicium
- APTES réchauffer à température ambiante avant l'ouverture. Si la bouteille est trop froid, la condensation peut se produire, entraînant l'hydrolyse des APTES pendant le stockage. Ajouter 1980 pi de 18 MQ x cm d'eau et 20 pi de APTES à un flacon de scintillation propre et agiter pour mélanger. Utiliser immédiatement cette solution.
- Placez une puce de silicium nettoyé réfléchissante côté-up dans le flacon de scintillation, couronner le, et laissez reposer pendant 20 min. Retirer la puce en utilisant une pince à épiler et rincer avec 200 pi d'eau et à sec pendant 1 min avec un courant de N2.
- Déposer l'ADN origami sur APTES fonctionnalisés Silicon
Remarque: Les étapes pour déposer l'ADN origami sur le silicium fonctionnalisés sont analogues à celles pour déposer sur le mica.- Mélanger rapidement le flacon ADN origami et la pipette 4 pi de solutionsur le substrat de silicium. Si nécessaire, augmenter le volume de solution d'ADN origami utilisé pour couvrir la totalité du substrat en silicium fonctionnalisés est plus hydrophobe que le substrat de mica. Utilisez une lamelle de verre à appuyer sur la solution de dépôt vers le bas et éviter l'évaporation pendant de longues dépositions.
- Laisser reposer la solution pendant le temps nécessaire à la concentration utilisée et la couverture souhaitée (voir les figures 2 et 3 pour l'effet du temps et de la concentration sur la couverture de surface). Rincez le substrat avec 100 ul de 18 MQ x cm d'eau stérile et sec avec N2 pendant 1 min.
- Répéter le rinçage avec un 100 ul supplémentaires de l'eau stérile et sécher le substrat avec N2 pendant 3 min.
- Conserver l'échantillon dans un récipient propre jusqu'à ce que d'autres expériences ou d'imagerie peuvent être effectuées. Les échantillons commencent à montrer accumulation de particules après environ une à deux semaines de stockage en fonction de la façon dont much ils sont manipulés.
7. AFM imagerie et l'analyse d'images d'échantillons d'ADN Origami
- Utilisez AFM en mode Tapping dans l'air à des fins d'imagerie. Mode de taraudage assure une force minimale sera appliquée aux nanostructures fragiles, par rapport au mode de contact.
Remarque: Les paramètres d'imagerie dépendront de l'instrument. Toutes les images présentées ont été capturées à l'aide d'un MultiMode Nanoscope IIIa. - Sélectionnez sondes AFM pour sans contact / mode dans de prélèvement d'air, avec revêtement réfléchissant d'or, une fréquence de résonance (nominal) de ~ 300 kHz, force constante de 40 N / m, les pourboires rayon <10 nm.
- Traiter et analyser les images AFM utilisant NanoScope logiciel d'analyse. Effectuer des calculs de couverture utilisant ImageJ. 19
Résultats
Deux variables dictent la couverture de l'ADN origami sur le substrat: concentration de la solution et de la durée d'exposition. Les caractéristiques d'adsorption de l'ADN origami sur le mica et APTES oxyde de silicium fonctionnalisés ont été précédemment rapporté. 13 La relation entre la concentration de l'ADN origami dans la solution de dépôt et les couvertures finales sur le mica sont résumées dans le tableau 1 et figure 2, montrant de plus en plus des résultats de concentration par une couverture accrue. La dépendance temporelle de liaison est vu dans la couverture de surface Figure 3. A été précédemment étudié pour quantifier le comportement de liaison de l'ADN origami mica et sur les surfaces d'oxyde de silicium modifiés. Pliage d'ADN dans 12 mM de magnésium dans le tampon TAE 1X a 83,3% ± 3,1% de couverture de mica temps d'absorption après 30 minutes sur la surface. La couverture maximale d'oxyde de silicium modifié avec APTES SAM est observée après 60 min, ce qui est inférieur à la couverture maximale de mica. UNplus de temps de dépôt est nécessaire si une couverture de surface élevée est nécessaire sur APTES oxyde de silicium fonctionnalisés.
Il ya plusieurs variables qui peuvent causer une mauvaise préparation de l'échantillon. Le plus gênant est un rinçage inadéquat et le séchage. Si la solution tampon n'a pas été correctement rincé, de grands agrégats se forment sur le substrat (figure 4A). 'origami îles d'ADN »sont observés lorsque les nanostructures adhèrent aux plaques de sels de magnésium à la surface (figure 4B). Enfin, avec des échantillons de couverture élevés, il est possible d'avoir des composants tampons excès de transition entre l'ADN origami individuelle (figure 4C), ce qui rend difficile de différencier nanostructures par AFM. Ces résultats peuvent être évités par suite d'un rinçage soigneux et un protocole de séchage pour les deux substrats de silicium et le mica.
Formation de films APTES sur des substrats d'oxyde de silicium peut poser des problèmes ainsi. Les plaquettes de silicium ont unecouche d'oxyde de silicium brut qui doit être enlevé et, une couche d'oxyde de silicium plus mince lisse réformé avant de pouvoir être fonctionnalisé. Un substrat de silicium convenablement nettoyé est illustré sur la figure 5A. Pendant le nettoyage de la tranche de silicium, il est important d'assurer que le nombre de puces de silicium ne soit pas trop élevée, car il est possible pour les deux jetons à se coincer les uns aux autres, le blocage exposition aux réactifs (Figure 5B). Si le silicium nettoyée a été stocké dans 18 MQ x cm d'eau pour plus d'une semaine, la couche contaminant réformer et recleaning est nécessaire. L'alimentation APTES peut également causer des problèmes avec la préparation des échantillons. APTES polymérise facilement par hydrolyse, qui est la base pour la formation de la monocouche. 20 L'ampleur de cette polymérisation dépend de la concentration de l'eau que l'APTES est exposé. Au fil du temps et par une utilisation répétée, il est possible que l'eau se condense à l'intérieur de la bouteille de APTES et contaminer laapprovisionnement. La polymérisation produit résultant gros agrégats qui adhèrent au substrat (Figure 5C). La rugosité accrue et la présence d'agrégats rend l'identification des nanostructures d'ADN par AFM difficile. Il est de bonne pratique pour stocker la bouteille APTES dans un sac en plastique dans le réfrigérateur, et de laisser le APTES bouteille chaude à la température ambiante avant de l'ouvrir pour éviter la condensation.
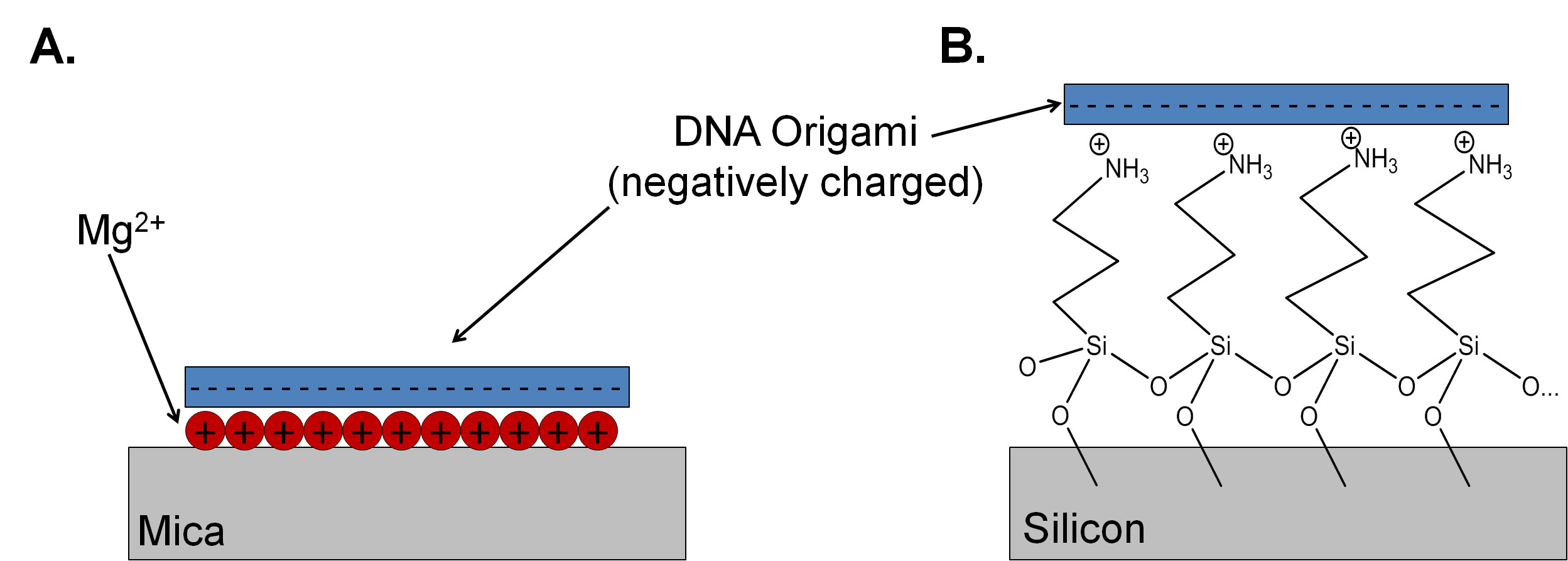
Figure 1. Un oxyde de silicium comparer le mécanisme de liaison pour l'ADN origami sur (A) et de mica (B) APTES fonctionnalisé schématique (non à l'échelle). La liaison avec le mica est médiée par la présence de cations divalents, généralement Mg 2+. Une monocouche de l'aminé protonée terminé le 3-aminopropyltriéthoxysilane est utilisé pour favoriser l'adhérence sur le substrat en oxyde de silicium.

Figure 2. images AFM illustrant la couverture variable après 10 min dépositions de (A) 2 nM, (B) 4 nM, et (C) 6 nM de mica. L'échelle de hauteur pour toutes les images est de 5 nm.

Figure 3. Tendances de la couverture de surface pour 2 nM ADN origami dans un tampon TAE 1X, 12 mM de Mg 2+. Marqueurs MICA = ligne violet et cercle, APTES = lignes jaunes et les marqueurs de triangle. N = 3 dans la détermination de l'erreur-type.

Figure 4. Mauvais rinçage et le séchage peuvent causer (A) l'agrégation de solution sur le substrat, (B) îles ADN origami, et (C) pontage des nanostructures suréchantillons de couverture élevés en présence de sels de tampon en excès. L'échelle de hauteur pour toutes les images est de 5 nm.

Figure 5. (A) silicium Clean devrait avoir rugosité RMS inférieure à 0,5 Å de plus de 1 micron carré. Un APTES SAM complète déposée sur une bonne oxyde natif doit avoir une rugosité RMS inférieure à 1 A sur 1 micron carré. (B) APTES film formé sur un oxyde de silicium incomplètement nettoyé avec une rugosité RMS de 2,29 nm sur 1 micron carré. . Notez les lacunes et la rugosité du film APTES (C) APTES surface formée à partir d'un échantillon de APTES contaminés par des déchets condensé; hydrolyse dans les formes APTES de bouteilles de grandes particules. L'échelle de la hauteur pour toutes les images est de 5 nm.
Tableau 1. Pourcentage de mesures de couverture pour les substrats de mica avec varying ADN concentrations de la solution de l'origami. Toutes les heures de dépôt sont 10 min.
| Solution ADN Origami | Couverture% sur Mica Substrat |
| 2 nM | 8,49 ± 2,67 (n = 5) |
| 4 nM | 55,89 ± 5,65 (N = 3) |
| 6 nM | 77,44 ± 1,89 (N = 4) |
Discussion
Il ya plusieurs étapes qui doivent être souligné pour atteindre des résultats cohérents et idéales. Pour les échantillons de mica, après un rinçage stricte et approfondie et de séchage régime, comme dans les étapes 3.3 et 3.4, fera en sorte que des images de haute qualité de personne origami d'ADN peuvent être atteints en utilisant l'AFM sans les divers problèmes décrits dans la section des résultats représentatifs. D'une importance primordiale pour des échantillons de silicium est la propreté du substrat. En suivant les procédures de nettoyage décrites dans l'étape 5.2 soigneusement et méticuleusement veillera à ce que une surface d'oxyde de silicium nettoyée de manière appropriée sera atteint. En outre, la surveillance de la qualité et l'efficacité des produits chimiques, tels que le peroxyde d'hydrogène, l'acide fluorhydrique, et APTES, fera en sorte que la procédure se déroule bien.
Les techniques décrites ne sont pas limités seulement à des solutions aqueuses d'APTES. Une monocouche mixte de APTES et de chlorure de trimethylaminopropyltrimethoxysilyl (TMAC) peut nous êtreed pour régler la charge de surface de silicium et de promouvoir la variable de l'ADN origami adhérence. 11 Le TMAC contient un quaternaire terminale chargée permanence amine -N (CH 3) 3 +, par rapport à la charge dépendant du pH sur APTES. Parce que l'environnement de la solution ne modifie pas l'état de protonation ou la charge de TMAC, la variation de la concentration de la solution de TMAC permet de syntoniser la charge de surface de la monocouche mixte et affecte l'interaction entre le substrat et l'origami d'ADN. Optimal origami d'ADN liaison a été observée pour les monocouches ayant une charge de surface de 0,75-1,5 charges / nm 2, ce qui correspond à SAM contenant 100% à 40% de concentration TMAC. Cette charge optimale de la surface incité couvertures ADN origami d'environ 110 origami / um 2 sur APTES SAM et 120 origami / um 2 sur TMAC SAM.
Un avantage de silicium est sa compatibilité avec les processus de formation de motifs lithographiques. Haute teneur en magnésiumLes concentrations peuvent être utilisées avec des oxydes de silicium traité au plasma pour favoriser la liaison sélective de l'ADN origami de silicium. Il faut faire attention lors de la suppression de l'échantillon à partir de la solution de dépôt à éviter rinçage ions magnésium à une distance et à déformer la origami d'ADN. 21,22 Les formes de traitement APTES fixés par covalence des cations sur la surface d'oxyde de silicium, de sorte que le lavage ou le rinçage avec du tampon ou est de l'eau ne pas endommager l'origami ADN fixé. La méthode du «décollage moléculaire» est une autre voie possible pour modeler des substrats de silicium et la promotion de l'ADN origami adhérence. Le substrat de silicium est structurée en utilisant la lithographie par faisceau d'électrons et APTES est déposée sur le substrat exposé. À la suite de décollement de la résine photosensible, origami ADN peut être déposée sur la surface à motifs, de préférence de liaison à l'APTES. 23
L'interaction de l'ADN origami avec un substrat change sa stabilité, l'ouverture de nouvelles avenues pour la recherche et les applications. ADN Origami adhéré à mica peut être chauffé à 150 ° C sans altération visible de dimensions des nanostructures et des changements chimiques minimes. 24 Ceci est en contraste frappant avec la fragilité de l'ADN origami en solution, où les nanostructures sont complets déshybridées-dessus de 70 ° C. 25, 26 Cette stabilité est maintenue sur des supports d'oxyde de silicium chauffées. 27 Même dans divers systèmes de solvants tels que l'hexane, le toluène et l'éthanol, la forme et la couverture des nanostructures sont maintenues. La stabilité surprenante de pliage d'ADN indique que les applications qui étaient auparavant considérées incompatibles, tels que plasma dépôt en phase vapeur, l'utilisation de résines photosensibles et des solvants courants, et des environnements uniques de dépôt chimique, peuvent être utilisés en conjonction avec l'ADN origami. Toutefois, si l'origami d'ADN maintenir leur fonctionnalité est encore inconnue et peut limiter les applications possibles.
Bien que la stabilité de l'ADN origami à température élevées et dans un nombre limité de systèmes de solvants a été déterminée, la stabilité à long terme de l'ADN origami sur des substrats est encore inconnue. L'utilisation de techniques stériles est nécessaire pour éviter une éventuelle contamination, mais cela ne peut pas être évitée après le dépôt de surface. Imagerie et l'analyse de l'échantillon doivent être remplis presque immédiatement après la préparation des échantillons; si l'échantillon est stocké pendant trop longtemps (plus d'une semaine) divers exemples de la dégradation de l'échantillon sont souvent identifiés, y compris l'accumulation de particules et de nanostructures d'ADN cassées. Avenues possibles de la recherche en nanoélectronique, les biocapteurs, et d'autres applications d'origami d'ADN de substrat à base peuvent être limités par la déstabilisation en fonction du temps de l'ADN. Identifier les limites de ces techniques pour les applications nécessitant une stabilité pendant des périodes de temps plus longues nécessite une enquête plus approfondie.
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
The authors thank Dr. Gary Bernstein for use of the AFM.
matériels
| Name | Company | Catalog Number | Comments |
| Eppendorf epT.I.P.S. Reloads, capacity 2-200 μl | VWR International, LLC | 22491733 | 10 reload tray of 96 tips |
| Microcentrifuge Tubes, Polypropylene | VWR International, LLC | 87003-290 | 0.65 ml, natural |
| Research Plus Pippete - Single Channel - 20-200 μl | A. Daigger & Company, Inc. | EF8960F-3120000054 EACH | Adjustable Volume |
| Research Plus Pippete - Single Channel - 2-20 μl | A. Daigger & Company, Inc. | EF8960D-3120000038 EACH | Adjustable Volume |
| Scotch 237 Permanent Double-Sided Tape | Office Depot, Inc. | 602710 | 3/4" x 300", Pack of 2 |
| Vortex Mixer | Thermo Scientific | M37610-33Q | |
| Wafer container single, 2" (50 mm), 60 mm x 11 mm | Electron Microscopy Sciences | 64917-2 | 6 per pack |
| 6" Wafer, P-type, <100> orientation, w/ primary flat | Nova Electronic Materials, Ltd. | GC49266 | |
| Powder-Free Nitrile Examination Gloves | VWR International, LLC | 82062-428 | Catalog number is for size large |
| High Accuracy Noncontact probes with Au reflective coating | K-Tek Nanotechnology, Inc. | HA_NC/15 | |
| Autoclave Pan | A. Daigger & Company, Inc. | NAL692-5000 EF25341C | |
| Sol-Vex II Aggressive Gloves, Size: 9-9.5; 15 mil, 13 inch - 1 dz | Spectrum Chemical Mfg. Corp. | 106-15055 | Before use, rinse with water and scrub together until no bubbles form on the gloves. |
| Tweezers PTFE 200 mm Square | Dynalon Corp. | 316504-0002 | |
| Muscovite Mica Sheets V-5 Quality | Electron Microscopy Sciences | 71850-01 | 10 per pack |
| Mica Disc, 10 mm | Ted Pella, Inc | 50 | Mica discs are optional |
| Scriber Diamon Pen for Glassware | VWR International, LLC | 52865-005 | |
| Scintillation Vials, Borosilicate Glass, with Screw Cap - 20 ml | VWR International, LLC | 66022-060 | Case of 500, with attached polypropylene cap and pulp foil liner |
| 4 x 5 Inch Top PC-200 Hot Plate, 120 V/60 Hz | Dot Scientific, Inc. | 6759-200 | |
| Straight-Sided Glass Jars, Wide Mouth | VWR International, LLC | 89043-554 | Case of 254, caps with pulp/vinyl liner attached |
| Standar-Grade Glass Beaker, 250 ml Capacity | VWR International, LLC | 173506 | |
| Beakers, PTFE | VWR International, LLC | 89026-022 | For use with HF |
| Shallow form watch glass, 3" | VWR International, LLC | 66112-107 | Case of 12 |
| Plastic Storage Container | VWR International, LLC | 470195-354 | For secondary container |
| General-Purpose Liquid-In-Glass Thermometers | VWR International, LLC | 89095-564 | |
| High precision and ultra fine tweezers | Electron Microscopy Sciences | 78310-0 | |
| Polycarbonate Faceshield | Fisher Scientific, Inc. | 18-999-4542 | |
| Neoprene Apron | Fisher Scientific, Inc. | 19-810-609 | |
| Calcium Gluconate, Calgonate | W.W Grainger, Inc. | 13W861 | Tube, 25 g |
| Hydrogen Peroxide 30% CR ACS 500 ml | Fisher Scientific, Inc. | H325 500 | HARMFUL, TOXIC |
| 3-Aminopropyltriethoxysilane | Gelest Inc. | SIA0610.0-25GM | Let warm to room temperature before use. |
| Ammonium hydroxide, 2.5 L | Fisher Scientific, Inc. | A669-212 | HARMFUL, TOXIC |
| Hydrochloric acid | Fisher Scientific, Inc. | A144-212 | HARMFUL, TOXIC |
| Hydrofluoric acid | Fisher Scientific, Inc. | A147-1LB | HARMFUL, TOXIC |
| MultiMode Nanoscope IIIa | Veeco Instruments, Inc. | Any AFM capable of tapping mode is suitable for analysis | |
| Dunk basket | Made in lab | Made in lab | The dunk basket was made using the bottom of a PTFE bottle with holes drilled in, PTFE handle, and all PTFE screws. |
Références
- Rothemund, P. W. K. Folding DNA to create nanoscale shapes and patterns. Nature. 440, 297-302 (2006).
- Anderson, E. S., et al. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 459, 73-77 (2009).
- Wang, Z., Ding, B. Engineering DNA Self-Assemblies as Templates for Functional Nanostructures. Acc. Chem. Res. 47, 1654-1662 (2014).
- Xu, K., et al. Graphene Visualizes the First Water Adlayers on Mica at Ambient Conditions. Science. 329, 1188-1191 (2010).
- Bustamante, C., et al. Circular DNA-molecules imaged in air by scanning force microscopy. Biochemistry. 31, 22-26 (1992).
- Hsueh, C., et al. Localized Nanoscopic Surface Measurements of Nickel-Modified Mica for Single-Molecule DNA Sequence Sampling. ACS Appl Mater. Interfaces. 2, 3249-3256 (2010).
- Pastre, D., et al. Anionic polyelectrolyte adsorption on mica mediated by multivalent cations: A solution to DNA imaging by atomic force microscopy under high ionic strengths. Langmuir. 22, 6651-6660 (2006).
- Woo, S., et al. Self-assembly of two-dimensional DNA origami lattices using cation-controlled surface diffusion. Nature Communications. 5, 4889 (2014).
- Vesenka, J., et al. Substrate preparation for reliable imaging of DNA molecules with the scanning force microscope. Ultramicroscopy. 42-44, 1243-1249 (1992).
- Albrechts, B., et al. Adsorption studies of DNA origami on silicon dioxide. , (2010).
- Sarveswaran, K., et al. Adhesion of DNA Nanostructures and DNA Origami to lithographically patterned self-assembled monolayers in Si[100]. Proc. of SPIE-Soc. Opt. Eng. 7637, 76370M-1 (2010).
- Kern, W., Puotien, D. A. Cleaning solutions based on hydrogen peroxide for use in silicon semiconductor technology. RCA Rev. 31, 187-206 (1970).
- Pillers, M., Goss, V., Lieberman, M. Electron-Beam Lithography and Molecular Liftoff for Directed Attachment of DNA Nanostructures on Silicon: Top-down Meets Bottom-up. Acc. Chem. Res. 47, 1759-1767 (2014).
- Saccá, B., Niemery, C. M. DNA Origami: The Art of Folding DNA. Angew. Chem. Int. Ed. 51, 58-66 (2012).
- Douglas, S. M., et al. Rapid prototyping of 3D DNA-origami shapes with caDNAno. Nucleic Acids Res. 37, 5001-5006 (2009).
- Ben-Ishay, E., et al. Designing a Bio-responsive Robot from DNA. Origami. J. Vis. Exp. (77), e50268 (2013).
- Woo, S., et al. Programmable molecular recognition based on the geometry of DNA nanostructures. Nature Chemistry. 3, 620-627 (2011).
- Schlegel, M. L., et al. Cation sorption on the muscovite (001) surface in chloride solutions using high-resolution X-ray reflectivity. Geochim. Cosmochim. Acta. 70, 3549-3565 (2006).
- Rasband, W. S., Howarter, J. A., et al. National Institutes of Health. Langmuir. 22, 11142-11147 (2006).
- Kershner, R. J., et al. Placement and orientation of individual DNA shapes on lithographically patterned surfaces. Nature Nanotechnology. 4, 557-561 (2009).
- Hung, A. M., et al. Large-area spatially ordered arrays of gold nanoparticles directed by lithographically confined DNA origami. Nature Nanotechnology. 5, 121-126 (2010).
- Sarveswaran, K., et al. et al.Adhesion of DNA nanostructure and DNA origami to lithographically patterned self-assembled monolayers on Si[100. Proc. SPIE-Int. Soc. Opt. Eng. 7637, 76370M (2010).
- Pillers, M. A., Lieberman, M. Thermal stability of DNA origami on mica. J. Vac. Sci. Technol. B. 32, 040602 (2014).
- Song, J., et al. Direct Visualization of Transient Thermal Response of a DNA. Origami. J. Am. Chem. Soc. 134, 9844 (2012).
- Wei, X., et al. Mapping the thermal behavior of DNA origami nanostructures. J. Am. Chem. Soc. 135 (16), 6165-6176 (2013).
- Hyojeong Kim, ., et al. Stability of DNA Origami Nanostructures under Diverse Chemical Environments. Chem. Mater. 26, 5265-5273 (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon