Method Article
Desvendando os jogadores invisíveis no Oceano - Um Guia de Campo para Química da Água e Microbiologia Marinha
Neste Artigo
Resumo
Here, we present a comprehensive protocol to assess the organic and inorganic nutrient availability and the abundance and structure of microbial and viral communities in remote marine environments.
Resumo
Aqui apresentamos uma série de protocolos de pesquisa cuidadosamente testados e bem padronizados, adaptados para o uso em ambientes marinhos remotos. Os protocolos de amostragem incluem a avaliação dos recursos disponíveis para a comunidade microbiana (carbono orgânico dissolvido, matéria orgânica particulada, nutrientes inorgânicos), e uma descrição abrangente das comunidades virais e bacterianas (via contagem viral e microbianas diretos, enumeração de micróbios autofluorescentes, e construção de metagenomes virais e microbianos). Usamos uma combinação de métodos, que representam um campo disperso de disciplinas científicas que incluem protocolos já estabelecidos e algumas das técnicas mais recentes desenvolvidas. Técnicas de sequenciamento especialmente metagenomic utilizados para a caracterização da comunidade virais e bacterianas, só foram estabelecidas nos últimos anos, e são, portanto, ainda sujeita a constante melhoria. Isto levou a uma variedade de procedimentos de amostragem e processamento de amostras currently em uso. O conjunto de métodos apresentado aqui fornece uma abordagem até data para coletar e processar amostras ambientais. Parâmetros abordados com estes protocolos produzir o mínimo de informação essencial para caracterizar e compreender os mecanismos subjacentes da dinâmica da comunidade virais e microbianas. Dá fácil de seguir as orientações para a realização de pesquisas abrangentes e discute etapas críticas e possíveis ressalvas pertinentes a cada técnica.
Introdução
Os ecossistemas marinhos são submetidas a uma ampla gama de perturbações que resultam em alterações de disponibilidade de nutrientes para a biomassa de predadores. Durante a última década, vários estudos têm demonstrado a importância das comunidades microbianas em ecossistemas marinhos 1-4. É evidente que as alterações na comunidade bacteriana e viral estão intimamente associados com a degradação global dos ambientes marinhos 5. Estas alterações podem ser facilitada por geochemistry alterada na coluna de água, tais como a disponibilidade de oxigénio ou de carbono remineralização 6,7. Como resultado da interdependência entre o macroorganismos, química da água e loops de feedback atividade microbiana pode acelerar a taxa de degradação dos ecossistemas 8.
Nos anos 1970 e 80, várias tentativas foram feitas para desvendar os ciclos biogeoquímicos em ecossistemas marinhos 11/09. Um dos principais desafios para estes estudos iniciais foi a faltade abordagens padronizadas para medir os parâmetros biogeoquímicos avaliados. Embora houvesse protocolos disponíveis, principalmente sobre os nutrientes da água do mar 12,13, a avaliação da dinâmica de carbono e estrutura da comunidade microbiana foram limitados pelas ferramentas e métodos disponíveis. Com o JGOFS EqPac comparação métodos, a Sharp et al., 14 sugeriram pela primeira vez um protocolo fiável e normalizado para as avaliações (DOC) de concentração de carbono orgânico dissolvido. Até o início da década de 2000 os desafios analíticos para determinar os recursos disponíveis para a comunidade microbiana tinha, assim, em grande parte resolvidos e abordagens para caracterizar comunidades microbianas em ambientes controlados cultura tinha sido estabelecida (ver DeLong 15). Os métodos de sequenciamento tradicionais na época em grande parte se baseou em culturas clonais cultivadas. Seqüenciamento do gene ambiental precoce de amostras naturais revelou, no entanto, que a maior parte da biodiversidade microbiana tinha sido perdida por cultivmétodos baseados em ation 16. Em 2002, Breitbart et al., 17 utilizadas shotgun de sequenciação pela primeira vez para descrever a comunidade viral em amostras de água do mar ambientais que estabelecem um método para sequenciar o genoma inteiro de comunidades virais marinhos não cultivadas.
Dentro das avaliações microbianas existentes, os vírus ainda continua a ser particularmente estudado, como a maioria são difíceis de cultura e que falta um marcador universalmente conservado como o 16S RNA ribossomal (rRNA) genes normalmente utilizados para avaliar a diversidade de perfis e comunidades. A abordagem metagenômica sequenciamento fornece uma alternativa aos métodos dependentes de cultura e alvo de genes para analisar comunidades virais complexas. Enquanto os primeiros metagenomes virais geradas a partir de água do mar foram sequenciados utilizando sequenciamento Sanger 17, o desenvolvimento de tecnologias de sequenciamento de última geração e técnicas moleculares melhoria levou a um rápido aumento em estudos metagenomic 18 . O fluxo de trabalho laboratorial corrente para metagenomics virais envolve a separação, o enriquecimento, e / ou a concentração de partículas virais, seguido de ácido nucleico (DNA ou RNA) extracção, a preparação da biblioteca, e sequenciação de 19-21.
Para aprofundar o conhecimento existente sobre viral, bacteriana e funcionamento bioquímico em ecossistemas aquáticos, comparando conjuntos de dados em vários sistemas ao redor do mundo é essencial. No entanto, os protocolos estabelecidos têm sido largamente desenvolvido para ambientes costeiros com instalações laboratoriais acessíveis. Assim, a falta de métodos de campo padronizados dificulta as comparações entre estes ecossistemas. Aqui apresentamos exaustivamente testado e adaptações de estado dos protocolos de pesquisa de arte bem padronizado para uso em locais de campo remotas. Esses métodos foram provados com sucesso em vários estudos que medem a última década, 5,22 e são usadas atualmente por vários laboratórios marinhos, bem como sobre a NOAAAvaliação Reef e Programa de Monitoramento (RAMP) ao longo do Oceano Pacífico 4. Os protocolos de amostragem incluem parâmetros da água química (concentrações de carbono inorgânico e orgânico, concentração de nutrientes inorgânicos), abundância viral e microbiana (contagem bacteriana direta, contagem viral diretos, e citometria de fluxo para avaliar autotroph vs índices de Heterotroph) e estrutura da comunidade microbiana e viral e potencial metabólico através da análise metagenômica (para uma visão geral veja a Figura 1). Os resultados obtidos a partir dos métodos descritos aqui são necessárias a fim de elucidar principais parâmetros bioquímicos fundo necessários para caracterizar exaustivamente ecossistemas aquáticos.
Protocolo
1. Preparação de Instrumentos
- Preparação da instalação de Filtração
- Prepara-se uma solução a 5% de ácido clorídrico (HCl) (0,5-0,6 M; adicionar 250 ml de 32-36% HCl para 4.75 L de água destilada para fazer 5 L de solução final). Conserva-se a solução em um recipiente de polietileno de alta densidade (HDPE) por até 2 semanas.
- Deixe todas as partes que entram em contato com a amostra (excepto os filtros) para 24 horas na solução de HCl 5% a sanguessuga fora potenciais (DOC) contaminantes carbono orgânico dissolvido. Após cada evento de amostragem lavar todas as peças e lave as linhas com cerca de 30 ml de solução de HCl 5% para evitar a contaminação de carbono (por exemplo, a partir de fumos de gás, petróleo, barcos, mosquito spray).
- Manter todos os itens de amostragem pré-embrulhados em folha de alumínio queimado ou tapados até ao imediatamente antes da utilização.
- Precombust 25 milímetros GF / F filtros embrulhadas em papel alumínio em um forno de mufla a 550 ° C durante 12 horas para volatilizar fora todo o carbono e armazená-los em local limpo, secocolocar até à sua utilização. Use de ácido lavou-se fórceps estéreis e, luvas sem pó para lidar com os filtros precombusted em todos os momentos.
- Preparação de Materiais de amostragem virais metagenome
- Lave cuidadosamente quatro garrafões de 20 L recolhíveis polietileno de baixa densidade e quatro baldes de 20 L de polietileno de alta densidade e as tampas com 10% de água sanitária.
- Posteriormente lavar todos os itens 3x com água destilada e, em seguida, amostra e feche garrafões com torneira.
- Filtros de Lavagem de fluxo tangencial (TFF), após cada utilização, de acordo com o seguinte protocolo, e limpo preventivamente se o filtro não tem sido utilizado nos últimos 5 dias.
- Dissolve-se 50 g de pastilhas de NaOH em 5 L de água num balde de lavagem.
- Anexar um TFF molhado para a tubulação e montagem de acordo com a Figura 3.
- Execute bomba peristáltica em ~ 30 rpm sem contrapressão para mover um pouco da solução de lavagem NaOH através do sistema. Depois de lavagem flui da linha de retorno, mova transferência para lavar baldepara completar a circulação de solução.
- Aplicar contrapressão ao sistema através da colocação de um grampo sobre a linha de retorno a jusante da TFF. Isso vai forçar solução de lavagem a linha filtrado. Tempo excessivo (5 min) para inundar o lado de fora da TFF e produzir filtrado é indicativo de um TFF degradada.
- Executar o sistema até que toda a solução de NaOH está nas linhas. Em seguida, retire a contrapressão, a solução de correr para fora do sistema e descarte.
- Lavar o TFF por água corrente sob contrapressão até que o pH da água que flui a partir das linhas de retorno e filtrado é pH 7-8.
- Remover o tubo da bomba peristáltica e de TFF. Voltar a vedar TFF para armazenamento. Certifique-se de que a TFF permanece molhado até a próxima utilização.
Coleção 2. Amostra
NOTA: Durante o transporte de todas as amostras coletadas devem ser armazenadas legal (se possível 4 ° C) e não expostos à luz solar direta, até posterior processamento.
- Amostragempara carbono, nutrientes, microscopia, citometria de fluxo, e microbianas Metagenomics
- Tome duas unidades Hatay Niskin por evento de amostragem (resultando em 4 L de amostra de água). Abra os vasos de amostragem pouco antes de submergir (ar preso caso contrário irá impedir descendente).
- No respectivo local, lave o interior do recipiente de amostragem com a amostra de água através da abertura de ambas as extremidades do cilindro e movendo-se ao longo do eixo polar através da água.
- Pegue a amostra e fechar cuidadosamente o recipiente de amostragem. Verifique se o local de amostragem é a montante do barco e mergulhadores para evitar a contaminação.
- Amostragem para Virais Metagenomics
- No site alvo montar bomba de esgoto em 20 L garrafão.
- Encha garrafão com o objetivo de esgoto bomba de entrada na área e cada selo garrafão alvejado com a torneira correspondente.
- Colete 4 garrafões de água do mar não filtrada (todos juntos 80 L) de cada local de amostragem.
3. Samppreparação le
NOTA: Processe as amostras na ordem apresentada aqui, começando com o COD para minimizar a possibilidade de contaminação. Use pó luvas grátis para todo o procedimento.
- Carbono Orgânico Dissolvido (DOC)
- Monte uma das duas unidades em alíquotas Hatay Niskin respectiva ranhura do conjunto de filtração. Ligue o tubo de saída que conduz ao respectivo suporte de filtro. Ligue o tubo de ar pressurizado (0,2 bar) para a unidade de Hatay Niskin (Ver Figura 2).
- Lave todas as linhas com 100 ml de água de amostra antes de colocar filtros nos suportes dos filtros. Coloque 25 milímetros pré-combustão GF / F filtro no suporte do filtro com o ácido lavado fórceps. Lavar cada garrafa DOC e tampar 3 vezes com cerca de 20 ml de água filtrada amostragem.
- Preencha frasco com cerca de 40 ml (~ 2/3 cheio) de água de amostragem. Coletar pelo menos duplicatas de amostras DOC ter um backup em caso de contaminação ou perda potencial durante o transporte.
- Fgarrafas de amostragem reeze pé na posição vertical a -20 ° C até a análise.
- Matéria orgânica particulada (POM)
- Após as amostras foram coletadas DOC continuar filtração até um total de 500 ml passaram o filtro GF / F, incluindo o que passou por enquanto a coleta de amostras DOC.
- Suporte de filtro de linha Desaparafuse.
- Retire o filtro para análise POM usando uma pinça e filtro lugar dentro de um quadrado de pré-combustão papel alumínio, dobre-parte superior voltada para si mesma, e enrole.
- Congelar os filtros à temperatura de -20 ° C durante o armazenamento até que submetida a análise elementar e isotópica.
- Nutrientes inorgânicos
- Colocar um filtro de 0,2 m Gravado-Track em cada porta-filtro. Volte a colocar as cassetes de filtros.
- Lavar cada 20 ml frasco de cintilação de plástico três vezes com água de amostragem. Encher cada garrafa para o ombro (~ 18 ml).
- Congelar a -20 ° C até análise posterior.
- Microscopy (SYBR Ouro e DAPI)
- Retire o suporte do filtro.
- Recolhe 1 ml de água em cada um dos dois tubos de microcentrífuga.
- Adicionar 66 ul de 32% de paraformaldeído a uma das aliquotas (por coloração com ouro SYBR mais tarde). Adicionar 12 ul 25% de glutaraldeído para o outro correspondente (para mais tarde coloração DAPI).
- Misture delicadamente e deixe amostras para "consertar" por pelo menos 15 minutos à temperatura ambiente no escuro.
- Citometria de Fluxo
- Inserir um filtro de policarbonato de 8,0 pm em um dos suportes de filtro para excluir detritos e grandes células eucarióticas.
- Passe água de amostragem através de preencher dois criotubos de cada local de amostragem com 1 ml da amostra.
- Adicionam-se 5 ul de 25% de glutaraldeído para cada frasco de congelação (concentração final = 0,125%).
- Inverta os frascos para misturar.
- Deixar a amostra para fixar durante 15-30 min à temperatura ambiente. Não exceder 30 min.
- Amostras do Flash congelamento em nitrogênio líquido.
- Armazene as amostras à temperatura de -80 ° C ou em liquid nitrogênio shipper seco até a análise em um citômetro de fluxo.
- Amostra metagenômica Microbial
- Retirar em suportes de filtros de linha.
- Montar diretamente a 0,22 filtro cilíndrico para a respectiva linha.
- Filtro de água restante amostra de ambas as unidades Hatay Niskin de cada local (num total de 3-4 L por filtro) através de um filtro.
- Após a filtração empurrar a água restante de cada um dos filtros, utilizando uma seringa de 10 ml limpos cheios de ar.
- Coloque filtro de volta em sua embalagem original e feche o pacote com fita laboratório.
- Armazenar filtros embalados individualmente a -20 ° C.
- Amostra metagenômica Viral
- Transferir amostras virais metagenome imediatamente para os baldes lavados para garantir que não haja amostra for perdido ou contaminado pelo ambiente.
- Pré-filtro usando o grande tamanho dos poros da malha de nylon (25-100 mm) para remover os resíduos e material celular antes da concentração 19.
- Defina-se a TFF, como mostrado na Figura 3, colocando a linha de distribuição de amostra dentro de um balde, e deixando as linhas de retorno e filtrado em execução para uma pia.
- Ligue bomba peristáltica e lave as linhas com 1-2 L de água da amostra.
- Coloque linha de retorno no balde amostra para completar o ciclo, adicione 0,7 bar de contrapressão.
- Apesar de concentrar a água do mar, encher reservatório de amostra como o nível cai. Quando o nível de água descer abaixo da linha de entrada no balde, transferência concentrado em um lavado-água sanitária, lavagem tripla tripour copo e continuar se concentrando.
- Se reservatório copo está vazio, remova contrapressão, aumentar a taxa de bomba e empurrar toda a amostra através das linhas, recuperando-o no tripour.
- Passe o concentrado através de 0,45 filtros cilíndricos para remover a maioria das bactérias, enquanto não discriminar nenhum linhagens virais.
- Mudar os filtros cilíndricos de 0,45 mm após cada 150 ml. Recolher o 0,45-filconcentrado viral trou em tubos de 50 ml.
- Adicionar 250 ul de clorofórmio a cada alíquota de 50 ml de concentrado viral filtrada para eliminar bactérias residuais. Inverta para misturar e armazenar, em pé, a 4 ° C até o processamento subseqüente.
- Seque os 0,45 filtros e armazená-los como descrito nos passos 3.6.5 e 3.6.6.
4. Processamento das Amostras Microscopia
NOTA: Lavar torres de filtro com 10% de água sanitária, seguido por 95% de etanol para evitar manchas ou resíduos biológicos potencial entre as execuções. Amostras processo microscópio dentro de 1 hora e evitar a exposição manchas e filtros manchados à luz, se possível.
- Monte
- Adicione 100 ml de ácido ascórbico de 10% para 4,9 ml de PBS 1x, e misture bem.
- Adicionar 5 ml de 100% de glicerol, e misture bem.
- Filtro de montagem usando uma 0,02 mm matriz de alumina filtro de seringa descartável, alíquota e armazenar a -20 ° C.
- SYBROuro Coloração
- Pipetar uma alíquota de 500 ul de cada amostra fixo com 2% de paraformaldeído concentração final num tubo de microcentrífuga.
- Adicionar 0,5 uL de solução de 1.000x SYBR Gold cada uma das aliquotas.
- Misture delicadamente amostra e colocá-lo no escuro por 10 min.
- Coloque um filtro de 0,02 pm matriz de alumina com anelar anel de suporte de polipropileno em cada suporte de filtro do sistema de bomba de vácuo e anexar as torres de filtro (Figura 4). Verifique se o filtro de 0,02 mm é colocado em plástico do lado do suporte do filtro para cima, como esses filtros têm uma dupla porosidade.
- Com a bomba de vácuo desligada, adicionar os 500 ul de SYBR coradas amostra, juntamente com 2 ml de 0,02 mm de água filtrada de vírus livre para assegurar que a amostra é espalhada uniformemente ao longo do filtro.
- Ligar a bomba de vácuo para criar uma pressão negativa de não mais do que 1 bar para evitar vírus prejudiciais e micróbios serem filtrados.
- Permitir que toda a amostra a percorreratravés.
- Utilizando uma pinça (não o mesmo par, usado para DOC), retire cuidadosamente cada filtro da torre de filtragem, e passar sobre uma tarefa delicada limpar para assegurar que o filtro está seco.
- Pipeta de 10 mL de montagem sobre uma lâmina de microscópio e colocar o filtro seco contendo a amostra corada na gota de montagem.
- Adicionar 10 ul de montagem para a parte superior de cada filtro antes de adicionar uma lamela.
- Pressione suavemente para baixo lamela. Tente eliminar bolhas de ar, se possível, e evitar movimento lateral da lamela.
- Coloque slide em uma slidebox e armazenar a -20 ° C.
- DAPI Coloração
- Coloque um filtro de 0,2 m matriz de alumina com anelar anel de suporte de polipropileno em cada estande filtro e anexar as torres de filtro.
- Com a bomba de vácuo desligado, adicione 2 ml de 0,02 mm de água filtrada livre de vírus para cada torre utilizada.
- Pipeta de 1 ml da amostra fixado com glutaraldeído em cada torre, resultando em um volume total de of 3 ml em cada torre filtro.
- Ligar a bomba de vácuo para criar uma pressão negativa de não mais do que 1 bar.
- Filtre a amostra inteira para a 0,2 um filtro de matriz de alumina.
- Remova cuidadosamente filtro da torre de filtragem usando uma pinça (não o mesmo par, usado para DOC).
- Coloque o filtro em uma solução DAPI 100 queda de l [25 mg / ml] em uma placa de Petri manter o filtro do lado direito para cima e deixe a mancha de amostra para 15 min. Evite luz durante o processo de coloração.
- Após os 15 minutos, retire o filtro da mancha, passe-o sobre uma tarefa delicada wipe para secar e transfira para uma queda de 100 mL de água livre de 0,02 mm vírus filtrada, novamente mantendo o filtro do lado direito para cima, para lavar restante DAPI mancha durante 1 min. Repita esta lavagem uma vez.
- Filtro de montagem em microscópio deslizar a maneira idêntica como se descreveu para as amostras de SYBR Gold.
5. Extração de DNA
- Microbial metagenômica DNA
- Descongelar os filtros cilíndricos durante pelo menos 20 min à temperatura ambiente.
- Remova qualquer água do mar restante no filtro utilizando uma seringa de 10 ml. Tapar a extremidade inferior do filtro cilíndrico usando um tampão branqueada e autoclavado.
- Para cada filtro, misturar 360 ul T1 "pré-lise" tampão e 50 ul de proteinase K, e, em seguida, pipetar a mistura para dentro do filtro. Tampe a extremidade aberta.
- Incubar a reacção lise num forno de O / N (ou, pelo menos, 2 h) rotativo a 55 ° C.
- Remova uma das tampa e adicione 200 mL B3 "lise" tampão no filtro com a amostra pré-lise. Re-tampa de filtro e incubar a mistura num forno rotativo durante 10-20 min a 70 ° C.
- Extrair todo o volume do filtro cilíndrico utilizando uma seringa de 3 ml por sucção do líquido para fora com o filtro de cabeça para baixo.
- Expelir a amostra lisadas para um novo tubo de microcentrífuga, e adicionar 200 ul de etanol a 100%.
- Misture bem, coloque toda a amostra para o spincoluna, e proceda conforme as instruções do protocolo do fabricante.
- Quantificar ADN utilizando um ensaio fluorescente baseado em.
- Marinha Viral Metagenomics
- Purificação e concentração de partículas de fagos (modificado de Thurber et al. 19)
- Dissolve-se em água do mar de CsCl para preparar soluções de 1,7 g / mL, 1,5 g / mL, 1,35 g / mL, 1,2 g / mL (calibrado por pesagem de 1 ml de solução). Filtrar cada fracção com 0,02 mm de filtro antes de serem utilizados.
NOTA: Certifique-se que a densidade de cada fração for preciso para três algarismos significativos antes de usar as soluções e filtrar todas as soluções com um filtro de 0,02 mm antes do uso. A utilização de água do mar ou água do mar saturado com CsCl para diminuir ou aumentar a densidade, respectivamente. - Adicione uma lâmina de microscópio de 1 ml de reagentes combinados tal como descrito na secção 4.2 para assegurar que todos os reagentes são desprovidos de vírus antes de prosseguir.
- Adicionar 9 g de CsCl a 45 ml de concentrado viral para TFFuma densidade final de 1,12 g / ml. Leve à geladeira durante a configuração do gradiente de CsCl.
- Começando com o g / ml de solução 1,7 adicionar sucessivamente 1 ml de cada fracção sequencialmente menos denso lentamente em cada tubo, utilizando pipetas serológicas. Marque o nível de cada menisco individual e garantir que os pycnoclines entre fracções não sejam perturbados. Para mais detalhes ver o companheiro papel (Lim et al. 2014).
- Com uma pipeta serológica de carga de 7,5 ml de amostra em cada um dos 6 tubos e centrifugar a 83.000 ~ xg, 4 ° C durante 2 horas.
- Sem perturbar os gradientes de densidade de descarregar o rotor e furar o tubo imediatamente abaixo de 1,5 g / ml camada de densidade previamente marcado (Figura 5) com uma agulha de 18 G em uma seringa de 3 ml. Colhem-se 1,5 ml de VLP purificadas e transferir para um tubo de microcentrífuga novo. Repita o procedimento para todas as amostras.
- Adicionar 0,2 volume de clorofórmio para as VLP purificadas, misturar vigorosamente, incubar à TA durante 10 min.
- Adicionar 150 &# 181; l de tampão 10x DNAse a cada tubo de microcentrífuga.
- Gire a 16.000 xg por 5 min, e recolher a fase aquosa. Se o clorofórmio não agregada na parte inferior do tubo de microcentrífuga, adicionar mais tampão de DNAse, misturar e repetir.
- Adicionar ADNase I (concentração final = 2,5 unidade / ul) e incubar a 37 ° C durante 2 h, em seguida inactivar a actividade DNase por incubação a 65 ° C durante 15 min.
- Dissolve-se em água do mar de CsCl para preparar soluções de 1,7 g / mL, 1,5 g / mL, 1,35 g / mL, 1,2 g / mL (calibrado por pesagem de 1 ml de solução). Filtrar cada fracção com 0,02 mm de filtro antes de serem utilizados.
- Extracção de DNA a partir de partículas virais (VLPs)
- Uniformemente reunir os VLP purificadas de cada amostra em dois tubos de Oak Ridge limpos e autoclavados.
- Adicionar 0,1 volume de 2 M de Tris-HCl (pH 8,5) com EDTA 0,2 M, 0,01 volume de 0,5 M de EDTA, 1 volume de formamida, 10 ul de glicogénio (10 mg / ml). Misturar bem e incubar à TA durante 30 min.
- Referindo-se ao novo volume, adicionar 2 volumes de etanol RT 100%. Misturar e incubar a 4 ° C durante mais de 30 min.
- DNA Pellet girando os tubos de Oak Ridge no 17.200 xg por 60 min, a 4 & #176; C.
- Remover o sobrenadante. Lavar o sedimento de DNA duas vezes com etanol gelado a 70% usando pipetas serológicas.
- Remova todo o etanol (re-girando rapidamente, se necessário), tampa, e permitir pellet secar ao ar à temperatura ambiente por> 15 min.
- Ressuspender o sedimento de DNA para> 15 min em 567 ul de tampão TE 1X (pH 8,0). Transferir 567 ul de DNA ressuspenso para um novo tubo de microcentrífuga de 1,5 ml.
- Adicionar 30 ul de SDS a 10% (pré-aquecer a 65 ° C antes de usar) e 3 ul de proteinase K (20 ug / ml), homogeneizar e incubar durante 1 hora a 56 ° C.
- Adicione 100 ml de NaCl 5 M e misture bem. Adicionar 80 ul de solução pré-aquecida de CTAB de NaCl, vortex, e incubar durante 10 min a 65 ° C.
- Adicionar 0,2 partes de volume de clorofórmio, de vórtice e centrifugação a 16.000 xg durante 2 min. Transferir o sobrenadante para um novo tubo de microcentrífuga de 1,5 ml.
- Adicionar igual volume de fenol clorofórmio isoamílico 25: 24: 1 solução, vortex para misturar,e girar a 16.000 xg por 2 min. Transferir o sobrenadante para um novo tubo de microcentrifugação de 1,5 ml.
- Adicionar igual volume de clorofórmio, de vórtice, e centrifugação a 16.000 xg durante 2 min. Transferir o sobrenadante para um novo tubo de microcentrifugação de 1,5 ml.
- Adicionar 0,7 volume de isopropanol à fracção sobrenadante e incuba-se à temperatura de -20 ° C durante pelo menos 30 minutos para precipitar o ADN.
- Sedimentar o ADN por centrifugação a 16000 xg durante 20 min a 4 ° C. Pipetar fora o sobrenadante e lavar o sedimento duas vezes com 500 mL de etanol gelado a 70%.
- Girar a 16.000 xg e remover o etanol remanescente a partir do tubo. Secar o sedimento durante 15 min.
- Ressuspender o sedimento de DNA com 50 ul de tampão de eluição (Tris 5 mM, pH 7,5) ou Tris-EDTA pH 8,0 para, pelo menos, 5 min à TA.
- Quantificar o ADN utilizando um ensaio baseado em fluorescência de alta sensibilidade.
- Purificação e concentração de partículas de fagos (modificado de Thurber et al. 19)
Resultados
Microscopia
Amostras de microscopia pode e deve ser analisado imediatamente para garantir que eles tenham a qualidade desejada. Para medir a abundância ea distribuição de tamanho da comunidade bacteriana, lâminas DAPI será examinado por microscopia de epifluorescência (excitação / emissão: 358/461 nm, ver McDole et al 2012). (Figura 6A). Contagem de células e dimensões podem ser coletados por meio de software de imagem (por exemplo, ImagePro ou ImageJ). A partir de volumes de células medidas de comprimento e largura (V) são derivadas, fazendo a suposição de que todas as células têm a forma de cilindros com as tampas hemisféricas, utilizando a seguinte equação:
V = π / 4 × 2 w (L - w / 3)
onde L é o comprimento e w é a largura de cada célula 23,4. A biomassa microbiana pode ser estimada utilizando relações dependentes de tamanho previamente estabelecidos for comunidades microbianas marinhas 24. Geralmente bactérias marinhas variam em comprimento 0,1-4 mm, mas ir até ~ 8 mm em alguns locais.
Enquanto os filtros (0,2 um) corados com DAPI só irá mostrar as bactérias, os filtros utilizados para a mancha SYBR Gold (0,02 mm) contêm bactérias e vírus. Medindo a abundância de vírus segue o mesmo protocolo como para micróbios, no entanto uma excitação de 325-375 nm vai ser utilizado e o máximo de emissão é de 537 nm (Figura 6B).
A fim de gerar dados quantitativa do volume de amostragem pode necessitar de ser ajustado dependendo da abundância viral na amostra original. O volume correto para filtrar é melhor determinado empiricamente para uma dada massa de água. Exemplos de micrografias contendo amostras com diferentes concentrações de vírus encontram-se ilustrados na Figura 7.
Os resultados de estudos anteriores sugerem que vírus para Microberatios (VMR) variam geralmente de 1 a 50 em sistemas aquáticos 25-29, e entre 3 e 20, com uma média de cerca de 6 em sistemas de recifes de coral (Knowles dados não publicados).
Citometria de Fluxo
Além das contagens directas e tamanho estimativa da comunidade microbiana, as avaliações da proporção de autotrófico para micróbios heterotróficas através de citometria de fluxo pode ainda ser extraídos a partir das amostras recolhidas (citometria de fluxo exemplar de saída dada na Figura 8). Para determinar o número de células totais das bactérias, as amostras foram coradas com SYBR Green I e um tubo fotomultiplicador com um filtro de passagem de banda 530/30 é usado para a detecção. Um canal de clorofila (LP volta para espelhos de volta, resultando numa gama de 675-735 nm) e (filtro de passagem de banda 585/42) ficoeriterina é usado para contar a abundância de autotróficos em amostras não coradas. Para determinar a abundância de micróbios heterotróficas, as contagens autotróficas da não-manchado porção são então subtraída a contagem total de SYBR-corados (McDole et al., submetido).
Metagenomics virais
Metagenômica Viral utiliza uma abordagem molecular independente de cultura com a ecologia viral, utilizando as informações total de seqüência genômica para determinar a estrutura e função da comunidade. Metagenômica vem avançando a nossa compreensão da complexidade e diversidade das comunidades virais a nível local 17 a 30 escalas globais. O recente desenvolvimento de uma ampla gama de ferramentas de bioinformática para análise de dados metagenomic virais diminuiu gargalos computacionais, permitindo uma visão mais abrangente da do virosphere através da lente da metagenômica.
Os métodos apresentados aqui realçar o isolamento e enriquecimento de partículas virais a partir de água do mar utilizando uma combinação de pré-filtração com o grande tamanho dos poros da malha de nylon para remover os detritos e material celular, concentração deamostras com TFF (Figura 3) e uma subsequente filtração de 0,45 um para remover as células maiores. Este método produz cerca de um 100x amostra concentrada (Figuras 5B-5E), que nos permite realizar processos a jusante em volumes menores. A fim de evitar o crescimento de quaisquer células microbianas que permanecem no lisado viral e, por conseguinte, muda na abundância viral da amostra durante o longo tempo de trânsito entre a estação de campo e no laboratório, clorofórmio é frequentemente adicionado a uma concentração final de 2% para armazenamento. Após isolamento e purificação de partículas VLP, microscopia de epifluorescência com corantes ácidos nucleicos, tais como SYBR ouro são usados para verificar a presença e pureza das partículas virais (Figuras 5B-5E).
Aqui, apresentamos dois metagenomes virais do recife de coral do Sul Linha Island, especificamente, a Starbuck (STAR7) e Millennium (CAR9; anteriormente conhecido como Caroline) ilhas. Um volume total de 120 L sample água foi coletada de cada local, a uma profundidade de 10 metros e processados como descrito na seção 3.7 e 5.2. As VLP purificadas a partir da centrifugação em gradiente de cloreto de césio foram 3,3 x 10 8 de partículas / ml para CAR9 (Figura 5D) e de 2,9 x 10 9 por ml para partículas STAR7 como foi verificado de acordo com o método descrito no ponto 4 (Figura 5B). A quantidade total de DNA isolado a partir destas amostras foram de cerca de 400 ng, com base na medição Nanodrop. O DNA foi amplificado utilizando polimerase phi29 e sequenciados pela facilidade Ambiental Genomics núcleo em 2011. As características da sequência de dados é apresentada na Tabela 1. Com base em pesquisas de similaridade utilizando bases de dados existentes, a maioria (> 70%) das sequências frequentemente terminou descaracterizados, ou seja, origens e funções desconhecidas. Do mesmo modo, as duas viromes aqui apresentados apresentaram mais de 97% das sequências desconhecidas. Como resultado, não banco de dadosanálise dependente foi utilizado para análise e abriu um novo braço de oportunidade de pesquisa sobre a "matéria escura" em metagenômica virais (Seguritan et al. no prelo).
Microbial Metagenomics
A análise dos metagenomes microbianas permite a caracterização da comunidade microbiana presente ea descrição funcional destas comunidades. Exemplos incluem estimativas da presença de patógenos e virulência 5,22 ou comparações entre as mudanças na estrutura da comunidade microbiana ou nutrientes disponíveis e abundância de espécies e suas vias metabólicas predominantes (Figura 9; Kelly et al 2014)..
Química da Água
Parâmetros de química da água em geral, tem extensa processamento analítico para gerar os dados desejados; As amostras para análise do COD vai ser medido através de alta temperatura de oxidação catalítica 31,32, matéria orgânica particulada (POM), através de espectrometria de massa de razão isotópica acoplado a um analisador elementar 33-35 e nutrientes inorgânicos pela Flow Injection Analysis 36,12,37. Após a conclusão bem sucedida de todas as análises, informação como mostrado na Tabela 1 irá estar disponível para complementar a caracterização da comunidades microbianas e virais. Esta informação pode ser complementada com proporções de isótopos estáveis de carbono e nitrogênio amostras orgânicas (Veja Haas et al. 35) e relações entre autótrofos e heterótrofos (McDole et al. Submetido).
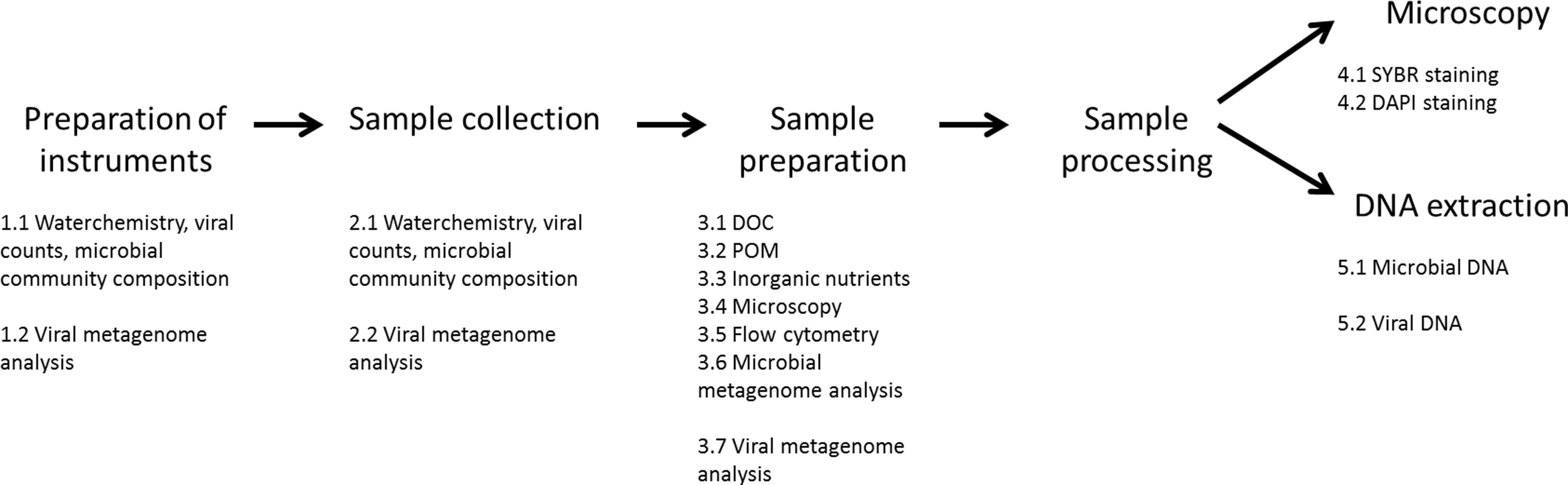
Figura 1. Visão geral dos métodos descritos na seção de protocolo. Por favor clique aqui para ver uma versão maior desta figure.

Figura 2. Exemplo de configuração do processamento de equipamento. A configuração de filtragem introduzido aqui (desenho esquemático esquerda, quadro painel direito real) não é necessariamente obrigatório para gerar amostras, mas vai acelerar o processo de forma significativa. A instalação é constituída por uma placa de apoio, em que as unidades de Hatay Niskin (1) estão montados. Na saída virada para baixo, as unidades de Hatay Niskin estão ligados com tubagem de silicone para os filtros de suportes (2) em-linha. Estes permitem a passagem de água amostrada através do respectivo filtro para os frascos de amostragem HDPE (3) montada logo abaixo. O processo de filtração é acelerada por meio de ar pressurizado (4), e que regulado por um indicador de pressão (5). Uma doca de expansão impede derramar água durante a troca dos frascos de PEAD ou filtração POM. Por favor,clique aqui para ver uma versão maior desta figura.

Água amostra Figura 3. programação filtro de fluxo tangencial (modificado a partir do Thurber et al. 19). Filtrou-se com tela de nylon é bombeado a partir do reservatório (1) através de uma bomba peristáltica (2) a um filtro de fluxo tangencial (3). As amostras de água retorna para o reservatório (1) ao longo da linha de retorno (4), na ausência de pressão de retorno. Quando a pressão de retorno é aplicada através de uma braçadeira de mangueira (5) na linha de retorno, a água passa através do filtro e é descartado ou entregues ao reservatório filtrado (6). Amostra de água é substituído como está concentrada a linha filtrado até toda a água da amostra é concentrado nas linhas de tubagem.
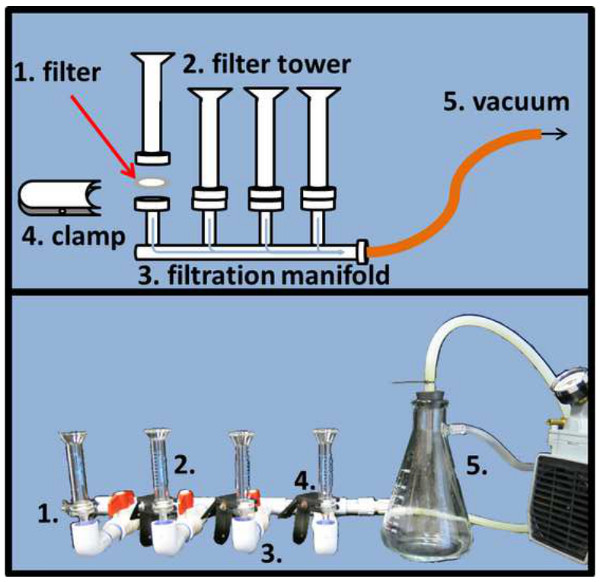
4. Microscópio Figura configuração filtração slide.Para distribuir uniformemente DAPI e SYBR ouro amostras coradas sobre o respectivo filtro de matriz de alumina (1), os filtros necessitam de ser colocado entre a torre de filtro (2) e da haste do colector de filtração e fixada com um grampo (4). Um vácuo regulado de pressão (5) vai acelerar o processo de filtração (top desenho esquemático, painel de fundo a fotografia original). Por favor, clique aqui para ver uma versão maior desta figura.
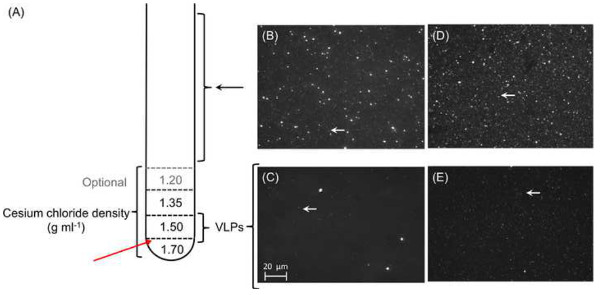
Figura 5. A purificação e concentração de partículas de fago. Um total de 1 ml de cada gradiente de densidade de cloreto de césio é mergulhado em cima uns dos outros antes de levando a amostra pré-tratada (A). Cada camada deve ser marcada no exterior do tubo para facilitar a extracção após ultracentrifugação. Marcas de seta vermelha onde o tubo será perfurada para extrato a fracção de VLP. (BE) Micrografias de concentrados virais: micrografias de epifluorescência 0,45 concentrados virais representativos micrometros de filtrados de STAR7 (B) e CAR9 (D). As grandes partículas, incluindo as células bacterianas eram visíveis a partir dos 0,45 mm a partir de ambos os filtrados-STAR7 (B) e (D) CAR9 amostras, enquanto que apenas as VLP (setas brancas) permanecem após ultracentrifugação de gradiente de cloreto de césio; Estrela 7 (C) e CAR9 (E). Por favor, clique aqui para ver uma versão maior desta figura.
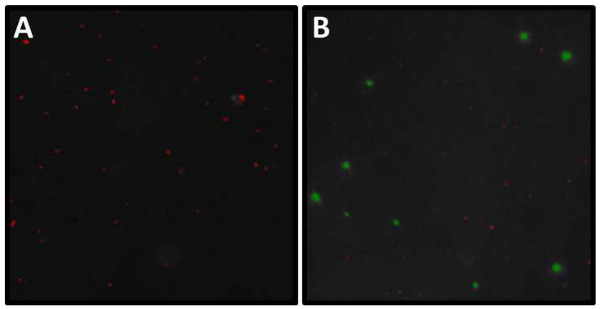
Figura 6. Exemplo de DAPI e análise micrografia SYBR Gold. Tela a partir da análise de (A) e DAPI (B) SYBR ouro coradas exemplo corrediça. (A) Comprimento e largura (mm) das partículas em destaque todas(= Micróbios) pode ser avaliada com a ajuda de um programa de imagem. Note-se a agregação no centro da imagem. Estes agrupamentos terão de ser excluídos da análise subseqüente. (B) Durante vírus análise e bactérias precisam ser finalmente resolvido por limiares em VLP e faixas de tamanho celular (VLP vermelho, verde celular). Note-se que normalmente há alguns VLP tênues Sem categoria pelo respectivo programa de imagem. Estes VLP que aparecem como pontos brancos leves devem ser contados e adicionados aos contagem viral automatizados manualmente. Por favor clique aqui para ver uma versão maior desta figura.

Figura 7. Concentrações de SYBR ouro manchado amostras. Micrografias de um filtro de 0,02 matriz de alumina contendo vários números de SYBR manchado amostras de vírus. O painel A mostra afILTER contendo uma quantidade adequada de água do mar filtrada, enquanto que as concentrações no filtro mostrados em B são muito altos e em C muito baixo para a contagem de confiança. Por favor clique aqui para ver uma versão maior desta figura.
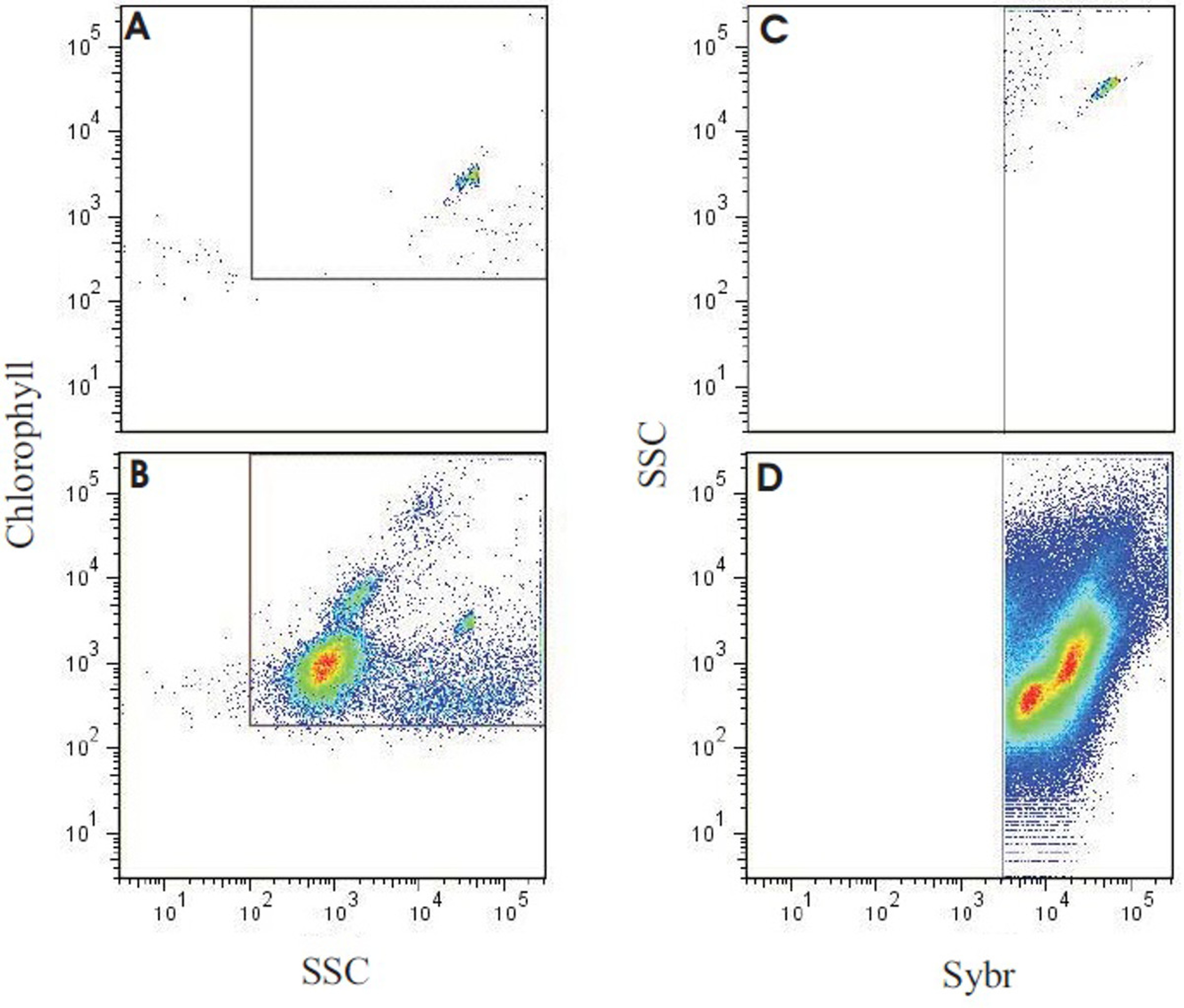
Figura 8. Os resultados de citometria de fluxo de saída da análise de uma amostra de recife-água. O painel (A) mostra a água de grau molecular só com microesferas amarelo-verdes fluorescentes (0,75 um), utilizadas para verificar a fundo mínima com as configurações do instrumento. Painel (B) mostra a saída do recife amostra de água correr com configurações idênticas e layout como em A. Note-se que as contas ainda podem ser vistos no mesmo local, junto com várias populações de autótrofos. (C) A mesma amostra como em (B), usado para fazer-gate o citômetro de fluxo, visando eventos positivos SYBR (autotrófica + contagem de heterotróficos) ea minimização de fundo. (D) A amostra representativa recife-água coradas com SYBR Green I. Usando o gating estabelecido em (C), a contagem total de micróbios foram gerados, e micróbios heterotróficas e autotróficas particionado pela clorofila autofluorescência (ver McDole et al. 4). Por favor, clique aqui para ver uma versão maior desta figura.
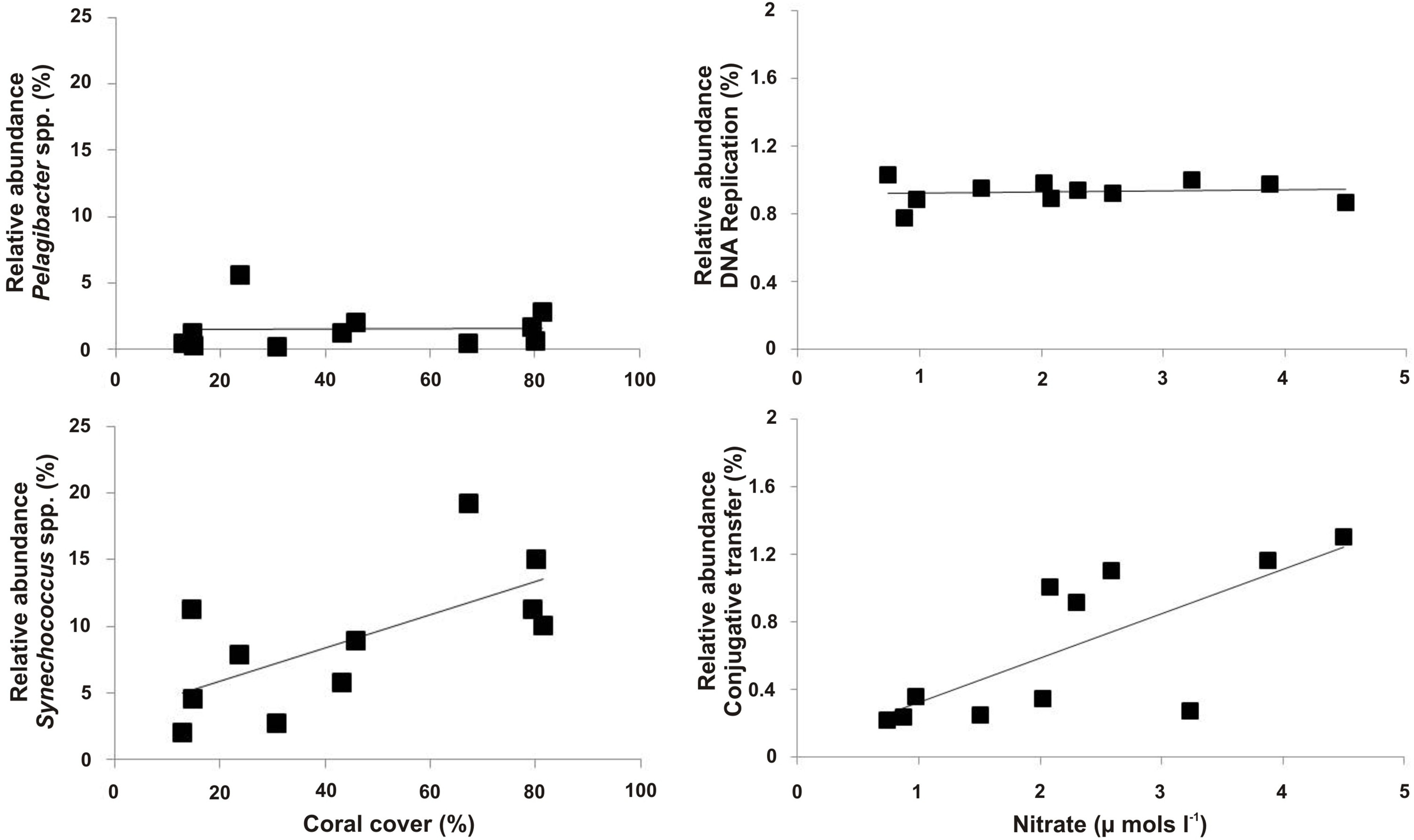
Figura 9. Exemplo de dados metagenome microbianas. Padrões entre os dados da seqüência metagenomic microbianas e do site variáveis dependentes. Aqui, a abundância relativa de Synechococcus spp. foi positivamente correlacionada com a tampa por cento do coral, enquanto Pelagibacter spp. não foi (painéis da esquerda). A via metabólica para a transferência conjugative foi positivamente correlacionada com as concentrações de nitrato, enquanto que a via metabólica para o reparo do DNA foi consistente entre todos os metagenomes (painéis à direita). Por favor, clique aqui para ver uma versão maior desta figura.
| Estrela 7 | CAR9 | |
| Número total de Lê | 939.311 | 591.600 |
| Número de pré-processados Lê | 579.664 | 360.246 |
| A média de Sequence Length (pb) | 395 ± 131 | 451 ± 159 |
| Sequências anotados (%) | 42,218 (7,3%) | 9117 (2,5%) |
| Desconhecido (%) | 537446 (92,7%) | 351129 (97,5%) |
Tabela 1. Características dos dados de dois viromes gerados a partir das Ilhas da Linha do Sul, Starbuck e Millennium. O grau de anotação é baseada em BLAT usando o servidor MG-RAST.
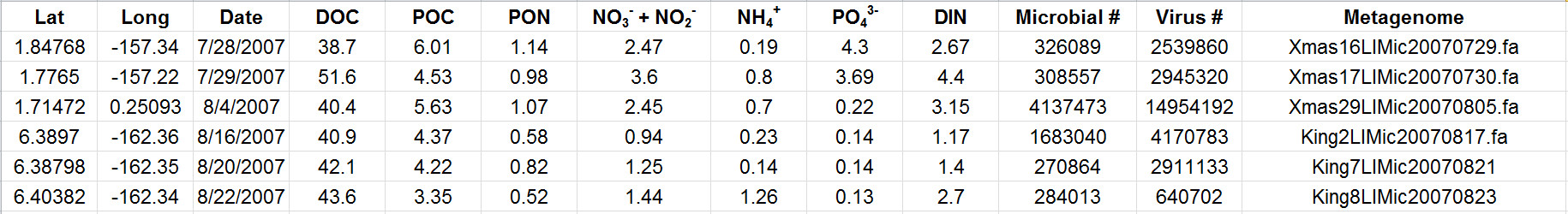
Tabela 2. Os resultados representativos mostrando dados de vários locais durante uma expedição. Os parâmetros aqui analisados incluem medições química da água emparelhado com microbiana e abundâncias virais. A tabela contém ainda os nomes dos arquivos referentes aos respectivos dados metagenomic para cada local de amostragem. As concentrações de carbono e nutrientes inorgânicos são dadas em nmol / L. Por favorclique aqui para ver uma versão maior desta tabela.
Discussão
Os métodos apresentados aqui vai fornecer uma ferramenta para gerar uma avaliação abrangente da dinâmica viral e microbianas em ecossistemas aquáticos. Eles são direcionados para quantificar o estoque de pé de recursos orgânicos e inorgânicos disponíveis para comunidades microbianas e caracterizar a composição da atual comunidade viral e microbiana. Ao lado de quantificação viral abundância ea abundância e biomassa microbiana, informações seqüência genômica revela sua estrutura e função da comunidade. Todos os métodos têm sido desenvolvidos ou modificados para permitir a aplicação de campo em locais remotos. No entanto, a falta de definições controladas de laboratório inerentemente cria algum potencial para erros. Aqui vamos discutir algumas ressalvas associadas a cada um dos parâmetros avaliados. Próximo ao potencial de contaminação durante a manipulação de amostra alguns dos parâmetros também produzir potencial de erro no processo analítico.
Carbono Orgânico Dissolvido
Carbono contaminação pode ocorrer durante os procedimentos de amostragem (amostragem a jusante, ou na vizinhança de um barco ou um mergulhador), durante a preparação da amostra (a contaminação do equipamento ou manipulação inadvertida), e, mesmo durante o armazenamento da amostra (no congelador outras amostras contendo orgânica Os solventes voláteis /). Para evitar as diversas fontes de contaminação conduta como alguns passos de manuseio quanto possível, uma vez que cada deslocalização amostra representa uma fonte adicional de contaminação. A amostra não deve entrar em contato com qualquer item não ácido lavado ou queimado. Finalmente, designa um congelador de armazenamento exclusivamente para amostras que não contenham substâncias orgânicas voláteis vai garantir a integridade das amostras durante os períodos de armazenagem mais longos. Se as amostras não pode ser mantido congelado durante um período prolongado de viagens de amostras DOC acidificação com HCl concentrado (como descrito 38-41) pode ser uma alternativa aplicável. Para verificar a precisão da medição materiais de referência consenso DOC deve ser usado regcularmente durante a medição DOC é executado. Especialmente água do mar profundo (> 2000 m) referências são importantes na avaliação do desempenho da máquina analítica, pois é muito estável em seu conteúdo DOC 42.
Matéria orgânica particulada
Além de evitar a contaminação de carbono orgânico, a amostragem POM requer uma atenção especial para garantir a precisão do volume filtrado. Se o volume de 500 ml orientada deve desviar-se por qualquer motivo, este potencial deve ser anotado para relacionar a quantidade de POM capturado no filtro para o filtrado da qual foi derivada.
Nutrientes inorgânicos
Tal como acontece com a amostragem de carbono orgânico, deve ser dada atenção à possível contaminação da amostra de nutrientes gerados por várias fontes, como barcos ou descargas de águas residuais 12. Filtros usados para a amostragem de carbono orgânico com um tamanho de poros nominais de 0,8 mm, não podem excluir toda a biomassa microbiana e shodia, por conseguinte, ser alterado para filtros de 0,2 um para este passo de filtração. Além disso, os limites de detecção constituem um problema com as amostras de nutrientes; especialmente na água de superfície oligotrófico em torno de muitos locais de recifes de coral (por exemplo, Cotner et al. 43). Os limites de detecção são mais ou menos em torno de 0,1, 0,2, e 0,1 mmol / L para o de amónio, nitrato e nitrito, e orto-fosfato, respectivamente (ver 36,12,37)
Microscopia
Ao lado de variações na concentração de análise de imagem a partir de amostras de lâminas de microscopia, permite maior potencial de erros. Por exemplo, as imagens podem ficar fora de foco em algumas áreas do campo de visão, e no foco em outros. Como um elevado grau de pureza do filtro matriz de alumina é um tipo de filtro muito rígida, quaisquer detritos sob o filtro pode fazer com que todo o filtro para se sentar em um ângulo para a corrediça. Como resultado, as imagens podem ser consistentemente fora de foco em várias partes do campo de visão para um dado filtro, reindependente- de alinhamento microscópio. Remontagem filtros, assegurando o lado de baixo do filtro está limpo, pode melhorar esta se for observada. Além disso, em alguns casos, pode haver dois planos focais em que os vírus e micróbios podem ser encontrados. Este é o resultado de se tornarem manchadas objectos separado do filtro quando da montagem, e que flutuam até à parte de baixo da lâmina de cobertura que pode tornar as contagens não confiável. Por isso recomenda-se sempre de verificar a qualidade das amostras durante o processo.
Citometria de Fluxo
Tal como acontece com as amostras de microscopia, pode haver diferenças entre amostras de citometria de fluxo, que exigem ajustamentos do processo analítico que ocorre naturalmente. Por exemplo, dependendo da sensibilidade do instrumento, fracamente as células fluorescentes Prochlorococcus pode ser inferior ao nível de ruído e não irá ser quantificada. Grânulos servir como uma amostra de controlo interno (por exemplo, para confirmar que resp do instrumentoonse de sinais fluorescentes é consistente do dia a dia (e permanece estável durante a própria corrida). Se adicionado à amostra em concentrações conhecidas, que também pode servir como um controlo interno para verificar o volume da amostra de execução. No entanto, recomenda-se rodar sempre uma alíquota de um "padrão" de amostra de água do mar previamente congelada que é descongelada e coradas juntamente com as amostras de interesse para proporcionar um controle biológico adicional para manipulação de amostra entre 96 poços é executado placa. Dependendo da localização, as amostras de água do mar pode parecer muito diferente uma da outra. Classificando populações "representativas" e, em seguida, visualização sob um microscópio de epifluorescência (EM) pode ser uma boa maneira de verificar rapidamente as populações de fitoplâncton como quer Synechococcus sp. Ou eucariotas fotossintéticos. Prochlorococcus células geralmente não são visíveis sob EM porque eles desaparecem muito rápido. Além de clorofila a, Synechococcus sp., Também contêm o phphycoeurythrin otopigment (PE), que emite luz em comprimentos de onda mais curtos (540-630 nm) do que a clorofila A (660-700 nm). Quando as células Synechococcus são excitados com luz azul / verde (470-490 nm), eles vão aparecer de ouro, se visto sob um filtro de emissão que remove todos os comprimentos de onda de luz abaixo de 510 nm. Por comparação, a fracção eucariótica vai ficar vermelha, quando excitado com o mesmo comprimento de onda da luz.
Metagenomics virais
É importante notar que o processo de armazenamento e concentração de VLP influencia a comunidade recuperado. Perda de partículas virais podem ocorrer em todos os passos do processo, e, assim, a selecção de protocolos de purificação e de enriquecimento virais podem em última análise afectar a composição taxonômica observada e diversidade dos metagenomes virais resultantes 44,45,18. A concentração das amostras pode ser feita utilizando TFF 19 ou 20 à base de floculação química. Na abordagem baseada em química, posittivamente carregado iões de ferro ligar as partículas virais naturalmente carregados negativamente que formam grandes (> 8 mm) complexos de ferro-viral que floculam para fora da solução e pode ser recuperado utilizando filtros de 8 pm. Subsequentemente, o ferro é quelado fora das partículas virais e re-dissolvido através de magnésio, ácido ascórbico, e EDTA, deixando partículas virais concentrados para a extracção de ácido nucleico 20. Depois de comparar estes dois métodos atuais, concluímos que o método de cloreto de ferro é menos demorado do que a concentração viral TFF, mas pode render algumas ressalvas. Descobrimos que a dissociação e a dissolução do ferro-viral requer um duplo solução mais concentrada de magnésio-ácido ascórbico-EDTA 20 do que o relatado para a dissolução, a fim de proceder antes degrada os ácidos ascórbico. Além deste problema, o protocolo purifica partículas virais de fraccionamento por tamanho por si só, a remoção de contaminantes microbianos usando filtração de 0,2 um. Vírus grandes não passam através do filter (por exemplo, Yang et al. 46) e, portanto, não vai ser detectado, no metagenome, enquanto que algumas bactérias fazer (McDole, dados não publicados). Isso resulta em contaminação bacteriana ao discriminar contra vírus grandes.
Esse problema específico no entanto, também é relevante para a remoção de micróbios em ligação com a concentração de TFF. Como o tratamento de clorofórmio foi mostrado para remover vírus sensível ao clorofórmio com membranas lipídicas externas 47 alguns estudos sugerem que 0,22 um de filtração pode ser utilizada para remover a maioria de bactérias. Também deve ser notado que o método apresentado tem como alvo principalmente bacteriófago, o transportador mais abundante de material genético em ambientes marinhos. Porque fagos são geralmente pensados para não contêm comumente lipídios 48 eles são mais resistentes ao tratamento clorofórmio. Para o nosso conhecimento de um método "catch-all" não existe até o momento. O método aqui apresentado é uma adaptação da UOexperiência de campo r ao longo dos últimos 10 anos em vários ambientes marinhos medem tropical para sistemas árticas.
Microbial Metagenomics
A construção de bibliotecas metagenomic de micróbios (por exemplo, bactérias e archaea) é mais simples do que metagenomes virais, uma vez que é mais fácil de gerar as concentrações mínimas de ADN necessárias para a preparação da biblioteca. Amplificação espingarda bibliotecas Linker (LASLs) amplificação 17,49,50 e todo o genoma com base em múltiplos de amplificação de deslocamento (MDA) são os dois métodos mais comumente usados para gerar DNA suficiente para o seqüenciamento. O uso de MDA para obter concentrações mais elevadas de ADN em metagenomes microbianas é por vezes necessário. Este, porém, pode causar artefatos nos dados de seqüência, como a overamplification de minicírculos de dinoflagelados. MDA são conhecidos métodos para amplificar preferencialmente ADN de cadeia simples e circular, resultando em resultados não-quantitativos 51,52. Os LASLs otimizados aproximar 50 pode, assim, serve como uma alternativa melhor na amplificação tanto microbiana e DNA viral metagenômica para o seqüenciamento. É importante notar que a abordagem LASLs tem múltiplos passos, requer equipamento sofisticado, e está limitado aos modelos de dsDNA. Além disso, o estojo de extracção de ADN de tecidos tem sido mostrado para extrair DNA de ambos filos negativo Gram positivas e Gram, mas não foi verificada a taxa microbiana lisar eficazmente recalcitrante ou suas estruturas associadas (por exemplo, certos archaea, endosporos, ou esporos de fungos) . Por conseguinte, pode ser necessário um passo de batimento talão para obter o DNA de certos micróbios.
Estas avaliações abrangentes resultará em uma melhor compreensão do funcionamento dos ecossistemas viral e microbiana. Embora poucos estudos têm empreendido um esforço para avaliar todos os parâmetros propostos, muitos têm vindo a investigar os selecionados. Nelson et al. 53 sugeriram uma ligação entreen habitats de recife específico e esgotamento em ambas as concentrações de DOC e bacterioplâncton relativos às águas offshore. Essas mudanças de concentração foram acompanhadas por diferenciações distintos de bacterioplâncton. Dinsdale et al. 5 mostraram que o aumento impacto antropogénico foi acompanhada por 10x maior abundância de células microbianas e partículas semelhantes a vírus. Comunidades microbianas em águas marinhas vizinhas ilhas habitadas também teve frações maiores de heterotróficos e potenciais patógenos 5. Finalmente McDole et al. 4 mostraram, ao introduzir a pontuação microbialization, que as atividades humanas estão mudando energia para os micróbios, em detrimento dos macróbios. Estes estudos, com foco em parâmetros microbiológicos e químicos da água específicos, fornecer novos insights sobre a estrutura bioquímica dos ecossistemas marinhos. Combinando dados tradicionais de esforços de monitoramento de ecossistemas - como valores de temperatura e pH, biomassa de peixes e diversity, cobertura bentônica, ou exposição ao impacto humano - com a química da água e avaliações microbianas, provavelmente resultará em mais sensíveis esforços de monitoramento ambiental, o que pode levar a esforços de conservação, mais especificamente direcionados.
Divulgações
The authors have nothing to disclose.
Agradecimentos
We thank Elisha Wood-Charlson, Jackie Mueller, Karen Weynberg, and Kathy Morrow for their help and constructive input on this manuscript. We also thank the crew and captain of the Hanse explorer which provided us with a perfect working environment to conduct large parts of this research. Further we would like to thank the three anonymous reviewers for their time and helpful comments to improve the manuscript. This work was funded by the NSF Dimensions of Biodiversity and PIRE awards DEB-1046413 and OISE/IIA-1243541, respectively, the Gordon and Betty Moore Foundation, Investigator Award 3781, and the CIFAR Integrated Microbial Diversity Fellowship IMB-ROHW-141679, all to FR.
Materiais
| Name | Company | Catalog Number | Comments |
| Niskin collection and processing units (Hatay Niskins) | Figure 1 | ||
| Filtration setup | Figure 1 | ||
| 32-36% Hydrochloric acid (HCl) | Fisher Sci | A508-212 | |
| High-density polyethylene (HDPE) container | e.g., 5 Gallon HDPE UN Certified Pail with Snap-On Lid | ||
| Combustion oven (550 °C) | |||
| 60 ml HDPE vials | Fisher Sci | 02-896-2B | |
| 25 mm GF/F filter | Fisher Sci | 09-874-64 | |
| Forceps | Fisher Sci | XX62-000-06 | |
| Sterile, non-powdered gloves | Fisher Sci | 19-170-010B | |
| Bilge pump | e.g., Thirsty Mate 124PF | ||
| 20 L collapsible low-density polyethylene carboy | e.g., Reliance Fold A Carrier II | ||
| Tangential flow filter (TFF) ultrafiltration hollow fiber cartridges | Amersham | CFP-2-E-9A | |
| Platinum-cured silicon tubing for TFF | Cole Parmer | 96440-82 | |
| Hose clamps | Cole Parmer | P-68007-23 | |
| Peristaltic pump | Cole Parmer | EW-77410-10 | |
| Sodium hydroxide (NaOH) solution | Electron Microscopy Sciences | 21162 | |
| 0.2 µm Polycarbonate track-etched membrane filter | Fisher Sci | 09-300-61 | |
| 8.0 µm Polycarbonate track-etched membrane filter | Fisher Sci | 09-300-57 | |
| 0.22 µm Polyvinylidene difluoride cylindrical filter | Fisher Sci | SVGP01050 | |
| 0.45 µm Polyvinylidene difluoride cylindrical filter | Fisher Sci | SVHV010RS | |
| Synthetic nylon mesh | Sefar | 03-125-37 | |
| 20 ml Plastic scintillation bottle | Fisher Sci | 03-341-72C | |
| Clean bucket (10-20 L) | |||
| 32% Paraformaldehyde | Electron Microscopy Science | 15714 | |
| 25% Glutaraldehyde | Electron Microscopy Science | 16220 | |
| Liquid nitrogen | |||
| 0.02 µm High-purity alumina matrix filter with annular polypropylene support ring | Fisher Sci | 6809-6002 | |
| 0.2 µm High-purity alumina matrix filter with annular polypropylene support ring | Fisher Sci | 6809-6022 | |
| 10,000x SYBR Gold nucleic acid stain | Invitrogen | S11494 | |
| DAPI solution 25 µg/ml | Invitrogen | D1306 | |
| Molecular grade water | sigma aldrich | W-4502 | |
| Ascorbic acid | Fisher Sci | BP351-500 | |
| Phosphate Buffered Saline (PBS) | Fisher Sci | BP399-500 | |
| Glycerol | Fisher Sci | G31-500 | |
| Microscope slide | Fisher Sci | 12-550-123 | |
| Coverslip | Fisher Sci | 12-548-5M | |
| Delicate task wipe | Fisher Sci | 06-666A | |
| Slide station with vacuum pump | Figure 3 | ||
| DNA extraction kit | Macherey-Nagel | 740952.1 | Nucleospin tissue kit |
| Proteinase K (20 mg/ml) | Lifetechnologies | AM2546 | |
| 200-proof Ethanol | Electron Microscopy Sciences | 15058 | |
| Red cap: Male Luer with Lock Ring x Female Luer Coupler | Cole Parmer | EW-45503-88 | |
| Cesium chloride (CsCl) | Fisher Sci | bp1595-500 | |
| 60 ml Syringe | Fisher Sci | 13-6898 | |
| 0.02 µm Alumina matrix disposable syringe filter | Fisher Sci | O992613 | |
| Ultra-clear centrifuge tubes | Beckman Coulter | 344059 | |
| Ultra-swinging bucket rotor | Beckman Coulter | 333790 | SW41 Ti Rotor |
| DNase I (100 units/µl) | Calbiochem | 260913 | |
| 0.5 M EDTA | Fisher Sci | BP2482-500 | |
| Formamide | Fisher Sci | BP228-100 | |
| Glycogen | sigma aldrich | G-1767 | |
| 1x Tris-EDTA (TE) pH 8.0 | Fisher Sci | BP2473-500 | |
| Sodium Dodecyl Sulfate (SDS) | Fisher Sci | 02674-25 | |
| 20 μg/ml Proteinase K | Fisher Sci | BP1700-500 | |
| Chloroform | Fisher Sci | BP1145-1 | |
| Phenol:Chloroform:Isoamyl alcohol 25:24:1 | sigma aldrich | p3803 | |
| Isopropanol | Fisher Sci | BP2618-500 | |
| 50 ml Oak Ridge tube | Fisher Sci | 05-562-16B |
Referências
- Fuhrman, J. A. Marine viruses and their biogeochemical and ecological effects. Nature. 399 (6736), 541-548 (1999).
- Azam, F., Malfatti, F. Microbial structuring of marine ecosystems. Nature Reviews Microbiology. 5 (10), 782-791 (2007).
- Huber, J. A., et al. Microbial population structures in the deep marine biosphere. Science. 318 (5847), 97-100 (2007).
- McDole, T., et al. Assessing coral reefs on a Pacific-wide scale using the microbialization score. PloS ONE. 7 (9), (2012).
- Dinsdale, E. A., et al. Microbial ecology of four coral atolls in the Northern Line Islands. PloS ONE. 3 (2), (2008).
- Nelson, C. E., et al. Coral and macroalgal exudates vary in neutral sugar composition and differentially enrich reef bacterioplankton lineages. The ISME Journal. 7 (5), 962-979 (2013).
- Haas, A. F., et al. Influence of coral and algal exudates on microbially mediated reef metabolism. PeerJ. 1, (2013).
- Smith, J. E., et al. Indirect effects of algae on coral: algae‐mediated, microbe‐induced coral mortality. Ecology Letters. 9 (7), 835-845 (2006).
- Burdige, D. J., Martens, C. S. Biogeochemical cycling in an organic-rich coastal marine basin: 10. The role of amino acids in sedimentary carbon and nitrogen cycling. Geochimica et Cosmochimica Acta. 52 (6), 1571-1584 (1988).
- Capone, D. G., Kiene, R. P. Comparison of microbial dynamics in marine and freshwater sediments: Contrasts in anaerobic carbon catabolism. Limnology and Oceanography. 33 (4), 725-749 (1988).
- Krom, M. D., Sholkovitz, E. R. Nature and reactions of dissolved organic matter in the interstitial waters of marine sediments. Geochimica et Cosmochimica Acta. 41 (11), 1565-1574 (1977).
- Grasshoff, K. Methods of sea-water analysis. Verlag Chemie. , Weinheim and New York. (1976).
- Strickland, J. D. Practical handbook of seawater analysis. Bulletin Fisheries Research Board of Canada. 167, (1968).
- Sharp, J. H., et al. Analyses of dissolved organic carbon in seawater: the JGOFS EqPac methods comparison. Marine Chemistry. 48 (2), 91-108 (1995).
- DeLong, E. F. Microbial population genomics and ecology. Current Opinion in Microbiology. 5 (5), 520-524 (2002).
- Hugenholz, P., Goebel, B. M., Pace, N. R. Impact of Culture-Independent Studies on the Emerging Phylogenetic View of Bacterial Diversity. Journal of Bacteriology. 180 (18), 4765-4774 (1998).
- Breitbart, M., et al. Genomic analysis of uncultured marine viral communities. Proceedings of the National Academy of Sciences. 99 (22), 14250-14255 (2002).
- Willner, D., Hugenholtz, P. From deep sequencing to viral tagging: Recent advances in viral metagenomics. BioEssays. 35, 436-442 (2013).
- Thurber, R. V., Haynes, M., Breitbart, M., Wegley, L., Rohwer, F. Laboratory procedures to generate viral metagenomes. Nature Protocols. 4, 470-483 (2009).
- John, S. G., et al. A simple and efficient method for concentration of ocean viruses by chemical flocculation. Environmental Microbiology Reports. 3, 195-202 (2011).
- Lim, Y. W., et al. The Microbiome and Virome of CF. Pediatric Pulmonology. 47, 321-322 (2012).
- Kelly, L. W., et al. Black reefs: iron-induced phase shifts on coral reefs. The ISME Journal. 6 (3), 638-649 (2012).
- Bjornsen, P. K. Automatic determination of bacterioplankton biomass by image analysis. Applied Environmental Microbiology. 51, 1199 (1986).
- Simon, M., Azam, F. Protein content and protein synthesis rates of planktonic marine bacteria. Marine Ecology Progress Series. 51 (3), 201-213 (1989).
- Hara, S., Koike, I., Terauchi, K., Kamiya, H., Tanoue, E. Abundance of viruses in deep oceanic waters. Marine Ecology Progress Series. 145 (1), 269-277 (1996).
- Wommack, K. E., Colwell, R. R. Virioplankton: viruses in aquatic ecosystems. Microbiology and Molecular Biology Reviews. 64 (1), 69-114 (2000).
- Weinbauer, M. G., Brettar, I., Hofle, M. G. Lysogeny and virus-induced mortality of bacterioplankton in surface, deep, and anoxic marine waters. Limnology and Oceanography. 48 (4), 1457-1465 (2003).
- Clasen, J. L., Brigden, S. M., Payet, J. P., Suttle, C. A. Evidence that viral abundance across oceans and lakes is driven by different biological factors. Freshwater Biology. 53 (6), 1090-1100 (2008).
- Parada, V., Baudoux, A. C., Sintes, E., Weinbauer, M. G., Herndl, G. J. Dynamics and diversity of newly produced virioplankton in the North Sea. The ISME Journal. 2 (9), 924-936 (2008).
- Angly, F. E., et al. The marine viromes of four oceanic regions. PLoS Biology. 4 (11), (2006).
- Hansell, D. A., Carlson, C. A. Deep ocean gradients in dissolved organic carbon concentrations. Nature. 263 (266), (1998).
- Carlson, C. A., et al. Dissolved organic carbon export and subsequent remineralization in the mesopelagic and bathypelagic realms of the North Atlantic basin. Deep Sea Research Part II: Topical Studies in Oceanography. 57 (16), 1433-1445 (2010).
- Sharp, J. H. Improved analysis for particulate organic carbon and nitrogen from seawater. Limnology and Oceanography. 19 (6), 984-989 (1974).
- Ehrhardt, M., Koeve, W. Determination of particulate organic carbon and nitrogen. Methods of Seawater Analysis. Third Edition, 437-444 (2007).
- Haas, A. F., et al. Organic matter release by coral reef associated benthic algae in the Northern Red Sea. Journal of Experimental Marine Biology and Ecology. 389 (1), 53-60 (2010).
- Guildford, S. J., Hecky, R. E. Total nitrogen, total phosphorus, and nutrient limitation in lakes and oceans: Is there a common relationship. Limnology and Oceanography. 45 (6), 1213-1223 (2000).
- Allison, S. D., Chao, Y., Farrara, J. D., Hatosy, S., Martiny, A. C. Fine-scale temporal variation in marine extracellular enzymes of coastal southern California. Frontiers in Microbiology. 3, (2012).
- Van Duyl, F. C., Gast, G. J. Linkage of small-scale spatial variations in DOC, inorganic nutrients and bacterioplankton growth with different coral reef water types. Aquatic Microbial Ecology. 24 (1), 17-26 (2001).
- De Goeij, J. M., Van Duyl, F. C. Coral cavities are sinks of dissolved organic carbon (DOC). Limnology and Oceanography. 52 (6), 2608-2617 (2007).
- De Goeij, J. M., et al. Major bulk dissolved organic carbon (DOC) removal by encrusting coral reef cavity sponges. Marine Ecology Progress Series. 357, 139-151 (2008).
- Mueller, B., et al. Natural diet of coral-excavating sponges consists mainly of dissolved organic carbon (DOC). PLoS ONE. 9 (2), (2014).
- Carlson, C. A., Ducklow, H. W., Hansel, D. A. Organic carbon partitioning during spring phytoplankton blooms in the Ross Sea polynya and the Sargasso Sea. Oceanography. 43 (3), 375-386 (1998).
- Cotner, J. B., et al. Nutrient limitation of heterotrophic bacteria in Florida Bay. Estuaries. 23 (5), 611-620 (2000).
- Willner, D., et al. Metagenomic detection of phage-encoded platelet-binding factors in the human oral cavity. Proceedings of the National Academy of Science USA. 108 (1), 4547-4553 (2011).
- Hall, R. J., et al. Evaluation of rapid and simple techniques for the enrichment of viruses prior to metagenomic virus discovery. Journal of Virological Methods. 195, 194-204 (2013).
- Yang, S. Y., et al. Nanoporous membranes with ultrahigh selectivity and flux for the filtration of viruses. Advanced Materials. 18 (6), 709-712 (2006).
- Feldman, H. A., Wang, S. S. Sensitivity of various viruses to chloroform. Proceedings of the Society for Experimental Biology and Medicine. 106 (4), 736-738 (1961).
- Ackermann, H. W. The Bacteriophages., Second Edition. Calendar, R. , Oxford University Press. New York. 8-16 (2006).
- Henn, M. R., et al. Analysis of High-Throughput Sequencing and Annotation Strategies for Phage Genomes. PLoS ONE. 5 (2), (2010).
- Duhaime, M. B., Deng, L., Poulos, B. T., Sullivan, M. B. Towards quantitative metagenomics of wild viruses and other ultra-low concentration DNA samples: a rigorous assessment and optimization of the linker amplification method. Environmental Microbiology. 14 (9), 2526-2537 (2012).
- Yilmaz, S., Allgaier, M., Hugenholtz, P. Multiple displacement amplification compromises quantitative analysis of metagenomes. Nature Methods. 7 (12), 943-944 (2010).
- Kim, K. H., Bae, J. W. Amplification Methods Bias Metagenomic Libraries of Uncultured Single-Stranded and Double-Stranded DNA Viruses. Applied Environmental Microbiology. 77 (21), 7663-7668 (2011).
- Nelson, C. E., Alldredge, A. L., McCliment, E. A., Amaral-Zettler, L. A., Carlson, C. A. Depleted dissolved organic carbon and distinct bacterial communities in the water column of a rapid-flushing coral reef ecosystem. The ISME Journal. 5 (8), 1374-1387 (2011).
- Kelly, L. W., et al. Local genomic adaptation of coral reef-associated microbiomes to gradients of natural variability and anthropogenic stressors. Proceedings of the National Academy of Sciences. 111 (28), 10227-10232 (2014).
- Lim, Y. W., Haynes, M., Furlan, M., Robertson, C. E., Harris, J. K., Rohwer, F. Purifying the Impure: Sequencing Metagenomes and Metatranscriptomes from Complex Animal-associated Samples. J. Vis. Exp. , (2014).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados