Method Article
Démêler les joueurs invisibles dans l'océan - un guide de terrain à la chimie de l'eau et de microbiologie marine
Dans cet article
Résumé
Here, we present a comprehensive protocol to assess the organic and inorganic nutrient availability and the abundance and structure of microbial and viral communities in remote marine environments.
Résumé
Ici, nous introduisons une série de protocoles de recherche et soigneusement testés et normalisés conçus pour être utilisés dans des environnements marins éloignés. Les protocoles d'échantillonnage comprennent l'évaluation des ressources à la disposition de la communauté microbienne (carbone organique dissous, la matière organique particulaire, les nutriments inorganiques), et une description complète des communautés virales et bactériennes (via compte virales et microbiennes directs, le dénombrement des microbes autofluorescentes, et construction de métagénomes virales et microbiennes). Nous utilisons une combinaison de méthodes, qui représentent un champ dispersé des disciplines scientifiques comprenant des protocoles déjà établis et certaines des techniques les plus récentes développées. Techniques de séquençage métagénomique surtout utilisés pour la caractérisation virale et bactérienne communautaire, ont été mis en place que depuis quelques années, et sont donc encore soumis à l'amélioration constante. Cela a conduit à une variété de méthodes d'échantillonnage et de traitement de l'échantillon CurrenTLY en utilisation. L'ensemble des méthodes présentées ici fournit une mise à jour approche pour collecter et traiter les échantillons environnementaux. Les paramètres adressés à ces protocoles donnent le minimum sur les informations essentielles pour caractériser et comprendre les mécanismes sous-jacents de la dynamique des communautés virales et microbiennes. Il donne des directives faciles à suivre pour mener des enquêtes complètes et décrit les étapes critiques et les mises en garde potentiels pertinents pour chaque technique.
Introduction
Les écosystèmes marins sont soumis à une large gamme de perturbations qui entraînent des changements de la disponibilité des nutriments à la biomasse des grands prédateurs. Au cours de la dernière décennie, plusieurs études ont démontré l'importance des communautés microbiennes dans les écosystèmes marins 1-4. Il est évident que les changements dans la communauté bactérienne et virale sont étroitement associés à la dégradation globale de l'environnement marin 5. Ces changements peuvent être facilitées par la géochimie altérée dans la colonne d'eau tels que la disponibilité de l'oxygène ou la reminéralisation du carbone 6,7. En raison de l'interdépendance entre la macro-organismes, la chimie de l'eau et activité microbienne des boucles de rétroaction peut accélérer la vitesse de dégradation de l'écosystème 8.
Dans les années 1970 et 80, plusieurs tentatives ont été faites pour démêler les cycles biogéochimiques dans les écosystèmes marins 9-11. L'un des principaux défis pour ces premières études était le manquedes approches normalisées pour mesurer les paramètres biogéochimiques évalués. Bien qu'il y avait des protocoles disponibles, principalement sur les éléments nutritifs de l'eau de mer 12,13, l'évaluation de la dynamique du carbone et la structure de la communauté microbienne ont été limitées par les outils et méthodes disponibles. Avec la comparaison des méthodes JGOFS EqPac, Sharp et al. 14 a suggéré pour la première fois un protocole fiable et standardisée pour les évaluations carbone organique dissous (COD) de concentration. Au début des années 2000 les défis analytiques pour déterminer les ressources disponibles pour la communauté microbienne avait donc été en grande partie résolus et les approches pour caractériser les communautés microbiennes dans les milieux de culture contrôlées ont été mis en place (voir DeLong 15). Les méthodes de séquençage traditionnelles à cette époque largement appuyée sur les cultures clonales cultivées. Tôt le séquençage des gènes de l'environnement d'échantillons naturels a toutefois révélé que la majorité de la biodiversité microbienne a été manqué par Cultivméthodes basées ation-16. En 2002, Breitbart et al. 17 Séquençage du fusil de chasse pour la première fois pour décrire la communauté virale dans les échantillons d'eau de mer de l'environnement d'établir une méthode de séquençage du génome entier des communautés virales marins incultes.
Dans les évaluations actuelles de micro, les virus restent particulièrement sous-étudié, car la plupart sont difficiles à cultiver et ils manquent un marqueur universellement conservées comme la ARN ribosomique 16S (ARNr) des gènes habituellement utilisés pour évaluer la diversité et de la communauté profilage. L'approche de séquençage métagénomique offre une alternative aux méthodes de culture-dépendante et gène-cible pour l'analyse des communautés virales complexes. Alors que les premiers métagénomes virales générées à partir de l'eau de mer ont été séquences en utilisant le séquençage Sanger 17, le développement des technologies de séquençage de prochaine génération et l'amélioration des techniques de biologie moléculaire a conduit à une augmentation rapide dans les études de métagénomique 18 . Le flux de travail de laboratoire en cours pour la métagénomique virales implique la séparation, l'enrichissement et / ou de la concentration de particules virales, puis de l'acide nucléique (ADN ou ARN) extraction, la préparation bibliothèque, et le séquençage 19-21.
Pour approfondir les connaissances actuelles sur virale, microbienne et le fonctionnement biochimique dans les écosystèmes aquatiques, en comparant les ensembles de données dans différents systèmes dans le monde entier est essentiel. Cependant, les protocoles établis ont été largement développée pour les environnements proches du rivage avec des installations de laboratoire facilement accessibles. Ainsi, l'absence de méthodes de terrain standardisés empêche ces comparaisons entre les écosystèmes. Ici, nous introduisons testé et adaptations de l'état des protocoles de recherche d'art bien standardisée pour utilisation dans des endroits éloignés de terrain. Ces méthodes ont fait leurs preuves avec succès dans plusieurs études couvrant la dernière décennie 5,22 et sont actuellement utilisées par les différents laboratoires marins ainsi que sur la NOAAÉvaluation de corail et Programme de surveillance (RAMP) dans tout l'océan Pacifique 4. Les protocoles d'échantillonnage comprennent des paramètres de l'eau de la chimie (concentrations organiques et inorganiques carbone, la concentration en éléments nutritifs inorganiques), l'abondance virale et microbienne (chiffres bactériennes directs, compte virales directs, et la cytométrie de flux pour évaluer autotroph vs ratios hétérotrophes), et la structure des communautés virales et microbiennes et potentiel métabolique par l'analyse métagénomique (pour un aperçu, voir la figure 1). Les résultats obtenus à partir des méthodes décrites ici sont nécessaires afin d'élucider les principaux paramètres de fond biogéochimiques nécessaires à la caractérisation complète des écosystèmes aquatiques.
Protocole
1. Préparation des instruments
- Préparation de l'installation de filtration
- Préparer un acide chlorhydrique (HCl) dilué à 5% (de 0,5 à 0,6 M, ajouter 250 ml de 32 à 36% de HCl à 4,75 L d'eau distillée pour faire 5 litres de solution finale). Conserver cette solution dans un récipient en polyéthylène haute densité (HDPE) pour un maximum de 2 semaines.
- Laissez toutes les pièces entrant en contact avec l'échantillon (à l'exception des filtres) pour 24 heures dans la solution de HCl à 5% à la sangsue sur le carbone organique dissous (COD) contaminants potentiels. Après chaque prélèvement rincer toutes les parties et les lignes de niveau avec environ 30 ml de solution HCl à 5% pour éviter la contamination de carbone (par exemple, des fumées de gaz, de pétrole, de bateaux, spray anti-moustique).
- Conservez tous les éléments d'échantillonnage d'emballages en aluminium pré-combustion aluminium ou plafonnés à jusqu'à immédiatement avant l'utilisation.
- Precombust 25 mm filtres GF / F enveloppés dans du papier d'aluminium dans un four à moufle à 550 ° C pendant 12 heures pour volatiliser hors tout carbone et de les stocker dans un endroit propre, secplacer jusqu'à utilisation. Utilisez Acid Washed pinces et gants stériles, non poudrés pour gérer les filtres precombusted à tout moment.
- Préparation du métagénome virales échantillonnage des matières
- Laver soigneusement les quatre 20 L pliables bonbonnes de polyéthylène basse densité et quatre 20 L haute densité de polyéthylène seaux et couvercles avec l'eau de Javel à 10%.
- Ensuite rincer tous les éléments 3x avec de l'eau distillée, puis l'échantillon et refermez bonbonnes avec robinet.
- Filtres lavage à flux tangentiel (TFF) après chaque utilisation selon le protocole suivant, et propre préventivement si le filtre n'a pas été utilisé au cours des 5 derniers jours.
- Dissoudre 50 g de NaOH en pastilles dans 5 litres d'eau dans un seau de lavage.
- Joindre une FFT humide à la tubulure et assembler selon la figure 3.
- Faire fonctionner la pompe péristaltique à ~ 30 min, sans contre-pression à déplacer une partie de la solution de lavage à travers le système de NaOH. Une fois lavage découle de la conduite de retour, déplacer transfert à laver seaupour compléter la circulation de la solution.
- Appliquer une contre-pression dans le système en plaçant un dispositif de serrage sur la conduite de retour en aval de la FFT. Cela va forcer la solution de lavage sur la ligne de filtrat. Temps excessif (5 min) à inonder l'extérieur de la FFT et produire filtrat est indicative d'une dégradation de la FFT.
- Faire fonctionner le système jusqu'à ce que la totalité de la solution de NaOH est entre les lignes. Ensuite, retirer la contre-pression, solution de fonctionner du système et le jeter.
- Rincer la FFT par l'eau courante sous contre-pression jusqu'à ce que le pH de l'eau qui coule dans les conduites de retour et le filtrat est pH 7-8.
- Retirer les tubes de la pompe péristaltique et la FFT. Refermer la FFT pour le stockage. Assurez-vous que la FFT reste humide jusqu'à la prochaine utilisation.
Collection 2. Exemple
REMARQUE: Pendant le transport, tous les échantillons prélevés doivent être conservés au frais (si possible 4 ° C) et non exposés à la lumière directe du soleil jusqu'à leur traitement ultérieur.
- Échantillonnagepour le carbone, éléments nutritifs, microscopie, cytométrie en flux, et microbiennes métagénomique
- Prenez deux unités Hatay Niskin par événement d'échantillonnage (résultant dans 4 L d'eau de l'échantillon). Ouvrez les vaisseaux échantillonnage juste avant l'immersion (air emprisonné autrement empêchera décroissant).
- Sur le site respectif, rincer l'intérieur du récipient d'échantillonnage avec l'échantillon d'eau en ouvrant les deux extrémités du cylindre et se déplaçant le long de l'axe polaire dans l'eau.
- Prenez l'échantillon et fermer soigneusement le récipient d'échantillonnage. Assurez-vous que le site d'échantillonnage est en amont du bateau et des plongeurs pour éviter la contamination.
- Échantillonnage pour virales métagénomique
- Sur le site cible monter pompe de cale sur 20 L bonbonne.
- Remplissez bonbonne visant cale entrée de la pompe à chaque zone et le sceau bonbonne ciblée avec l'embout correspondant.
- Recueillir 4 bonbonnes d'eau de mer non filtrée (ensemble 80 L) de chaque site d'échantillonnage.
3. SampLe préparation
REMARQUE: traiter les échantillons dans l'ordre présenté ici, à commencer par le DOC pour minimiser le risque de contamination. Utiliser des gants non poudrés pour l'ensemble de la procédure.
- Le carbone organique dissous (DOC)
- Montez une des deux unités Hatay Niskin aliquote en fente respective de l'ensemble de filtration. Raccorder la tubulure de sortie qui conduit au support de filtre respectif. Connectez le tuyau d'air sous pression (0,2 bar) à l'unité Hatay Niskin (voir la figure 2).
- Rincer toutes les lignes avec 100 ml d'eau de l'échantillon avant d'insérer des filtres dans le porte-filtre. Placez 25 mm pré-combustion filtre GF / F dans le porte-filtre en utilisant l'acide lavé forceps. Rincer chaque bouteille de DOC et plafonner 3 fois avec environ 20 ml eau filtrée d'échantillonnage.
- Remplir la bouteille avec environ 40 ml (~ 2/3) de l'eau d'échantillonnage. Recueillir au moins des doubles des échantillons de DOC d'avoir une sauvegarde en cas de contamination ou perte potentiel pendant le transport.
- Fabouteilles reeze d'échantillonnage tenant debout à -20 ° C jusqu'à l'analyse.
- Matière organique particulaire (POM)
- Après échantillons de DOC ont été perçus continuent filtration jusqu'à un total de 500 ml ont passé le filtre GF / F, y compris ce qui se passait à travers tout en collectant des échantillons de DOC.
- Dévissez le porte-filtre en ligne.
- Retirez le filtre de l'analyse de POM aide de pinces et lieu filtre à l'intérieur d'un carré de papier pré-combustion aluminium, pliez haut-côté tourné sur lui-même, et envelopper.
- Congeler les filtres à -20 ° C pour le stockage jusqu'à ce que l'objet de l'analyse élémentaire et isotopique.
- Nutriments inorganiques
- Placer un filtre de 0,2 pm Track-Gravé dans chaque support de filtre. Replacez les cartouches filtrantes.
- Rincer chaque 20 ml bouteille en plastique de scintillation trois fois à l'eau d'échantillonnage. Remplir chaque bouteille à l'épaule (~ 18 ml).
- Congeler à -20 ° C jusqu'à l'analyse plus tard.
- Microscopy (SYBR or et DAPI)
- Retirer le support de filtre.
- Prélever 1 ml d'eau dans chacun des deux tubes à centrifuger.
- Ajouter 66 ul de 32% de paraformaldéhyde à l'une des parties aliquotes (pour plus tard or SYBR coloration). Ajouter 12 ul 25% de glutaraldéhyde à l'autre correspondant (pour plus tard DAPI).
- Mélanger délicatement et laisser les échantillons de «réparer» pendant au moins 15 min à température ambiante dans l'obscurité.
- Cytométrie en flux
- Placer un filtre en polycarbonate de 8,0 um dans l'un des porte-filtres pour exclure les débris et les grosses cellules eucaryotes.
- Passer l'eau d'échantillonnage jusqu'à remplir deux cryotubes de chaque site d'échantillonnage avec échantillon de 1 ml.
- Ajouter 5 ul de glutaraldéhyde à 25% à chaque (concentration finale = 0,125%) cryovial.
- Inverser flacons à mélanger.
- Laisser l'échantillon de fixer pendant 15-30 min à température ambiante. Ne pas dépasser 30 minutes.
- échantillons de gel rapide dans l'azote liquide.
- Conserver les échantillons à -80 ° C ou en liquid azote expéditeur sec jusqu'à l'analyse sur un cytomètre de flux.
- Exemple métagénomique microbienne
- Retirer en porte-filtres de ligne.
- Directement monter un filtre cylindrique de 0,22 um sur la ligne correspondante.
- Filtre eau restante de l'échantillon des deux unités Hatay Niskin de chaque site (pour un total de 3-4 litres par filtre) à travers un filtre.
- Après la filtration, pousser l'eau restant sur chaque filtre à l'aide d'une seringue de 10 ml propre remplie d'air.
- Placer le filtre de retour dans son emballage d'origine et sceller l'emballage avec du ruban laboratoire.
- Rangez les filtres remplis individuellement à -20 ° C.
- Exemple de métagénomique virale
- Transfert des échantillons sur le métagénome virales immédiatement dans les seaux lavés à assurer qu'aucun échantillon est perdu ou contaminé par l'environnement.
- Pré-filtre en utilisant grand maillage de nylon de pores (25-100 um) pour enlever les débris et le matériel cellulaire avant concentration 19.
- Mettre en place la FFT comme le montre la figure 3, en plaçant la ligne de livraison dans un échantillon seau, et en laissant les conduites de retour et le filtrat fonctionnant dans un évier.
- Mettre la pompe péristaltique et rincer les conduites avec 1-2 L d'eau de l'échantillon.
- Placez la ligne de retour dans l'échantillon seau à terminer un cycle, ajouter 0,7 bar de contre-pression.
- Alors que la concentration de l'eau de mer, l'appoint réservoir d'échantillon que le niveau baisse. Lorsque le niveau de l'eau descend en dessous de la ligne d'admission dans le seau, le transfert concentré dans un agent de blanchiment à la chaux, rincés trois fois tripour bécher et continuer à se concentrer.
- Si bécher réservoir est vide, retirez la contre-pression, augmenter le débit de la pompe et poussez la totalité de l'échantillon à travers les lignes, la récupération dans le tripour.
- Passez le concentré de 0,45 um filtres cylindriques pour éliminer la plupart des bactéries sans discrimination contre des lignées virales.
- Changer de 0,45 m filtres cylindriques toutes les 150 ml. Recueillir le 0,45 um-filconcentré viral nominatives en tubes de 50 ml.
- Ajouter 250 ul de chloroforme à chaque 50 ml aliquote de concentré viral filtré pour éliminer les bactéries résiduelles. Mélanger par inversion, et magasin, debout à 4 ° C jusqu'à leur traitement ultérieur.
- Sécher les filtres de 0,45 m et de les stocker comme décrit dans les étapes 3.6.5 et 3.6.6.
4. Traitement des échantillons de microscopie
REMARQUE: Rincer tours de filtre avec l'eau de Javel à 10%, suivie par 95% d'éthanol pour éviter tache potentiel ou des résidus biologiques entre les pistes. Échantillons processus de microscope dans 1 heure et éviter les taches et les filtres colorés exposant pour éclairer si possible.
- Monture
- Ajouter 100 ul de 10% d'acide ascorbique à 4,9 ml de PBS 1x, et bien mélanger.
- Ajouter 5 ml de 100% de glycérol, et bien mélanger.
- Montez le filtre à l'aide d'une matrice d'alumine 0,02 um filtre de seringue jetable, aliquote et conserver à -20 ° C.
- SYBROr coloration
- Introduire à la pipette une partie aliquote de 500 ul de chaque échantillon fixé avec du paraformaldehyde à 2% de concentration finale dans un tube à centrifuger.
- Ajouter 0,5 pi de solution 1,000x SYBR or à chacune des parties aliquotes.
- Mélanger l'échantillon doucement et le mettre dans l'obscurité pendant 10 min.
- Placer un filtre de matrice d'alumine de 0,02 um avec bague annulaire de support de polypropylene sur chaque support de filtre du système de pompe à vide et attacher les tours de filtre (figure 4). Assurez-vous que le filtre de 0,02 um est placé sur le côté plastique de support de filtre en place, comme ces filtres ont une double porosité.
- Avec la pompe à vide éteint, ajouter les 500 pi de SYBR teinté échantillon, avec 2 ml de 0,02 um virus filtré l'eau libre à assurer que l'échantillon est réparti uniformément à travers le filtre.
- Mettre en marche la pompe à vide pour créer une pression négative de pas plus de 1 bar à éviter des virus et des microbes nuisibles filtrés.
- Permettre à l'ensemble de l'échantillon d'allerà travers.
- En utilisant une pince (pas la même paire que celle utilisée pour DOC), retirez soigneusement chaque filtre de la tour de filtrage, et passez une tâche délicate essuyer à assurer que le filtre est sec.
- Ajouter 10 ul de montage sur une lame de microscope et placer le filtre sec contenant l'échantillon coloré sur la baisse de la montagne.
- Ajouter 10 ul monter au sommet de chaque filtre avant d'ajouter une lamelle.
- Appuyez doucement sur la lamelle vers le bas. Essayez d'éliminer les bulles d'air si possible et éviter mouvement latéral de la lamelle.
- Placer les lames dans une boîte de lames et conserver à -20 ° C.
- La coloration DAPI
- Placer un filtre de matrice d'alumine de 0,2 um avec bague annulaire de support en polypropylène sur chaque stand de filtre et d'attacher les tours de filtre.
- Avec la pompe à vide éteint, ajouter 2 ml de 0,02 um de l'eau filtrée sans virus à chaque tour utilisé.
- Pipette de 1 ml de l'échantillon fixé au glutaraldéhyde dans chaque tour, résultant en un volume total de of 3 ml à chaque tour de filtre.
- Mettre la pompe à vide pour créer une pression négative de pas plus de 1 bar.
- Filtre échantillon complet sur le filtre de 0,2 um de matrice d'alumine.
- Retirez soigneusement le filtre de la tour de filtrage en utilisant une pince (pas la même paire que celle utilisée pour DOC).
- Placer le filtre sur une solution DAPI 100 pi baisse [25 pg / ml] dans une boîte de Pétri en gardant le filtre à l'endroit et laisser l'échantillon tache pendant 15 min. Evitez la lumière pendant le processus de coloration.
- Après la 15 min, retirer le filtre de la tache, passer sur une tâche délicate lingette pour sécher et de les transférer à une baisse de 100 pi d'eau libre de 0,02 um de virus filtré, en gardant à nouveau le filtre à l'endroit, à laver restant DAPI tache pendant 1 min. Répéter ce lavage une fois.
- Mont filtre sur la lame de microscope façon identique à celle décrite pour les échantillons SYBR Gold.
5. Extraction de l'ADN
- Microbienne ADN métagénomique
- Décongeler les filtres cylindriques pendant au moins 20 min à température ambiante.
- Enlever l'eau de mer tout en restant dans le filtre à l'aide d'une seringue de 10 ml. Boucher l'extrémité inférieure du filtre cylindrique à l'aide d'un capuchon et blanchie à l'autoclave.
- Pour chaque filtre, mélanger 360 pi T1 "pré-lyse« tampon et 50 pi protéinase K, puis la pipette le mélange dans le filtre. Cap l'extrémité ouverte.
- Incuber la réaction de lyse dans un four O / N (ou au moins 2 heures) tournant à 55 ° C.
- Retirez l'une des bouchon et ajouter 200 ul B3 "lyse" tampon dans le filtre à l'échantillon pré-lyse. Filtre Re-bouchon et incuber le mélange dans un four rotatif pour 10-20 min à 70 ° C.
- Extraire la totalité du volume du filtre cylindrique à l'aide d'une seringue de 3 ml par aspiration du liquide avec le filtre à l'envers.
- Expulser l'échantillon lysé dans un nouveau tube de centrifugeuse et ajouter 200 ul d'éthanol à 100%.
- Bien mélanger, charger la totalité de l'échantillon sur le spincolonne, et procéder comme indiqué par le protocole du fabricant.
- Quantifier l'ADN en utilisant un marquage fluorescent à base.
- Marine virale métagénomique
- Purifier et concentrer Phage particules (modifié d'après Thurber et al. 19)
- Dissoudre CsCl dans l'eau de mer de préparer des solutions de 1,7 g / ml, 1,5 g / ml, 1,35 g / ml, 1,2 g / ml (étalonnage en pesant 1 ml de solution). Filtre à chaque fraction de 0,02 um filtre avant utilisation.
REMARQUE: Assurez-vous que la densité de chaque fraction est précis à trois chiffres significatifs avant d'utiliser les solutions et filtrer toutes les solutions avec un filtre de 0,02 um avant utilisation. Utiliser l'eau de mer ou l'eau de mer saturés avec CsCl pour réduire ou augmenter la densité, respectivement. - Faire une lame de microscope à partir de 1 ml de réactifs combinés comme décrit dans la section 4.2 à veiller à ce que tous les réactifs sont exempts de virus avant de procéder.
- Ajouter 9 g de CsCl à 45 ml de concentré viral de la FFT àune densité finale de 1,12 g / ml. Réfrigérer pendant la mise en place du gradient de CsCl étape.
- A partir de la g / ml solution 1,7 ajouter successivement 1 ml de chaque fraction séquentielle moins dense lentement dans chaque tube en utilisant des pipettes sérologiques. Marquez le niveau de chaque individu ménisque et veiller à ce que les pycnoclines entre fractions ne sont pas perturbés. Pour plus de détails, voir le document d'accompagnement (Lim et al. 2014).
- Avec une charge de pipette sérologique 7,5 ml d'échantillon dans chacun des six tubes et centrifuger à ~ 83 000 x g, 4 ° C pendant 2 heures.
- Sans perturber les gradients de densité décharger le rotor et percent le tube juste au-dessous de 1,5 g / ml couche de densité préalablement marqué (figure 5) avec une aiguille de 18 G sur une seringue de 3 ml. Prélever 1,5 ml de VLP purifiées et transférer dans un nouveau tube. Répétez l'opération pour tous les échantillons.
- Ajouter 0,2 volume de chloroforme pour les VLP purifiées, mélanger vigoureusement, incuber à température ambiante pendant 10 min.
- Ajouter 150 &# 181; l de 10x DNAse tampon à chaque microtube.
- Tourner à 16 000 g pendant 5 min, et recueillir la phase aqueuse. Si le chloroforme ne pas total dans le fond du tube à centrifuger, ajouter davantage de tampon de DNAse, mélanger et répéter.
- Ajouter la DNase I (concentration finale = 2,5 unité / pl) et on incube à 37 ° C pendant 2 heures, puis inactiver l'activité de la DNase par incubation à 65 ° C pendant 15 min.
- Dissoudre CsCl dans l'eau de mer de préparer des solutions de 1,7 g / ml, 1,5 g / ml, 1,35 g / ml, 1,2 g / ml (étalonnage en pesant 1 ml de solution). Filtre à chaque fraction de 0,02 um filtre avant utilisation.
- D'extraction d'ADN à partir de particules pseudo-virales (VLP)
- Commun uniformément les VLP purifiées à partir de chaque échantillon en deux tubes Oak Ridge nettoyés et passés à l'autoclave.
- Ajouter 0,1 volume de 2 M de Tris-HCl (pH 8,5) avec 0,2 M d'EDTA, 0,01 volumes d'EDTA 0,5 M, 1 volume de formamide, 10 ul de glycogène (10 mg / ml). Bien mélanger et incuber à température ambiante pendant 30 min.
- En se référant à nouveau volume, ajouter 2 volumes RT 100% d'éthanol. Mélanger et incuber à 4 ° C pendant plus de 30 min.
- Culot d'ADN en faisant tourner les tubes Oak Ridge à 17 200 g pendant 60 min, à 4 & #176; C.
- Eliminer le surnageant. Laver le culot d'ADN deux fois avec de l'éthanol à 70% glacé en utilisant des pipettes sérologiques.
- Retirez tout l'éthanol (re-tourner brièvement le cas échéant), couvrir et laisser culot sécher à l'air à température ambiante pendant> 15 min.
- Remettre en suspension le culot d'ADN pour> 15 min dans 567 pi de tampon TE 1X (pH 8,0). Transférer les 567 ul d'ADN remis en suspension dans un nouveau tube de 1,5 ml.
- Ajouter 30 ul de SDS à 10% (pré-chaud à 65 ° C avant utilisation) de pi et 3 de la protéinase K (20 ug / ml), bien mélanger et laisser incuber pendant 1 heure à 56 ° C.
- Ajouter 100 ul de 5 M de NaCl et bien mélanger. Ajouter 80 ul de solution de pré-chauffé CTAB NaCl, vortex et incuber pendant 10 min à 65 ° C.
- Ajouter 0,2 parties de volume de chloroforme, vortex, et tournent à 16 000 g pendant 2 min. Transférer le surnageant dans un nouveau tube de 1,5 ml.
- Ajouter un volume égal de phénol chloroforme isoamylique 25: 24: une solution, vortex pour mélanger,et tourner à 16 000 g pendant 2 min. Transférer le surnageant dans un nouveau tube de 1,5 ml à centrifuger.
- Ajouter un volume égal de chloroforme, vortex, et tournent à 16 000 g pendant 2 min. Transférer le surnageant dans un nouveau tube de 1,5 ml à centrifuger.
- Ajouter 0,7 volume d'isopropanol à la fraction surnageante et on incube à -20 ° C pendant au moins 30 min pour précipiter l'ADN.
- Pellet l'ADN par centrifugation à 16 000 xg pendant 20 min à 4 ° C. Pipeter le surnageant et laver le culot deux fois avec 500 ul de glace-éthanol à 70% froid.
- Tourner à 16 000 xg et éliminer l'éthanol restant dans le tube. Sécher à l'air le culot pendant 15 min.
- Remettre en suspension l'ADN culot avec 50 pi de tampon d'élution (Tris 5 mM, pH 7,5) ou du Tris-EDTA, pH 8,0 pendant au moins 5 min à température ambiante.
- Quantifier l'ADN en utilisant une analyse par fluorescence à haute sensibilité.
- Purifier et concentrer Phage particules (modifié d'après Thurber et al. 19)
Résultats
Microscopie
échantillons de microscopie peuvent et doivent être analysés immédiatement pour assurer qu'ils sont de la qualité souhaitée. Pour mesurer l'abondance et la distribution de la taille de la communauté bactérienne, diapositives DAPI seront examinées par microscopie à épifluorescence (excitation / émission: 358/461 nm, voir McDole et al 2012). (Figure 6A). Les comptages cellulaires et les dimensions peuvent être recueillies à l'aide de logiciels d'imagerie (par exemple, ImagePro ou ImageJ). De volumes longueur et la largeur cellulaires (V) sont dérivés en faisant l'hypothèse que toutes les cellules ont la forme de cylindres avec des bouchons hémisphériques en utilisant l'équation suivante:
V = π / 4 × w 2 (L - w / 3)
où L est la longueur et w est la largeur de chaque cellule de 23,4. La biomasse microbienne peut alors être estimé en utilisant déjà établi des relations de taille dépendant FOr communautés microbiennes marines 24. Généralement vont bactéries marines en longueur de 0,1 à 4 pm, mais aller jusqu'à ~ 8 pm dans certains endroits.
Alors que les filtres (0,2 um) colorées au DAPI ne montre que les bactéries, les filtres utilisés pour la teinture SYBR Gold (0,02 um) contiennent des bactéries et des virus. La mesure de l'abondance de virus suit le même protocole que pour les micro-organismes, mais une excitation de 325 à 375 nm est utilisé, et le maximum d'émission est à 537 nm (Figure 6B).
Afin de générer les données quantitatives du volume de prélèvement peut être nécessaire d'ajuster en fonction de l'abondance du virus dans l'échantillon d'origine. Le volume exact de filtre est mieux déterminée empiriquement pour une masse d'eau. Des exemples de micrographies contenant des échantillons avec des concentrations variables virales sont illustrés à la Figure 7.
Les résultats des études précédentes suggèrent que le virus de Microberatios (VMR) varient généralement de 1 à 50 dans les systèmes aquatiques 25-29, et entre 3 et 20, avec une moyenne d'environ 6 dans les systèmes de récifs coralliens (Knowles données non publiées).
Cytométrie en flux
En plus des comptages directs et estimation de la taille de la communauté microbienne, les évaluations du rapport autotrophes à microbes hétérotrophes par cytométrie en flux peuvent en outre être extraits des échantillons prélevés (flux exemplaire cytométrie sortie donnée dans la figure 8). Pour déterminer le nombre total de cellules de bactéries, les échantillons sont colorés avec du SYBR Green I et un tube photomultiplicateur d'un filtre passe-bande 530/30 est utilisé pour la détection. Un canal pour la chlorophylle (retour à miroirs de LP dos résultant dans une gamme de 675 à 735 nm) et (filtre passe-bande 585/42) phycoérythrine est utilisée pour compter l'abondance des autotrophes dans des échantillons non colorés. Pour déterminer l'abondance de microbes hétérotrophes, les comtes autotrophes de la nonpartie tachée sont ensuite soustrait du nombre de SYBR teinté totale (McDole et al. soumis).
La métagénomique des virus
Métagénomique viral utilise une approche moléculaire indépendante de la culture de l'écologie virale en utilisant l'information totale de la séquence génomique de déterminer la structure et la fonction communauté. La métagénomique a été faire progresser notre compréhension de la complexité et de la diversité des communautés virales au niveau local 17 à l'échelle mondiale 30. Le développement récent d'un large éventail d'outils bioinformatiques pour l'analyse de données de métagénomique virale a diminué les goulets d'étranglement de calcul, permettant une vision plus globale de la de la virosphere à travers le prisme de la métagénomique.
Les méthodes présentées ici mettent en évidence l'isolement et l'enrichissement des particules virales à partir de l'eau de mer en utilisant une combinaison de pré-filtration de nylon large de la taille des pores de maille pour enlever les débris et la matière cellulaire, de la concentration deles échantillons avec de la FFT (figure 3) et une filtration subséquente de 0,45 um pour éliminer les cellules plus grandes. Cette méthode produit environ un 100x échantillon concentré (figures 5B-5E) qui nous permet d'effectuer des procédés en aval dans les petits volumes. Afin d'empêcher la croissance de toutes les cellules microbiennes qui restent dans le lysat viral et par conséquent des changements dans l'abondance viral de l'échantillon au cours du temps de transit de temps entre la station de terrain et en laboratoire, le chloroforme est souvent ajouté à une concentration finale de 2% pour le stockage. Suite à l'isolation et la purification des VLP, la microscopie à épifluorescence avec des colorants d'acides nucléiques tels que l'or SYBR sont utilisés pour vérifier la présence et la pureté des particules virales (figures 5B-5E).
Ici, nous présentons deux métagénomes virales de la barrière de corail sud de l'île de ligne, en particulier, le Starbuck (STAR7) et du Millénaire (CAR9; précédemment connu sous le nom Caroline) îles. Un volume total de 120 L sample eau ont été recueillies à partir de chaque site à une profondeur de 10 mètres et traité comme décrit dans la section 3.7 et 5.2. Les VLP purifiées à partir de la centrifugation sur gradient de chlorure de césium était 3,3 x 10 8 particules / ml pour CAR9 (Figure 5D) et 2,9 x 10 9 particules par ml pour STAR7 vérifiée selon la méthode décrite au paragraphe 4 (figure 5B). La quantité totale d'ADN isolé à partir de ces échantillons étaient d'environ 400 ng basée sur la mesure de Nanodrop. L'ADN a été amplifié à l'aide Phi29 polymérase et séquencé par le Fonds pour l'environnement génomique de base en 2011. Les caractéristiques des données de séquence est présentée dans le tableau 1. Sur la base de recherches de similarité en utilisant les bases de données existantes, la majorité (> 70%) des séquences souvent fini , à savoir les origines et les fonctions inconnues non caractérisés. De même, les deux viromes présentées ici ont présenté plus de 97% de séquences inconnues. En conséquence, non-base de donnéesanalyse dépendante a été utilisé pour l'analyse et a ouvert un nouveau bras de possibilité de recherche sur la «matière noire» dans la métagénomique virales (Seguritan et al. sous presse).
Microbienne métagénomique
L'analyse de métagénomes microbiennes permet de caractériser de la communauté microbienne présente et la description fonctionnelle de ces communautés. Les exemples incluent des estimations de la présence d'agents pathogènes et la virulence 5,22 ou des comparaisons entre les changements dans la structure des communautés macrobial ou de nutriments disponibles et l'abondance des espèces et de leurs voies métaboliques prédominent (figure 9; Kelly et al 2014)..
chimie de l'eau
paramètres chimiques de l'eau ont généralement un traitement complet d'analyse pour générer les données souhaitées; échantillons pour l'analyse du COD seront mesurés par oxydation à haute température catalytique 31,32, la matière organique particulaire (POM) par rapport isotopique par spectrométrie de masse couplée à un analyseur élémentaire 33-35, et les nutriments inorganiques par Flow Injection Analysis 36,12,37. Après une réussite de toutes les analyses, informations comme indiqué dans le tableau 1 sera disponible pour compléter la caractérisation du microbienne et des communautés virales. Cette information peut être complétée par des ratios d'isotopes stables d'échantillons de carbone et d'azote organique (Voir Haas et al. 35) et les rapports entre autotrophes et hétérotrophes (McDole et al. Soumis).
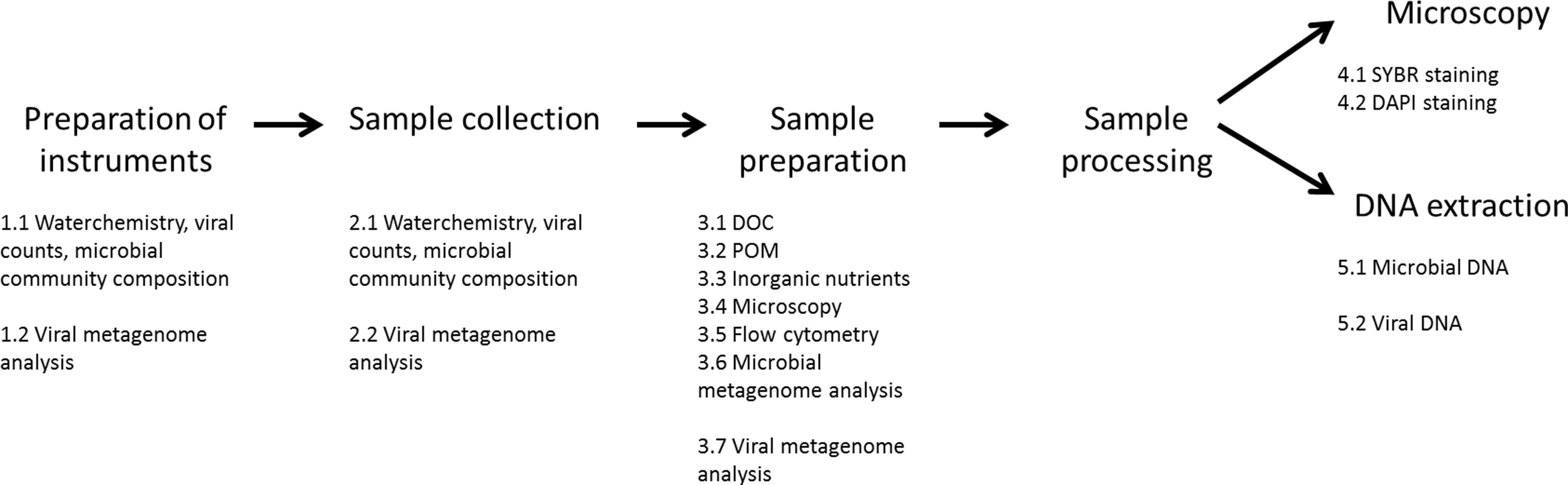
Figure 1. Vue d'ensemble des méthodes décrites dans la section de protocole. Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Figure 2. Exemple de configuration traitement de forage. L'installation de filtration introduit ici (schéma à gauche, image réelle panneau de droite) ne doit pas nécessairement pour générer des échantillons mais permettra d'accélérer le processus de manière significative. L'installation se compose d'une plaque, sur laquelle les unités Hatay Niskin (1) sont montés. A la sortie vers le bas, les unités Hatay Niskin sont connectés avec un tube de silicone pour les porteurs (2) filtre en ligne. Ces laissez-passer l'eau échantillonnée à travers le filtre respectif dans les flacons PEHD d'échantillonnage (3) monté juste en dessous. Le procédé de filtration est accéléré à travers l'air sous pression (4), qui et réglé par une jauge de pression (5). Un bassin de débordement empêche renverser de l'eau au cours de l'évolution des flacons en PEHD ou filtration de POM. Se il vous plaîtcliquez ici pour voir une version plus grande de cette figure.

L'eau de l'échantillon 3. La figure tangentielle gamme de filtration d'écoulement (modifié à partir de Thurber et al. 19). Maille de nylon filtrée est pompée à partir du réservoir (1) par l'intermédiaire d'une pompe péristaltique (2) à un filtre à flux tangentiel (3). L'échantillon d'eau revient au réservoir (1) le long de la ligne de retour (4) en l'absence de contre-pression. Lorsque la contre-pression est appliquée au moyen d'un collier de serrage (5) sur la ligne de retour, l'eau passe à travers le filtre et est délivrée au rebut ou vers le réservoir de filtrat (6). l'eau de l'échantillon est remplacé comme il est concentré sur la ligne filtrat jusqu'à ce que toute l'eau de l'échantillon est concentré dans les tubulures.
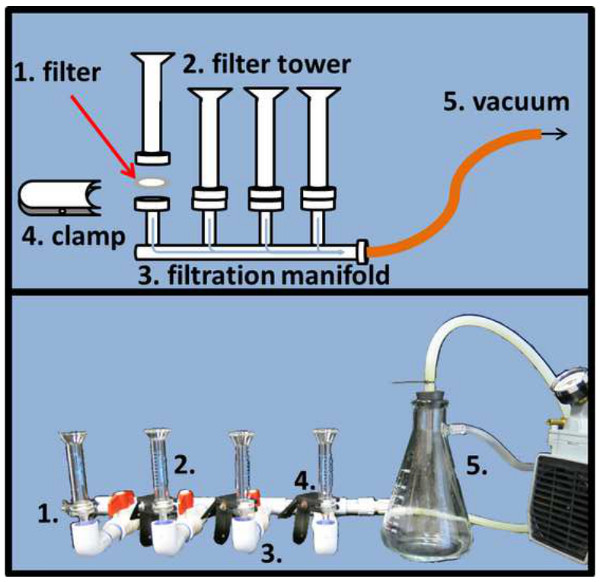
Figure 4. Microscope installation de filtration de diapositive.Pour répartir uniformément DAPI et SYBR or échantillons colorés sur le filtre de matrice d'alumine (1) respective, les filtres doivent être placés entre la tour de filtre (2) et de la tige du collecteur de filtration et fixé avec une pince (4). Un vide réglementé de pression (5) permettra d'accélérer le processus de filtration (en haut de schéma, réelle panneau inférieur de l'image). Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
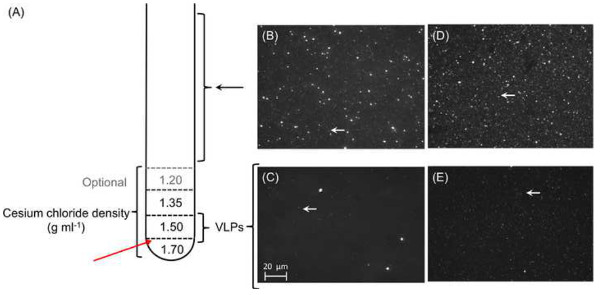
Figure 5. La purification et la concentration de particules de phage. Un total de 1 ml de chaque gradient de densité de chlorure de césium sont posés l'un sur l'autre avant de diriger l'échantillon pré-traité (A). Chaque couche doit être marquée à l'extérieur du tube pour faciliter l'extraction après ultracentrifugation. Les flèches rouges où le tube sera percé à l'extracter la fraction de VLP. (BE) micrographies des concentrés viraux: micrographies épifluorescence de 0,45 um filtrées concentrés viraux représentatifs de STAR7 (B) et CAR9 (D). Les grosses particules, y compris les cellules bactériennes ont été visibles à partir de 0,45 um les filtrats des deux-STAR7 (B) et (D) CAR9 échantillons, tandis que des VLP (flèches blanches) restent après gradient de chlorure de césium ultracentrifugation; 7 étoiles (C) et CAR9 (E). Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
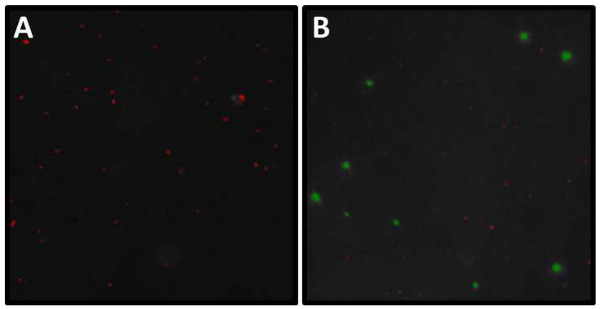
Figure 6. Exemple de DAPI et or SYBR analyse micrographique. D'écran à partir de l'analyse de (A) et DAPI (B) or SYBR teinté exemple de glissière. (A) La longueur et la largeur (um) de toutes les particules mises en évidence(= Microbes) peuvent être évalués à l'aide d'un programme d'imagerie. Noter l'agrégation dans le centre de l'image. Ces groupes doivent être exclus de l'analyse ultérieure. (B) Au cours de l'analyse des virus et des bactéries ont besoin d'être mis en cellule par des seuils de taille et de VLP dans des gammes de taille cellulaire (VLP rouge, vert cellulaire). Notez qu'il existe généralement des VLP faibles Non classé par le programme d'imagerie respectif. Ces VLP apparaissant points blancs faibles doivent être comptabilisés et ajoutés aux comptes virales automatisés manuellement. Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.

Figure 7. Les concentrations de SYBR or teinté échantillons. Micrographies d'un filtre 0,02 matrice alumine contenant différents nombres de SYBR teinté échantillons de virus. Le panneau A montre afilter contenant une quantité appropriée d'eau de mer filtrée, alors que les concentrations sur le filtre de la B sont trop élevés et en C trop faible pour compte fiables. Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
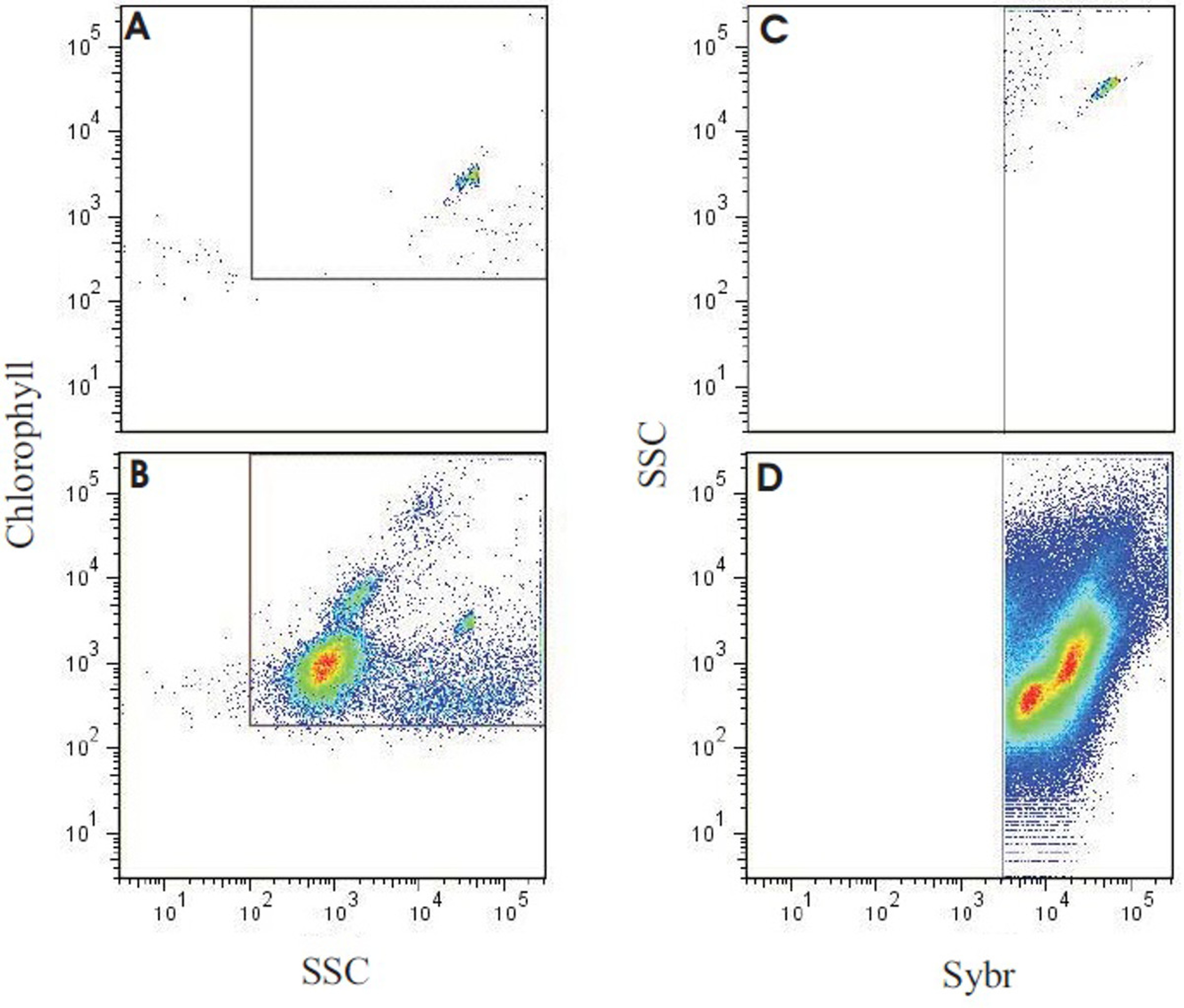
Figure 8. Résultats de cytométrie en flux de sortie de l'analyse d'un échantillon récif-eau. Panneau (A) montre l'eau moléculaire de qualité uniquement avec des microsphères fluorescentes jaune-vert (0,75 um), utilisés pour vérifier fond minimale avec les réglages de l'appareil. Groupe (B) représente la sortie de l'échantillon d'eau de récif terme avec des réglages identiques et la mise en page que dans A. Notez que les perles peuvent encore être vus dans le même endroit, ainsi que plusieurs populations de autotrophes. (C) Le même échantillon que dans (B), utilisé pour sauvegarder porte le cytomètre en flux, en ciblant les événements positifs de SYBR (autotrophes + comptage hétérotrophe) et en minimisant fond. (D) L'échantillon récif-eau représentant colorées avec SYBR Green I. Utilisation le déclenchement établi à (C), le nombre total de microbes ont été générés, et les microbes hétérotrophes et autotrophes cloisonnés par la chlorophylle autofluorescence (voir McDole et al. 4). Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
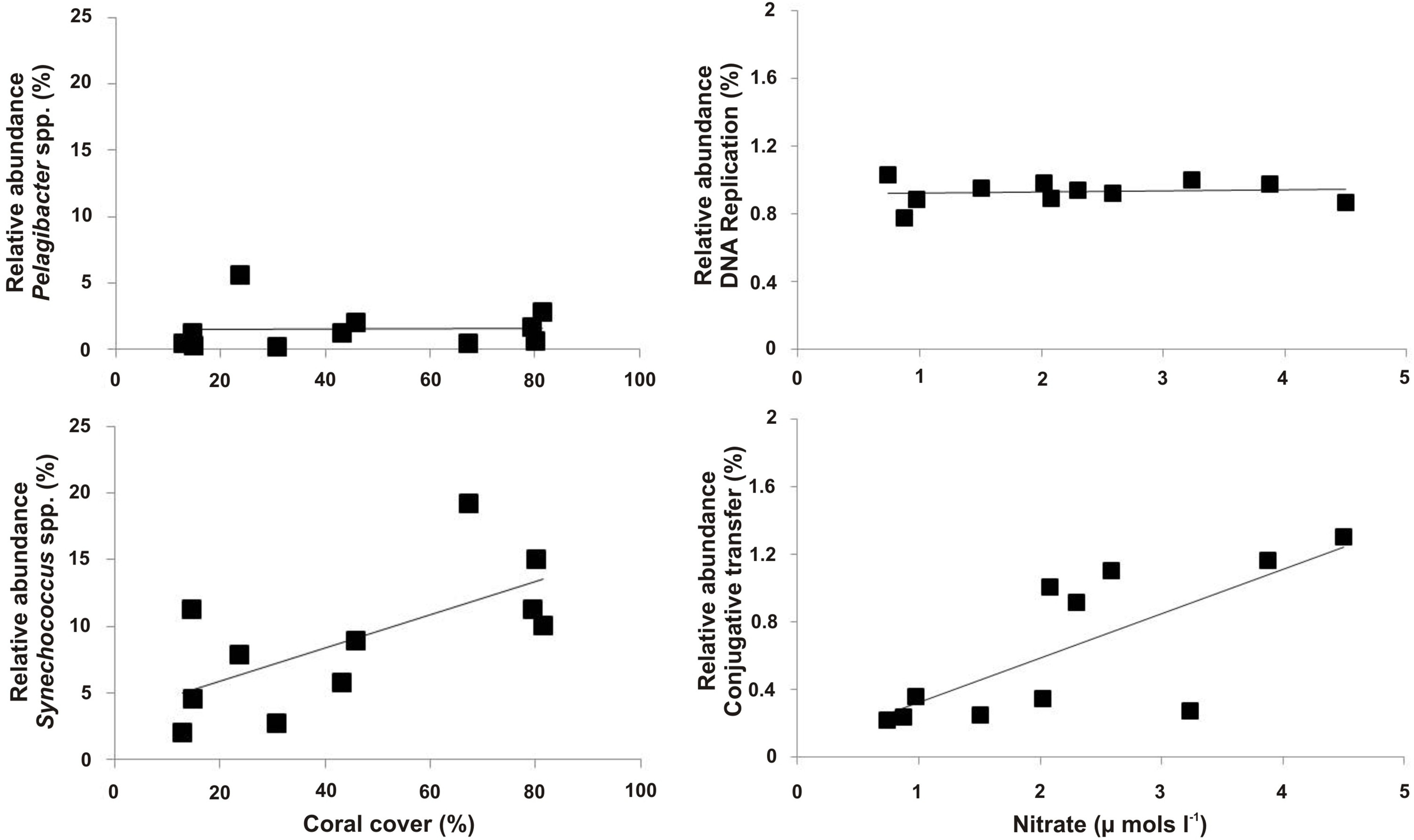
Figure 9. Exemple de données sur le métagénome microbien. Patterns entre les données de séquence de métagénomique microbienne et variables dépendantes du site. Ici, l'abondance relative de Synechococcus spp. est positivement corrélé avec le couvercle de pour cent de corail, tandis que Pelagibacter spp. n'a pas été (panneaux de gauche). La voie métabolique pour le transfert de conjugaison a été positivement corrélé avec les concentrations de nitrates, tandis que la voie métabolique de réparation de l'ADN était cohérente entre tous les métagénomes (panneaux de droite). Se il vous plaît cliquer ici pour voir une version plus grande de cette figure.
| 7 étoiles | CAR9 | |
| Nombre total de Lit | 939311 | 591600 |
| Nombre de Prétraitment Lit | 579664 | 360246 |
| Séquence longueur moyenne (pb) | 395 ± 131 | 451 ± 159 |
| Séquences annotées (en%) | 42 218 (7,3%) | 9117 (2,5%) |
| Inconnu (%) | 537 446 (92,7%) | 351 129 (97,5%) |
Tableau 1. Caractéristiques techniques de deux viromes générés par les îles de la Ligne du Sud, Starbuck et du Millénaire. Le degré d'annotation est basé sur BLAT utilisant le serveur MG-RAST.
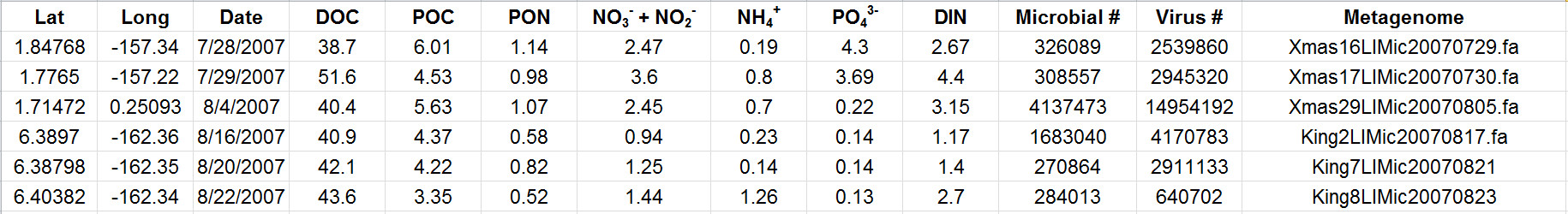
Tableau 2. Les résultats représentatifs indiquent les données de différents sites au cours d'une expédition. Les paramètres évalués ici de mesures de la chimie de l'eau jumelé avec microbienne et abondances virales. Le tableau contient en outre des noms de fichier se référant à des données de métagénomique respectifs pour chaque emplacement d'échantillonnage. concentrations de carbone et des nutriments inorganiques sont donnés en pmol / L. Se il vous plaîtcliquez ici pour voir une version plus grande de cette table.
Discussion
Les méthodes présentées ici fournissent un outil pour générer une évaluation complète de la dynamique virales et microbiennes dans les écosystèmes aquatiques. Ils visent à quantifier le stock permanent de ressources organiques et inorganiques disponibles pour les communautés microbiennes et de caractériser la composition de la communauté virale et microbienne présente. En plus de la quantification de l'abondance virale et de l'abondance et de la biomasse microbienne, des informations de séquence génomique révèle leur structure et leur fonction communauté. Toutes les méthodes ont été développés ou modifiés pour permettre l'application dans des endroits éloignés de terrain. Cependant, l'absence de paramètres de laboratoire contrôlées crée par nature un certain risque d'erreurs. Ici, nous discutons quelques mises en garde associées à chacun des paramètres évalués. Suivant à risque de contamination lors de la manipulation des échantillons de certains paramètres céder aussi risque d'erreur dans le processus analytique.
Le carbone organique dissous
contamination de carbone peut se produire pendant les procédures d'échantillonnage (échantillonnage en aval, ou dans le voisinage d'un bateau ou plongeur), au cours de la préparation de l'échantillon (contamination de l'équipement ou de la manipulation par inadvertance), et même au cours du stockage de l'échantillon (d'autres échantillons dans le congélateur contenant organique solvants volatils /). Pour éviter les différentes sources de contamination de conduite que quelques étapes de manipulation possible, que chaque échantillon délocalisation pose une source supplémentaire de contamination. L'échantillon ne doit pas entrer en contact avec un objet non lavé à l'acide ou brûlés. Enfin, la désignation d'un congélateur de stockage exclusivement pour les échantillons ne contenant pas de substances organiques volatiles sera d'assurer l'intégrité des échantillons pendant les périodes de stockage plus longues. Si les échantillons ne peuvent être congelés pendant une longue période de Voyage acidification des échantillons de DOC avec HCl concentré (comme décrit 38-41) peut être une alternative applicable. Pour vérifier les documents de référence DOC de consensus de précision de mesure doivent être utilisés regrement au cours de la mesure de la DOC fonctionne. En particulier l'eau de mer profonde (> 2000 m) références sont précieuses pour évaluer la performance de la machine analytique car il est très stable dans son contenu DOC 42.
Matière organique particulaire
En plus d'éviter la contamination de carbone organique, POM échantillonnage nécessite une attention particulière pour assurer l'exactitude du volume filtré. Si le volume prévu de 500 ml doit écarter pour quelque raison que ce potentiel doit être noté à rapporter la quantité de POM capturées sur le filtre pour le filtrat, dont il a été dérivé.
Nutriments inorganiques
Comme pour l'échantillonnage de carbone organique, il faut faire attention à une éventuelle contamination de l'échantillon en éléments nutritifs générés par diverses sources comme des bateaux ou des rejets d'eaux usées 12. Les filtres utilisés pour l'échantillonnage du carbone organique avec une tension nominale de tailles de pores de 0,8 um, peuvent ne pas exclure toute la biomasse microbienne et should donc être modifié de 0,2 pm pour les filtres de cette étape de filtration. En outre, les limites de détection posent un problème avec les échantillons des éléments nutritifs; en particulier dans l'eau de surface oligotrophe autour de nombreux endroits de récifs coralliens (par exemple, Cotner et al., 43). Les limites de détection sont à peu près autour de 0,1, 0,2, et 0,1 pmol / L pour l'ammonium, nitrate et nitrite, et ortho-phosphate respectivement (voir 36,12,37)
Microscopie
En plus des variations dans la concentration de l'analyse d'image des échantillons de lames de microscopie donne en outre risques d'erreurs. Par exemple, les images peuvent être de mise au point dans certaines régions du champ de vision, et la mise au point dans d'autres. Comme un filtre de matrice d'alumine de grande pureté est un type de filtre très rigide, tous les débris sous le filtre peut entraîner l'ensemble de filtre à reposer sur un angle par rapport à la glissière. En conséquence, les images peuvent être systématiquement hors foyer dans différentes parties du champ de vision pour un filtre donné, regardless de l'alignement du microscope. Remontage des filtres, assurant la face inférieure du filtre est propre, peut améliorer cette si elle est observée. En outre, dans certains cas, il peut y avoir deux plans focaux dans lequel les virus et les microbes peuvent être trouvés. Ceci est le résultat d'objets colorés se détacher du filtre lors du montage, et flottant à la face inférieure de la lamelle qui peut rendre compte peu fiables. Par conséquent, il est toujours recommandé de vérifier la qualité des échantillons au cours du processus.
Cytométrie en flux
Comme pour les échantillons de microscopie, il peut y avoir des différences entre la cytométrie de flux des échantillons qui nécessitent des ajustements du processus d'analyse d'origine naturelle. Par exemple, en fonction de la sensibilité de l'instrument, les cellules fluorescentes Prochlorococcus faiblement peuvent être en dessous du niveau de bruit et ne seront pas quantifiés. Perles de faire un contrôle de l'échantillon interne (par exemple, pour confirmer que le REEE de l'instrumentonse aux signaux fluorescents est conforme au jour le jour (et reste stable pendant la course elle-même). Si elles sont ajoutées à l'échantillon à des concentrations connues, ils peuvent également servir en tant que contrôle interne afin de vérifier l'exécution de volume de l'échantillon. Cependant, il est recommandé de toujours lancer une aliquote d'un échantillon préalablement congelés "standard" de l'eau de mer qui est décongelé et coloré avec les échantillons d'intérêt à fournir un contrôle biologique supplémentaire pour la manipulation entre plaque de 96 puits pistes échantillon. En fonction de l'emplacement, des échantillons d'eau de mer peuvent être très différents les uns des autres. Tri populations «représentatives» et puis regarder sous un microscope à épifluorescence (EM) peut être un bon moyen de vérifier rapidement les populations de phytoplancton que soit Synechococcus sp. Ou eucaryotes photosynthétiques. Prochlorococcus cellules ne sont généralement pas visibles sous EM car elles se fanent trop vite. En plus de la chlorophylle a, Synechococcus sp. Contenir aussi le phphycoeurythrin otopigment (PE), qui émet de la lumière à des longueurs d'onde plus courtes (540 à 630 nm) que la chlorophylle A (660-700 nm). Lorsque les cellules Synechococcus sont excités avec lumière bleue / verte (470-490 nm), ils apparaissent d'or si on la regarde sous un filtre d'émission qui supprime toutes les longueurs d'onde de lumière en dessous de 510 nm. Par comparaison, la fraction eucaryote cherchera rouge, lorsqu'il est excité avec la même longueur d'onde de la lumière.
La métagénomique des virus
Il est important de noter que le processus de concentration et de stockage VLP influence de la communauté récupéré. Perte de particule virale peut survenir à chaque étape du processus, et donc, le choix des protocoles de purification et d'enrichissement virales peut finalement affecter la composition taxonomique observée et la diversité des métagénomes virales résultant 44,45,18. Concentration des échantillons peut être fait en utilisant la FFT 19 ou 20 floculation à base chimique. Dans la base chimique approche, postulertivement chargée d'ions de fer se lient les particules virales naturellement chargées négativement formant grandes (> 8 um) des complexes de fer-virale qui floculent dans la solution et peuvent être récupérés en utilisant des filtres de 8 um. Par la suite le fer est chélaté les particules virales et re-dissous en utilisant du magnésium, de l'acide ascorbique, et de l'EDTA, en laissant les particules virales concentrées pour l'extraction d'acide nucléique 20. Après avoir comparé ces deux méthodes actuelles, nous avons conclu que la méthode de chlorure de fer est moins de temps que la FFT concentration virale, mais peut donner quelques mises en garde. Nous avons trouvé que la dissociation et la dissolution du fer-viral nécessite une deux fois la solution plus concentrée de magnésium de l'acide ascorbique, de l'EDTA à 20 rapporté dans le but de procéder à la dissolution avant se dégrade de l'acide ascorbique. Mis à part ce problème, le protocole purifie particules virales par taille fractionnement seule, éliminer les contaminants microbiens en utilisant 0,2 um filtration. Les grands virus ne passent pas par la FILTer (par exemple, Yang et al. 46) et ne sera donc pas détecté dans le métagénome, tandis que certaines bactéries ne (McDole, données non publiées). Il en résulte une contamination bactérienne tout en discriminant contre les grands virus.
Cette question spécifique est toutefois également pertinente pour l'élimination des microbes dans le cadre de la concentration de la FFT. Comme a été démontré que le traitement par le chloroforme pour éliminer les virus sensibles au chloroforme des membranes lipidiques externes 47 certaines études suggèrent que 0,22 pm filtration peut être utilisé pour éliminer la majorité des bactéries. Il convient également de noter que la méthode présentée vise principalement bactériophage, le support le plus abondant de matériel génétique dans des environnements marins. Parce phages sont pas habituellement considérée contenir communément lipides 48 ils sont plus résistants au traitement de chloroforme. À notre connaissance, une méthode «fourre-tout» ne existe pas à ce jour. La méthode présentée ici est adaptée de l'OUl'expérience de terrain de r au cours des 10 dernières années dans divers environnements marins couvrant tropical aux systèmes arctiques.
Microbienne métagénomique
La construction de banques metagénomiques de microbes (par exemple, les bactéries et les archées) est plus simple que métagénomes viraux comme il est plus facile de produire des concentrations d'ADN minimales requises pour la préparation de bibliothèque. Amplification fusil de chasse bibliothèques Linker (de) LASLs 17,49,50 et tout le génome amplification basée sur l'amplification par déplacement multiple (MDA) sont les deux méthodes les plus couramment utilisées pour générer suffisamment d'ADN pour le séquençage. L'utilisation de MDA pour obtenir des concentrations d'ADN plus élevées dans métagénomes microbiennes est parfois nécessaire. Il peut cependant provoquer des artefacts dans les données de séquence, telles que la suramplification de minicercles de dinoflagellés. MDA méthodes sont connues pour amplifier préférentiellement l'ADN simple brin et circulaire, résultant en résultats non-quantitatives 51,52. Les LASLs optimisés approchent 50 peut donc sert comme une meilleure alternative à amplifier la fois microbienne et de l'ADN métagénomique viral pour le séquençage. Il est important de noter que l'approche de LASLs a plusieurs étapes, nécessite un équipement sophistiqué, et se limite à des modèles ADN double brin. En outre, le kit d'extraction d'ADN de tissu a été montré pour extraire l'ADN des deux embranchements à Gram négatif positif et Gram, mais il n'a pas été vérifiée de taxons microbienne efficace lyse récalcitrant ou leurs structures associées (par exemple, certaines archées, endospores ou des spores de champignons) . Par conséquent, une étape de battage de talon peut être nécessaire pour obtenir de l'ADN à partir de certains microbes.
Ces évaluations globales se traduira par une meilleure compréhension du fonctionnement virale et microbienne des écosystèmes. Bien que peu d'études ont entrepris l'effort d'évaluer tous les paramètres proposés, beaucoup ont étudié sélectionnés. Nelson et al. 53 suggère une connexion entreen habitats récifaux spécifique, et un appauvrissement dans les deux concentrations de COD et bactérioplancton relatifs aux eaux extracôtières. Ces changements de concentration ont été accompagnés par des différenciations distinctes des communautés bactérioplancton. Dinsdale et al. 5 a montré que l'augmentation de l'impact anthropique a été accompagnée par une plus grande abondance de 10x cellules microbiennes et de particules pseudo-virales. Les communautés microbiennes dans les eaux marines entourant les îles habitées ont également de plus grandes fractions de bactéries hétérotrophes et des agents pathogènes potentiels 5. Enfin McDole et al. 4 a montré, en introduisant le score de microbialization, que les activités humaines se déplacent d'énergie pour les microbes, au détriment des macrobes. Ces études, centrées sur les paramètres microbiens et chimiques de l'eau spécifiques, fournissent de nouveaux aperçus sur la structure biochimique des écosystèmes marins. En combinant les données traditionnelles des efforts de surveillance des écosystèmes - comme température et de pH valeurs, la biomasse des poissons et diversiTy, couverture benthique, ou l'exposition à l'impact humain - avec la chimie de l'eau et des évaluations microbiens, se traduira probablement par les efforts de surveillance de l'environnement les plus sensibles, ce qui peut conduire à des efforts de conservation plus ciblés.
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
We thank Elisha Wood-Charlson, Jackie Mueller, Karen Weynberg, and Kathy Morrow for their help and constructive input on this manuscript. We also thank the crew and captain of the Hanse explorer which provided us with a perfect working environment to conduct large parts of this research. Further we would like to thank the three anonymous reviewers for their time and helpful comments to improve the manuscript. This work was funded by the NSF Dimensions of Biodiversity and PIRE awards DEB-1046413 and OISE/IIA-1243541, respectively, the Gordon and Betty Moore Foundation, Investigator Award 3781, and the CIFAR Integrated Microbial Diversity Fellowship IMB-ROHW-141679, all to FR.
matériels
| Name | Company | Catalog Number | Comments |
| Niskin collection and processing units (Hatay Niskins) | Figure 1 | ||
| Filtration setup | Figure 1 | ||
| 32-36% Hydrochloric acid (HCl) | Fisher Sci | A508-212 | |
| High-density polyethylene (HDPE) container | e.g., 5 Gallon HDPE UN Certified Pail with Snap-On Lid | ||
| Combustion oven (550 °C) | |||
| 60 ml HDPE vials | Fisher Sci | 02-896-2B | |
| 25 mm GF/F filter | Fisher Sci | 09-874-64 | |
| Forceps | Fisher Sci | XX62-000-06 | |
| Sterile, non-powdered gloves | Fisher Sci | 19-170-010B | |
| Bilge pump | e.g., Thirsty Mate 124PF | ||
| 20 L collapsible low-density polyethylene carboy | e.g., Reliance Fold A Carrier II | ||
| Tangential flow filter (TFF) ultrafiltration hollow fiber cartridges | Amersham | CFP-2-E-9A | |
| Platinum-cured silicon tubing for TFF | Cole Parmer | 96440-82 | |
| Hose clamps | Cole Parmer | P-68007-23 | |
| Peristaltic pump | Cole Parmer | EW-77410-10 | |
| Sodium hydroxide (NaOH) solution | Electron Microscopy Sciences | 21162 | |
| 0.2 µm Polycarbonate track-etched membrane filter | Fisher Sci | 09-300-61 | |
| 8.0 µm Polycarbonate track-etched membrane filter | Fisher Sci | 09-300-57 | |
| 0.22 µm Polyvinylidene difluoride cylindrical filter | Fisher Sci | SVGP01050 | |
| 0.45 µm Polyvinylidene difluoride cylindrical filter | Fisher Sci | SVHV010RS | |
| Synthetic nylon mesh | Sefar | 03-125-37 | |
| 20 ml Plastic scintillation bottle | Fisher Sci | 03-341-72C | |
| Clean bucket (10-20 L) | |||
| 32% Paraformaldehyde | Electron Microscopy Science | 15714 | |
| 25% Glutaraldehyde | Electron Microscopy Science | 16220 | |
| Liquid nitrogen | |||
| 0.02 µm High-purity alumina matrix filter with annular polypropylene support ring | Fisher Sci | 6809-6002 | |
| 0.2 µm High-purity alumina matrix filter with annular polypropylene support ring | Fisher Sci | 6809-6022 | |
| 10,000x SYBR Gold nucleic acid stain | Invitrogen | S11494 | |
| DAPI solution 25 µg/ml | Invitrogen | D1306 | |
| Molecular grade water | sigma aldrich | W-4502 | |
| Ascorbic acid | Fisher Sci | BP351-500 | |
| Phosphate Buffered Saline (PBS) | Fisher Sci | BP399-500 | |
| Glycerol | Fisher Sci | G31-500 | |
| Microscope slide | Fisher Sci | 12-550-123 | |
| Coverslip | Fisher Sci | 12-548-5M | |
| Delicate task wipe | Fisher Sci | 06-666A | |
| Slide station with vacuum pump | Figure 3 | ||
| DNA extraction kit | Macherey-Nagel | 740952.1 | Nucleospin tissue kit |
| Proteinase K (20 mg/ml) | Lifetechnologies | AM2546 | |
| 200-proof Ethanol | Electron Microscopy Sciences | 15058 | |
| Red cap: Male Luer with Lock Ring x Female Luer Coupler | Cole Parmer | EW-45503-88 | |
| Cesium chloride (CsCl) | Fisher Sci | bp1595-500 | |
| 60 ml Syringe | Fisher Sci | 13-6898 | |
| 0.02 µm Alumina matrix disposable syringe filter | Fisher Sci | O992613 | |
| Ultra-clear centrifuge tubes | Beckman Coulter | 344059 | |
| Ultra-swinging bucket rotor | Beckman Coulter | 333790 | SW41 Ti Rotor |
| DNase I (100 units/µl) | Calbiochem | 260913 | |
| 0.5 M EDTA | Fisher Sci | BP2482-500 | |
| Formamide | Fisher Sci | BP228-100 | |
| Glycogen | sigma aldrich | G-1767 | |
| 1x Tris-EDTA (TE) pH 8.0 | Fisher Sci | BP2473-500 | |
| Sodium Dodecyl Sulfate (SDS) | Fisher Sci | 02674-25 | |
| 20 μg/ml Proteinase K | Fisher Sci | BP1700-500 | |
| Chloroform | Fisher Sci | BP1145-1 | |
| Phenol:Chloroform:Isoamyl alcohol 25:24:1 | sigma aldrich | p3803 | |
| Isopropanol | Fisher Sci | BP2618-500 | |
| 50 ml Oak Ridge tube | Fisher Sci | 05-562-16B |
Références
- Fuhrman, J. A. Marine viruses and their biogeochemical and ecological effects. Nature. 399 (6736), 541-548 (1999).
- Azam, F., Malfatti, F. Microbial structuring of marine ecosystems. Nature Reviews Microbiology. 5 (10), 782-791 (2007).
- Huber, J. A., et al. Microbial population structures in the deep marine biosphere. Science. 318 (5847), 97-100 (2007).
- McDole, T., et al. Assessing coral reefs on a Pacific-wide scale using the microbialization score. PloS ONE. 7 (9), (2012).
- Dinsdale, E. A., et al. Microbial ecology of four coral atolls in the Northern Line Islands. PloS ONE. 3 (2), (2008).
- Nelson, C. E., et al. Coral and macroalgal exudates vary in neutral sugar composition and differentially enrich reef bacterioplankton lineages. The ISME Journal. 7 (5), 962-979 (2013).
- Haas, A. F., et al. Influence of coral and algal exudates on microbially mediated reef metabolism. PeerJ. 1, (2013).
- Smith, J. E., et al. Indirect effects of algae on coral: algae‐mediated, microbe‐induced coral mortality. Ecology Letters. 9 (7), 835-845 (2006).
- Burdige, D. J., Martens, C. S. Biogeochemical cycling in an organic-rich coastal marine basin: 10. The role of amino acids in sedimentary carbon and nitrogen cycling. Geochimica et Cosmochimica Acta. 52 (6), 1571-1584 (1988).
- Capone, D. G., Kiene, R. P. Comparison of microbial dynamics in marine and freshwater sediments: Contrasts in anaerobic carbon catabolism. Limnology and Oceanography. 33 (4), 725-749 (1988).
- Krom, M. D., Sholkovitz, E. R. Nature and reactions of dissolved organic matter in the interstitial waters of marine sediments. Geochimica et Cosmochimica Acta. 41 (11), 1565-1574 (1977).
- Grasshoff, K. Methods of sea-water analysis. Verlag Chemie. , Weinheim and New York. (1976).
- Strickland, J. D. Practical handbook of seawater analysis. Bulletin Fisheries Research Board of Canada. 167, (1968).
- Sharp, J. H., et al. Analyses of dissolved organic carbon in seawater: the JGOFS EqPac methods comparison. Marine Chemistry. 48 (2), 91-108 (1995).
- DeLong, E. F. Microbial population genomics and ecology. Current Opinion in Microbiology. 5 (5), 520-524 (2002).
- Hugenholz, P., Goebel, B. M., Pace, N. R. Impact of Culture-Independent Studies on the Emerging Phylogenetic View of Bacterial Diversity. Journal of Bacteriology. 180 (18), 4765-4774 (1998).
- Breitbart, M., et al. Genomic analysis of uncultured marine viral communities. Proceedings of the National Academy of Sciences. 99 (22), 14250-14255 (2002).
- Willner, D., Hugenholtz, P. From deep sequencing to viral tagging: Recent advances in viral metagenomics. BioEssays. 35, 436-442 (2013).
- Thurber, R. V., Haynes, M., Breitbart, M., Wegley, L., Rohwer, F. Laboratory procedures to generate viral metagenomes. Nature Protocols. 4, 470-483 (2009).
- John, S. G., et al. A simple and efficient method for concentration of ocean viruses by chemical flocculation. Environmental Microbiology Reports. 3, 195-202 (2011).
- Lim, Y. W., et al. The Microbiome and Virome of CF. Pediatric Pulmonology. 47, 321-322 (2012).
- Kelly, L. W., et al. Black reefs: iron-induced phase shifts on coral reefs. The ISME Journal. 6 (3), 638-649 (2012).
- Bjornsen, P. K. Automatic determination of bacterioplankton biomass by image analysis. Applied Environmental Microbiology. 51, 1199 (1986).
- Simon, M., Azam, F. Protein content and protein synthesis rates of planktonic marine bacteria. Marine Ecology Progress Series. 51 (3), 201-213 (1989).
- Hara, S., Koike, I., Terauchi, K., Kamiya, H., Tanoue, E. Abundance of viruses in deep oceanic waters. Marine Ecology Progress Series. 145 (1), 269-277 (1996).
- Wommack, K. E., Colwell, R. R. Virioplankton: viruses in aquatic ecosystems. Microbiology and Molecular Biology Reviews. 64 (1), 69-114 (2000).
- Weinbauer, M. G., Brettar, I., Hofle, M. G. Lysogeny and virus-induced mortality of bacterioplankton in surface, deep, and anoxic marine waters. Limnology and Oceanography. 48 (4), 1457-1465 (2003).
- Clasen, J. L., Brigden, S. M., Payet, J. P., Suttle, C. A. Evidence that viral abundance across oceans and lakes is driven by different biological factors. Freshwater Biology. 53 (6), 1090-1100 (2008).
- Parada, V., Baudoux, A. C., Sintes, E., Weinbauer, M. G., Herndl, G. J. Dynamics and diversity of newly produced virioplankton in the North Sea. The ISME Journal. 2 (9), 924-936 (2008).
- Angly, F. E., et al. The marine viromes of four oceanic regions. PLoS Biology. 4 (11), (2006).
- Hansell, D. A., Carlson, C. A. Deep ocean gradients in dissolved organic carbon concentrations. Nature. 263 (266), (1998).
- Carlson, C. A., et al. Dissolved organic carbon export and subsequent remineralization in the mesopelagic and bathypelagic realms of the North Atlantic basin. Deep Sea Research Part II: Topical Studies in Oceanography. 57 (16), 1433-1445 (2010).
- Sharp, J. H. Improved analysis for particulate organic carbon and nitrogen from seawater. Limnology and Oceanography. 19 (6), 984-989 (1974).
- Ehrhardt, M., Koeve, W. Determination of particulate organic carbon and nitrogen. Methods of Seawater Analysis. Third Edition, 437-444 (2007).
- Haas, A. F., et al. Organic matter release by coral reef associated benthic algae in the Northern Red Sea. Journal of Experimental Marine Biology and Ecology. 389 (1), 53-60 (2010).
- Guildford, S. J., Hecky, R. E. Total nitrogen, total phosphorus, and nutrient limitation in lakes and oceans: Is there a common relationship. Limnology and Oceanography. 45 (6), 1213-1223 (2000).
- Allison, S. D., Chao, Y., Farrara, J. D., Hatosy, S., Martiny, A. C. Fine-scale temporal variation in marine extracellular enzymes of coastal southern California. Frontiers in Microbiology. 3, (2012).
- Van Duyl, F. C., Gast, G. J. Linkage of small-scale spatial variations in DOC, inorganic nutrients and bacterioplankton growth with different coral reef water types. Aquatic Microbial Ecology. 24 (1), 17-26 (2001).
- De Goeij, J. M., Van Duyl, F. C. Coral cavities are sinks of dissolved organic carbon (DOC). Limnology and Oceanography. 52 (6), 2608-2617 (2007).
- De Goeij, J. M., et al. Major bulk dissolved organic carbon (DOC) removal by encrusting coral reef cavity sponges. Marine Ecology Progress Series. 357, 139-151 (2008).
- Mueller, B., et al. Natural diet of coral-excavating sponges consists mainly of dissolved organic carbon (DOC). PLoS ONE. 9 (2), (2014).
- Carlson, C. A., Ducklow, H. W., Hansel, D. A. Organic carbon partitioning during spring phytoplankton blooms in the Ross Sea polynya and the Sargasso Sea. Oceanography. 43 (3), 375-386 (1998).
- Cotner, J. B., et al. Nutrient limitation of heterotrophic bacteria in Florida Bay. Estuaries. 23 (5), 611-620 (2000).
- Willner, D., et al. Metagenomic detection of phage-encoded platelet-binding factors in the human oral cavity. Proceedings of the National Academy of Science USA. 108 (1), 4547-4553 (2011).
- Hall, R. J., et al. Evaluation of rapid and simple techniques for the enrichment of viruses prior to metagenomic virus discovery. Journal of Virological Methods. 195, 194-204 (2013).
- Yang, S. Y., et al. Nanoporous membranes with ultrahigh selectivity and flux for the filtration of viruses. Advanced Materials. 18 (6), 709-712 (2006).
- Feldman, H. A., Wang, S. S. Sensitivity of various viruses to chloroform. Proceedings of the Society for Experimental Biology and Medicine. 106 (4), 736-738 (1961).
- Ackermann, H. W. The Bacteriophages., Second Edition. Calendar, R. , Oxford University Press. New York. 8-16 (2006).
- Henn, M. R., et al. Analysis of High-Throughput Sequencing and Annotation Strategies for Phage Genomes. PLoS ONE. 5 (2), (2010).
- Duhaime, M. B., Deng, L., Poulos, B. T., Sullivan, M. B. Towards quantitative metagenomics of wild viruses and other ultra-low concentration DNA samples: a rigorous assessment and optimization of the linker amplification method. Environmental Microbiology. 14 (9), 2526-2537 (2012).
- Yilmaz, S., Allgaier, M., Hugenholtz, P. Multiple displacement amplification compromises quantitative analysis of metagenomes. Nature Methods. 7 (12), 943-944 (2010).
- Kim, K. H., Bae, J. W. Amplification Methods Bias Metagenomic Libraries of Uncultured Single-Stranded and Double-Stranded DNA Viruses. Applied Environmental Microbiology. 77 (21), 7663-7668 (2011).
- Nelson, C. E., Alldredge, A. L., McCliment, E. A., Amaral-Zettler, L. A., Carlson, C. A. Depleted dissolved organic carbon and distinct bacterial communities in the water column of a rapid-flushing coral reef ecosystem. The ISME Journal. 5 (8), 1374-1387 (2011).
- Kelly, L. W., et al. Local genomic adaptation of coral reef-associated microbiomes to gradients of natural variability and anthropogenic stressors. Proceedings of the National Academy of Sciences. 111 (28), 10227-10232 (2014).
- Lim, Y. W., Haynes, M., Furlan, M., Robertson, C. E., Harris, J. K., Rohwer, F. Purifying the Impure: Sequencing Metagenomes and Metatranscriptomes from Complex Animal-associated Samples. J. Vis. Exp. , (2014).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon