Method Article
水化学と海洋微生物学へのフィールドガイド - 海における幽玄選手の解明
要約
Here, we present a comprehensive protocol to assess the organic and inorganic nutrient availability and the abundance and structure of microbial and viral communities in remote marine environments.
要約
ここでは、遠隔海洋環境での使用に適合徹底的にテストされ、十分に標準化された研究プロトコールのシリーズをご紹介します。サンプリングプロトコルは、微生物群集(溶存有機炭素、粒子状有機物、無機栄養素)への利用可能なリソースの評価、およびダイレクトウイルスおよび微生物数を経由してウイルスおよび細菌コミュニティの包括的な説明(、自家微生物の列挙が含まれ、ウイルスおよび微生物メタゲノムの建設)。我々はすでに確立されたプロトコルと開発された最も最近の技術のいくつかを含んで科学分野の分散フィールドを表す方法の組み合わせを使用しています。ウイルスおよび細菌群集の特徴付けに使用される、特にメタゲノム配列決定技術は、近年でのみ確立されているため、依然として一定の改良が施される。これは、サンプリングの様々な導かれ、試料処理手順のカレンたTLY使用中。ここに提示される方法のセットが収集した環境サンプルを処理する日付のアプローチまで提供する。これらのプロトコルでアドレスパラメータが特徴付け、ウイルスおよび微生物群集ダイナミクスの基礎となるメカニズムを理解するために不可欠な情報の最小をもたらす。これは、総合的な調査を行うためのガイドラインに従うことを容易に与え、重要なステップとそれぞれの技術に関連する潜在的な注意事項について説明します。
概要
海洋生態系は頂点捕食者のバイオマスへの栄養素利用から変化をもたらす摂動の広い範囲に供される。過去10年間、複数の研究では、海洋生態系1-4微生物群集の重要性を実証している。これは、細菌およびウイルスのコミュニティの変化が密接に海洋環境5の全体的な低下と関連していることは明らかである。これらの変化は、酸素の可用性または炭素再石灰6,7として水柱に改変された地球化学によって容易にすることができる。マクロ生物間の相互依存性の結果として、水の化学的性質と微生物活動のフィードバックループは、生態系の劣化8の速度を加速することができる。
1970年代と80年代に多数の試みが海洋生態系9-11に物質循環を解明するために行われた。これらの初期の研究のための主要な課題の一つは不足だった査定生物地球化学的パラメータを測定するための標準化されたアプローチの。主に海水の栄養分12,13上で利用可能なプロトコル、ありましたけれども、炭素動態の評価と微生物群集構造が利用可能なツールおよび方法によって制限されていました。 JGOFS EqPac方法比較すると、シャープら 14は初めて溶存有機炭素(DOC)濃度評価のための信頼性の高い標準化されたプロトコルのために提案した。 2000年代初頭によって(デロング15を参照)微生物群集への利用可能なリソースを決定するための分析的な課題は、このように大部分が解決されていたし、制御培養環境中で微生物群集を特徴付けるためのアプローチが確立されていた。当時の伝統的な配列決定方法は、主に栽培クローンの文化に依存していました。天然試料の初期の環境遺伝子配列は、微生物多様性の大部分はcultivによって見逃されていたことが明らかになったATIONベースの方法16。 2002年には、Breitbart ら 17は、培養されていない海洋ウイルスのコミュニティの全ゲノムを配列決定する方法を確立し、環境海水試料中のウイルスのコミュニティを記述するために初めてショットガンシーケンシングを使用していました。
ほとんどは培養が困難であるとして、既存の微生物アセスメントの中では、ウイルスはまだ、特に下に研究されたままであり、彼らはそのような16SリボソームRNA(rRNAの)は、典型的には、多様性とコミュニティプロファイリングを評価するために使用される遺伝子として普遍的に保存されたマーカーを欠いている。メタゲノムシーケンシングアプローチは、複雑なウイルスのコミュニティを分析するための文化に依存すると遺伝子標的方法に対する代替手段を提供します。海水から生成された最初のウイルスメタゲノムはサンガー配列17を使用して配列決定したが、次世代配列決定技術および改善された分子技術の開発は、メタゲノム研究18の急速な増加につながっているまで。ウイルスメタゲノミクスの現在の研究室のワークフローは、核酸(DNAまたはRNA)を抽出、ライブラリの準備、および配列決定19-21に続くウイルス粒子の分離、濃縮、および/ または濃度を伴う。
水界生態系におけるウイルス、微生物および生化学的機能に対する既存の知識を促進するため、世界中の様々なシステムのデータセットを比較することが不可欠です。しかし、確立されたプロトコルは、主に容易にアクセス可能な研究施設とニアショア環境向けに開発されてきた。したがって、標準化されたフィールド法の欠如は、これらのクロス生態系の比較を妨げる。ここでは、遠隔フィールド位置で使用するための芸術研究プロトコルの状態から徹底的にテストし、うまく標準化された適応をご紹介します。これらの方法は、過去10年間5,22にまたがる複数の研究において成功することが証明され、現在NOAA上だけでなく、様々な海洋研究所によって使用されている太平洋全域4リーフの評価と監視プログラム(RAMP)。サンプリングプロトコルは、水質パラメータ(無機および有機炭素濃度は、無機栄養濃度)、ウイルス及び微生物の存在量(直接細菌数、直接ウイルス数、従属栄養比対独立栄養性を評価するためにフローサイトメトリー)、及びウイルス及び微生物群集構造を含み、メタゲノム解析により代謝電位(概要については図1を参照)。ここに記載された方法から得られた結果を総合的に水界生態系を特徴づけるために必要な鍵生物地球化学的バックグラウンドのパラメータを解明するために必要とされる。
プロトコル
楽器の調製
- ろ過セットアップの準備
- (0.5〜0.6 M;最終溶液5リットルを作るために4.75 Lの蒸留水に250ミリリットル32から36パーセントのHClを追加)を5%塩酸(HCl)溶液を調製する。最大2週間の高密度ポリエチレン容器(HDPE)でこの溶液を保存する。
- 潜在的な溶存有機炭素(DOC)の汚染物質を奪っ5%HCl溶液中で24時間(フィルターを除く)は、サンプルと接触するすべての部品のままにしておきます。各サンプリング事象後に炭素汚染を防止するために約30mlの5%HCl溶液で全ての部およびフラッシュラインをすすぐ( 例えば 、ガス煙、油、ボート、蚊スプレーから)。
- プレ燃焼し、アルミホイルに包んで、または使用直前になるまで蓋をし、すべてのサンプリングアイテムを保持。
- Precombust 25mmのGF / 12時間550℃でマッフル炉中でアルミホイルに包まれたFフィルタは全ての炭素が揮散し、清潔で乾燥し、それらを格納する使用するまで置く。すべての回でprecombustedフィルタを処理するために、酸洗浄鉗子および滅菌、非粉末状の手袋を使用してください。
- ウイルスメタゲノムサンプリング材料の調製
- 徹底的4 20 L折りたたみ式の低密度ポリエチレンカーボイ四20 L高密度ポリエチレンバケット10%漂白剤と蓋を洗う。
- その後、蒸留を3倍した後、水をサンプリングし、スピゴットとカーボイを再シールすべての項目をすすぐ。
- 以下のプロトコルに従って、各使用後に洗浄タンジェンシャルフローフィルター(TFF)、および先制クリーンフィルタは、過去5日間に使用されていない場合。
- 洗浄バケツ水5LにNaOHペレット50gを溶解する。
- チューブに濡れたTFFを取り付け、 図3に従って組み立てる。
- システムを介してのNaOH洗浄溶液の一部を移動するには、[いいえ背圧で〜30rpmで蠕動ポンプを実行します。洗浄は戻りラインから流れたら、バケツを洗浄するために転送を移動溶液の循環を完成させる。
- TFFの下流のリターンライン上のクランプを配置することによって、システムに背圧を適用します。これは、ろ液ラインから洗浄液を強制します。 TFFの外にあふれ、ろ液を生成するために過度の時間(5分)は、劣化したTFFを示す。
- NaOH溶液のすべてがラインになるまでシステムを実行する。次に、背圧を削除するには、システムの外にソリューションを実行して捨てる。
- リターンし、ろ液ラインから流れる水のpHがpHが7-8になるまで背圧で水を実行することにより、TFFをすすぐ。
- 蠕動ポンプおよびTFFからチューブを外します。ストレージ用TFFを再シール。 TFFは、次の使用まで湿ったままであることを確認してください。
2.サンプル採取
注:輸送中に収集されたすべてのサンプルは、冷静さを格納する必要があります(4℃可能であれば)、さらなる処理まで直射日光にさらされていない。
- サンプリングカーボン、栄養素、顕微鏡、フローサイトメトリー、および微生物メタゲノムのため
- (4Lの試料水を生じる)のサンプリングイベントごとに2つのハタイNiskin単位を取る。ちょうど(降順を防ぐことができますそれ以外の場合は捕捉された空気)を沈める前にサンプリング容器を開きます。
- 各サイトでは、両端を開き、水を介して極性軸に沿って円柱を移動させることにより、試料水で採取容器の内部を洗い流す。
- サンプルを取り、慎重にサンプリングコンテナを閉じます。サンプリング部位は、汚染を避けるために、ボートやダイバーから上流にあることを確認します。
- ウイルスメタゲノム解析のためのサンプリング
- 標的部位に20Lのカーボイにビルジポンプをマウントします。
- 対応するインローでインクルード対象の各エリアとシールカーボイでビルジポンプ入口を目指しカーボイを埋める。
- 各サンプリングサイトからフィルタリングされていない海水の4カーボイ(すべて一緒に80 L)を収集します。
3. SAMPル準備
注:汚染の機会を最小限にするため、DOCを皮切りに、ここで紹介するために、サンプルを処理する。全体の手順のために粉末フリーの手袋を使用してください。
- 溶存有機炭素(DOC)
- マウントろ過セットのそれぞれのスロット内の2つのアリコートハタイNiskinユニットの一つ。それぞれのフィルターホルダーにつながる出口チューブを接続します。ハタイNiskinユニットに加圧された空気(0.2バール)チューブを接続します( 図2を参照してください)。
- フィルターホルダーにフィルターを挿入する前に、試料水100mlですべての行をフラッシュします。酸を用いたフィルターホルダーが25mmのプレ燃焼さGF / Fフィルターを置き、鉗子を洗浄した。サンプリング水フィルタリング約20mlの各DOCボトルとキャップを3回すすいでください。
- サンプリングの水を約40ml(〜2/3フル)でボトルを埋める。輸送中に潜在的な汚染や紛失の場合のバックアップを持っているのDOCのサンプルのうち、重複に集まる。
- F分析まで-20℃で直立reezeサンプリングボトル。
- 粒子状有機物(POM)
- DOCのサンプルが収集された後500ミリリットルの合計は、DOCのサンプルを収集しながら通過ものを含め、GF / Fフィルタを経過するまでのフィルタリングを続ける。
- インラインフィルターホルダーを外します。
- 予備燃焼さのアルミニウム箔の正方形の内部に鉗子と場所フィルタを使ってPOM分析用フィルターを取り外し、トップ側自体でオン倍、およびラップ。
- 元素や同位体分析に供されるまで貯蔵のために-20℃でフィルターをフリーズします。
- 無機栄養素
- 各フィルターホルダーに0.2μmのトラックエッチングフィルタを配置します。フィルタカセットを再接続します。
- 各20ミリリットルのプラスチックシンチレーション瓶にサンプリング水で3回すすいでください。肩(〜18ミリリットル)に各ボトルを埋める。
- 後で分析するまで-20℃で凍結します。
- MicroscopY(SYBRゴールドおよびDAPI)
- フィルターホルダーを脱ぐ。
- 2マイクロ遠心管のそれぞれに1mlの水を収集する。
- (後SYBRゴールド染色用)アリコートの一つに66μlの32%パラホルムアルデヒドを追加します。 (後にDAPI染色のために)対応する他に12μlの25%グルタルアルデヒドを追加します。
- 穏やかに混合し、サンプルは暗闇の中で、室温で少なくとも15分間、「修正」することができます。
- フローサイトメトリー
- 破片と大きな真核細胞を除外するためにフィルターホルダーのいずれかで8.0μmのポリカーボネートフィルターを置きます。
- 1ミリリットルの試料と各サンプリングサイトから2クライオバイアルを埋めるために通ってサンプリング水を渡します。
- 各クライオバイアル(最終濃度= 0.125%)を25%グルタルアルデヒドの5μlを添加する。
- ミックスするバイアルを反転。
- サンプルは室温で15〜30分間修正することができます。 30分を超えないようにしてください。
- 液体窒素中でフラッシュ凍結サンプル。
- -80℃またはリキに保管サンプルフローサイトメーター上で分析するまでdの窒素ドライ荷主。
- 微生物メタゲノムサンプル
- ラインフィルタホルダーに取り外します。
- 直接それぞれのライン上に0.22μmの円筒形フィルターをマウントします。
- 一つのフィルターを通して各サイト(フィルターごとに3-4 L合計)の両方からハタイNiskinユニットの試料水を残りのフィルタ。
- 濾過した後、空気で満たさクリーンた10mlシリンジを用いて各フィルタのうち残りの水を押し出す。
- 元の包装にフィルタを配置し、ラボテープでパッケージを密封する。
- -20℃で個別にパックされたフィルターを保管してください。
- ウイルスメタゲノムサンプル
- 何サンプルは環境によって失われたり、汚染されていることを確認するために、すぐに洗浄してバケットにウイルスメタゲノムサンプルを転送します。
- 濃縮前19に残骸および細胞材料を除去するために大きな孔サイズのナイロンメッシュ(25〜100μm)を用いて、プレフィルタ。
- 図3に示すように、TFFを設定するサンプルバケットに供給ラインを配置し、ヒートシンクに実行し、リターンし、ろ液ラインを残す。
- 蠕動ポンプと試料水の1-2 Lと同一面にラインをオンにします。
- 背圧の0.7バーを追加し、サイクルを完了するサンプルバケツに戻りラインを配置します。
- 海水を濃縮する一方で、レベルとしてトップアップサンプルリザーバは低下します。水位が、漂白洗浄、すすぎ、トリプルtripourビーカーにバケット内の吸気ライン、トランスファ濃縮物を下回ったときに集中し続ける。
- リザーバビーカーが空の場合、背圧を取り外し、ポンプ速度を増加させ、ラインを介してサンプル全体を押し、tripourでそれを回収することを含む。
- 任意のウイルスの系統を差別しないが、ほとんどの細菌を除去するために0.45μmの円筒形フィルターを通して濃縮物を渡す。
- すべての150ミリリットルの後に0.45μmの円筒形のフィルターを変更します。 0.45μmの-FILを集める50mlチューブ中のウイルス濃縮物をtered。
- 残留細菌を除去するためにフィルタリング、ウイルス濃縮物のそれぞれ50ミリリットルのアリコートにクロロホルム250μlを添加する。混合するために反転させ、店舗、その後の処理まで4℃で直立。
- 0.45μmのフィルターを乾燥させ、3.6.5と3.6.6の手順で説明するように、それらを格納します。
顕微鏡試料の4処理
注:実行間の潜在的または生物学的ステイン残基を防ぐために、95%エタノールに続いて、10%の漂白剤でフィルターの塔をすすぐ。 1時間内のプロセス顕微鏡サンプルと、可能な場合に点灯するように汚れやステンドフィルタをさらすことは避けてください。
- マウント
- 1×PBS 4.9 mlに10%のアスコルビン酸を100μlを追加し、完全に混合する。
- 100%のグリセロールの5ミリリットルを追加し、完全に混合する。
- フィルタは、-20℃で0.02μmのアルミナマトリックスディスポーザブルシリンジフィルター、アリコートとストアを使用してマウントします。
- SYBRゴールド染色
- マイクロチューブ内最終濃度2%パラホルムアルデヒドで固定し、各サンプルの500μlのアリコートをピペットで。
- アリコートのそれぞれに1,000倍SYBRゴールド溶液0.5μLを加える。
- 穏やかにサンプルを混合し、10分間、暗所に置く。
- 真空ポンプシステムの各フィルタ台に環状ポリプロピレン支持リングと0.02μmのアルミナマトリックスフィルタを配置し、フィルター塔( 図4)に取り付ける。これらのフィルタは二重の多孔性を持っているように0.02μmのフィルターは、フィルタースタンドプラスチック側アップに置かれていることを確認します。
- 真空ポンプの電源を切った状態で、フィルタに均等に分散されているサンプルを確保するために、0.02μmのフィルター、ウイルスフリーの水2mlと一緒に、SYBR染色標本の500μlを添加する。
- フィルタリングされているウイルスや微生物の損傷を防ぐためにこれ以上1バール未満の負圧を作成するために、真空ポンプの電源をオンにします。
- 試料全体が行くことを許可スルー。
- 鉗子(DOCのために使用されるようではないと同じペア)を使用して、慎重にフィルタリング塔から各フィルタを削除し、繊細なタスク上を通過フィルタが乾燥していることを確認するために拭いてください。
- 顕微鏡スライド上のマウントのピペットを10μlと、マウントのドロップに染色されたサンプルを含む乾燥フィルターを配置。
- 10μlをカバースリップを追加する前に、各フィルタの上部にマウントを追加します。
- そっとカバースリップを押します。可能ならば、空気の泡を除去し、カバースリップの動き横向き避けるようにしてください。
- -20℃でslideboxと店舗内の場所スライド。
- DAPI染色
- 各フィルタスタンドに環状のポリプロピレンサポートリングを持つ0.2μmのアルミナマトリックスフィルターを置き、フィルターの塔を添付します。
- 真空ポンプの電源を切った状態で、使用される各塔には0.02μmフィルター、ウイルスを含まない水の2ミリリットルを追加します。
- 総体積oを結果としてピペット各タワーにグルタルアルデヒド固定サンプル1mlのfは各フィルタ塔内の3ミリリットル。
- せいぜい、1バールの負圧を作成するために、真空ポンプの電源をオンにします。
- 0.2μmのアルミナマトリックスフィルター上にサンプル全体をフィルタリングします。
- 慎重に鉗子を用いたフィルタリング塔(DOCのために使用しないように同じペア)からフィルタを削除します。
- 右のアップサイドフィルタを保ちシャーレ内100μlのドロップDAPI溶液[25 / mlの]にフィルターを置き、15分間のサンプル染色をしてみましょう。染色処理中に光を避けてください。
- 15分後、汚れからフィルタを削除繊細なタスクにそれを渡すワイプ乾燥して、再度フィルターを維持し、0.02ミクロンフィルター、ウイルスを含まない水の100μlドロップに転送するために右のアップサイド、DAPI染色を、残りの洗い流し1分間。一度この洗浄を繰り返します。
- SYBRゴールドのサンプルについて説明したように、顕微鏡上のマウント·フィルタは、同一の方法をスライドさせます。
5. DNA抽出
- 微生物のメタゲノムDNA
- RTで少なくとも20分間、円筒形のフィルタを解凍する。
- 10ミリリットルの注射器を使用してフィルタの残りの海水を削除します。キャップ漂白し、オートクレーブキャップを使用して、円筒形フィルターの底端。
- 各フィルタの場合は、360μlのT1」プレ溶解を「ミックスバッファーと50μlのプロテイナーゼK、次にフィルターに混合物をピペット。開口端をキャップ。
- 55℃で、回転オーブンO / N(または少なくとも2時間)で溶解反応をインキュベートする。
- キャップの1つを削除して、200μlのB3の追加」溶菌「プレ溶解サンプルにフィルタにバッファ。再キャップフィルターと70℃で10〜20分間回転させ、オーブン中の混合物をインキュベートする。
- 逆さまのフィルターで液体を吸い出すことで3ミリリットル注射器を用いて、円筒状フィルタからボリューム全体を抽出します。
- 新しいマイクロ遠心チューブに溶解サンプルを追放し、100%エタノール200μlを添加する。
- よく混ぜ、スピンの上に、サンプル全体をロード列、および製造業者のプロトコルによって指示されるように進行する。
- 蛍光ベースのアッセイを用いてDNAを定量化する。
- マリンウイルスメタゲノム
- ファージ粒子(Thurber らから変更。19)を精製し、濃縮
- 1.7グラム/ mLの溶液を調製するために海水中のCsClを溶解さが1.5g / mlで1.35グラム/ mlで1.2グラム/ミリリットル(溶液1mlを秤量することによって較正)。使用前に0.02μmのフィルターを用いて各分画をフィルタリングします。
注:各画分の密度がソリューションを使用する前に、有効数字3桁まで正確であることを確認し、使用前に0.02μmのフィルターを用いて、すべてのソリューションをフィルタリングします。使用海水または海水がそれぞれ低下または密度を増加させるためにCsClを飽和させた。 - セクション4.2で説明したように全ての試薬は、先に進む前に、ウイルスを欠いていることを保証するために組み合わされた試薬の1ミリリットルから顕微鏡スライドを作成します。
- 9グラムのCsClは、ウイルスへのTFF濃縮物の45ミリリットルに追加1.12グラム/ mlの最終濃度。冷蔵のCsCl段階勾配を設定中。
- 1.7グラム/ mlの溶液を用いて開始し、順次、血清学的ピペットを用いてゆっくりと各チューブに密度の低い分、各順次の1ミリリットルを追加します。各個別のメニスカスのレベルをマークし、フラクション間pycnoclinesが乱れていないことを確認。詳細はコンパニオンペーパーを参照してください(LIM ら 2014)。
- サンプルの血清学的ピペット負荷7.5ミリリットルと2時間〜83000×gで、4℃で6管や遠心分離機のそれぞれに。
- 中断することなく、密度勾配がローターをアンロードし、わずか3 mlシリンジに18Gの針を用いて、以前にマークされた1.5グラム/ mlの密度層( 図5)の下にチューブを貫通。精製されたVLPの1.5ミリリットルをオフに描画し、新しいマイクロチューブに移す。すべてのサンプルについて繰り返します。
- 室温で10分間インキュベート激しく混ぜ、精製されたVLPへのクロロホルム0.2ボリュームを追加します。
- 追加150#181;各マイクロチューブに10倍のDNAseバッファlの。
- 5分間、16,000×gでスピンし、水相を集め。クロロホルムをマイクロ遠心チューブの底にない集計を行う場合、よりDNアーゼ緩衝液を追加混合して繰り返す。
- I(最終濃度= 2.5ユニット/μl)をDNアーゼを添加し、15分間65℃でインキュベートすることによって、デオキシリボヌクレアーゼ活性を不活性化、次いで、2時間37℃でインキュベートする。
- 1.7グラム/ mLの溶液を調製するために海水中のCsClを溶解さが1.5g / mlで1.35グラム/ mlで1.2グラム/ミリリットル(溶液1mlを秤量することによって較正)。使用前に0.02μmのフィルターを用いて各分画をフィルタリングします。
- ウイルス様粒子からのDNA抽出(VLPは)
- 均等に2清掃し、オートクレーブオークリッジチューブに各試料から精製されたVLPをプール。
- 0.2MのEDTA、0.01体積の0.5M EDTA、ホルムアミドの1の体積、10μlのグリコーゲンた(10mg / ml)で0.1容量の2Mトリス塩酸(pH値8.5)を追加します。よく混合し、室温で30分間インキュベートする。
- 新しいボリュームを参照すると、2容量のRT 100%エタノールを加える。混合し、30分以上4℃でインキュベートする。
- 4&#で、60分間17200×gでオークリッジチューブをスピニングすることにより、ペレットのDNA176; C。
- 上清を取り除きます。血清学的ピペットを用いて氷冷70%エタノールで2回DNAペレットを洗浄する。
- すべてのエタノール(必要に応じて簡単に再スピニング)、カバーを取り外し、> RTで15分間空気乾燥へのペレットを可能にします。
- 1×TE緩衝液(pH 8.0)567μlの> 15分間、DNAペレットを再懸濁する。新しい1.5 mlのマイクロチューブに再懸濁DNAの567μlのを転送します。
- (使用前に65℃でのプレ暖かい)を10%、30μlのSDSを追加し、プロテイナーゼK(20μg/ ml)を3μlの、完全に混合し、56℃で1時間インキュベートする。
- 5M NaClを100μlのを追加し、完全に混合する。予め温めCTAB NaCl溶液を80μl、渦を追加し、65℃で10分間インキュベートする。
- クロロホルムの容積の0.2部、渦を追加し、2分間16000×gでスピン。新しい1.5 mlのマイクロチューブに上清を移し。
- 、1溶液、混合するボルテックス:24:25アミルフェノール等量のクロロホルムを追加します。そして2分間16000×gでスピン。新しい1.5ミリリットルの微量遠心チューブに上清を移し。
- 、等量のクロロホルムを追加し、ボルテックスし、2分間16,000×gでスピン。新しい1.5ミリリットルの微量遠心チューブに上清を移し。
- 上清画分にイソプロパノール0.7容量を添加し、DNAを沈殿させ、少なくとも30分間、-20℃でインキュベートする。
- 4℃で20分間16,000×gで回転させることによって、DNAをペレット化。上清をピペットし、氷冷70%エタノール500μlでペレットを2回洗浄する。
- 16000×gでスピンし、チューブから残りのエタノールを除去。 15分間、ペレットを空気乾燥。
- 室温で少なくとも5分間、溶出緩衝液(5mMのトリス、pH7.5)中またはTris-EDTA pH8.0の50μlの再懸濁によるDNAペレット。
- 高感度の蛍光に基づくアッセイを用いてDNAを定量する。
- ファージ粒子(Thurber らから変更。19)を精製し、濃縮
結果
顕微鏡検査
顕微鏡試料は、それらが所望の品質であることを確認するためにすぐに分析すべきであることができる。豊かさと細菌群集のサイズ分布を測定するために、DAPIスライドを落射蛍光顕微鏡で検査されます( 図6A)(励起/発光。461分の358 nmの、McDole ら 2012を参照)。細胞数および寸法は、イメージングソフトウェア( 例えば 、イメージプロ(ImagePro)またはImageJの)を使用して収集することができる。長さおよび幅の測定値から細胞体積(V)は、全ての細胞が、以下の式を使用して半球状のキャップをシリンダーの形状を有していることを仮定することによって導出される。
V =π/ 4×W 2(L - / 3 ワット )
Lは長さであり、wは 、各セル23,4の幅である。微生物バイオマスは、その後foの以前に確立サイズ依存関係を用いて推定することができるR海洋微生物群集24。一般的に海洋細菌は、0.1〜4程度の長さの範囲であるが、いくつかの場所で〜8μmのに上がる。
DAPIで染色されたフィルター(0.2μm)が細菌だけを表示しますが、SYBRゴールドステイン(0.02μm)のために使用されるフィルターは、細菌やウイルスが含まれています。ウイルスの存在量を測定する微生物と同じプロトコルに従う、しかし325から375の励起が使用され、発光極大は537 nmの( 図6B)である。
定量的データを生成するために、サンプルボリュームは、元の試料中のウイルスの存在量に応じて調整する必要があるかもしれない。フィルタへの正しいボリュームが最もよく水の所与体について経験的に決定される。ウイルスの濃度を変化させたサンプルを含む顕微鏡写真の例を図7に示されている。
以前の研究からの結果は、微生物にそのウイルスを提案比(VMR)は、一般的に約6サンゴ礁システム(ノウルズ未発表データ)中の平均で、25-29水生システムで1から50までの範囲であり、3〜20で。
フローサイトメトリー
直接カウントおよび微生物群集の大きさの推定に加えて、フローサイトメトリーを介して従属微生物に対する栄養の比率の評価は、さらに収集されたサンプル( 図8に示す例示的なフローサイトメトリー出力)から抽出することができる。総細菌細胞の数を決定するために、サンプルは、SYBRグリーンIで染色し、530/30バンドパスフィルタを有する光電子増倍管は、検出のために使用される。クロロフィルおよびフィコエリトリン(585/42バンドパスフィルタ)(背面675から735ナノメートルの範囲で得られたバックLPミラーへ)チャネルが未染色試料における独立栄養の存在量をカウントするために使用される。従属栄養微生物の豊富さ、非から独立栄養カウントを決定するために、染色された部分は、その後、SYBR染色された合計カウント(McDole ら提出された)から減算される。
ウイルスメタゲノム
ウイルスメタゲノムは、コミュニティの構造と機能を決定するために全ゲノム配列情報を使用して、ウイルスの生態に培養非依存的な分子アプローチを利用する。メタゲノミクスは、地元の17〜30地球規模へのウイルスで社会の複雑さと多様性の我々の理解を進めてきた。ウイルスメタゲノムデータの分析のためのバイオインフォマティクスツールの広い配列の最近の開発は、メタゲノミクスのレンズを通して、より包括的なビューvirosphereのを可能にする、計算ボトルネックを減少している。
ここに提示される方法は、残骸および細胞材料を除去するために大きな孔サイズのナイロンメッシュで濾過前の組み合わせを用いて海水からの濃度のウイルス粒子の単離および濃縮を強調TFF( 図3)より大きな細胞を除去する後続の0.45μmでろ過した試料。この方法は、私たちはより少量で下流プロセスを実行することができ、ほぼ100倍濃縮サンプル( 図5B-5E)を生成します。ウイルス溶解物内の残りの微生物細胞の成長、したがって、フィールド·ステーションと実験の間の長い輸送時間中のサンプルのウイルスの存在量の変化を防止するために、クロロホルムは多くの場合、保存のために2%の最終濃度まで添加される。 VLPの単離および精製に続いて、そのようなSYBRゴールド、核酸色素と落射蛍光顕微鏡検査は、ウイルス粒子( 図5B-5E)の存在および純度を確認するために使用される。
ここでは、南線島サンゴ礁から2つのウイルスメタゲノムを提示し、具体的には、スターバックス(STAR7)とミレニアム(CAR9、以前キャロラインとして知られている)の島。 120 Lサムの総体積PLE水は10メートルの深さで、各サイトから収集し、セクション3.7および5.2で説明したように処理した。パラグラフ4( 図5B)に記載の方法に従って検証として塩化セシウム勾配遠心分離から精製されたVLPはCAR9( 図5D)は3.3×10 8粒子/ mlとSTAR7ために、1mlあたり2.9×10 9粒子であった。 DNAの総量は、ナノドロップ測定値に基づいて、約400 ngのあったこれらのサンプルから単離した。 DNAは、配列データの特性を表1に提示されている。既存のデータベースを使用して、類似性検索に基づくファージPhi29ポリメラーゼを用いて増幅され、2011年に環境ゲノミクスコア施設によって配列決定し、配列の大部分(> 70%)は、多くの場合、結局特徴付けられていない、 すなわち 、未知の起源と機能。同様に、ここで紹介する2 viromesは未知の配列の97%以上を提示した。その結果、非データベース依存解析は、解析のために使用し、(ウイルスメタゲノミクスの「暗黒物質」の研究機会の新しい腕を開いたSeguritan ら 。 印刷中)。
微生物メタゲノム
微生物メタゲノムの解析は、微生物群集存在し、これらのコミュニティの機能説明の特徴づけが可能になる。例としては、macrobial群集構造の変化や利用可能な栄養素と種の豊富さとその優勢な代謝経路(;ケリーら2014。 図9)との間に病原体や病原性5,22や比較の存在の推定値が含まれています。
水化学
水質パラメータは、一般に、所望のデータを生成する大規模な分析処理を行う。 DOC分析用試料は、高温触媒酸化31,3を介して測定される図2に示すように、フローインジェクション分析36,12,37による元素分析33-35、および無機栄養素に結合された同位体比質量分析を介して、粒子状有機物(POM)。すべての分析が正常に完了した後、 表1に示すように、情報は、微生物およびウイルスのコミュニティの特性を補完するために利用可能であろう。この情報は、有機炭素と窒素のサンプルの安定同位体比で賞賛することができ、独立栄養と従属栄養の比(McDole ら提出)(ハースら 35参照 )。
プロトコルセクションに記載されている方法の図1.概要。 このfiguの拡大版をご覧になるにはこちらをクリックしてください再。

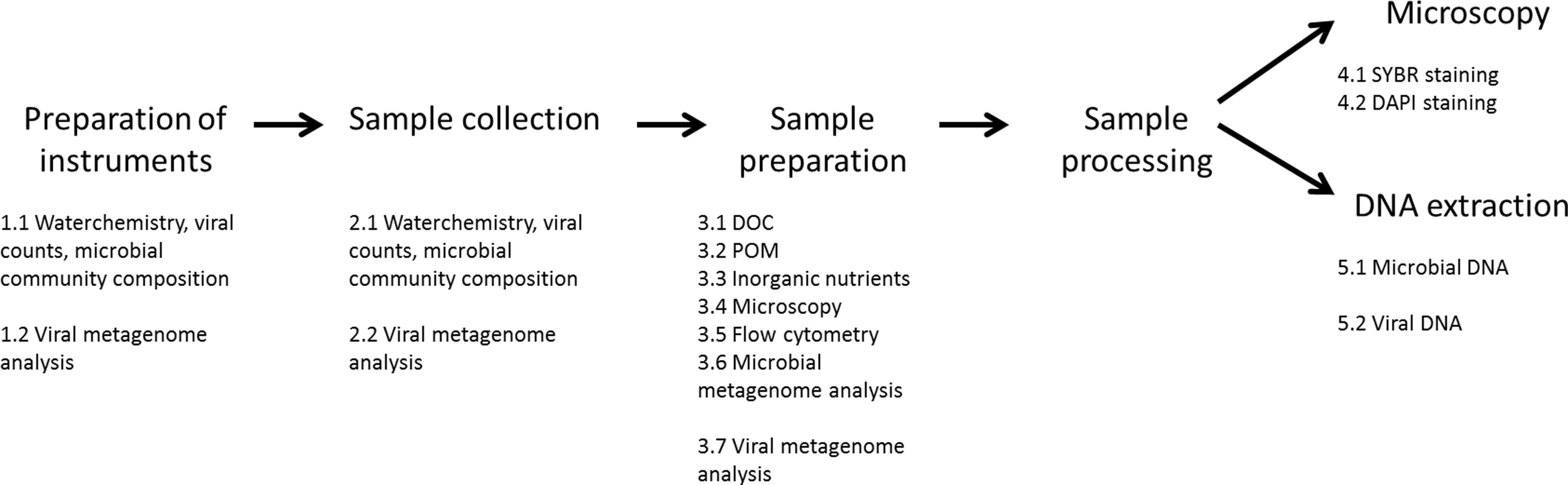
図2.サンプル処理リグのセットアップ。ここで紹介ろ過セットアップ(模式図の左側、実際の画像右側のパネル)は、必ずしもサンプルを生成するために必要とされるものではなく、大幅にプロセスを促進します。セットアップ(1)ハタイNiskinユニットが搭載されたバックプレートから成る。下向きの出口に、ハタイNiskinユニットは、インラインフィルターホルダーにシリコンチューブ(2)に接続されている。これらは、HDPEサンプリングバイアルにそれぞれのフィルタを介してサンプリングされた水のパス(3)の右下に取り付けられてみましょう。濾過プロセスは、圧力計(5)によって調節され、加圧された空気(4)を通って加速される。オーバーフロー盆地はHDPEバイアルまたはPOMろ過の変更中に水をこぼす防ぐことができます。 してくださいこの図の拡大版をご覧になるにはこちらをクリックしてください。

ナイロン(Thurber ら 19から変更)図3の接線流フィルタのラインナップを濾過メッシュ試料水は、接線流フィルタに蠕動ポンプ(2)を介してリザーバ(1)から圧送される(3)。背圧の不在下での戻りライン(4)に沿って貯留槽(1)の試料水に戻る。背圧は、戻りライン上のホースクランプ(5)を介して印加されると、水がフィルタを通過し、廃棄またはろ液リザーバ(6)へ送られる。すべての試料水がチューブラインに集中しているまで、ろ液ラインを集中するように、サンプル水が交換される。
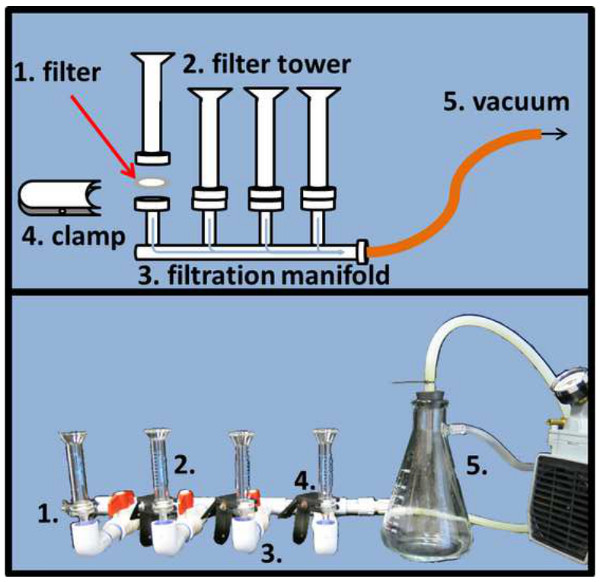
図4.顕微鏡スライドろ過セットアップ。均等に、それぞれアルミナマトリックスフィルタにDAPI及びSYBRゴールドで染色サンプルを配布する(1)、フィルタはクランプ(4)でろ過マニホールドのフィルタ塔(2)とステムの間に配置して固定する必要がある。圧力規制真空(5)は濾過プロセス(概略図トップ、実際の画像下のパネル)を迅速化します。 この図の拡大版をご覧になるにはこちらをクリックしてください。
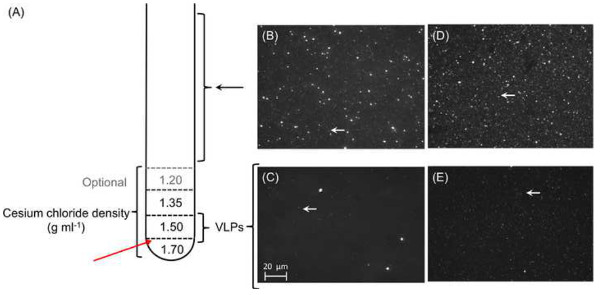
図5.精製し、ファージ粒子の濃度は、それぞれ 、塩化セシウム密度勾配を1mlの合計は、前前処理された試料(A)をリードすると、互いの上に積層されている。各層は、超遠心分離後の抽出を容易にするために、管の外側にマークされるべきである。チューブが元に貫通されます赤い矢印マークVLP画分を消化管。 STAR7(B)とCAR9(D)の0.45μmの濾過された代表的なウイルス濃縮物の落射蛍光顕微鏡写真:ウイルス濃縮物の顕微鏡写真(BE)。唯一のVLP(白矢印)は塩化セシウム勾配超遠心分離後に残るのに対し、細菌細胞を含む大きな粒子は、STAR7(B)とCAR9(D)のサンプルの両方から0.45μmの-濾液から見ることができた。スター7(C)およびCAR9(E)。 この図の拡大版をご覧になるにはこちらをクリックしてください。
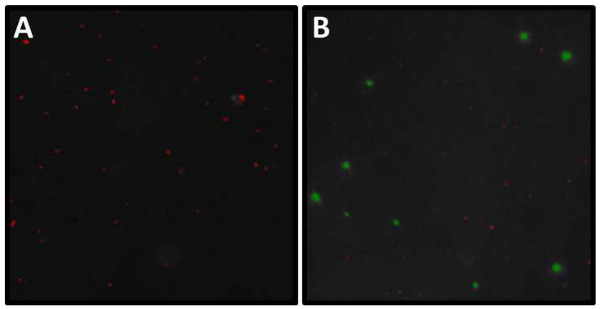
図(A)及びDAPI(B)SYBRゴールドで染色スライド例の分析から、DAPIおよびSYBRゴールド顕微鏡分析。スクリーンの6例 。すべての(A)の長さと幅(μm)は、粒子を強調(=微生物)は画像プログラムの助けを借りて評価することができる。画像の中央に凝集を注意してください。これらのクラスターは、分析の間、ウイルス(B)。その後の分析から除外される必要があり、細菌は、VLPおよび細胞サイズの範囲(赤色VLP、セルラ緑)にサイズのしきい値により選別される必要がある。通常、各撮像プログラムによって未分類一部のかすかなVLPがあることに注意してください。としてかすかな白のドットを現れるこれらのVLPは、手動で数え、自動化されたウイルスのカウントに追加される必要があります。 この図の拡大版をご覧になるにはこちらをクリックしてください。

図SYBRゴールドの7濃度を染色したサンプル。SYBR染色ウイルスサンプルの様々な数字を含む0.02アルミナマトリックスフィルタの顕微鏡写真。パネルAは、AFを示していILTERは、Bに示したフィルタ上の濃度は信頼性の高いカウントのためには低すぎる高すぎるとCであるのに対し、フィルタリング海水の適切な量を含んでいる。 この図の拡大版をご覧になるにはこちらをクリックしてください。
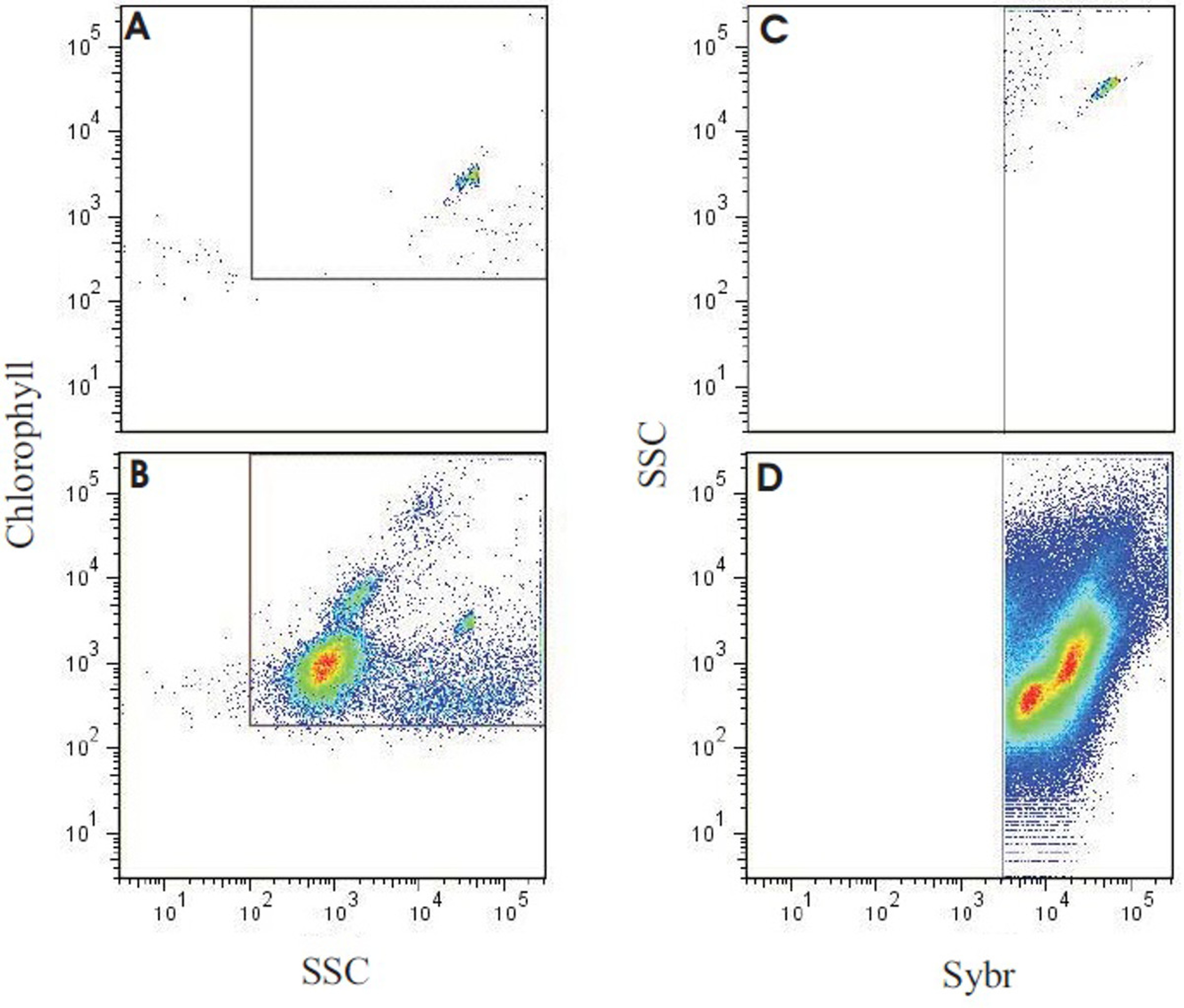
図サンゴ礁-水試料の分析出力フローサイトメトリーの8の結果。パネル(A)は、唯一の機器の設定と最小限の背景を検証するために使用される黄緑色の蛍光マイクロスフェア(0.75ミクロン)で分子グレードの水を示しています。パネル(B)は、ビーズが依然として独立栄養のいくつかの集団と一緒に、同じ場所で見ることができる。(C)A.ノートのように同一の設定やレイアウトを使用して実行リーフ水サンプルの出力を示している、(B)のように>同じサンプル、SYBR陽性事象(独立栄養+従属カウント)をターゲットとし、バックグラウンドを最小限に抑えることができる。(D)を使用してのSYBR Green Iで染色された代表的なサンゴ礁水試料ゲーティング(C)に設立され、総微生物カウントが生成された、とクロロフィル自家蛍光によって仕切ら従属栄養と独立栄養微生物(McDole ら4参照 )。 この図の拡大版をご覧になるにはこちらをクリックしてください。
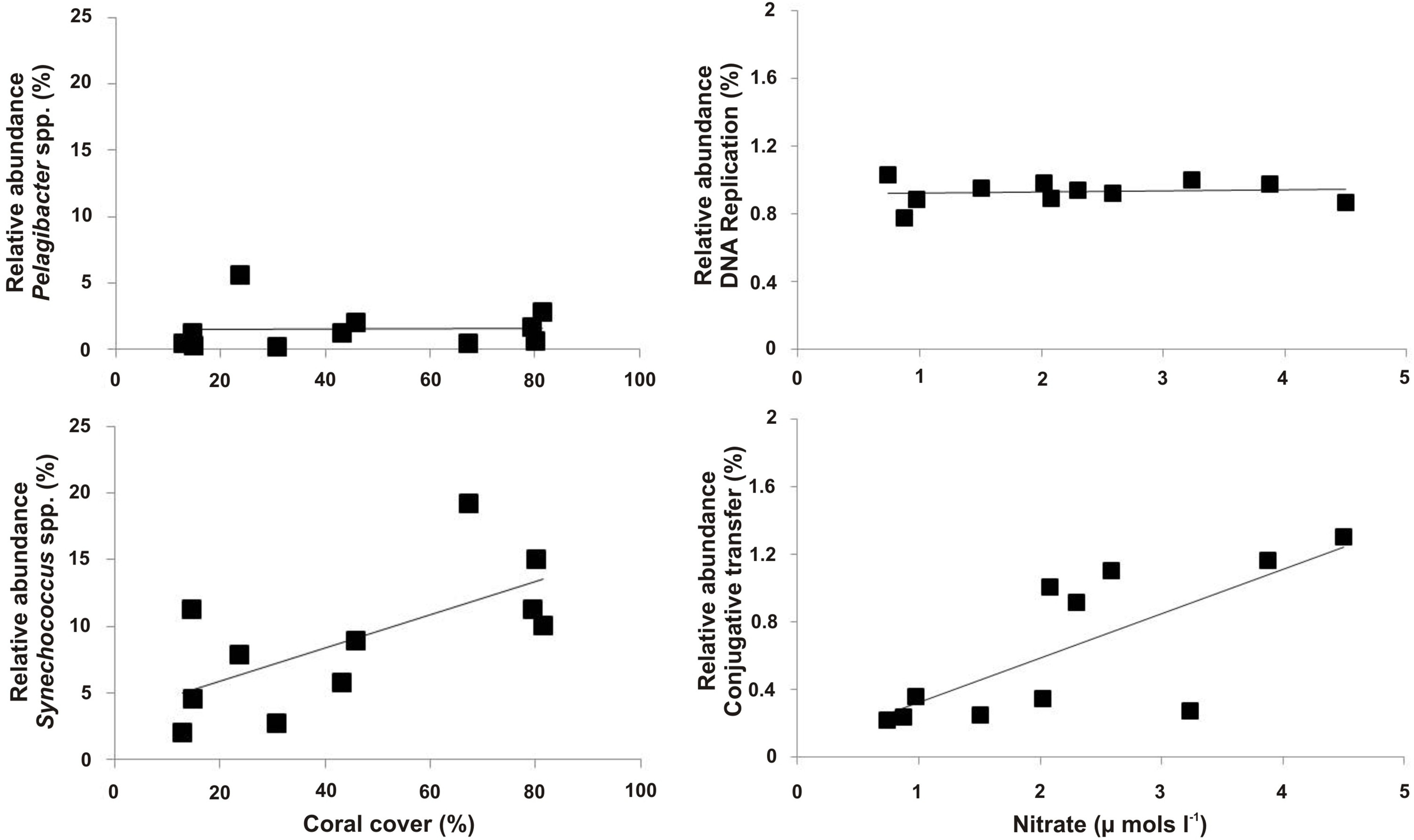
図微生物メタゲノムデータの9例。微生物のメタゲノム配列データとサイト従属変数間のパターン。ここでは、 シネココッカスの相対量属。積極Pelagibacter属のに対し、サンゴのパーセントカバーと相関していた。 (パネル左)ではありませんでした。 DNA修復のための代謝経路は、すべてのメタゲノム(右パネル)の間で一致していた一方、接合伝達のための代謝経路は正に、硝酸塩濃度と相関していた。 この図の拡大版をご覧になるにはこちらをクリックしてください。
| スター7 | CAR9 | |
| の総数は読み込み | 939311 | 591600 |
| プリプロセスの数は、読み込み | 579664 | 360246 |
| シーケンスの長さを意味する(BP) | 395±131 | 451±159 |
| 注釈付き配列(%) | 42218(7.3%) | 9117(2.5%) |
| 不明(%) | 537446(92.7%) | 351129(97.5%) |
南ライン諸島、スターバックス、ミレニアムから生成された2つのviromes表1.データの特性。注釈の程度はMG-RASTサーバーを使用してBLATに基づいています。
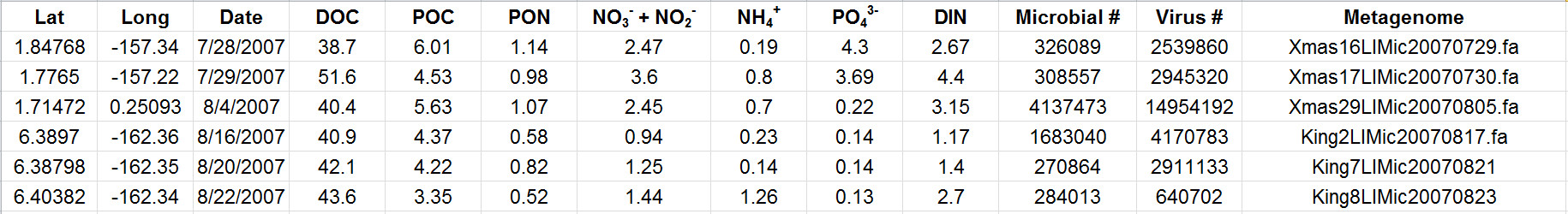
1遠征中に様々なサイトからのデータを示す表2.代表的な結果。ここで評価されたパラメータは、微生物およびウイルスの存在量とペア水質測定を含む。テーブルには、さらに、各サンプリング位置に対するそれぞれのメタゲノムのデータを参照するファイル名が含まれています。炭素と無機栄養濃度がマイクロモル/ Lに記載されている。 してくださいこの表の拡大版をご覧になるにはこちらをクリックしてください。
ディスカッション
ここで紹介する方法は、水生生態系におけるウイルスおよび微生物動態を総合的に評価を生成するためのツールを提供します。これらは微生物群集への利用可能な有機および無機資源の立って株式を定量化すると、ウイルスや微生物群集の存在メーキャップを特徴づけるためにターゲットとしている。次にウイルス豊かさと微生物の豊かさとバイオマスを定量化するために、ゲノム配列情報は、彼らのコミュニティの構造と機能を明らかにしている。すべての方法が開発またはリモートフィールド位置における適用を可能にするように改変されている。しかし制御された実験室の設定の欠如は、本質的なエラーのためのいくつかの可能性が生じる。ここでは、評価されたパラメータのそれぞれに関連付けられたいくつかの注意点について説明します。次汚染の可能性への試料中にも分析プロセスにおいてエラーの可能性をもたらすいくつかのパラメータを処理する。
溶存有機炭素
炭素汚染は(下流サンプリング、またはボートやダイバーの近くで)、試料調製中(機器または不注意な取り扱いの汚染)、さらには、試料の保存中に(他のサンプルを冷凍庫で有機含むサンプリング手順の間に発生する可能性が溶剤/揮発性物質)。各サンプルの再配置は、汚染の追加のソースを提起するように、可能な限り少ない操作ステップとして汚染行為のさまざまな情報源を避けるために。サンプルを洗浄したり燃焼し酸ではない任意の項目と接触して取得するべきではありません。最後に、揮発性有機物質を含有しないサンプル専用のストレージ·冷凍庫を指定すると、より長い貯蔵期間中にサンプルの完全性を確保します。サンプルは、濃HClでDOCサンプルの拡張移動期間酸性化中に凍結保存することができない場合(38〜41に記載のように)適用される代替することができる。測定精度DOCコンセンサス標準物質がregのに使用されるべきであることを確認するにはDOC測定実行中にularly。特に海洋深層水(> 2000メートル)の参照、そのDOCコンテンツ42に非常に安定しているような分析機械の性能を評価する上で貴重である。
粒子状有機物
有機炭素の汚染を回避することに加えて、POMサンプリングを濾過容積の正確さを保証するために特別な注意を必要とする。 500ミリリットルのターゲットボリュームは、潜在的な理由で外れる必要がある場合、これは、それが由来したのろ液に、フィルタ上に捕捉されたPOMの量を関連付けることに注意する必要があります。
無機栄養素
有機炭素のサンプリングと同じように、注意がボートや排水放電12のような様々な情報源で生成された可能性栄養素試料汚染を払わなければならない。 0.8μmの公称孔径を有する有機炭素サンプリングに使用されるフィルタは、すべての微生物バイオマスおよびshを除外しないかもしれないウルドは、したがって、この濾過工程のために0.2μmのフィルタに変更すること。また、検出限界は、栄養サンプルに問題を提起する。特に多くのサンゴ礁の場所( 例えば 、Cotner ら 43)の周りに貧栄養表層水で。検出限界は、おおよその周りに0.1、0.2、およびアンモニウム、硝酸塩や亜硝酸塩、およびオルト-リン酸0.1モル/ Lのそれぞれ(36,12,37を参照)である
顕微鏡検査
顕微鏡スライドからのサンプル画像分析物の濃度の変化の横さらなるエラーの可能性をもたらす。例えば、画像は、視野の一部の地域では、焦点から外れて、その他の焦点である。高純度アルミナマトリックスフィルタは、非常に剛性のフィルタタイプであるように、フィルタの下のごみは、全体のフィルタがスライドに角度の上に座る可能性があります。その結果、画像は、特定のフィルタの視野の様々な部分に焦点から外れ一貫することができ、再顕微鏡整列のgardless。それが観察されている場合、フィルタを再マウント、フィルターの下の確保がきれいで、これを改善することができます。また、場合によっては、ウイルスや微生物が見出されることができる2つの焦点面が存在してもよい。これは実装時のフィルタから離脱しつつ、かつカウントが信頼できないことができますカバースリップの下側に浮上染色オブジェクトの結果である。したがって、常にプロセスの間に、サンプルの品質をチェックすることをお勧めします。
フローサイトメトリー
顕微鏡の試料と同様に、分析プロセスの調整を必要とサンプルをフローサイトメトリーとの間の天然に存在する違いがあるかもしれません。例えば、機器の感度に応じて、薄暗い蛍光Prochlorococcus細胞がノイズレベル以下とすることができ、定量化されない。ビーズは、測定器のRESPことを確認するために、 例えば 、(試料内部対照として役立つ蛍光シグナルへオンセは日から一貫している(そして、実行自体の間に安定したまま)。既知の濃度で試料に加えた場合、彼らはまた、試料体積の実行を確認するための内部対照として役立つことができる。しかし、それは常に96ウェルプレート実行間サンプルの取り扱いのための追加の生物学的コントロールを提供するために、関心のあるサンプルと一緒に解凍し、染色されている以前に凍結された「標準」海水試料のアリコートを実行することをお勧めします。場所によっては、海水試料は、互いに非常に異なって見える場合があります。迅速シネココッカス·エスピーまたは光合成真核生物のいずれかとして植物プランクトンの個体数を確認するには良い方法であることができる「代表」集団をソートして、落射蛍光顕微鏡(EM)の下に表示する。Prochlorococcus細胞は、彼らが速すぎてフェードので、EMの下で、通常は表示されません。また、 シネココッカス·エスピークロロフィルする。また、pHを含めるクロロフィルA(660から700ナノメートル)より短い波長(540から630 nm)の光を発光するotopigment phycoeurythrin(PE)。 シネココッカス細胞は青/緑色光(470〜490ナノメートル)で励起されると510 nm以下の光のすべての波長を取り除く発光フィルターの下で見た場合、彼らは黄金色に表示されます。光の同じ波長で励起されたときの比較により、真核画分は、赤になります。
ウイルスメタゲノム
これは、VLPの濃度および貯蔵の過程を回収コミュニティに影響を与えることに留意することが重要である。ウイルス粒子の損失は、プロセス内のすべての段階で発生できるので、ウイルスの精製および濃縮プロトコルの選択は、最終的には結果としてウイルスメタゲノム44,45,18の観察分類学的組成や多様性に影響を与えることができる。サンプルの濃度は、TFF 19または化学ベースの凝集20を用いて行うことができる。化学ベースのアプローチでは、電着物ively帯電した鉄イオンは自然に負に帯電したウイルス粒子が溶液から凝集し8ミクロンのフィルターを用いて回収することができる大規模な(>8μmの)鉄 - ウイルス複合体を形成する結合する。続いて、鉄は核酸抽出20の濃縮ウイルス粒子を残し、マグネシウム、アスコルビン酸、およびEDTAを使用してウイルス粒子をオフにキレート化され、再溶解される。これら二つの現在の方法を比較した後、我々は、塩化鉄法はTFFウイルス濃度未満で時間がかかるが、いくつかの注意点をもたらすことができると結論付けた。我々は、鉄ウイルスの解離および溶解が、アスコルビン酸が分解する前に進める溶解するために20を報告したよりも2倍以上濃縮されたマグネシウム-アスコルビン酸-EDTA溶液を必要とすることを見出した。さておき、この問題から、このプロトコルは、0.2μmの濾過を用いて微生物の汚染物を除去する、単独のサイズ分画によりウイルス粒子を精製する。大型のウイルスはFILTを通過しないER( 例えば 、Yang ら 46)、従ってメタゲノム中に検出されませんが、いくつかの細菌が行う一方、(McDole、未発表データ)。大ウイルスを差別しながらこれは細菌汚染が生じる。
その特定の問題がしかしTFF濃度との接続中の微生物の除去にも関連しています。クロロホルム処理、外部脂質膜47をクロロホルムに敏感なウイルスを除去することが示されているように、いくつかの研究では、0.22μmのろ過細菌の大部分を除去するために使用できることを示唆している。また、提示された方法は、主に、バクテリオファージ、海洋環境における遺伝物質の最も豊富なキャリアをターゲットに留意すべきである。ファージは、一般的に、それらがクロロホルム処理に対してより耐性である脂質48を一般的に含んでいないと考えられるからである。我々の知る限りでは「キャッチオール」の方法は、これまでに存在しません。ここで紹介する方法は、OUから適応されている北極のシステムにまたがる熱帯様々な海洋環境の最後の10年間のRフィールドでの経験。
微生物メタゲノム
それは、ライブラリ調製に必要な最小限のDNA濃度を生成することが容易であるように、微生物のためのメタゲノムライブラリーの構築( すなわち 、バクテリアや古細菌)ウイルスのメタゲノムよりも簡単です。複数置換増幅(MDA)に基づくリンカー増幅ショットガンライブラリ(LASLs)17,49,50および全ゲノム増幅は、配列決定のために十分なDNAを生成するために、2つの最も一般的に使用される方法である。微生物メタゲノムより高いDNA濃度を得るためにMDAの使用が必要な場合がある。しかし、これはそのような渦鞭毛藻ミニサークルのoveramplificationとして配列データ内のアーチファクトを引き起こす可能性があります。 MDA法は、非定量的な結果51,52、結果として優先的に一本鎖および環状DNAを増幅することが知られている。最適化されたLASLsは50に近づき、微生物および配列決定のためのウイルスメタゲノムDNAの両方を増幅する中、より良い代替手段として役立つことがあります。これはLASLsアプローチは、複数のステップを有する精巧な装置を必要とし、二本鎖DNAテンプレートに限定されていることに留意することが重要である。さらに、組織DNA抽出キットは、グラム陽性およびグラム陰性の門の両方からDNAを抽出することが示されているが、それは効果的に溶解する難治微生物の分類群またはそれらに関連する構造( 例えば 、ある種の古細菌、胞子、または真菌胞子)に検証されていない。したがって、ビーズビーティングステップある種の微生物からDNAを得るために必要であり得る。
これらの総合的な評価は、ウイルスおよび微生物生態系の機能をより良く理解することになります。いくつかの研究が提案されたすべてのパラメータを評価するための努力を行ってきたが、多くは選択されたものを調査してきた。 Nelson ら 53は betw接続を示唆沖合からの相対の両方のDOCとbacterioplankton濃度の具体的なサンゴ礁の生息地や枯渇をEEN。これらの濃度変化がbacterioplanktonコミュニティの明確な分化を伴っていた。 Dinsdale ら 5が増加人為的影響が微生物細胞およびウイルス様粒子の10倍より高い存在量を伴っていたことを示した。有人島を囲む海域における微生物群集も従属し、潜在的な病原体5の大きな画分を持っていた。最後McDole ら 4は、人間活動がmacrobesを犠牲にして、微生物にエネルギーをシフトしていること、microbializationスコアを導入することによって示した。これらの研究は、海洋生態系の生化学的構造に小説洞察を提供、特定の微生物と水の化学パラメータに焦点を当てた。温度およびpH値は、魚のバイオマスと多様化のように - 生態系モニタリング努力の伝統的なデータを組み合わせるTY、底生カバー、またはヒトへの影響への暴露 - 水化学及び微生物アセスメントで、おそらくより特異的に標的保全活動につながる可能性がより敏感な環境モニタリングへの取り組み、になります。
開示事項
The authors have nothing to disclose.
謝辞
We thank Elisha Wood-Charlson, Jackie Mueller, Karen Weynberg, and Kathy Morrow for their help and constructive input on this manuscript. We also thank the crew and captain of the Hanse explorer which provided us with a perfect working environment to conduct large parts of this research. Further we would like to thank the three anonymous reviewers for their time and helpful comments to improve the manuscript. This work was funded by the NSF Dimensions of Biodiversity and PIRE awards DEB-1046413 and OISE/IIA-1243541, respectively, the Gordon and Betty Moore Foundation, Investigator Award 3781, and the CIFAR Integrated Microbial Diversity Fellowship IMB-ROHW-141679, all to FR.
資料
| Name | Company | Catalog Number | Comments |
| Niskin collection and processing units (Hatay Niskins) | Figure 1 | ||
| Filtration setup | Figure 1 | ||
| 32-36% Hydrochloric acid (HCl) | Fisher Sci | A508-212 | |
| High-density polyethylene (HDPE) container | e.g., 5 Gallon HDPE UN Certified Pail with Snap-On Lid | ||
| Combustion oven (550 °C) | |||
| 60 ml HDPE vials | Fisher Sci | 02-896-2B | |
| 25 mm GF/F filter | Fisher Sci | 09-874-64 | |
| Forceps | Fisher Sci | XX62-000-06 | |
| Sterile, non-powdered gloves | Fisher Sci | 19-170-010B | |
| Bilge pump | e.g., Thirsty Mate 124PF | ||
| 20 L collapsible low-density polyethylene carboy | e.g., Reliance Fold A Carrier II | ||
| Tangential flow filter (TFF) ultrafiltration hollow fiber cartridges | Amersham | CFP-2-E-9A | |
| Platinum-cured silicon tubing for TFF | Cole Parmer | 96440-82 | |
| Hose clamps | Cole Parmer | P-68007-23 | |
| Peristaltic pump | Cole Parmer | EW-77410-10 | |
| Sodium hydroxide (NaOH) solution | Electron Microscopy Sciences | 21162 | |
| 0.2 µm Polycarbonate track-etched membrane filter | Fisher Sci | 09-300-61 | |
| 8.0 µm Polycarbonate track-etched membrane filter | Fisher Sci | 09-300-57 | |
| 0.22 µm Polyvinylidene difluoride cylindrical filter | Fisher Sci | SVGP01050 | |
| 0.45 µm Polyvinylidene difluoride cylindrical filter | Fisher Sci | SVHV010RS | |
| Synthetic nylon mesh | Sefar | 03-125-37 | |
| 20 ml Plastic scintillation bottle | Fisher Sci | 03-341-72C | |
| Clean bucket (10-20 L) | |||
| 32% Paraformaldehyde | Electron Microscopy Science | 15714 | |
| 25% Glutaraldehyde | Electron Microscopy Science | 16220 | |
| Liquid nitrogen | |||
| 0.02 µm High-purity alumina matrix filter with annular polypropylene support ring | Fisher Sci | 6809-6002 | |
| 0.2 µm High-purity alumina matrix filter with annular polypropylene support ring | Fisher Sci | 6809-6022 | |
| 10,000x SYBR Gold nucleic acid stain | Invitrogen | S11494 | |
| DAPI solution 25 µg/ml | Invitrogen | D1306 | |
| Molecular grade water | sigma aldrich | W-4502 | |
| Ascorbic acid | Fisher Sci | BP351-500 | |
| Phosphate Buffered Saline (PBS) | Fisher Sci | BP399-500 | |
| Glycerol | Fisher Sci | G31-500 | |
| Microscope slide | Fisher Sci | 12-550-123 | |
| Coverslip | Fisher Sci | 12-548-5M | |
| Delicate task wipe | Fisher Sci | 06-666A | |
| Slide station with vacuum pump | Figure 3 | ||
| DNA extraction kit | Macherey-Nagel | 740952.1 | Nucleospin tissue kit |
| Proteinase K (20 mg/ml) | Lifetechnologies | AM2546 | |
| 200-proof Ethanol | Electron Microscopy Sciences | 15058 | |
| Red cap: Male Luer with Lock Ring x Female Luer Coupler | Cole Parmer | EW-45503-88 | |
| Cesium chloride (CsCl) | Fisher Sci | bp1595-500 | |
| 60 ml Syringe | Fisher Sci | 13-6898 | |
| 0.02 µm Alumina matrix disposable syringe filter | Fisher Sci | O992613 | |
| Ultra-clear centrifuge tubes | Beckman Coulter | 344059 | |
| Ultra-swinging bucket rotor | Beckman Coulter | 333790 | SW41 Ti Rotor |
| DNase I (100 units/µl) | Calbiochem | 260913 | |
| 0.5 M EDTA | Fisher Sci | BP2482-500 | |
| Formamide | Fisher Sci | BP228-100 | |
| Glycogen | sigma aldrich | G-1767 | |
| 1x Tris-EDTA (TE) pH 8.0 | Fisher Sci | BP2473-500 | |
| Sodium Dodecyl Sulfate (SDS) | Fisher Sci | 02674-25 | |
| 20 μg/ml Proteinase K | Fisher Sci | BP1700-500 | |
| Chloroform | Fisher Sci | BP1145-1 | |
| Phenol:Chloroform:Isoamyl alcohol 25:24:1 | sigma aldrich | p3803 | |
| Isopropanol | Fisher Sci | BP2618-500 | |
| 50 ml Oak Ridge tube | Fisher Sci | 05-562-16B |
参考文献
- Fuhrman, J. A. Marine viruses and their biogeochemical and ecological effects. Nature. 399 (6736), 541-548 (1999).
- Azam, F., Malfatti, F. Microbial structuring of marine ecosystems. Nature Reviews Microbiology. 5 (10), 782-791 (2007).
- Huber, J. A., et al. Microbial population structures in the deep marine biosphere. Science. 318 (5847), 97-100 (2007).
- McDole, T., et al. Assessing coral reefs on a Pacific-wide scale using the microbialization score. PloS ONE. 7 (9), (2012).
- Dinsdale, E. A., et al. Microbial ecology of four coral atolls in the Northern Line Islands. PloS ONE. 3 (2), (2008).
- Nelson, C. E., et al. Coral and macroalgal exudates vary in neutral sugar composition and differentially enrich reef bacterioplankton lineages. The ISME Journal. 7 (5), 962-979 (2013).
- Haas, A. F., et al. Influence of coral and algal exudates on microbially mediated reef metabolism. PeerJ. 1, (2013).
- Smith, J. E., et al. Indirect effects of algae on coral: algae‐mediated, microbe‐induced coral mortality. Ecology Letters. 9 (7), 835-845 (2006).
- Burdige, D. J., Martens, C. S. Biogeochemical cycling in an organic-rich coastal marine basin: 10. The role of amino acids in sedimentary carbon and nitrogen cycling. Geochimica et Cosmochimica Acta. 52 (6), 1571-1584 (1988).
- Capone, D. G., Kiene, R. P. Comparison of microbial dynamics in marine and freshwater sediments: Contrasts in anaerobic carbon catabolism. Limnology and Oceanography. 33 (4), 725-749 (1988).
- Krom, M. D., Sholkovitz, E. R. Nature and reactions of dissolved organic matter in the interstitial waters of marine sediments. Geochimica et Cosmochimica Acta. 41 (11), 1565-1574 (1977).
- Grasshoff, K. Methods of sea-water analysis. Verlag Chemie. , Weinheim and New York. (1976).
- Strickland, J. D. Practical handbook of seawater analysis. Bulletin Fisheries Research Board of Canada. 167, (1968).
- Sharp, J. H., et al. Analyses of dissolved organic carbon in seawater: the JGOFS EqPac methods comparison. Marine Chemistry. 48 (2), 91-108 (1995).
- DeLong, E. F. Microbial population genomics and ecology. Current Opinion in Microbiology. 5 (5), 520-524 (2002).
- Hugenholz, P., Goebel, B. M., Pace, N. R. Impact of Culture-Independent Studies on the Emerging Phylogenetic View of Bacterial Diversity. Journal of Bacteriology. 180 (18), 4765-4774 (1998).
- Breitbart, M., et al. Genomic analysis of uncultured marine viral communities. Proceedings of the National Academy of Sciences. 99 (22), 14250-14255 (2002).
- Willner, D., Hugenholtz, P. From deep sequencing to viral tagging: Recent advances in viral metagenomics. BioEssays. 35, 436-442 (2013).
- Thurber, R. V., Haynes, M., Breitbart, M., Wegley, L., Rohwer, F. Laboratory procedures to generate viral metagenomes. Nature Protocols. 4, 470-483 (2009).
- John, S. G., et al. A simple and efficient method for concentration of ocean viruses by chemical flocculation. Environmental Microbiology Reports. 3, 195-202 (2011).
- Lim, Y. W., et al. The Microbiome and Virome of CF. Pediatric Pulmonology. 47, 321-322 (2012).
- Kelly, L. W., et al. Black reefs: iron-induced phase shifts on coral reefs. The ISME Journal. 6 (3), 638-649 (2012).
- Bjornsen, P. K. Automatic determination of bacterioplankton biomass by image analysis. Applied Environmental Microbiology. 51, 1199 (1986).
- Simon, M., Azam, F. Protein content and protein synthesis rates of planktonic marine bacteria. Marine Ecology Progress Series. 51 (3), 201-213 (1989).
- Hara, S., Koike, I., Terauchi, K., Kamiya, H., Tanoue, E. Abundance of viruses in deep oceanic waters. Marine Ecology Progress Series. 145 (1), 269-277 (1996).
- Wommack, K. E., Colwell, R. R. Virioplankton: viruses in aquatic ecosystems. Microbiology and Molecular Biology Reviews. 64 (1), 69-114 (2000).
- Weinbauer, M. G., Brettar, I., Hofle, M. G. Lysogeny and virus-induced mortality of bacterioplankton in surface, deep, and anoxic marine waters. Limnology and Oceanography. 48 (4), 1457-1465 (2003).
- Clasen, J. L., Brigden, S. M., Payet, J. P., Suttle, C. A. Evidence that viral abundance across oceans and lakes is driven by different biological factors. Freshwater Biology. 53 (6), 1090-1100 (2008).
- Parada, V., Baudoux, A. C., Sintes, E., Weinbauer, M. G., Herndl, G. J. Dynamics and diversity of newly produced virioplankton in the North Sea. The ISME Journal. 2 (9), 924-936 (2008).
- Angly, F. E., et al. The marine viromes of four oceanic regions. PLoS Biology. 4 (11), (2006).
- Hansell, D. A., Carlson, C. A. Deep ocean gradients in dissolved organic carbon concentrations. Nature. 263 (266), (1998).
- Carlson, C. A., et al. Dissolved organic carbon export and subsequent remineralization in the mesopelagic and bathypelagic realms of the North Atlantic basin. Deep Sea Research Part II: Topical Studies in Oceanography. 57 (16), 1433-1445 (2010).
- Sharp, J. H. Improved analysis for particulate organic carbon and nitrogen from seawater. Limnology and Oceanography. 19 (6), 984-989 (1974).
- Ehrhardt, M., Koeve, W. Determination of particulate organic carbon and nitrogen. Methods of Seawater Analysis. Third Edition, 437-444 (2007).
- Haas, A. F., et al. Organic matter release by coral reef associated benthic algae in the Northern Red Sea. Journal of Experimental Marine Biology and Ecology. 389 (1), 53-60 (2010).
- Guildford, S. J., Hecky, R. E. Total nitrogen, total phosphorus, and nutrient limitation in lakes and oceans: Is there a common relationship. Limnology and Oceanography. 45 (6), 1213-1223 (2000).
- Allison, S. D., Chao, Y., Farrara, J. D., Hatosy, S., Martiny, A. C. Fine-scale temporal variation in marine extracellular enzymes of coastal southern California. Frontiers in Microbiology. 3, (2012).
- Van Duyl, F. C., Gast, G. J. Linkage of small-scale spatial variations in DOC, inorganic nutrients and bacterioplankton growth with different coral reef water types. Aquatic Microbial Ecology. 24 (1), 17-26 (2001).
- De Goeij, J. M., Van Duyl, F. C. Coral cavities are sinks of dissolved organic carbon (DOC). Limnology and Oceanography. 52 (6), 2608-2617 (2007).
- De Goeij, J. M., et al. Major bulk dissolved organic carbon (DOC) removal by encrusting coral reef cavity sponges. Marine Ecology Progress Series. 357, 139-151 (2008).
- Mueller, B., et al. Natural diet of coral-excavating sponges consists mainly of dissolved organic carbon (DOC). PLoS ONE. 9 (2), (2014).
- Carlson, C. A., Ducklow, H. W., Hansel, D. A. Organic carbon partitioning during spring phytoplankton blooms in the Ross Sea polynya and the Sargasso Sea. Oceanography. 43 (3), 375-386 (1998).
- Cotner, J. B., et al. Nutrient limitation of heterotrophic bacteria in Florida Bay. Estuaries. 23 (5), 611-620 (2000).
- Willner, D., et al. Metagenomic detection of phage-encoded platelet-binding factors in the human oral cavity. Proceedings of the National Academy of Science USA. 108 (1), 4547-4553 (2011).
- Hall, R. J., et al. Evaluation of rapid and simple techniques for the enrichment of viruses prior to metagenomic virus discovery. Journal of Virological Methods. 195, 194-204 (2013).
- Yang, S. Y., et al. Nanoporous membranes with ultrahigh selectivity and flux for the filtration of viruses. Advanced Materials. 18 (6), 709-712 (2006).
- Feldman, H. A., Wang, S. S. Sensitivity of various viruses to chloroform. Proceedings of the Society for Experimental Biology and Medicine. 106 (4), 736-738 (1961).
- Ackermann, H. W. The Bacteriophages., Second Edition. Calendar, R. , Oxford University Press. New York. 8-16 (2006).
- Henn, M. R., et al. Analysis of High-Throughput Sequencing and Annotation Strategies for Phage Genomes. PLoS ONE. 5 (2), (2010).
- Duhaime, M. B., Deng, L., Poulos, B. T., Sullivan, M. B. Towards quantitative metagenomics of wild viruses and other ultra-low concentration DNA samples: a rigorous assessment and optimization of the linker amplification method. Environmental Microbiology. 14 (9), 2526-2537 (2012).
- Yilmaz, S., Allgaier, M., Hugenholtz, P. Multiple displacement amplification compromises quantitative analysis of metagenomes. Nature Methods. 7 (12), 943-944 (2010).
- Kim, K. H., Bae, J. W. Amplification Methods Bias Metagenomic Libraries of Uncultured Single-Stranded and Double-Stranded DNA Viruses. Applied Environmental Microbiology. 77 (21), 7663-7668 (2011).
- Nelson, C. E., Alldredge, A. L., McCliment, E. A., Amaral-Zettler, L. A., Carlson, C. A. Depleted dissolved organic carbon and distinct bacterial communities in the water column of a rapid-flushing coral reef ecosystem. The ISME Journal. 5 (8), 1374-1387 (2011).
- Kelly, L. W., et al. Local genomic adaptation of coral reef-associated microbiomes to gradients of natural variability and anthropogenic stressors. Proceedings of the National Academy of Sciences. 111 (28), 10227-10232 (2014).
- Lim, Y. W., Haynes, M., Furlan, M., Robertson, C. E., Harris, J. K., Rohwer, F. Purifying the Impure: Sequencing Metagenomes and Metatranscriptomes from Complex Animal-associated Samples. J. Vis. Exp. , (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved