Method Article
Entwirren der unsichtbaren Spieler im Ozean - ein Feld Führer zu Wasserchemie und Marine Mikrobiologie
In diesem Artikel
Zusammenfassung
Here, we present a comprehensive protocol to assess the organic and inorganic nutrient availability and the abundance and structure of microbial and viral communities in remote marine environments.
Zusammenfassung
Hier stellen wir eine Reihe von sorgfältig getestet und gut standardisierte Forschung Protokolle für den Einsatz in abgelegenen Meeresumgebungen angepasst. Die Probenahmeprotokolle umfassen die Beurteilung der zur Verfügung stehenden Ressourcen der mikrobiellen Gemeinschaft (gelöster organischer Kohlenstoff, partikulären organischen Substanz, anorganische Nährstoffe) und eine umfassende Beschreibung der viralen und bakteriellen Gemeinschaften (über direkte virale und Keimzahlen, Aufzählung autofluoreszierenden Mikroben und Bau von viralen und mikrobiellen Metagenomen). Wir verwenden eine Kombination von Verfahren, die eine dispergierte Gebiet der wissenschaftlichen Disziplinen umfasst bereits etablierten Protokollen und einige der neuesten Techniken entwickelt darzustellen. Besonders metagenomische Sequenzierungstechniken für virale und bakterielle Gemeinschaft Charakterisierung verwendet, haben erst in den letzten Jahren etabliert und werden daher noch zu ständigen Verbesserung unterzogen. Dies hat zu einer Vielzahl von Probennahme und Probenverarbeitung Verfahren curren führtently in Gebrauch. Der Satz von hier vorgestellten Methoden bietet eine aktuelle Ansatz zu sammeln und zu verarbeiten Umweltproben. Parameter mit diesen Protokollen angesprochen ergeben das Minimum auf wesentliche Informationen zu charakterisieren und zu verstehen, die zugrunde liegenden Mechanismen der viralen und mikrobiellen Gemeinschaft Dynamik. Es gibt einfach zu Richtlinien, umfassende Umfragen durchzuführen, folgen und diskutiert kritische Schritte und mögliche Vorbehalte relevant für jede Technik.
Einleitung
Meeresökosysteme sind auf ein breites Spektrum von Störungen, die in Veränderungen von Nährstoffverfügbarkeit auf die Biomasse des Topraeuber führen ausgesetzt. Im Laufe des letzten Jahrzehnts haben mehrere Studien die Bedeutung der mikrobiellen Gemeinschaften in marinen Ökosystemen 4.1 demonstriert. Es ist offensichtlich, dass Veränderungen in der bakteriellen und viralen Gemeinschaft sind eng mit der allgemeinen Verschlechterung der Meeresumwelt 5 verbunden. Diese Veränderungen können durch veränderte geochemischen in der Wassersäule, wie die Verfügbarkeit von Sauerstoff oder Kohlenstoff Remineralisierung 6,7 erleichtert. Als Folge der Wechselwirkung zwischen dem Makroorganismen, Wasserchemie und mikrobielle Aktivität Rückkopplungsschleifen können die Rate der Verschlechterung des Ökosystems 8 beschleunigen.
In den 1970er und 80er Jahren wurden mehrere Versuche unternommen, um Stoffkreisläufe in marinen Ökosystemen 9-11 entwirren. Eine der größten Herausforderungen für diese frühen Studien war der Mangelstandardisierter Ansätze, die beurteilt biogeochemische Parameter zu messen. Zwar gab es Protokolle zur Verfügung, vor allem auf Meerwasser Nährstoffe 12,13 wurden die Bewertung der Kohlenstoffdynamik und der mikrobiellen Gemeinschaftsstruktur durch die Werkzeuge und Methoden zur Verfügung beschränkt. Mit der JGOFS EQPac Methoden Vergleich Sharp et al. 14 vorgeschlagen, zum ersten Mal eine zuverlässige und standardisierten Protokoll für gelöste organische Kohlenstoff (DOC) Konzentration Abschätzungen. Durch den frühen 2000er Jahren die analytischen Herausforderungen zur Verfügung stehenden Ressourcen der mikrobiellen Gemeinschaft war damit weitgehend gelöst zu bestimmen und Ansätze zur mikrobiellen Gemeinschaften in kontrollierten Kulturumgebungen charakterisieren gegründet worden war (siehe DeLong 15). Die traditionellen Sequenzierungsverfahren zu diesem Zeitpunkt weitgehend verließ sich auf kultivierte klonalen Kulturen. Frühumwelt Gen-Sequenzierung von natürlichen Proben zeigten jedoch, dass die Mehrheit der mikrobiellen biologischen Vielfalt war von cultiv verpasst wordenation-basierte Methoden 16. Im Jahr 2002 Breitbart et al. 17 verwendet Shotgun-Sequenzierung zum ersten Mal, um die virale Community im Umweltseewasserproben zur Festlegung eines Verfahrens, das gesamte Genom von unkultivierten Meeres viralen Gemeinden sequenzieren zu beschreiben.
Innerhalb der bestehenden mikrobiellen Assessments, bleiben Viren besonders untersuchte, wie die meisten schwierig zu kultivieren sind und es fehlt ein universell konservierten Marker wie die 16S ribosomale RNA (rRNA) Gene in der Regel für die Beurteilung der Vielfalt und Gemeinschaft Profilierung eingesetzt. Die metagenomic Sequenzierung Ansatz bietet eine Alternative zur Kultur abhängig und Gen-Targeting Verfahren zur Analyse komplexer viraler Gemeinden. Während die ersten viralen Metagenomen aus Meerwasser erzeugt wurden mittels Sanger-Sequenzierung 17 sequenziert, hat die Entwicklung der nächsten Generation Sequenzierungstechnologien und verbesserte molekulare Techniken, um einen raschen Anstieg der metagenomische Studien 18 LED . Die aktuelle Labor-Workflow für die virale Metagenomik beinhaltet die Trennung, Anreicherung und / oder Konzentration von Viruspartikeln, gefolgt von Nukleinsäure (DNA oder RNA) Extraktion, Bibliothek Vorbereitung, und Sequenzierung 19-21.
Um das vorhandene Wissen auf die virale, mikrobielle und biochemische Funktion in aquatischen Ökosystemen zu fördern, den Vergleich von Daten-Sets in verschiedenen Systemen auf der ganzen Welt ist unerlässlich. Doch die etablierten Protokollen weitgehend für Nearshore-Umgebungen mit leicht zugänglichen Laboreinrichtungen entwickelt worden. Damit ist der Mangel an standardisierten Feldmethoden behindert diese Quer Ökosystem Vergleiche. Hier stellen wir ausführlich getestet und gut standardisiert Anpassungen aus dem Stand der Technik Forschungsprotokolle für den Einsatz in Fernfeldpositionen. Diese Methoden wurden in mehreren Studien überspannt den letzten zehn Jahren 5,22 bewährt und werden derzeit von verschiedenen Meereslabors auf der NOAA verwendet sowieReef Abschätzung und Monitoring Program (RAMP) in der gesamten pazifischen Ozean 4. Die Probenahmeprotokolle sind Wasser-chemischen Parametern (anorganische und organische Kohlenstoffkonzentrationen, anorganischen Nährstoffkonzentration), virale und mikrobielle Abundanz (direkte Keimzahlen, direkte virale zählt, und Durchflusszytometrie zu autotroph vs heterotroph Kennziffern analysieren) und viralen und mikrobiellen Gemeinschaftsstruktur und Stoffwechselpotenzial durch Metagenom-Analyse (für eine Übersicht siehe Abbildung 1). Die aus den hier beschriebenen Methoden erhaltenen Ergebnisse sind, um die Schlüssel biogeochemischen Hintergrund Parameter benötigt, um umfassend zu charakterisieren aquatischen Ökosystemen aufzuklären erforderlich.
Protokoll
1. Herstellung der Instrumente
- Vorbereitung der Filtration-Setup
- Eine 5% ige Salzsäure (HCl) -Lösung (0,5-0,6 M; fügen 250 ml 32-36% HCl auf 4,75 L destilliertem Wasser auf 5 l Endlösung zu machen). Diese Stammlösung wird in einer hochdichten Polyethylenbehälter (HDPE) für bis zu 2 Wochen.
- Verlassen alle Teile, die in Kontakt mit der Probe (mit Ausnahme der Filter) kommen wird 24 h in 5% HCl-Lösung auf Blutegel potentielle gelöstem organischen Kohlenstoff (DOC) Verunreinigungen. Nach jeder Probenahme Ereignis gründlich alle Teile und bündig Linien mit etwa 30 ml 5% HCl-Lösung auf Kohlenstoffverunreinigung zu verhindern (zB von Abgasen, Öl, Boote, Mückenspray).
- Bewahren Sie alle Sampling Artikel im vorge verbrannt Aluminiumfolie eingewickelt oder verkappt, bis unmittelbar vor der Verwendung.
- Precombust 25 mm GF / F-Filter in Aluminiumfolie eingewickelt in einem Muffelofen bei 550 ° C für 12 h bis verflüchtigen alle Kohlenstoff und speichern sie in einem sauberen, trockenenlegen bis zur Verwendung. Verwenden Säure gewaschen Pinzette und steril, nicht gepuderte Handschuhe, um die precombusted Filter jederzeit umgehen.
- Herstellung von Viral Metagenom Sampling Materialien
- Gründlich waschen vier 20 L zusammenklappbar Polyethylen niedriger Dichte Glasballons und vier 20 l Polyethylen hoher Dichte Eimer und Deckel mit 10% Bleichmittel.
- Anschließend spülen Sie alle Artikel 3x mit destilliertem und dann Probenwasser und verschließen Kanister mit Hahn.
- Wasch Tangential Flow-Filter (TFF) nach jedem Gebrauch nach dem folgenden Protokoll und sauber präventiv, wenn der Filter nicht in den letzten 5 Tagen verwendet wurden.
- Auflösen von 50 g NaOH-Pellets in 5 L Wasser in einem Wascheimer.
- Bringen Sie einen nassen TFF auf den Schlauch und montieren nach Abbildung 3.
- Laufen peristaltischen Pumpe bei ~ 30 min ohne Gegendruck etwas von der NaOH-Waschlösung durch das System zu bewegen. Sobald der Wasserfluss aus dem Rücklauf, bewegen Transfer zum Eimer zu waschenum die Zirkulation von Lösung zu vervollständigen.
- Gegendruck anzuwenden, um das System durch Anordnen einer Klemme auf der Rücklaufleitung stromab des TFF. Dies wird Waschlösung aus dem Filtrat Linie zwingen. Viel Zeit (5 Minuten), um die Außenseite des TFF fluten und produzieren Filtrat indikativ einer abgebauten TFF.
- Führen des Systems, bis das gesamte NaOH Lösung ist in den Zeilen. Dann entfernen Sie Gegendruck, führen Lösung aus dem System entfernen und entsorgen.
- Spülen Sie die TFF durch fließendes Wasser unter Gegendruck, bis der pH-Wert des Wassers aus den Rücklauf und Filtratleitungen fließende pH 7-8.
- Schlauch entfernen aus dem Schlauchpumpe und TFF. Reseal TFF für die Lagerung. Sicherzustellen, dass die TFF naß bleibt bis zum nächsten Einsatz.
2. Probenentnahme
HINWEIS: Während des Transports alle gesammelten Proben sollten kühl gelagert werden (wenn möglich 4 ° C) und bis zur weiteren Verarbeitung nicht dem direkten Sonnenlicht ausgesetzt ist.
- Samplingfor Carbon, Nährstoffe, Mikroskopie, Durchflusszytometrie und Mikrobielle Metagenomics
- Nehmen Sie zwei Hatay Niskin Einheiten pro Probenahme Ereignis (was in 4 L Probenwasser). Öffnen Sie die Probenahmegefäße kurz vor Untertauchen (sonst eingeschlossene Luft wird absteigend verhindern).
- Am jeweiligen Standort, spülen Sie das Innere des Probengefäß mit der Probe Wasser durch Öffnen beider Enden und Bewegen des Zylinders entlang der polaren Achse durch das Wasser.
- Nehmen Sie die Probe und Schließen Sie vorsichtig die Probenbehälter. Sicherstellen, dass die Probenahmestelle stromaufwärts von Boot und Taucher, um eine Kontamination zu vermeiden.
- Die Probenahme für Viral Metagenomics
- Auf der Zielstelle montieren Lenzpumpe auf 20 L Glasballon.
- Füllen Sie carboy Ziel Lenzpumpe Einlass an der jeweiligen Zielregion und Dichtung Glasballon mit der entsprechenden Zapfen.
- Sammle 4 Ballonflaschen von ungefiltertem Meerwasser (alle zusammen 80 L) aus jeder Probenahmestelle.
3. Sample Vorbereitung
HINWEIS: Verarbeiten Sie die Proben in der hier angegebenen Reihenfolge, beginnend mit DOC, um die Wahrscheinlichkeit einer Kontamination zu minimieren. Verwenden puderfrei Handschuhe für das gesamte Verfahren.
- Gelöstem organischen Kohlenstoff (DOC)
- Halterung einer der beiden Aliquote Hatay Niskin Einheiten in entsprechenden Schlitz des Filtersatzes. Schließen Sie den Ablaufschlauch an die jeweilige Filterhalter führt. Schließen Sie die Druckluft (0,2 bar) Schlauch an den Hatay Niskin Einheit (siehe Abbildung 2).
- Spülen Sie alle Zeilen mit 100 ml Probenwasser vor dem Einsetzen Filter in den Filterhalter. Zeigen 25 mm vor, verbrannt GF / F-Filter in Filterhalter mit Hilfe der Säure gewaschen Pinzette. Spülen Sie jede DOC Flasche und Verschluss 3-mal mit etwa 20 ml gefilterten Messwasser.
- Füllen Flasche mit ca. 40 ml (~ 2/3 voll) der Probenahme Wasser. Sammeln Sie mindestens Duplikate DOC Proben, um ein Backup im Falle eines potentiellen Kontamination oder Verlust während des Transports haben.
- FReeze Probenflaschen aufrecht stehend bei -20 ° C bis zur Analyse.
- Particulate Organic Matter (POM)
- Nach DOC Proben gesammelt wurden weiterhin Filterung, bis insgesamt 500 ml haben die GF / F-Filter geleitet, einschließlich dessen, was durchlaufen, während das Sammeln DOC Proben.
- Abschrauben Inline Filterhalter.
- Entfernen Sie den Filter für POM-Analyse mit einer Pinzette und Ort Filter in einem Quadrat von vorgebrannten Aluminiumfolie, falten top-Seite gedreht in sich selbst, und wickeln.
- Filter bei -20 ° C zur Lagerung bis zu elementarem und Isotopenanalyse unterzogen.
- Anorganische Nährstoffe
- Legen Sie eine 0,2 um spurgeätzten Filter in jedem Filterhalter. Bringen Sie die Filterkassetten.
- Spülen je 20 ml Kunststoff Scintallationsflasche dreimal mit Messwasser. Füllen Sie jede Flasche an der Schulter (~ 18 ml).
- Bei -20 ° C, bis eine spätere Analyse.
- Microscopy (SYBR Gold-und DAPI)
- Take off Filterhalter.
- Sammeln von 1 ml Wasser in jeden von zwei Mikrozentrifugenröhrchen.
- Fügen 66 ul 32% Paraformaldehyd zu einer der Teilmengen (für spätere SYBR Gold-Färbung). Werden 12 & mgr; l 25% Glutaraldehyd zu der entsprechenden anderen (später DAPI-Färbung).
- Vorsichtig mischen und ermöglichen Proben zu "reparieren" für mindestens 15 min bei RT im Dunkeln.
- Durchflusszytometrie
- Ein 8,0 & mgr; m Polycarbonat-Filter in einer der Filterhalter um Schmutz und großen eukaryotischen Zellen auszuschließen.
- Pass Probenahme Wasser durch bis 2 Kryoröhrchen aus jeder Probenahmestelle mit 1 ml Probe füllen.
- In 5 ul 25% Glutaraldehyd zu jedem Kryoröhrchen (Endkonzentration = 0,125%).
- Kehren Fläschchen zu mischen.
- Die Probe wird für 15-30 Minuten bei RT zu reparieren. Sie 30 Minuten nicht überschreiten.
- Blitzeis Proben in flüssigem Stickstoff.
- Proben bei -80 ° C oder in Liquid Stickstoff trocken Versender bis zur Analyse mit einem Durchflusszytometer.
- Mikrobielle Metagenomanalyse Probe
- Entfernen Sie in Zeile Filterhaltern.
- Direktmontage eines 0,22 um zylindrische Filter auf die jeweilige Zeile.
- Verbleibende Probenwasser sowohl Hatay Niskin Einheiten aus jeder Website-Filter (in Höhe von insgesamt 3-4 l pro Filter) durch einen Filter.
- Nach der Filtration schieben das restliche Wasser aus jedem Filter mit einem sauberen 10-ml-Spritze mit Luft gefüllt.
- Zeigen Filter wieder in Originalverpackung und verschließen Sie das Paket mit Laborband.
- Bewahren einzeln verpackt Filter bei -20 ° C.
- Viral Metagenomanalyse Probe
- Übertragen viralen Metagenoms Proben sofort in den gewaschenen Schaufeln sicherzustellen keine Probe wird durch die Umgebung verloren oder verunreinigt.
- Vorfilter mit großer Porengröße Nylon-Mesh (25-100 & mgr; m), um Schmutz und Zellmaterial vor Konzentration 19 zu entfernen.
- Die TFF auf, wie in Abbildung 3 dargestellt, indem die Förderleitung in einer Probe Eimer, und verlassen die Rückkehr und Filtratleitungen läuft in ein Waschbecken.
- Schalten Sie Schlauchpumpe und bündig Linien mit 1-2 l Probenwasser.
- Zeigen Rückleitung in der Probe Eimer Zyklus abzuschließen, fügen Sie 0,7 bar Gegendruck.
- Und konzentriert sich das Meerwasser nachfüllen Probenreservoir wie das Niveau sinkt. Wenn Wasserstand unter der Saugleitung in den Eimer, Trägerkonzentrat in eine Bleich-gewaschen, dreimal gespülte tripour Becher und weiter konzentriert.
- Wenn Reservoir Becher leer ist, ziehen Gegendruck, erhöhen die Pumprate und drücken Sie die gesamte Probe durch die Leitungen, erholt es im tripour.
- Übergeben Sie das Konzentrat durch 0,45 um zylindrische Filter können die meisten Bakterien zu entfernen, während nicht gegen virale Linien Unterscheiden.
- Ändern 0,45 um zylindrische Filter nach jeweils 150 ml. Sammeln Sie die 0,45 um-filNamenviruskonzentrat in 50-ml-Röhrchen.
- In 250 ul Chloroform je 50 ml Aliquot filtriert Viruskonzentrat, um restliche Bakterien zu eliminieren. Kehren Sie zu mischen und zu speichern, aufrecht bei 4 ° C bis zur nachfolgenden Verarbeitung.
- Trocknen Sie die 0,45 um Filter und lagern Sie sie wie in den Schritten 3.6.5 und 3.6.6 beschrieben.
4. Verarbeitung von Mikroskopie-Proben
HINWEIS: Spülen Filtertürme mit 10% Bleichmittel, gefolgt von 95% Ethanol, um mögliche Flecken oder biologische Rückstände zwischen den Läufen zu verhindern. Prozess Mikroskop Proben innerhalb 1 Stunde und vermeiden Flecken und gefärbten Filter können Sie, wenn möglich, zu beleuchten.
- Berg
- 100 l 10% Ascorbinsäure auf 4,9 ml 1x PBS, und gründlich mischen.
- 5 ml 100% Glycerin, und gründlich mischen.
- Filtergewinde mit einem 0,02 um Aluminiumoxid-Matrix Einwegspritzenfilter, aliquotieren und bei -20 ° C.
- SYBRGoldfärbung
- Pipettieren 500 & mgr; l-Aliquot jeder mit 2% Endkonzentration Paraformaldehyd in einem Mikrozentrifugenröhrchen befestigt Probe.
- Mit 0,5 ul 1,000x SYBR Gold-Lösung zu jedem der Aliquots.
- Mischprobe vorsichtig und legen Sie sie in der Dunkelheit für 10 min.
- Legen Sie eine 0,02 um Aluminiumoxid-Matrix-Filter mit Ring Polypropylen Stützring auf jedem Filter Stand der Vakuumpumpsystem und befestigen Sie die Filtertürme (Abbildung 4). Sicherzustellen, dass die 0,02-um-Filter auf dem Filterstandkunststoffseite nach oben gelegt, da diese Filter eine doppelte Porosität.
- Mit der Vakuumpumpe abgeschaltet wird, fügen Sie die 500 ul SYBR gefärbte Probe, zusammen mit 2 ml von 0,02 um gefiltert virenfrei Wasser, um die Probe zu gewährleisten wird gleichmäßig über den Filter verteilt.
- Einschalten der Vakuumpumpe auf einen Unterdruck von nicht mehr als 1 bar, um eine Beschädigung Viren und Mikroben gefiltert erstellen.
- Lassen Sie die gesamte Probe zu gehendurch.
- Mit einer Pinzette (nicht dasselbe Paar wie für DOC verwendet), sorgfältig jedes Filter von Filterturm zu entfernen, und übergeben Sie über eine heikle Aufgabe zu wischen, um sicherzustellen, der Filter trocken ist.
- Je 10 ul des Berges auf einen Objektträger und legen Sie die Trockenfilter den verschmutzten Probe auf dem Tropfen Halterung enthalten.
- Werden 10 & mgr; l zu montieren, um der Oberseite jeder Filter bevor ein Deckglas.
- Drücken Sie vorsichtig das Deckglas nach unten. Versuchen Sie, Luftblasen, wenn möglich zu beseitigen und zu vermeiden Seitwärtsbewegung des Deckglases.
- Den Objektträger in einer slidebox und bei -20 ° C.
- DAPI-Färbung
- Legen Sie eine 0,2 um Aluminiumoxid-Matrix-Filter mit Ring Polypropylen Stützring auf jedem Filter Ständer und befestigen Sie die Filtertürme.
- Mit der Vakuumpumpe ausgeschaltet, 2 ml von 0,02 um gefiltert virenfrei Wasser auf jedem Turm verwendet.
- Je 1 ml der glutaraldehydfixierten Probe in jedem Turm, was einem Gesamtvolumen of 3 ml in jeder Filterturm.
- Einzuschalten Vakuumpumpe auf einen Unterdruck von nicht mehr als 1 bar erzeugen.
- Filtern gesamte Probe auf die 0,2 um Aluminiumoxid-Matrix-Filter.
- Filter vorsichtig aus Filterturm mit einer Pinzette (nicht dasselbe Paar wie für DOC verwendet).
- Setzen Sie den Filter auf einer 100 ul Tropfen DAPI-Lösung [25 ug / ml] in einer Petrischale halten den Filter wieder richtig herum und ließ die Probe Fleck für 15 min. Licht während des Färbungsprozesses zu vermeiden.
- Nach der 15 min, entfernen Sie den Filter aus dem Fleck, geben sie über einen heiklen Aufgabe Tuch zu trocknen und in ein 100 ul Tropfen von 0,02 um gefiltert virenfrei Wasser, wieder halten die Filter wieder richtig herum, abzuwaschen restlichen DAPI-Färbung für 1 min. Wiederholen Sie diesen waschen.
- Berg Filter auf Objektträger die gleiche Weise wie für die SYBR Gold-Proben beschrieben.
5. DNA-Extraktion
- Mikrobielle Metagenom-DNA
- Auftauen der zylindrische Filter für mindestens 20 min bei RT.
- Entfernen Sie alle restlichen Meerwasser in den Filter mit einem 10-ml-Spritze. Verschließe das untere Ende des zylindrischen Filters unter Verwendung eines gebleichten und autoklaviert Kappe.
- Für jeden Filter, Mischungs 360 ul T1 "pre-Lyse" Puffer und 50 & mgr; l Proteinase K und pipettieren die Mischung in den Filter. Verschließen Sie die offenen Ende.
- Inkubiere die Lyse-Reaktion in einem Drehofen O / N (oder mindestens 2 h) bei 55 ° C.
- Entfernen Sie eine der Kappe und mit 200 ul B3 "Lyse" Puffer in den Filter an den Pre-Lyse Probe. Re-Kappenfilter und Inkubieren der Mischung in einem Drehofen für 10-20 min bei 70 ° C.
- Extrahieren des gesamten Volumens aus dem zylindrischen Filter mit einem 3-ml-Spritze durch Ansaugen der Flüssigkeit aus dem Filter Kopf.
- Vertreiben die lysierte Probe in ein neues Mikrozentrifugenröhrchen und mit 200 ul 100% Ethanol.
- Gut mischen, laden die gesamte Probe auf die SpinSpalte und gehen Sie durch Protokoll des Herstellers angewiesen.
- Quantifizierung DNA unter Verwendung eines fluoreszenzbasierten Assays.
- Meeres Viral Metagenomics
- Reinigung und Konzentrierung Phagenpartikel (von Thurber et al modifiziert. 19)
- Aufzulösen CsCl in Meerwasser, Lösungen von 1,7 g / ml herzustellen, 1,5 g / ml, 1,35 g / ml, 1,2 g / ml (kalibriert durch Auswiegen von 1 ml der Lösung). Filtern jede Fraktion mit 0,02 um-Filter vor der Verwendung.
HINWEIS: Stellen Sie sicher, dass die Dichte jeder Fraktion ist genau auf drei signifikante Stellen, bevor Sie die Lösungen und alle Lösungen filtern mit einem 0,02 um-Filter vor dem Gebrauch. Verwenden Sie Meerwasser oder Meerwasser mit CsCl gesättigt zu senken oder zu erhöhen Dichte auf. - Einen Objektträger aus 1 ml kombinierten Reagenzien wie in Abschnitt 4.2 beschrieben, um sicherzustellen, dass alle Reagenzien sind frei von Viren, bevor Sie fortfahren.
- Hinzufügen 9 g CsCl und 45 ml Viruskonzentrat TFFeine endgültige Dichte von 1,12 g / ml. Kühlen Sie beim Einrichten der CsCl Stufengradienten.
- Beginnend mit der 1,7 g / ml-Lösung nacheinander mit 1 ml jeder sequentiell weniger dichte Fraktion langsam in jedes Röhrchen mit serologischen Pipetten. Markieren Sie die Ebene der einzelnen Meniskus und sicherzustellen, dass die pycnoclines zwischen den Fraktionen nicht gestört werden. Weitere Einzelheiten finden Sie im Begleitpapier (Lim et al. 2014).
- Mit einer serologischen Pipette Last 7,5 ml Probe in jede der 6 Röhrchen und Zentrifugieren bei ~ 83.000 × g, 4 ° C für 2 Stunden.
- Ohne Unterbrechung des Dichtegradienten Entladen der Rotor und das Rohr durchstechen gerade unterhalb des zuvor markierten 1,5 g / ml Dichte Schicht (5) mit einer 18 G Nadel auf eine 3 ml Spritze. Beziehen Sie 1,5 ml gereinigtem VLPs und Transfer zu einem neuen Reaktionsgefäß. Wiederholen Sie dies für alle Proben.
- In 0,2 Volumen Chloroform zu den gereinigten VLPs, kräftig mischen, Inkubation bei RT für 10 min.
- In 150 &# 181; l 10x DNAse Puffer zu jedem Reaktionsgefäß.
- Spin bei 16.000 × g für 5 min und Sammeln der wässrigen Phase. Wenn das Chloroform nicht aggregiert in dem Boden des Mikrozentrifugenröhrchens, fügen mehrere DNAse-Puffer, zu mischen und zu wiederholen.
- In DNase I (Endkonzentration = 2,5 Einheiten / ul) und bei 37 ° C für 2 Stunden, dann inaktivieren DNase-Aktivität durch Inkubation bei 65 ° C für 15 min.
- Aufzulösen CsCl in Meerwasser, Lösungen von 1,7 g / ml herzustellen, 1,5 g / ml, 1,35 g / ml, 1,2 g / ml (kalibriert durch Auswiegen von 1 ml der Lösung). Filtern jede Fraktion mit 0,02 um-Filter vor der Verwendung.
- DNA Extraktion aus Viral-ähnliche Partikel (VLPs)
- Gleichmäßig bündeln die gereinigten VLPs aus jeder Probe in zwei gereinigt und autoklaviert Oak Ridge Röhren.
- Mit 0,1 Volumen 2 M Tris-HCl (pH 8,5) mit 0,2 M EDTA, 0,01 Volumen 0,5 M EDTA, 1 Volumen Formamid, 10 & mgr; l Glycogen (10 mg / ml). Gut mischen und Inkubation bei RT für 30 min.
- Bezugnehmend auf neues Volume, fügen 2 Bände RT 100% Ethanol. Mischen und Inkubieren bei 4 ° C für mehr als 30 min.
- Pellet DNA, durch Drehen der Oak Ridge-Röhrchen bei 17.200 × g für 60 min bei 4 & #176; C.
- Entfernen Sie den Überstand. Zweimal waschen das DNA-Pellet mit eiskaltem 70% Ethanol mit serologischen Pipetten.
- Entfernen Sie das gesamte Ethanol (re-Spinnerei kurz falls erforderlich), Deckel, und ermöglichen Pellet an der Luft trocknen bei RT> 15 min.
- Resuspendieren der DNA-Pellet zu> 15 min in 567 ul 1X TE Puffer (pH 8,0). Übertragen Sie die 567 ul resuspendierten DNA in ein neues 1,5 ml Reaktionsgefäß.
- In 30 ul 10% SDS (pre-warmen bei 65 ° C vor der Verwendung) und 3 ul Proteinase K (20 ug / ml), gut mischen und Inkubation für 1 h bei 56 ° C.
- 100 l 5 M NaCl und gründlich mischen. Werden 80 & mgr; l vorgewärmtem CTAB NaCl-Lösung, Vortex und Inkubation für 10 min bei 65 ° C.
- In 0,2 Teile Volumen Chloroform, Vortex und Spin bei 16.000 × g für 2 min. Überstand in ein neues 1,5 ml Reaktionsgefäß.
- Hinzufügen gleichen Volumen Phenol-Chloroform-Isoamylalkohol 25: 24: 1-Lösung, Vortex zu mischen,und Spin bei 16.000 × g für 2 min. Den Überstand in ein neues 1,5 ml Reaktionsgefäß.
- Hinzufügen gleichen Volumen Chloroform, Wirbel und Schleudern bei 16.000 xg für 2 min. Den Überstand in ein neues 1,5 ml Reaktionsgefäß.
- Hinzufügen von 0,7 Volumen Isopropanol zu dem Überstandsfraktion und Inkubation bei -20 ° C für mindestens 30 min, um DNA auszufällen.
- Pellet die DNA durch Zentrifugieren bei 16.000 xg für 20 min bei 4 ° C. Pipettieren des Überstandes und zweimal waschen das Pellet mit 500 ul eiskaltem 70% Ethanol.
- Spin bei 16.000 xg und entfernen Sie das restliche Ethanol aus der Tube. Luft trocknen den Pellet für 15 min.
- Resuspendieren DNA-Pellet mit 50 & mgr; l Elutionspuffer (5 mM Tris, pH 7,5) oder Tris-EDTA pH 8,0 für mindestens 5 Minuten bei RT.
- Quantifizierung der DNA mit hoher Empfindlichkeit auf Fluoreszenz basierenden Assay.
- Reinigung und Konzentrierung Phagenpartikel (von Thurber et al modifiziert. 19)
Ergebnisse
Mikroskopie
Mikroskopie Proben können und sollten sofort untersucht werden, um sicherzustellen, dass sie in der gewünschten Qualität sind. Um Fülle und Größenverteilung der Bakteriengemeinschaft zu messen, wird DAPI gleitet durch Epifluoreszenzmikroskopie untersucht werden (Anregung / Emission: 358/461 nm, siehe McDole et al 2012). (6A). Zellzahlen und Abmessungen können mit Bildbearbeitungssoftware (zB ImagePro oder ImageJ) gesammelt werden. Aus Länge und Breite Zellvolumen (V) werden, indem die Annahme, dass alle Zellen, die die Form von Zylindern mit halbkugelförmigen Kappen Verwendung der folgenden Gleichung abgeleitet sind:
V = π / 4 × B 2 (L - w / 3)
wobei L die Länge und die Breite w ist jeder Zelle 23,4. Mikrobielle Biomasse kann dann mit vorher festgelegten größenabhängigen Beziehungen fo geschätzt werdenr marine mikrobielle Gemeinschaften 24. Generell marinen Bakterien haben eine Länge von 0,1 bis 4 um, sondern gehen bis ~ 8 um in einigen Orten.
Während die Filter (0,2 um) mit DAPI gefärbt wird nur Bakterien zeigen, die für die SYBR Gold-Färbung (0,02 um) verwendeten Filter enthalten Bakterien und Viren. Messen der Häufigkeit von Viren nach dem gleichen Protokoll wie für Mikroben jedoch eine Anregung 325-375 nm verwendet werden und das Emissionsmaximum liegt bei 537 nm (Abbildung 6B).
Um quantitative Daten zu erzeugen, muss die Probenmenge in Abhängigkeit von der viralen Hülle und Fülle in der ursprünglichen Probe angepasst werden. Die richtige Lautstärke zu filtern wird am besten empirisch für eine bestimmte Wasserkörper bestimmt. Beispiele von Mikroaufnahmen enthaltenden Proben mit unterschiedlichen Viruskonzentrationen sind in Figur 7 dargestellt.
Ergebnisse aus früheren Studien legen nahe, dass Virus MicrobeVerhältnisse (VMR) reichen im Allgemeinen von 1 bis 50 in aquatischen Systemen 25-29, und in zwischen 3 und 20, mit einem Durchschnitt von etwa 6 in Korallenriffsystemen (Knowles unveröffentlichte Daten).
Durchflusszytometrie
Zusätzlich zu den direkten zählt und Größenabschätzung der mikrobiellen Gemeinschaft können Einschätzungen des Verhältnisses von autotrophen zu heterotrophen Mikroben über Durchflusszytometrie weiter von den gesammelten Proben extrahiert werden (beispielhafte Durchflusszytometrie in Abbildung 8 angegebenen Ausgang). Um die Anzahl der Gesamtbakterienzellen zu bestimmen, werden Proben mit SYBR Green I gefärbt und eine Photovervielfacherröhre mit einer 530/30 Bandpass-Filter wird zur Detektion verwendet. Ein Kanal für Chlorophyll (Rücken an Rücken LP Spiegel, was zu einer Reihe von 675 bis 735 nm) und Phycoerythrin (585/42 Bandpassfilter) wird verwendet, um die Fülle der autotrophs in ungefärbten Proben zählen. Um die Fülle der heterotrophen Mikroben, die autotrophen zählt von dem nicht festzustellen,Bunt Teil werden dann aus dem SYBR-gefärbten Gesamtkeimzahl (McDole et al. eingereicht) subtrahiert.
Viral Metagenomics
Viral Metagenomik nutzt eine kulturunabhängige molekular Ansatz zur viralen Ökologie, mit der gesamten genomischen Sequenzinformation zur Gemeinschaft Struktur und Funktion zu bestimmen. Metagenomics wurde Erweiterung der Kenntnisse über die Komplexität und Vielfalt der viralen Gemeinden auf lokaler 17 bis globaler Ebene 30. Die jüngste Entwicklung einer breiten Palette von bioinformatischen Tools für die Analyse von viralen metagenomische Daten Rechenengpässe verringert, so dass für eine umfassendere Sicht der der virosphere durch die Linse der Metagenomik.
Die hier vorgestellten Methoden markieren die Isolierung und Anreicherung von Viruspartikeln aus Meerwasser unter Verwendung einer Kombination aus Vorfiltration mit großer Porengröße Nylon Netz um Schmutz und Zellmaterial zu entfernen, KonzentrationProben mit TFF (Figur 3) und eine anschließende Filtration 0,45 um zu größeren Zellen zu entfernen. Dieses Verfahren erzeugt etwa eine 100x konzentrierte Probe (5B-5E), die uns an nachgelagerte Prozesse in kleineren Volumina durchführen können. Um das Wachstum der verbleibenden mikrobiellen Zellen in der viralen Lysat zu verhindern und daher Änderungen in der viralen Hülle und Fülle der Probe während der langen Laufzeit zwischen Bodenstation und Labor wird oft Chloroform bis zu einer Endkonzentration von 2% für die Lagerung gegeben. Nach der Isolierung und Reinigung von VLPs, Epifluoreszenzmikroskopie mit Nukleinsäure-Farbstoffe, wie SYBR Gold werden verwendet, um das Vorhandensein und die Reinheit der Viruspartikel (5B-5E) zu verifizieren.
Hier präsentieren wir zwei virale Metagenomen aus der südlichen Linie Insel Korallenriff, gesagt, die Starbuck (STAR7) und Millennium (CAR9; zuvor als Caroline bekannt) Inseln. Ein Gesamtvolumen von 120 L sample Wasser wurde von jedem Standort in einer Tiefe von 10 Metern erfasst und verarbeitet werden, wie in Kapitel 3.7 und 5.2 beschrieben. Die aus dem Cäsiumchlorid-Gradienten-Zentrifugation gereinigt VLPs wurden 3,3 x 10 8 Teilchen / ml für CAR9 (5D) und 2,9 x 10 9 Partikel pro ml für STAR7, wie nach dem in Absatz 4 (5B) beschriebenen Verfahren überprüft. Die Gesamtmenge der DNA aus diesen Proben isoliert wurden, wurden etwa 400 ng, bezogen auf das Nanodrop Messung. DNA wurde mit Phi29 Polymerase amplifiziert und durch die Environmental Genomics Core Facility in 2011 sequenziert Die Eigenschaften der Sequenzdaten ist in Tabelle 1 dargestellt. Basierend auf Ähnlichkeitssuchen mit bestehenden Datenbanken, die Mehrheit (> 70%) der Sequenzen oft landeten uncharakterisierte, dh unbekannter Herkunft und Funktionen. Ebenso sind die beiden hier dargestellten viromes präsentiert mehr als 97% der unbekannten Sequenzen. Als Ergebnis nicht-Datenbankabhängige Analyse wurde für die Analyse verwendet und eine neue Arm der Forschung Gelegenheit eröffnet auf der "dunklen Materie" im viralen Metagenomik (Seguritan et al. im Druck).
Mikrobielle Metagenomics
Die Analyse der mikrobiellen Metagenomen ermöglicht die Charakterisierung der mikrobiellen Gemeinschaft und der Funktionsbeschreibung dieser Gemeinschaften. Beispiele dafür sind Schätzungen des Vorhandensein von Krankheitserregern und Virulenz 5,22 oder Vergleiche zwischen den Veränderungen in macrobial Gemeinschaftsstruktur oder verfügbaren Nährstoffe und Fülle von Arten und ihre vorherrschende Stoffwechselwege (Abbildung 9; Kelly et al 2014)..
Wasserchemie
Wasserchemie Parameter der Regel die umfangreichen analytischen Verarbeitung für den gewünschten Daten zu erzeugen; Proben für die DOC-Analyse wird über Hochtemperatur-katalytische Oxidation 31,3 gemessen werden2, partikulären organischen Substanz (POM) über Isotopenverhältnis-Massenspektrometrie zu einem Elementaranalysator 33-35 und anorganische Nährstoffe von Flow Injection Analysis 36,12,37 verbunden. Nach einem erfolgreichen Abschluss aller Analysen werden in Tabelle 1 gezeigt Information verfügbar sein, um die Charakterisierung der mikrobiellen und viralen Gemeinschaften ergänzen. Diese Informationen können mit stabilen Isotopenverhältnisse des organischen Kohlenstoffs und Stickstoffproben ergänzt werden (siehe Haas et al. 35) und die Verhältnisse zwischen autotrophs und heterotrophs (McDole et al. Eingereicht).
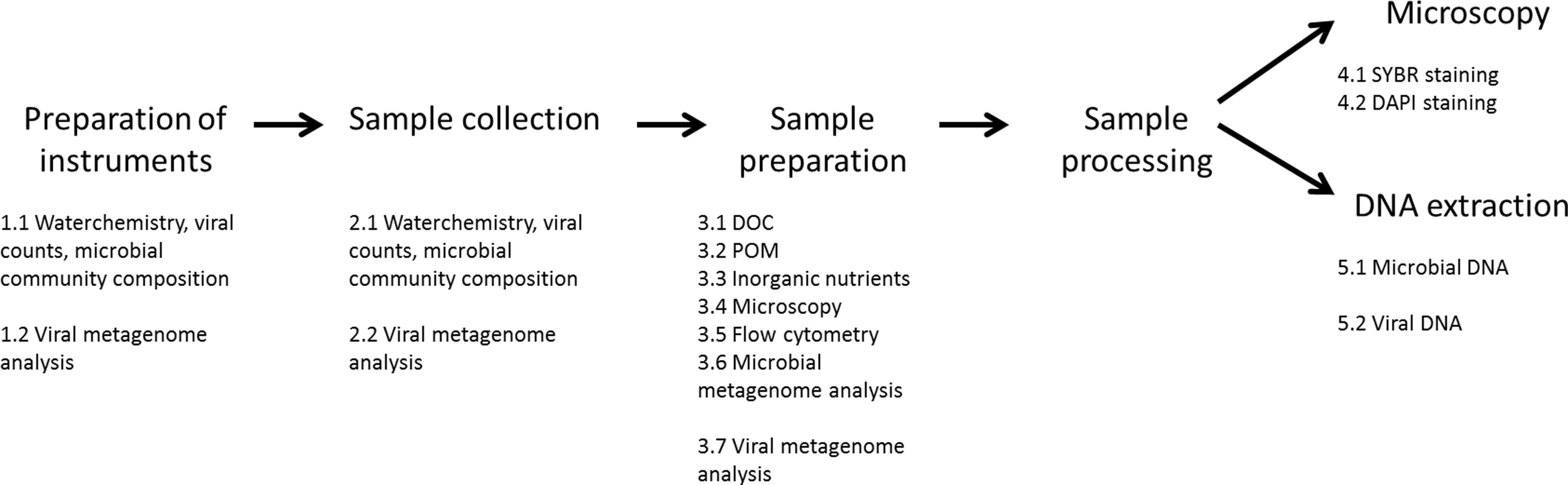
Abbildung 1. Übersicht über im Protokoll Abschnitt beschriebenen Methoden. Bitte klicken Sie hier, um eine größere Version dieses figu ansehenre.

Abbildung 2. Probenbearbeitung rig Setup. Die Filtration Setup hier eingeführt (schematische Zeichnung nach links, eigentliche Bild rechts) ist nicht unbedingt erforderlich, um Proben zu erzeugen, sondern wird den Prozess deutlich beschleunigen. Der Aufbau besteht aus einer Grundplatte, auf der die Hatay Niskin Einheiten (1) montiert sind. Auf der Austrittsöffnung nach unten, werden Hatay Niskin Einheiten mit Silikonschläuchen der in-line-Filterhalter (2) verbunden ist. Diese lassen das abgetastete Wasserdurchlauf durch die jeweiligen Filter in die HDPE Probenahmefläschchen (3) montiert rechts unter. Der Filtrationsprozess wird durch Druckluft (4), und durch einen Druckmesser (5) geregelt beschleunigt. Ein Überlaufbecken verhindert Verschütten von Wasser während der Wechsel der HDPE Fläschchen oder POM Filtration. BitteKlicken Sie hier, um eine größere Version dieser Figur zu sehen.

Abbildung 3 Tangentialstromfilter Formation. Nylon (von Thurber et al. 19 modifiziert) Sieb filtriert Probenwasser wird aus dem Behälter (1) über eine peristaltische Pumpe (2) zu einer tangentialen Strömungsfilter (3) gepumpt. Die Messwasser wieder in den Vorratsbehälter (1) entlang der Rücklaufleitung (4) in der Abwesenheit von Gegendruck. Bei Gegendruck wird durch eine Schlauchklemme (5) an der Rücklaufleitung angelegt wird, gelangt Wasser durch den Filter und wird verworfen bzw. den Filtratbehälter (6) geliefert wird. Probenwasser ersetzt wird, wie es das Filtrat Linie konzentriert wird, bis das gesamte Probenwasser in den Schlauchleitungen konzentriert.
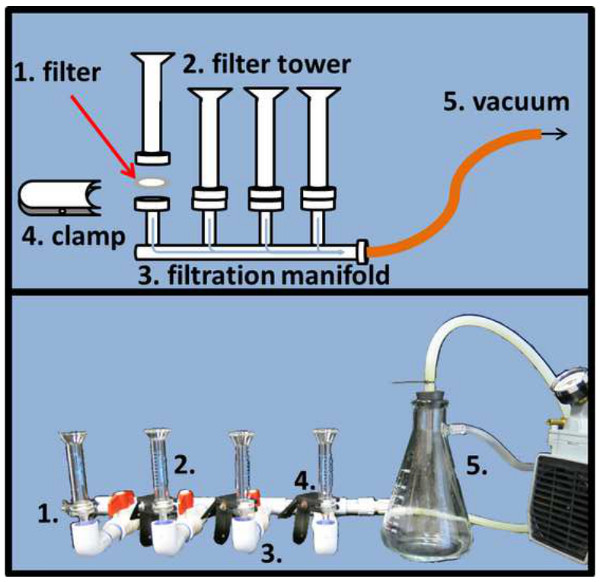
Abbildung 4. Objektträger Filtration Setup.Gleichmäßig zu verteilen DAPI und SYBR Gold-gefärbten Proben auf die jeweilige Matrix aus Aluminiumoxid-Filter (1), benötigen Filter, um zwischen dem Filterturm (2) und Schaft des Filtrationsverteilers mit einer Klemme (4) angeordnet und befestigt werden. Ein druckgeregelte Vakuum (5) wird die Filtrationsprozess (schematische Zeichnung oben, eigentliche Bild Bodenplatte) zu beschleunigen. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
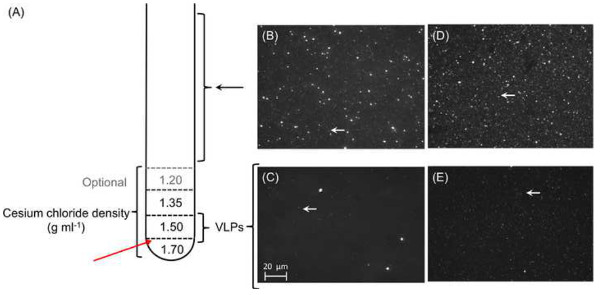
Figur 5. Reinigung und Konzentration von Phagenpartikeln. Insgesamt 1 ml jeder Cäsiumchlorid-Dichtegradienten auf einander vor führt die vorbehandelten Probe (A) überlagert. Jede Schicht sollte außerhalb der Röhre markiert werden, um die Extraktion nach Ultrazentrifugation erleichtern. Rote Pfeil markiert, wo das Rohr wird auf ex durchstochen werdenTrakt des VLP-Fraktion. (BE) Mikroskopische Aufnahmen von Viruskonzentrate: Epifluoreszenz-Aufnahmen von 0,45 & mgr; m filtriert repräsentativen Viruskonzentrate STAR7 (B) und CAR9 (D). Große Teilchen einschließlich Bakterienzellen wurden aus den 0,45 um-Filtrat von beiden STAR7 (B) und CAR9 (D) Proben sichtbar, während nur VLPs (weiße Pfeile) nach der Cäsiumchlorid-Ultrazentrifugation bleiben; Stern 7 (C) und CAR9 (E). Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
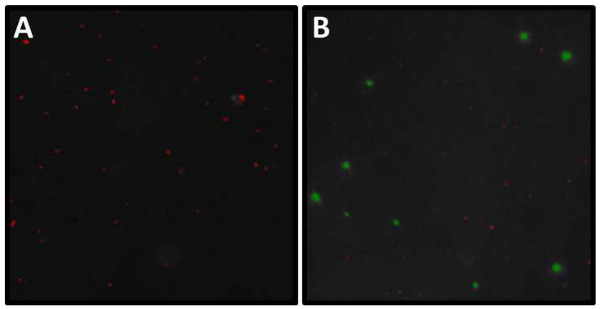
Abbildung 6. Beispiel für DAPI und SYBR Gold-Mikrograph Analyse. Screenshot aus der Analyse von (A) DAPI und (B) SYBR Gold-gefärbten Objektträger Beispiel. (A) Länge und Breite (um) der markierten Teilchen(= Mikroben) kann mit Hilfe einer Bilderzeugungsprogramm bewertet werden. Beachten Sie die Aggregation in der Mitte des Bildes. Diese Cluster müssen von nachfolgenden Analyse ausgeschlossen werden. (B) Bei der Analyse von Viren und Bakterien müssen durch Größenschwellen in VLP und zellulären Größenbereiche (VLP rot, zelluläre grün) klassierte werden. Beachten Sie, dass es in der Regel einige schwache VLPs durch den jeweiligen Bildbearbeitungsprogramm Uncategorized. Diese VLPs als schwache weiße Punkte erscheinen muss manuell gezählt und addiert, um die automatisierten virale zählt. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.

Abbildung 7. Konzentrationen SYBR Gold gefärbten Proben. Mikroskopische Aufnahmen einer 0,02 Aluminiumoxid-Matrix-Filter, die verschiedene Anzahlen von SYBR gefärbten Virusproben. Abbildung A zeigt afilter, die eine geeignete Menge an Meerwasser gefiltert, während die Konzentrationen auf dem Filter in B gezeigt zu hoch und in C für zuverlässige Werte zu niedrig sind. Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
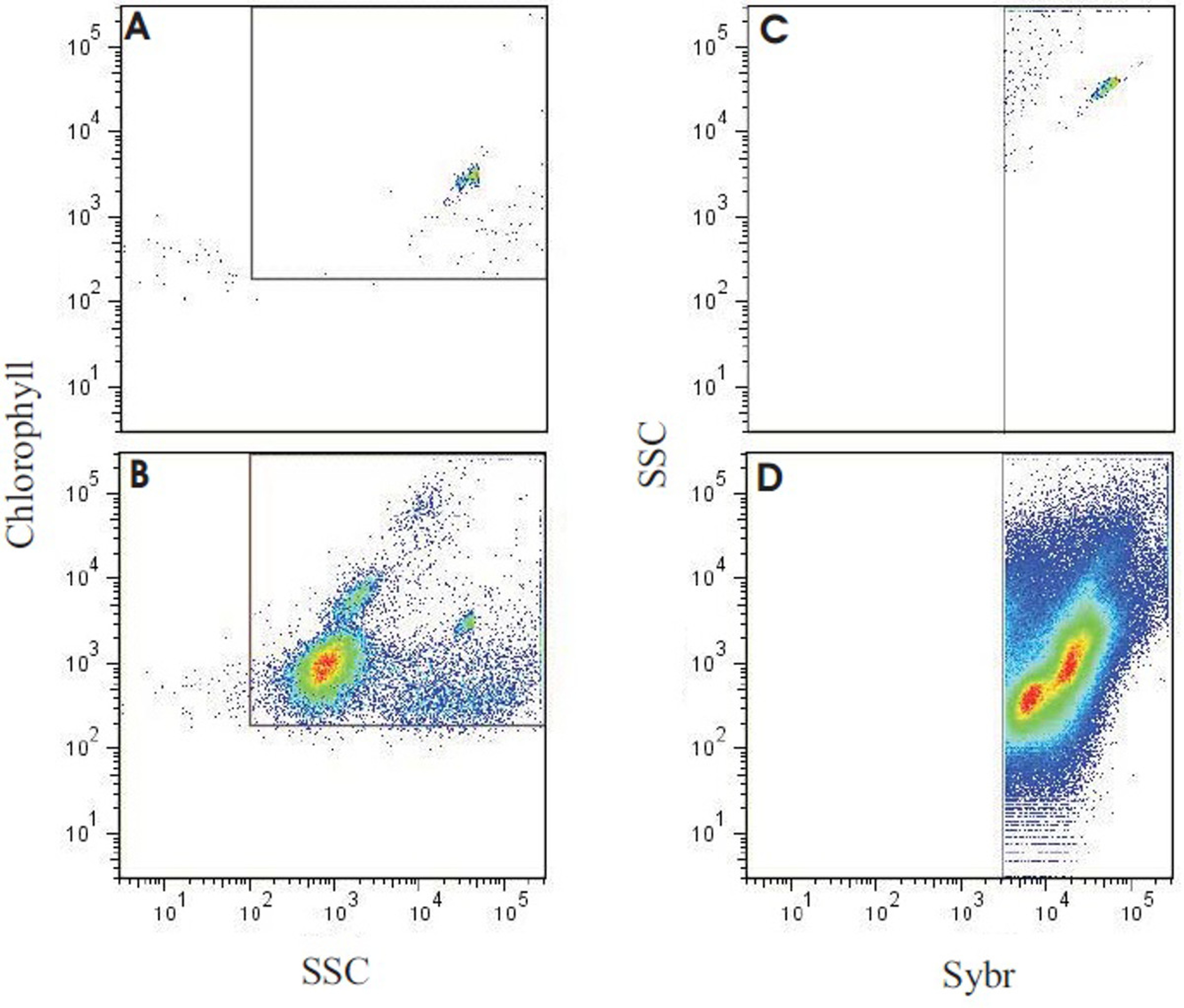
Abbildung 8. Ergebnisse der Durchflusszytometrie-Analyse Ausgang eines Riff-Wasserprobe. Panel (A) zeigt die Wassermolekül Grade nur mit gelb-grün fluoreszierende Mikrosphären (0,75 Mikrometer), verwendet werden, um eine minimale Hintergrund mit den Geräteeinstellungen zu überprüfen. Platte (B) zeigt das Ausgangssignal des Riffs Probe Lauf mit identischen Einstellungen und Layout wie in A. Man beachte, daß die Kügelchen immer noch an der gleichen Stelle mit mehreren Populationen von autotrophs gesehen werden, zusammen. (C) Die gleiche Probe wie in (B), verwendet, um Back-Gate-Durchflusszytometer, Targeting SYBR positive Ereignisse (autotrophe + heterotrophen Zahl) und der Minimierung Hintergrund. (D) Der Vertreter Riff-Wasserprobe mit SYBR Green I. Mit Bunt die in (C) fest Gating wurden insgesamt Keimzahlen erzeugt und heterotrophen und autotrophen Mikroben durch Chlorophyll Autofluoreszenz partitioniert (siehe McDole et al. 4). Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
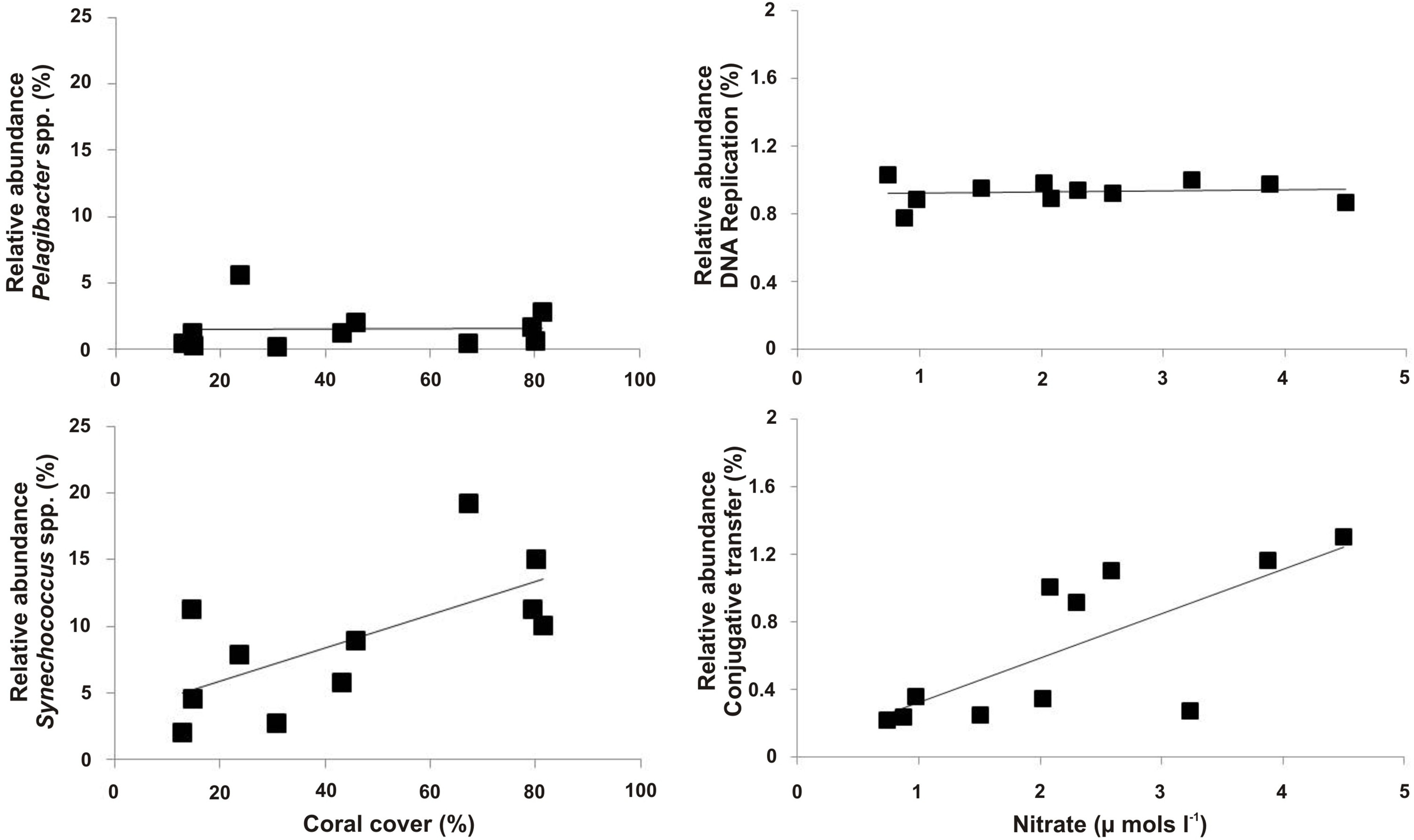
Abbildung 9. Beispiel der mikrobiellen Metagenom Daten. Muster zwischen den mikrobiellen metagenomische Sequenzdaten und Ort abhängigen Variablen. Hier ist die relative Häufigkeit von Synechococcus spp. positiv mit der prozentualen Abdeckung von Korallen korreliert, während Pelagibacter spp. nicht (linke Felder). Der Stoffwechselweg zur konjugativen Transfer positiv mit Nitratkonzentrationen korreliert, während der Stoffwechselweg zur DNA-Reparatur war konsistent zwischen allen Metagenomen (rechte Felder). Bitte klicken Sie hier, um eine größere Version dieser Figur zu sehen.
| Stern 7 | CAR9 | |
| Gesamtzahl der Reads | 939.311 | 591.600 |
| Anzahl der vorverarbeiteten Liest | 579.664 | 360.246 |
| Mittlere Sequenzlänge (bp) | 395 ± 131 | 451 ± 159 |
| Kommentierte Sequenzen (%) | 42.218 (7,3%) | 9,117 (2,5%) |
| Unbekannt (%) | 537.446 (92,7%) | 351.129 (97,5%) |
Tabelle 1. Daten Merkmale von zweirädrigen viromes aus den südlichen Line Islands, Starbuck und Millennium erzeugt. Der Grad der Annotation auf BLAT mit der MG-RAST Server.
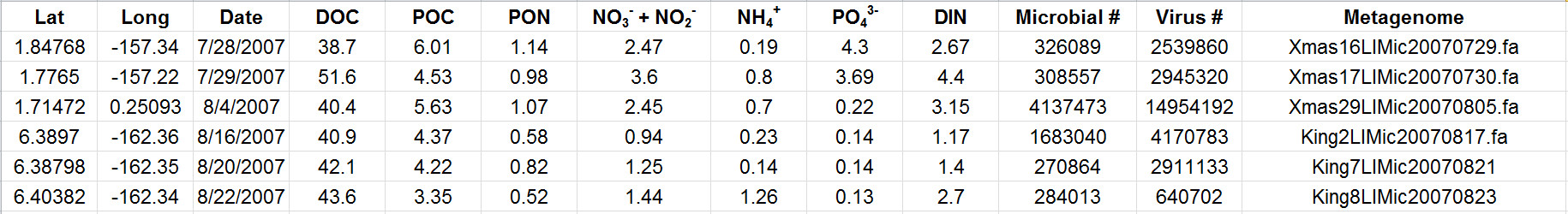
Tabelle 2. Repräsentative Ergebnisse zeigen Daten aus verschiedenen Websites, während einer Expedition. Die hier zu beurteile Parametern gehören Wasserchemie Messungen gepaart mit mikrobiellen und viralen Häufigkeiten. Die Tabelle enthält weitere Dateinamen, die sich auf den jeweiligen metagenomische Daten für jede Entnahmestelle. Kohlenstoff und anorganischen Nährstoffkonzentrationen sind in mmol / L angegeben. BitteKlicken Sie hier, um eine größere Version dieser Tabelle anzuzeigen.
Diskussion
Die hier vorgestellten Methoden liefert ein Werkzeug, um eine umfassende Bewertung der viralen und mikrobiellen Dynamik in aquatischen Ökosystemen zu generieren. Sie werden gezielt auf die stehend Lager von organischen und anorganischen Ressourcen zur Verfügung zu mikrobiellen Gemeinschaften zu quantifizieren und das Make-up der viralen und mikrobiellen Gemeinschaft zu charakterisieren. Neben der Quantifizierung viraler Fülle und mikrobielle Abundanz und Biomasse, genomische Sequenzinformation verrät ihre Gemeinschaft Struktur und Funktion. Alle Methoden wurden entwickelt oder geändert, um für den Einsatz in Fernfeldpositionen zu ermöglichen. Aber der Mangel an kontrollierten Umgebungs erzeugt inhärent eine Fehlerpotential. Hier diskutieren wir einige Vorbehalte mit jedem der untersuchten Parameter zugeordnet. Neben Potenzial für Kontamination während Probenhandling einige der Parameter auch ergeben Fehlerpotenzial in den analytischen Prozess.
Von gelöstem organischem Kohlenstoff
Kohlenstoffkontamination während der Probenahme erfolgen (Abtasten abwärts oder in der Nähe eines Bootes oder Taucher), während der Probenvorbereitung (Verschmutzung der Ausrüstung oder unbeabsichtigte Handhabung) und auch während der Lagerung der Probe (in anderen Proben, die organische Gefrier Lösungsmittel / flüchtigen). Die verschiedenen Verschmutzungsquellen Verhalten als wenige Handhabungsschritte wie möglich zu vermeiden, da jede Probe Verlagerung stellt eine zusätzliche Quelle der Verunreinigung. Die Probe sollte nicht in Kontakt mit irgendeinem Einzelteil nicht mit Säure gewaschen oder verbrannt zu bekommen. Schließlich Bezeichnen eines Speicher Gefrierschrank ausschließlich für Proben flüchtige organische Substanzen nicht enthalten, werden die Integrität der Proben bei längerer Lagerung zu gewährleisten. Wenn Proben nicht während einer längeren Reisezeit Versauerung der DOC-Proben mit konzentrierter HCl gefroren aufbewahrt werden (wie beschrieben 38-41) kann eine anwendbare Alternative sein. Um zu überprüfen, die Messgenauigkeit DOC Konsens Referenzmaterialien sollten reg verwendet werdenders während der DOC-Messung läuft. Besonders tiefe Meerwasser (> 2000 m) Referenzen sind wertvoll bei der Beurteilung der Leistung der analytischen Maschine, wie sie in ihrem DOC-Gehalt 42 sehr stabil ist.
Particulate Organic Matter
Neben der Vermeidung von organischen Kohlenstoffverunreinigung, erfordert POM Probenahme besonders auf die Genauigkeit des gefilterten Volumens zu gewährleisten. Falls die gewählte Volumen von 500 ml sollte für jede mögliche Ursache abweichen dies muss angemerkt, die Menge des POM auf dem Filter zu dem Filtrat von dem es abgeleitet wurde gefangen bezieht.
Anorganische Nährstoffe
Wie bei organischen Kohlenstoff Probenahme, muss man auf mögliche Nährstoffprobenkontamination durch verschiedene Quellen wie Boote oder Abwassereinleitungen 12 erzeugt bezahlt werden. Filter für organischen Kohlenstoff Probenahme mit einer Nennporengrößen von 0,8 um verwendet werden, können nicht alle mikrobiellen Biomasse und sh ausschließenould daher auf 0,2 & mgr; m-Filter für diesen Filtrationsschritt geändert werden. Ferner stellen Nachweisgrenzen ein Problem mit Nährstoffproben; vor allem in den nährstoffarmen Oberflächenwasser um viele Korallenriff Standorten (zB Cotner et al. 43). Nachweisgrenzen liegen etwa bei 0,1, 0,2 und 0,1 umol / L für Ammonium, Nitrat und Nitrit und ortho-Phosphat jeweils (siehe 36,12,37)
Mikroskopie
Neben Variationen in der Konzentration von Bildproben-Analyse von Objektträger weiterhin ergibt Fehlerpotential. Zum Beispiel können die Bilder unscharf in einigen Bereichen des Sichtfeldes ist, und in Ausrichtung in anderen. Als eine hochreine Aluminiumoxidmatrixfilters ist eine sehr starre Filtertyp kann jeder Schmutz unter dem Filter bewirken, dass der gesamte Filter auf einen Winkel zum Schlitten sitzen. Als Ergebnis können die Bilder unscharf konsistent in verschiedenen Teilen des Sichtfelds für eine gegebene Filter, wiederunabhängig von Mikroskop Ausrichtung. Remon Filter, wodurch die Unterseite der Filter sauber ist, kann dies zu verbessern, wenn sie beobachtet wird. Auch in einigen Fällen kann es zwei Brennebenen, in denen Viren und Mikroben gefunden werden. Dies ist das Ergebnis von gefärbten Gegenständen Ablösen von dem Filter bei der Montage und Aufschwimmen an der Unterseite des Deckglases, das zählt unzuverlässig machen kann. Daher ist es immer empfehlenswert, um die Qualität der Proben während des Prozesses zu überprüfen.
Durchflusszytometrie
Wie bei den Mikroskopie Proben kann es natürlich vorkommen Unterschiede zwischen Durchflusszytometrie Proben, die Anpassungen des analytischen Prozesses erforderlich. Zum Beispiel, abhängig von der Empfindlichkeit des Geräts, schwach fluoreszierende Prochlorococcus Zellen unterhalb des Rauschpegels sein und nicht quantifizierbar. Perlen dienen als interne Probenkontrolle (zB, um zu bestätigen, dass bzw. des InstrumentsOnse um Fluoreszenzsignale ist konsistent von Tag zu Tag (und bleibt während des Laufs selbst stabil). Wenn der Probe mit bekannter Konzentration zugesetzt wird, kann sie auch als eine interne Kontrolle, das Probenvolumen Lauf überprüfen zu dienen. Es wird jedoch empfohlen, immer ein Aliquot eines zuvor eingefroren "Standard" Meer Probe aufgetaut ist und zusammen mit den Proben von Interesse angefärbt, um eine zusätzliche Steuerung für biologische Probenhandhabungs zwischen 96-Well-Platte laufen Läufen bereitzustellen. Je nach Lage kann Seewasser Proben sehr unterschiedlich voneinander aussehen. Sortierung "Vertreter" der Bevölkerung und dann die Anzeige unter einem Epifluoreszenzmikroskop (EM) kann ein guter Weg, um Phytoplanktonpopulationen entweder Synechococcus sp. Oder photosynthetischen Eukaryoten schnell überprüfen. Prochlorococcus Zellen sind in der Regel nicht sichtbar unter EM, weil sie zu schnell verblassen. Neben einem, Synechococcus sp Chlorophyll. Den ph enthalten auchotopigment phycoeurythrin (PE), die Licht mit kürzeren Wellenlängen (540-630 nm) als Chlorophyll A (660-700 nm) emittiert. Wenn Synechococcus Zellen mit blau / grünes Licht (470-490 nm) angeregt wird, werden sie golden erscheinen, wenn unter einem Emissionsfilter, die alle Wellenlängen des Lichts unterhalb 510 nm beseitigt angesehen. Im Vergleich dazu wird die eukaryotische Fraktion rot aussehen, wenn sie mit der gleichen Wellenlänge von Licht angeregt.
Viral Metagenomics
Es ist wichtig zu beachten, dass der Prozess der VLP-Konzentration und Lagerung beeinflußt die Gemeinschaft gewonnen. Viruspartikelverlust bei jedem Schritt in dem Verfahren auftreten, und somit kann die Auswahl von Virusreinigung und Anreicherungsprotokolle letztlich auf die beobachtete taxonomische Zusammensetzung und Vielfalt der resultierenden viralen metagenomes 44,45,18. Konzentration der Proben kann mit TFF 19 oder chemischer Basis Ausflockung 20 erfolgen. In der chemisch-basierten Ansatz, postulierenhungsweise geladenen Eisenionen binden die natürlich negativ geladenen Viruspartikel bilden große (> 8 um) Eisenvirale Komplexe, die aus der Lösung ausflocken und kann mit 8 um Filter zurückgewonnen werden. Anschließend wird das Eisen von den Viruspartikeln chelatisiert und wieder gelöst unter Verwendung von Magnesium, Ascorbinsäure und EDTA, so konzentrierten viralen Partikel für die Nukleinsäureextraktion 20. Nach dem Vergleich dieser beiden gängigen Methoden schlossen wir, dass das Eisenchlorid-Verfahren ist weniger zeitaufwendig als TFF Viruskonzentration, kann jedoch einige Einschränkungen ergeben. Wir fanden, dass Dissoziation und Auflösung des Eisenvirale erfordert eine Zwei-fach konzentrierter Magnesium-Ascorbinsäure-EDTA-Lösung als berichtet 20, um zur Auflösung vor den Ascorbinsäure verschlechtert fortzufahren. Abgesehen von diesem Problem, reinigt das Protokoll Viruspartikel nach Größen Fraktionierung allein Entfernen mikrobiellen Verunreinigungen unter Verwendung von 0,2 & mgr; m-Filtration. Große Viren nicht durch die filt gebenER (zB Yang et al. 46) und wird daher nicht in der Metagenom nachgewiesen werden, während einige Bakterien zu tun (McDole, unveröffentlichte Daten). Dies führt zu bakterieller Kontamination während diskriminieren große Viren.
Das spezifische Problem aber ist auch zur Entfernung von Mikroben im Zusammenhang mit TFF Konzentration relevant. Da die Chloroformbehandlung wurde gezeigt, Chloroform empfindlichen Viren mit Lipidmembranen Außen 47 zu entfernen einige Studien darauf hin, dass 0,22 & mgr; m Filtration kann verwendet werden, um den Großteil der Bakterien zu entfernen. Es sollte auch beachtet werden, dass das vorgestellte Verfahren zielt vor allem auf Bakteriophagen, die am häufigsten vorkommende Träger des genetischen Materials in Meeresumgebungen werden. Weil Phagen werden in der Regel dachte nicht zu häufig enthalten Lipide 48 sie widerstandsfähiger gegen Chloroform-Behandlung sind. Nach unserem Wissen eine "catch-all" Methode nicht auf dem neuesten Stand existieren. Das hier vorgestellte Verfahren ist aus ou angepasstr Felderfahrung in den letzten 10 Jahren in verschiedenen Meeresumwelt Spanning tropisch bis arktisch-Systeme.
Mikrobielle Metagenomics
Die Konstruktion von Metagenombanken für Mikroben (dh Bakterien und Archaea) einfacher ist als das virale metagenomes da es einfacher ist, die minimale DNA-Konzentration für Bibliotheksvorbereitung zu erzeugen. Linker Verstärkungs shotgun Bibliotheken (LASLs) 17,49,50 und Amplifikation des gesamten Genoms anhand mehrerer Displacement Amplifikation (MDA) sind die beiden am häufigsten verwendeten Verfahren, um eine ausreichende DNA für die Sequenzierung zu erzeugen. Die Verwendung von MDA, höhere DNA-Konzentrationen in mikrobiellen metagenomes erhalten, ist es manchmal notwendig. Dies kann jedoch Artefakte in den Sequenzdaten, wie etwa die Übersteuerung von Dinoflagellaten Miniringe verursachen. MDA Verfahren sind bekannt, um vorzugsweise zu verstärken einzelsträngige DNA und Kreis, was zu nicht-quantitative Ergebnisse 51,52. Die optimierten LASLs Ansatz 50 kann daher als bessere Alternative Verstärken sowohl mikrobieller und viraler DNA zur Sequenzierung metagenomic dient. Es ist wichtig zu beachten, dass die LASLs Ansatz hat mehrere Schritte erfordert anspruchsvolle Ausstattung, und ist an dsDNA Vorlagen beschränkt. Ferner ist die Gewebe DNA-Extraktions-Kits wurde gezeigt, dass DNA sowohl von Gram-positiven und Gram-negativen Stämmen extrahiert, aber es wurde nicht überprüft, um effektiv lysieren widerspenstigen mikrobiellen Taxa und ihre zugehörigen Strukturen wurden (zB bestimmte Archaea, Endosporen oder Pilzsporen) . Daher kann eine Perle Schlagen Schritt notwendig sein, DNA von bestimmten Mikroben erhalten.
Diese umfassende Bewertung wird in einem besseren Verständnis der viralen und mikrobiellen Ökosystemfunktionen führen. Obwohl nur wenige Studien haben die Anstrengung, alle vorgeschlagenen Parameter einschätzen zu, viele wurden untersucht ausgewählte. Nelson et al. 53 vorgeschlagen, eine Verbindung zween spezifischen Riffhabitaten und Abreicherungen in beiden DOC und Bakterioplankton Konzentrationen relativ zu küstennahen Gewässern. Diese Konzentrationsänderungen wurden durch deutliche Differenzierungen Bakterioplankton Gemeinden begleitet. Dinsdale et al. 5 gezeigt, dass erhöhte anthropogenen Auswirkungen durch 10x höhere Häufigkeiten von Mikrobenzellen und virusähnlicher Partikel beigefügt. Mikrobiellen Gemeinschaften in Meeresgewässern umliegenden bewohnten Inseln hatte auch größere Fraktionen heterotrophs und potenzielle Krankheitserreger 5. Schließlich McDole et al. 4 zeigte, durch die Einführung des microbialization Score, dass menschliche Aktivitäten verlagern sich Energie auf die Mikroben, auf Kosten der macrobes. Diese Studien, zu speziellen mikrobiellen und Wasserchemie Parameter konzentriert, liefern neue Einblicke in die biochemische Struktur der marinen Ökosysteme. Die Kombination traditioneller Datenüberwachungs Ökosystem Bemühungen - wie Temperatur und pH-Werte, Fischbiomasse und diversity, benthische Abdeckung oder Exposition gegenüber menschlichen Einfluss - mit Wasserchemie und mikrobielle Assessments, wird wahrscheinlich in empfindlicher Umweltüberwachung Anstrengungen, die zu mehr gezielte Schutzbemühungen führen kann führen.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
We thank Elisha Wood-Charlson, Jackie Mueller, Karen Weynberg, and Kathy Morrow for their help and constructive input on this manuscript. We also thank the crew and captain of the Hanse explorer which provided us with a perfect working environment to conduct large parts of this research. Further we would like to thank the three anonymous reviewers for their time and helpful comments to improve the manuscript. This work was funded by the NSF Dimensions of Biodiversity and PIRE awards DEB-1046413 and OISE/IIA-1243541, respectively, the Gordon and Betty Moore Foundation, Investigator Award 3781, and the CIFAR Integrated Microbial Diversity Fellowship IMB-ROHW-141679, all to FR.
Materialien
| Name | Company | Catalog Number | Comments |
| Niskin collection and processing units (Hatay Niskins) | Figure 1 | ||
| Filtration setup | Figure 1 | ||
| 32-36% Hydrochloric acid (HCl) | Fisher Sci | A508-212 | |
| High-density polyethylene (HDPE) container | e.g., 5 Gallon HDPE UN Certified Pail with Snap-On Lid | ||
| Combustion oven (550 °C) | |||
| 60 ml HDPE vials | Fisher Sci | 02-896-2B | |
| 25 mm GF/F filter | Fisher Sci | 09-874-64 | |
| Forceps | Fisher Sci | XX62-000-06 | |
| Sterile, non-powdered gloves | Fisher Sci | 19-170-010B | |
| Bilge pump | e.g., Thirsty Mate 124PF | ||
| 20 L collapsible low-density polyethylene carboy | e.g., Reliance Fold A Carrier II | ||
| Tangential flow filter (TFF) ultrafiltration hollow fiber cartridges | Amersham | CFP-2-E-9A | |
| Platinum-cured silicon tubing for TFF | Cole Parmer | 96440-82 | |
| Hose clamps | Cole Parmer | P-68007-23 | |
| Peristaltic pump | Cole Parmer | EW-77410-10 | |
| Sodium hydroxide (NaOH) solution | Electron Microscopy Sciences | 21162 | |
| 0.2 µm Polycarbonate track-etched membrane filter | Fisher Sci | 09-300-61 | |
| 8.0 µm Polycarbonate track-etched membrane filter | Fisher Sci | 09-300-57 | |
| 0.22 µm Polyvinylidene difluoride cylindrical filter | Fisher Sci | SVGP01050 | |
| 0.45 µm Polyvinylidene difluoride cylindrical filter | Fisher Sci | SVHV010RS | |
| Synthetic nylon mesh | Sefar | 03-125-37 | |
| 20 ml Plastic scintillation bottle | Fisher Sci | 03-341-72C | |
| Clean bucket (10-20 L) | |||
| 32% Paraformaldehyde | Electron Microscopy Science | 15714 | |
| 25% Glutaraldehyde | Electron Microscopy Science | 16220 | |
| Liquid nitrogen | |||
| 0.02 µm High-purity alumina matrix filter with annular polypropylene support ring | Fisher Sci | 6809-6002 | |
| 0.2 µm High-purity alumina matrix filter with annular polypropylene support ring | Fisher Sci | 6809-6022 | |
| 10,000x SYBR Gold nucleic acid stain | Invitrogen | S11494 | |
| DAPI solution 25 µg/ml | Invitrogen | D1306 | |
| Molecular grade water | sigma aldrich | W-4502 | |
| Ascorbic acid | Fisher Sci | BP351-500 | |
| Phosphate Buffered Saline (PBS) | Fisher Sci | BP399-500 | |
| Glycerol | Fisher Sci | G31-500 | |
| Microscope slide | Fisher Sci | 12-550-123 | |
| Coverslip | Fisher Sci | 12-548-5M | |
| Delicate task wipe | Fisher Sci | 06-666A | |
| Slide station with vacuum pump | Figure 3 | ||
| DNA extraction kit | Macherey-Nagel | 740952.1 | Nucleospin tissue kit |
| Proteinase K (20 mg/ml) | Lifetechnologies | AM2546 | |
| 200-proof Ethanol | Electron Microscopy Sciences | 15058 | |
| Red cap: Male Luer with Lock Ring x Female Luer Coupler | Cole Parmer | EW-45503-88 | |
| Cesium chloride (CsCl) | Fisher Sci | bp1595-500 | |
| 60 ml Syringe | Fisher Sci | 13-6898 | |
| 0.02 µm Alumina matrix disposable syringe filter | Fisher Sci | O992613 | |
| Ultra-clear centrifuge tubes | Beckman Coulter | 344059 | |
| Ultra-swinging bucket rotor | Beckman Coulter | 333790 | SW41 Ti Rotor |
| DNase I (100 units/µl) | Calbiochem | 260913 | |
| 0.5 M EDTA | Fisher Sci | BP2482-500 | |
| Formamide | Fisher Sci | BP228-100 | |
| Glycogen | sigma aldrich | G-1767 | |
| 1x Tris-EDTA (TE) pH 8.0 | Fisher Sci | BP2473-500 | |
| Sodium Dodecyl Sulfate (SDS) | Fisher Sci | 02674-25 | |
| 20 μg/ml Proteinase K | Fisher Sci | BP1700-500 | |
| Chloroform | Fisher Sci | BP1145-1 | |
| Phenol:Chloroform:Isoamyl alcohol 25:24:1 | sigma aldrich | p3803 | |
| Isopropanol | Fisher Sci | BP2618-500 | |
| 50 ml Oak Ridge tube | Fisher Sci | 05-562-16B |
Referenzen
- Fuhrman, J. A. Marine viruses and their biogeochemical and ecological effects. Nature. 399 (6736), 541-548 (1999).
- Azam, F., Malfatti, F. Microbial structuring of marine ecosystems. Nature Reviews Microbiology. 5 (10), 782-791 (2007).
- Huber, J. A., et al. Microbial population structures in the deep marine biosphere. Science. 318 (5847), 97-100 (2007).
- McDole, T., et al. Assessing coral reefs on a Pacific-wide scale using the microbialization score. PloS ONE. 7 (9), (2012).
- Dinsdale, E. A., et al. Microbial ecology of four coral atolls in the Northern Line Islands. PloS ONE. 3 (2), (2008).
- Nelson, C. E., et al. Coral and macroalgal exudates vary in neutral sugar composition and differentially enrich reef bacterioplankton lineages. The ISME Journal. 7 (5), 962-979 (2013).
- Haas, A. F., et al. Influence of coral and algal exudates on microbially mediated reef metabolism. PeerJ. 1, (2013).
- Smith, J. E., et al. Indirect effects of algae on coral: algae‐mediated, microbe‐induced coral mortality. Ecology Letters. 9 (7), 835-845 (2006).
- Burdige, D. J., Martens, C. S. Biogeochemical cycling in an organic-rich coastal marine basin: 10. The role of amino acids in sedimentary carbon and nitrogen cycling. Geochimica et Cosmochimica Acta. 52 (6), 1571-1584 (1988).
- Capone, D. G., Kiene, R. P. Comparison of microbial dynamics in marine and freshwater sediments: Contrasts in anaerobic carbon catabolism. Limnology and Oceanography. 33 (4), 725-749 (1988).
- Krom, M. D., Sholkovitz, E. R. Nature and reactions of dissolved organic matter in the interstitial waters of marine sediments. Geochimica et Cosmochimica Acta. 41 (11), 1565-1574 (1977).
- Grasshoff, K. Methods of sea-water analysis. Verlag Chemie. , Weinheim and New York. (1976).
- Strickland, J. D. Practical handbook of seawater analysis. Bulletin Fisheries Research Board of Canada. 167, (1968).
- Sharp, J. H., et al. Analyses of dissolved organic carbon in seawater: the JGOFS EqPac methods comparison. Marine Chemistry. 48 (2), 91-108 (1995).
- DeLong, E. F. Microbial population genomics and ecology. Current Opinion in Microbiology. 5 (5), 520-524 (2002).
- Hugenholz, P., Goebel, B. M., Pace, N. R. Impact of Culture-Independent Studies on the Emerging Phylogenetic View of Bacterial Diversity. Journal of Bacteriology. 180 (18), 4765-4774 (1998).
- Breitbart, M., et al. Genomic analysis of uncultured marine viral communities. Proceedings of the National Academy of Sciences. 99 (22), 14250-14255 (2002).
- Willner, D., Hugenholtz, P. From deep sequencing to viral tagging: Recent advances in viral metagenomics. BioEssays. 35, 436-442 (2013).
- Thurber, R. V., Haynes, M., Breitbart, M., Wegley, L., Rohwer, F. Laboratory procedures to generate viral metagenomes. Nature Protocols. 4, 470-483 (2009).
- John, S. G., et al. A simple and efficient method for concentration of ocean viruses by chemical flocculation. Environmental Microbiology Reports. 3, 195-202 (2011).
- Lim, Y. W., et al. The Microbiome and Virome of CF. Pediatric Pulmonology. 47, 321-322 (2012).
- Kelly, L. W., et al. Black reefs: iron-induced phase shifts on coral reefs. The ISME Journal. 6 (3), 638-649 (2012).
- Bjornsen, P. K. Automatic determination of bacterioplankton biomass by image analysis. Applied Environmental Microbiology. 51, 1199 (1986).
- Simon, M., Azam, F. Protein content and protein synthesis rates of planktonic marine bacteria. Marine Ecology Progress Series. 51 (3), 201-213 (1989).
- Hara, S., Koike, I., Terauchi, K., Kamiya, H., Tanoue, E. Abundance of viruses in deep oceanic waters. Marine Ecology Progress Series. 145 (1), 269-277 (1996).
- Wommack, K. E., Colwell, R. R. Virioplankton: viruses in aquatic ecosystems. Microbiology and Molecular Biology Reviews. 64 (1), 69-114 (2000).
- Weinbauer, M. G., Brettar, I., Hofle, M. G. Lysogeny and virus-induced mortality of bacterioplankton in surface, deep, and anoxic marine waters. Limnology and Oceanography. 48 (4), 1457-1465 (2003).
- Clasen, J. L., Brigden, S. M., Payet, J. P., Suttle, C. A. Evidence that viral abundance across oceans and lakes is driven by different biological factors. Freshwater Biology. 53 (6), 1090-1100 (2008).
- Parada, V., Baudoux, A. C., Sintes, E., Weinbauer, M. G., Herndl, G. J. Dynamics and diversity of newly produced virioplankton in the North Sea. The ISME Journal. 2 (9), 924-936 (2008).
- Angly, F. E., et al. The marine viromes of four oceanic regions. PLoS Biology. 4 (11), (2006).
- Hansell, D. A., Carlson, C. A. Deep ocean gradients in dissolved organic carbon concentrations. Nature. 263 (266), (1998).
- Carlson, C. A., et al. Dissolved organic carbon export and subsequent remineralization in the mesopelagic and bathypelagic realms of the North Atlantic basin. Deep Sea Research Part II: Topical Studies in Oceanography. 57 (16), 1433-1445 (2010).
- Sharp, J. H. Improved analysis for particulate organic carbon and nitrogen from seawater. Limnology and Oceanography. 19 (6), 984-989 (1974).
- Ehrhardt, M., Koeve, W. Determination of particulate organic carbon and nitrogen. Methods of Seawater Analysis. Third Edition, 437-444 (2007).
- Haas, A. F., et al. Organic matter release by coral reef associated benthic algae in the Northern Red Sea. Journal of Experimental Marine Biology and Ecology. 389 (1), 53-60 (2010).
- Guildford, S. J., Hecky, R. E. Total nitrogen, total phosphorus, and nutrient limitation in lakes and oceans: Is there a common relationship. Limnology and Oceanography. 45 (6), 1213-1223 (2000).
- Allison, S. D., Chao, Y., Farrara, J. D., Hatosy, S., Martiny, A. C. Fine-scale temporal variation in marine extracellular enzymes of coastal southern California. Frontiers in Microbiology. 3, (2012).
- Van Duyl, F. C., Gast, G. J. Linkage of small-scale spatial variations in DOC, inorganic nutrients and bacterioplankton growth with different coral reef water types. Aquatic Microbial Ecology. 24 (1), 17-26 (2001).
- De Goeij, J. M., Van Duyl, F. C. Coral cavities are sinks of dissolved organic carbon (DOC). Limnology and Oceanography. 52 (6), 2608-2617 (2007).
- De Goeij, J. M., et al. Major bulk dissolved organic carbon (DOC) removal by encrusting coral reef cavity sponges. Marine Ecology Progress Series. 357, 139-151 (2008).
- Mueller, B., et al. Natural diet of coral-excavating sponges consists mainly of dissolved organic carbon (DOC). PLoS ONE. 9 (2), (2014).
- Carlson, C. A., Ducklow, H. W., Hansel, D. A. Organic carbon partitioning during spring phytoplankton blooms in the Ross Sea polynya and the Sargasso Sea. Oceanography. 43 (3), 375-386 (1998).
- Cotner, J. B., et al. Nutrient limitation of heterotrophic bacteria in Florida Bay. Estuaries. 23 (5), 611-620 (2000).
- Willner, D., et al. Metagenomic detection of phage-encoded platelet-binding factors in the human oral cavity. Proceedings of the National Academy of Science USA. 108 (1), 4547-4553 (2011).
- Hall, R. J., et al. Evaluation of rapid and simple techniques for the enrichment of viruses prior to metagenomic virus discovery. Journal of Virological Methods. 195, 194-204 (2013).
- Yang, S. Y., et al. Nanoporous membranes with ultrahigh selectivity and flux for the filtration of viruses. Advanced Materials. 18 (6), 709-712 (2006).
- Feldman, H. A., Wang, S. S. Sensitivity of various viruses to chloroform. Proceedings of the Society for Experimental Biology and Medicine. 106 (4), 736-738 (1961).
- Ackermann, H. W. The Bacteriophages., Second Edition. Calendar, R. , Oxford University Press. New York. 8-16 (2006).
- Henn, M. R., et al. Analysis of High-Throughput Sequencing and Annotation Strategies for Phage Genomes. PLoS ONE. 5 (2), (2010).
- Duhaime, M. B., Deng, L., Poulos, B. T., Sullivan, M. B. Towards quantitative metagenomics of wild viruses and other ultra-low concentration DNA samples: a rigorous assessment and optimization of the linker amplification method. Environmental Microbiology. 14 (9), 2526-2537 (2012).
- Yilmaz, S., Allgaier, M., Hugenholtz, P. Multiple displacement amplification compromises quantitative analysis of metagenomes. Nature Methods. 7 (12), 943-944 (2010).
- Kim, K. H., Bae, J. W. Amplification Methods Bias Metagenomic Libraries of Uncultured Single-Stranded and Double-Stranded DNA Viruses. Applied Environmental Microbiology. 77 (21), 7663-7668 (2011).
- Nelson, C. E., Alldredge, A. L., McCliment, E. A., Amaral-Zettler, L. A., Carlson, C. A. Depleted dissolved organic carbon and distinct bacterial communities in the water column of a rapid-flushing coral reef ecosystem. The ISME Journal. 5 (8), 1374-1387 (2011).
- Kelly, L. W., et al. Local genomic adaptation of coral reef-associated microbiomes to gradients of natural variability and anthropogenic stressors. Proceedings of the National Academy of Sciences. 111 (28), 10227-10232 (2014).
- Lim, Y. W., Haynes, M., Furlan, M., Robertson, C. E., Harris, J. K., Rohwer, F. Purifying the Impure: Sequencing Metagenomes and Metatranscriptomes from Complex Animal-associated Samples. J. Vis. Exp. , (2014).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten