Method Article
Recombineering Construções recombinação homóloga em
Neste Artigo
Resumo
Técnicas de recombinação homóloga avançar muito Drosophila Genética, permitindo a criação de mutações molecularmente precisas. A recente adopção recombineering permite manipular grandes pedaços de DNA e transformá-los em Drosophila 6. Os métodos aqui apresentados combinar estas técnicas para gerar rapidamente grandes vectores de recombinação homóloga.
Resumo
A continuação do desenvolvimento de técnicas para a manipulação rápida e em grande escala de loci de genes endógenos vai alargar a utilização de Drosophila melanogaster como um organismo modelo genético para investigação relacionada com a doença humana-. Nos últimos anos houve avanços técnicos e recombineering como recombinação homóloga. No entanto, a geração de mutações nulas inequívocos ou proteínas endógenas marcação continua a ser um esforço substancial para a maioria dos genes. Aqui, nós descrevemos e demonstrar as técnicas para a utilização de métodos de clonagem baseados em recombineering para gerar vectores que podem ser utilizados para alvejar e manipular loci endógenas in vivo Especificamente, nós estabelecemos uma combinação de três tecnologias: (1). BAC transgénese / recombineering, ( 2) termina-a recombinação homóloga e (3) de tecnologia de gateway para fornecer um método robusto, flexível e eficiente para a manipulação de loci genómicas endógenos. Neste protocolo, nós fornecemos detalhes passo-a-passo sobre como (1) projeto individuavetores l, (2) como clonar grandes fragmentos de DNA genômico no vetor recombinação homóloga utilizando reparação lacuna, e (3) como substituir ou marcar genes de interesse dentro desses vetores usando uma segunda rodada de recombineering. Finalmente, vamos também fornecer um protocolo de como mobilizar esses cassetes in vivo para gerar um nocaute ou um gene marcado via knock-in. Estes métodos podem ser facilmente adotado para alvos múltiplos em paralelo e fornecer meios para manipular o genoma de Drosophila em uma maneira oportuna e eficiente.
Introdução
Limpe manipulações molecularmente definidos de genes individuais em seus loci endógenos oferecer uma ferramenta de valor inestimável para o estudo de uma miríade de questões relevantes para a biologia dos eucariotas. Drosophila reverter técnicas genéticas para a geração de alelos de perda de função tinha provado ser um desafio até Golic e colegas introduziram em segmentação de genes in vivo utilizando recombinação homóloga de Drosophila 1-3. Eles demonstraram que o locus genómico específicos podem ser alvo utilizando um fragmento linear de ADN a partir de uma construção transgénica integrado. Este DNA linear "dador" é gerado in vivo por meio de recombinação FRT-mediada (para excisar o ADN do cromossoma como uma molécula circular), seguido por linearização com a meganuclease I-Scel. Embora esta metodologia tem sido utilizada com sucesso para gerar uma variedade de lesões definidos, a técnica não tem sido facilmente adaptar para a manipulação de vários genes em paralelo, porque cada indivíduoconstrução dupla de nocaute requer design distinto e personalizado. Por exemplo, as dificuldades em manipular facilmente grandes fragmentos de ADN (> 5 kb) in vitro, usando a enzima de restrição clássica / ligadura clonagem ou PCR, bem como as limitações de tamanho da tradicional, em vectores de transformação in vivo, interferem frequentemente com a criação rápida de recombinação homóloga segmentação vetores. Para superar essas limitações, nós combinamos a P [acman] sistema recombineering / transgenia, que permite a sub-clonagem e transgênese de até 100 kb de DNA, com o fim-out gene metodologia visando a estabelecer uma plataforma eficiente e relativamente rápida que facilita gene Drosophila segmentação.
Recombinação mediada por engenharia genética (recombineering) é um poderoso recombinação homóloga baseada em tecnologia de clonagem 4,5. Em contraste com a enzima / ligase de clonagem convencional restrição, recombineering não é limitado pela sequência ou size do DNA manipulado. Recombineering utiliza uma E. especial coli que alberga maquinaria de recombinação é fornecida por um profago defeituoso λ 4. Esta técnica foi recentemente aprovada para uso em Drosophila 6,7. Recombineering em Drosophila depende de um cromossoma alterado condicionalmente amplificável bacteriano artificial (BAC) vetor chamado P [acman] 6,7. Este vector transporta duas origens de replicação: oriV, que produz maior número de cópia por indução química, para a purificação de grandes quantidades de ADN necessárias para a sequenciação e injecção de embriões e oris, que mantém baixo número de cópia em condições basais. Além disso, o P [acman] vector é equipado com um acessório local bacteriana (attB). O local attB serve como um substrato para a integrase ΦC31 transgénese mediada que permite a incorporação de grandes fragmentos de ADN num local de pouso predeterminado dentro do genoma de Drosophila 8,9.
Geramos um P [acman] vetor (referido como P [acman]-KO 1.0) que pode ser usado como um vector-alvo termina para fora recombinação homóloga 10,11. Para incorporar fins-out gene targeting tecnologia no sistema, nós adicionamos dois FRT e dois locais I-SCEI. Incluímos também uma cassete gateway dentro desse vetor modificado para agilizar o processo de incorporação dos braços de homologia em P [acman]-KO 1.0. Isso fornece uma maneira rápida e simples para introduzir praticamente qualquer região genômica de interesse no vetor alvo. Neste protocolo, irá descrever como projetar um vetor alvo usando P [acman]-KO 1.0, e como mobilizar esse vetor in vivo para atingir o locus endógeno. Para o propósito deste protocolo usaremos o cassete RFP / Kan para substituir um gene de interesse, mas uma variedade de cassetes que contêm um marcador de selecção de antibiótico pode ser usada com este protocolo. Temos desenvolvido e utilizado com sucesso um conjunto de cassetes parasubstituição de genes de marcação e 10,11.
Protocolo
1. Seleção de BAC e Região para Target
- Para adquirir um BAC com o gene de interesse (GOI) (aqui CG32095 é usado como exemplo), procure CG32095 em www.flybase.org . De acordo com os estoques de seção e reagentes, verifique a seção "Clones genômicos" para BACs contendo CG32095. Certifique-se de que o BAC inclui pelo menos 10 kb a montante e 5 kb a jusante do gene de interesse. Alternativamente, os clones do P [acman] vetor 7 também pode ser encontrado em http://www.pacmanfly.org/libraries.html .
- A clones BAC vêm em células DH10B e P [acman] clones vêm em EPI300 células. Os clones genómicos precisam de ser transformado em células SW102, que são passíveis de recombineering. "Sujos minipreps" são realizadas (a minipreparações Qiagen sem utilizar uma coluna e precipitar o ADN, utilizando isopropanol), partindoml de uma cultura D / N 5 inoculado com as células contendo o CCB. Depois de re-suspensão da pelota de ADN recentemente feita em 20 uL de dH2O, 1 uL diluído em 100 jil de dH2O e utilizar 1 ml de electroporate SW102 electrocompetentes. Para tornar SW102 electrocompetentes usar o protocolo seguinte:
- Inocular SW102 células em 4 ml de LB. Crescer a cultura a 32 ° CO / N com agitação a 250 rpm Importante:. Células SW102 nunca deve ser exposto a uma temperatura superior a 32 ° C, desde que a indução da maquinaria de recombinação é desejada.
- Inocular 1 ml da cultura saturada SW102 em 44 ml de LB e crescer num agitador de 32 ° C a DO600 entre 0,2-0,3 (usualmente cerca de 1-1,5 horas) NOTA:. Outros protocolos recombineering recomendado crescimento da cultura num volume menor de DO600 de 0,6-0,7, no entanto o crescimento da bactéria com uma densidade inferior a um maior volume aumenta significativamente transformação efficiency 12. Além disso, esta modificação reduz o tempo de espera.
- Arrefece-se a cultura em suspensão de água-gelo durante 5 min Importante:. Durante as seguintes etapas células devem ser mantidos a baixas temperaturas. Isto é crucial para a obtenção de células electrocompetentes de boa qualidade.
- Centrifugar cultura a 4 ° C a 1500 xg durante 10 min. Rejeitar sobrenadante e lavar o sedimento em 25 mL de água fria (4 ° C), 10% de glicerol 13. Primeiro, Ressuspender as células em 5 ml tocando e agitando o tubo na lama de água gelada (NÃO pipeta cima e para baixo) e, em seguida, adicione os restantes 10% de glicerol (20 ml).
- Centrifugar a 4 ° C a 1.500 xg durante 10 minutos, rejeitar o sobrenadante e lavar o sedimento uma segunda vez com 25 mL de gelo frio de 10% de glicerol.
- Centrifugar a 4 ° C a 1.500 xg durante 10 minutos, rejeitar o sobrenadante e lavar o sedimento uma terceira vez com 25 mL de gelo frio de 10% de glicerol.
- Centrifugar a 4 ° C a 1500 xg durante 10 minutos e descartar supernatant. O pellet celular pode ser muito solto neste momento e deve ser tomado cuidado quando descartar o sobrenadante. Ressuspender o sedimento em 1 ml de glicerol a 10% frio. Transferir as células para um tubo de 1,5 ml e centrifugar durante 30 segundos a 12.000 x g numa microcentrífuga tampo a 4 ° C. Descartar a maior parte do sobrenadante deixando ≈ 90 ul de glicerol / células. As células são agora pronto para ser electroporada NOTA:. Neste ponto as células podem ser armazenadas a -80 ° C para uso posterior. No entanto, isto pode reduzir a competência.
- Electroporate ADN em células utilizando uma cuvete de 1 mm a 1.800 V, 25 uF, 200 Ω. Após a electroporação, adicionam-se 300 ul de SOC e deixar que as células recuperar durante 2 horas a 32 ° C (agitação não é necessária). Células da placa sobre ágar LB mais 25 ug / ml de cloranfenicol (ou outros antibióticos adequados, dependendo do CCB transformada ou PAC) e incubar a 32 ° C durante 24-30 hr.
- Braços de homologia para o gene alvo são projetados por selectção de 10 kb de DNA genômico a montante e jusante 5 kb relação ao GI. Seleccione 500 pb no final do 10 kb e 5 kb braços de homologia para amplificar (ver secção 2) (Figura 1). Estes 500 fragmentos pb servem como braços de homologia utilizados para recombinar a região genômica em torno do GOI em P [acman] KO 1.0. Estes fragmentos são referidos como o braço esquerdo (AE) para a região que corresponde ao montante 5 'do braço de homologia e o braço direito (RA), para a região que corresponde a jusante 3' do braço de homologia Importante:. Braços de homologia não deve conter BamHI sítios de restrição, tal como a linearização do plasmídeo (no passo 3.2) é realizado através de uma digestão de restrição BamHI.
2. Insira a homologia de Armas em P [acman]-KO 1.0
- Configure duas reações de PCR individuais, um para amplificar o LA e outro para amplificar o RA. Para a LA, utilizar iniciadores attB1-I-Scel-LA-F e BamHI-LA-R e utilizar os iniciadores de BamHI-RA-F e attB2-I-Scel-RA-Ipara amplificar a RA (Tabela 1). Use 1 ul de DNA de BAC passo 1.2 ou 0.5 ml de cultura bacteriana durante a noite fervida contendo TAS de interesse como um modelo. Certifique-se de usar uma polimerase de DNA com capacidade de revisão. Isto aplica-se a todos os PCRs posterior-com excepção do controlo de PCR nos passos 2.8 e 3,13 Observação:. Tempo de extensão por uma polimerase de ADN de revisão pode ser diferente de regular a Taq DNA polimerase, dependendo do fabricante. PfuUltra II HotStart fusão, que sofre polimerização a uma taxa de 15 seg / kb, pode ser usada nestes passos.
LA / RA PCR
As condições de PCR1 ul DNA de miniprep sujo do passo 1.2 do Bac 0,25 mL Cada 20 uM Primer 2,5 mL 10X PfuUltra II tampão 0,75 mL 10 mM (cada) de dNTP 0,5 mL PfuUltra II Fusão HotStart DNA Polymerase 19,75 mL Água 25 ul Volume Total Passo 1 95 ° C 2 min para activar enzima Passo 2 95 ° C 20 seg desnaturam Passo 3 60 ° C, 20 s de recozimento Passo 4 72 ° C 20 segundos extensão Passo 5 Vá para a etapa 2 e repita 29 ciclos Passo 6 4 ° C Segure - Executar amostras num gel de agarose 1% e extrai-se utilizando um kit de produtos Zymoclean recuperação do DNA. Eluir o ADN a partir da coluna Zymoclean com 10 ul de água estéril.
- Realize splicing por extensão sobreposição (PCR SOE), com os dois fragmentos, utilizando 10-30 ng de DNA total a partir de cada produto amplificado purificado de 2,2 (Figura 2). Isto é geralmente cerca de 1/20 de cada produto de PCR purificado:
LA / RA PCR-Soe
As condições de PCR0,5 mL cada LA e RA purificado do produto de PCR de aproximadamente 10-30 ng 0,25 mL cada 20 uM Primer 2,5 mL 10X PfuUltra II tampão 0,75 mL 10 mM (cada) de dNTP 0,5 mL PfuUltra II Fusão HotStart DNA Polymerase 19,25 mL Água 25 ul Volume Total Passo 1 95 ° C 2 min para activar enzima Passo 2 95 ° C 20 seg desnaturam Passo 3 55 ° C 20 seg recozimento (Baixa temperatura. Permitir braços para temperar) Passo 4 72 ° C 20 segundos extensão Passo 5 Vá para a etapa 2 e repita um ciclo Passo 6 95 ° C 20 seg desnaturam Passo 7 60 ° C, 20 s de recozimento Step8 72 ° C 20 segundos extensão Step9 Vá para a etapa 2 e repita 27 ciclos Passo 10 4 ° C Segure - Correr a amostra de PCR num gel de agarose e se recuperar a banda de 1,0 kb por extracção de gel. Esta é a cassete attB1-LA-BamHI-RA-attB2.
- Use gateway kit enzimático BP ClonaseII configurar uma reação BP seguindo o protocolo fornecido pelo fabricante.A cassete gerada em 2,4 actuará como o DNA dador e o P [acman]-KO 1.0 como o vector de destino.
- Dilui-se 50 ul de células electrocompetentes TransforMax EPI300 pela adição de 30 ul de glicerol a 10% frio ou frio dH 2 O. Transformação 1 ul da reacção da BP em células electrocompetentes seguindo o protocolo fornecido pelo fabricante. A adição de água dilui a concentração relativamente elevada de sal da reacção BP para impedir a descarga eléctrica (arco) durante electroporação. Electroporate ADN em células utilizando uma cuvete de 1 mm a 1.800 V, 25 uF, 200 Ω.
- Adicionar 300 ul de SOC e deixar as células recuperar durante 1 hora a 37 ° C (agitação não é necessária). Células da placa de LB-agar + 50 ug / ml de Amp (P [acman]-KO 1.0 contém um gene de resistência a Amp). Incubar as placas a 37 ° C durante 18-24 horas.
- PCR verificar colônias individuais de colônia PCR convencional utilizando T3 (T3 seqüência é a jusante da cassete Gateway o P [acman]-KO 1.0 (Figure 1) e attB1-I-Scel-LA-F iniciadores. Os clones positivos vai mostrar uma banda que funciona a 1,2 kb.
- Crescer uma cultura nocturna de um clone positivo em LB com 50 ug / ml de ampicilina.
- Amplificar o P [acman]-KO 1,0 vector contendo o LA e RA em 10 ml de LB com 50 ug / ml de ampicilina utilizando uma solução de cultura de controlo da cópia seguindo o protocolo do fabricante (Epicentre).
- Executar uma minipreparação Qiagen de acordo com protocolo do fabricante.
3. Recombinação a região genômica de interesse em P [acman]-KO 1.0
Dia 1
- Inocular SW102 células contendo o TAS de interesse em 4 ml de LB mais 25 ug / ml de cloranfenicol (Cl) ou a selecção antibiótica apropriada. Crescer a cultura a 32 ° CO / N com agitação a 250 rpm Importante:. Além disso, durante a indução na etapa 3.5, SW102 de células não deve ser exposto a uma temperatura superior a 32 ° C, com a temperatura elevada activa a recombinação maChinery destas células.
Dia 2
- Digest 0,4 ug do P [acman]-KO-1.0 contendo os braços LA e RA gerados na secção 2 com ≈ 20 unidades de BamHI, numa reacção de 25 ul. Incubar a 37 ° C durante pelo menos 3 horas Importante:. Comprida digestão é crítica para garantir a digestão de todo o ADN. Isto irá diminuir o número de clones falsos positivos mais tarde.
- Inocular 1,0 ml da cultura SW102/BAC saturada em 44 ml de LB-Chl (25 ug / ml) e crescer num agitador de 32 ° C a DO600 entre 0,2-0,3 (usualmente cerca de 1-1,5 horas). Incluir uma amostra idêntica adicional para controle não-HS. Esta amostra será tratado apenas como o experimental em todos os aspectos, com exceção do HS. Esta cultura não-calor-choque serve como controle negativo. O aparecimento de muitas colônias na placa de controle não-HS (passo 3.13) geralmente indica transformação com indigesto P [acman] KO 1,0 LA/ RA plasmídeo.
- Embora a cultura está a crescer, executar o BamHI-restrito P [acman]-KO num gel. Então gel-extrair e eluir o DNA em 10 ml de água aquecida a 55 ° C. NOTA: Não eluir DNA em qualquer buffer. Os sais presentes no tampão pode provocar uma descarga eléctrica durante electroporação.
- Choque de calor a cultura a 42 ° C durante exactamente 15 min no banho de água. Isso irá ativar o mecanismo de recombineering SW102 células. Não aqueça o choque da cultura controle.
- Imediatamente após o choque térmico, a transferência de calor em estado de choque e as culturas de controle não-SH, refrigerados 50 ml tubos canônicas e deixar o frio culturas em uma pasta de água e gelo por 5 min Importante:. Durante os seguintes passos células devem ser mantidos em temperaturas baixas . Isto é crucial para obter células electrocompetentes de boa qualidade.
- Centrifugar a cultura a 1.500 x g durante 10 min. Rejeitar sobrenadante e lavar o sedimento em 25 mL de água fria (4 ° C), 10% de glicerol 13. Em primeiro lugar,ressuspender as células em 5 ml tocando e agitando o tubo de pasta de água gelada (NÃO pipeta cima e para baixo) e, em seguida, adicione os restantes 10% de glicerol (20 ml).
- Centrifugar a 4 ° C a 1.500 xg durante 10 minutos, rejeitar o sobrenadante e lavar o sedimento com 25 ml de gelado a 10% de glicerol uma segunda vez.
- Centrifugar a 4 ° C a 1.500 xg durante 10 minutos, rejeitar o sobrenadante e lavar o sedimento com 25 ml de gelado a 10% de glicerol uma terceira vez.
- Centrifugar a 4 ° C a 1500 xg durante 10 min e descartar o sobrenadante. O pellet celular pode ser muito solto neste momento e deve ser tomado cuidado quando descartar o sobrenadante. Ressuspender o sedimento em 1 ml de glicerol a 10% frio. Transferir as células para um tubo de 1,5 ml e centrifugar durante 30 segundos a 12.000 x g numa microcentrífuga tampo a 4 ° C. Descartar a maior parte do sobrenadante deixando ≈ 90 pi de água / células NOTA:. No ponto destas células podem ser armazenadas a -80 ° C para uso posterior. No entanto, isto pode reduzir a competência.
- Adicionam-se 2 mL do digerido com BamHI P [acman] (objectivo de 50 ng) para as células, e misturar suavemente utilizando uma ponta de pipeta. Transferência de células numa cuvete de electroporação 1 mm e electroporate a 1.800 V, 25 uF, 200 Ω.
- Adicionar 300 ul de SOC e deixar que as células recuperar durante 2 horas a 32 ° C (agitação não é necessária). Células da placa sobre ágar LB mais 50 ug / ml de ampicilina e incubado a 32 ° C durante 24-30 hr.
Dia 3
- Tela para o bom gap-repair usando colônia PCR. Use T3 e RA verificar primers para a RA e T7 e LA cartilha de seleção para o LA (Tabela 1). Projetar a PCR-Check-RA e PCR-Check-AL primers 100-200 bp para a região alvo (Tabela 1). Colônias positivas de reações de PCR utilizando PCR-Check-RA com T3 ou PCR-Check-LA com T7 vai mostrar uma banda que funciona em 800-1.000 bp (Figura 3) NOTA:. Ele é fundamental para testar ambos os braços desde a incorporação de um braçomas não a outra é por vezes observada.
- Cresça PCR verificados colónias a 32 ° C em 4 ml de LB 50 ug / ml de ampicilina durante a noite. Adicione um estoque congeladas destas células para utilização posterior.
4. Substituir a região genômica com a cassete Segmentação
Dia 4
- Aumentar o cassete nocaute RFP / KAN por PCR usando pENTR-RFP/KAN (pré-digeridas com AscI e NheI para linearizar) como um modelo eo 5'HA-RFP / Kan-F e 3'HA-RFP / Kan-R primers ( Tabela 1). Cassete RFP / KAN pode ser solicitada a partir da referência 11.
- Gel de purificar o produto de PCR de eluição em 20 ul de água estéril. A cassete RFP-KAN mais os braços de homologia deve rodar em torno de 3 kb.
- Inocular 1 ml da saturado SW102 / P [acman]-KO cultura (passo 3.13) em 44 ml de LB-Amp (50 ug / ml), e crescem em 32 ° C a DO600 agitador entre 0,2-0,3. Incluir uma amostra idêntica adicional para controle não-HS. Esta amostra será tratado apenascomo o experimental em todos os aspectos, com exceção do HS.
- Após o [acman] cultura KO SW102 / P atinge a densidade desejada, siga os passos 3,5-3,10.
- Electroporate aproximadamente 100 ng da cassete de segmentação em 90 ul de células electrocompetentes SW102 / P [acman]-KO. Transferência de células numa cuvete de electroporação 1 mm e electroporate a 1.800 V, 25 uF, 200 Ω. Adicionar 300 ul de SOC e permitir que as células recuperar durante 2 horas a 32 ° C (agitação não é necessária) NOTA:. É altamente recomendada a utilização de um gel fresco cassete purificada.
- Células da placa de agar LB + Amp (50 ug / ml) + Kan (50 ug / ml) e incubar durante 24-30 horas a 32 ° C.
Dia 5
- Crescer 5 ml de cultura a partir de 5 colónias diferentes de 32 ° CO / N.
Dia 6
- Realizar uma "sujo miniprep" e correr metade do DNA obtido em gel de agarose 1% a olhar para o presence de um baixo peso molecular de plasmídeo. Não deve haver qualquer plasmídeo que corre fole 12 kb. (Figura 4)
- Fazer uma diluição 1:500 do ADN a partir de um clone positivo e utilizar 1 ml de electroporate em 50 ul de TransforMax EPI300 Importante:. Certifique-se para diluir o ADN para prevenir a electroporação de vários plasmídeos em uma célula. Células da placa de agar LB + Amp (50 ug / ml) + Kan (50 ug / ml) e incubar durante 18-24 horas a 37 ° C
Dia 7
- Inocule uma colónia simples em 10 mL de LB + e crescer S / N.
Dia 8
- Semente de 100 ml de meio LB com a cultura do passo 4.10 saturada. Adicionar antibióticos apropriados mais 100 ul de 1000 X solução de controle de cópias e incubar a 37 ° C durante 5-6 horas.
- Executar uma maxiprep e verificar a inserção da cassete no local correcto por sequenciação.
- Além de sequenciação, realizar uma restrição enzymteste e de digestão com ADN de um clone que não continha qualquer de baixo peso molecular e o seu DNA de plasmídeo parental. As enzimas de restrição seleccionadas será diferente para cada gene, mas o objectivo é o de encontrar um grupo de enzimas que permitem ao investigador para caracterizar o vector recombineered. A título de exemplo, o vetor de direcionamento CG32095 foi sequencialmente digerido com Pacl, Asei, BamHI e Aatll. O aparecimento de todas as bandas preditos e ausência de quaisquer bandas incorrectos confirma que o vector alvo foi correctamente recombineered (Figura 5).
5. Injetando Flies e Mobilização cassete In Vivo
- Injectar a cassete usando ΦC31 integrase, em um local de pouso pré-definido de escolha. Fornecedores externos, tais como o arco transgênicos ( http://www.rainbowgene.com ), podem ser utilizados para os serviços de injecção Importante:. O ADN deve ser recém-prepared antes da injeção. A maior eficiência de transformação é observada quando o DNA é preparado no local e injectada no mesmo dia.
- Para mobilizar a cassete do local de pouso, use Bloomington estoque número 25680 ou 25679. Estes stocks conter Flippase I-Scel e a jusante de um promotor de choque térmico no cromossoma, dois e três, respectivamente. Além disso, eles contêm um hs-hid transgene no cromossoma balanceador e do cromossoma Y. Colete homens do estoque escolhido e cruzar para fêmeas virgens que levam o cassete KO no local de pouso. . Set ~ 30 cruzes e permitem que as fêmeas põem ovos por 2-3 dias NOTA: Recomenda-se para escolher o estoque que está em um cromossomo diferente do local de destino.
- Pais de aleta para um novo conjunto de 30 frascos, e larvas de choque térmico a 37 ° C durante 1 hora, duas vezes por dia, durante três dias consecutivos. Os choques térmicos activar a produção das enzimas; flippase e I-Scel, e o gene de morte celular escondido , Matando os homens e as moscas que transportam cromossomas balanceador. Permitir que os pais nos frascos recém capotou para desovar por 2-3 dias, em seguida, repita os choques térmicos para os frascos, depois de virar os pais em novas taças novamente. Repita este procedimento para vários flips, até que os pais não põem ovos mais.
- Colete fêmeas virgens da descendência F1 das moscas calor chocado, e atravessar para y - w - homens. Apontar para 150-200 cruzes, três virgens e três homens por cruz.
- Tela de progênie F2 em um escopo fluorescente que permite a detecção de RFP no olho. Tela para RFP olhos positivos que são mutantes para os genes brancos e amarelos (y - w -). Este processo de selecção selecciona positivamente para a presença da cassete de RFP e selecciona para a ausência de P [acman] vetor (mini-branco) e o local de destino, o qual é marcado pelo gene NOTA amarelo de tipo selvagem:. É menos tempo -consuming para triagem de RFP no olho, do que a primeira tela para moscas yw. Os eventos de mobilização são muito eficientes durante os choques de calor, resultando em muitas moscas contendo menos RFP no olho, tal como em relação a y - w - moscas.
- Configurar acasalamentos par individuais com cada y - w - RFP + mosca, uma vez que cada evento de mobilização pode ser diferente. Mapear a RFP para o cromossomo correta. Correto direcionamento cromossomo é tipicamente observado em> 95% dos casos.
- Estabelecer ações e verificar se há eventos voltados corretas usando as técnicas padrão para a análise de Southern Blot. Projete a 2 kb sonda upstream (braço esquerdo) ea jusante (braço direito) do gene-alvo, e 2 kb (ou menor, se necessário), abrangendo a ORF excluído. As primeiras duas sondas irá produzir uma banda nos orifícios homozigóticos e heterozigóticos, que não esteja presente no tipo selvagem, enquanto que a ORF não irá produzir uma banda no knockout homozigóticos. Uma rápida PCR fou a ORF é também possível, no entanto, isto depende da ausência de uma banda.
Resultados
Ampliação da LA e os braços de homologia com AR deve produzir 500 produtos bp ea reação de PCR-SOE deve produzir um produto de 1,0 kb (Seções 2,1-2,4; Figura 2). A reação BP realizada na seção 2.5 é tipicamente transformação muito eficiente e bacteriana dos rendimentos produtos 5-100 colônias em média. Quase todas as colônias testadas com PCR verificar mostrar o produto de PCR esperado.
Durante a primeira rodada de recombineering (Seção 3) espera obter colônias 20-40 após a transformação do P digerido [acman]-KO-1.0 que contém o LA e os braços AR em SW102 células. 40-60% destes clones irá transportar o produto de recombinação desejado. O controlo de choque térmico não deve conter muito poucos, se quaisquer colónias, em comparação com as células de calor-chocados. O aparecimento do mesmo número de colónias em ambas as placas de amostra e de controlo normalmente indica a presença do plasmídeo não cortado ou outro contaminante. Neste caso, a experiência Should ser descartado e repetida, desta vez digerindo o P [acman]-KO-1.0 transportando o LA e os braços de AR para conclusão seguindo as instruções descritas na etapa 3.2. Um exemplo de um teste de PCR para a recuperação da região genómica de interesse em P [acman] 1,0-KO no qual foram obtidas colónias ≈ 60 é mostrada na Figura 3. Aqui, 60% das colônias testadas levou o product.Care recombinação desejado deve ser tomado para que os produtos de PCR observados executado no tamanho previsto, desde bandas de PCR aberrantes geralmente indicam eventos recombineering incorretas.
Ocasionalmente há colônias irá crescer após a primeira rodada de recombineering. Utilizando células com eficácia sub-óptima representa a causa mais comum para uma reacção recombineering falhou. Manter as células frias em todos os momentos durante a sua preparação e manipulação suave durante as lavagens (nunca vórtice) é fundamental para a obtenção de células competentes de alta qualidade. Tente sempre usar recém-digeridos / P [acman]-KO-purificada1,0 de ADN para se obter a máxima eficiência.
Às vezes, a primeira rodada dos rendimentos recombineering colônias, mas as colônias não passar no teste de verificação de PCR. Isto indica geralmente produtos recombineering incorrectos, mas um tal resultado pode também indicar problemas potenciais com um dos conjuntos de iniciadores. Para validar o primer PCR-Check-LA, criar uma reação de PCR, em combinação com o attB1-I-SCEI-LA-F. Para verificar a PCR-check-RA configurar uma reação de PCR com o attB2-I-SCEI-RA-R. Em ambos os casos, utilizar o BAC originais como um molde. Essas reações de PCR deverá produzir produtos do tamanho esperado, que irão variar dependendo da sequência dos primers de verificação. Os resultados positivos desta experiência de controle descarta a possibilidade de que uma incapacidade de detectar clones positivos é devido à ineficiência primer.
Se você já tentou a primeira rodada de recombineering várias vezes seguindo o protocolo acima, sem qualquer sucesso e ter realizado todo ocontroles sugeriu, recomendamos a escolha de um novo par de braços de homologia 500 pb. Por razões que não são claras, no momento presente, algumas regiões do genoma são altamente resistentes à recombinação do DNA. Simplesmente concepção de novos braços de homologia e de movê-los para fora ou a partir do gene de interesse, por vezes, resolve estes problemas.
A segunda rodada de recombineering (seção 4) é tipicamente muito mais eficiente. Normalmente, 30-50 colónias foram observadas após a transformação, com 90-95% dos quais contendo a construção desejada. O problema mais comum nesta etapa é a obtenção de colônias falso-positivos. Figura 4 mostra exemplos de falsos e verdadeiros clones positivos após segunda rodada de recombineering (seção 4).
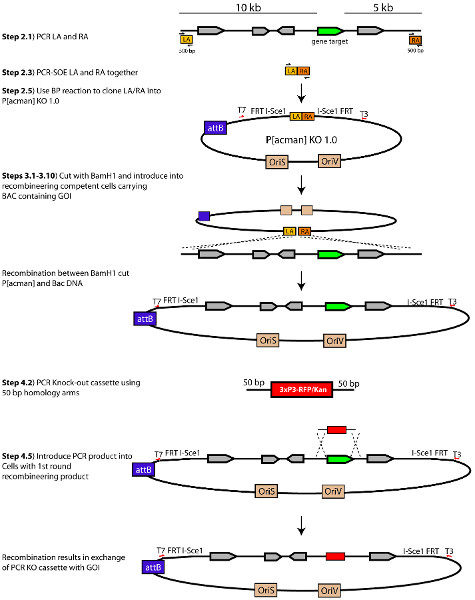
Figura 1. Esquemático do processo para gerar um P [acman]-KO 1,0 vector alvo. Modificado de Chan et ai. (2012). Clique aqui para ver a figura maior .

Figura 2. Esquema de emendas por sobreposição de PCR (PCR SOE), realizado na etapa 2.3. Clique aqui para ver a figura maior .
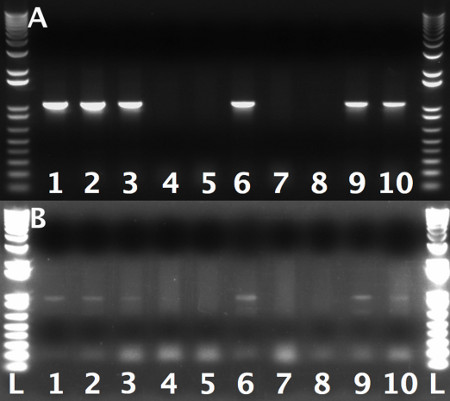
Figura 3. PCR-check gel mostrando a eficiência do primeiro evento recombineering Cada faixa representa uma única colônia A:.. Colônias testadas para o braço esquerdo. integração utilizando os iniciadores T7 e LA-verificação B: Os mesmos colónias como A, que foi testada para a integração braço direito, utilizando T3 e RA-cheque primers.1kb Além escada de DNA foi utilizado como um marcador de peso molecular NOTA:. devido à má iniciação de T3 cartilha em relação a T7, as bandas do cheque braço direito foram significativamente mais escura. Isto deve ser tido em conta durante a visualização do gel.

Figura 4. ADN de gel de agarose que mostra os plasmídeos de elevado peso molecular (P [acman]) e de baixo peso molecular, o plasmídeo (modelo não digerido) isolado de potenciais clones positivos após segunda ronda de recombineering. ADN foi isolado a partir de 4 potenciais clones positivos e correu num gel de agarose a 1% corado com brometo de etídio. Clones 1,3 e 4 são verdadeiros clones positivos. Clone 2 é um falso positivo como foi observado por todas as formas de ap que lasmid corre fole 12 kb (a maior banda de tamanho no DNA escada pista). Além disso, o ADN 1 kb Escada foi usada como um marcador de peso molecular.

Figura 5. Teste de evento de recombinação desejado por enzima de restrição digerir. No exemplo acima, é mostrado um computador previu bandas padrão do P [acman]-KO CG32095 antes e depois da segunda rodada de recombineering. Foram utilizados quatro diferentes enzimas: paci, AscI, Aatll e BamHI. Corretamente produto recombinada terá as mesmas bandas como seu parental P [acman]-KO CG32095 exceto para a mudança pretendida (setas). NEBcutter V2.0 foi utilizado para prever o padrão de banda.
0346/50346fig6highres.jpg "src =" / files/ftp_upload/50346/50346fig6.jpg "/>
Figura 6. Esquemática descrevendo a mobilização de uma cassete alvo in vivo. Clique aqui para ver a figura maior .

Tabela 1. Primers. Clique aqui para ampliar a tabela .
Discussão
O poder de organismos modelo genético em pesquisa biomédica é em grande parte baseado nas ferramentas disponíveis para a manipulação genética. Os modelos pequenos C. elegans e Drosophila em particular, permitir análises genéticas moleculares barato e rápido de caminhos completos e famílias de genes implicados no desenvolvimento multicelular ou função. Nos últimos anos houve avanços significativos no desenvolvimento de ferramentas para manipulação de genes em Drosophila 14,15. Por exemplo, recombineering, que é amplamente utilizado no rato genética para manipular Bac construções de ADN, foi recentemente adaptado para Drosophila, através do uso dos P [acman] vetor e ΦC31 transgénese mediada 6. Diferentes cassetes de seleção permitem a inserção ou deleção de seqüências específicas em qualquer lugar dentro de uma construção utilizando recombinação bactérias competentes 4,13. Temos modificado do original P [acman] vetor de modo que ele pode ser usado para a construção In vivo vectores de recombinação homóloga que visam loci endógenos em Drosophila. Isto permite que um novo vector para gerar o objectivo de cassetes com braços de homologia maior do que aqueles que podem ser facilmente feitas utilizando métodos de clonagem corte e colar. Além disso, uma cassete de clonagem Gateway também foi incorporada neste vector, o que permite uma rápida e eficiente para introduzir novo DNA genómico para o sistema.
Embora a maior parte da metodologia aqui descrita é simples, existem vários passos encontrámos crítico para o sucesso da técnica. Devido ao tamanho do DNA manipulado (> 15 kb), a primeira ronda de reacção recombineering (secção 3) coloca um obstáculo técnico significativo. Por isso, esta rodada de recombineering exige um protocolo de recombinação muito eficiente. Ajustamentos do protocolo aparentemente menor, como o crescimento das células de densidade mais baixa, a lavagem das células três vezes em vez de dois, e com 10% de glicerol em vez de 2 / Sub> O 13, que melhorou muito a eficiência de transformação de nossas células competentes recombineering. Além disso, tendo o cuidado de certificar-se da LA / RA contendo P [acman] é cortado até a conclusão com BamHI (Passo 3.1) elimina muitos falsos positivos.
Eventos de recombinação indesejáveis representam outro problema bastante comum durante recombineering. Por exemplo, temos observado casos de duplicações e translocações dentro de várias construções. Estes eventos podem levar a vários problemas durante a transformação Drosophila e no gene alvo vivo. Verificação PCR e análise de seqüenciamento não consegue detectar todos esses eventos. Assim, uma análise com enzimas de restrição deve ser realizada no produto final clonado antes da injecção de ADN em embriões. Esta verificação final provou crítico e nos permitiu evitar a injecção de construções que contêm aberrações indesejadas.
Maxi preparando-o vetor de direcionamento antes injexão e nunca congelamento do ADN melhora grandemente a eficiência de transformação destas grandes construções em Drosophila. Em suma, pequenos detalhes fazem uma grande diferença no sucesso destas técnicas. Os protocolos delineados e demonstrado aqui representar insights de um número de diferentes fontes e nossa própria experiência.
Enquanto esperamos que o protocolo apresentado aqui ajuda os outros a adotar métodos recombineering em seus próprios laboratórios, acreditamos que novas melhorias e aperfeiçoamentos na metodologia são ainda possíveis. Ocasionalmente, os fragmentos genómicos específicos são difíceis de manipular utilizando recombineering por razões que não são totalmente óbvias. Além disso, apesar de nossos melhores esforços para eliminar o fundo indesejado, ainda encontramos que certos pedaços de DNA genômico tendem a produzir produtos finais incorretamente recombineered. Uma compreensão mais profunda do processo de recombineering pode render mais protocolos ideais na discussão futuro e aberto derecombineering falhas e histórias de sucesso irá promover o sucesso do uso destas técnicas poderosas pela comunidade de pesquisa mais amplo.
Divulgações
Os autores não têm interesses conflitantes no que diz respeito às técnicas descritas aqui.
Agradecimentos
Gostaríamos de agradecer ao Banco de Bloomington Centro de reagentes e Hugo Bellen. Agradecemos mais Koen Venken, Hugo Bellen e todos os membros da Buszczak e Hiesinger laboratórios para discussões úteis. Este trabalho foi financiado por doações do Instituto Nacional de Saúde para ACR (T32GM083831), PRH (RO1EY018884) e MB (RO1GM086647), a concessão pelo Cancer Prevention Research Institute of Texas a MB e PRH (RP100516), eo Welch Fundação (I-1657) para PRH. MB é um EE e Greer Garson Fogelson Scholar em Investigação Biomédica e PRH é um Eugene McDermott Scholar em Investigação Biomédica pelo UT Southwestern Medical Center.
Materiais
| Name | Company | Catalog Number | Comments |
| Name of the reagent | Company | Catalogue number | Comments (optional) |
| SW102 Recombination competent bacteria | NCI-Frederick | Recombination Bacteria (SW102, SW105 and SW106) | http://ncifrederick.cancer.gov/research/brb/logon.aspx |
| TransforMax EPI300 electrocopmpetent E. coli | Epicentre | EC300110 | Includes CopyControl induction solution |
| PfuUltra II Fusion HS DNA Polymerase | Aligent Technology Inc. | 600670 | |
| BamHI-HF | New England Biolabs | R3136S | |
| Zymoclean Gel DNA Recovery Kit | Zymo Research | D4001 | |
| Use Gateway BP ClonaseII Enzyme kit | Invitrogen | 11789-020 | |
| P[acman]KO1.010 | Buszczak and Hiesinger Labs | Upon request | |
| pENTR RFP-Kan11 | Buszczak and Hiesinger Labs | Upon request | |
| Flystocks | Bloomington stock center | Stock numbers: 25680, 25679 | y1 w*/Dp(2;Y)G, P{hs-hid}Y; P{70FLP}11 P{70I-SceI}2B snaSco/CyO, P{hs-hid}4 y1 w*/Dp(2;Y)G, P{hs-hid}Y; P{70FLP}23 P{70I-SceI}4A/TM3, P{hs-hid}14, Sb1 |
| Electroporation machine | Biorad GenePulser Xcell with PC module | 165-2662 | |
| Cuvettes | Fisher Brand | #FB101 |
Referências
- Rong, Y. S., Golic, K. G. A targeted gene knockout in Drosophila. Genetics. 157, 1307-1312 (2001).
- Rong, Y. S., Golic, K. G. Gene targeting by homologous recombination in Drosophila. Science. 288, 2013-2018 (2000).
- Gong, W. J., Golic, K. G. Ends-out, or replacement, gene targeting in Drosophila. Proc. Natl. Acad. Sci. U.S.A. 100, 2556-2561 (1073).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36 (2005).
- Copeland, N. G., Jenkins, N. A., Court, D. L. Recombineering: a powerful new tool for mouse functional genomics. Nat. Rev. Genet. 2, 769-779 (2001).
- Venken, K. J., He, Y., Hoskins, R. A., Bellen, H. J. P[acman]: a BAC transgenic platform for targeted insertion of large DNA fragments in D. melanogaster. Science. 314, 1747-1751 (2006).
- Venken, K. J., et al. Versatile P[acman] BAC libraries for transgenesis studies in Drosophila melanogaster. Nat. Methods. 6, 431-434 (2009).
- Groth, A. C., Fish, M., Nusse, R., Calos, M. P. Construction of transgenic Drosophila by using the site-specific integrase from phage phiC31. Genetics. 166, 1775-1782 (2004).
- Bischof, J., Maeda, R. K., Hediger, M., Karch, F., Basler, K. An optimized transgenesis system for Drosophila using germ-line-specific phiC31 integrases. Proceedings of the National Academy of Sciences of the United States of America. 104, 3312-3317 (2007).
- Chan, C. C., et al. Systematic discovery of Rab GTPases with synaptic functions in Drosophila. Current Biology: CB. 21, 1704-1715 (2011).
- Chan, C. C., Scoggin, S., Hiesinger, P. R., Buszczak, M. Combining recombineering and ends-out homologous recombination to systematically characterize Drosophila gene families: Rab GTPases as a case study. Commun. Integr. Biol. 5, 179-183 (2012).
- Wu, N., Matand, K., Kebede, B., Acquaah, G., Williams, S. Enhancing DNA electrotransformation efficiency in Escherichia coli DH10B electrocompetent cells. Electronic Journal of Biotechnology. 13, (2010).
- Wang, S., Zhao, Y., Leiby, M., Zhu, J. A new positive/negative selection scheme for precise BAC recombineering. Mol. Biotechnol. 42, 110-116 (2009).
- Venken, K. J., Bellen, H. J. Transgenesis upgrades for Drosophila melanogaster. Development. 134, 3571-3584 (2007).
- Dietzl, G., et al. A genome-wide transgenic RNAi library for conditional gene inactivation in Drosophila. Nature. 448, 151-156 (2007).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados