Method Article
における相同組換えコンストラクトを遺伝子改変
要約
相同組換え技術は大きく前進ショウジョウバエ分子正確な突然変異の作成を可能にすることによって、遺伝学。コンビニアリングの最近の採用は1つがDNAの大部分を操作し、にそれらを変換することができますショウジョウバエ 6。方法は、急速に大規模な相同組換えベクターを生成するためにこれらの技術を組み合わせて、ここで提示した。
要約
内因性遺伝子座の高速、大規模な操作のための技術の継続的な開発は、ヒトの疾患関連の研究のための遺伝モデル生物としてキイロショウジョウバエの使用を広げるする。近年では、相同組換えとコンビニアリングなどの技術的な進歩を見てきました。しかし、明確なヌル突然変異またはタギング内因性タンパク質を生成することはほとんどの遺伝子のために相当な努力のまま。ここでは、 インビボで内因性遺伝子座を標的とし、操作するために使用することができるベクターを生成するためにコンビベースのクローニング方法を使用するための技術を記述し、実証具体的には、3つの技術の組み合わせを確立している:(1)BACの形質転換/コンビ、( 2)端部アウト相同組換えおよび(3)ゲートウェイ技術は、内因性遺伝子座を操作するための堅牢な、効率的で柔軟な方法を提供することにある。このプロトコルでは、我々はどのように(1)設計individuaの約ステップバイステップの詳細を提供Lベクトルは、(2)ギャップの修復を使用して相同組換えベクターにゲノムDNAの大断片のクローンを作成する方法、および(3)コンビニアリングの第二ラウンドを使用して、これらのベクター内に目的の遺伝子を交換するか、タグにどのように。最後に、我々はまた、ノックアウト、またはノックイン経由タグ付けされた遺伝子を生成するために、生体内でこれらのカセットを動員する方法のためのプロトコルを提供する。これらの方法は、簡単に並列に複数のターゲットを採用し、タイムリーかつ効率的にショウジョウバエのゲノムを操作するための手段を提供することができます。
概要
彼らの内因性遺伝子座にある単一の遺伝子の分子的に定義された操作をきれいにすることは、真核生物に関連する質問の無数を研究するための貴重なツールを提供しています。 ショウジョウバエは、機能喪失対立遺伝子を生成するための遺伝学的手法を逆Golicと同僚に導入するまで挑戦することが証明されていた生体内遺伝子はショウジョウバエ 1-3に相同組換えを利用したターゲット。彼らは、その特定のゲノム遺伝子座が統合トランスジェニック構築物からのDNAの線状断片を用いて標的を実証することができる。この線形"ドナー" DNAはメガヌクレアーゼI-SceIを持つ線形続いFRT媒介組換え(物品税に環状分子として染色体からDNA)を通して生体内で生成されます。この方法は正常に定義された病変の様々を生成するために使用されているが、この技術は、並列で多数の遺伝子を操作するために容易に拡張されていないため、各INDIVIデュアルノックアウトコンストラクトははっきりとカスタム設計を必要とします。シームレスクラシカル制限酵素/ライゲーションクローニングやPCRならびに生体形質転換ベクターの伝統のサイズ制限はしばしば標的相同組換えの迅速な作成に干渉を用いたin vitroでの DNAの大断片(> 5 KB)を操作することで例えば、困難ベクトル。これらの制限を克服するために、我々はそれを効率的かつ比較的急速なプラットフォームを確立するための方法論をターゲット両端アウト遺伝子とDNAの100キロバイトまでのサブ·クローニングと遺伝子導入を可能にしますコンビニアリング/トランスジェネシスP [acman]システムを、組み合わせショウジョウバエの遺伝子ターゲティングが容易になります。
組み換え媒介遺伝子工学(コンビニアリング)は4,5パワフル相同組換えベースのクローン技術です。従来の制限酵素/リガーゼクローニングとは対照的に、コンビは、シーケンスまたはSIZに限定されるものではない操作したDNAの電子。コンビニアリングは、特別なE.を使用しています不良λプロファージ4で提供される組換え機構を宿す大腸菌株 。この技術は、最近ショウジョウバエ 6,7に採用されている。 ショウジョウバエのコンビニアリングはP [acman] 6,7と呼ばれる修正された条件に応じて増幅細菌人工染色体(BAC)ベクターに依存しています。 ORIV、シーケンシングおよび胚の注入および基底条件下で低コピー数を維持オリスに必要なDNAの大量の精製のための化学誘導時に高コピー数を生成します。このベクターは、複製の二起源を運ぶ。さらに、P [acman]ベクターは細菌の付着(のattB)サイトが装備されています。のattBサイトは、 ショウジョウバエのゲノム8,9内の所定の着陸地点に大きなDNA断片の取り込みを可能にΦC31インテグ媒介形質転換のための基質として機能します。
我々は、両端アウト相同組換え10,11のためのターゲティングベクターとして使用することができるP [acman]ベクトル(P [acman]-KO 1.0と呼ぶ)が生成されている。システムに技術をターゲット両端アウト遺伝子組み込むには、我々は2つのFRTと2 I-SceI制限酵素サイトを追加しました。また、P [acman]-KO 1.0にホモロジーアームを組み込むプロセスを合理化するために、この修正されたベクター内のゲートウェイカセットが含まれている。これは、ターゲティングベクターに関心の事実上すべてのゲノム領域を導入するための迅速かつ簡単な方法を提供します。このプロトコルでは、P [acman]-KO 1.0、そしてどのように内因性遺伝子座を標的とするin vivoでこのベクトルを動員するを使用してターゲティングベクターを設計する方法について説明します。このプロトコルの目的のために、我々は、目的の遺伝子を置換するRFP /菅カセットを使用するが、抗生物質選択マーカーを含有するカセットの様々なこのプロトコルで使用することができる。私たちは、カセットのセットのために設計されており、正常に使用している遺伝子置換およびタギング10,11。
プロトコル
1。ターゲットへのBACと地域の選択
- 目的の遺伝子(GOI)(ここでCG32095を例として使用されている)、でCG32095の検索でBACを取得するwww.flybase.org 。セクションの株式や試薬の下では、CG32095を含むBACをするため、 "ゲノムクローン"と題するセクションを確認してください。 BACは、少なくとも10キロバイト、上流と5キロバイト関心の下流の遺伝子が含まれていることを確認してください。また、P [acman]ベクトル7のクローンはまたで見つけることができますhttp://www.pacmanfly.org/libraries.html 。
- BACクローンはDH10B細胞とEPI300細胞に来るP [acman]クローンで来る。ゲノムクローンはコンビニアリングのために適しているSW102細胞に変換する必要がある。 "ダーティミニプレップ"が始まる(カラムを使用し、イソプロパノールを用いてDNAを沈殿させずにキアゲンミニプレップ)が行われる5ミリリットルO / N培養からBACを含む細胞を接種した。のdH 2 O20μlにたてDNAペレットの再懸濁後、のdH 2 O100μlの中に1μLを希釈し、エレクトロSW102をエレクトロに1μlのを使用しています。エレクトロSW102は、以下のプロトコルを使用するには:
- LB 4mlのにSW102細胞に接種。 32で文化を育てる°CO / N 250 rpmで振盪重要:再結合機械の誘導が望まれている限り、SW102細胞は、32°C以上の温度にさらされるべきではありません。
- 44ミリリットルのLBに飽和SW102文化を1mlに接種し、0.2〜0.3(通常は約1〜1.5時間)の間のOD 600から 32°Cシェーカーに成長注:その他コンビニアリングプロトコルはに小さい音量で文化を成長勧めOD 600 0.6〜0.7ただし、より大きな容積に低い密度に細菌の成長が大幅変換efficieを増加させるNCY 12。また、本変形例では、待ち時間を低減する。
- 5分間氷水スラリー中の文化を冷やす重要:次の手順の間に細胞が低温に保たなければなりません。これは上質なエレクトロ細胞を得るために重要です。
- 10分間、1,500×gで4℃で文化を遠心分離します。上清を捨て、冷たい25mlの(4℃)を10%グリセリン13でペレットを洗浄する。氷水スラリーにチューブをタップし、渦巻くによって5ml中まず、再懸濁した細胞(UPピペットAND DOWNはいけません )し、残りの10%グリセロール(20ミリリットル)を追加します。
- 10分間、1,500×gで4℃で遠心分離し、上清を捨て、25ミリリットルの氷冷10%グリセロールでペレットをもう一度洗う。
- 10分間、1,500×gで4℃で遠心分離し、上清を捨て、25ミリリットルの氷冷10%グリセロールでペレットを3回洗う。
- 10分間1500×gで4℃で遠心し、商標捨てるernatant。細胞ペレットは、この時点で非常にゆるんでいる可能性があり、いつ破棄上澄み注意が必要です。冷10%グリセロール1mlにペレットを再懸濁します。 1.5mlチューブに細胞を移し、4℃で卓上遠心で12,000×gで30秒間遠心分離グリセロール/セルの≈90μLを残し上清のほとんどを捨てる。細胞は今やエレクトロする準備が整いました注:この時点で、細胞を°C、後で使用するために-80℃で保存することができます。しかし、これは能力を低下させることができる。
- 1,800 V、25μF、200Ωで1ミリメートルキュベットを用いて細胞にDNAをエレクトロ。エレクトロポレーション後、SOCの300μlを添加し、細胞が32°C(揺れは必要ありません)で2時間で回復しましょう。プレートLB寒天上の細胞を加えた25μgの/ mlのクロラムフェニコール(または変換BACまたはPACに応じて、他の適切な抗生物質)と24-30時間32℃でインキュベートする。
- 標的遺伝子に対するホモロジーアームは、選択によって設計されていますGOIの相対川下り上流のゲノムDNAの10キロバイトと5キロバイト増幅するために10キロバイトの終わりに500 bpのと5キロバイトホモロジーアーム(セクション2を参照)( 図1)を選択します。これらの500 bpの断片は、P [acman] KO 1.0にGOIを取り巻くゲノム領域を再結合するために使用されるホモロジーアームとして役立つ。これらのフラグメントは、ホモロジーアーム'下流の3に相当する地域のホモロジーアームと右腕(RA)'上流5に対応する領域のために左腕(LA)と呼ばれています重要 :ホモロジーアームをBamHIを含むべきではありません制限部位は、プラスミドのような線形化する(ステップ3.2)にBamHI制限酵素消化によって達成される。
2。 P [acman]-KO 1.0にホモロジーアームを挿入
- 2個々のPCR反応を設定し、1は、RAを増幅するためにLAと別のものを増幅する。 LAには、プライマーattB1-I-SceI制限酵素-LA-F及びBamHI-LA-Rを使用し、プライマーのBamHI-RA-FとattB2-I-SceI制限酵素-RA-Rを使用しRA( 表1)を増幅した。テンプレートとして関心のBACを含む茹で一晩細菌培養のステップ1.2または0.5μLからBAC DNAを1μlを使用する。能力を校正とDNAポリメラーゼを使用してください。これは、後続のすべてに適用されたPCR-でステップ2.8と3.13でのPCR検査の例外注:プルーフリーディングDNAポリメラーゼ用延長時間はメーカーによって、正規のTaq DNAポリメラーゼとは異なる場合があります。 15秒/ KBの割合で重合PfuUltra II融合HotStartのは、次の手順で使用することができます。
LA / RA PCR
PCR条件1μlの バクのステップ1.2から汚れたミニプレップからのDNA 0.25μlの各 20μMのプライマー 2.5μL 10X PfuUltra IIバッファ μlの0.75 10mMの(それぞれ)のdNTP 0.5μL PfuUltra II融合HotStartのDNAポリメラーゼ 19.75μlの 水 25μlの 全容積 ステップ1 酵素を活性化するために95℃で2分 STEP2 95℃20秒の変性 ステップ3 60℃、20秒アニーリング ステップ4 72℃20秒延長 ステップ5 ステップ2と29のサイクルを繰り返すに行く ステップ6 4°Cホールド - 1%アガロースゲル上でサンプルを実行するとZymoclean DNA回復キットを使用した製品を抽出します。滅菌水10μlのとZymocleanカラムからDNAを溶出する。
- (PCR S拡張を重ねてスプライシングを行う2.2( 図2)で精製した各増幅産物から全DNAの10〜30 ngの断片を用いて二付きOE)。これは通常の周りにそれぞれ精製したPCR産物の1/20 番目です。
LA / RA PCR-ソー
PCR条件0.5μlの各 LAおよびRA精製したPCR産物を約10〜30 ngの 0.25μlの各 20μMのプライマー 2.5μL 10X PfuUltra IIバッファ μlの0.75 10mMの(それぞれ)のdNTP 0.5μL PfuUltra II融合HotStartのDNAポリメラーゼ 19.25μlの 水 25μlの 全容積 ステップ1 酵素を活性化するために95℃で2分 STEP2 95℃20秒の変性 ステップ3 55℃で20秒(アニーリングする武器を可能にするために低い温度。)アニーリング ステップ4 72℃20秒延長 ステップ5 ステップ2と1サイクルを繰り返すに行く ステップ6 95℃20秒の変性 STEP7 60℃、20秒アニーリング ステップ8 72℃20秒延長 ステップ9 ステップ2と27のサイクルを繰り返すに行く STEP10 4°Cホールド - アガロースゲルでPCRサンプルを実行し、ゲル抽出により1.0キロバイトバンドを回復。これはattB1-LA-のBamHI-RA-attB2カセットです。
- メーカーが提供するプロトコルに従ってBP反応を設定するゲートウェイBP ClonaseII酵素キットを使用してください。2.4で生成されたカセットは、ドナーDNAおよび宛先ベクトルとしてP [acman]-KO 1.0として動作します。
- 冷10%グリセロールまたはコールドのdH 2 O30μlのを追加することによってTransforMax EPI300エレクトロ細胞の50μlの希釈メーカーが提供するプロトコルに従ってエレクトロ細胞にBP反応液1μlを変換します。水の添加は、エレクトロポレーション時の放電(アーク放電)を防止するために、BP反応のかなり高い塩濃度を希釈。 1,800 V、25μF、200Ωで1ミリメートルキュベットを用いて細胞にDNAをエレクトロ。
- SOCの300μlを添加し、細胞を37℃(動揺は必要ありません)で1時間で回復しましょう。 LB-寒天上で平板セル+アンプ50μgの/ mlの(P [acman]-KO 1.0アンプの耐性遺伝子が含まれています)。 18-24時間、37℃でプレートをインキュベートする。
- PCRは、T3用いた従来のコロニーPCRによって個々のコロニーを確認してください(T3シーケンスはP [acman]-KO 1.0( 図ゲートウェイカセットの下流にあるURE 1)及びattB1-I-SceI制限酵素-LA-Fプライマー。陽性クローンは1.2キロバイトで動作バンドが表示されます。
- 50μgの/ mlのアンピシリンを含むLBで陽性クローンの一晩培養を育てる。
- 製造元のプロトコール(エピセンター)に続いてコピー制御ソリューションを使用して50μgの/ mlのアンピシリン文化とを10mlのLBにLAやRAを含むP [acman]-KO 1.0ベクトルを増幅する。
- 製造業者のプロトコルに従ってキアゲンミニプレップを実行します。
3。 P [acman]-KO 1.0に関心のゲノム領域を再結合
1日目
- LB 4mlのプラス25μgの/ mlのクロラムフェニコール(クロロフィル)、または適切な選択抗生物質への関心のBACを含むSW102細胞に接種。 32で文化を育てる°CO / Nが250 rpmで振盪重要:高温組み換えMAを活性化するように加えてステップ3.5誘導中、SW102細胞は、32℃より高い温度にさらされるべきではありませんこれらの細胞のチナリー。
2日目
- 25μlの反応をBamHIの≈20単位でセクション2で生成されたLAやRA武器を含むP [acman]-KO-1.0のダイジェスト0.4μgの。少なくとも3時間37℃でインキュベートする重要:ロング消化はすべてDNAの消化を確保することが重要です。これは、後に偽陽性クローンの数が減少します。
- 44ミリリットルLB-クロロフィル(25μgの/ ml)に飽和SW102/BAC文化の1.0ミリリットルを接種すると、0.2〜0.3(通常は約1〜1.5時間)の間のOD 600から 32°Cシェーカーで育つ。非HS制御用の追加の同一のサンプルが含まれています。このサンプルは、単にHSを除いて、あらゆる局面での実験的なもののように扱われます。この非熱ショックを与えた文化は、ネガティブコントロールとして機能します。非HS制御板(ステップ3.13)に多くのコロニーの外観は通常消化P [acman] KO 1.0 LAで形質転換を示すプラスミド/ RA。
- 文化が成長している一方で、ゲル上のBamHI-制限P [acman]-KOを実行します。その後、ゲル抽出し水10μlのDNAを溶出は55まで温め℃まで注記:任意のバッファーでDNAを溶出しないでください。バッファ内に存在する塩は、エレクトロポレーション中に放電が発生することがあります。
- 水浴中で正確に15分間、42°Cで熱ショック文化。これは、SW102細胞のコンビニアリング機械がアクティブになります。対照培養ショックを加熱しないでください。
- すぐに冷やした50ミリリットル正規チューブに熱ショック、転送熱ショックを与えたと非HS制御培養した後、5分間氷水スラリー中の文化の寒さを聞かせて重要:次の手順の間に細胞が低温に保たれなければならない(MUST) 。これは上質なエレクトロ細胞を得ることが重要である。
- 10分間、1,500 xgで文化を遠心分離します。上清を捨て、冷たい25mlの(4℃)を10%グリセリン13でペレットを洗浄する。まず、氷水スラリー(UPピペットAND DOWNしないでください)、その後、残りの10%グリセロール(20ミリリットル)を追加でチューブをタップし、渦巻くによって5mlに、細胞を再懸濁します。
- 10分間、1,500×gで4℃で遠心分離し、上清を捨て、25ミリリットルの氷冷10%グリセロール二度目でペレットを洗浄。
- 10分間、1,500×gで4℃で遠心分離し、上清を捨て、25ミリリットルの氷冷10%グリセロール三度目でペレットを洗浄。
- 10分間、1,500×gで4℃で遠心し、上清を捨てる。細胞ペレットは、この時点で非常にゆるんでいる可能性があり、いつ破棄上澄み注意が必要です。冷10%グリセロール1mlにペレットを再懸濁します。 1.5mlチューブに細胞を移し、4℃で卓上遠心で12,000×gで30秒間遠心分離水/セルの≈90μLを残し上清のほとんどを捨て注:この時点で、細胞を-80℃で保存できます、後で使用するためのC。しかし、これは能力を低下させることができる。
- 細胞にBamHIで消化P [acman](50 ngのための目安)2μLを加え、穏やかにピペットチップを使用して混ぜる。 1ミリメートルエレクトロポレーションキュベットに細胞を移し、1,800 V、25μF、200Ωでエレクトロ。
- SOCの300μlを添加し、細胞が32°C(揺れは必要ありません)で2時間で回復しましょう。プレートLB寒天上の細胞プラス24-30時間、32℃で50μgの/ mlのアンピシリンとインキュベート。
3日目
- コロニーPCRを用いてギャップ修復適切用スクリーン。 T3使って、RAはRAとT7とLAのためにLAチェックプライマー( 表1)のためのプライマーを確認してください。 PCR-チェック-RAとターゲット地域に向けたPCR-チェックLAプライマー100-200 bpの( 表1)を設計。 T7とT3またはPCR-チェック-LAでPCR-チェック-RAを用いたPCR反応から陽性コロニーは、800〜1,000 bpの( 図3)で実行されるバンドが表示されます注:これは、1つの組み込み以来両腕をテストすることが重要ですアームそれ以外ではないが、時々観察される。
- LB +50μgの/一晩のためにアンピシリン4mlに32℃でPCR-検証コロニーを育てる。後で使用するために、これらの細胞の凍結ストックを作る。
4。ターゲットカセットとゲノム領域を交換する
4日目
- テンプレートと5'HA-RFP /菅-Fと3'HA-RFP /菅-Rプライマー(としてpENTR-RFP/KANを用いてPCR(線形化するためのAscI及びNheIで前消化)でRFP / KANノックアウトカセットを増幅表1)。 RFP / KANカセット参照11から要求することができます。
- ゲル滅菌水20μlの溶出PCR産物を精製する。 RFP-KANカセットプラスホモロジーアームは3キロバイトの周りを実行する必要があります。
- 44ミリリットルLB-アンプ(50μgの/ ml)に飽和SW102 / P [acman]-KOの文化(ステップ3.13)を1ml接種と°CシェーカーOD 600 0.2から0.3の間に32に成長。非HS制御用の追加の同一のサンプルが含まれています。このサンプルはわずかに扱われるHSを除いて、あらゆる局面での実験のような。
- SW102 / P [acman]-KO文化が所望の濃度に達した後、3.5から3.10までの手順に従ってください。
- エレクトロSW102 / P [acman]-KO細胞90μlのにカセットをターゲットに約100 ngのをエレクトロ。 1ミリメートルエレクトロポレーションキュベットに細胞を移し、1,800 V、25μF、200Ωでエレクトロ。 SOCの300μlを添加し、細胞が32°C(揺れは必要ありません)で2時間で回復することができます注:これは、高度に精製されたカセットを新鮮なゲルを使用することを推奨します。
- LB寒天+アンプ(50μgの/ ml)を+菅(50μgの/ ml)と32で24-30時間インキュベート℃の中でプレート細胞
5日目
- 32°CO / Nで5つの異なるコロニーから文化を5mlを育てる
6日目
- "ダーティミニプレップ"を実行し、PRを探すために、1%アガロースゲル上で得られたDNAの半分を実行プラスミド低分子量のesence。怒鳴る12キロバイトを実行する任意のプラスミドであってはならない。 ( 図4)
- 陽性クローンからのDNAの1:500希釈を行い、TransforMax EPI30050μlのにエレクトロに1μlの使用重要 :1つのセルに複数のプラスミドのエレクトロポレーションを防ぐために、DNAを希釈してください。 LB寒天+アンプ(50μgの/ ml)の中にプレート細胞+菅(50μgの/ ml)と37℃で18-24時間インキュベート°C
7日目
- LB 10mlのシングルコロニーを植菌+とO / Nを育てる
8日目
- ステップ4.10から飽和文化とLB培地の種子100ミリリットル。適切な抗生物質を加えた5-6時間、37℃で1,000 Xコピー制御ソリューションとインキュベートを100μlを加える。
- maxiprepを実行し、配列決定することにより、正しいサイト内のカセットの挿入を確認します。
- 配列決定に加えて、制限enzymを行う任意のプラスミド低分子量とその親のDNAが含まれていなかったクローンからのDNAとのe消化テスト。選択した制限酵素は、各遺伝子の異なるであろうしかし、ゴールはrecombineeredベクトルを特徴付けるために調査員にallow酵素群を見つけることである。一例として、CG32095ターゲティングベクターが直列PacIで、AscIで、BamHIとをAatIIで消化し た。間違ったバンドのすべての予測バンドや不在の外観は、ターゲティングベクターが正しく( 図5)recombineeredされたことを確認。
5。ハエを注入し、生体内でカセットを動員
- 選択肢の事前定義された着陸地点に、ΦC31インテグラーゼを用いたカセットを注入する。このようなレインボートランスジェニック(などの外部ベンダー、 http://www.rainbowgene.comが )、注射サービスに使用することができます重要:DNAがたてPでなければならない注入前repared。 DNAは、現場で調製され、同じ日に注入されると、より高い変換効率が観察される。
- 着陸地点からカセットを動員するには、ブルーミントン在庫番号25680または25679を使用しています。これらの株式は、それぞれ染色体2と3、上の熱ショックプロモーターのフリッパーゼとI-SceIを下流に、含まれています。さらに、彼らはバランサー染色体上とY染色体上のHS-HID導入遺伝子が含まれています。選択された在庫から男性を収集し、着陸地点でKOカセットを運ぶ処女雌に渡る。 〜30十字架を設定し、女性は2〜3日のために卵を産むことができ注:これは、標的遺伝子座とは異なる染色体上にある株を選択することをお勧めします。
- フリップ30バイアルの新しいセットに両親、三日間連続で1時間37℃で熱ショックの幼虫、1日2回。ヒートショックは、酵素の産生を活性化する;フリッパーゼとI-SceIは、細胞死遺伝子HIDを 、バランサー染色体を運ぶ男性とハエを殺し。 2〜3日のために卵を産むための新た反転バイアル中に両親を許可してから、再度新しいバイアルに両親を反転した後、それらのバイアル用熱ショックを繰り返す。両親はもう卵を産むなくなるまで、いくつかは、反転のためにこれを繰り返します。
- 熱ショックを与えたハエのF1子孫から処女雌を収集し、yにクロス- W -男性。 3処女とクロスあたり3人の男性; 150-200十字架を目指す。
- 目におけるRFPの検出を可能にする蛍光スコープの下にF2子孫をスクリーニング。 白と黄色の遺伝子( - W - y)のための変異体であるRFP陽性の目のための画面。このスクリーニングプロセスは、積極的にRFPカセットの有無を選択し、野生型黄色遺伝子によってマークされているP [acman]ベクトル(ミニホワイト)と着陸地点の不在のために選択されます注:これは、より少ない時間である-CONYWのハエのための最初の画面に比べて、目にRFPのための画面にsuming。 W - -ハエ動員イベントはyに比べて、目にRFPを含む多くの少ないハエで、その結果、熱ショック時に非常に効率的です。
- 各動員イベントは異なる場合がありますので、RFP +フライ- W -各Yとシングルペア交配を設定します。正しい染色体にRFPをマップします。正確な染色体ターゲティングは、典型的には、ケースの> 95%で観察される。
- 株式を確立し、サザンブロット解析のための標準的な技術を使用して正しいターゲットイベントを確認。 2キロバイトプローブ上流(左腕)と下流(右腕)遺伝子が標的とされているのと、削除されたORFにまたがる2キロバイト(または必要に応じてより短い)を設計。最初の二つのプローブは、ORFがホモ接合ノックアウトでバンドが得られないでしょうが、野生には存在しない、つまり、ホモ接合体とヘテロ接合体ノックアウトでバンドが得られます。迅速PCR FまたはORFも可能であるが、これはバンドの有無に依存している。
結果
LAやRAのホモロジーアームの増幅は500 bpの製品を生産する必要がありますし、PCR-SOE反応が1.0キロバイト製品(セクション2.1から2.4、 図2)を得る必要があります。セクション2.5で実行BP反応は、通常、製品の歩留まり平均で5-100コロニーの非常に効率的で、細菌の変換です。ほぼPCR検査で試験したすべてのコロニーは、予想されるPCR産物を示す。
コンビニアリングの最初のラウンド(第3節)の間、SW102細胞にLAやRA武器を含む消化P [acman]-KO-1.0の変換後の20〜40個のコロニーを得ることを期待しています。これらのクローンの40〜60%は、目的の組換え産物を運ぶでしょう。熱ショックを与えた細胞と比較して、非熱ショックコントロールはコロニー場合ごく少数含まれている必要があります。試料とコントロールプレート上のコロニーの両方に同じ数の外観は、通常、未切断プラスミドの存在下または他の汚染物質を示す。この場合、実験寿LDは破棄され、繰り返され、今回はP [acman]は-KO-1.0は、ステップ3.2で説明した命令の次の補完にLAやRA武器を運んで消化する。 ≈60のコロニーが得られたP [acman]-KO 1.0に関心のあるゲノム領域の検索のためのPCR検査の一例が図3に示されている。ここではコロニーの60%が実施し異常のPCRバンドは、通常、誤ったコンビニアリングのイベントを示すことから、目的の組換えproduct.Careが、観測されたPCR産物は、予測サイズで実行することを注意しなければならないテスト。
時折全くコロニーはコンビニアリングの最初のラウンドの後に成長しません。準最適効率で細胞を使用すると、失敗したコンビ反応のための最も一般的な原因を表す。洗浄中にそれらの製造と優しい取り扱い(NEVER渦)中にすべての回で風邪の細胞を保つことは、高品質コンピテント細胞を得るために重要である。いつも使用してみて新たに消化/ P [acman]-KO-の精製最高の効率を得るために1.0 DNA。
たまにコンビニアリング利回りコロニーの最初のラウンドが、それらのコロニーは、PCR検査検定に合格しない。これは通常、間違ったコンビ製品を示すが、このような結果はまた、プライマーセットのいずれかの潜在的な問題を示すことができる。 PCR-チェックインLAプライマーを検証するには、attB1-I-SceI制限酵素-LA-Fとの組み合わせでPCR反応を設定します。確認するにはPCR-チェック-RAはattB2-I-SceI制限酵素-RA-Rを用いたPCR反応を設定します。どちらの場合も、テンプレートとして元BACを使用しています。これらのPCR反応は、チェックプライマーの順序によって異なります予想されたサイズの製品が得られるはず。この対照実験の陽性結果は陽性クローンを検出することができないことは、プライマーの非効率性に起因する可能性を排除する。
何か成功することなく、上記のプロトコルに従って数回コンビニアリングの最初のラウンドを試してみましたが、すべて実行した場合コントロールを提案し、我々は500 bpの相同性アームの新しいペアを選択することをお勧めします。現時点では明確ではない理由のために、いくつかのゲノム領域は、組換えDNAに対して非常に耐性がある。単に新しいホモロジーアームを設計し、目的の遺伝子の方にまたはから離れてそれらを移動すると、時々、これらの問題を解決します。
コンビニアリングの第二ラウンド(第4節)は、一般的にはるかに効率的です。通常は30〜50個のコロニーを所望の構築物を含むこれらの90〜95%で、形質転換後に観察される。この段階で最も一般的な問題は、偽陽性コロニーを取得している。 図4はコンビニアリングの第二ラウンド(第4節)の後に真と偽陽性クローンの例を示します。
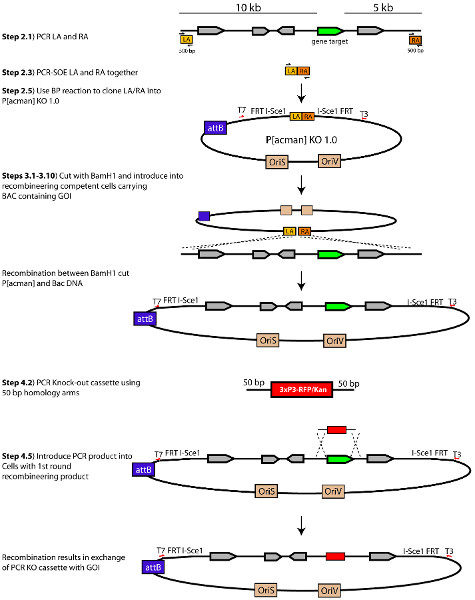
図1。 Pの概略rocessはP [acman]を生成する-KO 1.0ターゲティングベクター。チャンらから変更。 (2012)。 より大きい数字を表示するには、ここをクリックしてください 。

図2。ステップ2.3で実行PCR(PCR SOE)の重複によるスプライシングの概略は 大きい数字を表示するには、ここをクリックしてください 。
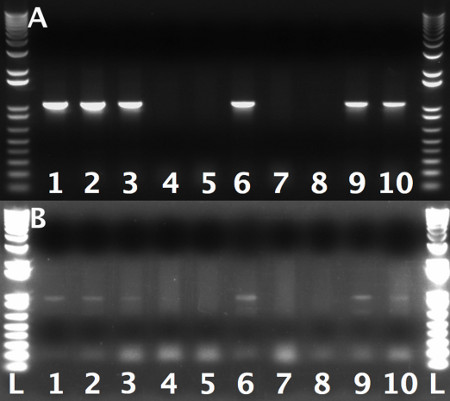
図3。最初のコンビニアリングイベントの効率を示すゲルをPCR-チェック各レーンは、単一のコロニーを表します。左腕をテストコロニーをT7とLA-チェックプライマーを用いて統合B:。、T3使って右腕の統合のためにテストし、RA-チェックprimers.1kbプラスDNAラダーは、分子量マーカーとして使用されたのと同じコロニーは注 :T3の貧しいプライミングによりT7に比べてプライマーは、右腕チェックのバンドがかなり暗くなりました。ゲルを表示するときに考慮されるべきである。

図4。 DNAアガロースゲルは、DNAは4潜在的な陽性クローンから単離した。高分子量のプラスミド(P [acman])とコンビニアリングの第二ラウンド後の潜在的な陽性クローンから分離された低分子量のプラスミド(未消化テンプレート)された表示と、1%アガロースゲル上で走ったエチジウムブロマイドで染色した。クローン1,3および4は、真の陽性クローンです。クローン2は、APのすべての形式で述べたように偽陽性の一つです lasmidそれは怒鳴る12キロバイト(DNAラダーレーンで最大サイズのバンド)を実行します。 1キロバイトプラスDNAラダーを、分子量マーカーとして使用した。

図5。制限酵素消化によって所望の組換え事象のためのテスト。上記の例で示されていますコンビニアリングの第二ラウンドの前後にP [acman]-KO CG32095のパターンをバンディングコンピュータが予測された。四つの異なる酵素が使用されました:PacIで、AscIで、をAatIIとBamHI。正しく組み換え製品が意図された変化(矢印)を除いて、その親のP [acman]-KO CG32095と同じバンドを持つことになります。 NEBcutter V2.0は、バンドパターンを予測するために使用された。
0346/50346fig6highres.jpg "SRC =" / files/ftp_upload/50346/50346fig6.jpg "/>
生体内で標的カセットの動員を記述する図6回路図。 より大きい数字を表示するには、ここをクリックしてください 。

表1プライマー。 大きいテーブルを表示するには、ここをクリック 。
ディスカッション
生物医学研究における遺伝的モデル生物の力は、主に遺伝子操作のために利用可能なツールに基づいています。小さ なモデルC.特に虫とショウジョウバエは、多細胞発達や機能に関与して、完全な経路および遺伝子ファミリーの、安価で高速な分子遺伝学的解析が可能になります。近年は、 ショウジョウバエ 14,15の遺伝子を操作するためのツールの開発に大きな進歩を見てきました。例えば、広くバクのDNA構築物を操作するためにマウス遺伝学で使用されるコンビは、最近、P [acman]ベクトルとΦC31-媒介形質転換6を使用することにより、 ショウジョウバエのために適合させた。異なる選択カセットはどこにも換え有能な細菌を4,13を使用して構造内の特定の配列の挿入または削除が可能になります。それは構築するために使用することができるように、我々は、元のP [acman]ベクトルを変更した ショウジョウバエにおける内因性遺伝子座を標的生体相同組換えベクターである。この新しいベクターは容易にカット&ペーストクローニング法を用いて作製することができるものよりも大きな相同性アームとターゲティングカセットを生成するために1つを可能にする。また、ゲートウェイクローニングカセットはまた、1つの迅速かつ効率的にシステム内に新たなゲノムDNAを導入することができ、このベクターに組み込まれている。
ここで説明する方法論のほとんどは簡単ですが、我々は技術の成功のための重要な発見したいくつかのステップがあります。操作DNA(> 15キロバイト)のサイズのため、コンビニアリング反応の最初のラウンド(第3節)が重要な技術的ハードルを提起。したがって、コンビニアリングのこのラウンドでは、非常に効率的な組換えプロトコルが必要です。三回の代わりに二つ、より低い密度に細胞を増殖する細胞を洗浄のような、代わりのdH 2の10%グリセロールで一見マイナープロトコルの調整、</サブ> O 13は 、非常に我々のコンビニアリングテントセルの形質転換効率を改善している。加えて、確認するために世話をしてLA / RA含むP [acman]をBamHIする(ステップ3.1)は、多くの偽陽性を排除で完全にカットされています。
望ましくない組換え事象はコンビニアリング中に別のかなり一般的な問題を表しています。例えば、様々な構築物の中に重複し、転座のケースを観察した。これらのイベントは、 ショウジョウバエの変態中および生体内標的遺伝子内の複数の問題につながることができます。 PCRの確認および配列決定分析は、これらすべてのイベントを検出することができない。従って、制限酵素分析は、胚へのDNAの注入の前にクローニングし、最終生成物に対して行われなければならない。この最終チェックは重要な証明していると私たちは望ましくない収差を含むコンストラクトを注入避けることができました。
ただINJ前ターゲティングベクターをマキシ準備中ECTIONとDNAを凍結することはありませんが大幅にショウジョウバエにこれらの大規模な構造物の形質転換効率を向上させます。要するに、小さな詳細が、これらの技術の成功に大きな違いを生む。プロトコルが概説され、さまざまなソースや私たち自身の経験の数から洞察力を表し、ここで明らかにした。
我々はここで紹介するプロトコルは、他の人が自分の研究室でコンビニアリングの方法を採用するのに役立ちます願っているが、我々は方法論のさらなる改善と改良は可能であると信じています。時々、特定のゲノム断片は、完全に自明ではない理由のためにコンビを使用して操作することが困難である。さらに、不要なバックグラウンドを除去するために最善の努力にもかかわらず、我々はまだゲノムDNAの特定の部分が誤っrecombineered最終製品を生産する傾向があることがわかります。コンビニアリングプロセスのより深い理解が、将来の、オープンな議論で、より最適なプロトコルをもたらすかもしれませんコンビニアリングの失敗と成功の物語はより広範な研究コミュニティによって、これらの強力な技術の使用の成功を促進します。
開示事項
著者は、ここで概説技術に関しての任意の競合する利益を持っていません。
謝辞
私たちは、ヒューゴBellenと試薬用ブルーミントンストックセンターに感謝したいと思います。我々はさらに有用な議論のために公園Venken、ヒューゴBellenとBuszczakとHiesingerラボのすべてのメンバーに感謝します。この作品は、ACRの国立衛生研究所(T32GM083831)、PRH(RO1EY018884)からの補助金によってとMB(RO1GM086647)、MBとPRH(RP100516)にテキサス州のがん予防研究所によって助成金、およびウェルチにサポートされていましたPRHへ財団(I-1657)。 MBは、生物医学研究におけるEEとグリア·ガーソンFogelson大学者で、PRHは、UTの南西医療センターの生物医学研究におけるユージンマクダーモット学者です。
資料
| Name | Company | Catalog Number | Comments |
| Name of the reagent | Company | Catalogue number | Comments (optional) |
| SW102 Recombination competent bacteria | NCI-Frederick | Recombination Bacteria (SW102, SW105 and SW106) | http://ncifrederick.cancer.gov/research/brb/logon.aspx |
| TransforMax EPI300 electrocopmpetent E. coli | Epicentre | EC300110 | Includes CopyControl induction solution |
| PfuUltra II Fusion HS DNA Polymerase | Aligent Technology Inc. | 600670 | |
| BamHI-HF | New England Biolabs | R3136S | |
| Zymoclean Gel DNA Recovery Kit | Zymo Research | D4001 | |
| Use Gateway BP ClonaseII Enzyme kit | Invitrogen | 11789-020 | |
| P[acman]KO1.010 | Buszczak and Hiesinger Labs | Upon request | |
| pENTR RFP-Kan11 | Buszczak and Hiesinger Labs | Upon request | |
| Flystocks | Bloomington stock center | Stock numbers: 25680, 25679 | y1 w*/Dp(2;Y)G, P{hs-hid}Y; P{70FLP}11 P{70I-SceI}2B snaSco/CyO, P{hs-hid}4 y1 w*/Dp(2;Y)G, P{hs-hid}Y; P{70FLP}23 P{70I-SceI}4A/TM3, P{hs-hid}14, Sb1 |
| Electroporation machine | Biorad GenePulser Xcell with PC module | 165-2662 | |
| Cuvettes | Fisher Brand | #FB101 |
参考文献
- Rong, Y. S., Golic, K. G. A targeted gene knockout in Drosophila. Genetics. 157, 1307-1312 (2001).
- Rong, Y. S., Golic, K. G. Gene targeting by homologous recombination in Drosophila. Science. 288, 2013-2018 (2000).
- Gong, W. J., Golic, K. G. Ends-out, or replacement, gene targeting in Drosophila. Proc. Natl. Acad. Sci. U.S.A. 100, 2556-2561 (1073).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36 (2005).
- Copeland, N. G., Jenkins, N. A., Court, D. L. Recombineering: a powerful new tool for mouse functional genomics. Nat. Rev. Genet. 2, 769-779 (2001).
- Venken, K. J., He, Y., Hoskins, R. A., Bellen, H. J. P[acman]: a BAC transgenic platform for targeted insertion of large DNA fragments in D. melanogaster. Science. 314, 1747-1751 (2006).
- Venken, K. J., et al. Versatile P[acman] BAC libraries for transgenesis studies in Drosophila melanogaster. Nat. Methods. 6, 431-434 (2009).
- Groth, A. C., Fish, M., Nusse, R., Calos, M. P. Construction of transgenic Drosophila by using the site-specific integrase from phage phiC31. Genetics. 166, 1775-1782 (2004).
- Bischof, J., Maeda, R. K., Hediger, M., Karch, F., Basler, K. An optimized transgenesis system for Drosophila using germ-line-specific phiC31 integrases. Proceedings of the National Academy of Sciences of the United States of America. 104, 3312-3317 (2007).
- Chan, C. C., et al. Systematic discovery of Rab GTPases with synaptic functions in Drosophila. Current Biology: CB. 21, 1704-1715 (2011).
- Chan, C. C., Scoggin, S., Hiesinger, P. R., Buszczak, M. Combining recombineering and ends-out homologous recombination to systematically characterize Drosophila gene families: Rab GTPases as a case study. Commun. Integr. Biol. 5, 179-183 (2012).
- Wu, N., Matand, K., Kebede, B., Acquaah, G., Williams, S. Enhancing DNA electrotransformation efficiency in Escherichia coli DH10B electrocompetent cells. Electronic Journal of Biotechnology. 13, (2010).
- Wang, S., Zhao, Y., Leiby, M., Zhu, J. A new positive/negative selection scheme for precise BAC recombineering. Mol. Biotechnol. 42, 110-116 (2009).
- Venken, K. J., Bellen, H. J. Transgenesis upgrades for Drosophila melanogaster. Development. 134, 3571-3584 (2007).
- Dietzl, G., et al. A genome-wide transgenic RNAi library for conditional gene inactivation in Drosophila. Nature. 448, 151-156 (2007).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved