Method Article
Recombineering costrutti omologhi ricombinazione
In questo articolo
Riepilogo
Tecniche di ricombinazione omologa notevolmente avanzare Drosophila Genetica, consentendo la creazione di mutazioni molecolarmente precise. La recente adozione del recombineering permette di manipolare grandi pezzi di DNA e di trasformarle in Drosophila 6. I metodi qui presentati combinare queste tecniche per generare rapidamente grandi vettori di ricombinazione omologa.
Abstract
Il continuo sviluppo di tecniche per veloce, manipolazione su larga scala di endogeni loci genici estenderà l'uso di Drosophila melanogaster come organismo modello genetico per la ricerca connessa umano-patologia. Gli ultimi anni hanno visto progressi tecnici come ricombinazione omologa e recombineering. Tuttavia, generando null mutazioni inequivocabili o proteine endogene di tagging rimane un notevole sforzo per la maggior parte dei geni. Qui, descriviamo e dimostrare le tecniche per l'utilizzo di metodi di clonazione recombineering-based per generare vettori che possono essere utilizzati per indirizzare e manipolare loci endogeni in vivo In particolare, abbiamo stabilito una combinazione di tre tecnologie:. (1) BAC transgenesi / recombineering, ( 2) le estremità-out ricombinazione omologa e (3) la tecnologia Gateway per fornire un robusto metodo, efficiente e flessibile per manipolare loci genomici endogeni. In questo protocollo, noi forniamo i dettagli passo-passo su come (1) disegno individuavettori l, (2) come clonare grossi frammenti di DNA genomico nel vettore di ricombinazione omologa utilizzando riparazione gap, e (3) come sostituire o tag geni di interesse all'interno di questi vettori che utilizzano un secondo giro di recombineering. Infine, ci sarà anche fornire un protocollo per il modo di mobilitare queste cassette in vivo per generare un ko, o di un gene con tag via knock-in. Possono essere facilmente adottate Questi metodi per obiettivi multipli in parallelo e forniscono un mezzo per manipolare il genoma di Drosophila in modo tempestivo ed efficiente.
Introduzione
Pulire manipolazioni molecolarmente definiti di singoli geni a loro loci endogene offrono uno strumento prezioso per lo studio di una miriade di domande pertinenti alla biologia degli eucarioti. Drosophila invertire tecniche genetiche per la generazione di alleli perdita-di-funzione avevano dimostrato di essere una sfida fino Golic e colleghi hanno introdotto in genica in vivo il targeting utilizzando la ricombinazione omologa di Drosophila 1-3. Hanno dimostrato che specifici loci genomici possono essere mirate utilizzando un frammento di DNA lineare da un costrutto transgenico integrato. Questo DNA lineare "donatore" viene generato in vivo mediante ricombinazione FRT-mediata (di asportare il DNA dal cromosoma come molecola circolare) seguita da linearizzazione con il meganuclease I-SCEI. Sebbene questa metodologia è stata utilizzata con successo per generare una varietà di lesioni definite, la tecnica non è facilmente scalabile per la manipolazione di numerosi geni in parallelo, poiché ciascun indivicostrutto doppio ko richiede design distinto e personalizzato. Per esempio, la difficoltà di manipolare grandi senza soluzione di frammenti di DNA (> 5 kb) in vitro usando enzimi di restrizione classica / legatura clonazione o PCR, nonché le limitazioni di dimensione dei tradizionali in vettori di trasformazione in vivo spesso interferiscono con la rapida realizzazione di ricombinazione omologa di targeting vettori. Per superare queste limitazioni, abbiamo unito il sistema P [acman] recombineering / transgenesi, che consente al sub-clonazione e transgenesi fino a 100 kb di DNA, con l'estremità-out gene targeting metodologia per stabilire una piattaforma efficiente e relativamente rapido che facilita gene Drosophila targeting.
Ricombinazione mediata da ingegneria genetica (recombineering) è un potente ricombinazione omologa a base di tecnologia di clonazione 4,5. In contrasto con l'enzima di restrizione convenzionale / ligasi clonazione, recombineering non è limitata dalla sequenza o size del DNA manipolato. Recombineering utilizza uno speciale E. coli ceppo che ospita macchinari ricombinazione fornito da un λ difettoso profago 4. Questa tecnica è stata recentemente adottata per l'utilizzo in Drosophila 6,7. Recombineering in Drosophila si basa su un condizionale amplificabile cromosoma artificiale batterico modificato (BAC) vettore chiamato P [acman] 6,7. Questo vettore porta due origini di replicazione: oppureiv, che produce numero elevato di copia dopo induzione chimica per la purificazione di grandi quantità di DNA necessarie per il sequenziamento e l'iniezione embrione e ORIS, che mantiene basso numero di copie in condizioni basali. Ulteriormente, il P [acman] vettore è dotato di un sito di attacco batterico (attB). Il sito attB serve come substrato per ΦC31 integrasi transgenesi mediata che permette l'incorporazione di grandi frammenti di DNA in un sito di atterraggio predeterminata all'interno del genoma di Drosophila 8,9.
Abbiamo generato un P [acman] vettore (denominato P [acman]-KO 1.0) che può essere utilizzato come vettore di targeting per fini-out ricombinazione omologa 10,11. Per incorporare le estremità-out dei geni della tecnologia nel sistema, abbiamo aggiunto due FRT e due siti di I-SCEI. Abbiamo incluso anche una cassetta Gateway all'interno di questo vettore modificata per semplificare il processo di integrazione tra le braccia di omologia in P [acman]-KO 1.0. Questo fornisce un modo rapido e semplice per introdurre virtualmente qualsiasi regione genomica di interesse nel vettore targeting. In questo protocollo si descrivono Come Engineer un vettore targeting utilizzando P [acman]-KO 1.0, e come mobilitare questo vettore in vivo di indirizzare il locus endogeno. Ai fini del presente protocollo useremo la cassetta RFP / Kan per sostituire un gene di interesse, ma una serie di cassette che contengono un marcatore di selezione antibiotico può essere utilizzato con questo protocollo. Abbiamo progettato e utilizzato con successo una serie di cassette persostituzione del gene e tagging 10,11.
Protocollo
1. Selezione di BAC e Regione a Target
- Per acquisire un tasso alcolemico con il gene di interesse (GOI) (qui CG32095 è usato come esempio), la ricerca di CG32095 a www.flybase.org . Sotto i titoli di sezione e dei reagenti, controllare la sezione intitolata "cloni genomici" per BAC contenenti CG32095. Assicurarsi che il tasso alcolemico comprende almeno 10 kb a monte e 5 kb a valle del gene di interesse. In alternativa, i cloni della P [acman] vettore 7 si possono trovare anche presso http://www.pacmanfly.org/libraries.html .
- La cloni BAC vengono in cellule DH10B e le p [acman] cloni vengono in EPI300 cellule. I cloni genomici hanno bisogno di essere trasformati in SW102 cellule, che sono suscettibili di recombineering. "Minipreps sporchi" vengono eseguite (a Qiagen miniprep senza l'utilizzo di una colonna e precipitare il DNA con isopropanolo) a partireda un ml di O / N cultura 5 inoculato con cellule contenenti il BAC. Dopo la risospensione del DNA appena fatto pellet in 20 ml di dH 2 O, diluire 1 ml in 100 ml di dH 2 O e usare 1 ml per l'elettroporazione di SW102 electrocompetent. Per rendere SW102 electrocompetent utilizzano il seguente protocollo:
- Seminare SW102 cellule in 4 ml di LB. Crescere la cultura a 32 ° CO / N agitazione a 250 rpm. Importante: le cellule SW102 non devono MAI essere esposti ad una temperatura superiore a 32 ° C a meno che non si desideri l'induzione di macchinari ricombinazione.
- Seminare 1 ml della cultura satura SW102 in 44 ml di LB e crescere su un 32 ° C shaker di OD 600 tra 0,2-0,3 (di solito circa 1-1,5 ore). NOTA: Altri protocolli recombineering consigliano crescere la cultura in un volume più piccolo di OD 600 0,6-0,7, tuttavia crescente i batteri per una densità minore in un volume più grande aumenta significativamente trasformazione efficieNCY 12. Inoltre, tale modifica riduce il tempo di attesa.
- Raffreddare la cultura in slurry acqua e ghiaccio per 5 min. Importante: Durante la procedura seguente cellule devono essere mantenuti a basse temperature. Questo è fondamentale per l'ottenimento di cellule electrocompetent buona qualità.
- Centrifugare cultura a 4 ° C a 1500 xg per 10 min. Scartare il surnatante e lavare pellet in 25 ml di freddo (4 ° C) 10% glicerolo 13. In primo luogo, risospendere le cellule in 5 ml toccando e vorticoso il tubo in sospensione di acqua e ghiaccio (Non pipettare su e giù) e poi aggiungere il restante 10% di glicerolo (20 ml).
- Centrifugare a 4 ° C a 1500 xg per 10 min, scartare il surnatante e lavare il pellet una seconda volta con 25 ml di ghiaccio freddo 10% glicerolo.
- Centrifugare a 4 ° C a 1500 xg per 10 min, scartare il surnatante e lavare il pellet una terza volta con 25 ml di ghiaccio freddo 10% glicerolo.
- Centrifugare a 4 ° C a 1500 xg per 10 minuti e scartare supernatant. Il pellet cellulare può essere molto sciolto, a questo punto e la cura dovrebbe essere presa quando scartare il surnatante. Risospendere il pellet in 1 ml di freddo 10% glicerolo. Trasferire le cellule in una provetta da 1,5 ml e centrifugare per 30 sec a 12.000 xg in una microcentrifuga da tavolo a 4 ° C. Scartare la maggior parte del surnatante lasciando ≈ 90 microlitri di glicerolo / cellule. Le cellule sono ora pronti per essere elettroporate. NOTA: A questo punto le cellule possono essere conservati a -80 ° C per un uso successivo. Tuttavia, questo può diminuire competenza.
- Elettroporazione di DNA in cellule usando una vaschetta da 1 mm a 1.800 V, 25 uF, 200 Ω. Dopo l'elettroporazione, aggiungere 300 ml di SOC e lasciare che le cellule recuperano per 2 ore a 32 ° C (agitazione non è richiesta). Cellule piastra su LB agar oltre 25 mg / ml di cloramfenicolo (o altri antibiotici appropriati a seconda del tasso alcolemico trasformato o PAC) e incubare a 32 ° C per 24-30 ore.
- Omologia armi per gene targeting sono stati progettati da selezionarezione 10 kb di DNA genomico a monte e 5 kb a valle rispetto al GOI. Selezionare 500 bp alla fine del 10 kb e 5 kb braccia di omologia per amplificare (cfr. sezione 2) (Figura 1). Questi 500 frammenti bp servono come le braccia di omologia utilizzati per ricombinare la regione genomica che circonda il governo indiano in P [acman] KO 1.0. Questi frammenti sono indicati come braccio sinistro (LA) per la regione corrispondente al monte 5 'omologia braccio e il braccio destro (RA) per la regione corrispondente alla valle 3' braccio omologia Importante:. Bracci omologia non dovrebbero contenere BamHI siti di restrizione, come la linearizzazione del plasmide (nel passo 3.2) avviene tramite una digestione di restrizione BamHI.
2. Inserire l'omologia Arms in P [acman]-KO 1.0
- Impostare due singoli reazioni PCR, uno per amplificare il Los Angeles e un altro per amplificare la RA. Per il LA, utilizzare primer attB1-I-SCEI-LA-F e BamHI-LA-R e usare il primer BamHI-RA-F e attB2-I-SCEI-RA-Rper amplificare il RA (Tabella 1). Usare 1 ml di BAC DNA dal punto 1.2 o 0.5 ml di coltura batterica bollito durante la notte contenente BAC di interesse come modello. Assicurarsi di utilizzare una DNA polimerasi con correzione di bozze capacità. Questo vale per tutte le successive PCR-con l'eccezione del controllo PCR in passi 2.8 e 3.13. NOTA: il tempo di estensione di una DNA polimerasi correzione può essere diverso dal DNA polimerasi Taq regolare a seconda del produttore. PfuUltra II Fusion HotStart, che polimerizza a una velocità di 15 sec / kb, può essere utilizzato in questi passaggi.
LA / RA PCR
Condizioni di PCR1 microlitri DNA da sporco miniprep dal punto 1.2 del Bac 0,25 ml Ogni 20 microns di primer 2,5 microlitri 10X PfuUltra II Buffer 0,75 ml 10mM (ciascuno) dNTP 0,5 microlitri PfuUltra II Fusion HotStart DNA polimerasi 19.75 ml Acqua 25 pl Totale Volume Step1 95 ° C 2 min per attivare enzima Step2 95 ° C 20 sec denaturare Fase 3 60 ° C 20 sec ricottura Fase 4 72 ° C 20 estensione sec Fase 5 Passare al punto 2 e ripetere 29 cicli Step6 4 ° C Tenere - Analizzare i campioni su un gel di agarosio all'1% ed estrarre i prodotti utilizzando un kit di ripristino del DNA Zymoclean. Eluire il DNA dalla colonna Zymoclean con 10 ml di acqua sterile.
- Eseguire la giunzione per sovrapposizione estensione (PCR SOE) con i due frammenti usando 10-30 ng di DNA totale da ciascun prodotto amplificato purificato a 2.2 (Figura 2). Questo è di solito circa 1/20 di ciascun prodotto di PCR purificato:
LA / RA PCR-Soe
Condizioni di PCR0,5 microlitri ciascuno LA e RA PCR purificato prodotto circa 10-30 ng 0,25 ml ciascuno 20 microns di primer 2,5 microlitri 10X PfuUltra II Buffer 0,75 ml 10mM (ciascuno) dNTP 0,5 microlitri PfuUltra II Fusion HotStart DNA polimerasi 19.25 ml Acqua 25 pl Totale Volume Step1 95 ° C 2 min per attivare enzima Step2 95 ° C 20 sec denaturare Fase 3 55 ° C 20 sec ricottura (temp. Per consentire le braccia per temprare) Fase 4 72 ° C 20 estensione sec Fase 5 Passare al punto 2 e ripetere 1 ciclo Step6 95 ° C 20 sec denaturare Step7 60 ° C 20 sec ricottura Step8 72 ° C 20 estensione sec Step9 Passare al punto 2 e ripetere 27 cicli Step10 4 ° C Tenere - Eseguire l'esempio di PCR su un gel di agarosio e recuperare la banda di 1,0 kb per estrazione gel. Questa è la cassetta attB1-LA-BamHI-RA-attB2.
- Utilizzare Gateway BP kit enzimatico ClonaseII impostare una reazione BP seguendo il protocollo fornito dal produttore.La cassetta generata in 2,4 agirà come il DNA del donatore e il P [acman]-KO 1.0 come il vettore di destinazione.
- Diluire 50 ml di TransforMax EPI300 cellule electrocompetent con l'aggiunta di 30 ml di freddo 10% glicerolo o freddo dH 2 O. Trasformare 1 microlitri della reazione BP in cellule electrocompetent seguendo il protocollo fornito dal produttore. L'aggiunta di acqua diluisce la concentrazione abbastanza elevata sale della reazione BP per evitare scariche elettriche (arco) durante elettroporazione. Elettroporazione di DNA in cellule usando una vaschetta da 1 mm a 1.800 V, 25 uF, 200 Ω.
- Aggiungere 300 ml di SOC e lasciare che le cellule recuperare per 1 ora a 37 ° C (agitazione non è richiesta). Cellule piastra su LB-agar + 50 mg / ml di Amp (P [acman]-KO 1.0 contiene un gene di resistenza Amp). Incubare le piastre a 37 ° C per 18-24 ore.
- PCR controllare le singole colonie da parte convenzionale colonia PCR utilizzando T3 (T3 sequenza è a valle della cassetta Gateway in P [acman]-KO 1.0 (Figure 1) e attB1-I-SCEI-LA-F primer. Cloni positivi mostreranno una band che funziona a 1.2 kb.
- Crescere una cultura durante la notte di un clone positivo in LB con 50 mg / ml ampicillina.
- Amplificare il P [acman]-KO 1.0 vettore contenente il LA e RA in un LB 10 ml con 50 mg / ml ampicillina cultura utilizzando la soluzione di controllo copia seguendo il protocollo del produttore (Epicentro).
- Eseguire un Qiagen miniprep seguendo il protocollo del produttore.
3. Ricombinando la regione genomica di interesse in P [acman]-KO 1.0
Giorno 1
- Seminare SW102 cellule contenenti il BAC di interesse in 4 ml di LB più 25 mcg / ml di cloramfenicolo (Chl), o l'appropriato antibiotico selezione. Crescere la cultura a 32 ° CO / N agitazione a 250 rpm. Importante: Oltre durante l'induzione al passo 3.5, SW102 cellule non dovrebbero essere esposti a una temperatura superiore a 32 ° C, come ad alta temperatura attiva la ricombinazione machine di queste cellule.
Giorno 2
- Digest 0,4 mg di P [acman]-KO-1.0 contenente le braccia LA e RA generati nella sezione 2 con ≈ 20 unità di BamHI in una reazione di 25 microlitri. Incubare a 37 ° C per almeno 3 ore. Importante: Lungo digestione è fondamentale per garantire la digestione di tutto il DNA. Ciò riduce il numero di falsi positivi cloni seguito.
- Inoculare 1,0 ml della coltura SW102/BAC satura in 44 ml di LB-Chl (25 mcg / ml) e crescere su 32 ° C agitatore a OD 600 tra 0,2-0,3 (di solito circa 1-1,5 ore). Includere un campione identico supplementare per il controllo non-HS. Tale campione sarà trattato come quello sperimentale in ogni singolo aspetto con l'eccezione del SA. Questa cultura non-calore-shock serve da controllo negativo. La comparsa di molte colonie sulla piastra di controllo non-HS (passo 3.13) di solito indica trasformazione con digerito P [acman] KO 1.0 LA/ RA plasmide.
- Mentre la cultura è in crescita, eseguire il BamHI-restricted P [acman]-KO su un gel. Poi gel-estrarre ed eluire il DNA in 10 ml di acqua riscaldata fino a 55 ° C. NOTA: non eluire il DNA in ogni buffer. Sali presenti nel buffer possono causare scariche elettriche durante l'elettroporazione.
- Heat-shock la coltura a 42 ° C per esattamente 15 minuti nel bagnomaria. Questo attiverà la macchina recombineering di SW102 cellule. Non riscaldare scosse la cultura del controllo.
- Subito dopo shock termico, trasferimento di calore-scioccato e colture di controllo non-SA, refrigerati provette da 50 ml canoniche e lasciare che il freddo le culture in un impasto di acqua e ghiaccio per 5 min. Importante: Durante la procedura seguente cellule devono essere mantenuti a basse temperature . Ciò è fondamentale per ottenere cellule electrocompetent di buona qualità.
- Centrifugare cultura a 1.500 xg per 10 min. Scartare il surnatante e lavare pellet in 25 ml di freddo (4 ° C) 10% glicerolo 13. Prima,risospendere le cellule in 5 ml toccando e agitando il tubo in sospensione di acqua e ghiaccio (Non pipettare su e giù) e poi aggiungere il restante 10% di glicerolo (20 ml).
- Centrifugare a 4 ° C a 1500 xg per 10 min, scartare il surnatante e lavare il pellet con 25 ml di ghiaccio freddo 10% glicerolo una seconda volta.
- Centrifugare a 4 ° C a 1500 xg per 10 min, scartare il surnatante e lavare il pellet con 25 ml di ghiaccio freddo 10% glicerolo una terza volta.
- Centrifugare a 4 ° C a 1500 xg per 10 minuti e scartare il surnatante. Il pellet cellulare può essere molto sciolto, a questo punto e la cura dovrebbe essere presa quando scartare il surnatante. Risospendere il pellet in 1 ml di freddo 10% glicerolo. Trasferire le cellule in una provetta da 1,5 ml e centrifugare per 30 sec a 12.000 xg in una microcentrifuga da tavolo a 4 ° C. Scartare la maggior parte del surnatante lasciando ≈ 90 microlitri di acqua / celle. NOTA: A questo punto le cellule possono essere conservati a -80 ° C per un uso successivo. Tuttavia, questo può diminuire competenza.
- Aggiungere 2 microlitri della BamHI-digerito P [acman] (scopo per il 50 ng) alle cellule e mescolare delicatamente con una pipetta. Trasferire le cellule in un 1 millimetro elettroporazione cuvetta ed elettroporazione a 1.800 V, 25 uF, 200 Ω.
- Aggiungere 300 ml di SOC e lasciare che le cellule recuperano per 2 ore a 32 ° C (agitazione non è richiesta). Cellule piastra su LB agar più 50 mg / ml ampicillina e incubare a 32 ° C per 24-30 ore.
Giorno 3
- Screen per una corretta gap-riparazione utilizzando colonia PCR. Utilizzare T3 e RA controllare primer per la RA e T7 e LA innesco di controllo per il LA (Tabella 1). Progettare il PCR-Check-RA e PCR-Check-LA primer 100-200 bp nei confronti della regione mirata (Tabella 1). Colonie positive da reazioni di PCR utilizzando PCR-Check-RA con T3 o PCR-Check-LA con T7 mostrerà una band che funziona a 800-1000 bp (Figura 3). NOTA: E 'fondamentale per testare entrambe le braccia da incorporazione di uno braccioma non l'altro si osserva talvolta.
- Crescere PCR verificati colonie a 32 ° C in 4 ml di LB 50 mcg / ml ampicillina per il pernottamento. Fare un brodo congelato di queste cellule per un uso successivo.
4. Sostituzione della regione genomica con la cassetta Targeting
Giorno 4
- Amplifica la cassetta ko RFP / KAN mediante PCR utilizzando pENTR-RFP/KAN (predigerito con AscI e NheI per linearizzare) come modello e la 5'HA-RFP / Kan-F e 3'HA-RFP / Kan-R primer ( Tabella 1). RFP / KAN cassetta può essere richiesto di riferimento 11.
- Gel purificare il prodotto di PCR eluizione in 20 ml di acqua sterile. La cassetta RFP-KAN più le braccia di omologia dovrebbero funzionare circa 3 kb.
- Seminare 1 ml di satura SW102 / P [acman] cultura-KO (passo 3.13) in 44 ml di LB-Amp (50 mg / ml) e crescere su 32 ° C shaker di OD 600 tra 0,2-0,3. Includere un campione identico supplementare per il controllo non-HS. Tale campione sarà trattato solocome quello sperimentale in ogni singolo aspetto con l'eccezione del SA.
- Dopo il [acman] cultura-KO SW102 / P raggiunge la densità desiderata, seguire i passaggi 3,5-3,10.
- Elettroporazione di circa 100 ng di mira cassetta in 90 microlitri di SW102 / P [acman] cellule-KO electrocompetent. Trasferire le cellule in un 1 millimetro elettroporazione cuvetta ed elettroporazione a 1.800 V, 25 uF, 200 Ω. Aggiungere 300 ml di SOC e consentono alle cellule di recuperare per 2 ore a 32 ° C (agitazione non è richiesta). NOTA: Si raccomanda vivamente di utilizzare un gel fresco purificata cassetta.
- Cellule piastra di agar LB + Amp (50 mcg / ml) + Kan (50 mcg / ml) e incubare per 24-30 ore a 32 ° C.
Giorno 5
- Crescere 5 ml di coltura da 5 diverse colonie a 32 ° CO / N.
Giorno 6
- Eseguire una "sporca miniprep" ed eseguire la metà del DNA ottenuto su un gel di agarosio all'1% per cercare il presence di un basso peso molecolare plasmide. Non ci dovrebbe essere alcun plasmide che corre a soffietto 12 kb. (Figura 4)
- Fare una diluizione 1:500 di DNA da un clone positivo e usare 1 ml per l'elettroporazione in 50 ml di TransforMax EPI300. Importante: Assicurarsi di diluire il DNA per impedire l'elettroporazione di più plasmidi in una cella. Cellule piastra di agar LB + Amp (50 mcg / ml) + Kan (50 mcg / ml) e incubare per 18-24 ore a 37 ° C
7 ° giorno
- Inoculare una singola colonia in 10 ml di LB + e crescere O / N.
Giorno 8
- Seed 100 ml di LB con la cultura dei media satura dal punto 4.10. Aggiungi antibiotici appropriati, più 100 ml di soluzione 1,000 X esemplare di controllo e incubare a 37 ° C per 5-6 ore.
- Eseguire un maxiprep e verificare l'inserimento della cassetta nel sito corretto per sequenziamento.
- In aggiunta a sequenziamento, eseguire un enzym restrizioneTest e digestione con DNA da un clone che non conteneva alcun basso peso molecolare e il suo DNA plasmidico parentale. Gli enzimi di restrizione selezionati sarà diverso per ogni gene, ma l'obiettivo è quello di trovare un gruppo di enzimi che permettono al ricercatore di individuare il vettore recombineered. A titolo di esempio, il vettore di targeting CG32095 stato serialmente digerito con Paci AscI, BamHI e AatII. L'aspetto di tutte le bande predetti ei assenza di qualunque banda errate conferma che il vettore targeting è stato recombineered correttamente (Figura 5).
5. Iniezione di mosche e mobilitando Cassette In Vivo
- Iniettare la cassetta con ΦC31 integrasi, in un sito di atterraggio predefinito di scelta. Fornitori esterni, come Arcobaleno transgenici ( http://www.rainbowgene.com ), possono essere utilizzati per i servizi di iniezione. Importante: Il DNA deve essere appena prepared prima dell'iniezione. Maggiore efficienza di trasformazione si osserva quando il DNA viene preparato in loco e iniettata lo stesso giorno.
- Per mobilitare la cassetta dal sito di atterraggio, utilizzare Bloomington magazzino numero 25680 o 25679. Queste scorte contengono flippase e I-SCEI a valle di un promotore shock termico, sul cromosoma due e tre, rispettivamente. Inoltre, essi contengono un hs-HID transgene sul cromosoma bilanciatore e sul cromosoma Y. Raccogliere i maschi del magazzino scelto e attraversare per femmine vergini che trasportano la cassetta KO nel sito di atterraggio. . Set ~ 30 croci e permettono femmine a deporre le uova per 2-3 giorni NOTA: Si raccomanda di scegliere l'azione che si trova su un cromosoma diverso dal luogo di destinazione.
- Capovolgere genitori in un nuovo insieme di 30 flaconcini e larve shock termico a 37 ° C per 1 ora, due volte al giorno, per tre giorni consecutivi. Gli shock termici attivano la produzione degli enzimi, flippase e I-SCEI, e il gene morte cellulare nascosti , Uccidendo i maschi e le mosche trasportano cromosomi bilanciatori. Lasciare i genitori nei flaconcini appena capovolto per deporre le uova per 2-3 giorni, quindi ripetere i colpi di calore per i flaconi, dopo lanciando i genitori in nuovi flaconi di nuovo. Ripetere questa operazione per diversi lanci, fino a quando i genitori non depongono più uova.
- Raccogliere femmine vergini della progenie F1 delle mosche calore scioccato, e attraversare a y - w - i maschi. Obiettivo per 150-200 croci, tre vergini e tre maschi per ogni croce.
- Schermare la progenie F2 in un ambito fluorescente che permette la rilevazione di RFP nell'occhio. Screen per RFP occhi positivi che sono mutanti per i geni bianco e giallo (y - w -). Questo processo di screening seleziona positivamente per la presenza della cassetta RFP e seleziona per l'assenza del [acman] vettoriale (mini-bianco) e atterraggio sito P, contrassegnato dal gene giallo di tipo selvatico. NOTA: È meno tempo -consuming a schermo per RFP negli occhi, che a prima schermata per yw mosche. Gli eventi di mobilitazione sono molto efficienti durante le scosse di calore, con conseguente molte meno mosche contenenti RFP negli occhi, rispetto a y - w - mosche.
- Impostare singoli accoppiamenti coppia con ogni y - w - RFP + fly, dal momento che ogni evento mobilitazione potrebbe essere diverso. Mappare la RFP per il corretto cromosoma. Il targeting corretto cromosoma è tipicamente osservata in> 95% dei casi.
- Stabilire le scorte e controllare gli eventi rivolti corrette utilizzando tecniche standard di analisi Southern Blot. Progettare un 2 kb sonda a monte (braccio sinistro) ed a valle (braccio destro) del gene nel mirino, e 2 kb (o meno se necessario) che abbracciano l'ORF cancellato. I primi due sonde produrrà una band nei fori omozigoti ed eterozigoti, che non è presente nel tipo selvatico, mentre la ORF non produrrà una banda nella knockout omozigote. Un rapido PCR fo la ORF è anche possibile, tuttavia questo si basa sulla mancanza di una banda.
Risultati
Amplificazione della LA e RA braccia di omologia dovrebbe produrre 500 prodotti bp e la reazione di PCR-SOE dovrebbe produrre un 1,0 kb prodotto (Sezioni 2.1-2.4; Figura 2). La reazione BP eseguita nella sezione 2.5 è in genere di trasformazione molto efficiente e batteriche delle rese di prodotto 5-100 colonie in media. Quasi tutte le colonie testati con PCR di controllo Mostra l'atteso prodotto di PCR.
Durante il primo giro di recombineering (sezione 3), si aspettano di ottenere colonie di 20-40 dopo la trasformazione del P digerito [acman]-KO-1.0 contenente il LA e RA braccia nelle cellule SW102. 40-60% di questi cloni porterà il prodotto desiderato ricombinazione. Il controllo di scossa non-calore dovrebbe contenere molto pochi eventuali colonie in confronto alle cellule calore scioccato. L'aspetto del medesimo numero di colonie su entrambi campione e piastre di comando normalmente indica la presenza di uncut plasmide o qualche altro contaminante. In questo caso, l'esperimento should essere scartato e ripetuta, questa volta digerire il P [acman]-KO-1.0 portando il LA e RA braccia al completamento seguendo le istruzioni descritte al punto 3.2. Un esempio di un assegno PCR per il recupero di regione genomica di interesse in P [acman]-KO 1.0 in cui sono ottenuti ≈ 60 colonie è mostrato in Figura 3. Qui il 60% delle colonie testato effettuata la product.Care ricombinazione desiderata occorre fare attenzione che i prodotti di PCR osservati corrono alla dimensione prevista, dal momento che le bande di PCR aberranti di solito indicano eventi recombineering errati.
Di tanto in tanto no colonie cresceranno dopo il primo turno di recombineering. Usando cellule con efficienza subottimale rappresenta la causa più comune per una reazione recombineering fallito. Mantenere le celle frigorifere, in ogni momento durante la loro preparazione e la manipolazione delicata durante i lavaggi (MAI vortice) è fondamentale per l'ottenimento di cellule competenti alta qualità. Cercate di usare sempre fresco digerito / purificata P [acman]-KO-1.0 DNA per avere la massima efficienza.
A volte il primo turno dei rendimenti recombineering colonie ma quelle colonie non passano il test di controllo PCR. Questo di solito indica prodotti recombineering errati ma un risultato del genere può anche indicare potenziali problemi con uno dei set di primer. Per convalidare il primer PCR-Check-LA, impostare una reazione PCR in combinazione con il attB1-I-SCEI-LA-F. Per verificare PCR-check-RA istituito una reazione di PCR con l'attB2-I-SCEI-RA-R. In entrambi i casi utilizzare il BAC originale come modello. Queste reazioni di PCR dovrebbero produrre prodotti della dimensione prevista, che varieranno a seconda della sequenza dei primer di controllo. I risultati positivi di questo esperimento di controllo si esclude la possibilità che l'incapacità di rilevare i cloni positivi è dovuta al fondo inefficienza.
Se avete provato il primo turno di recombineering più volte in seguito il protocollo di cui sopra, senza alcun successo e sono state eseguite tutte lesuggerito controlli, si consiglia di scegliere un nuovo paio di 500 bp braccia di omologia. Per ragioni che non sono chiare al momento attuale, alcune regioni genomiche sono altamente resistenti alla ricombinazione del DNA. Semplicemente la progettazione di nuovi bracci di omologia e spostandoli verso o lontano dal gene di interesse a volte risolve questi problemi.
Il secondo giro di recombineering (sezione 4) è in genere molto più efficiente. Normalmente 30-50 colonie sono osservate dopo la trasformazione, con 90-95% di questi contenente il costrutto desiderato. Il problema più comune in questa fase è ottenere colonie di falsi positivi. Figura 4 mostra esempi di veri e falsi cloni positivi dopo il secondo turno di recombineering (sezione 4).
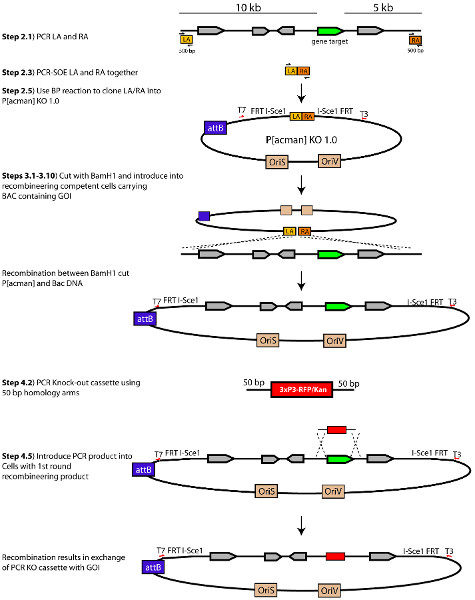
Figura 1. Schema del process per generare un P [acman]-KO 1.0 mira vettore. Modificato da Chan et al. (2012). Clicca qui per ingrandire la figura .

Figura 2. Schema di giunzione per sovrapposizione PCR (PCR SOE) eseguita su passo 2.3. Clicca qui per ingrandire la figura .
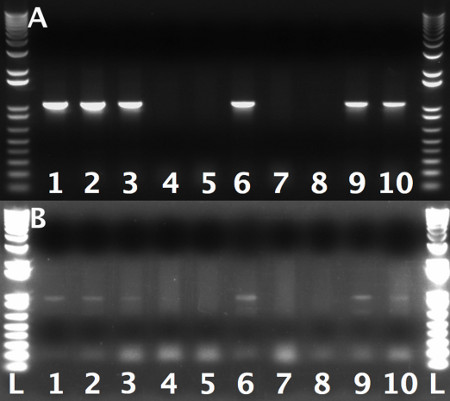
Figura 3. PCR-controllare gel che mostra l'efficienza del primo evento recombineering Ogni corsia rappresenta una singola colonia A:.. Colonie testati per il braccio sinistro. integrazione con T7 e primer LA-check B: Le stesse colonie come A, testati per l'integrazione del braccio destro con T3 e RA-check primers.1kb Inoltre DNA Ladder è stato utilizzato come marcatore di peso molecolare. NOTA: a causa della scarsa adescamento di T3 Primer relativa T7, i complessi del braccio destro controllo erano significativamente più scuro. Ciò deve essere tenuto in considerazione quando si visualizza il gel.

Figura 4. DNA gel di agarosio mostrando elevati plasmidi peso molecolare (P [acman]) e basso peso molecolare plasmide (template digerito) isolato da potenziali cloni positivi dopo secondo giro di recombineering. DNA è stato isolato dai quattro potenziali cloni positivi e corse su un gel di agarosio 1% colorati con bromuro di etidio. Cloni 1,3 e 4 sono dei veri cloni positivi. Clone 2 è un falso positivo, come rilevato dai tutte le forme di ap lasmid che corre a soffietto 12 kb (la banda di dimensione più grande della DNA ladder corsia). 1 kb Inoltre DNA Ladder è stato utilizzato come marcatore di peso molecolare.

Figura 5. Prova per evento di ricombinazione desiderato enzima di restrizione digest. Nell'esempio di cui sopra mostra il computer predetto banding modello della P [acman]-KO CG32095 prima e dopo il secondo turno di recombineering. Quattro diversi enzimi sono stati utilizzati: Paci AscI, AatII e BamHI. Correttamente prodotto ricombinato avrà le stesse bande di come il suo parentale P [acman]-KO CG32095 fatta eccezione per la modifica prevista (punte di freccia). NEBcutter V2.0 è stato utilizzato per predire il pattern band.
0346/50346fig6highres.jpg "src =" / files/ftp_upload/50346/50346fig6.jpg "/>
Figura 6. Schema che descrive la mobilitazione di una cassetta di targeting in vivo. Clicca qui per ingrandire la figura .

Tabella 1. Primer. Clicca qui per vedere tabella più grande .
Discussione
Il potere di organismi modello genetiche nella ricerca biomedica è largamente basato sugli strumenti disponibili per la manipolazione genetica. I piccoli modelli C. elegans e Drosophila consentire, in particolare economico e veloce molecolare le analisi genetiche dei percorsi completi e famiglie di geni implicati nello sviluppo o nella funzione multicellulare. Gli ultimi anni hanno visto progressi significativi nello sviluppo strumento per manipolare i geni in Drosophila 14,15. Per esempio, recombineering, che è ampiamente usato in genetica del mouse per manipolare Bac costrutti di DNA, è stato recentemente adattato per Drosophila, attraverso l'uso del P [acman] vettoriale e ΦC31 transgenesi mediata 6. Diverse cassette di selezione consentono l'inserimento o l'eliminazione di sequenze specifiche in qualsiasi punto all'interno di un costrutto utilizzando batteri competenti ricombinazione 4,13. Abbiamo modificato l'originale P [acman] vettore in modo che possa essere utilizzato per la costruzione In vivo omologhi vettori di ricombinazione che colpiscono loci endogeni in Drosophila. Questo nuovo vettore permette di generare rivolte cassette con omologia braccia più grandi di quelli che possono essere facilmente realizzati con metodi di clonazione di taglia e incolla. Inoltre, una cassetta Gateway-clonazione è stata integrata anche in questo vettore, permettendo di introdurre rapidamente e in modo efficiente nuovo DNA genomico nel sistema.
Sebbene la maggior parte della metodologia descritta qui è semplice, esistono diversi passaggi che abbiamo trovato critico per il successo della tecnica. A causa delle dimensioni del DNA manipolato (> 15 kb), il primo giro di reazione recombineering (Sezione 3) pone un significativo ostacolo tecnico. Quindi, questo giro di recombineering richiede un protocollo di ricombinazione molto efficiente. Aggiustamenti di protocollo apparentemente minori, come crescere le cellule a bassa densità, lavare le cellule tre volte invece di due e con il 10% glicerolo anziché DH 2 </ Sub> O 13, hanno notevolmente migliorato l'efficienza di trasformazione delle nostre cellule competenti recombineering. Inoltre, avendo cura di verificare che il LA / AR contenente P [acman] è tagliato a compimento con BamHI (Passo 3.1) elimina molti falsi positivi.
Ricombinazione eventi indesiderati rappresentano un altro problema abbastanza comune durante recombineering. Per esempio, abbiamo osservato casi di duplicazioni e traslocazioni all'interno di vari costrutti. Questi eventi possono portare a molteplici problemi durante la trasformazione Drosophila e in vivo gene targeting. Verifica PCR e analisi di sequenziamento non possono rilevare tutti questi eventi. Quindi, un enzima di restrizione analisi deve essere eseguita sul prodotto finale clonato prima dell'iniezione del DNA in embrioni. L'ultimo controllo è dimostrato critico e ha permesso di evitare l'iniezione di costrutti che contengono aberrazioni indesiderate.
Maxi-durante la preparazione del vettore targeting poco prima injezione e mai congelamento del DNA migliora notevolmente l'efficienza di trasformazione di questi grandi costrutti in Drosophila. In breve, i piccoli dettagli che fanno una grande differenza nel successo di queste tecniche. I protocolli descritti e dimostrati qui rappresentano intuizioni da un certo numero di fonti diverse e la nostra esperienza.
Mentre ci auguriamo che il protocollo presentato qui aiuta gli altri ad adottare metodi recombineering nei propri laboratori, crediamo che ulteriori miglioramenti e perfezionamenti della metodologia sono ancora possibili. Di tanto in tanto, specifici frammenti genomici sono difficili da manipolare utilizzando recombineering per ragioni che non sono del tutto evidenti. Inoltre, nonostante i nostri sforzi per eliminare fondo indesiderati, troviamo ancora che certi pezzi di DNA genomico tendono a produrre prodotti finali in modo non corretto recombineered. Una più profonda comprensione del processo recombineering può produrre protocolli più ottimali in futuro e aperta discussione direcombineering fallimenti e storie di successo favorirà il successo nell'uso di queste potenti tecniche da parte della comunità di ricerca più ampio.
Divulgazioni
Gli autori non hanno alcun conflitto di interessi in quanto riguarda le tecniche descritte qui.
Riconoscimenti
Vorremmo ringraziare Hugo Bellen e la Bloomington Stock Center per i reagenti. Ringraziamo inoltre Koen Venken, Hugo Bellen e tutti i membri del laboratorio Buszczak e Hiesinger per le discussioni utili. Questo lavoro è stato sostenuto da finanziamenti del National Institute of Health di ACR (T32GM083831), PRH (RO1EY018884) e MB (RO1GM086647), una sovvenzione dal Cancer Prevention Research Institute del Texas a MB e PRH (RP100516), e la Welch Fondazione (I-1657) a PRH. MB è un EE e Greer Garson Fogelson Scholar in Ricerca Biomedica e PRH è una Eugene McDermott Scholar in Ricerca Biomedica presso UT Southwestern Medical Center.
Materiali
| Name | Company | Catalog Number | Comments |
| Name of the reagent | Company | Catalogue number | Comments (optional) |
| SW102 Recombination competent bacteria | NCI-Frederick | Recombination Bacteria (SW102, SW105 and SW106) | http://ncifrederick.cancer.gov/research/brb/logon.aspx |
| TransforMax EPI300 electrocopmpetent E. coli | Epicentre | EC300110 | Includes CopyControl induction solution |
| PfuUltra II Fusion HS DNA Polymerase | Aligent Technology Inc. | 600670 | |
| BamHI-HF | New England Biolabs | R3136S | |
| Zymoclean Gel DNA Recovery Kit | Zymo Research | D4001 | |
| Use Gateway BP ClonaseII Enzyme kit | Invitrogen | 11789-020 | |
| P[acman]KO1.010 | Buszczak and Hiesinger Labs | Upon request | |
| pENTR RFP-Kan11 | Buszczak and Hiesinger Labs | Upon request | |
| Flystocks | Bloomington stock center | Stock numbers: 25680, 25679 | y1 w*/Dp(2;Y)G, P{hs-hid}Y; P{70FLP}11 P{70I-SceI}2B snaSco/CyO, P{hs-hid}4 y1 w*/Dp(2;Y)G, P{hs-hid}Y; P{70FLP}23 P{70I-SceI}4A/TM3, P{hs-hid}14, Sb1 |
| Electroporation machine | Biorad GenePulser Xcell with PC module | 165-2662 | |
| Cuvettes | Fisher Brand | #FB101 |
Riferimenti
- Rong, Y. S., Golic, K. G. A targeted gene knockout in Drosophila. Genetics. 157, 1307-1312 (2001).
- Rong, Y. S., Golic, K. G. Gene targeting by homologous recombination in Drosophila. Science. 288, 2013-2018 (2000).
- Gong, W. J., Golic, K. G. Ends-out, or replacement, gene targeting in Drosophila. Proc. Natl. Acad. Sci. U.S.A. 100, 2556-2561 (1073).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36 (2005).
- Copeland, N. G., Jenkins, N. A., Court, D. L. Recombineering: a powerful new tool for mouse functional genomics. Nat. Rev. Genet. 2, 769-779 (2001).
- Venken, K. J., He, Y., Hoskins, R. A., Bellen, H. J. P[acman]: a BAC transgenic platform for targeted insertion of large DNA fragments in D. melanogaster. Science. 314, 1747-1751 (2006).
- Venken, K. J., et al. Versatile P[acman] BAC libraries for transgenesis studies in Drosophila melanogaster. Nat. Methods. 6, 431-434 (2009).
- Groth, A. C., Fish, M., Nusse, R., Calos, M. P. Construction of transgenic Drosophila by using the site-specific integrase from phage phiC31. Genetics. 166, 1775-1782 (2004).
- Bischof, J., Maeda, R. K., Hediger, M., Karch, F., Basler, K. An optimized transgenesis system for Drosophila using germ-line-specific phiC31 integrases. Proceedings of the National Academy of Sciences of the United States of America. 104, 3312-3317 (2007).
- Chan, C. C., et al. Systematic discovery of Rab GTPases with synaptic functions in Drosophila. Current Biology: CB. 21, 1704-1715 (2011).
- Chan, C. C., Scoggin, S., Hiesinger, P. R., Buszczak, M. Combining recombineering and ends-out homologous recombination to systematically characterize Drosophila gene families: Rab GTPases as a case study. Commun. Integr. Biol. 5, 179-183 (2012).
- Wu, N., Matand, K., Kebede, B., Acquaah, G., Williams, S. Enhancing DNA electrotransformation efficiency in Escherichia coli DH10B electrocompetent cells. Electronic Journal of Biotechnology. 13, (2010).
- Wang, S., Zhao, Y., Leiby, M., Zhu, J. A new positive/negative selection scheme for precise BAC recombineering. Mol. Biotechnol. 42, 110-116 (2009).
- Venken, K. J., Bellen, H. J. Transgenesis upgrades for Drosophila melanogaster. Development. 134, 3571-3584 (2007).
- Dietzl, G., et al. A genome-wide transgenic RNAi library for conditional gene inactivation in Drosophila. Nature. 448, 151-156 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon