Method Article
Recombinería homólogos Construcciones recombinación en
En este artículo
Resumen
Técnicas de recombinación homóloga en gran medida avanzar Drosophila Genética al permitir la creación de mutaciones molecularmente precisas. La reciente adopción de recombineering permite para manipular grandes trozos de ADN y transformarlos en Drosophila 6. Los métodos presentados aquí combinar estas técnicas para generar rápidamente grandes vectores de recombinación homóloga.
Resumen
El continuo desarrollo de técnicas para la manipulación rápida, a gran escala de genes endógenos loci ampliará el uso de Drosophila melanogaster como organismo modelo genético para la investigación relacionada con la enfermedad humana. Los últimos años han visto avances técnicos como la recombinación homóloga y recombineering. Sin embargo, la generación de mutaciones nulas inequívocos o proteínas endógenos de etiquetado sigue siendo un esfuerzo sustancial para la mayoría de los genes. A continuación, describimos y demostrar las técnicas para el uso de métodos de clonación basados en recombinería para generar vectores que se pueden utilizar para apuntar y para manipular los loci endógenos in vivo Específicamente, hemos establecido una combinación de tres tecnologías:. (1) BAC transgénesis / recombinería, ( 2) extremos-a cabo la recombinación homóloga y (3) la tecnología de puerta de enlace para proporcionar un método robusto, eficaz y flexible para la manipulación de los loci genómicos endógenos. En este protocolo, proporcionamos instrucciones paso a paso sobre la forma de (1) el diseño individuaVectores l, (2) cómo clonar grandes fragmentos de ADN genómico en el vector de recombinación homóloga con reparación brecha, y (3) la forma de reemplazar o etiquetar genes de interés dentro de estos vectores con una segunda ronda de recombineering. Por último, también vamos a proporcionar un protocolo de cómo movilizar estos cassettes in vivo para generar un golpe de gracia, o un gen marcado por knock-in. Estos métodos se pueden adoptar fácilmente para múltiples objetivos en paralelo y proporcionan un medio para manipular el genoma de Drosophila en una manera oportuna y eficiente.
Introducción
Limpie manipulaciones molecularmente definidas de genes individuales en los loci endógenos ofrecen una valiosa herramienta para estudiar una gran variedad de cuestiones relacionadas con la biología eucariótica. Drosophila técnicas de genética inversa para generar alelos de pérdida de función había demostrado ser un reto hasta Golic y sus colegas introdujeron en génica in vivo focalización utilizando recombinación homóloga para Drosophila 1-3. Ellos demostraron que los loci genómicos específicas podrían seleccionarse utilizando un fragmento lineal de ADN a partir de un constructo transgénico integrado. Este ADN "donante" lineal se genera en vivo a través de recombinación FRT-mediada (para escindir el ADN del cromosoma como una molécula circular) seguido por linealización con la meganucleasa I-SCE. Aunque esta metodología se ha utilizado con éxito para generar una variedad de lesiones definidas, la técnica no ha sido fácilmente escalable para la manipulación de numerosos genes en paralelo, ya que cada indiviconstrucción de doble knockout requiere un diseño distinto y personalizado. Por ejemplo, las dificultades en la manipulación sin problemas grandes fragmentos de ADN (> 5 kb) in vitro utilizando enzima clásica restricción / ligadura de la clonación o la PCR, así como las limitaciones de tamaño de tradicional en vectores de transformación in vivo a menudo interfieren con la rápida creación de recombinación homóloga focalización vectores. Para superar estas limitaciones, hemos combinado el sistema recombineering P / transgénesis [ACMAN], que permite la sub-clonación y transgénesis de hasta 100 kb de ADN, con los extremos de salida génico metodología para establecer una plataforma eficiente y relativamente rápida que facilita la orientación del gen de Drosophila.
Recombinación mediada por ingeniería genética (recombinería) es un potente recombinación homóloga basada en la tecnología de clonación 4,5. En contraste con enzima / ligasa clonación restricción convencional, recombinería no está limitado por la secuencia o SIZe de la ADN manipulado. Recombinería utiliza un E. especial cepa de E. coli que alberga maquinaria de recombinación proporcionada por un profago λ defectuoso 4. Esta técnica se ha aprobado recientemente para el uso en Drosophila 6,7. Recombinería en Drosophila se basa en un cromosoma modificado condicionalmente amplifiable artificial bacteriano (BAC) vector llamado P [ACMAN] 6,7. Este vector lleva dos orígenes de replicación: OriV, lo que produce alto número de copias tras la inducción química para la purificación de grandes cantidades de ADN necesarias para la secuenciación y la inyección embrión y oris, que mantiene bajo número de copias bajo condiciones basales. Además, el P [ACMAN] vectorial está equipado con un sitio de unión bacteriano (attB). El sitio attB sirve como un sustrato para C31 transgénesis mediada por integrasa que permite la incorporación de grandes fragmentos de ADN en un sitio de aterrizaje predeterminada dentro del genoma de Drosophila 8,9.
Hemos generado un P [ACMAN] vectorial (referido como P [ACMAN]-KO 1.0) que puede ser usado como vector de abordaje para termina-a cabo la recombinación homóloga 10,11. Incorporar extremos Salida génico tecnología en el sistema, hemos añadido dos sitios FRT y dos I-SCEI. También hemos incluido un casete de puerta de enlace dentro de este vector modificado para agilizar el proceso de incorporación de los brazos de homología en P [ACMAN]-KO 1.0. Esto proporciona una manera rápida y sencilla para introducir prácticamente cualquier región genómica de interés en el vector de abordaje. En este protocolo vamos a describir cómo diseñar un vector de orientación utilizando P [ACMAN]-KO 1.0, y cómo movilizar este vector in vivo para apuntar el locus endógeno. A los efectos del presente Protocolo se utilizará el cartucho RFP / Kan para reemplazar un gen de interés, sino una variedad de cassettes que contienen un marcador de selección de antibióticos se pueden utilizar con este protocolo. Hemos diseñado y utilizado con éxito una serie de casetes desustitución de genes y marcado de 10,11.
Protocolo
1. Selección de BAC y la Región de Objetivo
- Para adquirir un BAC con el gen de interés (GOI) (aquí CG32095 se utiliza como un ejemplo), la búsqueda de CG32095 en www.flybase.org . En virtud de los stocks de las secciones y reactivos, consulte la sección titulada "Los clones genómicos" de BAC que contiene CG32095. Asegúrese de que el BAC incluye al menos 10 kb aguas arriba y 5 kb aguas abajo del gen de interés. Alternativamente, los clones en la P [ACMAN] vectorial 7 también se puede encontrar en http://www.pacmanfly.org/libraries.html .
- Los clones BAC vienen en células DH10B y el P [] ACMAN clones vienen en células EPI300. Los clones genómicos necesitan ser transformado en células SW102, que son susceptibles de recombinería. Se llevan a cabo "minipreparaciones sucios" (una miniprep de Qiagen y sin el uso de una columna y precipitar el ADN con isopropanol) a partirml de un cultivo de 5 S / N inoculados con células que contienen el BAC. Después de la resuspensión del sedimento de ADN recién hecho en 20 l de dH2O, diluir 1 l en 100 l de dH2O y el uso de 1 l para electroporar SW102 electrocompetentes. Para hacer SW102 electrocompetentes utilizan el protocolo siguiente:
- Se inoculan las células SW102 en 4 ml de LB. Grow cultivo a 32 ° CO / N con agitación a 250 rpm Importante:. Células SW102 nunca deben ser expuestas a una temperatura superior a 32 ° C a menos que se desea la inducción de la maquinaria de recombinación.
- Inocular 1 ml del cultivo saturado SW102 en 44 ml de LB y crecer en un agitador de 32 ° C a OD 600 entre 0,2-0,3 (generalmente alrededor de 1-1,5 horas) NOTA:. Otros protocolos recombinería recomiendan crecer el cultivo en un volumen más pequeño a OD 600 0.6-0.7, sin embargo el crecimiento de las bacterias a una densidad más baja en un volumen más grande aumenta significativamente transformación efficiency 12. Además, esta modificación reduce el tiempo de espera.
- Se enfría la cultura en la suspensión de hielo-agua durante 5 min Importante:. Durante los siguientes pasos células deben mantenerse a bajas temperaturas. Esto es crucial para la obtención de células electrocompetentes de buena calidad.
- Centrifugar de cultivo a 4 ° C a 1500 xg durante 10 min. Desechar el sobrenadante y lavar el pellet en 25 ml de frío (4 ° C) 10% de glicerol 13. En primer lugar, volver a suspender las células en 5 ml tocando y girando el tubo de suspensión de hielo-agua (no se puede pipetear arriba y abajo) y luego añadir el 10% de glicerol restante (20 ml).
- Centrifugar a 4 ° C a 1500 xg durante 10 min, desechar el sobrenadante y lavar el sedimento una segunda vez con 25 ml de hielo frío 10% de glicerol.
- Centrifugar a 4 ° C a 1500 xg durante 10 min, desechar el sobrenadante y lavar el sedimento una tercera vez con 25 ml de hielo frío 10% de glicerol.
- Centrifugar a 4 ° C a 1500 xg durante 10 min y descartar supernatant. El sedimento celular puede ser muy floja en este momento y se debe tener cuidado al desechar el sobrenadante. Resuspender el precipitado en 1 ml de glicerol al 10% frío. Transferir las células a un tubo de 1,5 ml y centrifugar durante 30 segundos a 12.000 xg en una microcentrífuga de mesa a 4 º C. Eliminar la mayor parte del sobrenadante dejando ≈ 90 l de glicerol / células. Las células están listas para ser electroporación NOTA:. Llegados a este punto las células pueden ser almacenadas a -80 ° C para su uso posterior. Sin embargo, esto puede reducir la competencia.
- Electroporación de ADN en las células usando una cubeta de 1 mm a 1800 V, 25 mF, 200 Ω. Después de la electroporación, añadir 300 l de SOC y dejar que las células se recuperan durante 2 horas a 32 ° C (sacudidas que no es necesario). Células de la placa de agar LB, más 25 mg / ml de cloranfenicol (u otros antibióticos adecuados en función de la BAC o PAC transformada) y se incuba a 32 ° C durante 24-30 horas.
- Brazos de homología para la orientación de genes han sido diseñados por selecciónción 10 kb de ADN genómico aguas arriba y 5 kb abajo en relación con el Gobierno de la India. Seleccionar 500 pb en el extremo de la 10 kb y 5 kb brazos de homología para amplificar (Ver la Sección 2) (Figura 1). Estos 500 bp fragmentos sirven como los brazos de homología utilizados para recombinar la región genómica que rodea el Gobierno de la India en P [ACMAN] KO 1.0. Estos fragmentos se conocen como brazo izquierdo (LA) para la región que corresponde a la corriente arriba 5 'brazo de homología y el brazo derecho (RA) para la región que corresponde a la corriente abajo 3' brazo de homología Importante:. Brazos de homología no deben contener BamHI sitios de restricción, como la linealización del plásmido (en el paso 3.2) se lleva a cabo a través de una digestión de restricción BamHI.
2. Inserte los brazos de homología en P [ACMAN]-KO 1.0
- La creación de dos reacciones de PCR individuales, uno para amplificar la LA y otro para ampliar la RA. Para la AI, utilice primers att B1-II CPE-LA-F y BamHI-LA-R y utilizar los cebadores BamHI-RA-F y attB2-II CPE-RA-Rpara amplificar la AD (Tabla 1). Usar 1 l de ADN de BAC de la etapa 1.2 o 0,5 l de cultivo bacteriano durante la noche hervida que contiene BAC de interés como una plantilla. Asegúrese de que utiliza una ADN polimerasa de corrección de textos capacidad. Esto se aplica a todos los posteriores PCR-con la excepción de la comprobación de la PCR en los pasos 2,8 y 3,13 NOTA:. Tiempo de extensión para una ADN polimerasa corrección de pruebas puede ser diferente de regular de Taq ADN polimerasa según el fabricante. PfuUltra II Fusión HotStart, que se polimeriza a una velocidad de 15 seg / kb, se puede utilizar en estos pasos.
LA / RA PCR
Condiciones de PCR1 l ADN de miniprep sucia desde el paso 1.2 de Bac 0,25 l cada 20 Primer mM 2,5 l 10X PfuUltra II Buffer 0,75 l 10 mM (cada uno) dNTP 0,5 l PfuUltra II Fusión HotStart ADN polimerasa 19.75 l Agua 25 l Volumen Total Paso 1 95 ° C 2 min para activar la enzima Paso 2 95 ° C 20 seg desnaturalizan Paso 3 60 ° C 20 seg de recocido Paso 4 72 ° C de extensión 20 sec Paso 5 Vaya al paso 2 y repita 29 ciclos Paso 6 4 ° C Sostenga - Ejecutar las muestras en un gel de agarosa al 1% y extraer productos utilizando un kit de recuperación de ADN Zymoclean. Eluir el ADN de la columna Zymoclean con 10 l de agua estéril.
- Realizar empalme por extensión superposición (PCR SOE) con los dos fragmentos utilizando 10-30 ng de ADN total de cada producto amplificado se purificó de 2,2 (Figura 2). Esto es por lo general alrededor de 1/20 ª de cada producto de PCR purificado:
LA / RA PCR-Soe
Condiciones de PCR0,5 l cada uno LA y RA purificado producto de PCR de aproximadamente 10-30 ng 0,25 l cada uno 20 Primer mM 2,5 l 10X PfuUltra II Buffer 0,75 l 10 mM (cada uno) dNTP 0,5 l PfuUltra II Fusión HotStart ADN polimerasa 19.25 l Agua 25 l Volumen Total Paso 1 95 ° C 2 min para activar la enzima Paso 2 95 ° C 20 seg desnaturalizan Paso 3 55 ° C 20 seg recocido (Baja temp. Para permitir brazos para recocer) Paso 4 72 ° C de extensión 20 sec Paso 5 Vaya al paso 2 y repita el ciclo 1 Paso 6 95 ° C 20 seg desnaturalizan Step7 60 ° C 20 seg de recocido Paso 8 72 ° C de extensión 20 sec Paso 9 Vaya al paso 2 y repita 27 ciclos Paso 10 4 ° C Sostenga - Ejecutar la muestra de PCR en un gel de agarosa y recuperar la banda de 1,0 kb por extracción en gel. Este es el cassette att B1-LA-BamHI-RA-attB2.
- Utilice el equipo de puerta de enlace de la enzima BP ClonaseII crear una reacción BP siguiendo el protocolo proporcionado por el fabricante.El casete generada en 2,4 actuará como el ADN del donante y el P [ACMAN]-KO 1.0 como el vector de destino.
- Diluir 50 l de TransforMax EPI300 células electrocompetentes mediante la adición de 30 l de glicerol al 10% frío o caliente dH 2 O. Transformar 1 l de la reacción de BP en células electrocompetentes siguiendo el protocolo proporcionado por el fabricante. La adición de agua diluye la concentración bastante alta de sal de la reacción BP para evitar la descarga eléctrica (arco) durante la electroporación. Electroporación de ADN en las células usando una cubeta de 1 mm a 1800 V, 25 mF, 200 Ω.
- Añadir 300 l de SOC y dejar que las células se recuperan durante 1 hora a 37 ° C (sacudidas que no es necesario). Células de la placa de LB-agar + 50 mg / ml de Amp (P [ACMAN]-KO 1.0 contiene un gen de resistencia Amp). Incubar las placas a 37 ° C durante 18-24 horas.
- PCR comprobar colonias individuales por PCR colonia convencional utilizando la secuencia T3 T3 (está abajo del cassette de puerta de enlace en el P [ACMAN]-KO 1.0 (Fig.ure 1) y att B1-II CPE-LA-F cebadores. Los clones positivos se mostrarán una banda que funciona a 1,2 kb.
- Crecer un cultivo de una noche de un clon positivo en LB con 50 mg / ml de ampicilina.
- Amplificar el [ACMAN]-KO vector de 1,0 P que contiene el LA y RA en un LB 10 ml con 50 mg / ml de cultivo ampicilina usando solución de control de copia siguiendo el protocolo del fabricante (Epicentre).
- Realizar una miniprep de Qiagen siguiendo el protocolo del fabricante.
3. Recombinación de la región genómica de interés en P [ACMAN]-KO 1.0
Día 1
- Se inoculan las células SW102 contiene el BAC de interés en 4 ml de LB más de 25 mg / ml de cloranfenicol (CHL) o el antibiótico de selección adecuado. Grow cultivo a 32 ° CO / N con agitación a 250 rpm Importante:. Además durante la inducción en el paso 3.5, células SW102 no deben ser expuestos a una temperatura superior a 32 ° C, como alta temperatura activa la recombinación maChinery de estas células.
Día 2
- Recopilación de 0,4 g de la P [ACMAN]-KO-1.0 que contiene los brazos de LA y RA generados en la sección 2 con ≈ 20 unidades de BamHI en una reacción de 25 l. Incubar a 37 ° C durante al menos 3 horas Importante:. Digestión larga es crítico para asegurar la digestión de todo el ADN. Esto disminuirá el número de falsos positivos clones más adelante.
- Inocular 1,0 ml del cultivo SW102/BAC saturado en 44 ml de LB-Chl (25 g / ml) y crecer en un agitador de 32 ° C a OD 600 entre 0,2-0,3 (generalmente alrededor de 1-1,5 horas). Incluya una muestra idéntica adicional para el control no-HS. Esta muestra será tratado igual que el experimental en todos los aspectos, con la excepción del SA. Esta cultura no-calor-shock sirve como control negativo. La aparición de muchas colonias en la placa de control no-HS (paso 3,13) por lo general indica la transformación con no digeridos P [ACMAN] KO 1.0 LA/ Plásmido RA.
- Mientras que la cultura es cada vez mayor, ejecute el BamHI-restringido P [ACMAN]-KO en un gel. Luego de gel extraer y eluir el ADN en 10 l de agua calentado a 55 ° C. NOTA: no se produzca la elución de ADN en cualquier tampón. Las sales presentes en la memoria intermedia pueden causar una descarga eléctrica durante la electroporación.
- Heat-shock de la cultura a 42 ° C durante exactamente 15 minutos en el baño de agua. Esto activará el mecanismo recombineering de células SW102. No caliente el choque de la cultura de control.
- Inmediatamente después del golpe de calor, transferencia de control de las culturas no-HS-shock de calor y fríos tubos canónicos de 50 ml y dejar que el frío de las culturas en una suspensión de agua con hielo durante 5 min Importante:. Durante los siguientes pasos células deben mantenerse a bajas temperaturas . Esto es crucial para la obtención de células electrocompetentes de buena calidad.
- Centrifugar cultivo a 1500 xg durante 10 min. Desechar el sobrenadante y lavar el pellet en 25 ml de frío (4 ° C) 10% de glicerol 13. En primer lugar,volver a suspender las células en 5 ml tocando y girando el tubo de suspensión de hielo-agua (no se puede pipetear arriba y abajo) y luego añadir el restante 10% de glicerol (20 ml).
- Centrifugar a 4 ° C a 1500 xg durante 10 min, desechar el sobrenadante y lavar el precipitado con 25 ml de hielo frío 10% de glicerol por segunda vez.
- Centrifugar a 4 ° C a 1500 xg durante 10 min, desechar el sobrenadante y lavar el precipitado con 25 ml de hielo frío 10% de glicerol por tercera vez.
- Centrifugar a 4 ° C a 1500 xg durante 10 minutos y desechar el sobrenadante. El sedimento celular puede ser muy floja en este momento y se debe tener cuidado al desechar el sobrenadante. Resuspender el precipitado en 1 ml de glicerol al 10% frío. Transferir las células a un tubo de 1,5 ml y centrifugar durante 30 segundos a 12.000 xg en una microcentrífuga de mesa a 4 º C. Deseche la mayor parte del sobrenadante dejando ≈ 90 l de agua / células NOTA:. En este punto las células pueden ser almacenadas a -80 ° C para su uso posterior. Sin embargo, esto puede reducir la competencia.
- Añadir 2 l de la digerido con BamHI P [ACMAN] (objetivo de 50 ng) a las células y mezclar suavemente usando una punta de pipeta. Transferir las células en una cubeta de electroporación de 1 mm y electroporar a 1.800 V, 25 uF, 200 Ω.
- Añadir 300 l de SOC y dejar que las células se recuperan durante 2 horas a 32 ° C (sacudidas que no es necesario). Células de la placa de agar LB, más 50 mg / ml de ampicilina y se incuba a 32 ° C durante 24-30 horas.
Día 3
- Pantalla para el buen brecha de reparación mediante PCR de colonias. Usar T3 y RA comprobar cebadores para la AR y T7 y el cebador de verificación LA para el LA (Tabla 1). El diseño de la PCR-Check-RA y PCR-Check-LA cebadores 100-200 pb hacia la región específica (Tabla 1). Las colonias positivas de reacciones de PCR usando PCR-Check-RA con T3 o PCR-Check-LA con T7 se muestran una banda que funciona a 800-1.000 pb (Figura 3) NOTA:. Es muy importante para poner a prueba los dos brazos ya que la incorporación de una brazopero no el otro se observa a veces.
- Grow PCR-verificados colonias a 32 ° C en 4 ml de LB 50 g / ml de ampicilina durante la noche. Hacer una reserva congelada de estas células para su uso posterior.
4. Sustitución de la región genómica con el cassette de Orientación
Día 4
- Amplificar el casete nocaut RFP / KAN por PCR usando pENTR-RFP/KAN (previamente digerido con AscI y NheI para linealizar) como una plantilla y la 5'HA-RFP / Kan-F y 3'HA-RFP / Kan-R cebadores ( Tabla 1). RFP / KAN casete puede ser solicitada por la referencia 11.
- Gel de purificar el producto de la PCR eluyendo en 20 l de agua estéril. El casete RFP-KAN más los brazos de homología deben correr 3 kb.
- Inocular 1 ml de la saturada SW102 / P [ACMAN] cultura-KO (paso 3,13) en 44 ml de LB-Amp (50 g / ml) y crecer en 32 ° C agitador a OD 600 entre 0,2-0,3. Incluya una muestra idéntica adicional para el control no-HS. Esta muestra se trata simplementecomo la experimentación en todos los aspectos, con la excepción del SA.
- Después de la SW102 / P [ACMAN] cultura-KO alcanza la densidad deseada, siga los pasos 3,5 a 3,10.
- Electroporar aproximadamente 100 ng de apuntar casete en 90 l de SW102 / P células-KO [ACMAN] electrocompetent. Transferir las células en una cubeta de electroporación de 1 mm y electroporar a 1.800 V, 25 uF, 200 Ω. Añadir 300 l de SOC y permiten que las células se recuperen durante 2 horas a 32 ° C (no se requiere agitación) NOTA:. Es muy recomendable utilizar un gel fresco casete purificada.
- Células de la placa en agar LB + Amp (50 g / ml) + Kan (50 ug / ml) e incubar durante 24-30 horas a 32 ° C.
Día 5
- Crecer 5 ml de cultivo de 5 colonias diferentes a 32 ° CO / N.
Día 6
- Realizar un "sucio miniprep" y ejecutar medio de la ADN obtenido en un gel de agarosa al 1% para buscar la presencia de un bajo peso molecular plásmido. No debería haber ningún plásmido que corre fuelle 12 kb. (Figura 4)
- Hacer una dilución 1:500 de ADN de un clon positivo y el uso de 1 l para electroporar en 50 l de TransforMax EPI300 Importante:. Asegúrese de que para diluir el ADN para prevenir la electroporación de múltiples plásmidos en una célula. Células de la placa en agar LB + Amp (50 g / ml) + Kan (50 ug / ml) e incubar durante 18-24 horas a 37 ° C
Día 7
- Se inocula una sola colonia en 10 ml de LB + y crecer S / N.
Día 8
- Seed 100 ml de medio LB con la cultura saturada de paso 4.10. Añadir antibióticos apropiados además de 100 l de 1000 X solución de control de copia y se incuba a 37 ° C durante 5-6 horas.
- Realizar una maxiprep y verificar la inserción del casete en el sitio correcto mediante secuenciación.
- Además de la secuenciación, realizar una Enzym restricciónprueba de la digestión de correo con el ADN de un clon que no contiene ningún plásmido de bajo peso molecular y su ADN parental. Las enzimas de restricción seleccionadas serán diferentes para cada gen, pero el objetivo es encontrar un grupo de enzimas que permiten al investigador para caracterizar el vector recombineered. A modo de ejemplo, el vector de orientación CG32095 se digirió en serie con PacI, AscI, BamHI y AatII. La aparición de las bandas de predicción y la ausencia de cualquier banda incorrectas confirma que el vector de orientación se ha recombineered correctamente (Figura 5).
5. Inyección de moscas y Movilización Cassette En Vivo
- Inyectar el casete utilizando C31 de la integrasa, en un lugar de aterrizaje predefinido de elección. Proveedores externos, como el arco iris transgénicos ( http://www.rainbowgene.com ), se pueden utilizar los servicios de inyección Importante:. El ADN debe ser recién pcorrigieron antes de la inyección. Mayor eficiencia de transformación se observa cuando el ADN se preparó en el lugar y se inyecta el mismo día.
- Para movilizar el cassette de la plataforma de aterrizaje, utilice Bloomington stock number 25680 o 25679. Estas poblaciones contienen flipasa y II CPE aguas abajo de un promotor de choque térmico, en el cromosoma dos y tres, respectivamente. Además, contienen un hs-hid transgén en el cromosoma equilibrador y en el Y-cromosoma. Recoge los hombres de la acción elegida y cruzar con las hembras vírgenes que llevan el casete KO en el lugar de aterrizaje. . Set ~ 30 cruces y permiten a las hembras pongan huevos durante 2-3 días NOTA: Se recomienda seleccionar la acción que se encuentra en un cromosoma distinto al lugar de destino.
- Los padres del tirón en un nuevo conjunto de 30 viales, y larvas de choque térmico a 37 ° C durante 1 hora, dos veces al día, durante tres días consecutivos. Los choques de calor activan la producción de las enzimas; flipasa y la I-SceI, y el gen de muerte celular se escondieron , Matando a los hombres y las moscas que llevan cromosomas equilibradores. Permitir a los padres en los viales recién volteado a poner huevos durante 2-3 días, luego repita los golpes de calor para los viales, después de mover de un tirón los padres en nuevos viales de nuevo. Repita esto para varias tiradas, hasta que los padres no ponen huevos más.
- Recoger hembras vírgenes de la progenie F1 de las moscas al calor conmocionado, y cruzar a y - w - varones. Trate de 150-200 cruces, tres vírgenes y tres varones por cada cruz.
- Pantalla de la progenie F2 bajo un microscopio fluorescente que permite la detección de RFP en el ojo. Pantalla de RFP ojos positivos que son mutantes de los genes blancos y amarillos (y - w -). Este proceso de selección selecciona positivamente para la presencia de la casete de RFP y selecciona para la ausencia de la [ACMAN] vectorial (mini-blanco) y el sitio de aterrizaje P, que está marcada por el gen de tipo salvaje amarillo NOTA:. Es menos tiempo -conasumiendo para detectar RFP en el ojo, que a primera pantalla para PA moscas. Los eventos de movilización son muy eficientes durante los choques de calor, lo que resulta en un menor número de moscas que contienen muchos RFP en el ojo, en comparación con Y - W - moscas.
- Configurar apareamientos solo par con cada Y - W - RFP + mosca, ya que cada evento de movilización puede ser diferente. Mapa del RFP para el cromosoma correcta. Correcta orientación cromosoma se observa típicamente en> 95% de los casos.
- Establecer acciones y comprobar los eventos dirigidos correctas utilizando técnicas estándar para el análisis de Southern Blot. Diseño de una sonda de 2 kb aguas arriba (brazo izquierdo) y aguas abajo (brazo derecho) del gen que se dirige, y 2 kb (o más corto si es necesario) que abarcan el ORF suprimido. Las dos primeras sondas dará lugar a una banda en los agujeros ciegos de homocigotos y heterocigotos, que no está presente en el tipo salvaje, mientras que el ORF no dará lugar a una banda en el nocaut homocigotos. Una rápida PCR fo la ORF también es posible, sin embargo, esto se basa en la ausencia de una banda.
Resultados
La amplificación de la LA y brazos de homología con AR debe producir 500 bp productos y la reacción de PCR-SOE debe producir un producto de 1,0 kb (Secciones 2.1 a 2.4, Figura 2). La reacción BP realizado en la sección 2.5 es típicamente muy eficiente y la transformación bacteriana de los rendimientos de los productos 5-100 colonias en promedio. Casi todas las colonias probadas con PCR de verificación Mostrar el producto de PCR esperado.
Durante la primera ronda de recombineering (Sección 3) esperar para obtener colonias de 20-40 después de la transformación de la P digerido [ACMAN]-KO-1.0 que contiene la LA y los brazos con AR en las células SW102. 40-60% de estos clones llevará el producto de recombinación deseado. El control de choque de calor no debe contener muy pocos o ninguno de colonias en comparación con las células al calor conmocionado. La aparición del mismo número de colonias en tanto la muestra como las placas de control por lo general indica la presencia de plásmido sin cortar o algún otro contaminante. En este caso, el experimento Should ser desechado y repitió, esta vez la digestión de la P [ACMAN]-KO-1.0 que lleva el LA y RA brazos hasta el final siguiendo las instrucciones descritas en el paso 3.2. Un ejemplo de un cheque de PCR para la recuperación de la región genómica de interés en P [ACMAN]-KO 1.0 en el que se obtuvieron ≈ 60 colonias se muestra en la Figura 3. Aquí 60% de las colonias ensayadas lleva a la product.Care recombinación deseado debe tener cuidado de que los productos de la PCR observados se ejecutan en el tamaño previsto, ya que las bandas de PCR aberrantes por lo general indican eventos recombinería incorrectos.
De vez en cuando hay colonias crecen después de la primera ronda de recombineering. El uso de células con una eficacia sub-óptima representa la causa más común para una reacción recombinería fallado. Mantener las celdas frías en todo momento durante su preparación y manipulación suave durante los lavados (NUNCA vórtice) es crítico para la obtención de células competentes de alta calidad. Trate de utilizar siempre recién digeridas / P [ACMAN]-KO-purificada1.0 ADN para obtener la máxima eficiencia.
A veces, la primera ronda de los rendimientos de las colonias recombinería pero esas colonias no pasan la prueba de verificación de PCR. Esto suele indicar productos recombinería incorrectos, pero tal resultado también puede indicar problemas potenciales con uno de los conjuntos de cebadores. Para validar el primer PCR-Check-LA, crear una reacción de PCR en combinación con el att B1-II CPE-LA-F. Para verificar PCR-check-RA creó una reacción de PCR con el attB2-II CPE-RA-R. En ambos casos utilizar el BAC original como una plantilla. Estas reacciones de PCR deben producir productos del tamaño esperado, que variarán dependiendo de la secuencia de los cebadores de verificación. Los resultados positivos de este experimento de control se descarta la posibilidad de que la incapacidad para detectar clones positivos se debe a la ineficiencia de imprimación.
Si usted ha tratado de la primera ronda de recombinería varias veces siguiendo el protocolo anteriormente sin ningún éxito y ha actuado por todo elsugirió controles, se recomienda elegir un nuevo par de brazos de homología 500 pb. Por razones que no están claras en el momento actual, algunas regiones del genoma son altamente resistentes a la recombinación de ADN. Simplemente el diseño de nuevos brazos de homología y moverlos hacia o desde el gen de interés a veces resuelve estos problemas.
La segunda ronda de recombineering (sección 4) suele ser mucho más eficiente. Normalmente 30-50 colonias se observaron después de la transformación, con 90-95% de estos que contiene la construcción deseada. El problema más común en este paso es la obtención de colonias de falsos positivos. Figura 4 muestra ejemplos de verdaderos y falsos clones positivos después de la segunda ronda de recombineering (sección 4).
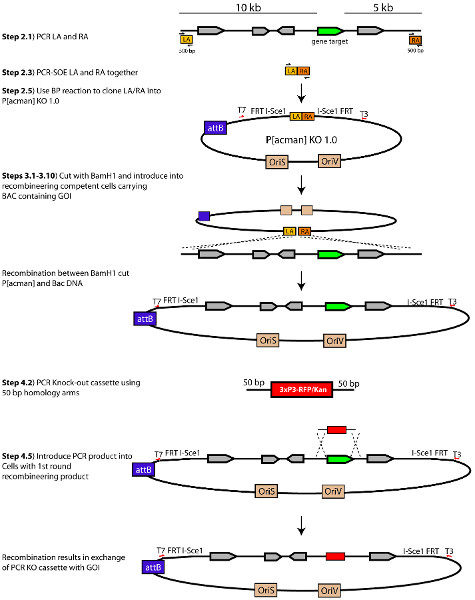
Figura 1. Esquema de la proceso de generar una P [ACMAN]-KO 1.0 vector dirigido. Modificado de Chan et al. (2012). Haga clic aquí para ver más grande la figura .

Figura 2. Esquema de empalme por la superposición de PCR (PCR SOE) realizó en el paso 2.3. Haz clic aquí para ver más grande la figura .
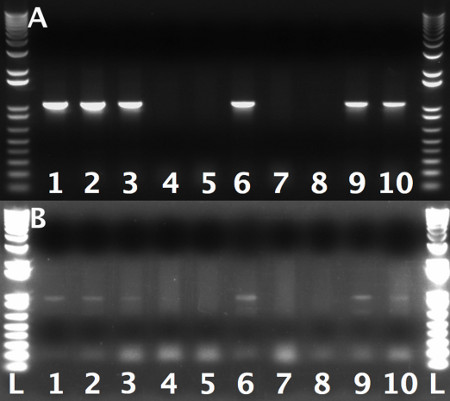
Figura 3. PCR en gel de comprobar la eficacia que muestra de primer evento recombineering Cada carril representa una sola colonia A:.. Colonias probados para el brazo izquierdo. integración utilizando T7 y LA-verificación cebadores B: Las mismas colonias como A, probados para la integración brazo derecho utilizando T3 y RA-cheque primers.1kb Plus DNA Ladder se utilizó como un marcador de peso molecular NOTA:. Debido a la mala cebado de T3 imprimación en relación con T7, las bandas de la verificación del brazo derecho fueron significativamente más oscuro. Esto debe tenerse en cuenta cuando se visualiza el gel.

La Figura 4. Gel de agarosa mostrando plásmidos de ADN de alto peso molecular (P [ACMAN]) y bajo peso molecular plásmido (plantilla sin digerir) aislado de posibles clones positivos después de la segunda ronda de recombinería. ADN fue aislado a partir de 4 posibles clones positivos y corrió en un gel de agarosa al 1% teñidos con bromuro de etidio. Los clones 1,3 y 4 son verdaderos clones positivos. Clone 2 es positivo falso como lo señaló la todas las formas de ap lasmid que corre fuelle 12 kb (el tamaño de la banda más grande de ADN escalera carril). 1 kb Plus DNA Ladder se utilizó como un marcador de peso molecular.

Figura 5. Prueba de evento de recombinación deseado por la enzima de restricción digestión. En el ejemplo anterior se muestra una computadora-predice patrón de bandas del P [ACMAN]-KO CG32095 antes y después de la segunda ronda de recombineering. Se utilizaron cuatro enzimas diferentes: PacI, AscI, AatII y BamHI. Correctamente producto recombinado tendrá las mismas bandas que su parental P [ACMAN]-KO CG32095 excepto por el cambio previsto (puntas de flecha). NEBcutter V2.0 se utilizó para predecir el patrón de bandas.
0346/50346fig6highres.jpg "src =" / files/ftp_upload/50346/50346fig6.jpg "/>
Figura 6. Esquema que describe la movilización de un cassette de selección in vivo. Haz clic aquí para ver más grande la figura .

Tabla 1. Imprimaciones. Haz clic aquí para ver la tabla más grande .
Discusión
El poder de los organismos modelos genéticos en la investigación biomédica se basa en gran medida de las herramientas disponibles para la manipulación genética. Los modelos pequeños C. elegans y Drosophila, en particular, permiten a los análisis genéticos moleculares de bajo costo y rápida de las rutas completas y familias de genes implicados en el desarrollo o función multicelular. Los últimos años han visto avances significativos en el desarrollo de herramientas para la manipulación de genes en Drosophila 14,15. Por ejemplo, recombinería, que es ampliamente utilizado en la genética del ratón para manipular Bac construcciones de ADN, se ha adaptado recientemente para Drosophila, a través del uso de los P [ACMAN] vector de transgénesis y C31-mediada 6. Las diferentes casetes de selección permiten la inserción o deleción de secuencias específicas en cualquier lugar dentro de una construcción usando bacterias competentes de recombinación 4,13. Hemos modificado el original P [ACMAN] vector de modo que se puede utilizar para construir De vectores de recombinación homóloga in vivo que se dirigen loci endógenos en Drosophila. Este nuevo vector capacita para generar dirigidos cassettes con brazos de homología más grandes que los que se pueden hacer fácilmente utilizando métodos de clonación corte y pegar. Además, un casete de clonación de puerta de enlace-también se ha incorporado en este vector, que permite una para introducir rápidamente y eficientemente nuevo ADN genómico en el sistema.
Aunque la mayor parte de la metodología descrita aquí es sencillo, hay varios pasos que hemos encontrado crítico para el éxito de la técnica. Debido al tamaño del ADN manipulado (> 15 kb), la primera ronda de reacción recombinería (sección 3) representa un obstáculo técnico importante. Por lo tanto, esta ronda de recombineering requiere un protocolo de recombinación muy eficiente. Ajustes de protocolo aparentemente menores, como el cultivo de las células a la densidad más baja, lavar las células tres veces en lugar de dos y con 10% de glicerol en lugar de dH 2 </ Sub> O 13, han mejorado en gran medida la eficiencia de transformación de nuestras células competentes recombinería. Además, teniendo cuidado de asegurarse de que el LA / RA que contiene P [ACMAN] se corta completamente con BamHI (Paso 3.1) elimina muchos falsos positivos.
Eventos de recombinación adversas representan otro problema bastante común durante recombineering. Por ejemplo, hemos observado casos de duplicaciones y translocaciones en diversas construcciones. Estos eventos pueden conducir a varios problemas durante la transformación Drosophila y en vivo de genes diana. Verificación de PCR y análisis de secuenciación no pueden detectar todos estos eventos. Por lo tanto, un análisis de enzima de restricción se debe realizar en el producto final clonado antes de la inyección de ADN en embriones. Esta comprobación final ha resultado fundamental y nos ha permitido evitar la inyección de construcciones que contienen aberraciones indeseadas.
Maxi-preparar el vector dirigido justo antes de injección y nunca congelar el ADN mejora en gran medida la eficiencia de transformación de estas grandes construcciones en Drosophila. En resumen, los pequeños detalles hacen una gran diferencia en el éxito de estas técnicas. Los protocolos descritos y demostrados aquí representan puntos de vista de una serie de diferentes fuentes y nuestra propia experiencia.
Mientras esperamos el protocolo presentado aquí ayuda a los demás a adoptar métodos recombinería en sus propios laboratorios, creemos nuevas mejoras y las mejoras de la metodología son todavía posibles. Ocasionalmente, fragmentos genómicos específicos son difíciles de manipular utilizando recombineering por razones que no son completamente evidentes. Por otra parte, a pesar de nuestros mejores esfuerzos para eliminar de fondo no deseado, todavía encontramos que ciertos fragmentos de ADN genómico tienden a producir productos finales incorrectamente recombineered. Una comprensión más profunda del proceso recombineering puede producir protocolos más óptimas en el futuro y abrir la discusión derecombinería fracasos y éxitos fomentará el uso exitoso de estas técnicas de gran alcance de la comunidad científica en general.
Divulgaciones
Los autores no tienen intereses en competencia en lo que respecta a las técnicas descritas aquí.
Agradecimientos
Nos gustaría dar las gracias a Hugo Bellen y la Bolsa Center Bloomington para los reactivos. Más Agradecemos Koen Venken, Hugo Bellen y todos los miembros de los laboratorios Buszczak y Hiesinger útil para los debates. Este trabajo fue apoyado por becas de los Institutos Nacionales de Salud de ACR (T32GM083831), PRH (RO1EY018884) y MB (RO1GM086647), una subvención por el Instituto de Investigación de la Prevención del Cáncer de Texas a MB y PRH (RP100516), y el Welch Fundación (I-1657) a PRH. MB es una EE y Greer Garson Fogelson Académico en Investigación Biomédica y PRH es un erudito Eugene McDermott en Investigación Biomédica de UT Southwestern Medical Center.
Materiales
| Name | Company | Catalog Number | Comments |
| Name of the reagent | Company | Catalogue number | Comments (optional) |
| SW102 Recombination competent bacteria | NCI-Frederick | Recombination Bacteria (SW102, SW105 and SW106) | http://ncifrederick.cancer.gov/research/brb/logon.aspx |
| TransforMax EPI300 electrocopmpetent E. coli | Epicentre | EC300110 | Includes CopyControl induction solution |
| PfuUltra II Fusion HS DNA Polymerase | Aligent Technology Inc. | 600670 | |
| BamHI-HF | New England Biolabs | R3136S | |
| Zymoclean Gel DNA Recovery Kit | Zymo Research | D4001 | |
| Use Gateway BP ClonaseII Enzyme kit | Invitrogen | 11789-020 | |
| P[acman]KO1.010 | Buszczak and Hiesinger Labs | Upon request | |
| pENTR RFP-Kan11 | Buszczak and Hiesinger Labs | Upon request | |
| Flystocks | Bloomington stock center | Stock numbers: 25680, 25679 | y1 w*/Dp(2;Y)G, P{hs-hid}Y; P{70FLP}11 P{70I-SceI}2B snaSco/CyO, P{hs-hid}4 y1 w*/Dp(2;Y)G, P{hs-hid}Y; P{70FLP}23 P{70I-SceI}4A/TM3, P{hs-hid}14, Sb1 |
| Electroporation machine | Biorad GenePulser Xcell with PC module | 165-2662 | |
| Cuvettes | Fisher Brand | #FB101 |
Referencias
- Rong, Y. S., Golic, K. G. A targeted gene knockout in Drosophila. Genetics. 157, 1307-1312 (2001).
- Rong, Y. S., Golic, K. G. Gene targeting by homologous recombination in Drosophila. Science. 288, 2013-2018 (2000).
- Gong, W. J., Golic, K. G. Ends-out, or replacement, gene targeting in Drosophila. Proc. Natl. Acad. Sci. U.S.A. 100, 2556-2561 (1073).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36 (2005).
- Copeland, N. G., Jenkins, N. A., Court, D. L. Recombineering: a powerful new tool for mouse functional genomics. Nat. Rev. Genet. 2, 769-779 (2001).
- Venken, K. J., He, Y., Hoskins, R. A., Bellen, H. J. P[acman]: a BAC transgenic platform for targeted insertion of large DNA fragments in D. melanogaster. Science. 314, 1747-1751 (2006).
- Venken, K. J., et al. Versatile P[acman] BAC libraries for transgenesis studies in Drosophila melanogaster. Nat. Methods. 6, 431-434 (2009).
- Groth, A. C., Fish, M., Nusse, R., Calos, M. P. Construction of transgenic Drosophila by using the site-specific integrase from phage phiC31. Genetics. 166, 1775-1782 (2004).
- Bischof, J., Maeda, R. K., Hediger, M., Karch, F., Basler, K. An optimized transgenesis system for Drosophila using germ-line-specific phiC31 integrases. Proceedings of the National Academy of Sciences of the United States of America. 104, 3312-3317 (2007).
- Chan, C. C., et al. Systematic discovery of Rab GTPases with synaptic functions in Drosophila. Current Biology: CB. 21, 1704-1715 (2011).
- Chan, C. C., Scoggin, S., Hiesinger, P. R., Buszczak, M. Combining recombineering and ends-out homologous recombination to systematically characterize Drosophila gene families: Rab GTPases as a case study. Commun. Integr. Biol. 5, 179-183 (2012).
- Wu, N., Matand, K., Kebede, B., Acquaah, G., Williams, S. Enhancing DNA electrotransformation efficiency in Escherichia coli DH10B electrocompetent cells. Electronic Journal of Biotechnology. 13, (2010).
- Wang, S., Zhao, Y., Leiby, M., Zhu, J. A new positive/negative selection scheme for precise BAC recombineering. Mol. Biotechnol. 42, 110-116 (2009).
- Venken, K. J., Bellen, H. J. Transgenesis upgrades for Drosophila melanogaster. Development. 134, 3571-3584 (2007).
- Dietzl, G., et al. A genome-wide transgenic RNAi library for conditional gene inactivation in Drosophila. Nature. 448, 151-156 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados