Method Article
Recombineering homologen Rekombination Konstrukte in
In diesem Artikel
Zusammenfassung
Homologe Rekombination Techniken stark voranbringen Drosophila Genetik, indem sie die Erstellung von molekular genauen Mutationen. Die jüngste Verabschiedung Rekombination ermöglicht es, große Stücke von DNA manipulieren und transformieren sie in Drosophila 6. Die hier vorgestellten Methoden kombinieren diese Techniken, um schnell erzeugen große homologe Rekombination Vektoren.
Zusammenfassung
Die Weiterentwicklung von Techniken für schnelle, großflächige Manipulation der endogenen Genorte verbreitern den Einsatz von Drosophila melanogaster als genetischer Modellorganismus für menschliche Krankheiten Forschung. In den letzten Jahren haben technische Fortschritte wie die homologe Rekombination und Rekombination gesehen. Allerdings erzeugt eindeutige Null-Mutationen oder Tagging endogenen Proteinen bleibt ein erheblicher Aufwand für die meisten Gene. Hier beschreiben wir demonstrieren und Techniken für den Einsatz Recombineering-basierte Klonen Methoden, um Vektoren, die verwendet werden, um gezielt zu manipulieren und endogenen Loci in vivo zu generieren Insbesondere haben wir eine Kombination von drei Technologien etabliert:. (1) BAC Transgenese / Rekombination, ( 2) Enden-out homologen Rekombination und (3) Gateway-Technologie, um eine stabile, effiziente und flexible Verfahren zum Manipulieren endogenen genomischen Loci. In diesem Protokoll, bieten wir Ihnen Schritt-für-Schritt-Anweisungen, wie man (1) Design individual Vektoren, (2) wie man große Fragmente genomischer DNA in der homologen Rekombination mit Vektor Lücke Reparatur, und (3) wie zu ersetzen oder zu kennzeichnen Gene von Interesse innerhalb dieser Vektoren mit einem zweiten Runde der Rekombination klonen. Schließlich werden wir auch ein Protokoll, wie Sie diese Kassetten in vivo zu mobilisieren, um einen Knockout oder eine markierte Gen über Knock-in generieren. Diese Methoden können leicht für mehrere Ziele parallel angenommen werden und ein Mittel zur Manipulation der Drosophila-Genom in eine rechtzeitige und effiziente Art und Weise.
Einleitung
Reinigen molekular definierten Manipulationen einzelner Gene auf ihre endogenen Loci bieten ein unschätzbares Werkzeug, um eine Vielzahl von relevanten Fragen eukaryotischen Biologie zu studieren. Drosophila umkehren genetische Techniken zur Erzeugung von loss-of-function-Allele hatten nachweislich eine Herausforderung sein, bis Golic und Kollegen in eingeführt vivo Gen-Targeting mit homologe Rekombination Drosophila 1-3. Sie zeigten, dass spezifische Loci konnten gezielt mit einem linearen DNA-Fragment von einem integrierten transgenen Konstrukts werden. Diese lineare "Donor"-DNA wird in vivo durch FRT-vermittelte Rekombination (Verbrauchsteuerpflichtige die DNA aus dem Chromosom eines ringförmigen Moleküls) durch Linearisierung der Meganuclease I-SceI gefolgt erzeugt. Obwohl diese Methode wurde erfolgreich verwendet worden, um eine Vielzahl von definierten Läsionen zu erzeugen, ist die Technik nicht leicht skalierbar zur Manipulation zahlreicher Gene in parallel, weil jeder einzelnenDual-Knockout-Konstrukt erfordert deutliche und individuelles Design. Beispielsweise Schwierigkeiten nahtlos Umgang mit großen DNA-Fragmente (> 5 kb) in vitro unter Verwendung klassischer Restriktionsenzym / Ligation Klonen oder PCR, sowie die Größe Einschränkungen herkömmlicher in vivo Transformationsvektoren oft die schnelle Entwicklung der homologen Rekombination gezielt beeinflussen Vektoren. Um diese Einschränkungen zu überwinden, kombinierten wir die Rekombination / Transgenese P [Pacman]-System, das die Sub-Klonierung und Transgenese von bis zu 100 kb ermöglicht der DNA, wobei die Enden-out Gen-Targeting-Methoden, um eine effiziente und relativ schnelle Plattform zu etablieren, dass erleichtert Drosophila Gen-Targeting.
Rekombination vermittelte Gentechnik (Rekombination) ist ein leistungsfähiges homologe Rekombination basierende Technologie des Klonens 4,5. Im Gegensatz zu herkömmlichen Restriktionsenzym / Ligase Klonen wird nicht durch Rekombination der Sequenz oder siz begrenzte der manipulierten DNA. Recombineering verwendet eine spezielle E. coli-Stamm, der Rekombination Maschinen durch eine defekte λ Prophagen 4 vorgesehen birgt. Diese Technik wurde kürzlich für den Einsatz in Drosophila 6,7 angenommen worden. Recombineering in Drosophila beruht auf einer modifizierten bedingt amplifizierbare bakterielle künstliche Chromosom (BAC) Vektor genannt P [Pacman] 6,7. Dieser Vektor trägt zwei Replikationsstartpunkte: oriV, die hohe Kopienzahl produziert auf chemische Induktion für die Reinigung von großen Mengen an DNA für die Sequenzierung und Embryo-Einspritzung und Oris, die Low-Kopienzahl unterhält unter basalen Bedingungen erforderlich. Zusätzlich wird die P [Pacman] Vektor wird mit einer bakteriellen Befestigung (attB) Website ausgestattet. Die attB-Stelle dient als Substrat für ΦC31 Integrase-vermittelten Gentransfer, dass der Einbau von großen DNA-Fragmente können in eine vorbestimmte Landeplatz im Drosophila Genom 8,9.
Wir haben ein P [Pacman] Vektor (bezeichnet als P [Pacman]-KO 1.0), die als Targeting-Vektor für Enden-out homologe Rekombination 10,11 verwendet werden können, erzeugt. Um übernehmen Enden-out-Gen-Targeting-Technologie in das System, haben wir zwei FRT und zwei I-SceI-Websites. Wir haben auch eine Gateway-Kassette in diesem modifizierten Vektor, um den Prozess der Einarbeitung der Homologie Arme in P [Pacman]-KO 1.0 straffen enthalten. Dies stellt eine schnelle und einfache Art und Weise nahezu jede genomischen Region von Interesse in den Targeting-Vektor einzuführen. In diesem Protokoll werden wir beschreiben, wie ein Targeting-Vektor mit P [Pacman]-KO 1.0, und wie man diesen Vektor in vivo mobilisieren, um die endogenen Locus gezielt konstruieren. Für die Zwecke dieses Protokolls verwenden wir den RFP / Kan-Kassette, ein Gen von Interesse zu ersetzen, sondern eine Vielzahl von Kassetten, die ein Antibiotikum Selektionsmarker enthalten, können mit diesem Protokoll verwendet werden. Wir haben entworfen und erfolgreich eingesetzt eine Reihe von Kassetten fürGen-Ersatz-und-Tagging 10,11.
Protokoll
1. Auswahl der BAC und Region an TARGET
- Um eine BAC mit dem Gen von Interesse (GOI) (hier CG32095 wird als Beispiel verwendet), die Suche nach CG32095 bei erwerben www.flybase.org . Unter der Rubrik Aktien und Reagenzien, überprüfen Sie den Abschnitt "Genomic Clones" für BACs mit CG32095. Stellen Sie sicher, dass die BAC mindestens 10 kb stromaufwärts und stromabwärts 5 kb das Gen von Interesse enthält. Alternativ können Klone in der P [Pacman] Vektor-7 auch zu finden unter http://www.pacmanfly.org/libraries.html .
- Die BAC-Klone in DH10B Zellen kommen und die P [Pacman] Klone in EPI300 Zellen kommen. Die genomische Klone müssen in SW102-Zellen, die zugänglich für Rekombination sind transformiert werden. "Schmutzige Minipreps" durchgeführt werden (a Qiagen Miniprep ohne Verwendung einer Säule und Ausfällen der DNA mit Isopropanol) abaus einer 5 ml O / N-Kultur mit Zellen, die das BAC geimpft. Nach der erneuten Aussetzung der frisch zubereiteten DNA-Pellet in 20 ul dH 2 O, verdünnter 1 ul in 100 ul dH 2 O und 1 ul verwenden, um elektrokompetente SW102 elektroporieren. Um elektrokompetente SW102 verwenden Sie den folgenden Protokoll:
- Impfen SW102 Zellen in 4 ml LB. Wachsen Kultur bei 32 ° CO / N Schütteln bei 250 UpM. Wichtig: SW102 Zellen sollten NIE auf eine Temperatur höher als 32 ° C ausgesetzt werden, wenn die Induktion der Rekombination Maschinen erwünscht ist.
- Impfen 1 ml der gesättigten SW102 Kultur in 44 ml LB und wachsen auf einem 32 ° C Schüttler bis OD 600 zwischen 0,2-0,3 (in der Regel ca. 1-1,5 Std.). HINWEIS: Weitere Recombineering Protokolle empfehlen wächst die Kultur in einem kleineren Volumen zu OD 600 0,6-0,7, jedoch das Wachstum der Bakterien zu einer niedrigeren Dichte in einem größeren Volumen erhöht Transformation efficiency 12. Außerdem verringert diese Modifikation die Wartezeit.
- Kühlen Sie die Kultur in Eis-Wasser-Schlamm für 5 min. Wichtig: Während der folgenden Schritte Zellen müssen bei niedrigen Temperaturen gehalten werden. Dies ist entscheidend für den Erhalt guter Qualität elektrokompetente Zellen.
- Zentrifuge Kultur bei 4 ° C bei 1.500 xg für 10 min. Überstand verwerfen und waschen Pellet in 25 ml kaltem (4 ° C) 10% Glycerin 13. Zunächst Zellen in 5 ml durch Antippen und wirbelnden das Rohr in Eis-Wasser-Schlamm (NICHT Pipette auf und ab) und fügen Sie dann die restlichen 10% Glycerin (20 ml).
- Zentrifuge bei 4 ° C bei 1.500 xg für 10 min, Überstand verwerfen und waschen das Pellet ein zweites Mal mit 25 ml eiskaltem 10% Glycerin.
- Zentrifuge bei 4 ° C bei 1.500 xg für 10 min, Überstand verwerfen und Waschen des Pellets ein drittes Mal mit 25 ml eiskaltem 10% Glycerin.
- Zentrifuge bei 4 ° C bei 1500 xg für 10 min und entsorgen supernatant. Das Zellpellet kann sehr lose an dieser Stelle und sollte darauf geachtet werden, wenn der Überstand verworfen werden. Resuspendieren des Pellets in 1 ml kaltem 10% Glycerin. Übertragen der Zellen in einem 1,5 ml Röhrchen und zentrifugieren für 30 Sekunden bei 12.000 × g in einer Tischplatte Mikrozentrifuge bei 4 ° C. Entsorgen Sie die meisten der Überstand verlassen ≈ 90 ul Glycerin / Zellen. Die Zellen sind nun bereit, Elektroporation werden. HINWEIS: An dieser Stelle können Zellen bei -80 ° C für eine spätere Verwendung gespeichert werden. Dies kann jedoch zu verringern Kompetenz.
- Elektroporieren DNA in Zellen mit einer 1 mm Küvette bei 1.800 V, 25 uF, 200 Ω. Nach der Elektroporation, fügen Sie 300 ul SOC und lassen die Zellen für 2 Stunden erholen bei 32 ° C (Schütteln ist nicht erforderlich). Platte Zellen auf LB-Agar plus 25 ug / ml Chloramphenicol (oder andere geeignete Antibiotika in Abhängigkeit von der transformierten BAC oder PAC) und Inkubation bei 32 ° C für 24-30 Stunden.
- Homologiearme für Gen-Targeting durch select ausgelegting 10 kb genomischer DNA stromaufwärts und 5 kb stromabwärts bezüglich der GOI. Wählen Sie 500 bp am Ende des 10 kb und 5 kb Homologie Arme zu verstärken (siehe Abschnitt 2) (Abbildung 1). Diese 500 bp-Fragmente dienen als Homologiearme verwendet, um die genomische Region um die indische Regierung in P [Pacman] KO 1.0 rekombinieren. Diese Fragmente werden als linken Arm (LA) für die Region, die zu den vorgelagerten 5 entspricht "Homologie Arm und den rechten Arm (RA) für die Region, die an die Downstream-3 'entspricht Homologiearm bezeichnet. Wichtig: Homology Arme sollten nicht enthalten BamHI Restriktionsschnittstellen, wie die Linearisierung des Plasmids (in Schritt 3.2) über eine BamHI-Restriktionsverdau durchgeführt.
2. Legen Sie die Arme in Homology P [Pacman]-KO 1.0
- Richten Sie zwei einzelnen PCR-Reaktionen, eine für die LA und andere zu verstärken, um die RA verstärken. Für die LA, verwenden Primer attB1-I-SceI-LA-F und BamHI-LA-R und verwenden Sie die Primer BamHI-RA-F und attB2-I-SceI-RA-Rdie RA (Tabelle 1) zu verstärken. Verwenden Sie 1 ul BAC DNA aus Schritt 1.2 oder 0.5 ul gekocht Nacht Bakterienkultur mit BAC von Interesse als Vorlage. Achten Sie darauf, eine DNA-Polymerase mit Korrekturlesen Fähigkeit zu verwenden. Dies gilt für alle nachfolgenden PCRs-mit Ausnahme des PCR-Check in den Schritten 2.8 und 3.13. HINWEIS: Verlängerung der Zeit für eine Korrektur-DNA-Polymerase kann von regelmäßigen Taq DNA Polymerase je nach Hersteller. PfuUltra II Fusion HotStart, die mit einer Rate von 15 sec / kb polymerisiert, kann in diesen Schritten verwendet werden.
LA / RA PCR
PCR-Bedingungen1 ul DNA aus schmutzigen miniprep aus Schritt 1.2 des Bac 0,25 ul Jeder 20 pM Primer 2,5 ul 10X PfuUltra II Buffer 0,75 ul 10mM (jeweils) dNTP 0,5 ul PfuUltra II Fusion HotStart DNA Polymerase 19.75 ul Wasser 25 ul Gesamtvolumen Schritt 1 95 ° C 2 min bis Enzym aktivieren Step2 95 ° C 20 sec Denaturierung Schritt 3 60 ° C 20 sec Glühen Schritt 4 72 ° C 20 sec Verlängerung Step5 Weiter zu Schritt 2 und wiederholen Sie 29 Zyklen Step6 4 ° C halten - Führen Proben auf einem 1% igen Agarosegel und Auszug mit einem Zymoclean DNA Recovery-Paket. Eluieren der DNA aus der Zymoclean Säule mit 10 ul sterilem Wasser.
- Führen Spleißen durch überlappende Verlängerung (PCR SOE) mit den beiden Fragmente mit 10-30 ng Gesamt-DNA aus jedem Produkt verstärkt in 2.2 (Abbildung 2) gereinigt. Dies ist in der Regel um 1/20 th jedes gereinigte PCR-Produkt:
LA / RA PCR-Soe
PCR-Bedingungen0,5 ul jeder LA und RA gereinigte PCR-Produkt etwa 10-30 ng 0,25 ul jeder 20 pM Primer 2,5 ul 10X PfuUltra II Buffer 0,75 ul 10mM (jeweils) dNTP 0,5 ul PfuUltra II Fusion HotStart DNA Polymerase 19.25 ul Wasser 25 ul Gesamtvolumen Schritt 1 95 ° C 2 min bis Enzym aktivieren Step2 95 ° C 20 sec Denaturierung Schritt 3 55 ° C 20 sec Glühen (Lower Temp. Für Arme ermöglichen Glühen) Schritt 4 72 ° C 20 sec Verlängerung Step5 Gehe zu Schritt 2 und wiederholen 1 Zyklus Step6 95 ° C 20 sec Denaturierung Step7 60 ° C 20 sec Glühen Step8 72 ° C 20 sec Verlängerung Step9 Weiter zu Schritt 2 und wiederholen Sie 27 Zyklen Step10 4 ° C halten - Führen Sie die PCR Probe auf einem Agarosegel und wieder das 1,0 kb-Bande durch Gel-Extraktion. Dies ist die attB1-LA-BamHI-RA-attB2 Kassette.
- Verwenden Gateway-BP ClonaseII Enzyme Kit, eine BP-Reaktion nach dem Protokoll des Herstellers.Die Kassette in 2.4 erzeugt wird als Spender-DNA und der P [Pacman]-KO 1.0 als Ziel Vektor handeln.
- Verdünnen Sie 50 ul TransforMax EPI300 elektrokompetente Zellen durch Zugabe von 30 ul kaltem 10% Glycerin oder kalt dH 2 O. Verwandeln 1 ul der BP-Reaktion in elektrokompetente Zellen nach dem Protokoll des Herstellers. Die Zugabe von Wasser verdünnt das relativ hohe Salzkonzentration des BP Reaktion auf elektrische Entladung (Lichtbogen) bei der Elektroporation zu verhindern. Elektroporieren DNA in Zellen mit einer 1 mm Küvette bei 1.800 V, 25 uF, 200 Ω.
- In 300 ul SOC und lassen Zellen für 1 Stunde erholen bei 37 ° C (Schütteln ist nicht erforderlich). Teller Zellen auf LB-Agar + 50 ug / ml Amp (P [Pacman]-KO 1.0 enthält eine Amp-Resistenz-Gen). Platten bei 37 ° C für 18-24 Stunden.
- PCR prüfen einzelnen Kolonien durch herkömmliche Kolonie-PCR unter Verwendung von T3 (T3-Sequenz stromabwärts der Gateway-Kassette in der P [Pacman]-KO 1.0 (Abb.Abbildung 1) und attB1-I-SceI-LA-F Primer. Positive Klone zeigen eine Band, die bei 1,2 kb läuft.
- Wachsen einer Übernachtkultur eines positiven Klons in LB mit 50 ug / ml Ampicillin.
- Erhöhen der P [Pacman]-KO 1.0 Vektor, der LA und RA in einem 10 ml LB mit 50 ug / ml Ampicillin Kultur mit Copy Control-Lösung nach dem Protokoll des Herstellers (Epicentre).
- Führen Sie eine Qiagen Miniprep nach dem Protokoll des Herstellers.
3. Rekombination der genomischen Region of Interest in P [Pacman]-KO 1.0
Tag 1
- Impfen SW102 Zellen mit dem BAC von Interesse in 4 ml LB plus 25 ug / ml Chloramphenicol (CHL) oder die entsprechende Auswahl Antibiotikum. Wachsen Kultur bei 32 ° CO / N Schütteln bei 250 Upm. Wichtig: Außer während der Einleitung in Schritt 3.5, sollte SW102 Zellen nicht auf eine höhere Temperatur als 32 ° C ausgesetzt werden, wie hohe Temperatur erfolgt die Rekombination maChinery dieser Zellen.
Tag 2
- Digest 0,4 ug des P [Pacman]-KO-1.0 mit den LA und RA Arme in Abschnitt 2 mit ≈ 20 Einheiten BamHI in einem 25 ul Reaktion erzeugt. Bei 37 ° C für mindestens 3 Stunden. Wichtig: Lange Verdauung ist entscheidend, um die Verdauung aller DNA sicherzustellen. Dadurch wird die Anzahl der Fehlalarme Klone später zu verringern.
- Impfen 1,0 ml der gesättigten SW102/BAC Kultur in 44 ml LB-Chl (25 ug / ml) und wachsen auf einem 32 ° C Schüttler bis OD 600 zwischen 0,2-0,3 (in der Regel ca. 1-1,5 h). Fügen Sie eine weitere identische Probe für nicht-HS Kontrolle. Diese Probe wird genau wie in dem experimentellen jedem einzelnen Aspekt mit Ausnahme des HS behandelt werden. Diese nicht-hitzegeschockten Kultur dient als negative Kontrolle. Das Auftreten vieler Kolonien auf dem nicht-HS Steuerplatte (Schritt 3.13) zeigt gewöhnlich Transformation mit unverdauten P [Pacman] KO 1,0 LA/ RA Plasmid.
- Während die Kultur wächst, laufen die BamHI-restricted P [Pacman]-KO auf einem Gel. Dann Gel zu extrahieren und zu eluieren die DNA in 10 ul Wasser erwärmt bis zu 55 ° C. HINWEIS: Nicht eluieren DNA in einem Puffer. Salze, die in dem Puffer können elektrische Entladung während der Elektroporation zu bewirken.
- Hitze-Schock die Kultur bei 42 ° C für genau 15 Minuten im Wasserbad. Dies aktiviert die Rekombination Maschinen von SW102 Zellen. Nicht erhitzen Schock die Kontrolle Kultur.
- Unmittelbar nach Hitzeschock-, Transfer hitzegeschockten und nicht-HS Kontroll-Kulturen zu gekühlten 50 ml Röhrchen kanonischen und ließ die Kulturen Chill in einem Eis-Wasser-Schlamm für 5 min. Wichtig: Während der folgenden Schritte Zellen müssen bei niedrigen Temperaturen gehalten werden . Dies ist entscheidend für gute Qualität elektrokompetente Zellen zu erhalten.
- Zentrifuge Kultur bei 1.500 xg für 10 min. Überstand verwerfen und waschen Pellet in 25 ml kaltem (4 ° C) 10% Glycerin 13. ZuerstDie Zellen in 5 ml durch Klopfen und Schütteln des Rohres in Eis-Wasser-Schlamm (NICHT Pipette auf und ab) und fügen Sie dann die restlichen 10% Glycerin (20 ml).
- Zentrifuge bei 4 ° C bei 1.500 xg für 10 min, Überstand verwerfen und das Pellet waschen mit 25 ml eiskaltem 10% Glycerin ein zweites Mal.
- Zentrifuge bei 4 ° C bei 1.500 xg für 10 min, Überstand verwerfen und das Pellet waschen mit 25 ml eiskaltem 10% Glycerin ein drittes Mal.
- Zentrifuge bei 4 ° C bei 1.500 xg für 10 min und Überstand verwerfen. Das Zellpellet kann sehr lose an dieser Stelle und sollte darauf geachtet werden, wenn der Überstand verworfen werden. Resuspendieren des Pellets in 1 ml kaltem 10% Glycerin. Übertragen der Zellen in einem 1,5 ml Röhrchen und zentrifugieren für 30 Sekunden bei 12.000 × g in einer Tischplatte Mikrozentrifuge bei 4 ° C. Entsorgen Sie die meisten der Überstand verlassen ≈ 90 l Wasser / Zellen. HINWEIS: An dieser Stelle können Zellen bei -80 ° C für eine spätere Verwendung gespeichert werden. Dies kann jedoch zu verringern Kompetenz.
- In 2 ul der BamHI-verdauten P [Pacman] (Ziel für 50 ng) zu den Zellen und vorsichtig mischen mit einer Pipettenspitze. Übertragen Zellen in einem 1 mm Elektroporationsküvette und elektroporieren bei 1.800 V, 25 uF, 200 Ω.
- In 300 ul SOC und lassen die Zellen für 2 Stunden erholen bei 32 ° C (Schütteln ist nicht erforderlich). Teller Zellen auf LB-Agar plus 50 ug / ml Ampicillin und Inkubation bei 32 ° C für 24-30 Std.
Tag 3
- Bildschirm für die richtige Lücke-Reparatur mit Kolonie-PCR. Verwenden T3 und RA überprüfen Primer für die RA und T7 und LA Check Primer für die LA (Tabelle 1). Entwerfen Sie die PCR-Check-RA und PCR-Check-LA Primer 100-200 bp zur Zielregion (Tabelle 1). Positive Kolonien von PCR-Reaktionen unter Verwendung von PCR-Check-RA mit T3 oder PCR-Check-LA mit T7 zeigt eine Band, die bei 800-1000 bp (Abbildung 3) HINWEIS läuft:. Es ist wichtig, beide Arme seit Einbau einer testen Armaber nicht die andere wird manchmal beobachtet.
- Wachsen PCR-geprüft Kolonien bei 32 ° C in 4 ml LB +50 ug / ml Ampicillin über Nacht. Einen gefrorenen dieser Zellen für die spätere Verwendung.
4. Austauschen der genomischen Region mit dem Targeting-Kassette
Tag 4
- Erhöhen der RFP / KAN Knockout-Kassette durch PCR unter Verwendung pENTR-RFP/KAN (vorverdauten mit AscI und NheI zur Linearisierung) als Vorlage und dem 5'HA-RFP / Kan-F und 3'HA-RFP / Kan-R-Primer ( Tabelle 1). RFP / KAN-Kassette kann aus Referenz 11 angefordert werden.
- Gel reinigt die PCR-Produkt Elution in 20 ul sterilem Wasser. Der RFP-KAN-Kassette sowie die Homologie Arme sollten etwa 3 kb laufen.
- Impfen 1 ml der gesättigten SW102 / P [Pacman]-KO Kultur (Schritt 3.13) in 44 ml LB-Amp (50 ug / ml) und wachsen auf 32 ° C Schüttler bis OD 600 zwischen 0,2-0,3. Fügen Sie eine weitere identische Probe für nicht-HS Kontrolle. Diese Probe wird nur behandelt werdenwie in dem experimentellen jedem einzelnen Aspekt mit Ausnahme des HS.
- Nach dem SW102 / P [Pacman]-KO Kultur erreicht die gewünschte Dichte, folgen Sie den Schritten von 3,5 bis 3,10.
- Elektroporieren etwa 100 ng von Targeting-Kassette in 90 ul elektrokompetente SW102 / P [Pacman]-KO-Zellen. Übertragen Zellen in einem 1 mm Elektroporationsküvette und elektroporieren bei 1.800 V, 25 uF, 200 Ω. In 300 ul SOC und lassen Zellen für 2 Stunden bei 32 ° C erholen (Schütteln ist nicht erforderlich). HINWEIS: Es wird dringend empfohlen, um eine frische Gel gereinigt Kassette verwenden.
- Teller Zellen in LB-Agar + Amp (50 ug / ml) + Kan (50 ug / ml) und Inkubation für 24-30 Stunden bei 32 ° C.
Tag 5
- Wachsen 5 ml Kultur aus 5 verschiedenen Kolonien bei 32 ° CO / N.
Tag 6
- Durchführen einer "dirty Miniprep" und führen Hälfte der erhaltenen DNA auf einem 1% Agarose-Gel für die pr aussehenEsence eines niedermolekularen Plasmid. Es sollte keine Plasmid, das Balg 12 kb läuft sein. (Abbildung 4)
- Machen Sie eine 1:500 Verdünnung der DNA von einem positiven Klon und verwenden Sie 1 ul in 50 ul TransforMax EPI300 elektroporieren. Wichtig: Achten Sie darauf, um die DNA zu verdünnen Elektroporation mehrerer Plasmide in einer Zelle zu verhindern. Teller Zellen in LB-Agar + Amp (50 ug / ml) + Kan (50 ug / ml) und Inkubation für 18-24 Stunden bei 37 ° C
Tag 7
- Impfen eine einzelne Kolonie in 10 ml LB + und wachsen O / N.
Tag 8
- Seed 100 ml LB-Medium mit gesättigten Kultur von Schritt 4.10. In geeigneten Antibiotika plus 100 ul von 1.000 X Kopie Kontroll-Lösung und bei 37 ° C für 5-6 Stunden.
- Durchführen einer maxiprep und überprüfen Einsetzen der Kassette in der richtigen Stelle durch Sequenzierung.
- Neben Sequenzierung, führen eine Beschränkung Enzyme Verdauung Test mit DNA aus einem Klon, nicht enthielt jede niedermolekulare Plasmid DNA und ihre Eltern. Die Restriktionsenzyme ausgewählt wird anders sein für jedes Gen, aber das Ziel ist es, eine Gruppe von Enzymen, die für den Forscher die recombineered Vektor charakterisieren lassen zu finden. Beispielsweise wurde die CG32095 Targeting-Vektor seriell mit PacI, AscI, BamHI und AatII verdaut. Das Erscheinungsbild aller vorhergesagten Bands und das Fehlen von falschen Bands bestätigt, dass die Targeting-Vektor wurde korrekt recombineered (Abbildung 5).
5. Spritzen Flies und Mobilisierung Cassette In Vivo
- Injizieren Sie die Cassette über ΦC31 Integrase, in einer vordefinierten Landeplatz der Wahl. Außerhalb Anbieter wie Regenbogen Transgenics ( http://www.rainbowgene.com ), kann zur Injektion Dienstleistungen verwendet werden. Wichtig: Die DNA muss frisch sein prepared vor der Injektion. Höhere Transformationseffizienz wird beobachtet, wenn die DNA vor Ort hergestellt wird, injiziert und die am selben Tag.
- Um die Kassette aus dem Landeplatz mobilisieren, verwenden Bloomington Bestellnummer 25680 oder 25679. Diese Bestände enthalten Flippase und I-SceI stromabwärts eines Hitzeschock-Promotor, auf Chromosom zwei bzw. drei. Darüber hinaus enthalten sie eine versteckte hs-Transgen auf dem Balancer-Chromosom und auf dem Y-Chromosom. Sammle Männchen vom gewählten Lager und überqueren jungfräulichen Weibchen tragen die KO-Kassette in den Landeplatz. . Set ~ 30 Kreuze und erlauben Weibchen Eier für 2-3 Tage lag HINWEIS: Es wird empfohlen, die Aktie, die auf einem anderen Chromosom vom anvisierten Ort zu wählen.
- Flip Eltern in eine neue Reihe von 30 Ampullen und Hitzeschock-Larven bei 37 ° C für 1 Stunde, zweimal am Tag, an drei aufeinanderfolgenden Tagen. Die Wärme Schocks aktivieren die Produktion der Enzyme; Flippase und I-SceI und die Zelltod-Gen versteckte , Töten die Männchen und Fliegen trägt Balancer Chromosomen. Lassen Sie die Eltern in den neu blätterte Fläschchen, um Eier für 2-3 Tage zu legen, dann wiederholen Sie die Wärme Schocks für die Fläschchen, nach Umklappen der Eltern in neue Gefäße wieder. Wiederholen Sie diesen Vorgang für mehrere Flips, bis die Eltern nicht mehr Eier legen.
- Sammle jungfräulichen Weibchen aus der F1 Nachkommen der hitzegeschockten Fliegen und überqueren y - w - Männer. Ziel für 150-200 Kreuze, drei Jungfrauen und drei Männern pro Kreuz.
- Bildschirm Der F2 Nachkommen unter einem Fluoreszenz-Bereich, der Nachweis von RFP im Auge ermöglicht. Schirm für RFP positive Augen, die Mutante für die weißen und gelben Gene (y - w -) sind. Dieses Screening positiv wählt für die Anwesenheit des RFP-Kassette und wählt für das Fehlen des P [Pacman] Vektor (Mini-weiß) und Landeplatz, der von der Wildtyp-Gen HINWEIS gelb markiert ist:. Es ist weniger Zeit -conSuming zum Screening auf RFP in die Augen, als zum ersten Bildschirm für yw Fliegen. Die Mobilisierung Veranstaltungen sind sehr effizient bei der Hitze Schocks, was in vielen weniger Fliegen mit RFP in die Augen, um y verglichen - w - Fliegen.
- Einrichten einziges Paar Paarungen mit jeweils y - w - RFP + fly, da jede Mobilisierung Veranstaltung unterschiedlich sein können. Ordnen Sie den RFP an die richtige Chromosom. Korrigieren Chromosom Targeting wird typischerweise in> 95% der Fälle beobachtet.
- Stellen Aktien und korrekte Targeting Ereignisse unter Verwendung von Standardverfahren für die Southern-Blot-Analyse. Entwerfen Sie ein 2 kb-Sonde vor (linker Arm) und stromabwärts (rechter Arm) des Gens, das gezielt und 2 kb (oder kürzer, wenn nötig) überspannt den ORF gelöscht. Die ersten beiden Sonden wird eine Band in den homozygoten und heterozygoten Knockouts ergeben, das ist nicht in der Wildtyp, während der ORF nicht nachgeben wird ein Band in der homozygoten Knockout. Eine schnelle PCR foder der ORF auch möglich ist, aber dies beruht auf der Abwesenheit eines Bandes.
Ergebnisse
Verstärkung des LA und RA Homologie Arme sollten produzieren 500 bp Produkte und die PCR-Reaktion sollte SOE eine 1,0 kb Produkt (Abschnitte 2.1-2.4; Abbildung 2) zu erhalten. Die BP-Reaktion in Abschnitt 2.5 durchgeführt wird, ist in der Regel sehr effizient und bakterielle Transformation der Ausbeuten 5-100 Kolonien im Durchschnitt. Fast alle Kolonien mit PCR getestet überprüfen die erwartete PCR-Produkt.
In der ersten Runde der Rekombination (§ 3) erwarten, 20-40 Kolonien nach der Transformation der verdauten P bekommen [Pacman]-KO-1.0 mit dem LA und RA Arme in SW102 Zellen. 40-60% dieser Klone tragen Sie die gewünschte Rekombination Produkt. Die Nicht-Hitzeschock-Kontrolle sollte enthalten sehr wenige oder gar keine Kolonien wie den hitzegeschockten Zellen verglichen. Das Aussehen der gleichen Anzahl der Kolonien auf den beiden Platten der Proben, Kontrollen in der Regel die Anwesenheit von ungeschnittenen Plasmid oder eine andere Verunreinigung. In diesem Fall wird das Experiment should verworfen und wiederholt werden, diesmal Verdauen der P [Pacman]-KO-1.0 Durchführung der LA und RA Arme bis zur Fertigstellung nach der Anweisung in Schritt 3.2 beschrieben. Ein Beispiel eines PCR-Check für Abruf von genomischen Region von Interesse in P [Pacman]-KO, in dem 1,0 ≈ 60 Kolonien erhalten, ist in 3 gezeigt. Hier sind 60% der getesteten Kolonien führte die gewünschte Rekombination product.Care getroffen werden müssen, dass die beobachteten PCR-Produkte bei der vorhergesagten Größe ausgeführt, da aberranten PCR-Banden in der Regel korrekt Recombineering Ereignisse zeigen.
Gelegentlich keine Kolonien wird nach der ersten Runde der Rekombination wachsen. Mit Zellen mit sub-optimalen Wirkungsgrad stellt die häufigste Ursache für eine fehlgeschlagene Recombineering Reaktion. Keeping die Zellen kalt jederzeit während ihrer Herstellung und schonende Handhabung während der Waschungen (NIE Vortex) ist von entscheidender Bedeutung für den Erhalt von hochwertigen zuständigen Zellen. Versuchen Sie, immer frisch verdaut / gereinigt P [Pacman]-KO-1.0 DNA, um höchste Effizienz zu erhalten.
Manchmal ist die erste Runde der Rekombination Ausbeuten Kolonien, sondern diejenigen Kolonien nicht bestehen, die PCR-Assay Scheck. Dies weist normalerweise auf falsche Recombineering Produkte, sondern ein solches Ergebnis kann auch bedeuten, potenzielle Probleme mit einem der Primer-Sets. Um die PCR-Check-LA Primer validieren, eine PCR-Reaktion in Kombination mit dem attB1-I-SceI-LA-F gesetzt. Um zu überprüfen, PCR-check-RA Einrichten einer PCR-Reaktion mit dem attB2-I-SceI-RA-R. In beiden Fällen können Sie die Original-BAC als Vorlage. Diese PCR-Reaktionen sollten ergeben Produkte der erwarteten Größe, die je nach der Reihenfolge der Scheck Primer. Positive Ergebnisse aus dieser Kontrolle Experiment wird die Möglichkeit auszuschließen, dass die Unfähigkeit, positive Klone erkennen durch Primer Ineffizienz ist.
Wenn Sie versucht haben die erste Runde der Rekombination mehrmals nach dem obigen Protokoll ohne Erfolg durchgeführt haben und die ganzevorgeschlagenen Kontrollen, empfehlen wir die Wahl eines neuen Paares von 500 bp Homologie Arme. Aus Gründen, die nicht klar sind in der heutigen Zeit sind einige genomischen Regionen sehr widerstandsfähig gegen DNA-Rekombination. Einfach Konzeption neuer Homologie Arme und bewegte sie hin zu oder weg von dem Gen von Interesse manchmal löst diese Probleme.
Die zweite Runde der Rekombination (Abschnitt 4) ist in der Regel sehr viel effizienter. Normalerweise 30-50 Kolonien werden nach der Transformation beobachtet, mit 90-95% von ihnen, die das gewünschte Konstrukt. Das häufigste Problem bei diesem Schritt ist der Erhalt falsch-positive Kolonien. Abbildung 4 zeigt Beispiele von wahr und falsch positive Klone nach der zweiten Runde der Rekombination (Abschnitt 4).
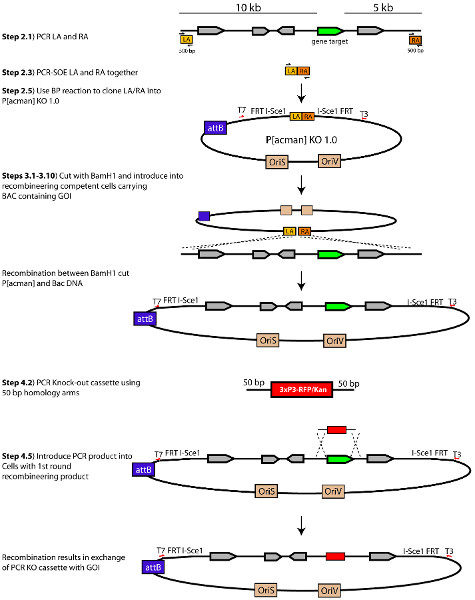
Abbildung 1. Schematische Darstellung des process zur Erzeugung eines P [Pacman]-KO 1.0 Targeting-Vektor. Geändert von Chan et al. (2012). Klicken Sie hier, um eine größere Abbildung anzuzeigen .

Abbildung 2. Schematische Darstellung des Spleißen durch überlappende PCR (PCR SOE) auf Schritt 2.3 durchgeführt. Klicke hier, um eine größere Abbildung anzuzeigen .
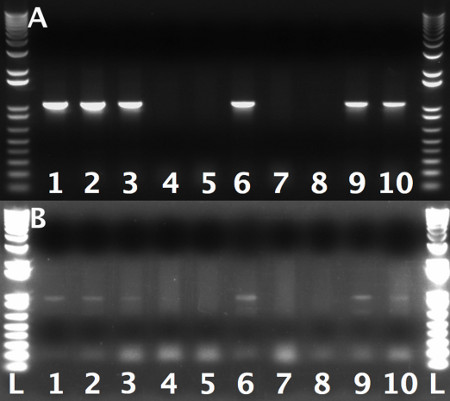
Abbildung 3. PCR-Check Gel zeigt Effizienz der ersten Veranstaltung Recombineering Jede Spur stellt eine einzelne Kolonie A:.. Kolonien für den linken Arm getestet. Integration unter Verwendung von T7 und LA-Check Primer B: Die gleichen Kolonien A, für den rechten Arm Integration mit T3 und RA-Plus-Check primers.1kb DNA Ladder wurde als Molekulargewichtsmarker verwendet HINWEIS getestet:. Aufgrund der schlechten Grundierung von T3 Primer gegenüber T7, waren die Bänder des rechten Armes Scheck deutlich dunkler. Dies sollte berücksichtigt werden, wenn sich das Gel.

Abbildung 4. DNA-Agarosegel zeigt hochmolekulare Plasmide (P [Pacman]) und niedermolekulare Plasmid (unverdaute Vorlage) von potenziellen positiven Klone nach der zweiten Runde der Rekombination isoliert. DNA wurde isoliert von 4 möglichen positiven Klone und lief auf einem 1% Agarosegel mit Ethidiumbromid gefärbt. Klone 1,3 und 4 sind echte positive Klone. Klon 2 ist eine falsche positive Einmaleffekte wie die alle Formen von ap beachten lasmid das läuft Balg 12 kb (die größte Größe Band in DNA-Leiter Spur). 1 kb plus DNA Ladder wurde als Molekulargewichtsmarker verwendet.

Abbildung 5. Test zur gewünschten Veranstaltung durch Rekombination Restriktionsenzymverdau. Im obigen Beispiel wird gezeigt, ein Computer-vorhergesagt Bandenmuster des P [Pacman]-KO CG32095 vor und nach der zweiten Runde der Rekombination. Vier verschiedene Enzyme wurden verwendet: Pacl, AscI, AatII und BamHI. Korrekt rekombinierten Produkt die gleichen Bands wie seine Eltern P [Pacman]-KO CG32095 Ausnahme der beabsichtigten Änderung (Pfeilspitzen) haben. NEBcutter V2.0 wurde verwendet, um das Band Muster vorherzusagen.
0346/50346fig6highres.jpg "src =" / files/ftp_upload/50346/50346fig6.jpg "/>
Abbildung 6. Schematische Beschreibung der Mobilisierung eines Targeting-Kassette in vivo. Klicke hier, um eine größere Abbildung anzuzeigen .

Tabelle 1. Grundierungen. Klicke hier, um eine größere Tabelle anzuzeigen .
Diskussion
Die Macht der genetischen Modellorganismen in der biomedizinischen Forschung wird weitgehend auf den verfügbaren Werkzeugen zur genetischen Manipulation basiert. Die kleinen Modelle C elegans und Drosophila insbesondere ermöglichen kostengünstige und schnelle molekulargenetische Analysen von kompletten Wege und Gen-Familien in vielzelligen Entwicklung oder Funktion gebracht. In den letzten Jahren bedeutende Fortschritte in der Entwicklung von Werkzeugen für die Manipulation von Genen in Drosophila 14,15 gesehen. Zum Beispiel wurde Rekombination, die weit in Mausgenetik wird Bac DNA-Konstrukte zu manipulieren, die vor kurzem für Drosophila angepasst, durch die Verwendung des P [Pacman]-Vektorpartikel und ΦC31-vermittelten Gentransfer 6. Verschiedene Selektionskassetten ermöglichen die Insertion oder Deletion von Sequenzen einer beliebigen Stelle innerhalb eines Konstrukts mit rekombinationskompetenten Bakterien 4,13. Wir haben das Original modifiziert P [Pacman] Vektor, so dass es für den Bau verwendet werden können In vivo homologen Rekombination Vektoren, die endogenen Loci in Drosophila Ziel. Dieser neue Vektor ermöglicht einem, Targeting Kassetten mit größeren Homologiearme als diejenigen, die einfach gemacht werden kann mit Ausschneiden und Einfügen Klonierungsmethoden erzeugen. Darüber hinaus wurde ein Gateway Klonierungskassette auch in diesen Vektor eingebaut, so dass man schnell und effizient einzuführen neue genomische DNA in das System.
Obwohl die meisten der hier beschriebenen Verfahren ist einfach, es gibt mehrere Schritte, die wir gefunden entscheidend für den Erfolg des Verfahrens haben. Aufgrund der Größe des manipulierten DNA (> 15 kb), stellt die erste Runde der Rekombination Reaktion (Abschnitt 3) eine signifikante technische Hürde. Daher erfordert diese Runde der Rekombination eine sehr effiziente Rekombination Protokoll. Scheinbar geringfügige Anpassungen Protokoll, wie Wachsen der Zellen zu niedriger Dichte, Waschen der Zellen dreimal anstelle von zwei und mit 10% Glycerin an Stelle von dH 2 </ Sub> O 13, haben stark die Transformation Effizienz unserer Recombineering kompetenten Zellen verbessert. Darüber hinaus kümmert sich sicherstellen, dass die LA / RA mit P [Pacman] ist vollständig mit BamHI (Schritt 3.1) beseitigt viele Fehlalarme geschnitten.
Unerwünschte Ereignisse stellen eine weitere Rekombination recht häufiges Problem während Rekombination. Zum Beispiel haben wir Fälle von Doppelarbeit und Translokationen innerhalb verschiedener Konstrukte beobachtet. Diese Ereignisse können auf mehrere Probleme bei Drosophila Transformation und in vivo Gen-Targeting führen. PCR und Sequenzierung Analyse Überprüfung kann nicht erkennen, alle diese Ereignisse. Daher muss eine Restriktionsanalyse auf die endgültige kloniert Produkt vor der Injektion von DNA in Embryonen durchgeführt werden. Diese Endkontrolle hat sich kritisch und hat uns erlaubt, zu vermeiden Einspritzen Konstrukte, die einen unerwünschten Abweichungen enthalten.
Maxi-prepping die Targeting-Vektor kurz vor injection und nie das Einfrieren der DNA verbessert die Transformation Effizienz dieser großen Konstrukte in Drosophila. Kurz gesagt, machen kleine Details einen großen Unterschied in den Erfolg dieser Techniken. Die Protokolle skizziert und gezeigt, hier stellen Erkenntnisse aus einer Reihe von verschiedenen Quellen und eigener Erfahrung.
Während hoffe, dass wir das Protokoll hier hilft anderen Recombineering Methoden in ihren eigenen Labors annehmen, glauben wir, weitere Verbesserungen und Verfeinerungen der Methode sind noch möglich. Gelegentlich sind bestimmte genomische Fragmente schwierig zu handhaben mittels Rekombination aus Gründen, die nicht vollständig klar. Darüber hinaus trotz unserer Bemühungen um unerwünschte Hintergrundgeräusche zu beseitigen, haben wir noch feststellen, dass bestimmte Teile der genomischen DNA zu falsch recombineered Endprodukte produzieren neigen. Ein tieferes Verständnis der Rekombination Prozess kann mehrere Protokolle optimal in die Zukunft und offene Diskussion ergebenRecombineering Misserfolge und Erfolge werden fördern den erfolgreichen Einsatz dieser leistungsfähigen Techniken, die von der breiteren Forschungsgemeinschaft.
Offenlegungen
Die Autoren haben keine konkurrierenden Interessen in Bezug auf den hier skizzierten Techniken.
Danksagungen
Wir möchten Hugo Bellen und Bloomington Stock Center für Reagenzien danken. Wir danken Koen weiter Venken, Hugo Bellen und alle Mitglieder der Buszczak und Hiesinger Labors für hilfreiche Diskussionen. Diese Arbeit wurde durch Zuschüsse aus dem National Institute of Health in ACR (T32GM083831), PRH (RO1EY018884) und MB (RO1GM086647), einen Zuschuss von der Cancer Prevention Research Institute of Texas in MB und PRH (RP100516) und der Welch unterstützt Foundation (I-1657), um PRH. MB ist ein EE und Greer Garson Fogelson Scholar in der biomedizinischen Forschung und PRH ist ein Eugene McDermott in Biomedical Research Scholar am UT Southwestern Medical Center.
Materialien
| Name | Company | Catalog Number | Comments |
| Name of the reagent | Company | Catalogue number | Comments (optional) |
| SW102 Recombination competent bacteria | NCI-Frederick | Recombination Bacteria (SW102, SW105 and SW106) | http://ncifrederick.cancer.gov/research/brb/logon.aspx |
| TransforMax EPI300 electrocopmpetent E. coli | Epicentre | EC300110 | Includes CopyControl induction solution |
| PfuUltra II Fusion HS DNA Polymerase | Aligent Technology Inc. | 600670 | |
| BamHI-HF | New England Biolabs | R3136S | |
| Zymoclean Gel DNA Recovery Kit | Zymo Research | D4001 | |
| Use Gateway BP ClonaseII Enzyme kit | Invitrogen | 11789-020 | |
| P[acman]KO1.010 | Buszczak and Hiesinger Labs | Upon request | |
| pENTR RFP-Kan11 | Buszczak and Hiesinger Labs | Upon request | |
| Flystocks | Bloomington stock center | Stock numbers: 25680, 25679 | y1 w*/Dp(2;Y)G, P{hs-hid}Y; P{70FLP}11 P{70I-SceI}2B snaSco/CyO, P{hs-hid}4 y1 w*/Dp(2;Y)G, P{hs-hid}Y; P{70FLP}23 P{70I-SceI}4A/TM3, P{hs-hid}14, Sb1 |
| Electroporation machine | Biorad GenePulser Xcell with PC module | 165-2662 | |
| Cuvettes | Fisher Brand | #FB101 |
Referenzen
- Rong, Y. S., Golic, K. G. A targeted gene knockout in Drosophila. Genetics. 157, 1307-1312 (2001).
- Rong, Y. S., Golic, K. G. Gene targeting by homologous recombination in Drosophila. Science. 288, 2013-2018 (2000).
- Gong, W. J., Golic, K. G. Ends-out, or replacement, gene targeting in Drosophila. Proc. Natl. Acad. Sci. U.S.A. 100, 2556-2561 (1073).
- Warming, S., Costantino, N., Court, D. L., Jenkins, N. A., Copeland, N. G. Simple and highly efficient BAC recombineering using galK selection. Nucleic Acids Res. 33, e36 (2005).
- Copeland, N. G., Jenkins, N. A., Court, D. L. Recombineering: a powerful new tool for mouse functional genomics. Nat. Rev. Genet. 2, 769-779 (2001).
- Venken, K. J., He, Y., Hoskins, R. A., Bellen, H. J. P[acman]: a BAC transgenic platform for targeted insertion of large DNA fragments in D. melanogaster. Science. 314, 1747-1751 (2006).
- Venken, K. J., et al. Versatile P[acman] BAC libraries for transgenesis studies in Drosophila melanogaster. Nat. Methods. 6, 431-434 (2009).
- Groth, A. C., Fish, M., Nusse, R., Calos, M. P. Construction of transgenic Drosophila by using the site-specific integrase from phage phiC31. Genetics. 166, 1775-1782 (2004).
- Bischof, J., Maeda, R. K., Hediger, M., Karch, F., Basler, K. An optimized transgenesis system for Drosophila using germ-line-specific phiC31 integrases. Proceedings of the National Academy of Sciences of the United States of America. 104, 3312-3317 (2007).
- Chan, C. C., et al. Systematic discovery of Rab GTPases with synaptic functions in Drosophila. Current Biology: CB. 21, 1704-1715 (2011).
- Chan, C. C., Scoggin, S., Hiesinger, P. R., Buszczak, M. Combining recombineering and ends-out homologous recombination to systematically characterize Drosophila gene families: Rab GTPases as a case study. Commun. Integr. Biol. 5, 179-183 (2012).
- Wu, N., Matand, K., Kebede, B., Acquaah, G., Williams, S. Enhancing DNA electrotransformation efficiency in Escherichia coli DH10B electrocompetent cells. Electronic Journal of Biotechnology. 13, (2010).
- Wang, S., Zhao, Y., Leiby, M., Zhu, J. A new positive/negative selection scheme for precise BAC recombineering. Mol. Biotechnol. 42, 110-116 (2009).
- Venken, K. J., Bellen, H. J. Transgenesis upgrades for Drosophila melanogaster. Development. 134, 3571-3584 (2007).
- Dietzl, G., et al. A genome-wide transgenic RNAi library for conditional gene inactivation in Drosophila. Nature. 448, 151-156 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten