Method Article
Mapeamento bacterianas redes funcionais e vias
Neste Artigo
Resumo
Sistemáticas e em grande escala genético sintético telas (gene-gene ou epistasia) interação pode ser usado para explorar a redundância genética e via de cross-talk. Aqui, descrevemos um alto rendimento quantitativo tecnologia de rastreamento genético sintético matriz, denominada eSGA que desenvolvemos para a elucidação de relações epistáticos e explorar redes de interação genéticos em Escherichia coli.
Resumo
Fenótipos são determinados por uma série complexa de física (por exemplo, proteína-proteína) e funcionais (por exemplo, gene-gene ou genética) interacções (GI) 1. Enquanto interações físicas podem indicar que as proteínas bacterianas estão associadas como complexos, eles não revelam necessariamente via de nível relationships1 funcionais. Telas de GI, em que o crescimento de mutantes duplos tendo dois genes eliminados ou inactivados é medida e comparada com os mutantes correspondentes individuais, pode iluminar dependências epistáticos entre loci e, portanto, proporcionam um meio para consultar e descobrir novas relações funcionais 2. Mapas de grande escala GI foram relatados por organismos eucarióticos como o fermento 3-7, mas a informação GI permanece escassa para procariontes 8, o que dificulta a anotação funcional de genomas bacterianos. Para este fim, e outros desenvolveram alto rendimento quantitativo bacterianas métodos de rastreio de IG 9, 10 Aqui, apresentamos os principais passos necessários para realizar E. quantitativa coli Genetic Matriz Sintética procedimento de triagem (eSGA) em um genoma de escala-9, utilizando-se a conjugação bacteriana natural e recombinação homóloga para gerar sistematicamente e medir a aptidão de um grande número de mutantes duplos em um formato de matriz de colónia. Resumidamente, um robô é usado para transferir , através de conjugação, cloranfenicol (Cm) - marcado alelos mutantes de Hfr engenharia (alta freqüência de recombinação) 'linhagens doadoras em um conjunto ordenado de canamicina (Kan) - marcadas F-receptor cepas. Normalmente, usamos a perda de função de mutantes simples tendo deleções de genes não essenciais (por exemplo, a recolha de 'Keio' 11) e as mutações de genes essenciais hypomorphic (isto é, os alelos que conferem a expressão da proteína reduzida, a estabilidade, ou a actividade de 9, 12, 13) para consultar as associações funcionais de genes essenciais e não essenciais, resvamente. Após a conjugação e subsequente troca genética mediada por recombinação homóloga, os duplos mutantes resultantes são seleccionadas em meio sólido contendo ambos os antibióticos. Depois de conseqüência, as placas são digitalmente fotografada e tamanhos colônia são quantitativamente registados com um sistema interno de tratamento automatizado de imagem 14. IG são reveladas quando a taxa de crescimento de um mutante duplo ou é significativamente melhor ou pior do que 9 esperada. SIG (ou negativo) agravante muitas vezes resultam entre a perda de função de mutações em pares de genes de vias compensatórias que incidem sobre o mesmo processo essencial 2. Aqui, a perda de um único gene é tamponada, tal que um ou outro mutante único é viável. No entanto, a perda de ambas as vias é prejudicial e resulta em letalidade sintética ou de doença (isto é, crescimento lento). Inversamente, aliviando (ou positivos) podem ocorrer interacções entre os genes na mesma via ou a proteína do complexo 2, como odeleção de um ou outro gene sozinho é frequentemente suficiente para perturbar o funcionamento normal da via ou complexo de tal forma que as perturbações adicionais não reduzem a actividade, e, consequentemente, o crescimento adicional. No geral, a identificação sistemática e análise de redes de GI pode fornecer imparciais, mapas globais de relações funcionais entre um grande número de genes, dos quais via de informações em nível de perdida por outras abordagens podem ser inferidas 9.
Protocolo
1. Construção de cepas mutantes HFR Doadores Cavalli por Recombineering 15, 16
Os passos para a construção dos corantes doadores eSGA são descritos abaixo. Resumidamente, usamos λ alvo - Red recombinação homóloga mediada 16 da cassete seleccionável amplificado de ADN marcador de fragmentos gerados por PCR, para criar não essenciais mutantes de deleção de genes (secção 1.1), ou de genes essenciais hypomorphic estirpes dadoras mutante (secção 1.2), que são então utilizados como "consultas" para definir redes GI.
Nota: Durante o processo de desenvolvimento de tecnologia, que avaliou a eficácia do uso de Hfr transferência mediada conjugativo para combinar mutações usando uma bem definida cepa doadora Hfr (Hfr Cavalli, Hfr Hayes, e Hfr 3000). Foram examinados (i) a capacidade para eficientemente fazer mutantes de doadores utilizando o método recombineering iniciada por Yu et al. (2000) 16, (ii) as eficiências relativasde transferência de DNA conjugativo de diferentes marcadores cromossómicos, e (iii) os efeitos de posição e orientação do gene cromossómico de consulta relativamente ao locus de transferência Hfr, oriT. Descobrimos que λ - Red mediada eficiência de recombinação homóloga foi muito maior no Hfr Cavalli que em Hfr Hayes ou Hfr3000. Se necessário, a transdução P1 ou uma estirpe Hfr Psuedo 17, pode ser usado para criar mutantes doadores. Conseguimos adaptado todos estes métodos para a produção e rastreio de grandes números de doadores. Uma vez que nas nossas experiências de conjugação de ensaio, a eficiência global da transferência e o número de ex-conjugantes observada foi significativamente maior com Hfr Cavalli, este fundo estirpe particular foi escolhido para a construção de doador e de grande escala eSGA. Todos os procedimentos para telas eSGA utilizando doadores HFR Cavalli são descritos.
- Amplificar o fragmento de DNA para posterior recombineering para eliminar um gene não essencial
Empregar dois passo-nested PCR amplicação (Figura 1) para criar uma cassete de deleção do gene alvo para a substituição do quadro de leitura aberto com um marcador de resistência a Cm do plasmídeo pKD3 (GI: 15554330; proteína ID: AAL02033.1) 15. O marcador é flanqueado pelo FRT-11 locais, permitindo a remoção do gene de resistência, se necessário, no seguimento experimentos.
Nota: se aninhada por PCR de dois passos para remover o plasmídeo a partir do produto amplificado que é usada posteriormente para transformar as células bacterianas. Removendo o plasmídeo (na primeira PCR) e, em seguida, criando a cassete de gene knockout na segunda PCR é necessário porque a transformação com um plasmídeo que é muito mais eficiente do que recombineering, e o objectivo é obter - tanto quanto possível - apenas as cepas que têm substituído com sucesso o ADN genómico alvo com o marcador seleccionável. Uma alternativa para a PCR aninhada é realizar uma digestão de restrição do plasmídeo fora da região amplificada na segunda PCRpasso. Em seguida, o produto da digestão de restrição é purificado e utilizado como molde na fase de PCR interno (1.1.2). Esta opção também é simples, mas requer mais trabalho o tempo.- Usar cerca de 45 ng de pKD3 como um molde em PCR com forward e reverse primers F1 R1 para produzir um produto de 1070 pb. Purifica-se o fragmento de PCR utilizando o protocolo de purificação da Qiagen PCR, e dilui-se o produto em água destilada estéril a 5 ng / uL de concentração.
- Utilizando o produto de PCR purificado como um modelo, criar uma segunda PCR com gene-específicos (por exemplo knockout) iniciadores encaixados, F2 e R2, que possuem (i) nt 45 de regiões de genes específicos de homologia no 5'-termina a permitir por recombinação homóloga, imediatamente a montante e a jusante do gene alvo para substituí-la com o marcador de resistência a Cm e (ii) 20 nt nas extremidades 3 'para iniciar a síntese do marcador de Cm. O tamanho do fragmento de 1123 pb é produzido. As regiões de homologia de genes específicos foram concebidos para eliminar o alvo open quadro de leitura, deixando todos os segmentos adjacentes ou parcialmente sobrepostos codificação intacta. Após esta amplificação, vá para o passo 1.3.
- Amplificação de uma cassete de marcação para a criação de uma mutação no gene essencial hypomorphic
- Para construir cepas mutantes doadores hypomorphic, adicione um C-terminal peptídeo seqüencial afinidade tag de fusão (SPA), que muitas vezes um pouco afeta a função da proteína essencial pela abundância transcrição desestabilizador (ou seja, do número de cópias de proteína) ou dobramento de proteínas / atividade. Para este fim, criar um modelo de DNA seleccionável SPA-tag (SPA-Cm) por conversão do marcador de resistência Kan pJL148 em 18 a Cm usando recombineering idênticos entre as sequências nucleotídicas que flanqueiam o marcador Kan em pJL148 e marcador Cm pKD3 em 15. O modelo resultante consiste em frame-SPA-tag, seguido por um marcador de resistência a Cm, e é adequado para amplificação por PCR, eSGA recombineering e posterior 12.
Nota: </ Strong> genes essenciais são, potencialmente, a mais interessante para estudos da função do gene. Entretanto, como esses genes não podem ser apagados, métodos inovadores teve que ser desenvolvido para investigar as relações funcionais de genes essenciais. Várias abordagens adequadas altamente bem sucedidos foram desenvolvidos inicialmente em levedura. Por exemplo, Davierwala et al. (2005) 19 realizadas GI telas utilizando um painel de 575 mutantes sensíveis à temperatura de genes essenciais, e encontraram que os genes essenciais exibiu cerca de cinco vezes o número de IG como genes não essenciais. Da mesma forma, Breslow et ai. (2008) 20 desenvolveu uma "perturbação abundância diminuiu mRNA '(DAMP) abordagem para gerar uma biblioteca de alelos hypomorphic cobrindo ~ 82% de genes de leveduras essenciais para estudar vias e complexos. A abordagem húmido modifica a extremidade 3 'do ARNm expresso, a redução do nível do gene alvo 6, provavelmente devido ao mRNA de desestabilização. Semelhante à abordagem úmido 6, nosso strategy tem sido o de explorar os efeitos subtis de perturbar a extremidade 3 'do mRNA expresso de proteínas essenciais.
Como descrevemos a seguir, a etiqueta, por si só não é susceptível de afectar o dobramento de proteínas ou de função, mas a perturbação subtil do mRNA pela etiqueta é suficiente, quando combinada com a tensão ambiental ou outras mutações, para revelar funcionalmente informativo gene-ambiente 13 e do gene gene-9, 12 interacções.
Em primeiro lugar, até o momento, foi utilizado SPA-tags para purificar ~ 3.000 essenciais e não-essenciais proteínas de E. coli 21-23, com mais de 80% dos 307 E. coli proteínas essenciais sendo representados entre nossas tensões SPA-8 com a tag. Dado que a etiqueta foi utilizada com sucesso para isolar complexos de proteínas conhecidas, que foram mutuamente validados (ou seja, o mesmo complexo pode ser isolado por meio de marcação e purificação de proteínas a partir de diferentes dentro do mesmo complexo), e os complexos isolados eram biologicamente relevante, razão pela qual as etiquetas fazergeralmente não afetam o dobramento de proteínas ou função 12. Da mesma forma, nossas observações não publicadas revelou que a marca por si só, não costuma afetar o crescimento tensão. No entanto, concluiu que alterando a 3'-UTR, integrando a etiqueta e a cassete de marcador, tal como foi relatado para as estirpes húmido 6, pode desestabilizar transcrições determinadas ao nível de ARN, ou, em casos raros, impedir a dobragem correcta ou a função do As proteínas de fusão. Assim, previmos que a combinação de tais perturbado alelos de genes essenciais com outras mutações funcionalmente relevantes em outras partes do genoma que resultaria em IG informativo, que de facto foi observado 9, 12. Alelos Além disso, SPA-etiquetados de genes essenciais exibiram interações gene-ambiente (por exemplo, hipersensibilidade a fármacos) de acordo com o trabalho de caracterização fenotípica por Nichols et ai. (2011) 13, que sujeita um subconjunto de SPA-tagged estirpes essenciais para uma variedade de condições de crescimento. O mesmo estudo confirmed que a maioria (54 de 58 testados) SPA-marcado cepas essenciais mostrou aptidão prejudicada em uma ou mais condições de cultura 13, que apoia a noção de que o SPA-tag muitas vezes cria leves alelos hypomorphic em genes essenciais que são úteis para genome-wide aplicativos de tela GI incluindo eSGA. - Amplificar a cassete de marcação a partir do modelo SPA-Cm utilizando primers específicos de gene de S1 e S2, cada uma contendo (i) uma região de 45 nt do gene específica para o homólogo recombineering na extremidade 5 ', seguido por (ii) um 27 pb SPA- sequência específica cm na extremidade 3 '.
- Para construir cepas mutantes doadores hypomorphic, adicione um C-terminal peptídeo seqüencial afinidade tag de fusão (SPA), que muitas vezes um pouco afeta a função da proteína essencial pela abundância transcrição desestabilizador (ou seja, do número de cópias de proteína) ou dobramento de proteínas / atividade. Para este fim, criar um modelo de DNA seleccionável SPA-tag (SPA-Cm) por conversão do marcador de resistência Kan pJL148 em 18 a Cm usando recombineering idênticos entre as sequências nucleotídicas que flanqueiam o marcador Kan em pJL148 e marcador Cm pKD3 em 15. O modelo resultante consiste em frame-SPA-tag, seguido por um marcador de resistência a Cm, e é adequado para amplificação por PCR, eSGA recombineering e posterior 12.
- Confirmando e purificação do produto de PCR
- Confirmar que a cassete de marcação tenha sido correctamente amplificado sujeitando 2-5 ul do produto de PCR a electroforese em gel de agarose e purificação do ADN restante seguindo o protocolo padrão do fabricante de purificação de PCR (Qiagen). Eluir o produto de PCR em 30 ul de água destilada estéril e diluída com ~ 50 ng / ul concentração. A purificaçãoed produto pode ser usado imediatamente em transformação (passo 1.5), ou armazenado a -20 ° C durante até 6 meses, até à utilização.
- Preparando células HFR Cavalli competentes para a construção de doadores
- Inocular um ml da cultura saturada durante a noite de uma estirpe Hfr Cavalli em 70 ml de fresco de Luria-Bertani (LB) com 35 ul de 50 ug / ml de ampicilina, num balão de 250 ml. Incubar a cultura a 32 ° C com agitação a 220 rpm até uma densidade óptica (OD) de 600 nm de ~ 0,5-0,6 é obtido (~ 2 h).
- Transferir a cultura para um banho de água para a indução de calor do sistema de recombinação λ vermelho 16 a 42 ° C durante 15 minutos com agitação a 160 rpm. Parar a indução através da transferência da cultura para um banho de água gelada gelo lama para 10-20 min a 160 rpm. Certifique-se de manter as células a frio a partir deste ponto até que após transformação, tubos de uso e tinas que foram arrefecidas em gelo durante pelo menos 15 min antes da utilização.
- Divida o cultura igualmente em duas pré-refrigerada 50 tubos de polipropileno ml (~ 35 ml de cultura por cada tubo) e centrifugar a 4400 g durante 6 min a 4 ° C.
- Descartar o sobrenadante e ressuspender o pellet celular por inversão suave em ~ 50 ml de gelo-água fria destilada e esterilizada. Centrifugar as células novamente a 4.400 xg durante 6 min a 4 ° C. Descartar o sobrenadante e ressuspender o pellet em cada célula 20 ml de gelado de glicerol estéril a 10% e centrifugar novamente, como descrito acima.
- Decantar o sobrenadante e ressuspender o sedimento de células em 500 ul de glicerol gelado a 10%. Alíquota das células preparadas competentes em volumes de 50 ul em cada pré-resfriada, de 1,5 ml de centrífuga de micro-tubos para transformação imediata. As células podem ser congeladas em gelo seco ou em azoto líquido e armazenado a -80 ° C durante até 3 meses. No entanto, a eficiência de transformação é mais elevada com as células competentes preparadas de fresco.
- Transformação
- Adicionam-se 100 ng do produto de PCR purificado a partir deo passo 1.3.1 para as células competentes. Flick o tubo e permitir a suspensão para se sentar em gelo durante 5 min.
- Transferir a suspensão de gelados células competentes e ADN para uma cuvete de electroporação de pré-arrefecida. Electroporate mistura de células com o kV 2,5, 25 mF, 200 ohms criação de uma lacuna cuvete 2 milímetros (ou seja, se com uma tina de diferente, consulte as instruções do fabricante para as configurações de eletroporação) e, imediatamente, adicionar 1 ml de meio ambiente temperatura SOC. Transferir as células electroporadas em meio SOC para um tubo de 15 ml de cultura e incuba-se a 32 ° C durante 1 hora com agitação orbital a 220 rpm.
- Após a incubação, centrifugar as células a 4400 xg durante 5 min à temperatura ambiente. Remover cerca de 850 ul do sobrenadante e ressuspender o sedimento de células no líquido restante.
- Espalhar as células sobre placas de LB pré-aquecido contendo 17 ug / ml de Cm, e incubar a 32 ° C durante a noite. Escolha duas colônias transformantes individuais e raia para fora em LBCm-placas para confirmação do mutante. Recomendamos evitar maiores colónias transformantes, a fim de reduzir a possibilidade de escolher as estirpes com mutações raras supressores. Também streak os mesmos transformantes em LB-Kan para confirmar que as estirpes não são resistentes Kan.
Nota: Uma vez que o marcador de resistência Cm é usada para fazer as cepas de doadores, não esperamos que os mutantes doadores para ser Kan resistente. Por formação de estrias em ambos os transformantes LB-Kan e LB-Cm placas, podemos garantir que a mutação em si não doador de algum modo provocar resistência Kan. Se este passo não foi realizado e as mutações alvo foram de afectar a capacidade da estirpe para sobreviver em Kan, mutantes duplos não seria feito de forma eficaz usando eSGA desde estirpes dadoras sobreviveriam todas as etapas de selecção. Esta é apenas uma medida de precaução lógica para pegar fenótipos inesperados ou quaisquer outros problemas que possam interferir com a conclusão das telas eSGA. Se essa etapa foram ignorados, acolherá outros controlesD ainda identificar telas de fracassados. Contudo, uma detecção precoce de um dador que é resistente à Kan eliminaria uma série de passos não produtivos e desnecessária. Outras razões possíveis para uma estirpe crescer em Kan podem incluir, por exemplo expirado antibiótico, assim como a utilização de um produto de PCR errado ou de uma estirpe para a transformação. Todos esses problemas podem ser identificados e tratados logo no início do procedimento, com a ajuda deste controle, que não é obrigatório, mas recomendado para eSGA.
- Confirmando a deleção do gene não essencial (passo 1.6.1) ou mutação do gene essencial hypomorphic (seção 1.6.2)
Nota: Quando confirmar a deleção do gene por PCR, o ADN genómico a partir de uma estirpe de tipo selvagem deve ser utilizada como um controlo.- Confirmando não-essencial deleção do gene
- Para confirmar a eliminação do gene por PCR, o ADN genómico isolado a partir das estirpes transformadas, knockout putativos cultivadas em meio LB-Cm líquido, após o manufactinstruções do urer (Promega).
- Para confirmar recombineering sucesso, amplificar o ADN de cada transformante com três diferentes conjuntos de primers de confirmação knockout. Realizar as reacções com ~ 150 ng de molde de ADN genómico, e confirmar os tamanhos dos fragmentos correctos, executando os produtos de PCR-sintetizados em um gel de agarose.
- O conjunto de iniciadores consiste num primeiro 20 nt-iniciadores flanqueando, localizado a 200 bp a montante da região de destino (KOCO-F), e Cm-R primer, que é complementar à sequência de cassete Cm. Esta amplificação é esperado para produzir um fragmento amplificado 445 nt no mutante, mas não na estirpe de tipo selvagem (Figura 1).
- O segundo conjunto inclui um iniciador directo (Cm-F), hibridação com a sequência de cassete Cm, e um reverso 20-nt flanqueando iniciador de confirmação (KOCO-R), o qual foi concebido para hibridar 200 pb a jusante da extremidade 3 'do suprimido do gene. Esta reacção de amplificação é esperado para produzir um fragmento amplificado 309 nt no mutante, mas nenhumat na estirpe de tipo selvagem (Figura 1).
- A PCR terceiro contém KOCO-F-R e KOCO primers. Esta reacção é necessária para confirmar que a estirpe seleccionada não é uma merodiploid, com um locus do gene ter sido substituída por a cassete e uma outra cópia duplicada, mas de outra forma do gene do tipo selvagem ainda presente. Esta amplificação a partir de um mutante de deleção correcta é esperado para produzir um produto de 1,4 kb (Figura 1).
Nota: Quando o gene alvo tem aproximadamente o mesmo comprimento em pares de bases, como a cassete de deleção do gene, não se observará uma diferença entre um local, o qual tenha sido substituído pela cassete, e um local, o qual não tem. Para uma confirmação definitiva em tais casos, é recomendável usar iniciadores adicionais para amplificar especificamente um segmento de ADN dentro do gene suprimido. Neste caso, se o gene foi eliminado, um produto de amplificação vai ser observado apenas na estirpe de tipo selvagem de controlo e não no mutante. Estes iniciadorespode ser manual ou computacionalmente concebido especificamente para amplificar um segmento de ADN dentro da região eliminada. Nós normalmente amplificar 140 pb regiões.
- Confirmando mutação do gene essencial hypomorphic
- Para confirmar a mutação hypomorphic por PCR, utilizar-gene específico de 20 nt a frente (KOCO-F) e os iniciadores reversos (KOCO-R) situado a 200 pb a montante e a jusante, respectivamente, do sítio de inserção SPA-Tag (Figura 2).
Nota: Este passo de amplificação é utilizado como controlo para garantir a ausência de uma cópia não marcada do gene alvo essencial. Quando apenas uma versão marcada está presente, uma única banda de 310 pb maior do que o amplicon SPA Cm-é observado. Um 400 bp do produto seria um sinal da presença do untagged versão do gene. - Em paralelo com a confirmação de PCR, utilizar transferência de Western para verificar a inserção em enquadramento do SPA-tag, utilizando um anticorpo anti-FLAG M2 específica para os epítopos do FLAG-da SPA tag-21.
- Para confirmar a mutação hypomorphic por PCR, utilizar-gene específico de 20 nt a frente (KOCO-F) e os iniciadores reversos (KOCO-R) situado a 200 pb a montante e a jusante, respectivamente, do sítio de inserção SPA-Tag (Figura 2).
- Confirmando não-essencial deleção do gene
- Armazenar a estirpe dadora confirmado
- Transferir a cultura durante a noite da estirpe dadora mutante recombinante confirmou que os criotubos rotulados, complementar com glicerol a 15% de concentração final, e misturar bem. Para armazenamento de longo prazo, manter os criotubos a -80 ° C.
2. Arraying E. coli F-beneficiário Mutantes para Telas eSGA
- Replica-o pino E. coli Keio único não essencial recolha de deleção do gene mutante gerado por Mori et ai. (3968 estirpes F-11; BW25113, pode ser obtido a partir de Open Biosystems) roboticamente para 24 384 poços de microplacas com 80 ul de meio LB líquido por poço , suplementado com 50 ng / ml Kan
Nota: Para a avaliação sistemática IG com genes essenciais, o destinatário Keio recolha 11 pode ser suplementado com o F-Kan-marcado gene essencial hypomorphcepas mutantes com ic C-terminal SPA-tags 22, 23. - Para dar espaço para a estirpe de controlo de fronteira, que irá actuar como um controlo positivo, a ajuda de dentro e entre as normalizações placa, bem como em quantizations colónias automatizado, remover o meio inoculado a partir dos poços de cada placa ultraperiféricas do passo anterior e de transferência para placas novas, mais uma vez de deixar os poços ultraperiféricas vazio.
Nota: A estirpe de controlo de fronteira JW5028 da colecção Keio 11 é suprimida por um pequeno gene pseudo que não deve, e não nos nossos genoma inteiro telas, exibem IG com quaisquer dadores. Portanto, esta é uma estirpe de controlo bem positivo para identificar casos raros em conjugação, prendendo, ou seleções falharam, resultando em crescimento ausentes ou esporádica fronteira tensão. Por exemplo, se as colónias de controlo de fronteira não se desenvolvem após a selecção duplo mutante, a estirpe de consulta pode não ser capaz de conjugação, a conjugação pode ter sido tentada ind condição que impediu a ter lugar (por exemplo, sobre uma placa contendo antibiótico), ou um antibiótico de errado podem ter sido adicionados à placa de selecção. Da mesma forma, se muito poucas colónias esporádicos, são observados em toda a placa, incluindo a borda, conjugação ou pode ter falha de pinagem e a tela deve ser repetido. Se esse crescimento fronteira esporádica é reprodutível, isso pode indicar a conjugação de doadores consistentemente pobres e, assim, casos em que mutantes duplos não são observadas são, possivelmente devido a uma falha de conjugação, não soldados verdade. Tais telas teriam de ser descartados a partir de uma análise mais aprofundada. Além disso, por estar presente em todas as placas, a estirpe de controlo fronteiriço permite que o software de quantização colónia para determinar automaticamente posições de colónias nas placas duplas mutantes e, assim, as posições de colónias não têm que ser demarcada manualmente. Por último, os auxiliares de controle de fronteiras de tensão na normalização de tamanhos colônia dentro (por exemplo, linhas diferentes dentro de um prato) e betw(por exemplo, placas een receptores diferentes preso com o mesmo doador em momentos diferentes) placas. - Do mesmo modo, criar pontos de controlo negativo, removendo quaisquer duas estirpes a partir de uma localização diferente no interior de cada placa (não de fronteira) e transferir as estirpes de uma nova placa. Preencher estes poços vazios com meio LB contendo 17 ug / ml de Cm. Estes pontos de controlo negativo devem estar vazio no recipiente e, por conseguinte, duplos mutantes placas e ajudar a assegurar que não havia nenhum tratamento ou erros de manuseamento da placa, quando a numeração, por imagem, ou a fixação das placas.
Nota: Estes lugares vazios são necessários controles negativos. Controles negativos ajudar a identificar casos quando a seleção não - quando cepas de controlo negativo crescer durante a seleção. Sugerimos que não só seleccionar únicos pontos de controlo negativo para cada uma das placas receptoras, mas também para escolher os pontos de controlo negativo no mesmo quadrante da placa inferior (por exemplo, para a direita). Deste modo, o padrãode pontos de controlo negativo seria identificar os erros possíveis placa mau uso (por exemplo, placas sendo erroneamente ou sendo preso de cabeça para baixo, onde a colônia superior esquerdo é fixado na posição inferior direita). - Inocular a cepa Keio JW5028 11, com uma deleção de pseudogene, para um balão de 500 ml com 200 ml de LB, contendo 50 ug / ml de Kan Com base nas nossas telas de todo o genoma eSGA, o crescimento do mutante particular, é o mais próximo de selvagem tipo BW25113 e é assim utilizado como um controlo de fronteira.
- Crescer as estirpes dispostas, assim como a cultura durante a noite a JW5028 32 ° C com 190 rpm de agitação orbital uma DO de ~ 0,4-0,6 a 600 nm. Após o crescimento durante a noite, preencha os poços de fronteira com cerca de 80 ul de JW5028 cultura durante a noite.
- As placas montadas podem ser utilizados no passo 3.2. Alternativamente, para armazenamento a longo prazo, completar cada poço nas placas receptoras com glicerol a 15%, misturar os meios de comunicação e glicerol, e manter as placasa -80 ° C.
3. Alta densidade Procedimento acasalamento Strain de Geração de E. Mutantes coli Duplo
- Crescer a estirpe dadora Hfr, tendo a mutação de consulta, marcado com Cm, durante a noite a 32 ° C em meio líquido rico LB-Cm (17 ng / ml de Cm) com agitação a 220 rpm.
- Pin a recolha destinatário ordenada mutante em 384 colónias de densidade em placas LB sólido suplementado com 50 ug / ml de Kan Em paralelo, o pino da estirpe mutante de consulta doador em 384 colónias de densidade para o mesmo número de placas de LB suplementado com 17 ug / ml de Cm. Incubar as placas durante a noite a 32 ° C.
Nota: As placas receptoras utilizadas para pinagem pode exigir até 36 horas, para a obtenção de colônias suficientemente grandes para as etapas subseqüentes. Quando o tamanho colônia média fronteira é de cerca de 2 mm de diâmetro (ou maior), as placas estão prontos para ser fixado. - Para conjugar as estirpes, fixar o doador de uma placa doador 384 colônias durante a noite ema placa de LB sólido. Posteriormente fixar uma placa único destinatário 384 colônias sobre o doador recém-fixado na placa de conjugação. Continue prendendo até que todas as placas de doador-receptor tem sido conjugado fixando em placas de conjugação sólidos LB. Incubar as placas de conjugação fixados a 32 ° C durante 16 a 24 horas.
Nota: individuais únicas estirpes dadoras mutantes podem apresentar gene-para-gene variabilidade na eficiência de acoplamento por causa dos possíveis efeitos das mutações sobre a conjugação, a eficiência de transferência de ADN, ou de fitness. O crescimento pode ser interrompida em qualquer momento compreendido entre 16 e 24 horas, quando suficientemente grandes colónias foram obtidos para os passos subsequentes de imobilização. Conjugações a duração de 16 a 24 horas produzir um número suficiente de ex-conjugantes para resultados reproduzíveis eSGA mesmo para os mutantes da tarde a transferência de genes mutantes ou com aptidão ligeiramente inferior. - Pin cada placa de conjugação 384 de densidade sobre uma placa de LB único sólido, contendo Cm (17 ug / mL) e Kan (50ug / ml), até que todas as placas tenham sido fixados de conjugação. Incubar os recém-fixadas placas primeira seleção para 16-36 horas a 32 ° C.
Nota: No que se refere aos passos de selecção, diferentes quantidades de tempo, pode ser necessário que as telas de doadores diferentes. Uma vez que todos os mutantes duplos em uma determinada tela têm a mutação mesmo doador, em média, um mutante mais lento crescimento dador origina crescimento mais lento mutantes duplos. Assim, dependendo da aptidão do doador, as etapas de selecção pode exigir entre 16 e 36 horas. O crescimento mutante duplo pode ser parado quando os mutantes duplos são suficientemente grandes para o passo subsequente ou imobilizando após a primeira selecção ou de imagem depois da segunda. Isso geralmente se traduz para a fronteira colônias de controle, em média, com pelo menos 2 mm de diâmetro. - Re-pin cada placa para uma primeira selecção de droga, segunda dupla (Cm e Kan), placa de selecção em 1536 colónias de formato, de modo que cada colónia primeira selecção é representado por quatro colónias ema chapa uma segunda selecção. Incubar as placas durante 16-36 horas a 32 ° C. Fotografar os finais duplas mutantes placas de selecção para medir quantitativamente a aptidão do crescimento do mutante e analisar as interacções entre todos os pares de genes (Figura 3).
Nota: Embora a frequência de merodiploidy (parciais duplicações cromossômicas) é baixa em cerca de 1/1, 000 células 24, merodiploidy pode mascarar GI. Por isso, recomendamos incluindo réplicas biológica em todas as telas eSGA. Felizmente, comerciais placas de plástico descartáveis e almofadas de imobilização pode ser reutilizado até três vezes por esterilização dos materiais como se segue: Agar das placas é rejeitada e as placas, bem como as almofadas são esterilizadas por imersão em lixívia a 10% durante a noite, seguido de uma lavar com água destilada, lavagem em etanol a 70%, e com ar de secagem a plasticware numa câmara de fluxo sob luz ultravioleta. As almofadas estéreis e as placas são armazenadas em sacos de plástico selados estéreis até utilização.
4. Processamento de dados e Derivando valores de GI
- Meça as colônias em cada placa em modo em lote ou individualmente usando um software de imagem especializada colônia 3, 9.
Nota: Uma adequada baseado em Java imagem colônia de alto rendimento e aplicação de pontuação agora está disponível livremente para acesso público ( http://srcollins77.users.sourceforge.net/ ). O software funciona em diferentes plataformas (ou seja Mac ou PC) e pode ser lançado tanto como um executável ou a partir de uma janela de terminal. - Normalizar as medidas de tamanho primas colônia, corrigindo desvios sistemáticos para dentro e entre as placas, como efeitos de borda, placa placa efeitos inter-variação, iluminação imagem irregular, artefatos devido à curvatura física da superfície do ágar, efeitos de concorrência para colônias vizinhas e possíveis defeitos fixando , assim como as diferenças de tempo de crescimento p> 9. Esses artefatos sistemáticas são independentes da aptidão duplo mutante e deve ser corrigido como eles dão origem a estimativas de crescimento da aptidão espúrias.
- As colónias em linhas e colunas de ponta tendem a ser maiores devido à menor competição por nutrientes do que os encontrados no centro da placa. Assim, para corrigir para este efeito, dimensionar o tamanho das colónias de ponta para ser tal que o tamanho médio, em linha ou coluna que é igual ao tamanho médio das colónias no centro da placa.
- Analisar os resultados estatisticamente para ter em conta a reprodutibilidade e a variância padrão a partir dos tamanhos médios das medições de colónias replicadas. Pelo menos, três experiências duplicadas biológicos independentes são necessárias para avaliar a reprodutibilidade experimental.
- Finalmente, a utilização dos tamanhos normalizados mediana de colónias de crescimento aptidão para gerar uma contagem GI (S), para cada par de genes usando a seguinte fórmula:
n 1 "src =" files/ftp_upload/4056/4056eq1.jpg / "/>
Onde, S = var (var Exp x (n Exp -1) + var Cont x (n Cont -1)) / (n + n Exp Cont-2); var Exp = a variância máxima dos tamanhos normalizados de colónias para o duplo mutante; var Cont mediana = das variações nos tamanhos normalizados duplas colônia mutantes a partir do conjunto de referência; Exp n = número de medições de duplas tamanhos colônia mutantes; Cont n = o número médio de réplicas experimental sobre todos os experimentos; Exp μ = Dimensões médias das colónias normalizados dos mutantes duplos, e Cont μ = média de tamanhos de colónias normalizadas para todos os mutantes duplos resultantes da estirpe mutante único doador. O S-score reflecte quer a confiança estatística de uma interacção digênica putativa, bem como a força da interacção biológica. Fortes positivos S-scores indicam efeitos aliviar, suggesting que os genes interagem participar na mesma via 9, enquanto negativo significativo S-scores refletir doença sintético ou letalidade, que muitas vezes é sugestivo de associação em paralelo caminhos redundantes 9. Para a determinação das relações de via nível funcional, um único S-score para cada par de genes testados é produzido a partir do conjunto de dados final.
Nota: No nosso estudo anterior 9, verificou-se que a recombinação tende a ocorrer menos frequentemente entre os genes dentro de 30 kpb do outro. Portanto, para a análise a jusante, após normalização e geração de pontuação, que remove as interações entre os genes dentro de 30 kpb do outro. Além de soldados si, relações funcionais entre os pares de genes pode ser investigado por olhar para a semelhança entre os perfis GI dos dois genes são. O perfil GI de um gene é o conjunto de todas as interacções do gene que com todos os outros genes do genoma fora doárea de ligação do gene. Genes funcionalmente semelhantes tendem a ter maiores correlações de perfis genéticos de interação 12, 25. Assim, ao calcular os coeficientes de correlação para todos os genes no conjunto de dados experimentais pode-se utilizar para investigar eSGA relações funcionais mesmo entre os genes encontram-se próximos um do outro no cromossoma.
Resultados
GIs reveal functional relationships between genes. Similarly, since genes in the same pathway display similar GI patterns and the GI profile similarity represents the congruency of phenotypes, we can group functionally related genes into pathways by clustering their GI profiles. Integrating GI and GI correlation networks with physical interaction information or other association data, such as genomic context (GC) relationships can also reveal the organization of higher-order functional modules that define core biological systems (and system crosstalk) in bacteria. For example, as with yeast4, 6, 7, 26, E. coli gene pairs exhibiting alleviating interactions that also have highly correlated GI profiles tend to encode proteins that are either physically associated (e.g. form a complex; Figure 4a) or that act coherently in a common biochemical pathway (e.g. components in a linear cascade). A representative example is shown in Figure 4b, where the components of the functionally redundant Isc and Suf pathways, which jointly participate in the essential Fe-S biosynthesis process, form distinct clusters that are linked together by extensive aggravating interactions (i.e. synthetic lethality). A statistical measure (e.g. hypergeometric distribution function27) can be used on GI or GI correlation data to find significant enrichment for interactions between and within pathways (Figure 4c). Cluster analysis can also be applied to functional networks derived using eSGA to predict the functions of genes lacking annotations5, 6, 9, 28-30 (Figure 4d). Since clustering algorithms vary, however, putative functional assignments determined through clustering require independent experimental verification.
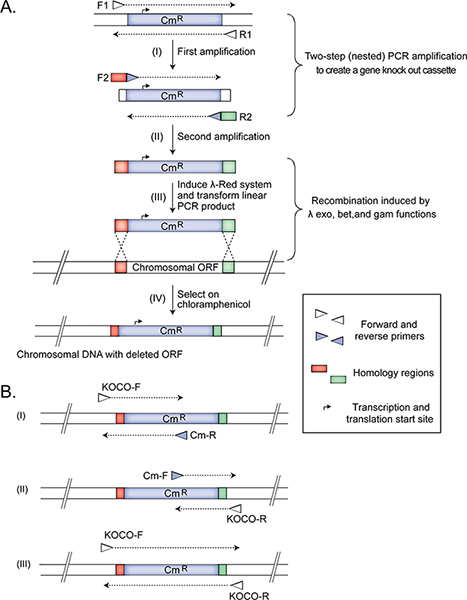
Figure 1. The construction and confirmation of Hfr Cavalli non-essential single gene deletion donor strains. The panels (adapted from14) illustrate the deletion of E. coli chromosomal ORF (A) and the three primer sets used for confirming (B) the correctly generated mutant Hfr Cavalli donor strain. Amplifications from a correctly constructed deletion donor strain should produce (i) 445 bp, (ii) 309 bp, and (iii) 1.4 kb products. See protocol text for details. Click here to view larger figure.

Figure 2. The construction and confirmation strategies of essential hypomorphic donor mutant strains in an Hfr Cavalli genetic background. The panels illustrate the creation of hypomorphic mutations of essential genes through recombineering technology (A) and the primer sets (B) used for the PCR confirmation of essential gene hypomorphic mutations. See protocol text for details.

Figure 3. Schematic summary of key eSGA steps. The Hfr donor (marked with chloramphenicol resistance, CmR) and recipient F- (marked with kanamycin resistance, KanR) mutant strains are grown in 384-colony density on LB-Cm and LB-Kan plates, respectively. The donor and the recipient strains are conjugated by pinning them over each other onto an LB plate, which is then incubated overnight at 32 °C. Consequently, the conjugants are pinned onto plates containing both Kan and Cm to select double mutants. The double mutant plates are digitally imaged and the growth fitness of the colony sizes is quantitatively scored to identify aggravating and alleviating interactions.

Figure 4. Representative computational analyses of genetic interaction (GI) scores for determining pathway-level functional relationships. (A) Panel I, Distribution of correlation coefficients between the GI profiles of gene pairs encoding proteins linked by protein-protein interactions (PPI) versus randomly drawn gene pairs. The p-value was computed using the two-sample Kolmogorov-Smirnov (KS) test; Panel II, Scatter plot of correlated genetic profiles from rich media (RM) for two transporters (mdtI, mdtJ) that form a heterodimeric complex required for spermidine excretion. (B) The hierarchical clustering of the GI sub-network adapted from Butland et al.9 highlights the functional connectivity between components of the previously known Isc and Suf pathways with similar GI patterns. Pink represents aggravating (negative S-score) interactions, green represents alleviating (positive S-score) interactions and black represents absence of GI. A predicted novel component of the SUF pathway, ydhD, displays GIs with the members of the Isc pathway. (C) Pathway cross-talk recorded among cell envelope processes that are significantly enriched for aggravating or alleviating GI according to the hypergeometric enrichment analysis27. (D) A GI sub-network predicts the role of two functionally unannotated genes, yceG and yebA, in peptidoglycan splitting based on their pattern of strong alleviating interactions with well-known cell division peptidoglycan hydrolases. The figures shown in panels A, C and D are adapted from Babu et al. (2011)12. Click here to view larger figure.
Discussão
Temos delineado um protocolo passo a passo para a utilização de triagem eSGA robótico para investigar funções de genes bacterianos em um nível caminho interrogando GI. Esta abordagem pode ser usada para estudar os genes individuais, bem como os sistemas biológicos inteiras em E. coli. Cuidadosamente executar as etapas experimentais descritas acima, incluindo todos os controles apropriados, e rigorosamente análise e validação dos dados de forma independente GI são aspectos fundamentais para o sucesso de eSGA em fazer novas descobertas funcionais. Além de eSGA, um método conceptualmente semelhante para estudar GI em E. coli, denominado GIANT coli-17, pode ser usado para iluminar novas relações funcionais que são frequentemente perdidas por outras abordagens, tais como a proteómica 23 ou 13 ecrãs fenotípicas sozinho. No entanto, como com qualquer abordagem de todo o genoma, não existem limitações sobre a aplicabilidade da eSGA ou gigante-coli, por exemplo, para outras espécies bacterianas. Isto é, em parte, sercausar a indisponibilidade de genoma único em toda a deleção coleções cepa mutante para a maioria das espécies de bactérias, especialmente para espécies que a mutagênese dirigida é dificultada por uma baixa frequência natural de recombinação homóloga. Em tais casos, as interrupções de genes, utilizando métodos tais como mutagénese aleatória rastreáveis ensaios baseados como TnSeq 31 pode potencialmente ser utilizada para investigar as funções de genes bacterianos. Para uma visão geral das novas alternativas bacterianas procedimentos de triagem genética, ver artigo de revisão de Gagarinova e Emili (2012) 32.
Embora os métodos de GI muitas vezes revelam muitas conexões interessantes sugestivo de novas ligações mecanicistas, a integração destes dados com outras informações, como perfil fenotípico 13, as redes de interação física 22, 23 e GC-inferidas associações podem ser particularmente informativo. Por exemplo, com as redes de GI, associações de GC, os quais são baseados na conservaçãoda ordem dos genes (operons), fusões de genes bacterianos, frequências de operão de recombinação a partir de distâncias derivadas intergénicas de operons preditas através genomas, bem como filogenética perfilamento 33 pode fornecer informações adicionais sobre como uma célula bacteriana organiza complexos de proteína em vias funcionais para mediar e coordenar major processos celulares 12, 23.
A E. coli é um modelo laborioso para entender a biologia molecular de outras bactérias Gram-negativas. Para este efeito, os dados eSGA GI pode ser usado em conjunto com a informação genómica comparativa (por exemplo, perfis filogenética, o perfil de expressão de genes) para investigar a conservação evolutiva de relações funcionais descobertos por eSGA através de outras espécies e taxa proteobacterial Prokaryotic. Além disso, os dados de interacção gerada por E. coli pode ser utilizado para obter insights sobre a arquitetura caminho de micróbios descobertos por metagenomicrofones, para o qual as anotações funcionais estão faltando. Uma vez que muitos genes são amplamente conservada entre todos os 23 micróbios, e porque a sensibilidade aos antibióticos pode ser aumentada por certas perturbações genéticas 34, as relações funcionais iluminadas por eSGA podem mesmo vir a ser explorados para a concepção de terapias de combinação com medicamentos inovadores.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Este trabalho foi financiado por fundos do Genome Canada, o Instituto Ontário Genomics, e os Institutos Canadenses de Pesquisa em Saúde de bolsas para JG e AG AE é um receptor de Vanier de Bolsas de Pós-Graduação no Canadá.
Materiais
| Name | Company | Catalog Number | Comments |
| I. Antibiotics | |||
| Chloramphenicol | Bioshop | #CLR201 | |
| Kanamycin | #KAN201 | ||
| Ampicillin | # AMP201 | ||
| 2. Luria-Bertani medium | |||
| LB powder | Bioshop | #LBL405 | |
| Agar | Bioshop | #AGR003 | |
| 3. Bacterial Strains and Plasmids | |||
| Hfr Cavalli strain λred system (JL238) | Babu et al.14. | ||
| pKD3 | E. coli Genetic Stock Centre, Yale | ||
| Keio E. coli F- recipient collection | National BioResource Project (NBRP) of Japan11 | ||
| Hypomorphic E. coli F- SPA-tag strains | Open biosystems; Babu et al.14 | ||
| 4. Primers | |||
| pKD3-based desalted constant primers | F1: 5'-GGCTGACATGGGAATTAGC-3' R1: 5'-AGATTGCAGCATTACACGTCTT-3' | ||
| Desalted custom primers | Cm-R: 5'-TTATACGCAAGGCGACAAGG-3' Cm-F: 5'- GATCTTCCGTCACAGGTAGG-3' | ||
| Desalted custom primers | F2 and R2: 20 nt constant regions based on pKD3 sequence and 45 nt custom homology regions F2 constant region: 5'-CATATGAATATCCTCCTTA-3' R2 constant region: 5'-TGTGTAGGCTGGAGCTGCTTC-3'S1 and S2: 27 nt constant regions for priming the amplification of the SPA-Cm cassette and 45 nt custom homology regions S1 constant region: 5'AGCTGGAGGATCCATGGAAAAGAGAAG -3' S2 constant region: 5'- GGCCCCATATGAATATCCTCCTTAGTT -3' KOCO-F and KOCO-C: 20 nt primers 200 bp away from the non-essential gene deletion site or the essential gene SPA-tag insertion site | ||
| 5. PCR and Electrophoresis Reagents | |||
| Taq DNA polymerase | Fermentas | # EP0281 | |
| 10X PCR buffer | Fermentas | # EP0281 | |
| 10 mM dNTPs | Fermentas | # EP0281 | |
| 25 mM MgCl2 | Fermentas | # EP0281 | |
| Agarose | Bioshop | # AGA002 | |
| Loading dye | NEB | #B7021S | |
| Ethidium bromide | Bioshop | # ETB444 | |
| 10X TBE buffer | Bioshop | # ETB444.10 | |
| Tris Base | Bioshop | # TRS001 | |
| Boric acid | Sigma | # T1503-1KG | |
| 0.5 M EDTA (pH 8.0) | Sigma | # B6768-500G | |
| DNA ladder | NEB | #N3232L | |
| 6. DNA isolation and Clean-up Kits | |||
| Genomic DNA isolation and purification kit | Promega | #A1120 | |
| Plasmid Midi kit | Qiagen | # 12143 | |
| QIAquick PCR purification kit | Qiagen | #28104 | |
| 7. Equipment for PCR, Transformation and Replica-pinning | |||
| Thermal cycler | BioRad, iCycler | ||
| Agarose gel electrophoresis | BioRad | ||
| Electroporator | Bio-Rad GenePulser II | ||
| 0.2 cm electroporation cuvette | Bio-Rad | ||
| 42 °C water bath shaker | Innova 3100 | ||
| Beckman Coulter TJ-25 centrifuge | Beckman Coulter | ||
| 32 °C shaker | New Brunswick Scientific, USA | ||
| 32 °C plate incubator | Fisher Scientific | ||
| RoToR-HDA benchtop robot | Singer Instruments | ||
| 96, 384 and 1,536 pin density pads | Singer Instruments | ||
| 96 or 384 long pins | Singer Instruments | ||
| 8. Imaging Equipments | |||
| Camera stand | Kaiser | ||
| Digital camera, 10 megapixel | Any Vendor | ||
| Light boxes, Testrite 16" x 24" units | Testrite | ||
| 9. Pads or Plates Recycling | |||
| 10% bleach | Any Vendor | ||
| 70% ethanol | Any Vendor | ||
| Sterile distilled water | Any Vendor | ||
| Flow hood | Any Vendor | ||
| Ultraviolet lamp | Any Vendor | ||
| 10. Labware | |||
| 50 ml polypropylene tubes | Any Vendor | ||
| 1.5 ml micro-centrifuge tubes | Any Vendor | ||
| 250 ml conical flaks | VWR | # 29140-045 | |
| 15 ml sterile culture tubes | Thermo Scientific | # 366052 | |
| Cryogenic vials | VWR | # 479-3221 | |
| Rectangular Plates | Singer Instruments | ||
| 96-well and 384-well microtitre plates | Singer Instruments | Nunc | |
| Plate roller for sealing multi-well | Sigma | #R1275 | |
| plates | ABgene | # AB-0580 | |
| Adhesive plate seals | Fisher Scientific | # 13-990-14 | |
| -80 °C freezer | Any Vendor | ||
Referências
- Bandyopadhyay, S., Kelley, R., Krogan, N. J., Ideker, T. Functional maps of protein complexes from quantitative genetic interaction data. PLoS Comput. Biol. 4, e1000065(2008).
- Costanzo, M., Baryshnikova, A., Myers, C. L., Andrews, B., Boone, C. Charting the genetic interaction map of a cell. Curr. Opin. Biotechnol. 22, 66-74 (2011).
- Costanzo, M., et al. The genetic landscape of a cell. Science. 327, 425-4231 (2010).
- Fiedler, D., et al. Functional organization of the S. cerevisiae phosphorylation network. Cell. 136, 952-963 (2009).
- Roguev, A., et al. Conservation and rewiring of functional modules revealed by an epistasis map in fission yeast. Science. 322, 405-4010 (2008).
- Schuldiner, M., et al. Exploration of the function and organization of the yeast early secretory pathway through an epistatic miniarray profile. Cell. 123, 507-519 (2005).
- Wilmes, G. M., et al. A genetic interaction map of RNA-processing factors reveals links between Sem1/Dss1-containing complexes and mRNA export and splicing. Mol. Cell. 32, 735-746 (2008).
- Babu, M., et al. Systems-level approaches for identifying and analyzing genetic interaction networks in Escherichia coli and extensions to other prokaryotes. Mol. Biosyst. 12, 1439-1455 (2009).
- Butland, G., et al. coli synthetic genetic array analysis. Nat. Methods. 5, 789-7895 (2008).
- Typas, A., et al. Regulation of peptidoglycan synthesis by outer-membrane proteins. Cell. 143, 1097-10109 (2010).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Mol. Syst. Biol. 2, 2006-200008 (2006).
- Babu, M., et al. Genetic interaction maps in Escherichia coli reveal functional crosstalk among cell envelope biogenesis pathways. PLoS Genet. 7, e1002377(2011).
- Nichols, R. J., et al. Phenotypic landscape of a bacterial cell. Cell. 144, 143-156 (2011).
- Babu, M., Gagarinova, A., Greenblatt, J., Emili, A. Array-based synthetic genetic screens to map bacterial pathways and functional networks in Escherichia coli. Methods Mol Biol. 765, 125-153 (2011).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc. Natl. Acad. Sci. U.S.A. 97, 6640-665 (2000).
- Yu, D., et al. An efficient recombination system for chromosome engineering in Escherichia coli. Proc. Natl. Acad. Sci. U.S.A. 97, 5978-5983 (2000).
- Typas, A., et al. quantitative analyses of genetic interactions in. E. coli. Nat. Methods. 5, 781-787 (2008).
- Zeghouf, M., et al. Sequential Peptide Affinity (SPA) system for the identification of mammalian and bacterial protein complexes. J. Proteome Res. 3, 463-468 (2004).
- Davierwala, A. P., et al. , 1147-1152 (2005).
- Breslow, D. K., et al. A comprehensive strategy enabling high-resolution functional analysis of the yeast genome. Nat. Methods. 5, 711-718 (2008).
- Babu, M., et al. Sequential peptide affinity purification system for the systematic isolation and identification of protein complexes from Escherichia coli. Methods Mol. Biol. 564, 373-400 (2009).
- Butland, G., et al. Interaction network containing conserved and essential protein complexes in Escherichia coli. Nature. 433, 531-537 (2005).
- Hu, P., Janga, S. C., Babu, M., Diaz-Mejia, J. J., Butland, G., et al. Global functional atlas of Escherichia coli encompassing previously uncharacterized proteins. PLoS Biol. 7, (2009).
- Anderson, R. P., Roth, J. R. Tandem genetic duplications in phage and bacteria. Annu. Rev. Microbiol. 31, 473-505 (1977).
- Boone, C., Bussey, H., Andrews, B. J. Exploring genetic interactions and networks with yeast. Nat. Rev. Genet. 8, 437-449 (2007).
- Collins, S. R., et al. Functional dissection of protein complexes involved in yeast chromosome biology using a genetic interaction map. Nature. 446, 806-8010 (2007).
- Le Meur, N., Gentleman, R. Modeling synthetic lethality. Genome Biol. 9, R135(2008).
- Tong, A. H., et al. Systematic genetic analysis with ordered arrays of yeast deletion mutants. Science. 294, 2364-2368 (2001).
- Tong, A. H., et al. Global mapping of the yeast genetic interaction network. Science. 303, 808-813 (2004).
- Wong, S. L., et al. Combining biological networks to predict genetic interactions. Proc. Natl. Acad. Sci. U.S.A. 101, 15682-15687 (2004).
- van Opijnen, T., Bodi, K. L., Camilli, A. Tn-seq: high-throughput parallel sequencing for fitness and genetic interaction studies in microorganisms. Nat. Methods. 6, 767-772 (2009).
- Gagarinova, A., Emili, A. Genome-scale genetic manipulation methods for exploring bacterial molecular biology. Mol. Biosyst. 8, 1626-1638 (2012).
- Dewey, C. N., et al. Positional orthology: putting genomic evolutionary relationships into context. Brief Bioinform. 12, 401-412 (2011).
- St Onge, R. P., et al. Systematic pathway analysis using high-resolution fitness profiling of combinatorial gene deletions. Nat. Genet. 39, 199-206 (2007).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados
Usamos cookies para melhorar sua experiência em nosso site.
Ao continuar usando nosso site ou clicando em 'continuar', você concorda em aceitar nossos cookies.