Method Article
رسم خرائط الشبكات الوظيفية البكتيرية والممرات في
In This Article
Summary
ويمكن استخدام منهجية واسعة النطاق الاصطناعية الوراثية شاشات التفاعل (الجينات الجين أو قشوة) لاستكشاف التكرار الجيني وعبر مسار الحديث. ووصف eSGA هنا، نحن تصف عالية الإنتاجية الكمية الاصطناعية الفحص الجيني مجموعة التكنولوجيا، التي قمنا بتطويرها لتوضيح العلاقات واستكشاف روكبي شبكات التفاعل الوراثية في كولاي.
Abstract
Phenotypes are determined by a complex series of physical (e.g. protein-protein) and functional (e.g. gene-gene or genetic) interactions (GI)1. While physical interactions can indicate which bacterial proteins are associated as complexes, they do not necessarily reveal pathway-level functional relationships1. GI screens, in which the growth of double mutants bearing two deleted or inactivated genes is measured and compared to the corresponding single mutants, can illuminate epistatic dependencies between loci and hence provide a means to query and discover novel functional relationships2. Large-scale GI maps have been reported for eukaryotic organisms like yeast3-7, but GI information remains sparse for prokaryotes8, which hinders the functional annotation of bacterial genomes. To this end, we and others have developed high-throughput quantitative bacterial GI screening methods9, 10.
Here, we present the key steps required to perform quantitative E. coli Synthetic Genetic Array (eSGA) screening procedure on a genome-scale9, using natural bacterial conjugation and homologous recombination to systemically generate and measure the fitness of large numbers of double mutants in a colony array format. Briefly, a robot is used to transfer, through conjugation, chloramphenicol (Cm) - marked mutant alleles from engineered Hfr (High frequency of recombination) 'donor strains' into an ordered array of kanamycin (Kan) - marked F- recipient strains. Typically, we use loss-of-function single mutants bearing non-essential gene deletions (e.g. the 'Keio' collection11) and essential gene hypomorphic mutations (i.e. alleles conferring reduced protein expression, stability, or activity9, 12, 13) to query the functional associations of non-essential and essential genes, respectively. After conjugation and ensuing genetic exchange mediated by homologous recombination, the resulting double mutants are selected on solid medium containing both antibiotics. After outgrowth, the plates are digitally imaged and colony sizes are quantitatively scored using an in-house automated image processing system14. GIs are revealed when the growth rate of a double mutant is either significantly better or worse than expected9. Aggravating (or negative) GIs often result between loss-of-function mutations in pairs of genes from compensatory pathways that impinge on the same essential process2. Here, the loss of a single gene is buffered, such that either single mutant is viable. However, the loss of both pathways is deleterious and results in synthetic lethality or sickness (i.e. slow growth). Conversely, alleviating (or positive) interactions can occur between genes in the same pathway or protein complex2 as the deletion of either gene alone is often sufficient to perturb the normal function of the pathway or complex such that additional perturbations do not reduce activity, and hence growth, further. Overall, systematically identifying and analyzing GI networks can provide unbiased, global maps of the functional relationships between large numbers of genes, from which pathway-level information missed by other approaches can be inferred9.
Protocol
1. بناء سلالات HFR المانحة كافالي المسخ بواسطة Recombineering 15 و 16
يتم وصف الخطوات لبناء البقع المانحة eSGA أدناه. لفترة وجيزة، ونحن نستخدم λ المستهدفة - الأحمر توسط إعادة التركيب مثلي 16 من تضخيم الحمض النووي كاسيت اختيار علامة الشظايا الناتجة من PCR لخلق طفرات غير الضرورية حذف الجينات (القسم 1.1) أو الجينات الأساسية hypomorphic سلالات متحولة المانحة (القسم 1.2) التي يتم استخدامها بعد ذلك "الاستعلامات" لتحديد شبكات GI.
ملاحظة: خلال عملية تطوير التكنولوجيا، ونحن تقييم فعالية استخدام HFR بوساطة نقل conjugative للجمع بين الطفرات باستخدام المانحة HFR واضحة المعالم سلالة (HFR كافالي، هايز HFR، وHFR 3000). درسنا (ط) القدرة على اتخاذ بكفاءة المسوخ المانحة باستخدام طريقة recombineering رائدها يو وآخرون. (2000) 16، (الثاني) والكفاءة النسبيةنقل DNA من علامات conjugative الكروموسومات المختلفة، و (الثالث). آثار موقف الجينات الصبغية الاستعلام والتوجيه بالنسبة للمكان نقل HFR، أوريت وجدنا أن λ - الأحمر بوساطة كفاءة إعادة التركيب مثلي كان أعلى من ذلك بكثير في كافالي HFR من HFR في هايز أو Hfr3000. إذا لزم الأمر، يمكن استخدام تنبيغ P1 أو سلالة HFR الزائف 17، الجهات المانحة لإنشاء متحولة. تكيفنا بنجاح جميع هذه الطرق لصنع وفرز أعداد كبيرة من المانحين. منذ تجاربنا في تصريف المحاكمة، الكفاءة العامة للنقل وعدد من السابقين لاحظ conjugants كان أكبر بكثير مع كافالي HFR، وقد تم اختيار هذه الخلفية سلالة معينة للبناء المانحة وeSGA على نطاق واسع. يتم وصف كل الإجراءات لشاشات eSGA باستخدام HFR المانحين كافالي.
- تضخيم الحمض النووي للجزء recombineering بعد حذف الجينات غير الضرورية
توظيف خطوتين متداخلة PCR AMPLIfication (الشكل 1) لإنشاء راديو كاسيت حذف الجينات لاستبدال الإطار الهدف القراءة مفتوحة مع علامة المقاومة سم من البلازميد pKD3 و(GI: 15554330؛ معرف البروتين: AAL02033.1) 15. ويحيط علامة ال 11 من FRT مواقع بجواز عزل الجين المقاومة، إذا لزم الأمر، في متابعة التجارب.
ملاحظة: نحن لا تتداخل خطوتين PCR لإزالة بلازميد من المنتج تضخيم يستخدم فيما بعد لتحويل الخلايا البكتيرية. إزالة بلازميد (PCR في الأولى) وإنشاء ثم كاسيت خروج المغلوب الجينات في PCR ثان ضروري لأن التحول بلازميد مع أكثر فاعلية من recombineering، والهدف من ذلك هو الحصول على - قدر الإمكان - سلالات فقط التي لديها استبدال بنجاح الحمض النووي الجيني المستهدفة مع علامة اختيار. بديل لPCR المتداخلة هو لأداء خلاصة القيد من خارج المنطقة البلازميد تضخيم PCR في 2الخطوة. ثم يتم تنقيته من المنتجات من هضم وتقييد استخدام كقالب في الخطوة PCR المتداخلة (1.1.2). هذا الخيار هو أيضا بسيطة، ولكنها تتطلب المزيد من العمل وقتا.- استخدام حوالي 45 نانوغرام من pKD3 كقالب في PCR مع الاشعال R1 F1 إلى الأمام وعكس لإنتاج منتج BP 1070. تنقية جزء PCR PCR باستخدام تنقية QIAGEN البروتوكول، وتمييع المنتج في الماء المقطر المعقم إلى 5 نانوغرام / ميكرولتر التركيز.
- استخدام المنتج PCR تنقية كقالب، إعداد PCR الثاني مع الاشعال (مثل خروج المغلوب) الجينات المحددة متداخلة، F2 وR2، التي لديها (ط) 45 NT من المناطق التماثل الجيني محددة في نهايات-5'للسماح لإعادة التركيب مثلي المنبع والمصب على الفور من الجين المستهدف ليحل محله مع علامة المقاومة سم و(الثاني) الإقليم الشمالي 20 في نهايات 3'-لرئيس تجميع للعلامة سم. حجم إنتاج جزء هو 1123 سنة مضت. تم تصميم مناطق محددة تماثل الجينات لإزالة المرجع الهدفEN قراءة الإطار مع ترك أي قطاعات متجاورة أو متداخلة جزئيا الترميز سليمة. بعد هذا التضخيم، انتقل إلى الخطوة 1.3.
- تضخيم شريط علامات لخلق طفرة جينية أساسية hypomorphic
- لبناء سلالات متحولة hypomorphic المانحة، إضافة محطة C-متتابعة الببتيد تقارب العلامة الانصهار (SPA)، والتي غالبا ما تؤثر قليلا الأساسية وظيفة البروتين من وفرة نص زعزعة الاستقرار (أي عدد النسخ البروتين) أو البروتين للطي / النشاط. تحقيقا لهذه الغاية، إنشاء قالب DNA اختيار SPA-علامة (SPA-CM) عن طريق تحويل علامة المقاومة في اساسه pJL148 18 إلى سم باستخدام تسلسل النوكليوتيدات recombineering بين متطابقة المرافقة علامة اساسه في pJL148 وعلامة سم في pKD3 15. القالب الناتج يتكون من في الإطار TAG-SPA تليها علامة المقاومة سم، ومناسبة لالتضخيم PCR، eSGA recombineering واللاحقة 12.
ملاحظة: </ STRONG> الجينات الأساسية يمكن أن تكون الأكثر إثارة للاهتمام للدراسات وظائف الجينات. ومع ذلك، لأنه لا يمكن حذف هذه الجينات، كان أساليب مبتكرة لتوضع للتحقيق العلاقات الوظيفية من الجينات الأساسية. وقد وضعت عدة نهج مناسبة ناجحة للغاية في البداية في الخميرة. على سبيل المثال، Davierwala وآخرون. العثور على (2005) 19 شاشات GI تنفيذها باستخدام لوحة من 575 حساسة للحرارة المسوخ الجينات الأساسية، وأن الجينات الأساسية معارضها حول عدد من الجنود خمس مرات والجينات غير الضرورية. وبالمثل، بريسلو وآخرون. (2008) 20 وضعت 'فرة بنسبة اضطراب مرنا' (الخام) نهج لإنشاء مكتبة من الأليلات hypomorphic تغطي ~ 82٪ من الجينات الخميرة الأساسية لدراسة مسارات والمجمعات. النهج رطبة تعديل 3'end من مرنا وأعرب، خفض مستوى الجين الهدف 6، من المحتمل بسبب مرنا زعزعة الاستقرار. على غرار النهج رطبة 6، الاستراتيجيه لديناكان ذ لاستغلال الآثار الخفية للاحداث بلبلة نهاية 3 'من مرنا وأعرب البروتينات الأساسية.
كما وصفنا أدناه، علامة في حد ذاته لا يؤثر على الأرجح للطي البروتين أو وظيفة، ولكن اضطراب خفية من مرنا من قيمة البطاقة هي كافية، وعندما يقترن الضغوطات البيئية أو غيرها من الطفرات، لتكشف وظيفيا بالمعلومات الجينات والبيئة 13 و الجين الجينات 9 و 12 التفاعلات.
أولا، حتى الآن، كنا SPA-به لتنقية ~ 3000 كولاي الأساسية وغير الأساسية البروتينات 21-23، مع أكثر من 80٪ من كولاي التي يمثلها 307 البروتينات الضرورية بين سلالات لدينا SPA-الموسومة 8. لانه كان يستخدم بنجاح العلامة لعزل بروتين يعرف المجمعات التي تم التحقق من صحتها بشكل متبادل (أي يمكن عزل نفسه عن طريق وضع علامات معقدة وتنقية البروتينات المختلفة من داخل المجمع نفسه)، ومجمعات معزولة وذات الصلة بيولوجيا، ونحن السبب في أن العلامات فعللا يؤثر عادة طي البروتين أو وظيفة 12. وبالمثل، كشفت ملاحظاتنا غير منشورة أن العلامة في حد ذاته لا يؤثر عادة نمو السلالة. ومع ذلك، نحن مسبب أن تغيير 3'-UTR من خلال دمج العلامة والعلامة الكاسيت، كما ذكرت لسلالات رطبة 6، قد تؤدي إلى زعزعة بعض النصوص على مستوى RNA أو، في حالات نادرة، وتعوق قابلة للطي مناسبة أو وظيفة الانصهار البروتينات. ولذلك، توقعنا أن الجمع بين هذه مضطرب الأليلات من الجينات الضرورية مع الطفرات الأخرى ذات الصلة وظيفيا في أي مكان آخر في الجينوم من شأنه أن يؤدي في نظم المعلومات الجغرافية بالمعلومات، التي كانت في الواقع لوحظ 9 و 12. عرض الأليلات وعلاوة على ذلك، SPA-الموسومة من الجينات الضرورية الجينات والبيئة التفاعلات (على سبيل المثال المخدرات فرط الحساسية) وفقا لأعمال التنميط المظهري من نيكولز وآخرون. (2011) 13، الذي تعرض مجموعة فرعية من SPA-الموسومة سلالات أساسية لمجموعة متنوعة من ظروف النمو. ويتأكد نفس الدراسةد أن معظم (54 من أصل 58 اختبار) SPA-الموسومة أظهرت السلالات الأساسية للياقة البدنية في ظروف ضعف ثقافة واحدة أو أكثر 13، مما يدعم فكرة أن في كثير من الأحيان علامة SPA-يخلق الأليلات hypomorphic معتدل في الجينات الأساسية التي هي مفيدة للالجينوم على نطاق تطبيقات الشاشة GI بما في ذلك eSGA. - تضخيم علامات كاسيت من القالب SPA-CM باستخدام الجينات محددة الاشعال S1 و S2، تحتوي كل منها على (ط) من 45 NT الجينات المحددة للمنطقة recombineering مثلي في نهاية 5 '، تليها (الثاني) في 27 BP-SPA تسلسل محدد الطول في نهاية 3 '.
- لبناء سلالات متحولة hypomorphic المانحة، إضافة محطة C-متتابعة الببتيد تقارب العلامة الانصهار (SPA)، والتي غالبا ما تؤثر قليلا الأساسية وظيفة البروتين من وفرة نص زعزعة الاستقرار (أي عدد النسخ البروتين) أو البروتين للطي / النشاط. تحقيقا لهذه الغاية، إنشاء قالب DNA اختيار SPA-علامة (SPA-CM) عن طريق تحويل علامة المقاومة في اساسه pJL148 18 إلى سم باستخدام تسلسل النوكليوتيدات recombineering بين متطابقة المرافقة علامة اساسه في pJL148 وعلامة سم في pKD3 15. القالب الناتج يتكون من في الإطار TAG-SPA تليها علامة المقاومة سم، ومناسبة لالتضخيم PCR، eSGA recombineering واللاحقة 12.
- مؤكدا وتنقية المنتج PCR
- تأكيد أنه تم تضخيم بشكل صحيح كاسيت علامات بإخضاع 2-5 ميكرولتر من المنتج PCR لهلام الاغاروز الكهربائي وتنقية DNA المتبقية بعد بروتوكول الشركة المصنعة معيار تنقية PCR (QIAGEN). أزل المنتج PCR في 30 ميكرولتر من الماء المقطر المعقم وتمييع لنانوغرام 50 ~ / ميكرولتر التركيز. وpurifiويمكن استخدام المنتج على الفور في إد التحول (الخطوة 1.5) أو تخزينها في -20 درجة مئوية لمدة تصل إلى 6 أشهر حتى الاستخدام.
- إعداد HFR الخلايا كافالي المختصة للبناء المانحة
- تطعيم واحد مل من ثقافة ليلة وضحاها من سلالة المشبعة كافالي HFR في 70 مل من المتوسط الطازجة (LB) لوريا Bertani-مع 35 ميكرولتر من 50 ميكروغرام الأمبيسلين مل / في قارورة 250 مل. احتضان الثقافة في C ° 32 مع اهتزاز في 220 دورة في الدقيقة حتى في كثافة الضوئية (OD) 600 نيوتن متر من ~ 0،5-0،6 يتم الحصول على (~ 2 ساعة).
- نقل الثقافة إلى حمام الماء لتحريض حرارة إعادة التركيب λ نظام الحمراء 16 في 42 ° C لمدة 15 دقيقة مع اهتزاز عند 160 دورة في الدقيقة. وقف التحريض عن طريق نقل الثقافة إلى حمام الماء المبرد الثلج لمدة 10-20 دقيقة الطين في 160 دورة في الدقيقة. تأكد للحفاظ على الخلايا الباردة من هذه النقطة حتى بعد التحول؛ أنابيب واستخدام cuvettes التي تم برود على الجليد لمدة 15 دقيقة قبل الاستخدام.
- تقسيم جulture على قدم المساواة إلى 2 قبل المبردة 50 مل أنابيب البولي بروبلين (~ الثقافة مل 35 في أنبوب) وأجهزة الطرد المركزي في 4400 x ج لمدة 6 دقائق في 4 درجات مئوية.
- تجاهل طاف، وإعادة تعليق بيليه الخلية عن طريق انعكاس لطيف في ~ 50 مل من الماء المقطر الجليد الباردة المعقمة. أجهزة الطرد المركزي الخلايا مرة أخرى في 4400 x ج لمدة 6 دقائق في C. ° 4 تجاهل طاف واعادة تعليق كل بيليه خلية في 20 مل من الجلسرين العقيمة الجليد الباردة 10٪ وأجهزة الطرد المركزي مرة أخرى كما هو موضح أعلاه.
- صب طاف وإعادة تعليق بيليه الخلية في 500 ميكرولتر من الجليد الباردة الجلسرين 10٪. قسامة الخلايا المعدة المختصة في 50 مجلدا ميكرولتر إلى الفردية، قبل المبردة 1،5 أنابيب الطرد المركزي الصغيرة مل للتحول فورا. قد يتم تجميد الخلايا على الثلج الجاف أو النتروجين السائل في وتخزينها في -80 ° C لمدة تصل إلى 3 أشهر. ومع ذلك، وكفاءة التحول هو أعلى مع الخلايا المختصة الطازجة.
- تحول
- إضافة 100 نانوغرام من الناتج PCR تنقيته منالخطوة 1.3.1 إلى الخلايا المختصة. نفض الغبار الأنبوب والسماح التعليق على الجلوس على الجليد لمدة 5 دقائق.
- نقل التعليق من الجليد الباردة الخلايا المختصة وDNA إلى كوفيت الصعق الكهربائي قبل مبردة. Electroporate خليط الخلية التي تحتوي على كيلو فولت 2.5، المكثف 25، 200 أوم وضع في كوفيت ملم الفجوة 2 (أي إذا باستخدام كوفيت مختلفة، الرجاء راجع إرشادات الشركة المصنعة للإعدادات الصعق الكهربائي)، وعلى الفور إضافة 1 مل من درجة حرارة الغرفة SOC المتوسط. نقل الخلايا electroporated في المتوسط SOC في أنبوب 15 مل الثقافة واحتضان في C ° 32 لمدة 1 ساعة مع الهز المدارية في 220 دورة في الدقيقة.
- بعد الحضانة، أجهزة الطرد المركزي الخلايا في 4400 x ج لمدة 5 دقائق في درجة حرارة الغرفة. إزالة نحو 850 ميكرولتر من طاف، وإعادة تعليق بيليه خلية في السائل المتبقي.
- تنتشر الخلايا LB على لوحات قبل تحسنت تحتوي على 17 ميكروغرام / مل سم، واحتضان عند 32 درجة مئوية خلال الليل. اختيار اثنين من المستعمرات transformant الفردية ومسحة من على LBسم لوحات لتأكيد متحولة. نوصي تجنب المستعمرات أكبر transformant من أجل الحد من إمكانية اختيار سلالات نادرة مع الطفرات القامع. أيضا في خط transformants نفسه على اساسه-LB للتأكد من أن السلالات المقاومة ليست اساسه.
ملاحظة: بما أن يتم استخدام العلامة المقاومة سم لجعل سلالات المانحة، ونحن لا نتوقع أن تكون المسوخ المانحة اساسه مقاومة. من streaking وtransformants على حد سواء اساسه-LB LB-وحات وسم، علينا أن نضمن أن الطفرة نفسها المانحة لم يتسبب إلى حد ما اساسه المقاومة. إذا لم تكن تنفيذ هذه الخطوة والطفرات المستهدفة كانت لتؤثر على قدرة سلالة من أجل البقاء على اساسه، لن تكون المسوخ مزدوجة الاستخدام الفعال eSGA منذ سلالات المانحة سيبقى على قيد الحياة جميع الخطوات الاختيار. هذا هو مجرد خطوة احترازية منطقية للقبض على الظواهر غير متوقعة أو أي قضايا أخرى قد تتداخل مع الانتهاء من شاشات eSGA. إذا تم تخطي هذه الخطوة، عوا عناصر التحكم الأخرىد تحديد تزال شاشات الفاشلة. ومع ذلك، فإن وجود كشف في وقت سابق من إحدى الجهات المانحة التي هي مقاومة للقضاء على اساسه سلسلة من الخطوات غير المنتجة وغير ضرورية. انتهت الأسباب المحتملة الأخرى لسلالة لتنمو على اساسه يمكن أن تشمل على سبيل المثال المضادات الحيوية وكذلك استخدام منتج PCR الخطأ أو سلالة لهذا التحول. ويمكن تحديد كل هذه القضايا والتعامل معها في وقت مبكر من الإجراء مع المعونة من هذه السيطرة، والتي ليست مطلوبة، ولكنها أوصت لeSGA.
- تأكيد الحذف الجينات غير الضرورية (الخطوة 1.6.1) أو طفرة جينية أساسية hypomorphic (القسم 1.6.2)
ملاحظة: عندما يؤكد حذف الجينات بواسطة PCR، ينبغي استخدام الحمض النووي الجيني من سلالة النوع البري كمجموعة تحكم.- مؤكدا غير الضرورية حذف الجينات
- لتأكيد الحذف من الجينات PCR، عزل الحمض النووي الجيني من تحول، سلالات خروج المغلوب المفترضة نمت في المتوسط LB-CM السائل، وبعد التصنurer في التعليمات (Promega).
- لتأكيد recombineering ناجحة، تضخيم الحمض النووي من كل transformant مع ثلاث مجموعات مختلفة من الاشعال تأكيد خروج المغلوب. أداء التفاعلات مع نانوغرام 150 ~ من قالب الحمض النووي الجيني، وتأكيد أحجام جزء الصحيح عن طريق تشغيل المنتجات PCR-توليفها على هلام الاغاروز.
- مجموعة التمهيدي الأول يتكون من 20 مادة اولية NT-المرافقة، على بعد 200 BP المنبع من المنطقة المستهدفة (KOCO-F)، وسم التمهيدي-R، والذي هو مكمل للتسلسل كاسيت سم. ومن المتوقع هذا التضخيم لإنتاج AMPLICON NT 445 في متحولة، ولكن ليس في النوع البري سلالة (الشكل 1).
- المجموعة الثانية تضم التمهيدي إلى الأمام (سم-F)، الصلب للتسلسل كاسيت سم، والعكس 20-NT المرافقة التمهيدي تأكيد (KOCO-R)، الذي يهدف إلى يصلب 200 المصب بي بي نهاية 3 'من حذف الجينات. ومن المتوقع رد الفعل هذا التضخيم لإنتاج AMPLICON NT 309 في متحولة، ولكن لار في سلالة النوع البري (الشكل 1).
- والثالث يحتوي PCR-F KOCO والاشعال KOCO-R. مطلوب رد الفعل هذا للتأكد من أن سلالة مختارة ليست merodiploid، حيث تم استبدال أحد الجينات من مكان الكاسيت وتكرار آخر ولكن على خلاف ذلك الجين نسخة من النوع البري لا تزال موجودة. ومن المتوقع هذا التضخيم من متحولة الحذف الصحيح لإنتاج منتج KB 1.4 (الشكل 1).
ملاحظة: عند الجين المستهدف لديها نحو نفس الطول في أزواج قاعدة الجين كما كاسيت الحذف، واحد لن تلاحظ الفرق بين موضع التي حلت محلها الكاسيت، والتي لم موضع. تأكيد نهائي في مثل هذه الحالات، ونحن نوصي باستخدام الاشعال إضافية لتضخيم تحديدا شريحة DNA داخل الجين حذف. في هذه الحالة، إذا تم حذف هذا الجين، سيتم وحظ منتج التضخيم فقط في نوع عنصر التحكم سلالة البرية وليس في متحولة. هذه الاشعاليمكن أن يكون يدويا أو مصممة خصيصا لتضخيم حسابيا جزء من الحمض النووي داخل المنطقة المحذوفة. ونحن عادة تضخيم المناطق BP 140.
- يؤكد الطفرة الجينية الأساسية hypomorphic
- لتأكيد الطفرة hypomorphic بواسطة PCR، استخدم الجينات المحددة 20-NT إلى الأمام (KOCO-F) والاشعال العكسي (KOCO-R) BP بعد 200 المنبع والمصب، على التوالي، من موقع الإدراج SPA-علامة (الشكل 2).
ملاحظة: يتم استخدام هذه الخطوة التضخيم كعنصر تحكم لضمان عدم وجود نسخة من هذا الجين untagged الهدف الأساسي. فقط عندما نسخة الموسومة موجودة، لكن لوحظ أنه واحد الفرقة 310 BP أكبر من AMPLICON SPA-CM. ومن شأن المنتج BP 400 إشارة وجود نسخة من هذا الجين untagged. - بالتوازي مع تأكيد PCR، استخدم الغربية النشاف للتحقق من الإدراج في الإطار للمجتمع طلال أبوغزاله للSPA باستخدام الأجسام المضادة لمكافحة FLAG M2 محددة إلى الحواتم-FLAG للSPA-21 علامة.
- لتأكيد الطفرة hypomorphic بواسطة PCR، استخدم الجينات المحددة 20-NT إلى الأمام (KOCO-F) والاشعال العكسي (KOCO-R) BP بعد 200 المنبع والمصب، على التوالي، من موقع الإدراج SPA-علامة (الشكل 2).
- مؤكدا غير الضرورية حذف الجينات
- تخزين سلالة المانحة أكدت
- نقل الثقافة بين عشية وضحاها من سلالة متحولة أكد المانحة المؤتلف إلى cryovials المسمى، مع الجلسرين لاستكمال تركيز النهائي 15٪، وتخلط جيدا. على المدى الطويل تخزين، والحفاظ على cryovials في -80 ° C.
2. Arraying E. القولونية F-مستلم المسوخ لشاشات eSGA
- طبق دبوس وE. (يمكن الحصول عليها من النظم البيولوجية فتح سلالات 3968 11؛؛ F-BW25113) آليا إلى 384 microplates جيدا 24 تحتوي على 80 ميكرولتر من وسائل الإعلام LB السائل لكل بئر واحدة القولونية كيو حذف الجينات غير الضرورية التي تم إنشاؤها بواسطة مجموعة متحولة موري وآخرون. ، على أن تستكمل مع 50 ميكروغرام / مل كانساس
ملاحظة: للحصول على تقييم منهجي نظم المعلومات الجغرافية مع الجينات الأساسية، يمكن استكمال كيو المتلقي جمع 11 مع لF-اساسه وضع علامات ناقص المفعول الجينات الأساسيةسلالات متحولة مع جيم C-SPA-محطة به 22 و 23. - لإفساح المجال لمراقبة الحدود سلالة، والذي سيكون بمثابة مراقبة إيجابية، والمساعدات في داخل وبين التطبيع لوحة وكذلك في quantizations مستعمرة الآلي، وإزالة وسائل الإعلام تلقيح من الآبار الأبعد من كل لوحة من الخطوة أعلاه ونقلها إلى لوحات جديدة، وترك مرة أخرى الآبار الأبعد فارغة.
ملاحظة: يتم حذف سلالة مراقبة الحدود JW5028 من مجموعة كيو 11 لجين زائفة الصغيرة التي لا ينبغي، ولم شاشات في كامل الجينوم لدينا، يحمل نظم المعلومات الجغرافية مع أي الجهات المانحة. لذلك، هذا هو جيد السلالة مراقبة إيجابية لتحديد حالات نادرة عندما الاقتران، تعلق، أو تحديدات قد فشلت، مما أدى إلى غياب أو متفرقة الحدود النمو التوتر. على سبيل المثال، إذا المستعمرات مراقبة الحدود لا تنمو بعد اختيار متحولة مزدوجة، سلالة الاستعلام قد لا تكون قادرة على تصريف الافعال، قد تم اقتران حاولتقد يكون NA شرط أن منعها من الحدوث (مثل المضادات الحيوية على صفيحة تحتوي على)، أو مضاد حيوي خاطئ تضاف إلى لوحة الاختيار. وبالمثل، إذا لوحظ عدد قليل جدا، والمستعمرات المتفرقة على لوحة كاملة، بما في ذلك الحدود، قد فشلت الاقتران أو تعلق الشاشة ويحتاج إلى تكرار. إذا كانت هذه هي النمو الحدود متفرقة استنساخه، فإنه قد تشير إلى الفقراء باستمرار اقتران المانحة وبالتالي الحالات التي لا تراعى المسوخ المزدوجة ربما بسبب فشل الاقتران، وليس صحيحا البيانات الجغرافية. ومثل هذه الشاشات تحتاج إلى التخلص من مزيد من التحليل. وعلاوة على ذلك، من خلال التواجد في جميع لوحات، وسلالة مراقبة الحدود يسمح البرنامج تكميم مستعمرة لتحديد المواقع تلقائيا مستعمرة على لوحات مزدوجة متحولة، وبالتالي المواقف مستعمرة ليس من الضروري أن يتم ترسيم يدويا. وأخيرا، فإن الإيدز سلالة مراقبة الحدود في تطبيع الأحجام داخل مستعمرة (على سبيل المثال صفوف مختلفة داخل لوحة) وbetwEEN (مثل لوحات مختلفة المتلقي يشبك مع الجهة المانحة نفسها في أوقات مختلفة) لوحات. - وبالمثل، وخلق بؤر المراقبة السلبية عن طريق إزالة أي سلالتين من موقع مختلف في كل لوحة (لا حدود) ونقل إلى لوحة سلالات جديدة. ملء هذه الآبار فارغة مع وسائل الإعلام التي تحتوي على LB 17 ميكروغرام / مل من سم. ومن المتوقع أن تحكم هذه البقع السلبية لتكون فارغة في المتلقي وبالتالي لوحات مزدوجة متحولة والمساعدة على ضمان عدم وجود أي أخطاء أو التعامل مع تجهيز لوحة الترقيم عند والتصوير، أو تعلق لوحات.
ملاحظة: هذه مطلوبة بقع فارغة الضوابط السلبية. الضوابط السلبية تساعد في تحديد حالات عندما فشل الاختيار - عندما سلالات المراقبة السلبية تنمو خلال التحديد. نقترح ليس فقط تحديد المواقع الفريدة المراقبة السلبية لكل من لوحات المتلقي، ولكن أيضا لاختيار المواقع المراقبة السلبية داخل نفس الربع من لوحة (على سبيل المثال اليمنى السفلى). بهذه الطريقة، ونمطوالبقع من المراقبة السلبية تحديد الأخطاء المحتملة لوحة سوء (يتم تسميتها لوحات معلقة على سبيل المثال أو يجري رأسا على عقب، حيث يتم شبك المستعمرة العلوي الأيسر في وضع اليمنى السفلى). - تطعيم سلالة كيو JW5028 11، مع حذف جين كاذب، إلى قارورة 500 مل مع LB 200 مل، يحتوي على 50 ميكروغرام / مل من كانساس وبناء على شاشاتنا eSGA كامل الجينوم، ونمو هذا المسخ خاصة هو الأقرب إلى البرية يتم استخدام نوع وبالتالي BW25113 وأنها مراقبة الحدود.
- زراعة سلالات العريض فضلا عن الثقافة بين عشية وضحاها في JW5028 32 ° C مع 190 دورة في الدقيقة المدارية تهز إلى OD ~ من 0،4 حتي 0،6 في 600 نانومتر. بعد النمو بين عشية وضحاها، وملء في الآبار الحدودية مع حوالي 80 ميكرولتر من JW5028 الثقافة بين عشية وضحاها.
- ويمكن استخدام لوحات تجميعها في الخطوة 3.2. بدلا من ذلك، على المدى الطويل تخزين، يكمل كل منهما جيدا في لوحات المتلقي مع الجلسرين 15٪، مزيج وسائل الإعلام والجلسرين، والحفاظ على لوحاتفي -80 ° C.
3. عالية الكثافة إجراء التزاوج سلالة لتوليد E. المسوخ القولونية مزدوجة
- تنمو سلالة المانحة HFR، مع الطفرة الاستعلام، مع وضع علامة سم، بين عشية وضحاها في 32 درجة مئوية في المتوسط الغنية السائل LB-CM (17 ميكروغرام / مل من سم) مع اهتزاز في 220 دورة في الدقيقة.
- دبوس جمع المتلقية أمر متحولة في 384 مستعمرة على لوحات الكثافة الصلبة LB تستكمل مع 50 ميكروغرام / مل من كانساس وفي موازاة ذلك، دبوس سلالة متحولة الاستعلام المانحة في 384 مستعمرة على كثافة نفس العدد من لوحات LB، على أن تستكمل مع 17 ميكروغرام / مل من سم. احتضان لوحات في ليلة وضحاها C. ° 32
ملاحظة: لوحات تستخدم لتعلق المتلقي قد تتطلب تصل إلى 36 ساعة للحصول على مستعمرات كبيرة بما فيه الكفاية لاتخاذ خطوات لاحقة. عندما متوسط حجم مستعمرة الحدود حوالي 2 مم في القطر (أو أكثر)، لوحات جاهزة للشبك. - لالمتقارن السلالات، يعلقون عليه من المانحين لوحة 384 مستعمرة المانحة ليلة وضحاها علىلوحة LB الصلبة. دبوس واحد في وقت لاحق لوحة المستفيدة 384 مستعمرة على مدى المانحة الطازجة معلقة على لوحة الاقتران. تواصل تعلق حتى لوحات كل المانح والمتلقي وكانت تعلق على مترافق بواسطة الصلبة لوحات اقتران LB. احتضان لوحات معلقة في الاقتران C ° 32 من 16 إلى 24 ساعة.
ملاحظة: فرد واحد سلالات متحولة المانحة قد تظهر الجينات الجين إلى التباين في كفاءة التزاوج بسبب الآثار المحتملة للطفرات على الاقتران، DNA كفاءة نقل، أو اللياقة البدنية. يمكن إيقاف النمو في أي وقت ما بين 16 و 24 ساعة، وعندما تم الحصول على مستعمرات كبيرة بما فيه الكفاية لاحقة الخطوات تعلق. الإقتران استمرت 16 حتي 24 ساعة إنتاج أعداد كافية من conjugants السابقين لنتائج eSGA استنساخه حتى بالنسبة للطفرات الجينات في وقت متأخر من نقل أو المسوخ مع انخفاض طفيف للياقة البدنية. - كل لوحة دبوس اقتران كثافة على 384 لوحة LB الصلبة واحدة، تحتوي على سم (17 ميكروغرام / مل) واساسه (50قد علقت ميكروغرام / مل) حتى جميع لوحات الاقتران. احتضان لوحات معلقة طازجة التحديد الأول ل16-36 ساعة عند C. ° 32
ملاحظة: في ما يخص الخطوات الاختيار، كميات مختلفة من الوقت قد تكون ضرورية لشاشات المانحة المختلفة. لأن جميع المسوخ مزدوجة في شاشة معينة لديها طفرة المانحة نفسها، في المتوسط، أبطأ متحولة المانحة المتزايد يؤدي إلى تباطؤ النمو طفرات مزدوجة. وهكذا، اعتمادا على اللياقة البدنية المانحة، قد تتطلب خطوات اختيار ما بين 16 و ساعة 36. يمكن إيقاف نمو مزدوج متحولة عندما المسوخ مزدوجة كبيرة بما فيه الكفاية للخطوة اللاحقة إما تعلق بعد اختيار الأول أو الثاني بعد التصوير. هذا يترجم عادة من الحدود المستعمرات السيطرة في المتوسط مع لا يقل عن 2 مم بأقطار. - إعادة يعلقون كل لوحة التحديد الأول على دواء، والثانية مزدوجة (سم واساسه)، لوحة التحديد في شكل مستعمرة-1536، بحيث يتم تمثيل كل مستعمرة من قبل أربعة التحديد الأول على المستعمراتلوحة اختيار الثاني. احتضان لوحات ل16-36 ساعة عند C. ° 32 تصوير لوحات مزدوجة النهائي اختيار متحولة لقياس كميا لياقة نمو متحولة وتحليل التفاعلات بين أزواج الجينات (الشكل 3).
ملاحظة: على الرغم من أن وتيرة merodiploidy (الكروموسومات الازدواجية جزئي) منخفض في حوالي 1/1 خلايا 000، 24، merodiploidy يمكن أن تحجب GI. وبالتالي، فإننا نوصي بما يعيد البيولوجي في جميع شاشات eSGA. لحسن الحظ، يمكن إعادة استخدامها لوحات بلاستيكية يمكن التخلص منها التجارية ومنصات تعلق تصل إلى ثلاث مرات من قبل تعقيم المواد على النحو التالي: يتم تجاهل آجار من لوحات ويتم تعقيم لوحات وكذلك منصات بواسطة نقع عليها في التبييض 10٪ بين عشية وضحاها، تليها شطف مع الماء المقطر، وغسل في الايثانول 70٪، والهواء تجفيف بلستيكور في غطاء تدفق تحت الضوء فوق البنفسجي. يتم تخزين معقمة ومنصات لوحات في حقائب مختومة بلاستيكية معقمة حتى الاستخدام.
4. معالجة البيانات واستخلاص النتائج GI
- قياس المستعمرات على كل لوحة في دفعة واسطة أو باستخدام التصوير فردي مستعمرة البرمجيات المتخصصة 3 و 9.
ملاحظة: A مناسبة جافا القائمة على التصوير مستعمرة عالية الإنتاجية وتطبيق التسجيل الآن متاحة بحرية للاطلاع الجمهور ( http://srcollins77.users.sourceforge.net/ ). البرنامج يعمل على منصات مختلفة (أي ماك أو PC) ويمكن إطلاقها إما للتنفيذ أو من نافذة الصالة. - تطبيع حجم مستعمرة الخام عن طريق تصحيح القياسات للتحيزات منتظمة داخل وبين لوحات مثل لوحة حافة الآثار، والآثار الاختلاف بين لوحة، صورة متفاوتة الإضاءة، والتحف بسبب انحناء سطح المادية للأجار، والآثار التنافس على المستعمرات المجاورة وعيوب ممكن تعلق ، فضلا عن الاختلافات في النمو مرة ف> 9. هذه التحف منهجية مستقلة عن اللياقة البدنية المزدوجة متحولة وينبغي تصحيحه كما أنها تؤدي إلى تقديرات زائفة اللياقة البدنية النمو.
- المستعمرات في الصفوف والأعمدة حافة تميل إلى أن تكون أكبر بسبب انخفاض المنافسة على المواد الغذائية من وجدت في وسط اللوحة. وهكذا، لتصحيح لهذا الغرض، توسيع نطاق أحجام المستعمرات حافة أن تكون مثل هذه أن حجم متوسط في هذا الصف أو العمود يساوي حجم متوسط من المستعمرات في وسط اللوحة.
- تحليل النتائج إحصائيا أن تأخذ في الاعتبار إمكانية استنساخ والتباين القياسية من الأحجام وسيطة من تكرار القياسات مستعمرة. على الأقل ثلاث تجارب مستقلة تكرار البيولوجية ضرورية لتقييم استنساخ التجارب.
- وأخيرا، استخدام أحجام النمو تطبيع مستعمرة اللياقة البدنية متوسط لإنشاء نقاط GI (S) لكل زوج الجينات باستخدام الصيغة التالية:
ن 1 "SRC =" / files/ftp_upload/4056/4056eq1.jpg "/>
حيث S = فار (فار التصدير X (ن إكسب -1) + فار المقاولات X (ن المقاولات -1)) / (ن + ن التصدير المقاولات -2)؛ فار = إكسب التباين الحد الأقصى لحجم مستعمرة للتطبيع متحولة مزدوجة؛ فار = متوسط المقاولات من الفروق في أحجام طبيعية مزدوجة مستعمرة متحولة من مجموعة المرجعية، ن = إكسب عدد من القياسات من أحجام مزدوجة مستعمرة متحولة، ن = عدد المقاولات وسيطة من خلال التجارب التجريبية يعيد للجميع؛ التصدير μ = متوسط أحجام مستعمرة تطبيع من المسوخ مزدوجة؛ والمقاولات μ = متوسط أحجام مستعمرة تطبيع للجميع المسوخ المزدوجة الناجمة عن سلالة متحولة متبرع واحد. وS-يعكس درجة الثقة الإحصائية للتفاعل digenic المفترضة فضلا عن قوة البيولوجي للتفاعل. إيجابية قوية S-عشرات تبين وجود آثار التخفيف، قuggesting أن الجينات تتفاعل المشاركة في المسار نفسه 9، في حين سلبي كبير S-عشرات تعكس المرض تركيبية أو الفتك، والتي غالبا ما توحي العضوية في مسارات متوازية زائدة 9. لتحديد مسار العلاقات على مستوى وظيفي، ويتم إنتاج واحد S-نقاط لكل زوج من الجينات اختبار من مجموعة البيانات النهائية.
ملاحظة: في دراستنا السابقة 9، وجدنا أن إعادة التركيب يميل إلى أن يحدث في كثير من الأحيان أقل بين الجينات في غضون 30 KBP عن بعضها البعض. لذلك، لتحليل المصب، بعد التطبيع وتوليد نقاط، ونحن إزالة التفاعلات بين الجينات في غضون 30 KBP عن بعضها البعض. بالإضافة إلى الجنود أنفسهم، ويمكن تحقيق العلاقات الوظيفية بين أزواج من الجينات من خلال النظر في كيفية مشابهة لمحات GI من الجينات هما. الملف الشخصى GI من الجين هو مجموعة من التفاعلات كل ذلك الجين مع جميع الجينات الأخرى في الجينوم خارجالجين منطقة الربط. الجينات مماثلة وظيفيا تميل إلى أن تكون أعلى من الارتباطات الجينية ملامح التفاعل 12 و 25. وهكذا، عن طريق حساب معاملات الارتباط لجميع الجينات في مجموعة البيانات التجريبية يمكن للمرء استخدام eSGA للتحقيق العلاقات الوظيفية بين الجينات حتى الكذب على مقربة من بعضها البعض على الكروموسوم.
النتائج
GIs reveal functional relationships between genes. Similarly, since genes in the same pathway display similar GI patterns and the GI profile similarity represents the congruency of phenotypes, we can group functionally related genes into pathways by clustering their GI profiles. Integrating GI and GI correlation networks with physical interaction information or other association data, such as genomic context (GC) relationships can also reveal the organization of higher-order functional modules that define core biological systems (and system crosstalk) in bacteria. For example, as with yeast4, 6, 7, 26, E. coli gene pairs exhibiting alleviating interactions that also have highly correlated GI profiles tend to encode proteins that are either physically associated (e.g. form a complex; Figure 4a) or that act coherently in a common biochemical pathway (e.g. components in a linear cascade). A representative example is shown in Figure 4b, where the components of the functionally redundant Isc and Suf pathways, which jointly participate in the essential Fe-S biosynthesis process, form distinct clusters that are linked together by extensive aggravating interactions (i.e. synthetic lethality). A statistical measure (e.g. hypergeometric distribution function27) can be used on GI or GI correlation data to find significant enrichment for interactions between and within pathways (Figure 4c). Cluster analysis can also be applied to functional networks derived using eSGA to predict the functions of genes lacking annotations5, 6, 9, 28-30 (Figure 4d). Since clustering algorithms vary, however, putative functional assignments determined through clustering require independent experimental verification.
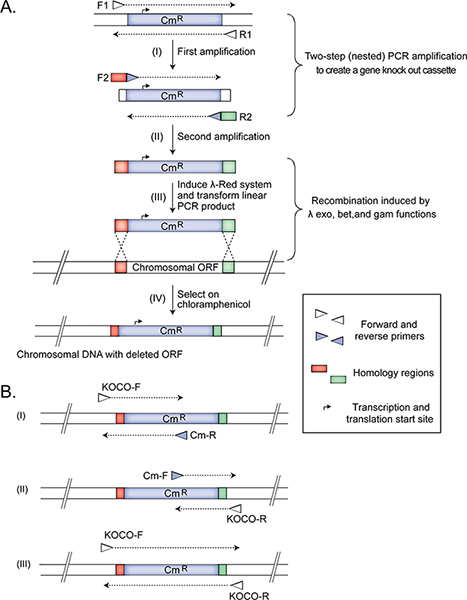
Figure 1. The construction and confirmation of Hfr Cavalli non-essential single gene deletion donor strains. The panels (adapted from14) illustrate the deletion of E. coli chromosomal ORF (A) and the three primer sets used for confirming (B) the correctly generated mutant Hfr Cavalli donor strain. Amplifications from a correctly constructed deletion donor strain should produce (i) 445 bp, (ii) 309 bp, and (iii) 1.4 kb products. See protocol text for details. Click here to view larger figure.

Figure 2. The construction and confirmation strategies of essential hypomorphic donor mutant strains in an Hfr Cavalli genetic background. The panels illustrate the creation of hypomorphic mutations of essential genes through recombineering technology (A) and the primer sets (B) used for the PCR confirmation of essential gene hypomorphic mutations. See protocol text for details.

Figure 3. Schematic summary of key eSGA steps. The Hfr donor (marked with chloramphenicol resistance, CmR) and recipient F- (marked with kanamycin resistance, KanR) mutant strains are grown in 384-colony density on LB-Cm and LB-Kan plates, respectively. The donor and the recipient strains are conjugated by pinning them over each other onto an LB plate, which is then incubated overnight at 32 °C. Consequently, the conjugants are pinned onto plates containing both Kan and Cm to select double mutants. The double mutant plates are digitally imaged and the growth fitness of the colony sizes is quantitatively scored to identify aggravating and alleviating interactions.

Figure 4. Representative computational analyses of genetic interaction (GI) scores for determining pathway-level functional relationships. (A) Panel I, Distribution of correlation coefficients between the GI profiles of gene pairs encoding proteins linked by protein-protein interactions (PPI) versus randomly drawn gene pairs. The p-value was computed using the two-sample Kolmogorov-Smirnov (KS) test; Panel II, Scatter plot of correlated genetic profiles from rich media (RM) for two transporters (mdtI, mdtJ) that form a heterodimeric complex required for spermidine excretion. (B) The hierarchical clustering of the GI sub-network adapted from Butland et al.9 highlights the functional connectivity between components of the previously known Isc and Suf pathways with similar GI patterns. Pink represents aggravating (negative S-score) interactions, green represents alleviating (positive S-score) interactions and black represents absence of GI. A predicted novel component of the SUF pathway, ydhD, displays GIs with the members of the Isc pathway. (C) Pathway cross-talk recorded among cell envelope processes that are significantly enriched for aggravating or alleviating GI according to the hypergeometric enrichment analysis27. (D) A GI sub-network predicts the role of two functionally unannotated genes, yceG and yebA, in peptidoglycan splitting based on their pattern of strong alleviating interactions with well-known cell division peptidoglycan hydrolases. The figures shown in panels A, C and D are adapted from Babu et al. (2011)12. Click here to view larger figure.
Discussion
وقد أوجزنا بروتوكول تدريجي لاستخدام الروبوتية eSGA الفحص الجيني للتحقيق في وظائف البكتيرية على مستوى المسار من خلال استجواب GI. ويمكن استخدام هذا النهج لدراسة الجينات الفردية وكذلك في النظم البيولوجية بأكمله E. القولونية. تنفيذ الخطوات بعناية التجريبية المذكورة أعلاه، بما في ذلك جميع الضوابط المناسبة، وتحليل دقيق والتحقق من صحة البيانات بشكل مستقل GI هي الجوانب الرئيسية لنجاح eSGA في تحقيق اكتشافات جديدة وظيفية. بالإضافة إلى eSGA، وهي طريقة مشابهة لدراسة المفهوم GI في E. كولاي، ووصف GIANT-القولونية 17 لا يمكن استخدامها لإضاءة الرواية العلاقات الوظيفية التي غاب في كثير من الأحيان المناهج الأخرى، مثل البروتين 23 أو 13 المظهري شاشات وحدها. ومع ذلك، كما هو الحال مع أي نهج الجينوم على نطاق والقيود موجودة على تطبيق eSGA أو الإشريكية GIANT، على سبيل المثال لالأنواع البكتيرية الأخرى. هذا ويكون جزئياسبب عدم وجود الجينوم على نطاق حذف جين واحد سلالة متحولة مجموعات لمعظم الأنواع البكتيرية، وخاصة بالنسبة للأنواع التي أعاقت الطفرات المستهدفة حسب التردد طبيعية منخفضة من إعادة التركيب مثلي. في مثل هذه الحالات، يمكن أن يحتمل تعطل الجينات باستخدام أساليب مثل المقايسات الطفرات العشوائية القائمة على تتبعها مثل TnSeq 31 أن تستخدم للتحقيق في وظائف الجينات البكتيرية. لمحة عامة عن بديل البكتيرية الناشئة إجراءات الفحص الجيني، انظر ورقة مراجعة من قبل Gagarinova وEmili (2012) 32.
على الرغم من أن كثيرا ما تكشف أساليب GI اتصالات كثيرة مثيرة للاهتمام يمكن أن توحي الرواية الروابط الآلية، ودمج هذه البيانات مع معلومات أخرى مثل التنميط المظهري 13، شبكات التفاعل الجسدي 22، 23، والجمعيات GC-الاستدلال تكون مفيدة بشكل خاص. على سبيل المثال، كما هو الحال مع الشبكات GI والجمعيات GC، والتي تقوم على الحفظمن أجل الجين (operons)، اندماج الجينات البكتيرية، الترددات إعادة التركيب OPERON المستمدة من مسافات بين الجينات من الجينوم عبر operons توقع، وكذلك يمكن النشوء والتطور التنميط 33 تقديم رؤى إضافية في كيفية خلية بكتيرية تنظم مجمعات البروتين إلى مسارات وظيفية للتوسط وتنسيق الرئيسية العمليات الخلوية 12 و 23.
E. القولونية هو نموذج العمود الفقري لفهم البيولوجيا الجزيئية من البكتيريا سالبة الجرام الأخرى. تحقيقا لهذه الغاية، يمكن استخدام البيانات GI eSGA جنبا إلى جنب مع معلومات علم الجينوم المقارن (مثل التنميط النشوء والتطور، التنميط الجيني التعبير) للتحقيق في حفظ التطورية للعلاقات الوظيفية التي اكتشفها eSGA عبر الأنواع الأخرى proteobacterial والأصناف بروكاريوتيك. وعلاوة على ذلك، فإن البيانات الناتجة عن التفاعل E. ويمكن استخدام القولونية إلى التبصر في العمارة مسار الميكروبات اكتشفه metagenoالبلدان المتوسطة الدخل، والتي تعاني من نقص وظيفي شروحه. من الحفظ على نطاق واسع منذ العديد من الجينات في جميع الميكروبات 23 عاما، وأنه يمكن تعزيز الحساسية للمضادات الحيوية بواسطة بعض الاضطرابات الوراثية 34 يجوز، حتى يحتمل أن تكون العلاقات الوظيفية تنيره eSGA يمكن استغلالها لتصميم علاجات العقاقير المبتكرة الجمع.
Disclosures
الإعلان عن أي تضارب في المصالح.
Acknowledgements
وأيد هذا العمل من جانب صناديق من كندا الجينوم، ومعهد أونتاريو علم الجينوم، والمعاهد الكندية لأبحاث الصحة لمنح JG وAG AE هي المستفيدة من منحة الدراسات العليا فانييه كندا.
Materials
| Name | Company | Catalog Number | Comments |
| I. Antibiotics | |||
| Chloramphenicol | Bioshop | #CLR201 | |
| Kanamycin | #KAN201 | ||
| Ampicillin | # AMP201 | ||
| 2. Luria-Bertani medium | |||
| LB powder | Bioshop | #LBL405 | |
| Agar | Bioshop | #AGR003 | |
| 3. Bacterial Strains and Plasmids | |||
| Hfr Cavalli strain λred system (JL238) | Babu et al.14. | ||
| pKD3 | E. coli Genetic Stock Centre, Yale | ||
| Keio E. coli F- recipient collection | National BioResource Project (NBRP) of Japan11 | ||
| Hypomorphic E. coli F- SPA-tag strains | Open biosystems; Babu et al.14 | ||
| 4. Primers | |||
| pKD3-based desalted constant primers | F1: 5'-GGCTGACATGGGAATTAGC-3' R1: 5'-AGATTGCAGCATTACACGTCTT-3' | ||
| Desalted custom primers | Cm-R: 5'-TTATACGCAAGGCGACAAGG-3' Cm-F: 5'- GATCTTCCGTCACAGGTAGG-3' | ||
| Desalted custom primers | F2 and R2: 20 nt constant regions based on pKD3 sequence and 45 nt custom homology regions F2 constant region: 5'-CATATGAATATCCTCCTTA-3' R2 constant region: 5'-TGTGTAGGCTGGAGCTGCTTC-3'S1 and S2: 27 nt constant regions for priming the amplification of the SPA-Cm cassette and 45 nt custom homology regions S1 constant region: 5'AGCTGGAGGATCCATGGAAAAGAGAAG -3' S2 constant region: 5'- GGCCCCATATGAATATCCTCCTTAGTT -3' KOCO-F and KOCO-C: 20 nt primers 200 bp away from the non-essential gene deletion site or the essential gene SPA-tag insertion site | ||
| 5. PCR and Electrophoresis Reagents | |||
| Taq DNA polymerase | Fermentas | # EP0281 | |
| 10X PCR buffer | Fermentas | # EP0281 | |
| 10 mM dNTPs | Fermentas | # EP0281 | |
| 25 mM MgCl2 | Fermentas | # EP0281 | |
| Agarose | Bioshop | # AGA002 | |
| Loading dye | NEB | #B7021S | |
| Ethidium bromide | Bioshop | # ETB444 | |
| 10X TBE buffer | Bioshop | # ETB444.10 | |
| Tris Base | Bioshop | # TRS001 | |
| Boric acid | Sigma | # T1503-1KG | |
| 0.5 M EDTA (pH 8.0) | Sigma | # B6768-500G | |
| DNA ladder | NEB | #N3232L | |
| 6. DNA isolation and Clean-up Kits | |||
| Genomic DNA isolation and purification kit | Promega | #A1120 | |
| Plasmid Midi kit | Qiagen | # 12143 | |
| QIAquick PCR purification kit | Qiagen | #28104 | |
| 7. Equipment for PCR, Transformation and Replica-pinning | |||
| Thermal cycler | BioRad, iCycler | ||
| Agarose gel electrophoresis | BioRad | ||
| Electroporator | Bio-Rad GenePulser II | ||
| 0.2 cm electroporation cuvette | Bio-Rad | ||
| 42 °C water bath shaker | Innova 3100 | ||
| Beckman Coulter TJ-25 centrifuge | Beckman Coulter | ||
| 32 °C shaker | New Brunswick Scientific, USA | ||
| 32 °C plate incubator | Fisher Scientific | ||
| RoToR-HDA benchtop robot | Singer Instruments | ||
| 96, 384 and 1,536 pin density pads | Singer Instruments | ||
| 96 or 384 long pins | Singer Instruments | ||
| 8. Imaging Equipments | |||
| Camera stand | Kaiser | ||
| Digital camera, 10 megapixel | Any Vendor | ||
| Light boxes, Testrite 16" x 24" units | Testrite | ||
| 9. Pads or Plates Recycling | |||
| 10% bleach | Any Vendor | ||
| 70% ethanol | Any Vendor | ||
| Sterile distilled water | Any Vendor | ||
| Flow hood | Any Vendor | ||
| Ultraviolet lamp | Any Vendor | ||
| 10. Labware | |||
| 50 ml polypropylene tubes | Any Vendor | ||
| 1.5 ml micro-centrifuge tubes | Any Vendor | ||
| 250 ml conical flaks | VWR | # 29140-045 | |
| 15 ml sterile culture tubes | Thermo Scientific | # 366052 | |
| Cryogenic vials | VWR | # 479-3221 | |
| Rectangular Plates | Singer Instruments | ||
| 96-well and 384-well microtitre plates | Singer Instruments | Nunc | |
| Plate roller for sealing multi-well | Sigma | #R1275 | |
| plates | ABgene | # AB-0580 | |
| Adhesive plate seals | Fisher Scientific | # 13-990-14 | |
| -80 °C freezer | Any Vendor | ||
References
- Bandyopadhyay, S., Kelley, R., Krogan, N. J., Ideker, T. Functional maps of protein complexes from quantitative genetic interaction data. PLoS Comput. Biol. 4, e1000065 (2008).
- Costanzo, M., Baryshnikova, A., Myers, C. L., Andrews, B., Boone, C. Charting the genetic interaction map of a cell. Curr. Opin. Biotechnol. 22, 66-74 (2011).
- Costanzo, M., et al. The genetic landscape of a cell. Science. 327, 425-4231 (2010).
- Fiedler, D., et al. Functional organization of the S. cerevisiae phosphorylation network. Cell. 136, 952-963 (2009).
- Roguev, A., et al. Conservation and rewiring of functional modules revealed by an epistasis map in fission yeast. Science. 322, 405-4010 (2008).
- Schuldiner, M., et al. Exploration of the function and organization of the yeast early secretory pathway through an epistatic miniarray profile. Cell. 123, 507-519 (2005).
- Wilmes, G. M., et al. A genetic interaction map of RNA-processing factors reveals links between Sem1/Dss1-containing complexes and mRNA export and splicing. Mol. Cell. 32, 735-746 (2008).
- Babu, M., et al. Systems-level approaches for identifying and analyzing genetic interaction networks in Escherichia coli and extensions to other prokaryotes. Mol. Biosyst. 12, 1439-1455 (2009).
- Butland, G., et al. coli synthetic genetic array analysis. Nat. Methods. 5, 789-7895 (2008).
- Typas, A., et al. Regulation of peptidoglycan synthesis by outer-membrane proteins. Cell. 143, 1097-10109 (2010).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Mol. Syst. Biol. 2, 2006-200008 (2006).
- Babu, M., et al. Genetic interaction maps in Escherichia coli reveal functional crosstalk among cell envelope biogenesis pathways. PLoS Genet. 7, e1002377 (2011).
- Nichols, R. J., et al. Phenotypic landscape of a bacterial cell. Cell. 144, 143-156 (2011).
- Babu, M., Gagarinova, A., Greenblatt, J., Emili, A. Array-based synthetic genetic screens to map bacterial pathways and functional networks in Escherichia coli. Methods Mol Biol. 765, 125-153 (2011).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc. Natl. Acad. Sci. U.S.A. 97, 6640-665 (2000).
- Yu, D., et al. An efficient recombination system for chromosome engineering in Escherichia coli. Proc. Natl. Acad. Sci. U.S.A. 97, 5978-5983 (2000).
- Typas, A., et al. quantitative analyses of genetic interactions in. E. coli. Nat. Methods. 5, 781-787 (2008).
- Zeghouf, M., et al. Sequential Peptide Affinity (SPA) system for the identification of mammalian and bacterial protein complexes. J. Proteome Res. 3, 463-468 (2004).
- Davierwala, A. P., et al. . , 1147-1152 (2005).
- Breslow, D. K., et al. A comprehensive strategy enabling high-resolution functional analysis of the yeast genome. Nat. Methods. 5, 711-718 (2008).
- Babu, M., et al. Sequential peptide affinity purification system for the systematic isolation and identification of protein complexes from Escherichia coli. Methods Mol. Biol. 564, 373-400 (2009).
- Butland, G., et al. Interaction network containing conserved and essential protein complexes in Escherichia coli. Nature. 433, 531-537 (2005).
- Hu, P., Janga, S. C., Babu, M., Diaz-Mejia, J. J., Butland, G., et al. Global functional atlas of Escherichia coli encompassing previously uncharacterized proteins. PLoS Biol. 7, (2009).
- Anderson, R. P., Roth, J. R. Tandem genetic duplications in phage and bacteria. Annu. Rev. Microbiol. 31, 473-505 (1977).
- Boone, C., Bussey, H., Andrews, B. J. Exploring genetic interactions and networks with yeast. Nat. Rev. Genet. 8, 437-449 (2007).
- Collins, S. R., et al. Functional dissection of protein complexes involved in yeast chromosome biology using a genetic interaction map. Nature. 446, 806-8010 (2007).
- Le Meur, N., Gentleman, R. Modeling synthetic lethality. Genome Biol. 9, R135 (2008).
- Tong, A. H., et al. Systematic genetic analysis with ordered arrays of yeast deletion mutants. Science. 294, 2364-2368 (2001).
- Tong, A. H., et al. Global mapping of the yeast genetic interaction network. Science. 303, 808-813 (2004).
- Wong, S. L., et al. Combining biological networks to predict genetic interactions. Proc. Natl. Acad. Sci. U.S.A. 101, 15682-15687 (2004).
- van Opijnen, T., Bodi, K. L., Camilli, A. Tn-seq: high-throughput parallel sequencing for fitness and genetic interaction studies in microorganisms. Nat. Methods. 6, 767-772 (2009).
- Gagarinova, A., Emili, A. Genome-scale genetic manipulation methods for exploring bacterial molecular biology. Mol. Biosyst. 8, 1626-1638 (2012).
- Dewey, C. N., et al. Positional orthology: putting genomic evolutionary relationships into context. Brief Bioinform. 12, 401-412 (2011).
- St Onge, R. P., et al. Systematic pathway analysis using high-resolution fitness profiling of combinatorial gene deletions. Nat. Genet. 39, 199-206 (2007).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved