Method Article
Mapping Bakterielle Functional Netzwerke und Pathways in
In diesem Artikel
Zusammenfassung
Systematische, groß angelegte synthetische genetische (Gen-Gen-oder Epistase) Interaktion Bildschirme können verwendet werden, um genetische Redundanz und Weg cross-talk zu erkunden. Hier haben wir eine High-Throughput-quantitative synthetischen genetischen array Screening-Technologie beschreiben, bezeichnet ESGA dass wir für die Aufklärung epistatische Beziehungen und erforschen genetische Interaktion Netze in den entwickelten Escherichia coli.
Zusammenfassung
Phänotypen durch eine komplexe Reihe von physikalischen (zB Protein-Protein) und funktionelle (zB Gen-Gen oder genetisches) Wechselwirkungen (GI) 1 bestimmt. Während körperliche Interaktionen können angeben, welche bakterielle Proteine als Komplexe verbunden sind, müssen sie nicht unbedingt enthüllen Weg-level funktionalen Beziehungen1. GI Bildschirmen, in denen das Wachstum der Doppelmutanten, die zwei gelöschte oder inaktivierte Gene gemessen und verglichen mit den entsprechenden einzelnen Mutanten, beleuchten kann epistatischen Abhängigkeiten zwischen Loci und somit ein Mittel zum Abfragen und entdecken neuartigen funktionalen Beziehungen 2. Large-scale GI Karten für eukaryotische Organismen wie Hefe 3-7 berichtet worden, aber GI Informationen bleiben spärlich für Prokaryoten 8, die die funktionellen Annotation von bakteriellen Genome behindert. Zu diesem Zweck haben wir und andere Hochdurchsatz-quantitative bakteriellen GI Screeningverfahren 9, 10 entwickelten Hier präsentieren wir die wichtigsten Schritte erforderlich, um quantitative E. durchführen coli Genetic Synthetische Array (ESGA) Screening-Verfahren auf einem genomweite 9, mit natürlichen bakteriellen Konjugation und homologe Rekombination, um systemisch zu erzeugen und zu messen, das die Eignung einer großen Anzahl von Doppel-Mutanten in einer Kolonie Anordnungsformat. Kurz gesagt, wird ein Roboter verwendet, um zu übertragen durch Konjugation, Chloramphenicol (Cm) - F-markierten Rezipientenstämme - Mutante Allele aus engineered Hfr (High Häufigkeit der Rekombination) 'Donorstämme' in eine geordnete Anordnung von Kanamycin (Kan) markiert. Normalerweise verwenden wir loss-of-function einzelnen Mutanten tragen nicht wesentlicher Deletionen (zB die "Keio" Sammlung 11) und essentielles Gen hypomorphen Mutationen (dh Allele verleihen reduzierte Protein Expression, die Stabilität oder Aktivität 9, 12, 13) fragen die funktionalen Zusammenhänge der nicht-essentielle und essentielle Gene, resziehungsweise. Nach Konjugation und anschließenden genetischen Austausch durch homologe Rekombination vermittelten werden die resultierenden Doppelmutanten auf festem Medium, das sowohl Antibiotika ausgewählt ist. Nach Auswachsen werden die Platten digital bebildert und Koloniegrößen quantitativ erzielt unter Verwendung einer in-house automatisierten Bildverarbeitungssystem 14. GIs enthüllt werden, wenn die Wachstumsrate einer Doppelmutante entweder signifikant besser oder schlechter als erwarteten 9 ist. Erschwerende (oder negative) GIs oft zwischen loss-of-function Mutationen in Paaren von Genen aus kompensatorischen Wege, die auf der gleichen grundlegenden Prozess 2 auftrifft führen. Hier wird der Verlust eines einzelnen Gens gepuffert, so dass entweder einzelne Mutante lebensfähig ist. Allerdings ist der Verlust der beiden Signalwege schädlichen und resultiert in synthetischen Letalität oder Krankheit (dh langsame Wachstum). Umgekehrt kann Linderung (oder positiven) Wechselwirkungen zwischen Genen in der gleichen Weg oder Proteinkomplexes 2 als der auftretenDeletion von entweder Gen allein oft ausreicht, um die normale Funktion des Weges oder komplexer, so dass zusätzliche Störungen nicht verringern Aktivität und damit Wachstum, weiter stören. Insgesamt lassen sich systematisch identifiziert und analysiert GI-Netzwerke bieten unvoreingenommene, globale Karten der funktionalen Zusammenhänge zwischen einer großen Anzahl von Genen, von denen Signalweg-Level-Informationen durch andere Ansätze verpasst 9 entnommen werden kann.
Protokoll
Ein. Konstruieren Hfr Cavalli Donor Mutantenstämme durch Recombineering 15, 16
Die Schritte zum Aufbau der ESGA Donor Flecken werden nachfolgend beschrieben. Kurz gesagt, werden wir gezielt λ - Red vermittelt homologe Rekombination 16 der verstärkten wählbaren DNA-Marker-Kassette Fragmente durch PCR nicht-essentielle Gen-Deletionsmutanten (Abschnitt 1.1) oder essentielle Gen hypomorphen mutierten Donorstämme (Abschnitt 1.2), die dann als verwendet werden erstellen "Abfragen", um GI-Netzwerken definieren.
Hinweis: Während der technologischen Entwicklung, untersuchten wir die Wirksamkeit der Verwendung Hfr-vermittelte konjugativen Transfer für die Kombination von Mutationen mit einem wohldefinierten Hfr Donorstamm (Hfr Cavalli, Hfr Hayes und Hfr 3000). Wir untersuchten (i) die Fähigkeit, effizient zu machen Spenders Mutanten mit der Rekombination Verfahren Pionierarbeit von Yu et al. (2000) 16, (ii) die relativen Wirkungsgradeder konjugative Transfer von DNA verschiedene chromosomale Marker, und (iii) die aus der Abfrage und chromosomale Gen Position Orientierung relativ zu der Übertragung Hfr Locus, oriT. Wir fanden, dass λ - Red-vermittelte homologe Rekombination Effizienz war deutlich höher in Hfr Cavalli als in Hfr Hayes oder Hfr3000. Bei Bedarf kann P1-Transduktion oder ein Psuedo Hfr Stamms 17, verwendet werden, um mutierte Spender zu schaffen. Wir haben erfolgreich alle diese Verfahren zur Herstellung und Screening einer großen Anzahl von Spendern ausgelegt ist. Da in unserer Studie Konjugation Experimenten beobachteten die gesamte Effizienz des Transfers und die Anzahl der Ex-Konjuganten war signifikant größer Hfr Cavalli, wurde diese besondere Belastung Hintergrund für Spender Bau und großen ESGA gewählt. Alle Verfahren für ESGA Bildschirme mit Hfr Cavalli Geber beschrieben.
- Amplifizieren des DNA-Fragments für die anschließende Rekombination um eine nicht-essentielle Gen löschen
Beschäftigen zwei-Schritt-nested PCR Amplibeschreibung (Abbildung 1) zur Schaffung eines Gendeletion Kassette zum Auswechseln des Target offenen Leserahmen mit einer Cm Resistenzmarker vom pKD3 Plasmid (GI: 15.554.330; Protein-ID: AAL02033.1) 15. Der Marker wird durch den FRT-Stellen 11, der die Entfernung des Resistenzgens, falls erforderlich, in Weiterverfolgung Experimenten flankiert.
Anmerkung: Es sind Zwei-Schritt-PCR verschachtelt, um das Plasmid aus dem amplifizierten Produkt, das anschließend verwendet wird, um die Bakterienzellen zu transformieren entfernen. Entfernen der Plasmid (in der ersten PCR) und dann Erstellen des Gen-Knockout-Kassette in der zweiten PCR ist notwendig, da die Transformation mit einem Plasmid, ist viel effizienter als Rekombinationstechnologie, und das Ziel ist, erhalten - soweit wie möglich - nur die Stämme haben erfolgreich ersetzt die zielgerichtete genomische DNA mit dem Selektionsmarker. Eine Alternative zu nested PCR ist es, einen Restriktionsverdau des Plasmids außerhalb des Bereichs in der zweiten PCR durchzuführenfort. Dann wird das Produkt aus dem Restriktionsverdau wird gereinigt und als eine Matrize in der verschachtelten PCR-Schritt (1.1.2). Diese Option ist auch einfach, erfordert aber mehr Arbeit up-Zeit.- Verwenden etwa 45 ng pKD3 als Matrize in PCR mit Vorwärts und Rückwärts-F1 R1 Primer eine 1070 bp-Produkt zu erzeugen. Reinigen das PCR-Fragment mit Hilfe des Qiagen PCR Aufreinigungsprotokolls und verdünnt das Produkt in sterilem destilliertem Wasser auf 5 ng / ul Konzentration.
- Verwendung des gereinigten PCR-Produkts als Matrize, eine zweite PCR mit genspezifischen (zB Knockout) verschachtelte Primer, F2 und R2, die (i) 45 nt der genspezifischen Homologie Regionen am 5'-Enden so eingestellt, dass für die homologe Rekombination unmittelbar stromaufwärts und stromabwärts des Zielgens es mit dem Cm Resistenzmarker und (ii) 20 nt an den 3'-Enden, um die Synthese des Markers Cm primen ersetzen. Die Größe der erzeugten Fragment ist 1123 bp. Die genspezifischen Homologiebereiche ausgebildet sind, um das Ziel zu entfernen open Leseraster, während die benachbarten oder teilweise überlappend kodierenden Segmenten intakt. Nach dieser Verstärkung, fahren Sie mit Schritt 1.3.
- Verstärken eines Tagging-Kassette für die Schaffung eines essentiellen Gens hypomorphen Mutation
- Um hypomorphen Donor Mutantenstämme konstruieren, einen C-terminalen Peptid sequentiellen Affinität (SPA) Fusions-Tag, der oft leicht beeinflusst essentielles Protein Funktion durch destabilisierenden Transkript Fülle (dh Protein Kopienzahl) oder Proteinfaltung / Aktivität. Zu diesem Zweck erzeugen einen selektierbaren SPA-tag (SPA-Cm) DNA-Template durch Umwandeln des Kan Resistenzmarker in pJL148 18 bis Cm Verwendung Rekombinationstechnologie zwischen identischen Nukleotidsequenzen flankierenden Kan Markers in pJL148 und Cm Markers in pKD3 15. Die resultierende Vorlage besteht aus In-Frame-SPA-Tag durch einen Cm Resistenzmarker gefolgt, und ist geeignet für die PCR-Amplifikation und anschließende Rekombinationstechnologie ESGA 12.
Hinweis: </ Strong> Essential Gene sind möglicherweise die interessantesten für die Gen-Funktions-Studien. Da diese Gene nicht gelöscht werden kann, musste innovative Methoden für die Untersuchung der funktionellen Beziehungen von essentiellen Genen entwickelt werden. Mehrere geeignete höchst erfolgreiche Ansätze wurden zunächst in Hefe entwickelt. Beispielsweise Davierwala et al. (2005) 19 durchgeführt GI-Bildschirme mit einem Panel von 575 temperaturempfindlichen essentielles Gen-Mutanten, und festgestellt, dass essentielle Gene etwa das Fünffache der Anzahl der GIs als essentiellen Genen ausgestellt. Ebenso Breslow et al. (2008) 20 entwickelt eine "verminderte Häufigkeit von mRNA Störung" (DAMP) Ansatz, um eine Bibliothek von hypomorphen Allele abdeckt ~ 82% ätherisches Hefegene, Wege und Komplexe zu untersuchen generieren. Die feuchten Ansatz ändert das 3'-Ende der mRNA exprimiert, wodurch das Niveau des Zielgens 6, wahrscheinlich aufgrund einer Destabilisierung der mRNA. Ähnlich wie bei der feuchten Ansatz 6, unseren strategy ist, um die subtilen Effekte von störenden das 3 'Ende des exprimierten mRNA von Proteinen zu nutzen.
Wie wir weiter unten beschrieben, wird das Tag von selbst nicht wahrscheinlich beeinträchtigt Proteinfaltung oder-Funktion, aber die subtile Störung der mRNA durch die Tag genügt, wenn sie mit Umweltstressoren oder andere Mutationen kombiniert werden, um funktionell informativen Gen-Umwelt Gen 13 und offenbaren -Gen 9, 12 Interaktionen.
Erstens, bisher haben wir SPA-tags bis ~ 3.000 essentielle und nicht-essentielle E. coli Proteine 21-23 reinigen, mit über 80% der 307 E. coli essentielle Proteine unter unseren SPA-markierten Stämme 8 dargestellt wird. Weil das Tag wurde erfolgreich verwendet, um Protein-Komplexe bekannt, die wechselseitig validiert wurden (dh die gleichen Komplexes durch Tagging isoliert werden konnte und Reinigung verschiedener Proteine aus dem gleichen Komplex) zu isolieren, und die isolierten Komplexe waren biologisch relevanten, wir Grund, dass die Tags tunnormalerweise keinen Einfluss Proteinfaltung oder Funktion 12. Ebenso zeigte unsere unveröffentlichte Beobachtungen, dass der Tag selbst in der Regel nicht beeinflussen Belastung Wachstum. Allerdings begründeten wir, dass Veränderung der 3'-UTR durch die Integration der Tag und die Markierung Kassette, wie für feuchte Stämme 6 berichtet wurde, könnten bestimmte Transkripte auf RNA-Ebene zu destabilisieren oder in seltenen Fällen behindern die korrekte Faltung oder der Funktion des Fusionsproteine. Deshalb sagten wir voraus, dass die Kombination von solchen gestörten Allele von essentiellen Genen mit anderen funktionell relevante Mutationen an anderer Stelle im Genom würde informative GIs, die in der Tat 9, 12 beobachtet wurde führen. Weiterhin SPA-markierten Allele von essentiellen Gene zeigten Gen-Umwelt Wechselwirkungen (zB Arzneimittelüberempfindlichkeit) gemäß der phänotypischen Profilierarbeiten von Nichols et al. (2011) 13, der eine Teilmenge von unterworfen SPA-tagged wesentlichen Stämme zu einer Vielzahl von Wachstumsbedingungen. Die gleiche Studie confirmed, dass die meisten (54 von 58 getesteten) SPA-markierten wesentliche Stämme zeigten beeinträchtigt Fitness in einem oder mehreren Kulturbedingungen 13, die die Vorstellung, dass der SPA-tag schafft oft mild hypomorphen Allele in essentiellen Genen, die nützlich sind für genomweite unterstützt GI-Screen-Anwendungen einschließlich ESGA. - Verstärken des Tagging Kassette aus dem SPA-Cm Vorlage mit genspezifischen Primern S1 und S2, die jeweils (i) eine 45 nt genspezifischen Region für eine homologe Rekombination am 5'-Ende, gefolgt von (ii) ein 27 bp-SPA cm spezifischen Sequenz am 3 'Ende.
- Um hypomorphen Donor Mutantenstämme konstruieren, einen C-terminalen Peptid sequentiellen Affinität (SPA) Fusions-Tag, der oft leicht beeinflusst essentielles Protein Funktion durch destabilisierenden Transkript Fülle (dh Protein Kopienzahl) oder Proteinfaltung / Aktivität. Zu diesem Zweck erzeugen einen selektierbaren SPA-tag (SPA-Cm) DNA-Template durch Umwandeln des Kan Resistenzmarker in pJL148 18 bis Cm Verwendung Rekombinationstechnologie zwischen identischen Nukleotidsequenzen flankierenden Kan Markers in pJL148 und Cm Markers in pKD3 15. Die resultierende Vorlage besteht aus In-Frame-SPA-Tag durch einen Cm Resistenzmarker gefolgt, und ist geeignet für die PCR-Amplifikation und anschließende Rekombinationstechnologie ESGA 12.
- Bestätigung und Reinigen des PCR-Produkts
- Bestätigen, dass das Tagging Kassette korrekt von 2-5 ul Unterwerfen des PCR-Produkts eine Agarose-Gelelektrophorese und Reinigung der restlichen DNA nach Angaben des Herstellers Standard-PCR Aufreinigungsprotokolls (Qiagen) amplifiziert. Eluieren das PCR-Produkt in 30 ul sterilem destilliertem Wasser lösen und auf ~ 50 ng / ul Konzentration. Die Aufreinigunged Produkt kann sofort in der Transformation verwendet werden (Schritt 1.5) oder bei -20 ° C für bis zu 6 Monate bis zur Verwendung.
- Vorbereiten Hfr Cavalli kompetenten Zellen für Spender Bau
- Beimpfen ein ml der gesättigten Übernachtkultur eines Hfr Cavalli Stamm in 70 ml frischem Luria-Bertani (LB)-Medium mit 35 ul von 50 ug / ml Ampicillin in einer 250 ml Flasche. Inkubieren der Kultur bei 32 ° C unter Schütteln bei 220 rpm, bis eine optische Dichte (OD) 600 nm von ~ 0,5 bis 0,6 erreicht ist (~ 2 h).
- Übertragung der Kultur in ein Wasserbad für Wärme Induktion des λ rot Rekombinationssystem 16 bei 42 ° C für 15 min unter Schütteln bei 160 UpM. Stoppen des Induktion durch Überführen der Kultur auf eine gekühlte Eis-Aufschlämmung Wasserbad für 10 bis 20 min bei 160 UpM. Achten Sie darauf, um die Zellen Kälte von diesem Punkt bis nach der Transformation, die Nutzung Rohre und Küvetten auf Eis wurden für mindestens 15 min gekühlt vor der Verwendung.
- Teilen Sie die cULTUR gleichmäßig in 2 vorgekühlte 50 ml Polypropylenröhrchen (~ 35 ml Kultur pro Röhrchen) und Zentrifuge bei 4.400 xg für 6 Minuten bei 4 ° C.
- Überstand verwerfen und erneut zu suspendieren das Zellpellet durch vorsichtiges Schwenken in ~ 50 ml eiskaltem sterilem destilliertem Wasser. Zentrifugation der Zellen erneut bei 4400 xg für 6 Minuten bei 4 ° C. Überstand verwerfen und wieder auszusetzen jedes Zellpellet in 20 ml eiskaltem, sterilen 10% Glycerin und zentrifugieren wie oben beschrieben.
- Den Überstand umfüllen und wieder aussetzen Zellpellet in 500 ul eiskaltem 10% Glycerin. Aliquot die vorbereiteten kompetenten Zellen in 50 ul Volumen in einzelne, vorgekühlte 1,5 ml Mikrozentrifugenröhrchen für den sofortigen Transformation. Die Zellen können auf Trockeneis oder in flüssigem Stickstoff eingefroren und bei -80 ° C für bis zu 3 Monate. Allerdings ist Transformationseffizienz höchsten mit frisch zubereiteten kompetenten Zellen.
- Transformation
- Fügen Sie 100 ng gereinigtes PCR-Produkt ausSchritt 1.3.1 die kompetenten Zellen. Flick die Röhre und ermöglichen Suspension auf Eis für 5 min stehen lassen.
- Übertragen Sie die Aussetzung der eiskalten zuständigen Zellen und DNA, um ein vorgekühltes Elektroporationsküvette. Elektroporieren Zellgemisch mit dem 2,5 kV, 25 uF, 200 Ohm Einstellung in einem Abstand von 2 mm Küvette (dh wenn Sie eine andere Küvette entnehmen Sie bitte den Anweisungen des Herstellers für die Elektroporation Einstellungen), und sofort 1 ml der Raumtemperatur SOC-Medium. Übertragen der elektroporierten Zellen in SOC-Medium in eine 15 ml-Kultur Rohr und inkubiere bei 32 ° C für 1 Stunde unter kreisförmigen Schütteln bei 220 UpM.
- Nach der Inkubation der Zellen zentrifugieren bei 4.400 × g für 5 min bei Raumtemperatur. Entfernen ungefähr 850 ul des Überstandes und Resuspendieren des Zellpellets in der verbleibenden Flüssigkeit.
- Verbreiten Sie die Zellen auf vorgewärmten LB-Platten mit 17 ug / ml Cm, und inkubieren bei 32 ° C über Nacht. Wählen Sie zwei einzelne Transformantenkolonien und streifenfrei auf LB-CM-Platten für die Mutante Bestätigung. Wir empfehlen die Vermeidung größten Transformantenkolonien um die Möglichkeit der Kommissionierung Stämme mit seltenen Suppressormutationen reduzieren. Auch Streifen die gleichen Transformanten auf LB-Kan, um zu bestätigen, dass die Stämme nicht Kan beständig.
Hinweis: Da die Cm-Resistenz-Marker verwendet wird, um die Donorstämme machen, wir erwarten nicht, dass die Spender-Mutanten Kan beständig sein. Durch Ausstreichen der Transformanten auf beide LB-Kan und LB-Cm Platten stellen wir sicher, dass der Spender Mutation selbst nicht irgendwie verursachen Kan Widerstand. Wenn dieser Schritt nicht durchgeführt wurde und die Target-Mutationen waren, um die Fähigkeit des Stammes auf Kan überleben beeinträchtigen würde Doppelmutanten nicht gemacht effektiv mit ESGA seit Donorstämme alle Selektionsschritte überleben würden. Dies ist lediglich eine Vorsichtsmaßnahme logische Schritt, um unerwartete Phänotypen oder andere Probleme, die mit der Fertigstellung der ESGA Bildschirmen stören kann fangen. Wenn dieser Schritt übersprungen wurden, andere Steuerelemente would noch identifizieren failed Bildschirme. Allerdings würde eine frühere Erkennung von einem Spender, die resistent gegen Kan ist beseitigt eine Reihe von unproduktiven und unnötige Schritte. Andere mögliche Gründe für ein Stamm auf Kan wachsen könnten beispielsweise abgelaufen Antibiotikum sowie die Verwendung einer falschen PCR-Produkt oder einem Stamm für die Transformation. All diese Fragen können identifiziert und behandelt werden frühzeitig in das Verfahren mit Hilfe dieser Kontrolle, die nicht benötigt wird, aber empfehlenswert für ESGA.
- Bestätigung der nicht-essentielle Gen-Deletion (Schritt 1.6.1) oder essentielle Gen hypomorphen Mutation (Abschnitt 1.6.2)
Anmerkung: Bei der Bestätigung der Deletion durch PCR von genomischer DNA aus einem Wildtypstamm sollte als Kontrolle verwendet werden.- Bestätigung nicht wesentlicher Gendeletion
- Um Gendeletion durch PCR bestätigt, Isolieren der genomischen DNA aus den transformierten, putativen Knockout-Stämme in flüssigem LB-Medium gezüchtet Cm nach dem manufacturer Anweisungen (Promega).
- Um eine erfolgreiche Rekombination bestätigen, verstärken die DNA aus jeder Transformante mit drei verschiedenen Sets von Knockout Bestätigung Primer. Führen Reaktionen mit ~ 150 ng genomische DNA-Matrize, und Bestätigung der korrekten Fragmentgrößen durch Ausführen der PCR-Produkte synthetisiert auf einem Agarosegel.
- Der erste Primer Set besteht aus einem 20-nt flankierenden Primer, 200 bp stromaufwärts von der Zielregion (KOCO-F) gelegen, und Cm-R-Primer, die komplementär zu der Kassette Cm Sequenz ist. Diese Verstärkung soll eine 445 nt Amplikon in der Mutante zu produzieren, jedoch nicht in dem Wildtyp-Stamm (Abbildung 1).
- Der zweite Satz umfasst eine Vorwärtsprimer (Cm-F), Annealen an die Kassette Cm Sequenz, und ein 20-nt umgekehrter flankierenden Primer Bestätigung (KOCO-R), die entworfen bis 200 bp stromabwärts des 3'-Ende der Glühung wird deletierte Gen. Diese Amplifikationsreaktion soll eine 309 nt Amplikon in der Mutante zu produzieren, aber keinet im Wildtyp-Stamm (Abbildung 1).
- Die dritte PCR enthält KOCO-F und KOCO-R-Primer. Diese Reaktion benötigt wird, um zu bestätigen, dass der ausgewählte Stamm kein merodiploid, mit einem Genlokus nachdem durch die Kassette ersetzt wurde und eine andere kopiert, aber ansonsten Wildtyp-Gen-Kopie noch vorhanden. Diese Verstärkung von einem korrekten Deletionsmutante soll eine 1,4 kb Produkt (1) zu erzeugen.
Anmerkung: Wenn das Target-Gen über die gleiche Länge in Basenpaaren als Gen Deletionskassette hat, eine nicht bemerken einer Differenz zwischen einem Ort, das von der Kassette ersetzt wurde, und einen Ort, der nicht aufweist. Für eine endgültige Bestätigung in solchen Fällen empfiehlt sich die Verwendung zusätzlicher Primer zur spezifischen Amplifizierung eines DNA-Segments innerhalb des Gens gestrichen. In diesem Fall, wenn das Gen deletiert worden ist, wird ein Amplifikationsprodukt nur im Wildtyp Kontrollstamm und nicht in der Mutante beobachtet werden. Diese Primerkann manuell oder rechentechnisch zur spezifischen Amplifizierung einer DNA-Segment innerhalb der deletierten Region. Wir in der Regel zu verstärken 140 bp Regionen.
- Bestätigen essentielles Gen hypomorphen Mutation
- Um die hypomorphen Mutation durch PCR bestätigt, verwenden Gen-spezifischen 20-nt vorne (KOCO-F) und Reverse-Primer (KOCO-R) 200 bp stromaufwärts und stromabwärts befindet bzw. des SPA-tag Insertionsstelle (Abbildung 2).
Anmerkung: Der Amplifikationsschritt wird als eine Kontrolle verwendet, um die Abwesenheit eines unbezeichnet Kopie der Ziel-essentielles Gen gewährleisten. Wenn nur eine markierte Version vorhanden ist, wird eine einzelne Bande 310 bp größer als der SPA-Cm Amplikon beobachtet. Ein 400 bp-Produkt würde signalisieren die Anwesenheit des untagged Version des Gens. - Parallel zu dem PCR bestätigt, verwenden Westernblotting die In-Frame-Insertion des SPA-tag verifizieren Verwendung eines anti-FLAG M2-Antikörper spezifisch für die FLAG-Epitope des SPA-tag 21.
- Um die hypomorphen Mutation durch PCR bestätigt, verwenden Gen-spezifischen 20-nt vorne (KOCO-F) und Reverse-Primer (KOCO-R) 200 bp stromaufwärts und stromabwärts befindet bzw. des SPA-tag Insertionsstelle (Abbildung 2).
- Bestätigung nicht wesentlicher Gendeletion
- Speichern der bestätigten Donorstamm
- Übertragen Sie die Übernacht-Kultur der bestätigten rekombinanten Spender mutierten Stamm zu den markierten Kryoröhrchen, zu ergänzen, mit Glycerin zu 15% Endkonzentration, und gut mischen. Für die langfristige Lagerung, halten die Kryoröhrchen bei -80 ° C.
2. Anordnen E. coli F-Recipient Mutanten für ESGA Screens
- Replica-pin der E. coli Keio einzigen nicht-essentielles Gen Deletionsmutante Kollektion von Mori et al generiert. (3968 Stämme 11; F-BW25113; kann von Open Biosystems erhältlich) maschinell auf 24 384-well Mikrotiterplatten mit 80 ul von flüssigem LB-Medium pro Well , ergänzt mit 50 ug / ml Kan
Hinweis: Für systematischen Beurteilung GIs mit essentiellen Genen, kann der Empfänger Keio collection 11 mit dem F-Kan-Kennzeichnung essentielles Gen hypomorph ergänzt werdenIC Mutantenstämme mit C-terminalen SPA-Tags 22, 23. - Um Platz für den Rand Kontrollstamm, der als positive Kontrolle dienen soll machen, Beihilfe innerhalb und zwischen Platte Normalisierungen sowie in automatisierten Kolonie Quantisierungen, entfernen die inokuliert Medien von den äußersten Vertiefungen jeder Platte aus der obigen Stufe und Übertragung auf neue Platten, wieder verlassen die äußersten Brunnen leer.
Hinweis: Die Grenzkontrollen Belastung JW5028 von der Keio-Sammlung 11 ist für einen kleinen Pseudo-Gen, das nicht, und sollte nicht in unserem gesamten Genoms Bildschirme zeigen GIs mit allen Spendern gelöscht. Daher ist dies ein guter positive Kontrolle zur seltenen Fällen zu identifizieren, wenn die Konjugation, Pinning oder Auswahlen versagt haben, was fehlt oder sporadische Grenze Belastung Wachstum. Zum Beispiel, wenn die Grenzkontrollen Kolonien nicht nach der Doppelmutante Auswahl wächst, die Abfrage Stamm kann nicht in der Lage Konjugation, kann Konjugation versucht worden ina Bedingung, die ihn von stattfindet (zB auf einem antibiotikahaltigen Platte) oder eine falsche Antibiotikum verhindert können der Selektionsplatte hinzugefügt wurden. Und falls sehr wenige, vereinzelte Kolonien auf der gesamten Platte, einschließlich der Grenze beachtet, kann Konjugation oder Pinning gescheitert und der Schirm muss wiederholt werden. Wenn diese sporadischen Grenze Wachstum ist reproduzierbar, kann dies konsequent armen Spender Konjugation und damit Fälle, in denen Doppelmutanten nicht eingehalten werden, sind möglicherweise aufgrund der Konjugation Versagen, nicht wahr GIs. Solche Bildschirme müssten von der weiteren Analyse verworfen werden. Ferner, indem es auf allen Platten vorhanden ist, kann der Rand Kontrollstamm die Kolonie Quantisierung Software automatisch zu bestimmen Kolonie Positionen auf den Platten Doppelmutante und damit Kolonie Positionen nicht mehr manuell abgegrenzt werden. Schließlich werden die Grenzkontrollen Belastung hilft bei der Normalisierung der Kolonie Größen innerhalb (z. B. unterschiedliche Zeilen innerhalb einer Platte) und zween (zB verschiedene Empfänger Platten mit dem gleichen Spender zu unterschiedlichen Zeiten Pins) Platten. - Ebenso erstellen negative Kontrolle Flecken durch Entfernen von zwei Stämmen von einem anderen Standort innerhalb jeder Platte (keine Grenze) und übertragen die Belastungen auf eine neue Platte. Füllen Sie diese leeren Brunnen mit LB-Medium mit 17 ug / ml Cm. Diese negative Kontrolle Spots werden voraussichtlich in den Empfänger und damit Doppelmutante Platten leer und um sicherzustellen, dass es keine Verarbeitung oder Plattenhandling Fehler, wenn die Nummerierung, Imaging, oder Anheften der Platten.
Hinweis: Diese leeren Flecken sind negative Kontrollen erforderlich. Negative Kontrollen zu identifizieren Fällen, wenn die Auswahl nicht - wenn negative Kontrolle Stämme bei der Auswahl zu wachsen. Wir schlagen vor, nicht nur wählen Sie einzigartige negative Kontrolle Spots für jede der begünstigten Platten, sondern auch wählen negative Kontrolle Stellen innerhalb des gleichen Quadranten der Platte (zB rechts unten). Auf diese Weise wird das Musterder negativen Kontrolle Spots würden identifizieren mögliche Platte Misshandlung Fehler (zB Platten falsch oder verkehrt herum, merken sich, wo die obere linke Kolonie in der rechten unteren Position fixiert ist). - Inokulieren Keio Belastung JW5028 11 mit einer Pseudogen Löschung, in einen 500 ml-Kolben mit 200 ml LB, die 50 ug / ml Kan auf unserer gesamten Genoms ESGA Bildschirmen Grundlage ist das Wachstum dieses speziellen Mutante in der Nähe von wilden Typ BW25113 und es wird somit als eine Grenze Kontrolle verwendet.
- Wachsen die angeordneten Stämme sowie die JW5028 Kultur über Nacht bei 32 ° C mit 190 rpm orbitalen Schütteln bis zu einer OD von ~ 0,4 bis 0,6 bei 600 nm aufweist. Nach dem Wachstum über Nacht, in den grenznahen Brunnen mit rund 80 ul JW5028 Übernacht-Kultur zu füllen.
- Die zusammengefügten Platten können in Schritt 3.2 verwendet werden. Alternativ für die langfristige Lagerung, ergänzen sich auch in den Empfängerländern Platten mit 15% Glycerin, mischen die Medien und Glycerin, und halten die Plattenbei -80 ° C.
3. High-Density-Dehnungs-Mating Verfahren zur Erzeugung E. coli Doppelmutanten
- Wachsen die Hfr Donorstamm, trägt die Abfrage Mutation, mit Cm, über Nacht bei 32 ° C in den reichen LB-Cm flüssigen Medium (17 ug / ml Cm) unter Schütteln bei 220 rpm markiert.
- Merken der geordneten Sammlung Empfängers Mutante in 384-Koloniendichte auf feste LB-Platten mit 50 ug / ml Kan Parallel ergänzt, stift den Spender Abfrage Mutantenstamm in 384-Kolonie Dichte auf die gleiche Anzahl von LB-Platten, supplementiert mit 17 ug / ml Cm. Inkubieren Sie die Platten über Nacht bei 32 ° C.
Anmerkung: Die Platten für Pinning Empfängers verwendet kann bis zu 36 Stunden zur Gewinnung hinreichend große Kolonien für nachfolgende Schritte. Wenn durchschnittliche Grenze Kolonie Größe ca. 2 mm im Durchmesser (oder höher) ist, sind die Platten bereit, fixiert werden. - Um die Stämme konjugieren, Pin des Spenders aus einer 384-Kolonie Nacht Spender Platte aufzu festen LB-Platte. Anschließend Pin einen einzigen 384-Kolonie Empfänger Platte über dem frisch merken Spender auf die Konjugation Platte. Weiter Pinning, bis alle Spender-Empfänger-Platten gewesen konjugiert durch Pinning auf festen LB Konjugation Platten. Inkubieren merken Konjugation Platten bei 32 ° C für 16 bis 24 Stunden.
Hinweis: Einzelne einzigen mutierten Donorstämme kann Gen-zu-Gen Variabilität in Paarungseffizienz wegen der möglichen Auswirkungen der Mutationen auf Konjugation, den DNA-Transfer Effizienz, Qualität oder Eignung zu zeigen. Wachstum kann zu jeder Zeit zwischen 16 und 24 h, wenn genügend große Kolonien für nachfolgende Pinning Schritte gewonnen wurden gestoppt werden. Conjugations dauert 16 bis 24 Stunden zu produzieren ausreichende Zahl von Ex-Konjuganten für reproduzierbare ESGA Ergebnisse auch für die Mutanten der späten Übertragung von Genen oder Mutanten mit etwas geringeren Fitness. - PIN jedes 384-Dichte Konjugation Platte auf einer einzigen festen LB-Platte, enthaltend Cm (17 ug / ml) und Kan (50pg / ml), bis alle Konjugation Platten wurden festgesteckt. Inkubieren der frisch gemerkt ersten Auswahl Platten für 16 bis 36 Stunden bei 32 ° C.
Hinweis: Im Hinblick auf die Auswahlschritte, unterschiedliche Mengen an Zeit kann erforderlich sein, für unterschiedliche Donor Bildschirmen. Da alle Doppelmutanten in einem bestimmten Bildschirm die gleichen Spender Mutation haben, im Durchschnitt ergibt eine langsamere wachsenden Donor Mutante Anlass zu langsamer wachsende Doppelmutanten. So können je nach dem Spender Fitness kann die Auswahl Stufen zwischen 16 und 36 h erforderlich. Die Doppelmutante Wachstum kann gestoppt werden, wenn die Doppelmutanten ausreichend groß sind für den nachfolgenden Schritt entweder Pinning nach der ersten Auswahl oder nach der zweiten bildgebenden werden. Diese Regel übersetzt die Kontrolle Kolonien im Durchschnitt mit mindestens 2 mm Durchmesser angrenzen. - Erneut Pin jedes erste Selektionsplatte auf einen zweiten, doppelt Drug (Cm und Kan), Auswahlplatte in 1.536-Kolonie-Format, so dass jedes erste Auswahl Kolonie durch vier Kolonien auf dargestellt wirddie zweite Auswahl Platte. Die Platten für 16-36 Stunden bei 32 ° C. Fotografieren Sie die letzten Doppelmutante Auswahl Platten quantitativ zu messen, das Wachstum Fitness der Mutante und die Wechselwirkungen zwischen Gen-Paare (Abbildung 3) zu analysieren.
Hinweis: Obwohl die Häufigkeit der merodiploidy (teilweise chromosomale Duplikationen) liegt bei etwa 1/1, 000 Zellen 24 niedrig, merodiploidy kann GI maskieren. Wir empfehlen daher, einschließlich der biologischen Replikate in allen ESGA Bildschirme. Glücklicherweise können kommerzielle wegwerfbare Platten und Kunststoff Pinning Pads bis zu dreimal wiederverwendet werden durch Sterilisieren der Materialien wie folgt: Agar von den Platten wird verworfen und die Platten sowie Pads sind durch Einweichen in 10% Bleichmittel über Nacht sterilisiert, gefolgt von einem mit destilliertem Wasser spülen, Waschen in 70% Ethanol und Lufttrocknung die Kunststoffprodukte in einem Flow-Haube unter UV-Licht. Die sterile Pads und Platten werden in geschlossenen, sterilen Plastikbeutel bis zur Verwendung gelagert.
4. Verarbeitung von Daten und Ableitung GI Scores
- Messen Sie die Kolonien auf jeder Platte im Batch-Modus oder einzeln mit Hilfe eines speziellen Kolonie Bildbearbeitungssoftware 3, 9.
Hinweis: Eine geeignete Java-basierten Hochdurchsatz-Kolonie Bildgebung und Scoring-Anwendung ist nun frei verfügbar für den öffentlichen Zugriff ( http://srcollins77.users.sourceforge.net/ ). Die Software arbeitet auf verschiedenen Plattformen (zB Mac oder PC) und kann entweder als ausführbare Datei oder von einem Terminal-Fenster gestartet werden. - Normalisieren die rohen Kolonie Messungen durch Korrektur systematischer Fehler innerhalb und zwischen den Platten wie Plattenrand Effekte inter-Platte Variation-Effekte, unebenen Bild Beleuchtung, Artefakte aufgrund körperlicher Krümmung der Agar-Oberfläche, Wettbewerb Effekte für benachbarten Kolonien und mögliche Pinning Mängeln sowie Unterschiede im Wachstum der Zeit p> 9. Diese systematischen Artefakte sind unabhängig von der Doppelmutante Fitness und sollte korrigiert, da sie zu falschen Wachstum Fitness-Schätzungen geben.
- Die Kolonien im Rand Zeilen und Spalten in der Regel größer aufgrund geringerer Konkurrenz für Nährstoffe als in der Mitte der Platte vorhanden. Somit, um diesen Effekt zu korrigieren, skaliert die Größen der Kante Kolonien zu sein, so dass die mittlere Größe in dieser Reihe oder Spalte gleich der mittleren Größe der Kolonien in der Mitte der Platte ist.
- Analyse der Ergebnisse statistisch zu berücksichtigen die Reproduzierbarkeit und den Standard Varianz aus den Median Größen der Replikat Kolonie Messungen. Mindestens drei unabhängigen biologischen Wiederholungsversuchen sind notwendig, um die experimentellen Reproduzierbarkeit auszuwerten.
- Schließlich verwenden die normierten mittleren Koloniewachstum Fitness Größen ein GI-Score (S) für jedes Gen Paar sich nach folgender Formel zu erzeugen:
n 1 "src =" / files/ftp_upload/4056/4056eq1.jpg "/>
Wobei S = var (var Exp. x (n Exp. -1) + var Cont x (n Cont -1)) / (n + n Exp. Cont -2); var Exp. = die maximale Varianz der normierten Größen für die Kolonie Doppelmutante; var Cont = Median der Varianzen in den normalisierten Doppelmutante Koloniegrößen aus der Bezugsmenge; n Exp. = Anzahl der Messungen der Doppelmutante Koloniegrößen; n Cont = die mittlere Zahl der experimentellen repliziert über alle Experimente; μ Exp = Median normalisierten Kolonie Größen der Doppelmutanten und μ Cont = Median der normierten Kolonie Größen für alle Doppelmutanten aus dem einzigen Spender Mutante. Die S-Score spiegelt sowohl die statistische Sicherheit eines mutmaßlichen digenen Interaktion sowie die biologische Stärke der Wechselwirkung. Starke positive S-Werte zeigen Linderung Effekte, suggesting daß die zusammenwirkenden Gene im gleichen Signalweg 9 teilnehmen, während signifikante negative S-Scores synthetischen Krankheit oder Letalität, die oft deutet auf Mitgliedschaft in parallele redundante Pfade 9 Rechnung zu tragen. Zur Bestimmung der Stoffwechselweg-Ebene funktionalen Beziehungen wird eine einzelne S-Punktzahl für jedes Paar von getesteten Gene aus dem endgültigen Datensatz erzeugt.
Anmerkung: In unserer früheren Studie 9, fanden wir, dass Rekombination weniger häufig zwischen Genen innerhalb von 30 kbp voneinander neigt. Daher ist für nachgeschaltete Analyse, nach Normalisierung und Punktzahl Generation entfernen wir Wechselwirkungen zwischen Genen innerhalb von 30 kbp voneinander. Neben GIs selbst können funktionale Beziehungen zwischen Paaren von Genen durch suchen, wie ähnlich die GI Profile der beiden Gene untersucht werden. Der GI-Profil eines Gens ist die Menge aller Wechselwirkungen dieses Gens mit allen anderen Genen im Genom außerhalbdes Gens Verknüpfung Bereich. Funktionell ähnliche Gene tendenziell höhere Korrelationen genetische Interaktion Profile 12, 25 haben. Somit ist es durch Berechnen Korrelationskoeffizienten für alle Gene im experimentellen Datensatz kann ESGA zur Untersuchung funktionalen Beziehungen zwischen Genen liegen sogar nahe beieinander auf dem Chromosom zu verwenden.
Ergebnisse
GIs reveal functional relationships between genes. Similarly, since genes in the same pathway display similar GI patterns and the GI profile similarity represents the congruency of phenotypes, we can group functionally related genes into pathways by clustering their GI profiles. Integrating GI and GI correlation networks with physical interaction information or other association data, such as genomic context (GC) relationships can also reveal the organization of higher-order functional modules that define core biological systems (and system crosstalk) in bacteria. For example, as with yeast4, 6, 7, 26, E. coli gene pairs exhibiting alleviating interactions that also have highly correlated GI profiles tend to encode proteins that are either physically associated (e.g. form a complex; Figure 4a) or that act coherently in a common biochemical pathway (e.g. components in a linear cascade). A representative example is shown in Figure 4b, where the components of the functionally redundant Isc and Suf pathways, which jointly participate in the essential Fe-S biosynthesis process, form distinct clusters that are linked together by extensive aggravating interactions (i.e. synthetic lethality). A statistical measure (e.g. hypergeometric distribution function27) can be used on GI or GI correlation data to find significant enrichment for interactions between and within pathways (Figure 4c). Cluster analysis can also be applied to functional networks derived using eSGA to predict the functions of genes lacking annotations5, 6, 9, 28-30 (Figure 4d). Since clustering algorithms vary, however, putative functional assignments determined through clustering require independent experimental verification.
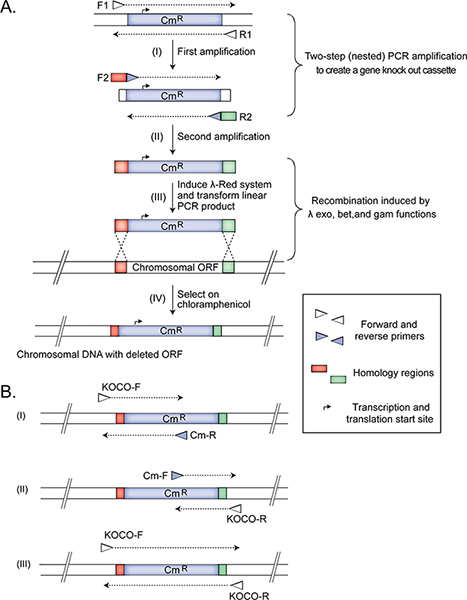
Figure 1. The construction and confirmation of Hfr Cavalli non-essential single gene deletion donor strains. The panels (adapted from14) illustrate the deletion of E. coli chromosomal ORF (A) and the three primer sets used for confirming (B) the correctly generated mutant Hfr Cavalli donor strain. Amplifications from a correctly constructed deletion donor strain should produce (i) 445 bp, (ii) 309 bp, and (iii) 1.4 kb products. See protocol text for details. Click here to view larger figure.

Figure 2. The construction and confirmation strategies of essential hypomorphic donor mutant strains in an Hfr Cavalli genetic background. The panels illustrate the creation of hypomorphic mutations of essential genes through recombineering technology (A) and the primer sets (B) used for the PCR confirmation of essential gene hypomorphic mutations. See protocol text for details.

Figure 3. Schematic summary of key eSGA steps. The Hfr donor (marked with chloramphenicol resistance, CmR) and recipient F- (marked with kanamycin resistance, KanR) mutant strains are grown in 384-colony density on LB-Cm and LB-Kan plates, respectively. The donor and the recipient strains are conjugated by pinning them over each other onto an LB plate, which is then incubated overnight at 32 °C. Consequently, the conjugants are pinned onto plates containing both Kan and Cm to select double mutants. The double mutant plates are digitally imaged and the growth fitness of the colony sizes is quantitatively scored to identify aggravating and alleviating interactions.

Figure 4. Representative computational analyses of genetic interaction (GI) scores for determining pathway-level functional relationships. (A) Panel I, Distribution of correlation coefficients between the GI profiles of gene pairs encoding proteins linked by protein-protein interactions (PPI) versus randomly drawn gene pairs. The p-value was computed using the two-sample Kolmogorov-Smirnov (KS) test; Panel II, Scatter plot of correlated genetic profiles from rich media (RM) for two transporters (mdtI, mdtJ) that form a heterodimeric complex required for spermidine excretion. (B) The hierarchical clustering of the GI sub-network adapted from Butland et al.9 highlights the functional connectivity between components of the previously known Isc and Suf pathways with similar GI patterns. Pink represents aggravating (negative S-score) interactions, green represents alleviating (positive S-score) interactions and black represents absence of GI. A predicted novel component of the SUF pathway, ydhD, displays GIs with the members of the Isc pathway. (C) Pathway cross-talk recorded among cell envelope processes that are significantly enriched for aggravating or alleviating GI according to the hypergeometric enrichment analysis27. (D) A GI sub-network predicts the role of two functionally unannotated genes, yceG and yebA, in peptidoglycan splitting based on their pattern of strong alleviating interactions with well-known cell division peptidoglycan hydrolases. The figures shown in panels A, C and D are adapted from Babu et al. (2011)12. Click here to view larger figure.
Diskussion
Wir haben eine stufenweise Protokoll für die Verwendung robotischen ESGA Screening, um bakterielles Gen Funktionen auf einen Stoffwechselweg Ebene durch Abfragen GI untersuchen skizziert. Dieser Ansatz kann verwendet werden, um einzelne Gene als auch ganzer biologischer Systeme in E. studieren coli. Sorgfältig Ausführung der experimentellen oben beschriebenen Schritte, einschließlich aller geeigneten Kontrollen und konsequent analysieren und unabhängig validiert die GI-Daten sind wesentliche Aspekte für den Erfolg der ESGA bei der Herstellung neuer funktionaler Entdeckungen. Neben ESGA, ein konzeptionell ähnliche Methode zur Untersuchung GI in E. coli, bezeichnet GIANT-coli 17 kann verwendet werden, um neuartige funktionelle Beziehungen, die oft von anderen Ansätzen, wie Proteomik 23 oder phänotypische 13 Bildschirmen allein verpasst zu beleuchten. Dennoch, wie bei jedem genomweite Ansatz müssen Beschränkungen der Anwendbarkeit des ESGA oder GIANT-coli existieren, zum Beispiel zu anderen Bakterienarten. Dies ist teilweise daraufUrsache der Nichtverfügbarkeit der genomweiten Gen Deletionsmutante Stammsammlungen für die meisten Bakterienarten, insbesondere für Arten, bei denen gezielte Mutagenese durch eine niedrige Eigenfrequenz der homologen Rekombination behindert wird. In solchen Fällen kann Gendisruptionen mit Methoden wie zufällige Mutagenese verfolgbaren-basierten Assays wie TnSeq 31 potenziell zur Untersuchung bakterielles Gen-Funktionen verwendet werden. Für einen Überblick der aufstrebenden alternativen bakterielle genetische Screening-Verfahren, siehe Kritik Papier Gagarinova und Emili (2012) 32.
Obwohl GI Methoden oft viele interessante Verbindungen zu offenbaren suggestive neuartiger mechanistische Links, die Integration dieser Daten mit anderen Informationen wie phänotypische Profilierung 13, physikalische Interaktion Netze 22, 23 und GC-abgeleitete Verbände können besonders informativ. Zum Beispiel, wie mit GI-Netzwerke, GC Verbände, die zur Erhaltung beruhenGen-Reihenfolge (Operons), bakterielle Genfusionen, Operons Rekombination Frequenzen von intergenischen Distanzen von vorhergesagten Operons in Genomen abgeleitet sind, können ebenso wie phylogenetischen Profilierung 33 zusätzliche Einblicke, wie eine Bakterienzelle organisiert Proteinkomplexe in funktionale Bahnen zu vermitteln und zu koordinieren großen bereitzustellen zellulären Prozessen 12, 23.
E. coli ist ein Arbeitstier Modell für das Verständnis der molekularen Biologie der andere Gram-negative Bakterien. Zu diesem Zweck können die ESGA GI-Daten in Verbindung mit vergleichenden Genomik Informationen (zB phylogenetische Profilierung, Gene Expression Profiling), um die evolutionäre Konservierung der funktionalen Beziehungen durch ESGA über andere proteobakteriellen Arten entdeckt und prokaryotischen Taxa untersucht werden. Darüber hinaus sind die Wechselwirkungen Daten für E. generiert coli kann verwendet werden, um einen Einblick in den Weg Architektur Mikroben durch metageno entdeckt zu gewinnenMikrofone, bei denen funktionale Annotationen fehlen. Da viele Gene häufig in allen Mikroben 23 konserviert sind, und weil antibiotische Empfindlichkeit kann durch bestimmte genetische Störungen 34 erhöht werden, können die funktionellen Beziehungen durch ESGA beleuchtet sogar potenziell zum Entwerfen neuartige Kombination Arzneimitteltherapien ausgenutzt werden.
Offenlegungen
Keine Interessenskonflikte erklärt.
Danksagungen
Diese Arbeit wurde durch Mittel aus Genome Kanada, Ontario Genomics Institute und der Canadian Institutes of Health Research gewährt JG und AE AG unterstützt wird, ist ein Empfänger von Vanier Canada Graduate Scholarship.
Materialien
| Name | Company | Catalog Number | Comments |
| I. Antibiotics | |||
| Chloramphenicol | Bioshop | #CLR201 | |
| Kanamycin | #KAN201 | ||
| Ampicillin | # AMP201 | ||
| 2. Luria-Bertani medium | |||
| LB powder | Bioshop | #LBL405 | |
| Agar | Bioshop | #AGR003 | |
| 3. Bacterial Strains and Plasmids | |||
| Hfr Cavalli strain λred system (JL238) | Babu et al.14. | ||
| pKD3 | E. coli Genetic Stock Centre, Yale | ||
| Keio E. coli F- recipient collection | National BioResource Project (NBRP) of Japan11 | ||
| Hypomorphic E. coli F- SPA-tag strains | Open biosystems; Babu et al.14 | ||
| 4. Primers | |||
| pKD3-based desalted constant primers | F1: 5'-GGCTGACATGGGAATTAGC-3' R1: 5'-AGATTGCAGCATTACACGTCTT-3' | ||
| Desalted custom primers | Cm-R: 5'-TTATACGCAAGGCGACAAGG-3' Cm-F: 5'- GATCTTCCGTCACAGGTAGG-3' | ||
| Desalted custom primers | F2 and R2: 20 nt constant regions based on pKD3 sequence and 45 nt custom homology regions F2 constant region: 5'-CATATGAATATCCTCCTTA-3' R2 constant region: 5'-TGTGTAGGCTGGAGCTGCTTC-3'S1 and S2: 27 nt constant regions for priming the amplification of the SPA-Cm cassette and 45 nt custom homology regions S1 constant region: 5'AGCTGGAGGATCCATGGAAAAGAGAAG -3' S2 constant region: 5'- GGCCCCATATGAATATCCTCCTTAGTT -3' KOCO-F and KOCO-C: 20 nt primers 200 bp away from the non-essential gene deletion site or the essential gene SPA-tag insertion site | ||
| 5. PCR and Electrophoresis Reagents | |||
| Taq DNA polymerase | Fermentas | # EP0281 | |
| 10X PCR buffer | Fermentas | # EP0281 | |
| 10 mM dNTPs | Fermentas | # EP0281 | |
| 25 mM MgCl2 | Fermentas | # EP0281 | |
| Agarose | Bioshop | # AGA002 | |
| Loading dye | NEB | #B7021S | |
| Ethidium bromide | Bioshop | # ETB444 | |
| 10X TBE buffer | Bioshop | # ETB444.10 | |
| Tris Base | Bioshop | # TRS001 | |
| Boric acid | Sigma | # T1503-1KG | |
| 0.5 M EDTA (pH 8.0) | Sigma | # B6768-500G | |
| DNA ladder | NEB | #N3232L | |
| 6. DNA isolation and Clean-up Kits | |||
| Genomic DNA isolation and purification kit | Promega | #A1120 | |
| Plasmid Midi kit | Qiagen | # 12143 | |
| QIAquick PCR purification kit | Qiagen | #28104 | |
| 7. Equipment for PCR, Transformation and Replica-pinning | |||
| Thermal cycler | BioRad, iCycler | ||
| Agarose gel electrophoresis | BioRad | ||
| Electroporator | Bio-Rad GenePulser II | ||
| 0.2 cm electroporation cuvette | Bio-Rad | ||
| 42 °C water bath shaker | Innova 3100 | ||
| Beckman Coulter TJ-25 centrifuge | Beckman Coulter | ||
| 32 °C shaker | New Brunswick Scientific, USA | ||
| 32 °C plate incubator | Fisher Scientific | ||
| RoToR-HDA benchtop robot | Singer Instruments | ||
| 96, 384 and 1,536 pin density pads | Singer Instruments | ||
| 96 or 384 long pins | Singer Instruments | ||
| 8. Imaging Equipments | |||
| Camera stand | Kaiser | ||
| Digital camera, 10 megapixel | Any Vendor | ||
| Light boxes, Testrite 16" x 24" units | Testrite | ||
| 9. Pads or Plates Recycling | |||
| 10% bleach | Any Vendor | ||
| 70% ethanol | Any Vendor | ||
| Sterile distilled water | Any Vendor | ||
| Flow hood | Any Vendor | ||
| Ultraviolet lamp | Any Vendor | ||
| 10. Labware | |||
| 50 ml polypropylene tubes | Any Vendor | ||
| 1.5 ml micro-centrifuge tubes | Any Vendor | ||
| 250 ml conical flaks | VWR | # 29140-045 | |
| 15 ml sterile culture tubes | Thermo Scientific | # 366052 | |
| Cryogenic vials | VWR | # 479-3221 | |
| Rectangular Plates | Singer Instruments | ||
| 96-well and 384-well microtitre plates | Singer Instruments | Nunc | |
| Plate roller for sealing multi-well | Sigma | #R1275 | |
| plates | ABgene | # AB-0580 | |
| Adhesive plate seals | Fisher Scientific | # 13-990-14 | |
| -80 °C freezer | Any Vendor | ||
Referenzen
- Bandyopadhyay, S., Kelley, R., Krogan, N. J., Ideker, T. Functional maps of protein complexes from quantitative genetic interaction data. PLoS Comput. Biol. 4, e1000065 (2008).
- Costanzo, M., Baryshnikova, A., Myers, C. L., Andrews, B., Boone, C. Charting the genetic interaction map of a cell. Curr. Opin. Biotechnol. 22, 66-74 (2011).
- Costanzo, M., et al. The genetic landscape of a cell. Science. 327, 425-4231 (2010).
- Fiedler, D., et al. Functional organization of the S. cerevisiae phosphorylation network. Cell. 136, 952-963 (2009).
- Roguev, A., et al. Conservation and rewiring of functional modules revealed by an epistasis map in fission yeast. Science. 322, 405-4010 (2008).
- Schuldiner, M., et al. Exploration of the function and organization of the yeast early secretory pathway through an epistatic miniarray profile. Cell. 123, 507-519 (2005).
- Wilmes, G. M., et al. A genetic interaction map of RNA-processing factors reveals links between Sem1/Dss1-containing complexes and mRNA export and splicing. Mol. Cell. 32, 735-746 (2008).
- Babu, M., et al. Systems-level approaches for identifying and analyzing genetic interaction networks in Escherichia coli and extensions to other prokaryotes. Mol. Biosyst. 12, 1439-1455 (2009).
- Butland, G., et al. coli synthetic genetic array analysis. Nat. Methods. 5, 789-7895 (2008).
- Typas, A., et al. Regulation of peptidoglycan synthesis by outer-membrane proteins. Cell. 143, 1097-10109 (2010).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Mol. Syst. Biol. 2, 2006-200008 (2006).
- Babu, M., et al. Genetic interaction maps in Escherichia coli reveal functional crosstalk among cell envelope biogenesis pathways. PLoS Genet. 7, e1002377 (2011).
- Nichols, R. J., et al. Phenotypic landscape of a bacterial cell. Cell. 144, 143-156 (2011).
- Babu, M., Gagarinova, A., Greenblatt, J., Emili, A. Array-based synthetic genetic screens to map bacterial pathways and functional networks in Escherichia coli. Methods Mol Biol. 765, 125-153 (2011).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc. Natl. Acad. Sci. U.S.A. 97, 6640-665 (2000).
- Yu, D., et al. An efficient recombination system for chromosome engineering in Escherichia coli. Proc. Natl. Acad. Sci. U.S.A. 97, 5978-5983 (2000).
- Typas, A., et al. quantitative analyses of genetic interactions in. E. coli. Nat. Methods. 5, 781-787 (2008).
- Zeghouf, M., et al. Sequential Peptide Affinity (SPA) system for the identification of mammalian and bacterial protein complexes. J. Proteome Res. 3, 463-468 (2004).
- Davierwala, A. P., et al. . , 1147-1152 (2005).
- Breslow, D. K., et al. A comprehensive strategy enabling high-resolution functional analysis of the yeast genome. Nat. Methods. 5, 711-718 (2008).
- Babu, M., et al. Sequential peptide affinity purification system for the systematic isolation and identification of protein complexes from Escherichia coli. Methods Mol. Biol. 564, 373-400 (2009).
- Butland, G., et al. Interaction network containing conserved and essential protein complexes in Escherichia coli. Nature. 433, 531-537 (2005).
- Hu, P., Janga, S. C., Babu, M., Diaz-Mejia, J. J., Butland, G., et al. Global functional atlas of Escherichia coli encompassing previously uncharacterized proteins. PLoS Biol. 7, (2009).
- Anderson, R. P., Roth, J. R. Tandem genetic duplications in phage and bacteria. Annu. Rev. Microbiol. 31, 473-505 (1977).
- Boone, C., Bussey, H., Andrews, B. J. Exploring genetic interactions and networks with yeast. Nat. Rev. Genet. 8, 437-449 (2007).
- Collins, S. R., et al. Functional dissection of protein complexes involved in yeast chromosome biology using a genetic interaction map. Nature. 446, 806-8010 (2007).
- Le Meur, N., Gentleman, R. Modeling synthetic lethality. Genome Biol. 9, R135 (2008).
- Tong, A. H., et al. Systematic genetic analysis with ordered arrays of yeast deletion mutants. Science. 294, 2364-2368 (2001).
- Tong, A. H., et al. Global mapping of the yeast genetic interaction network. Science. 303, 808-813 (2004).
- Wong, S. L., et al. Combining biological networks to predict genetic interactions. Proc. Natl. Acad. Sci. U.S.A. 101, 15682-15687 (2004).
- van Opijnen, T., Bodi, K. L., Camilli, A. Tn-seq: high-throughput parallel sequencing for fitness and genetic interaction studies in microorganisms. Nat. Methods. 6, 767-772 (2009).
- Gagarinova, A., Emili, A. Genome-scale genetic manipulation methods for exploring bacterial molecular biology. Mol. Biosyst. 8, 1626-1638 (2012).
- Dewey, C. N., et al. Positional orthology: putting genomic evolutionary relationships into context. Brief Bioinform. 12, 401-412 (2011).
- St Onge, R. P., et al. Systematic pathway analysis using high-resolution fitness profiling of combinatorial gene deletions. Nat. Genet. 39, 199-206 (2007).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten