Method Article
Mappatura batteriche reti funzionali e sulle vie di
In questo articolo
Riepilogo
Sistematiche e su larga scala di sintesi genetica (gene-gene o epistasi) schermi di interazione può essere utilizzato per esplorare la ridondanza genetica e via di cross-talk. Qui, descriviamo un high-throughput quantitativa sintetica genetica tecnologia di retinatura array, definito eSGA che abbiamo sviluppato per chiarire i rapporti epistatiche ed esplorare reti di interazione genetici in Escherichia coli.
Abstract
Fenotipi sono determinati da una serie complessa di fisica (ad esempio proteina-proteina) e funzionali (ad esempio gene-gene o genetico) interazioni (GI) 1. Mentre interazioni fisiche in grado di indicare quali proteine batteriche sono associati come complessi, non necessariamente rivelano percorso a livello di relationships1 funzionali. GI schermi, in cui si misura la crescita dei doppi mutanti recanti due geni cancellati o inattivato e rispetto ai corrispondenti singoli mutanti, in grado di illuminare le dipendenze epistatiche tra loci e, quindi, fornire un mezzo per interrogare e scoprire nuove relazioni funzionali 2. Mappe a grande scala GI sono stati riportati per gli organismi eucarioti, come lievito 3-7, ma le informazioni GI rimangono sparse per procarioti 8, che ostacola l'annotazione funzionale dei genomi batterici. A tal fine, noi e altri hanno sviluppato high-throughput quantitative batteriche metodi di screening GI 9, 10 Qui, presentiamo i passaggi chiave necessari per eseguire quantitativa E. coli sintetico Genetic Array (eSGA) Procedura di screening su un genoma scala 9, utilizzando naturale coniugazione batterica e ricombinazione omologa per generare sistemica e misurare l'idoneità di un gran numero di doppi mutanti in un formato matrice colonia. Brevemente, un robot è utilizzato per trasferire , attraverso la coniugazione, cloramfenicolo (Cm) - segnato da alleli mutanti Hfr ingegneria (ad alta frequenza di ricombinazione) 'ceppi donatori in un array ordinato di kanamicina (Kan) - marcati F-destinatario ceppi. In genere, si usa la perdita di funzione singoli mutanti recanti non essenziali delezioni del gene (ad esempio, la collezione 'Keio' 11) e le mutazioni geniche essenziali hypomorphic alleli (cioè conferiscono ridotta espressione della proteina, la stabilità, o attività 9, 12, 13) a interrogare le associazioni funzionali di geni non essenziali ed essenziali, resture rispettivamente. Dopo coniugazione mediata e conseguente scambio genetico mediante ricombinazione omologa, i mutanti risultanti doppie sono selezionati su terreno solido contenente entrambi gli antibiotici. Dopo conseguenza, le piastre vengono create digitalmente delle immagini e le dimensioni delle colonie sono quantitativamente valutato con un proprio sistema automatico di elaborazione delle immagini 14. IG si rivelano quando il tasso di crescita di un doppio mutante o è significativamente migliore o peggiore del previsto 9. Aggravanti (o negativo) GIS spesso il risultato tra la perdita-di-funzione mutazioni in coppie di geni provenienti da percorsi di compensazione che incidono sullo stesso processo essenziale 2. Qui, la perdita di un singolo gene è tamponata, in modo tale che sia mutante singolo è praticabile. Tuttavia, la perdita di entrambi i percorsi è deleteria e provoca letalità sintetica o malattia (ovvero crescita lenta). Viceversa, alleviare (o positivo) possono verificarsi interazioni tra i geni della via stessa o proteina complessa 2 comedelezione del gene o da solo è spesso sufficiente a perturbare la normale funzione della via o complesso tale che perturbazioni supplementari non ridurre l'attività, e quindi la crescita, ulteriormente. Nel complesso, l'identificazione sistematica e l'analisi delle reti GI in grado di fornire, località turistiche, mappe globali delle relazioni funzionali tra un gran numero di geni, da cui percorso informazioni a livello di perdere per altri approcci possono dedurre 9.
Protocollo
1. Costruire HFR ceppi donatori Mutant Cavalli da Recombineering 15, 16
I passi per costruire le macchie donatori eSGA sono descritti di seguito. In breve, usiamo λ mirata - Red mediata ricombinazione omologa 16 di cassette amplificato marcatore selezionabile frammenti di DNA generati da PCR per creare non essenziali mutanti di delezione del gene (punto 1.1) o di geni essenziali ceppi mutanti hypomorphic donatori (paragrafo 1.2), che vengono poi utilizzati come 'query' per definire le reti GI.
Nota: Durante il processo di sviluppo della tecnologia, abbiamo valutato l'efficacia dell'uso di Hfr mediato trasferimento coniugativo per combinare le mutazioni con un ben definito ceppo donatore Hfr (Hfr Cavalli, Hfr Hayes, e Hfr 3000). Abbiamo esaminato (i) la capacità di rendere efficiente mutanti donatori utilizzando il metodo recombineering lanciato da Yu et al. (2000) 16, (ii) le efficienze relativedi trasferimento di DNA coniugativo differenti marcatori cromosomici, e (iii) gli effetti di posizione e orientamento interrogazione gene cromosomico relativo al locus trasferimento Hfr, Orit. Abbiamo trovato che λ - rosso-mediata efficienza di ricombinazione omologa era molto più alto in Hfr Cavalli che in Hfr Hayes o Hfr3000. Se necessario, trasduzione P1 o un ceppo Psuedo Hfr 17, può essere utilizzato per creare donatori mutanti. Abbiamo adattato con successo tutti questi metodi per rendere lo screening e un gran numero di donatori. Poiché nei nostri esperimenti di prova coniugazione, l'efficienza complessiva del trasferimento e il numero di ex conjugants osservato era significativamente maggiore con Hfr Cavalli, questo sfondo particolare ceppo è stato scelto per la costruzione donatore e grande scala eSGA. Tutte le procedure per schermi eSGA con HFR donatori Cavalli sono descritti.
- Amplificare il frammento di DNA di recombineering successiva per eliminare un gene non essenziale
Impiegare in due fasi nested PCR amplizione (Figura 1) per creare una cassetta delezione del gene per la sostituzione del frame di destinazione di lettura aperta con un marcatore di resistenza Cm dal pKD3 plasmide (GI: 15554330; id proteine: AAL02033.1) 15. Il marcatore è fiancheggiato da siti FRT-11 permettendo la rimozione del gene di resistenza, se necessario, in seguito esperimenti.
Nota: Noi annidate due fasi PCR per rimuovere il plasmide dal prodotto amplificato che viene successivamente utilizzato per trasformare le cellule batteriche. Rimozione del plasmide (nel primo PCR) e quindi creare la cassetta knockout gene nella seconda PCR è necessaria perché la trasformazione con un plasmide è molto più efficiente rispetto recombineering, e l'obiettivo è di ottenere - per quanto possibile - solo ceppi che hanno sostituito con successo il DNA genomico mirato con il marcatore selezionabile. Un'alternativa a nested PCR è effettuare un digest restrizione del plasmide di fuori della regione amplificata nella seconda PCRpasso. Quindi il prodotto del digest restrizione viene purificato e usato come stampo nella fase nested PCR (1.1.2). Questa opzione è anche semplice, ma richiede più tempo di lavoro.- Usare circa 45 ng di pKD3 come modello in PCR con primer forward R1 F1 e inversa per produrre un prodotto di 1070 bp. Purificare il frammento di PCR utilizzando il protocollo di purificazione Qiagen PCR, e diluire il prodotto in acqua distillata sterile a 5 ng / pl concentrazione.
- Utilizzando il prodotto di PCR purificato come modello, impostare una seconda PCR gene-specifici (ad esempio knockout) primer nested, F2 e R2, che hanno (i) 45 nt di gene-specifiche regioni di omologia al 5'-end per consentire per la ricombinazione omologa immediatamente a monte ea valle del gene bersaglio per sostituirlo con il marcatore di resistenza Cm e (ii) 20 nt a 3'-end per innescare la sintesi del marcatore Cm. La dimensione del frammento di 1123 bp è prodotta. Il gene-specifiche regioni di omologia sono progettati per rimuovere l'op bersaglioit reading frame, lasciando tutti i segmenti adiacenti o parzialmente sovrapposte di codifica intatto. Dopo questa amplificazione, passare al punto 1.3.
- Amplificare una cassetta codifica per la creazione di una mutazione del gene essenziale hypomorphic
- Per costruire ceppi donatori hypomorphic mutanti, aggiungere un C-terminale sequenziale peptide affinità (SPA) tag fusion, che spesso incide leggermente la funzione delle proteine essenziali abbondanza trascrizione destabilizzante (numero di proteine ad esempio copia) o del ripiegamento delle proteine / attività. A tal fine, creare un selezionabile SPA-tag (SPA-Cm) stampo di DNA convertendo il marcatore di resistenza Kan in pJL148 18 a cm usando recombineering identici tra sequenze nucleotidiche che fiancheggiano il marcatore Kan in pJL148 e marcatore Cm in pKD3 15. Il modello risultante consiste in-frame SPA-tag seguito da un marcatore di resistenza Cm, ed è adatto per l'amplificazione PCR, recombineering e successiva eSGA 12.
Nota: </ Strong> geni essenziali sono potenzialmente più interessanti per gli studi di funzione del gene. Tuttavia, poiché questi geni non possono essere cancellati, metodi innovativi doveva essere sviluppato per indagare le relazioni funzionali di geni essenziali. Diversi approcci adatti grande successo sono stati sviluppati inizialmente in lievito. Per esempio, Davierwala et al. (2005) 19 schermi GI eseguite utilizzando un pannello di 575 termosensibili mutanti geniche essenziali, e ha scoperto che i geni essenziali esposti circa cinque volte il numero delle IG come geni non essenziali. Allo stesso modo, Breslow et al. (2008) 20 ha sviluppato un 'abbondanza è diminuita di perturbazione mRNA' (umido) approccio per generare una libreria di alleli hypomorphic coprono ~ 82% dei geni di lievito essenziali per studiare percorsi e complessi. L'approccio DAMP modifica la 3'end del mRNA espresso, riducendo il livello del gene bersaglio 6, probabilmente a causa di mRNA destabilizzazione. Come per l'approccio DAMP 6, la nostra strategy è stata quella di sfruttare gli effetti sottili di perturbare l'estremità 3 'del mRNA espresso di proteine essenziali.
Come descritto di seguito, l'etichetta di per sé non possono incidere folding delle proteine o funzione, ma la perturbazione sottile del mRNA dal tag è sufficiente, quando combinato con fattori di stress ambientale o mutazioni di altri, per rivelare funzionalmente informativo gene-ambiente 13 e gene gene-9, 12 interazioni.
In primo luogo, fino ad oggi, abbiamo usato SPA-tag per purificare ~ 3.000 essenziali e non essenziali proteine di E. coli 21-23, con oltre l'80% delle 307 E. coli essere rappresentate proteine essenziali tra i nostri ceppi SPA-etichetta 8. Poiché il tag è stato utilizzato con successo per isolare complessi proteici noti, che sono stati convalidati reciprocamente (cioè il complesso stesso potrebbe essere isolato mediante codifica e purificare proteine diverse all'interno della stessa), ed i complessi isolati erano biologicamente rilevante, abbiamo ragione che i tag faredi solito non influenzano il folding delle proteine o la funzione 12. Analogamente, le nostre osservazioni non pubblicate rivelato che il tag di per sé di solito non pregiudica la crescita ceppo. Tuttavia, abbiamo pensato che l'alterazione della 3'-UTR integrando il tag e la cassetta marcatore, come è stato segnalato per i ceppi umidi 6, potrebbe destabilizzare alcune trascrizioni a livello di RNA o, in rare occasioni, impediscono il ripiegamento corretto o la funzione del proteine di fusione. Pertanto, abbiamo previsto che unisce tale perturbato alleli di geni essenziali con altre mutazioni funzionalmente rilevanti altrove nel genoma comporterebbe IG informativo, che effettivamente è stato osservato 9, 12. Alleli Inoltre, SPA-etichetta di geni essenziali esposti interazioni gene-ambiente (ad es ipersensibilità farmaco) secondo il lavoro profiling fenotipica da Nichols et al. (2011) 13, che sottoposto un sottoinsieme di SPA-etichetta ceppi essenziali per una varietà di condizioni di crescita. Lo stesso studio confirmed che la maggior parte (54 su 58 testati) SPA-tagged ceppi essenziali mostrato palestra insufficiente in una o più condizioni di coltura 13, che sostiene l'idea che la SPA-tag spesso crea miti hypomorphic alleli dei geni essenziali che sono utili per il genoma applicazioni a schermo GI tra eSGA. - Amplificare la cassetta di tagging dal SPA-Cm template usando primers S1 e S2 gene-specifici, ciascuno contiene (i) a 45 nt gene-specifico per regione omologa recombineering alla estremità 5 ', seguita da (ii) a 27 bp SPA- cm sequenza specifica alla estremità 3 '.
- Per costruire ceppi donatori hypomorphic mutanti, aggiungere un C-terminale sequenziale peptide affinità (SPA) tag fusion, che spesso incide leggermente la funzione delle proteine essenziali abbondanza trascrizione destabilizzante (numero di proteine ad esempio copia) o del ripiegamento delle proteine / attività. A tal fine, creare un selezionabile SPA-tag (SPA-Cm) stampo di DNA convertendo il marcatore di resistenza Kan in pJL148 18 a cm usando recombineering identici tra sequenze nucleotidiche che fiancheggiano il marcatore Kan in pJL148 e marcatore Cm in pKD3 15. Il modello risultante consiste in-frame SPA-tag seguito da un marcatore di resistenza Cm, ed è adatto per l'amplificazione PCR, recombineering e successiva eSGA 12.
- Confermare e purificare il prodotto di PCR
- Verificare che la cassetta codifica è stata correttamente amplificato sottoponendo 2-5 microlitri del prodotto PCR ad elettroforesi su gel di agarosio e purificare il DNA rimanente dopo l'costruttore standard di protocollo di purificazione PCR (Qiagen). Eluire il prodotto di PCR in 30 ml di acqua distillata sterile e diluire a ~ 50 ng / pl concentrazione. La purificazioneprodotto ed è possibile utilizzare immediatamente nella trasformazione (passo 1.5) o conservato a -20 ° C per un massimo di 6 mesi fino al momento dell'uso.
- Preparazione HFR Cavalli cellule competenti per la costruzione dei donatori
- Inoculare un ml di coltura overnight saturo di un ceppo Hfr Cavalli in 70 ml di fresco Luria-Bertani (LB) di media con 35 pl di 50 ug / ml ampicillina in un pallone da 250 ml. Incubare la coltura a 32 ° C con agitazione a 220 rpm fino ad una densità ottica (OD) 600 nm di ~ 0,5-0,6 è ottenuta (~ 2 ore).
- Trasferire la cultura di un bagno di acqua per l'induzione di calore del sistema di ricombinazione rosso λ 16 a 42 ° C per 15 minuti con agitazione a 160 rpm. Arrestare l'induzione mediante il trasferimento della cultura di un freddo di ghiaccio liquami bagnomaria per 10-20 min a 160 giri al minuto. Assicurarsi di mantenere le cellule a freddo da questo punto fino a dopo la trasformazione; tubi di uso e le cuvette che sono stati raffreddati in ghiaccio per almeno 15 minuti prima dell'uso.
- Dividere il cCULTURA ugualmente in 2 pre-refrigerati provette da 50 ml in polipropilene (~ 35 ml cultura per provetta) e centrifugare a 4.400 xg per 6 min a 4 ° C.
- Eliminare il supernatante e risospendere il pellet di cellule delicatamente per inversione in ~ 50 ml di acqua ghiacciata distillata sterile. Centrifugare le cellule nuovamente a 4.400 xg per 6 minuti a 4 ° C. Eliminare il supernatante e risospendere ogni cella pellet in 20 ml di ghiaccio freddo glicerolo sterile 10% e centrifugare nuovamente come descritto sopra.
- Decantare il surnatante e risospendere il pellet cellulare in 500 ml di ghiaccio freddo glicerolo 10%. Aliquotare le cellule preparate competenti in 50 volumi in singoli pl, pre-refrigerati 1,5 ml micro-centrifuga tubi per la trasformazione immediata. Le cellule possono essere congelate in ghiaccio secco o in azoto liquido e conservati a -80 ° C per 3 mesi. Tuttavia, l'efficienza di trasformazione è più elevata con preparati cellule competenti.
- Trasformazione
- Aggiungere 100 ng del prodotto di PCR purificato dalpunto 1.3.1 alle cellule competenti. Flick il tubo e consentire la sospensione di sedersi su ghiaccio per 5 min.
- Trasferire la sospensione di cellule competenti ghiacciate e DNA per una pre-raffreddata cuvette elettroporazione. Electroporate miscela cella con il 2,5 kV, 25 uF, 200 ohm mettendo in una cuvetta da 2 mm gap (cioè se con una cuvetta diverso, si prega di consultare le istruzioni del produttore per le impostazioni di elettroporazione), e subito aggiungere 1 ml di terreno temperatura ambiente SOC. Trasferire le cellule elettroporate in mezzo SOC in una provetta da 15 ml e incubare a 32 ° C per 1 ora con agitazione orbitale a 220 rpm.
- Dopo l'incubazione, centrifugare le cellule a 4.400 xg per 5 min a temperatura ambiente. Rimuovere circa 850 pl del supernatante e risospendere il pellet cellulare nel liquido rimanente.
- Stendere le celle pre-riscaldate piastre LB contenente 17 mcg / ml Cm, e incubare a 32 ° C per una notte. Scegli due colonie trasformanti individuali e strisciare su LB-Cm piastre per conferma mutante. Si consiglia di evitare più grandi colonie trasformanti, al fine di ridurre la possibilità di scegliere i ceppi con rare mutazioni soppressori. Anche striscia trasformanti stessi LB-Kan per confermare che i ceppi resistenti non sono Kan.
Nota: Dal momento che il marcatore di resistenza Cm è usato per fare i ceppi donatori, non ci aspettiamo che i mutanti donatori per essere resistente Kan. Con striature trasformanti su entrambi LB-Kan e LB-Cm piatti, ci assicuriamo che la mutazione donatore stesso non ha in qualche modo causare resistenza Kan. Se questo passaggio non sono state eseguite e le mutazioni bersaglio erano a pregiudicare la capacità del ceppo di sopravvivere su Kan, doppi mutanti non verrebbero effettuate efficacemente utilizzando eSGA poiché ceppi donatori sopravviverebbero tutte le fasi di selezione. Questo è solo un passo logico per la cattura di precauzione fenotipi imprevisti o eventuali altri problemi che possono interferire con il completamento degli schermi eSGA. Se questo passaggio sono stati saltati, woul altri controllid ancora identificare schermi falliti. Tuttavia, una diagnosi precoce di un donatore che è resistente a Kan eliminerebbe una serie di passaggi improduttivi e inutili. Altre possibili ragioni per un ceppo di crescere su Kan potrebbero ad esempio scaduta antibiotico nonché l'utilizzo di un prodotto di PCR errato o un ceppo per la trasformazione. Tutti questi problemi possono essere individuati e affrontati nella fase iniziale della procedura, con l'aiuto di questo controllo, che non è obbligatoria, ma consigliata per eSGA.
- Confermare il non essenziale delezione del gene (fase 1.6.1) o mutazione del gene essenziale hypomorphic (sezione 1.6.2)
Nota: Quando si conferma la delezione del gene mediante PCR, il DNA genomico di un ceppo di tipo selvatico dovrebbe essere usato come controllo.- Conferma non essenziali delezione del gene
- Per confermare la cancellazione del gene mediante PCR, il DNA genomico isolato dai ceppi trasformati, knockout putativi coltivate in liquido LB-Cm mezzo, seguendo il manufattourer le istruzioni (Promega).
- Per confermare recombineering successo, amplificare il DNA di ogni trasformante con tre diversi set di primer di conferma eliminazione diretta. Eseguire le reazioni con ~ 150 ng di DNA stampo genomico, e confermare le dimensioni dei frammenti corrette eseguendo i prodotti PCR-sintetizzati su un gel di agarosio.
- La serie di primer prima consiste in un-20 nt innesco fiancheggiante, situato 200 bp a monte della regione di destinazione (KOCO-F), e Cm-R primer, che è complementare alla sequenza cassetta Cm. Questa amplificazione dovrebbe produrre un amplicone 445 nt nel mutante, ma non nel ceppo di tipo selvatico (Figura 1).
- Il secondo gruppo comprende un primer forward (Cm-F), ricottura alla sequenza cassetta Cm, ed un 20-nt inversa innesco fiancheggiante conferma (KOCO-R), che è progettato per temprare 200 bp a valle della estremità 3 'del soppresso gene. Questa reazione di amplificazione si prevede di produrre un amplicone 309 nt nel mutante, ma nessunt nel ceppo di tipo selvatico (Figura 1).
- La terza PCR contiene KOCO-F e KOCO-R primers. Questa reazione è richiesto di confermare che il ceppo selezionato non è un merodiploid, con un locus genico essendo stato sostituito da un altro cassetto e ripetuto ma altrimenti wild-type di copie del gene ancora presente. Questa amplificazione da un mutante delezione corretta dovrebbe produrre un prodotto di 1,4 kb (Figura 1).
Nota: Quando il gene bersaglio ha circa la stessa lunghezza in paia di basi, come la cassetta delezione del gene, uno non noterà la differenza tra un locus che è stato sostituito con la cassetta, e un locus che non ha. Per una conferma definitiva in tali casi, si consiglia di utilizzare primer aggiuntivi per amplificare specificamente un segmento di DNA all'interno del gene soppresso. In questo caso, se il gene è stato cancellato, un prodotto di amplificazione viene osservata solo nel ceppo selvatico controllo di tipo e non nel mutante. Questi primerpuò essere manuale oppure computazionalmente specificamente progettati per amplificare un segmento di DNA all'interno della regione soppresso. Noi di solito amplificare regioni 140 bp.
- Confermando mutazione del gene essenziale hypomorphic
- Per confermare la mutazione hypomorphic mediante PCR, utilizzare gene-specifico 20-nt forward (KOCO-F) e reverse primer (KOCO-R) situati 200 bp a monte ea valle, rispettivamente, del SPA-tag sito di inserimento (Figura 2).
Nota: Questo passaggio di amplificazione viene utilizzato come controllo per garantire l'assenza di una copia senza tag del gene bersaglio essenziale. Quando solo una versione con tag è presente, una singola banda di 310 bp più grande della SPA-Cm amplicone si osserva. Un prodotto di 400 bp avrebbe segnalare la presenza della versione untagged del gene. - Parallelamente alla conferma PCR, Western blotting utilizzare per verificare l'in-frame inserimento del SPA-tag utilizzando un anticorpo anti-FLAG M2 specifico per i FLAG-epitopi della SPA-tag 21.
- Per confermare la mutazione hypomorphic mediante PCR, utilizzare gene-specifico 20-nt forward (KOCO-F) e reverse primer (KOCO-R) situati 200 bp a monte ea valle, rispettivamente, del SPA-tag sito di inserimento (Figura 2).
- Conferma non essenziali delezione del gene
- Conservazione del ceppo donatore confermato
- Trasferire la coltura overnight del ceppo ha confermato donatore ricombinante mutante ai cryovials etichettati, integrare con glicerolo al 15% concentrazione finale, e mescolare bene. Per la conservazione a lungo termine, mantenere le cryovials a -80 ° C.
2. Formare gli array E. coli F-Recipient Mutanti per schermi eSGA
- Replica-pin E. coli Keio singolo non essenziale raccolta delezione del gene mutante generato da Mori et al. (3968 ceppi F-11; BW25113; può essere ottenuto da Open Biosystems) tramite macchina 24 384 pozzetti contenenti 80 ml di LB mezzi liquidi per pozzetto , supplementato con 50 pg / ml Kan
Nota: per valutare sistematicamente soldati con i geni essenziali, il destinatario Keio di raccolta 11 può essere integrato con l'F-Kan-segnato hypomorph gene essenzialeceppi mutanti con ic C-terminale SPA-tag 22, 23. - Per fare spazio al ceppo di controllo di frontiera, che fungerà da controllo positivo, aiuto all'interno e tra le normalizzazioni piastra così come in quantizzazioni colonia automatizzati, rimuovere i supporti inoculati dai pozzi più esterni di ogni piatto dal passaggio precedente e trasferimento piatti nuovi, lasciando ancora una volta i pozzi più esterni vuoto.
Nota: Il ceppo di controllo delle frontiere JW5028 della collezione Keio 11 è soppresso per un gene piccolo pseudo che non deve, e non è nelle nostre intero genoma schermi, mostrano soldati con eventuali donatori. Pertanto, si tratta di un ceppo di controllo positivo buona per identificare rari casi in cui coniugazione, pinning, o selezioni hanno fallito, con conseguente crescita bordo assente o sporadico sforzo. Per esempio, se le colonie di frontiera non crescono dopo la selezione doppio mutante, il ceppo query potrebbe non essere in grado di coniugazione, coniugazione può essere tentato ina condizione che impediva corso (ad esempio su un antibiotico contenente piastra), o un antibiotico errato potrebbe essere stato aggiunto alla piastra di selezione. Allo stesso modo, se molto pochi, sporadici si osservano colonie sulla piastra intera, compreso il bordo, coniugazione o pinning può essere guasto e lo schermo deve essere ripetuta. Se questa crescita confine sporadica è riproducibile, può indicare la coniugazione dei donatori costante scarsa e quindi i casi in cui doppi mutanti non sono osservate sono probabilmente a causa della mancata coniugazione non, IG vero. Tali schermi dovrebbero essere eliminati dalla successiva analisi. Inoltre, essendo presente in tutti i piatti, il ceppo di controllo delle frontiere permette al software di quantizzazione colonia di determinare automaticamente la posizione delle colonie sulle piastre doppio mutante e quindi posizioni colonia non devono essere delimitate manualmente. Infine, le frontiere aiuti di controllo di deformazione nella normalizzazione delle dimensioni delle colonie all'interno (ad esempio righe diverse all'interno di un piatto) e tra leEEN (ad esempio piastre di destinatari diversi appuntato con lo stesso donatore in tempi diversi) piatti. - Allo stesso modo, creare punti di controllo negativo, eliminando qualsiasi due ceppi da una posizione diversa all'interno di ciascuna piastra (non di confine) e trasferire i ceppi di una nuova piastra. Riempire questi pozzetti vuoti con supporti LB contenenti 17 ug / ml di Cm. Questi punti di controllo negativo dovrebbero essere vuoto nel ricevente e le piastre mutanti quindi doppie e fare in modo che non vi era alcuna trasformazione o errori nella gestione delle piastre quando la numerazione, l'imaging, o pinning le piastre.
Nota: Questi spazi vuoti sono necessari controlli negativi. I controlli negativi aiutare a identificare i casi in cui la selezione non riesce - quando i ceppi di controllo negativi crescere durante la selezione. Si consiglia di selezionare non solo i punti unici di controllo negativo per ciascuna delle piastre di destinatari, ma di scegliere anche i punti di controllo negativi all'interno stesso quadrante della piastra (ad esempio, in basso a destra). In questo modo, il patterndei punti di controllo negativo sarebbe identificare gli errori possibili maltratta piastra (ad esempio lastre di essere scambiati o di essere bloccato a testa in giù, dove la colonia in alto a sinistra è bloccato in posizione bassa a destra). - Inoculare il ceppo Keio JW5028 11, con una delezione pseudogene, in un pallone da 500 ml con 200 ml di LB, contenente 50 ug / ml di Kan base ai nostri intero genoma schermi eSGA, la crescita di questo mutante particolare è il più vicino al selvatico Tipo BW25113 ed è pertanto utilizzato come controllo di frontiera.
- Crescere i ceppi disposti in serie così come la cultura JW5028 tutta la notte a 32 ° C con 190 rpm orbitale si stringono ad un OD di ~ ,4-,6 a 600 nm. Dopo la crescita durante la notte, riempire i pozzetti di confine con circa 80 ml di JW5028 cultura durante la notte.
- Le piastre assemblate possono essere usati nella fase 3.2. In alternativa, per la conservazione a lungo termine, si integrano a vicenda e nei piatti dei destinatari con il 15% glicerolo, mescolare i mezzi di comunicazione e di glicerolo, e mantenere le piastrea -80 ° C.
3. Ad alta densità di accoppiamento Procedura di sforzo per la generazione di E. Mutants coli doppia
- Coltivare il ceppo donatore Hfr, recante la mutazione query, contrassegnati con Cm, tutta la notte a 32 ° C in ricco LB-Cm mezzo liquido (17 mg / ml di Cm) con agitazione a 220 rpm.
- Pin il destinatario raccolta ordinata mutante in 384-colonia su piastre LB densità solidi supplementati con 50 ug / ml di Kan Parallelamente, pin il ceppo mutante interrogazione donatore 384-colonia densità sullo stesso numero di piastre LB, supplementato con 17 mg / ml di Cm. Incubare le piastre per una notte a 32 ° C.
Nota: Le piastre destinatari utilizzati per il pinning può richiedere fino a 36 ore per ottenere colonie sufficientemente grandi per i passaggi successivi. Quando dimensione media delle colonie di confine è di circa 2 mm di diametro (o superiore), i piatti sono pronti per essere appuntato. - Per coniugare i ceppi, pin il donatore da una colonia di 384 piastra donatore durante la notte sualla piastra LB solido. Successivamente bloccare un singolo piatto da 384 colonia beneficiaria rispetto del donatore appena appuntato sulla piastra coniugazione. Continuare fino a quando tutte pinning donatore-ricevente piastre sono state coniugate da appuntare su piastre solide coniugazione LB. Incubare le piastre coniugazione bloccate a 32 ° C per 16 a 24 ore.
Nota: I singoli donatori singoli ceppi mutanti possono presentare gene per gene variabilità in termini di efficienza di accoppiamento a causa dei potenziali effetti delle mutazioni sulla coniugazione, l'efficienza di trasferimento del DNA, o idoneità. La crescita può essere interrotta in qualsiasi momento tra il 16 e 24 ore, quando le colonie sufficientemente grandi sono stati ottenuti per le successive fasi pinning. Coniugazioni durata di 16 a 24 ore di produrre un numero sufficiente di ex-conjugants per risultati eSGA riproducibili anche per i geni mutanti di fine trasferimento o mutanti con palestra leggermente inferiore. - 384-pin ciascuna densità piastra coniugazione su una singola piastra LB solido, cm contenente (17 mcg / ml) e Kan (50mcg / ml) finché tutte le piastre di coniugazione sono stati bloccati. Incubare le piastre appena appuntato prima selezione per 16-36 ore a 32 ° C.
Nota: Per quanto riguarda le fasi di selezione, diverse quantità di tempo può essere necessario per schermi diversi donatori. Poiché tutti i mutanti doppi in una schermata dato la stessa mutazione donatore, in media, un mutante lento donatore crescente comporta lenti mutanti doppi crescenti. Pertanto, a seconda della idoneità donatore, fasi di selezione può richiedere tra 16 e 36 ore. La crescita doppio mutante può essere fermato quando i doppi mutanti sono sufficientemente grandi per la fase successiva sia pinning dopo la prima selezione o imaging dopo il secondo. Questo si traduce di solito per delimitare colonie di controllo in media con almeno 2 mm di diametro. - Re-pin ogni piatto prima selezione su un secondo farmaco doppio (Cm e Kan), piastra di selezione nel 1536 le colonie formato, in modo tale che ogni colonia prima selezione è rappresentato da quattro colonie sula piastra seconda selezione. Incubare le piastre per 16-36 ore a 32 ° C. Fotografare le finali doppia piastra selezione dei mutanti per misurare quantitativamente l'idoneità crescita del mutante e analizzare le interazioni tra coppie di geni (Figura 3).
Nota: Anche se la frequenza di merodiploidy (parziali duplicazioni cromosomiche) è bassa a circa 1/1, 000 cellule 24, merodiploidy può mascherare GI. Pertanto, si consiglia anche replica biologica in tutte le schermate eSGA. Fortunatamente, commerciali e piatti monouso plastica pad pinning può essere riutilizzato fino a tre volte con la sterilizzazione dei materiali come segue: Agar dalle piastre viene scartato e le piastre così come pastiglie sono sterilizzati immergendoli in candeggina al 10% durante la notte, seguito da un risciacquo con acqua distillata, lavaggio in etanolo al 70%, e essiccazione del plasticware in una cappa a flusso sotto luce ultravioletta. I tamponi sterili e le piastre sono memorizzati in sacchi di plastica sterili fino al momento dell'uso.
4. Elaborazione dei dati e Derivazione punteggi GI
- Misurare le colonie su ogni piatto in modalità batch o singolarmente utilizzando uno speciale software di imaging colonia 3, 9.
Nota: Un adeguato Java-based high-throughput di imaging colonia e l'applicazione di punteggio è ora liberamente disponibile per l'accesso pubblico ( http://srcollins77.users.sourceforge.net/ ). Il software funziona su diverse piattaforme (ad esempio Mac o PC) e può essere lanciato sia come un file eseguibile o da una finestra di terminale. - Normalizzare le prime misurazioni dimensioni delle colonie, correggendo distorsioni sistematiche all'interno e tra le piastre, come effetti di bordo piatto, inter-piastra effetti di variazione di luce, immagine irregolare, artefatti dovuti alla curvatura fisica della superficie dell'agar, gli effetti della concorrenza per le colonie vicine e possibili difetti pinning , nonché differenze nel tempo di crescita p> 9. Questi artefatti sistematici sono indipendenti del fitness doppio mutante e dovrebbe essere corretto in quanto danno luogo a stime di crescita di fitness spuri.
- Le colonie in righe e colonne di bordo tendono ad essere più grandi a causa della minore competizione per nutrienti che si trovano al centro della piastra. Così, per correggere questo effetto, scalare le dimensioni delle colonie di bordo deve essere tale che il valore medio in quella riga o colonna è uguale alla dimensione mediana delle colonie nel centro della piastra.
- Analizzare i risultati statisticamente per tener conto della riproducibilità e la varianza standard dalla dimensione mediana delle misurazioni ripetute colonia. Almeno tre esperimenti indipendenti replicati biologici sono necessari per valutare la riproducibilità sperimentale.
- Infine, utilizzare i normalizzati mediani colonia dimensioni idoneità crescita per generare un punteggio GI (S) per ogni coppia di geni utilizzando la formula seguente:
n 1 "src =" / files/ftp_upload/4056/4056eq1.jpg "/>
Dove, S = var (var Exp x (n Exp -1) + var Cont x (n Cont -1)) / (n + n Exp Cont -2); var Exp = il massimo scostamento delle dimensioni colonie normalizzati per l' doppio mutante; var = Cont mediana delle variazioni nelle dimensioni normalizzate doppie colonie mutanti dal gruppo di riferimento; Exp n = numero di misurazioni di dimensioni doppie colonie mutanti; Cont n = il numero medio di repliche sperimentali su tutti gli esperimenti; μ Esp = dimensioni mediane delle colonie normalizzati dei doppi mutanti, e Cont μ = media delle dimensioni delle colonie normalizzati di tutti i doppi mutanti derivanti dal ceppo mutante singolo donatore. La S-score riflette sia la confidenza statistica di un'interazione digenic putative così come la forza di interazione biologica. Fortemente positivo S-punteggi indicano effetti alleviare, suggesting che i geni che interagiscono partecipare allo stesso percorso 9, mentre negativo significativo S punteggi riflettono malattia o letalità sintetica, che è spesso indicativa di appartenenza paralleli percorsi ridondanti 9. Per determinare i pathway relazioni a livello funzionale, un singolo S-punteggio per ogni coppia di geni testati è prodotto dal dataset finale.
Nota: Nel nostro studio precedente 9, abbiamo scoperto che la ricombinazione tende a verificarsi meno frequentemente tra i geni entro 30 kbp l'uno dall'altro. Pertanto, per analisi a valle, dopo la normalizzazione e generazione punteggio, rimuoviamo interazioni tra geni entro 30 kbp uno dall'altro. Oltre ai soldati stessi, le relazioni funzionali tra coppie di geni possono essere studiate, cercando in modo simile i profili GI dei due geni sono. Il profilo GI di un gene è l'insieme di tutte le interazioni di quel gene con tutti gli altri geni nel genoma esternozona di collegamento del gene. Funzionalmente geni simili tendono ad avere più alti correlazioni di profili genetici di interazione 12, 25. Così, calcolando i coefficienti di correlazione per tutti i geni nel dataset sperimentale si può utilizzare eSGA per investigare le relazioni funzionali tra geni anche situata vicino all'altro sul cromosoma.
Risultati
GIs reveal functional relationships between genes. Similarly, since genes in the same pathway display similar GI patterns and the GI profile similarity represents the congruency of phenotypes, we can group functionally related genes into pathways by clustering their GI profiles. Integrating GI and GI correlation networks with physical interaction information or other association data, such as genomic context (GC) relationships can also reveal the organization of higher-order functional modules that define core biological systems (and system crosstalk) in bacteria. For example, as with yeast4, 6, 7, 26, E. coli gene pairs exhibiting alleviating interactions that also have highly correlated GI profiles tend to encode proteins that are either physically associated (e.g. form a complex; Figure 4a) or that act coherently in a common biochemical pathway (e.g. components in a linear cascade). A representative example is shown in Figure 4b, where the components of the functionally redundant Isc and Suf pathways, which jointly participate in the essential Fe-S biosynthesis process, form distinct clusters that are linked together by extensive aggravating interactions (i.e. synthetic lethality). A statistical measure (e.g. hypergeometric distribution function27) can be used on GI or GI correlation data to find significant enrichment for interactions between and within pathways (Figure 4c). Cluster analysis can also be applied to functional networks derived using eSGA to predict the functions of genes lacking annotations5, 6, 9, 28-30 (Figure 4d). Since clustering algorithms vary, however, putative functional assignments determined through clustering require independent experimental verification.
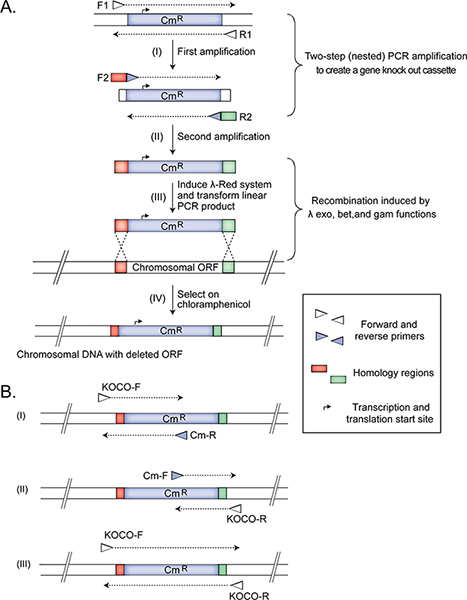
Figure 1. The construction and confirmation of Hfr Cavalli non-essential single gene deletion donor strains. The panels (adapted from14) illustrate the deletion of E. coli chromosomal ORF (A) and the three primer sets used for confirming (B) the correctly generated mutant Hfr Cavalli donor strain. Amplifications from a correctly constructed deletion donor strain should produce (i) 445 bp, (ii) 309 bp, and (iii) 1.4 kb products. See protocol text for details. Click here to view larger figure.

Figure 2. The construction and confirmation strategies of essential hypomorphic donor mutant strains in an Hfr Cavalli genetic background. The panels illustrate the creation of hypomorphic mutations of essential genes through recombineering technology (A) and the primer sets (B) used for the PCR confirmation of essential gene hypomorphic mutations. See protocol text for details.

Figure 3. Schematic summary of key eSGA steps. The Hfr donor (marked with chloramphenicol resistance, CmR) and recipient F- (marked with kanamycin resistance, KanR) mutant strains are grown in 384-colony density on LB-Cm and LB-Kan plates, respectively. The donor and the recipient strains are conjugated by pinning them over each other onto an LB plate, which is then incubated overnight at 32 °C. Consequently, the conjugants are pinned onto plates containing both Kan and Cm to select double mutants. The double mutant plates are digitally imaged and the growth fitness of the colony sizes is quantitatively scored to identify aggravating and alleviating interactions.

Figure 4. Representative computational analyses of genetic interaction (GI) scores for determining pathway-level functional relationships. (A) Panel I, Distribution of correlation coefficients between the GI profiles of gene pairs encoding proteins linked by protein-protein interactions (PPI) versus randomly drawn gene pairs. The p-value was computed using the two-sample Kolmogorov-Smirnov (KS) test; Panel II, Scatter plot of correlated genetic profiles from rich media (RM) for two transporters (mdtI, mdtJ) that form a heterodimeric complex required for spermidine excretion. (B) The hierarchical clustering of the GI sub-network adapted from Butland et al.9 highlights the functional connectivity between components of the previously known Isc and Suf pathways with similar GI patterns. Pink represents aggravating (negative S-score) interactions, green represents alleviating (positive S-score) interactions and black represents absence of GI. A predicted novel component of the SUF pathway, ydhD, displays GIs with the members of the Isc pathway. (C) Pathway cross-talk recorded among cell envelope processes that are significantly enriched for aggravating or alleviating GI according to the hypergeometric enrichment analysis27. (D) A GI sub-network predicts the role of two functionally unannotated genes, yceG and yebA, in peptidoglycan splitting based on their pattern of strong alleviating interactions with well-known cell division peptidoglycan hydrolases. The figures shown in panels A, C and D are adapted from Babu et al. (2011)12. Click here to view larger figure.
Discussione
Abbiamo delineato un graduale protocollo per l'utilizzo di robot per studiare lo screening eSGA funzioni geniche batteriche a livello percorso interrogando GI. Questo approccio può essere utilizzato per studiare singoli geni sia interi sistemi biologici in E. coli. Cura l'esecuzione delle fasi sperimentali di cui sopra, tra cui tutti i controlli del caso, e rigorosamente in maniera indipendente l'analisi e la validazione dei dati GI sono aspetti chiave per il successo di eSGA nel fare nuove scoperte funzionali. Oltre a eSGA, un metodo concettualmente simile per studiare GI in E. coli, denominato GIANT-coli 17, può essere utilizzato per illuminare nuove relazioni funzionali che sono spesso mancati da altri approcci, come proteomica 23 o 13 schermate fenotipici soli. Tuttavia, come con qualsiasi approccio genome-wide, esistono limitazioni in merito all'applicabilità di eSGA o GIANT-coli, ad esempio per le altre specie batteriche. Ciò è in parte esserela causa della indisponibilità di genome-wide singolo gene collezioni ceppo mutante di delezione per la maggior parte delle specie batteriche, in particolare per le specie in cui è ostacolato mutagenesi di mira da una bassa frequenza naturale di ricombinazione omologa. In tali casi, le interruzioni gene usando metodi quali la mutagenesi casuale tracciabili saggi basati come TnSeq 31 può potenzialmente essere usato per indagare funzioni geniche batteriche. Per una panoramica delle nuove procedure alternative batteriche screening genetico, vedi articolo di rassegna di Gagarinova e Emili (2012) 32.
Anche se i metodi GI spesso rivelano molti interessanti connessioni suggestiva di nuovi collegamenti meccanicistici, l'integrazione di questi dati con altre informazioni come profilo fenotipico 13, reti di interazione fisica 22, 23, e GC-inferiti associazioni può essere particolarmente informativo. Per esempio, come per le reti GI, associazioni GC, che si basano sulla conservazionedi ordine gene (operoni), fusioni di geni batterici, frequenze di ricombinazione operone derivati da distanze intergeniche di operoni previsti attraverso genomi, nonché profiling filogenetico 33 possono fornire informazioni aggiuntive su come una cellula batterica organizza complessi proteici in percorsi funzionali di mediare e coordinare principali processi cellulari 12, 23.
E. coli è un modello cavallo di lavoro per comprendere la biologia molecolare di altri batteri Gram-negativi. A tal fine, i dati GI eSGA può essere utilizzato in combinazione con le informazioni comparative genomica (filogenetica ad esempio profili, profili di espressione genica) per studiare la conservazione evolutiva delle relazioni funzionali scoperti da eSGA in altre specie e taxa proteobacterial procariota. Inoltre, i dati generati per interazione E. coli possono essere utilizzati al fine di conoscere l'architettura via dei microbi scoperti da metagenomicrofoni, per il quale le annotazioni funzionali sono carenti. Poiché molti geni sono ampiamente conservata tra tutti i microbi 23, e poiché la sensibilità agli antibiotici può essere migliorata da certe perturbazioni genetiche 34, le relazioni funzionali illuminate da eSGA può anche potenzialmente essere sfruttata per progettare innovative terapie farmacologiche combinate.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Questo lavoro è stato sostenuto da fondi del Genome Canada, l'Ontario Institute Genomica e Istituti canadesi di borse di ricerca per la salute e JG AE AG è un destinatario della borsa di studio Vanier Canada Graduate.
Materiali
| Name | Company | Catalog Number | Comments |
| I. Antibiotics | |||
| Chloramphenicol | Bioshop | #CLR201 | |
| Kanamycin | #KAN201 | ||
| Ampicillin | # AMP201 | ||
| 2. Luria-Bertani medium | |||
| LB powder | Bioshop | #LBL405 | |
| Agar | Bioshop | #AGR003 | |
| 3. Bacterial Strains and Plasmids | |||
| Hfr Cavalli strain λred system (JL238) | Babu et al.14. | ||
| pKD3 | E. coli Genetic Stock Centre, Yale | ||
| Keio E. coli F- recipient collection | National BioResource Project (NBRP) of Japan11 | ||
| Hypomorphic E. coli F- SPA-tag strains | Open biosystems; Babu et al.14 | ||
| 4. Primers | |||
| pKD3-based desalted constant primers | F1: 5'-GGCTGACATGGGAATTAGC-3' R1: 5'-AGATTGCAGCATTACACGTCTT-3' | ||
| Desalted custom primers | Cm-R: 5'-TTATACGCAAGGCGACAAGG-3' Cm-F: 5'- GATCTTCCGTCACAGGTAGG-3' | ||
| Desalted custom primers | F2 and R2: 20 nt constant regions based on pKD3 sequence and 45 nt custom homology regions F2 constant region: 5'-CATATGAATATCCTCCTTA-3' R2 constant region: 5'-TGTGTAGGCTGGAGCTGCTTC-3'S1 and S2: 27 nt constant regions for priming the amplification of the SPA-Cm cassette and 45 nt custom homology regions S1 constant region: 5'AGCTGGAGGATCCATGGAAAAGAGAAG -3' S2 constant region: 5'- GGCCCCATATGAATATCCTCCTTAGTT -3' KOCO-F and KOCO-C: 20 nt primers 200 bp away from the non-essential gene deletion site or the essential gene SPA-tag insertion site | ||
| 5. PCR and Electrophoresis Reagents | |||
| Taq DNA polymerase | Fermentas | # EP0281 | |
| 10X PCR buffer | Fermentas | # EP0281 | |
| 10 mM dNTPs | Fermentas | # EP0281 | |
| 25 mM MgCl2 | Fermentas | # EP0281 | |
| Agarose | Bioshop | # AGA002 | |
| Loading dye | NEB | #B7021S | |
| Ethidium bromide | Bioshop | # ETB444 | |
| 10X TBE buffer | Bioshop | # ETB444.10 | |
| Tris Base | Bioshop | # TRS001 | |
| Boric acid | Sigma | # T1503-1KG | |
| 0.5 M EDTA (pH 8.0) | Sigma | # B6768-500G | |
| DNA ladder | NEB | #N3232L | |
| 6. DNA isolation and Clean-up Kits | |||
| Genomic DNA isolation and purification kit | Promega | #A1120 | |
| Plasmid Midi kit | Qiagen | # 12143 | |
| QIAquick PCR purification kit | Qiagen | #28104 | |
| 7. Equipment for PCR, Transformation and Replica-pinning | |||
| Thermal cycler | BioRad, iCycler | ||
| Agarose gel electrophoresis | BioRad | ||
| Electroporator | Bio-Rad GenePulser II | ||
| 0.2 cm electroporation cuvette | Bio-Rad | ||
| 42 °C water bath shaker | Innova 3100 | ||
| Beckman Coulter TJ-25 centrifuge | Beckman Coulter | ||
| 32 °C shaker | New Brunswick Scientific, USA | ||
| 32 °C plate incubator | Fisher Scientific | ||
| RoToR-HDA benchtop robot | Singer Instruments | ||
| 96, 384 and 1,536 pin density pads | Singer Instruments | ||
| 96 or 384 long pins | Singer Instruments | ||
| 8. Imaging Equipments | |||
| Camera stand | Kaiser | ||
| Digital camera, 10 megapixel | Any Vendor | ||
| Light boxes, Testrite 16" x 24" units | Testrite | ||
| 9. Pads or Plates Recycling | |||
| 10% bleach | Any Vendor | ||
| 70% ethanol | Any Vendor | ||
| Sterile distilled water | Any Vendor | ||
| Flow hood | Any Vendor | ||
| Ultraviolet lamp | Any Vendor | ||
| 10. Labware | |||
| 50 ml polypropylene tubes | Any Vendor | ||
| 1.5 ml micro-centrifuge tubes | Any Vendor | ||
| 250 ml conical flaks | VWR | # 29140-045 | |
| 15 ml sterile culture tubes | Thermo Scientific | # 366052 | |
| Cryogenic vials | VWR | # 479-3221 | |
| Rectangular Plates | Singer Instruments | ||
| 96-well and 384-well microtitre plates | Singer Instruments | Nunc | |
| Plate roller for sealing multi-well | Sigma | #R1275 | |
| plates | ABgene | # AB-0580 | |
| Adhesive plate seals | Fisher Scientific | # 13-990-14 | |
| -80 °C freezer | Any Vendor | ||
Riferimenti
- Bandyopadhyay, S., Kelley, R., Krogan, N. J., Ideker, T. Functional maps of protein complexes from quantitative genetic interaction data. PLoS Comput. Biol. 4, e1000065 (2008).
- Costanzo, M., Baryshnikova, A., Myers, C. L., Andrews, B., Boone, C. Charting the genetic interaction map of a cell. Curr. Opin. Biotechnol. 22, 66-74 (2011).
- Costanzo, M., et al. The genetic landscape of a cell. Science. 327, 425-4231 (2010).
- Fiedler, D., et al. Functional organization of the S. cerevisiae phosphorylation network. Cell. 136, 952-963 (2009).
- Roguev, A., et al. Conservation and rewiring of functional modules revealed by an epistasis map in fission yeast. Science. 322, 405-4010 (2008).
- Schuldiner, M., et al. Exploration of the function and organization of the yeast early secretory pathway through an epistatic miniarray profile. Cell. 123, 507-519 (2005).
- Wilmes, G. M., et al. A genetic interaction map of RNA-processing factors reveals links between Sem1/Dss1-containing complexes and mRNA export and splicing. Mol. Cell. 32, 735-746 (2008).
- Babu, M., et al. Systems-level approaches for identifying and analyzing genetic interaction networks in Escherichia coli and extensions to other prokaryotes. Mol. Biosyst. 12, 1439-1455 (2009).
- Butland, G., et al. coli synthetic genetic array analysis. Nat. Methods. 5, 789-7895 (2008).
- Typas, A., et al. Regulation of peptidoglycan synthesis by outer-membrane proteins. Cell. 143, 1097-10109 (2010).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Mol. Syst. Biol. 2, 2006-200008 (2006).
- Babu, M., et al. Genetic interaction maps in Escherichia coli reveal functional crosstalk among cell envelope biogenesis pathways. PLoS Genet. 7, e1002377 (2011).
- Nichols, R. J., et al. Phenotypic landscape of a bacterial cell. Cell. 144, 143-156 (2011).
- Babu, M., Gagarinova, A., Greenblatt, J., Emili, A. Array-based synthetic genetic screens to map bacterial pathways and functional networks in Escherichia coli. Methods Mol Biol. 765, 125-153 (2011).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc. Natl. Acad. Sci. U.S.A. 97, 6640-665 (2000).
- Yu, D., et al. An efficient recombination system for chromosome engineering in Escherichia coli. Proc. Natl. Acad. Sci. U.S.A. 97, 5978-5983 (2000).
- Typas, A., et al. quantitative analyses of genetic interactions in. E. coli. Nat. Methods. 5, 781-787 (2008).
- Zeghouf, M., et al. Sequential Peptide Affinity (SPA) system for the identification of mammalian and bacterial protein complexes. J. Proteome Res. 3, 463-468 (2004).
- Davierwala, A. P., et al. . , 1147-1152 (2005).
- Breslow, D. K., et al. A comprehensive strategy enabling high-resolution functional analysis of the yeast genome. Nat. Methods. 5, 711-718 (2008).
- Babu, M., et al. Sequential peptide affinity purification system for the systematic isolation and identification of protein complexes from Escherichia coli. Methods Mol. Biol. 564, 373-400 (2009).
- Butland, G., et al. Interaction network containing conserved and essential protein complexes in Escherichia coli. Nature. 433, 531-537 (2005).
- Hu, P., Janga, S. C., Babu, M., Diaz-Mejia, J. J., Butland, G., et al. Global functional atlas of Escherichia coli encompassing previously uncharacterized proteins. PLoS Biol. 7, (2009).
- Anderson, R. P., Roth, J. R. Tandem genetic duplications in phage and bacteria. Annu. Rev. Microbiol. 31, 473-505 (1977).
- Boone, C., Bussey, H., Andrews, B. J. Exploring genetic interactions and networks with yeast. Nat. Rev. Genet. 8, 437-449 (2007).
- Collins, S. R., et al. Functional dissection of protein complexes involved in yeast chromosome biology using a genetic interaction map. Nature. 446, 806-8010 (2007).
- Le Meur, N., Gentleman, R. Modeling synthetic lethality. Genome Biol. 9, R135 (2008).
- Tong, A. H., et al. Systematic genetic analysis with ordered arrays of yeast deletion mutants. Science. 294, 2364-2368 (2001).
- Tong, A. H., et al. Global mapping of the yeast genetic interaction network. Science. 303, 808-813 (2004).
- Wong, S. L., et al. Combining biological networks to predict genetic interactions. Proc. Natl. Acad. Sci. U.S.A. 101, 15682-15687 (2004).
- van Opijnen, T., Bodi, K. L., Camilli, A. Tn-seq: high-throughput parallel sequencing for fitness and genetic interaction studies in microorganisms. Nat. Methods. 6, 767-772 (2009).
- Gagarinova, A., Emili, A. Genome-scale genetic manipulation methods for exploring bacterial molecular biology. Mol. Biosyst. 8, 1626-1638 (2012).
- Dewey, C. N., et al. Positional orthology: putting genomic evolutionary relationships into context. Brief Bioinform. 12, 401-412 (2011).
- St Onge, R. P., et al. Systematic pathway analysis using high-resolution fitness profiling of combinatorial gene deletions. Nat. Genet. 39, 199-206 (2007).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon