Method Article
La cartographie des réseaux fonctionnels bactériens et voies de
Dans cet article
Résumé
Systématiques et à grande échelle de synthèse génétiques écrans d'interaction gène-gène (ou épistasie) peut être utilisé pour explorer la redondance génétique et la voie de la diaphonie. Ici, nous décrivons un haut débit quantitatif synthétique de la technologie génétique de dépistage tableau, appelé ESGA que nous avons développé pour élucider les relations épistatiques et l'exploration des réseaux d'interactions génétiques Escherichia coli.
Résumé
Phénotypes sont déterminés par une série complexe de physique (par exemple protéine-protéine) et fonctionnelle (p. ex gène-gène ou génétique) des interactions (GI) 1. Alors que les interactions physiques peuvent indiquer quelles protéines bactériennes sont associées sous forme de complexes, ils ne révèlent pas nécessairement la voie au niveau relationships1 fonctionnels. Écrans GI, dans lequel la croissance des doubles mutants porteurs de deux gènes délétés ou inactivés est mesurée et comparée aux simples mutants correspondants, peut éclairer les dépendances entre les loci épistatiques et donc de fournir un moyen pour interroger et découvrir de nouveaux liens fonctionnels 2. Cartes à grande échelle GI ont été rapportés pour les organismes eucaryotes comme les levures 3-7, mais les informations restent rares IG pour les procaryotes 8, ce qui entrave l'annotation fonctionnelle des génomes bactériens. À cette fin, nous et d'autres avons développé à haut débit quantitatives bactériennes méthodes de dépistage GI 9, 10 Nous présentons ici les principales étapes requises pour effectuer quantitative E. coli synthétique procédure de dépistage génétique Array (ESGA) sur une échelle du génome 9, en utilisant la conjugaison bactérienne naturelle et la recombinaison homologue pour générer et systémique pour évaluer la justesse d'un grand nombre de doubles mutants dans un format de tableau colonie. bref, un robot est utilisé pour transférer , grâce à la conjugaison, le chloramphénicol (Cm) - a marqué allèles mutants de Hfr ingénierie (haute fréquence de recombinaison) «souches donateurs dans un ensemble ordonné de kanamycine (Kan) - marqués F-bénéficiaires souches. Typiquement, nous utilisons la perte de fonction simples mutants portant des délétions de gènes non essentiels (par exemple, la collection «Keio '11) et des mutations de gènes essentiels hypomorphes (allèles conférant à savoir l'expression des protéines réduite, la stabilité ou l'activité 9, 12, 13) interroger les associations fonctionnelles de gènes non essentiels et indispensables, respectivement. Après conjugaison et qui a suivi l'échange génétique induite par recombinaison homologue, les mutants résultants doubles sont sélectionnés sur milieu solide contenant deux antibiotiques. Après conséquence, les plaques sont numériquement imagée et la taille des colonies sont quantitativement classé par un système interne automatisé de traitement d'image 14. Les IG sont révélés lorsque le taux de croissance d'un double mutant est soit significativement meilleure ou pire que 9 attendu. Aggravante (ou négative) des IG entraînent souvent entre la perte de fonction des mutations dans les gènes de paires de voies compensatoires qui empiètent sur le même processus essentiel 2. Ici, la perte d'un seul gène est tamponnée, de sorte que soit seul mutant est viable. Cependant, la perte des deux voies est délétère et les résultats de létalité synthétique ou de maladie (c.-à croissance lente). A l'inverse, apaisantes (ou positif) des interactions entre les gènes peuvent se produire dans la même voie ou de protéines complexes 2 commedélétion de gène supporte seul est souvent suffisante pour perturber le fonctionnement normal de la voie ou complexes tels que des perturbations supplémentaires ne réduisent pas l'activité, et donc la croissance, en outre. Dans l'ensemble, l'identification systématique et l'analyse des réseaux IG peut fournir sans parti pris, des cartes mondiales des relations fonctionnelles entre un grand nombre de gènes, d'où la voie au niveau de l'information manquée par d'autres approches peuvent être inférées 9.
Protocole
1. Construction de souches Hfr donatrices Cavalli Mutant par Recombineering 15, 16
Les étapes de la construction des taches donateurs ESGA sont décrits ci-dessous. En bref, nous utilisons λ ciblée - Rouge médiation recombinaison homologue 16 de cassette d'ADN amplifié marqueur de sélection des fragments générés par PCR pour créer non essentiels mutants de délétion de gènes (section 1.1) ou de gènes essentiels hypomorphes souches mutantes donateurs (section 1.2), qui sont ensuite utilisés comme "requêtes" pour définir les réseaux gastro-intestinaux.
Remarque: Pendant le processus de développement de la technologie, nous avons évalué l'efficacité de l'aide Hfr médiée par transfert conjugatif pour combiner des mutations en utilisant une souche bien défini donateurs Hfr (Hfr Cavalli, Hfr Hayes, et Hfr 3000). Nous avons examiné (i) la capacité de faire des mutants efficace des bailleurs de fonds en utilisant la méthode recombineering mis au point par Yu et al. (2000) 16, (ii) l'efficacité relativede transfert d'ADN par conjugaison de différents marqueurs chromosomiques, et (iii) les effets de la position et de l'orientation du gène chromosomique de requête par rapport à la Hfr lieu de transfert, oriT. Nous avons constaté que λ - Rouge médiée par l'efficacité de recombinaison homologue était beaucoup plus élevé dans Hfr Cavalli que dans Hfr Hayes ou Hfr3000. Si nécessaire, la transduction P1 ou une souche Hfr Psuedo 17, peut être utilisé pour créer des mutants donateurs. Nous avons adapté avec succès tous ces procédés de fabrication et dépister un grand nombre de bailleurs de fonds. Comme de nos expériences de conjugaison d'essai, l'efficacité globale du transfert et le nombre des ex-conjuguants observée était significativement plus élevée avec Hfr Cavalli, cette toile de fond souche particulière a été choisi pour la construction des bailleurs de fonds et à grande échelle ESGA. Toutes les procédures d'écrans ESGA utilisant Hfr Cavalli donateurs sont décrits.
- Amplifier le fragment d'ADN à la suite de recombineering pour supprimer un gène non essentiel
Employer deux étapes de PCR nichée amplication (Figure 1) pour créer une cassette de délétion génique pour le remplacement du cadre de lecture ouvert cible avec un marqueur de résistance à Cm partir du plasmide pKD3 (GI: 15554330; protéines Identifiant: AAL02033.1) 15. Le marqueur est flanqué par l'un des sites FRT-11 permettant l'élimination du gène de résistance, si nécessaire, dans des expériences de suivi.
Remarque: Nous ne imbriqué à deux étapes de PCR pour éliminer le plasmide à partir du produit amplifié qui est utilisé par la suite pour transformer des cellules bactériennes. Retrait du plasmide (dans la première PCR) et en créant ensuite la cassette inactivation du gène dans la deuxième PCR est nécessaire parce que la transformation avec un plasmide est beaucoup plus efficace que recombineering, et l'objectif est d'obtenir - autant que possible - les souches qui ont réussi à remplacer l'ADN génomique cible avec le marqueur de sélection. Une alternative à la PCR nichée est d'effectuer une digestion par restriction de l'extérieur de la région du plasmide amplifié dans la seconde PCRl'étape. Puis le produit de la digestion de restriction est purifié et utilisé comme matrice dans l'étape de PCR nichée (1.1.2). Cette option est également simple, mais nécessite plus de travail le temps.- Utiliser environ 45 ng de pKD3 comme matrice dans une PCR avec de l'avant et de marche arrière amorces F1 R1 pour produire un produit de 1.070 pb. Purifier le fragment de PCR en utilisant le protocole Qiagen PCR purification, et de diluer le produit dans de l'eau distillée stérile à 5 ng / ul concentration.
- Utilisation du produit de PCR purifié comme modèle, mis en place une seconde PCR avec spécifiques du gène (knock-out par exemple) des amorces emboîtées, F2 et R2, qui ont (i) 45 nt des régions d'homologie spécifiques du gène à l'extrémité 5 se termine pour permettre pour la recombinaison homologue immédiatement en amont et en aval du gène cible à la remplacer par le marqueur de résistance Cm et (ii) 20 nt au niveau des extrémités 3 'pour amorcer la synthèse du marqueur Cm. La taille du fragment est produit 1.123 pb. Les régions d'homologie spécifiques du gène sont conçus pour éliminer l'op cibleen cadre de lecture tout en laissant les segments adjacents ou se chevauchant partiellement codage intacte. Après cette amplification, passez à l'étape 1.3.
- Amplifiant une cassette de marquage pour la création d'une mutation du gène essentiel hypomorphes
- Pour construire des souches mutantes hypomorphes donateurs, ajoutez un peptide C-terminal séquentielle marqueur d'affinité de fusion (SPA), qui ont souvent un impact légèrement la fonction des protéines essentielles par abondance des transcrits de déstabilisation (nombre de protéines copie) ou repliement des protéines / activité. À cette fin, de créer une matrice d'ADN sélectionnable SPA-tag (SPA-Cm) en convertissant le marqueur de résistance Kan à pJL148 18 à Cm en utilisant recombineering entre séquences nucléotidiques identiques flanquant le marqueur Kan à pJL148 et marqueurs Cm dans pKD3 15. Le modèle résultant comprend en cadre SPA-tag suivi d'un marqueur de résistance à Cm, et est approprié pour l'amplification par PCR, et les suivants recombineering ESGA 12.
Remarque: </ Strong> gènes essentiels sont potentiellement les plus intéressants pour les études de la fonction des gènes. Cependant, parce que ces gènes ne peuvent pas être supprimés, des méthodes innovantes ont dû être développés pour étudier les relations fonctionnelles de gènes essentiels. Plusieurs démarches idoines très réussies ont été développées initialement dans la levure. Par exemple, Davierwala et al. (2005) 19 écrans effectuées à l'aide d'un panneau GI de 575 mutants sensibles à la température de gènes essentielles, et constaté que les gènes essentiels exposé d'environ cinq fois le nombre des IG en tant que gènes non essentiels. De même, Breslow et al. (2008) 20 développé une «abondance a diminué de perturbation ARNm» (humide) approche pour générer une bibliothèque d'allèles hypomorphes couvrant environ 82% de gènes de levure essentielles pour étudier les voies et complexes. L'approche humide modifie l'extrémité 3 'de l'ARNm exprimé en réduisant le niveau du gène cible 6, probablement en raison de l'ARNm de déstabilisation. Similaire à l'approche humide 6, notre stratégy est d'exploiter l'effet perturbateur de subtils extrémité 3 'de l'ARNm exprimé de protéines essentielles.
Comme nous le décrivons ci-dessous, l'étiquette en elle-même ne devrait affecter le repliement des protéines ou de la fonction, mais la perturbation subtile de l'ARNm par la balise est suffisante, lorsqu'il est combiné avec les facteurs de stress environnementaux ou d'autres mutations, de révéler fonctionnellement informative gène-environnement 13 et du gène 9-gène, interactions 12.
Tout d'abord, à ce jour, nous avons utilisé SPA-tags pour purifier ~ 3.000 essentiels et non essentiels protéines de E. coli 21-23, avec plus de 80% des E. coli 307 protéines essentielles étant représentés parmi nos souches SPA-étiquette 8. Parce que l'étiquette a été utilisée avec succès pour isoler les complexes protéiques connues, qui ont été validés réciproquement (c'est à dire le même complexe a pu être isolé par le marquage et la purification de protéines différentes à partir du même complexe), et les complexes isolés étaient biologiquement pertinente, nous raisonnons que les balises fairedevrait pas nuire à repliement des protéines ou de la fonction 12. De même, nos observations non publiées ont révélé que l'étiquette en elle-même ne devrait pas nuire à la croissance souche. Cependant, nous avons pensé que la modification de la 3'-UTR en intégrant l'étiquette et la cassette de marqueur, comme cela a été rapporté pour les souches humides 6, pourrait déstabiliser certaines transcriptions sur le niveau de l'ARN ou, en de rares occasions, empêchent le repliement correct ou la fonction du des protéines de fusion. Par conséquent, nous avons prédit que la combinaison de ces perturbé allèles de gènes essentiels avec d'autres mutations fonctionnellement pertinents ailleurs dans le génome se traduirait par des IG informative, qui fait a été observé 9, 12. Allèles Par ailleurs, SPA-étiquette de gènes essentiels exposés interactions gènes-environnement (hypersensibilité au médicament, par exemple) en fonction du travail de profilage phénotypique par Nichols et al. (2011) 13, qui a soumis un sous-ensemble de SPA-étiquette souches essentiels à une variété de conditions de croissance. La même étude CONFIRMEd que la plupart (54 sur 58 testés) SPA-étiqueté souches a montré essentiels de remise en forme altérée dans une ou plusieurs des conditions de culture 13, qui soutient l'idée que le SPA-tag crée souvent bénins allèles hypomorphes de gènes essentiels qui sont utiles pour l'ensemble du génome applications à écran GI, y compris ESGA. - Amplifier la cassette de marquage à partir du modèle SPA-cm à l'aide d'amorces spécifiques du gène S1 et S2, contenant chacun (i) un 45 nt de la région spécifique du gène homologue à recombineering à l'extrémité 5 ', suivi par (ii) un 27 pb SPA- cm séquence spécifique à l'extrémité 3 '.
- Pour construire des souches mutantes hypomorphes donateurs, ajoutez un peptide C-terminal séquentielle marqueur d'affinité de fusion (SPA), qui ont souvent un impact légèrement la fonction des protéines essentielles par abondance des transcrits de déstabilisation (nombre de protéines copie) ou repliement des protéines / activité. À cette fin, de créer une matrice d'ADN sélectionnable SPA-tag (SPA-Cm) en convertissant le marqueur de résistance Kan à pJL148 18 à Cm en utilisant recombineering entre séquences nucléotidiques identiques flanquant le marqueur Kan à pJL148 et marqueurs Cm dans pKD3 15. Le modèle résultant comprend en cadre SPA-tag suivi d'un marqueur de résistance à Cm, et est approprié pour l'amplification par PCR, et les suivants recombineering ESGA 12.
- Confirmer et purifier le produit de PCR
- Assurez-vous que la cassette de marquage a été correctement amplifié en soumettant 2-5 ul du produit de PCR pour électrophorèse sur gel agarose et purification de l'ADN restant après la norme du fabricant protocole de purification PCR (Qiagen). Éluer le produit de PCR dans 30 ul d'eau distillée stérile et diluer à environ 50 ng / ul concentration. La purificationproduit ed peuvent être utilisées immédiatement dans la transformation (étape 1.5) ou stockés à -20 ° C pendant jusqu'à 6 mois jusqu'à leur utilisation.
- Préparation des cellules Hfr Cavalli compétentes pour la construction des bailleurs de fonds
- Inoculer un ml de la culture saturée du jour au lendemain d'une souche Hfr Cavalli dans 70 ml de frais de Luria-Bertani (LB) avec 35 ul de 50 pg d'ampicilline / ml dans un flacon de 250 ml. Incuber la culture à 32 ° C sous agitation à 220 rpm jusqu'à une densité optique (DO) à 600 nm de 0,5 à 0.6 ~ est obtenue (~ 2 h).
- Transférer la culture à un bain d'eau pour l'induction de chaleur du système de recombinaison λ rouge 16 à 42 ° C pendant 15 min sous agitation à 160 rpm. Arrêter le transfert de l'induction de la culture à un bain d'eau glacée coulis de glace pour 10-20 min à 160 tours par minute. Assurez-vous de garder les cellules à froid à partir de ce point jusqu'à ce que, après transformation; tubes utilisation et des cuvettes qui ont été emballées dans de la glace pendant au moins 15 min avant de les utiliser.
- Divisez la culture également en 2 pré-réfrigérés tubes de 50 ml en polypropylène (~ 35 ml de culture par tube) et centrifuger à 4400 xg pendant 6 min à 4 ° C.
- Jeter le surnageant et remettre en suspension dans la cellule par inversion douce en tablettes ~ 50 ml d'eau glacée distillée stérile. Centrifuger les cellules à nouveau à 4400 xg pendant 6 min à 4 ° C. Jeter le surnageant et remettre en suspension chaque culot cellulaire dans 20 ml de glace-froid glycérol 10% stérile et centrifuger à nouveau comme décrit ci-dessus.
- Décanter le surnageant et remettre en suspension le culot cellulaire dans 500 ul de glace froide glycerol à 10%. Aliquote des cellules compétentes préparées dans des volumes de 50 ul dans chaque pré-réfrigérés 1,5 ml de micro-centrifugation des tubes pour la transformation immédiate. Les cellules peuvent être congelés sur glace sèche ou dans l'azote liquide et conservés à -80 ° C pendant 3 mois. Toutefois, le rendement énergétique est la plus élevée fraîchement préparés avec des cellules compétentes.
- Transformation
- Ajouter 100 ng de produit PCR purifié à partir del'étape 1.3.1 pour les cellules compétentes. Basculez le tube et permettre la suspension de s'asseoir sur la glace pendant 5 min.
- Transférer la suspension de la glace-froid et de l'ADN des cellules compétentes d'une cuvette d'électroporation pré-réfrigérés. Électroporer mélange de cellules avec l'kV 2,5, 25 uF, 200 ohms plaçant dans un espace de 2 mm cuvette (c'est à dire si en utilisant une cuvette différente, s'il vous plaît voir les instructions du fabricant pour les paramètres d'électroporation), et ajouter immédiatement 1 ml de milieu SOC température ambiante. Transférer les cellules électroporées en milieu SOC dans un tube de culture de 15 ml et incuber à 32 ° C pendant 1 heure sous agitation orbitale à 220 rpm.
- Après incubation, centrifuger les cellules à 4400 g pendant 5 min à température ambiante. Retirez environ 850 pl du surnageant et remettre en suspension le culot cellulaire dans le liquide restant.
- Etaler les cellules sur des plaques LB préchauffées contenant 17 pg / ml Cm, et incuber à 32 ° C pendant la nuit. Choisissez deux colonies transformantes individuelles et traînée sur LB-Cm plaques de confirmation mutant. Nous vous recommandons d'éviter plus grandes colonies transformées afin de réduire la possibilité de choisir des souches de rares mutations suppresseurs. Aussi série des transformants sur LB-mêmes Kan pour confirmer que les souches résistantes ne sont pas Kan.
Remarque: Étant donné que le marqueur de résistance à Cm est utilisé pour fabriquer les souches donneuses, nous ne pensons pas que les mutants donateurs d'être Kan résistant. En striant les transformants sur les deux LB-Kan et LB-Cm plaques, nous nous assurons que la mutation donateur lui-même n'a pas en quelque sorte provoquer une résistance Kan. Si cette étape n'a pas été réalisée et les mutations cibles étaient d'affecter la capacité de la souche à survivre Kan, doubles mutants ne se ferait pas de manière efficace en utilisant ESGA depuis souches donneuses survivraient toutes les étapes de sélection. Il s'agit simplement d'une mesure de précaution logique pour attraper phénotypes inattendus ou tout autre problème qui pourrait nuire à la réalisation des écrans ESGA. Si cette étape ont été ignorés, woul autres contrôlesD Still identifier les écrans défaillants. Cependant, une détection plus précoce d'un donneur qui est résistant à Kan permettrait d'éliminer une série de mesures improductives et inutiles. Autres raisons possibles pour une souche à croître sur Kan pourrait inclure par exemple expiré antibiotiques ainsi que l'utilisation d'un mauvais produit PCR ou d'une souche de la transformation. Tous ces problèmes peuvent être identifiés et traités dès le début de la procédure à l'aide de cette commande, qui n'est pas obligatoire, mais recommandé pour ESGA.
- Confirmation de la suppression du gène non essentiel (étape 1.6.1) ou mutation du gène essentiel hypomorphes (section 1.6.2)
Remarque: Lors de la confirmation de la délétion du gène par PCR, l'ADN génomique d'une souche de type sauvage doit être utilisé comme un contrôle.- Confirmant la délétion du gène non essentiel
- Pour confirmer la suppression du gène par PCR, isoler l'ADN génomique à partir des souches transformées, knockout putatifs cultivées en liquide LB-Cm milieu, à la suite de la Marquerier les instructions (Promega).
- Pour confirmer recombineering succès, amplifier l'ADN de chaque transformant avec trois différents ensembles d'amorces de confirmation huitièmes de finale. Effectuez les réactions avec ~ 150 ng de matrice d'ADN génomique, et de confirmer la taille des fragments corrects en exécutant les produits PCR synthétisés sur un gel d'agarose.
- Le jeu d'amorces comprend une première 20-nt amorce latérale, située à 200 pb en amont de la région ciblée (KOCO-F), et Cm-R amorce qui est complémentaire de la séquence de la cassette Cm. Cette amplification est prévu pour produire un amplicon 445 nt dans le mutant, mais pas dans la souche de type sauvage (figure 1).
- Le deuxième ensemble comprend une amorce sens (Cm-F), le recuit de la séquence de cassette cm, et une marche arrière 20-nt flanquant amorce de confirmation (KOCO-R), qui est conçue pour s'hybrider 200 pb en aval de l'extrémité 3 'de l' supprimé gène. Cette réaction d'amplification est prévu pour produire un amplicon 309 nt dans le mutant, mais past dans la souche de type sauvage (figure 1).
- Le troisième PCR contient KOCO-F et-R KOCO amorces. Cette réaction est nécessaire pour confirmer que la souche sélectionnée n'est pas un merodiploid, avec un locus du gène ayant été remplacé par la cassette et un autre dupliqué mais sinon copie du gène de type sauvage toujours présent. Cette amplification d'un mutant de délétion correcte est prévu pour produire un produit de 1,4 kb (figure 1).
Remarque: lorsque le gène cible a environ la même longueur que les paires de bases dans la cassette de délétion du gène, on ne constate une différence entre un lieu, qui a été remplacée par la cassette, et un lieu, qui n'a pas. Pour une confirmation définitive dans de tels cas, nous recommandons utilisant des amorces supplémentaires pour amplifier spécifiquement un segment d'ADN dans le gène supprimé. Dans ce cas, si le gène a été supprimé, un produit d'amplification sera observée que dans la souche témoin de type sauvage et non chez le mutant. Ces amorcespeut être manuelle ou de calcul conçu pour amplifier spécifiquement un segment d'ADN à l'intérieur de la région délétée. En général, nous amplifient 140 régions pb.
- Confirmant mutation du gène essentiel hypomorphes
- Pour confirmer la mutation hypomorphes par PCR, utilisez spécifique du gène 20-nt avant (KOCO-F) et amorces inverses (KOCO-R) situé à 200 pb en amont et en aval, respectivement, du site d'insertion SPA-tag (figure 2).
Remarque: Cette étape d'amplification est utilisé comme contrôle pour s'assurer de l'absence d'une copie non balisé du gène cible essentielle. Lorsque seule une version étiquetée est présent, une seule bande 310 pb plus grand que l'amplicon SPA-Cm est observée. Produit de 400 pb indiquerait la présence de la version du gène non marqué. - En parallèle à la confirmation par PCR, utilisez Western blot pour vérifier l'insertion dans la trame de la SPA-étiquette à l'aide d'un anticorps anti-FLAG M2 spécifique à l'option-épitopes de la SPUn tag-21.
- Pour confirmer la mutation hypomorphes par PCR, utilisez spécifique du gène 20-nt avant (KOCO-F) et amorces inverses (KOCO-R) situé à 200 pb en amont et en aval, respectivement, du site d'insertion SPA-tag (figure 2).
- Confirmant la délétion du gène non essentiel
- Stockage de la souche donneuse confirmé
- Transférer la culture d'une nuit de la souche donneuse recombinant mutant a confirmé les cryotubes étiquetés, compléter avec du glycérol à une concentration finale de 15%, et bien mélanger. Pour stockage à long terme, maintenir les cryotubes à -80 ° C.
2. Rangement de E. coli F-Recipient Mutants pour écrans ESGA
- Réplique broches de l'E. Keio coli unique non essentiel collection délétion du gène mutant généré par Mori et al. (3968 souches 11; F-BW25113, peuvent être obtenus auprès Ouvrir Biosystems) robotisée à vingt-quatre de 384 puits des microplaques contenant 80 pi de milieu LB liquide par puits , complété avec 50 ug / ml Kan
Remarque: Pour évaluer systématiquement les indications géographiques avec des gènes essentiels, le destinataire Keio collecte 11 peut être complétée par le F-Kan-marquée gène essentiel hypomorphsouches mutantes avec ic C-terminaux SPA-tags 22, 23. - Pour faire de la place pour la souche contrôle des frontières, qui agira comme témoin positif, l'aide à l'intérieur et entre les normalisations plaque ainsi que dans les quantifications colonie automatisés, retirez les milieux inoculés à partir des puits de chaque plaque ultrapériphériques de l'étape ci-dessus et le transfert de nouvelles plaques, laissant à nouveau les puits ultrapériphériques vide.
Remarque: La souche de contrôle aux frontières JW5028 de la collection Keio 11 est supprimé pour un petit gène pseudo qui ne devrait pas, et n'a pas dans nos génome entier écrans, présentent IG avec les bailleurs de fonds. Par conséquent, il s'agit d'une souche témoin positive pour une bonne identifier les rares cas où conjugaison, épingler ou les sélections ont échoué, entraînant une croissance frontière absent souche ou sporadique. Par exemple, si les colonies de contrôle aux frontières ne poussent pas après la sélection double mutant, la souche requête peut ne pas être capable de conjugaison, orthographe peut-être été tenté icondition de na qui l'a empêché d'avoir lieu (par exemple sur une plaque contenant des antibiotiques), ou un antibiotique mal peut avoir été ajouté à la plaque de sélection. De même, si très peu de colonies sporadiques sont observés sur toute la plaque, y compris la frontière, la conjugaison ou l'épinglage peut avoir échoué et l'écran doit être répété. Si cette croissance sporadique frontière est reproductible, cela peut indiquer la conjugaison des donateurs constamment faible et donc des cas où des doubles mutants ne sont pas observées sont peut-être due à une défaillance de conjugaison, pas vrai géographiques. Ces écrans devraient être mis au rebut de l'analyse. De plus, en étant présent sur toutes les plaques, la souche de contrôle aux frontières permet au logiciel de quantification colonie de déterminer automatiquement les positions des colonies sur les plaques doubles mutants et donc les positions des colonies n'ont pas à être délimitées manuellement. Enfin, les aides de contrôle des frontières de contrainte dans la normalisation de la taille des colonies à l'intérieur (par exemple, des lignes différentes dans une assiette) et entreen (par exemple des plaques de destinataires différents épinglé avec le même donneur à des moments différents) plaques. - De même, créer des points de contrôle négatif en supprimant les deux souches à partir d'un emplacement différent dans chaque plaque (pas de frontière) et de transférer les souches d'une nouvelle plaque. Remplissez ces puits vides avec du milieu LB contenant 17 pg / ml de Cm. Ces points de contrôle négatif doivent être vides chez le receveur et donc doubles mutants plaques et faire en sorte qu'il n'y avait pas de traitement ou des erreurs de manipulation lors de la numérotation de plaques, l'imagerie, ou épinglant les plaques.
Remarque: Ces espaces vides sont tenus contrôles négatifs. Les contrôles négatifs aider à identifier les cas où la sélection échoue - lorsque des souches de contrôle négatif se développer lors de la sélection. Nous vous suggérons de sélectionner non seulement uniques points de contrôle négatif pour chacune des plaques de bénéficiaires, mais aussi de choisir des taches de contrôle négatif dans le même quadrant de la plaque (par exemple, en bas à droite). De cette façon, le modèledes points de contrôle négatif serait d'identifier les erreurs éventuelles erreurs de manipulation de plaques (p.ex. aux plateaux classés à tort ou être coincé à l'envers, où la colonie en haut à gauche est tombé en position inférieure droite). - Inoculer la souche Keio JW5028 11, avec une suppression pseudogène, dans un flacon de 500 ml avec 200 ml de LB contenant 50 ug / ml de Kan Basé sur nos écrans ESGA du génome entier, la croissance de ce mutant particulier est le plus proche de sauvage Type BW25113 et il est donc utilisé comme un contrôle des frontières.
- Cultiver les souches mis en réseau ainsi que la culture JW5028 une nuit à 32 ° C avec 190 tr agitation orbitale à une DO d'environ 0,4 à 0,6 à 600 nm. Après la croissance durant la nuit, remplir les puits frontalières avec environ 80 pi de JW5028 culture d'une nuit.
- Les plaques assemblées peuvent être utilisés dans l'étape 3.2. Sinon, pour stockage à long terme, de compléter chaque puits dans les plaques bénéficiaires avec 15% de glycerol, mélanger les médias et le glycérol, et de garder les plaquesà -80 ° C.
3. Haute densité procédure d'accouplement de traction pour Génération E. Mutants coli double
- Cultiver la souche donneuse Hfr, portant la mutation requête, marqué par Cm, pendant une nuit à 32 ° C dans du milieu LB liquide riche-cm (17 ug / ml de Cm) avec agitation à 220 tours par minute.
- Epingler la collection destinataire a ordonné mutant 384-colonie densité sur des plaques LB solide additionné de 50 pg / ml de Kan En parallèle, épingler la souche mutante requête des bailleurs de fonds dans 384 colonies de densité sur le même nombre de plaques LB additionné de 17 pg / ml de Cm. Incuber les plaques pendant la nuit à 32 ° C.
Remarque: Les plaques receveuses utilisées pour l'épinglage peut nécessiter jusqu'à 36 heures pour l'obtention de colonies suffisamment grandes pour les étapes ultérieures. Lorsque la taille moyenne des colonies frontière est d'environ 2 mm de diamètre (ou plus), les plaques sont prêtes à être épinglées. - Pour conjuguer les souches, épingler le donateur d'une plaque 384-colonie donneuse nuit surà la plaque LB solide. Par la suite épingler un destinataire unique plaque 384-colonie sur le donateur fraîchement épinglé sur la plaque de conjugaison. Continuer jusqu'à ce que toutes les plaques épinglant entre donateurs et bénéficiaires ont été conjugués en épinglant sur de solides plaques LB conjugaison. Incuber les plaques à 32 épinglés conjugaison ° C pendant 16 à 24 heures.
Remarque: seul individu souches mutantes donateurs peuvent montrer gène-à-gène variabilité d'efficacité accouplement en raison des effets potentiels des mutations sur la conjugaison, l'efficacité de transfert d'ADN ou d'aptitudes. La croissance peut être arrêtée à tout moment entre 16 h et 24 h, lorsque les colonies suffisamment importantes ont été obtenues pour les étapes ultérieures d'ancrage. Conjugaisons d'une durée de 16 à 24 h produire un nombre suffisant d'ex-conjuguants des résultats reproductibles ESGA même pour les mutants de la fin du transfert de gènes ou des mutants de remise en forme avec un peu plus bas. - Pin chaque plaque de conjugaison 384-densité sur une plaque LB solide unique, contenant Cm (17 pg / ml) et Kan (50ug / ml) jusqu'à ce que toutes les plaques de conjugaison ont été épinglés. Incuber les plaques fraîchement épinglés première sélection pour 16-36 heures à 32 ° C.
Remarque: En ce qui concerne les étapes de sélection, différentes quantités de temps peut être nécessaire pour les écrans des différents donateurs. Comme tous les doubles mutants dans un écran donné la mutation même donneur, en moyenne, un mutant lent croissante des donateurs donne lieu à croissance plus lente des doubles mutants. Ainsi, en fonction de l'aptitude des bailleurs de fonds, les étapes de sélection peut exiger entre 16 et 36 heures. La croissance double mutant peut être arrêté lorsque les doubles mutants sont suffisamment grands pour l'étape suivante, soit après la sélection épinglage première ou après la seconde image. Cela se traduit généralement à border colonies de commande en moyenne d'au moins 2 mm de diamètre. - Une autre broche de chaque plaque de sélection sur une première seconde, deux médicaments (Cm et Kan), la plaque de sélection de 1536 colonies de format, de telle sorte que chaque colonie première sélection est représenté par quatre coloniesla plaque de sélection secondes. Incuber les boîtes pendant 16-36 h à 32 ° C. Photographier les finales doubles plaques de sélection de mutants de mesurer quantitativement l'aptitude croissance du mutant et d'analyser les interactions entre les paires de gènes (figure 3).
Remarque: Bien que la fréquence des merodiploidy (duplications partielles chromosomiques) est faible, à environ 1/1, 000 cellules 24, merodiploidy peut masquer GI. Par conséquent, nous recommandons d'inclure reproduit biologique dans tous les écrans ESGA. Heureusement, commerciaux et plaques en matière plastique à usage unique blocs d'épinglage peut être réutilisée jusqu'à trois fois par stérilisation des matériaux comme suit: des plaques de gélose est éliminé et les plaques ainsi que les tampons sont stérilisés en les trempant dans l'eau de Javel à 10% pendant une nuit, suivie d'une rincer à l'eau distillée, lavage à l'éthanol 70%, et séchage à l'air de la vaisselle en plastique dans une hotte à flux sous la lumière ultraviolette. Les compresses stériles et les plaques sont stockées dans des sacs en plastique scellés stériles jusqu'à leur utilisation.
4. Traitement des données et Issu scores GI
- Mesurer les colonies sur chaque plaque en mode batch ou individuellement à l'aide d'un logiciel spécialisé colonie d'imagerie 3, 9.
Remarque: Un système adéquat basé sur Java imagerie colonie à haut débit et l'application de pointage est maintenant disponible gratuitement pour un accès public ( http://srcollins77.users.sourceforge.net/ ). Le logiciel fonctionne sur différentes plates-formes (par exemple Mac ou PC) et peut être lancé comme un fichier exécutable ou d'une fenêtre de terminal. - Normaliser les mesures brutes taille des colonies en corrigeant les erreurs systématiques au sein et entre les plaques tels que les effets de bord de plaque, les effets de la variation inter-plaques, de l'éclairage, des artefacts d'image inégale en raison de la courbure physique de la surface de la gélose, les effets de la concurrence pour les colonies voisines et possibles défauts épinglage , ainsi que des différences de temps de croissance p> 9. Ces artefacts systématiques sont indépendants de la remise en forme mutante double et doit être corrigée, car elles donnent lieu à des estimations erronées de remise en forme de croissance.
- Les colonies dans les rangées et les colonnes de pointe ont tendance à être plus faible en raison de la compétition pour les nutriments que l'on trouve dans le centre de la plaque. Ainsi, afin de corriger cet effet, à l'échelle les dimensions des colonies de bord pour être telle que la taille médiane en ce que la ligne ou la colonne est égale à la taille moyenne des colonies dans le milieu de la plaque.
- Analyser les résultats statistiquement pour tenir compte de la reproductibilité et de la variance standard de la taille médiane des mesures répétées colonie. Au moins trois expériences répétées indépendantes biologiques sont nécessaires pour évaluer la reproductibilité expérimentale.
- Enfin, utilisez les médianes normalisées taille des colonies de croissance de conditionnement physique pour générer un score GI (S) pour chaque paire de gènes à l'aide de la formule suivante:
n 1 "src =" / files/ftp_upload/4056/4056eq1.jpg "/>
Où, S = var (var x exp (exp n -1) + var Cont x (n -1 Cont)) / (n + n Exp Cont -2); var = Exp la variance maximale de la taille des colonies normalisées pour l' double mutant; var cont = médiane des écarts dans les doubles normalisées taille des colonies mutantes de l'ensemble de référence; Exp n = nombre de mesures de la taille des colonies mutantes doubles; n = Cont le nombre médian de expérimental reproduit sur toutes les expériences; Exp μ = médiane taille des colonies normalisées des doubles mutants, et Cont μ = moyenne de la taille des colonies normalisés pour tous les doubles mutants provenant de la souche mutante seul donneur. Le S-score reflète à la fois la fiabilité statistique de l'interaction putative digénique ainsi que la force de l'interaction biologique. Fortement positifs S-scores indiquent des effets apaisants, suggesting que les gènes interagissent participer dans la même voie 9, alors que négatif significatif S-scores reflètent la maladie synthétique ou de létalité, qui est souvent suggestive de l'adhésion en parallèle voies redondantes 9. Pour déterminer les relations fonctionnelles au niveau du parcours, un seul S-score pour chaque paire de gènes testés est produit à partir de l'ensemble de données final.
Remarque: Dans notre étude précédente 9, nous avons trouvé que la recombinaison a tendance à être moins fréquentes entre les gènes à moins de 30 kpb de l'autre. Par conséquent, pour l'analyse en aval, après la normalisation et la génération de score, nous supprimons les interactions entre les gènes à moins de 30 kpb de l'autre. En plus des indications géographiques elles-mêmes, les relations fonctionnelles entre les paires de gènes peuvent être étudiés en regardant comment même les profils IG des deux gènes sont. Le profil GI d'un gène est l'ensemble de toutes les interactions de ce gène avec tous les autres gènes dans le génome de l'extérieurzone de liaison au gène. Gènes fonctionnellement similaires ont tendance à avoir des corrélations entre les profils génétiques d'interaction 12, 25. Ainsi, en calculant les coefficients de corrélation pour tous les gènes dans l'ensemble de données expérimentales, on peut utiliser pour rechercher ESGA relations fonctionnelles entre les gènes se trouvant encore près de l'autre sur le chromosome.
Résultats
GIs reveal functional relationships between genes. Similarly, since genes in the same pathway display similar GI patterns and the GI profile similarity represents the congruency of phenotypes, we can group functionally related genes into pathways by clustering their GI profiles. Integrating GI and GI correlation networks with physical interaction information or other association data, such as genomic context (GC) relationships can also reveal the organization of higher-order functional modules that define core biological systems (and system crosstalk) in bacteria. For example, as with yeast4, 6, 7, 26, E. coli gene pairs exhibiting alleviating interactions that also have highly correlated GI profiles tend to encode proteins that are either physically associated (e.g. form a complex; Figure 4a) or that act coherently in a common biochemical pathway (e.g. components in a linear cascade). A representative example is shown in Figure 4b, where the components of the functionally redundant Isc and Suf pathways, which jointly participate in the essential Fe-S biosynthesis process, form distinct clusters that are linked together by extensive aggravating interactions (i.e. synthetic lethality). A statistical measure (e.g. hypergeometric distribution function27) can be used on GI or GI correlation data to find significant enrichment for interactions between and within pathways (Figure 4c). Cluster analysis can also be applied to functional networks derived using eSGA to predict the functions of genes lacking annotations5, 6, 9, 28-30 (Figure 4d). Since clustering algorithms vary, however, putative functional assignments determined through clustering require independent experimental verification.
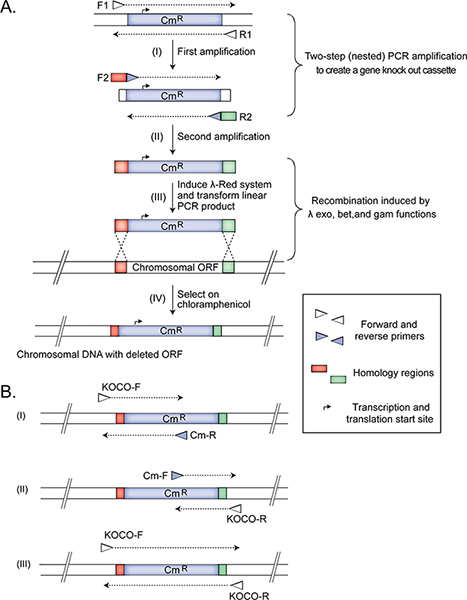
Figure 1. The construction and confirmation of Hfr Cavalli non-essential single gene deletion donor strains. The panels (adapted from14) illustrate the deletion of E. coli chromosomal ORF (A) and the three primer sets used for confirming (B) the correctly generated mutant Hfr Cavalli donor strain. Amplifications from a correctly constructed deletion donor strain should produce (i) 445 bp, (ii) 309 bp, and (iii) 1.4 kb products. See protocol text for details. Click here to view larger figure.

Figure 2. The construction and confirmation strategies of essential hypomorphic donor mutant strains in an Hfr Cavalli genetic background. The panels illustrate the creation of hypomorphic mutations of essential genes through recombineering technology (A) and the primer sets (B) used for the PCR confirmation of essential gene hypomorphic mutations. See protocol text for details.

Figure 3. Schematic summary of key eSGA steps. The Hfr donor (marked with chloramphenicol resistance, CmR) and recipient F- (marked with kanamycin resistance, KanR) mutant strains are grown in 384-colony density on LB-Cm and LB-Kan plates, respectively. The donor and the recipient strains are conjugated by pinning them over each other onto an LB plate, which is then incubated overnight at 32 °C. Consequently, the conjugants are pinned onto plates containing both Kan and Cm to select double mutants. The double mutant plates are digitally imaged and the growth fitness of the colony sizes is quantitatively scored to identify aggravating and alleviating interactions.

Figure 4. Representative computational analyses of genetic interaction (GI) scores for determining pathway-level functional relationships. (A) Panel I, Distribution of correlation coefficients between the GI profiles of gene pairs encoding proteins linked by protein-protein interactions (PPI) versus randomly drawn gene pairs. The p-value was computed using the two-sample Kolmogorov-Smirnov (KS) test; Panel II, Scatter plot of correlated genetic profiles from rich media (RM) for two transporters (mdtI, mdtJ) that form a heterodimeric complex required for spermidine excretion. (B) The hierarchical clustering of the GI sub-network adapted from Butland et al.9 highlights the functional connectivity between components of the previously known Isc and Suf pathways with similar GI patterns. Pink represents aggravating (negative S-score) interactions, green represents alleviating (positive S-score) interactions and black represents absence of GI. A predicted novel component of the SUF pathway, ydhD, displays GIs with the members of the Isc pathway. (C) Pathway cross-talk recorded among cell envelope processes that are significantly enriched for aggravating or alleviating GI according to the hypergeometric enrichment analysis27. (D) A GI sub-network predicts the role of two functionally unannotated genes, yceG and yebA, in peptidoglycan splitting based on their pattern of strong alleviating interactions with well-known cell division peptidoglycan hydrolases. The figures shown in panels A, C and D are adapted from Babu et al. (2011)12. Click here to view larger figure.
Discussion
Nous avons établi un protocole étape par étape pour l'utilisation de robotique de dépistage ESGA pour enquêter sur les fonctions des gènes bactériens à un niveau voie en interrogeant GI. Cette approche peut être utilisée pour étudier les gènes individuels ainsi que les systèmes biologiques entières dans E. coli. Attentivement l'exécution des étapes expérimentales décrites ci-dessus, y compris tous les contrôles appropriés, et rigoureusement l'analyse et la validation des données indépendamment GI sont des aspects essentiels pour le succès de ESGA à faire de nouvelles découvertes fonctionnels. En plus de ESGA, une méthode conceptuellement similaire pour l'étude de GI dans E. coli, appelé GIANT-coli 17, peuvent être utilisés pour éclairer de nouvelles relations fonctionnelles qui sont souvent oubliés par d'autres approches, telles que la protéomique 23 ou 13 écrans phénotypiques seuls. Néanmoins, comme pour toute démarche à l'échelle du génome, des limitations existent sur l'applicabilité de ESGA ou GIANT-coli, par exemple pour d'autres espèces bactériennes. Ceci est en partie êtrecause de l'indisponibilité de l'ensemble du génome simples collections de suppression de contrainte gènes mutants pour la plupart des espèces bactériennes, en particulier pour les espèces dont la mutagenèse ciblée est entravée par une faible fréquence naturelle de la recombinaison homologue. Dans de tels cas, les perturbations de gènes en utilisant des méthodes telles que la mutagenèse aléatoire traçables essais basés comme TnSeq 31 peut potentiellement être utilisée pour étudier les fonctions des gènes bactériens. Pour un aperçu des nouvelles procédures alternatives de dépistage génétique bactérienne, voir le document d'examen par Gagarinova et Emili (2012) 32.
Bien que les méthodes GI révèlent souvent de nombreux liens intéressants suggestive de nouveaux liens mécanistes, l'intégration de ces données avec d'autres informations telles que le profilage phénotypique 13, les réseaux d'interactions physiques 22, 23 et GC-inférées associations peuvent être particulièrement instructif. Par exemple, comme les réseaux, les associations de GI du GC, qui sont fondés sur la conservationde l'ordre des gènes (opérons), des fusions de gènes bactériens, les fréquences de recombinaison provenant de l'opéron distances intergéniques des opérons prédits dans les génomes, ainsi que le profilage phylogénétique 33 peut fournir des informations supplémentaires sur la manière dont une cellule bactérienne organise des complexes de protéines dans les voies fonctionnelles pour arbitrer et coordonner majeur processus cellulaires 12, 23.
E. coli est un bourreau de travail modèle pour la compréhension de la biologie moléculaire des autres bactéries Gram-négatives. À cette fin, les données ESGA IG peut être utilisée en conjonction avec l'information génomique comparative (profilage phylogénétique par exemple, le profil d'expression des gènes) d'enquêter sur la conservation évolutive des relations fonctionnelles découverts par ESGA dans d'autres espèces et des taxons protéobactéries procaryotes. En outre, les données d'interaction généré pour E. coli peuvent être utilisés pour mieux comprendre l'architecture voie de microbes découverts par metagenomicros, pour lequel les annotations fonctionnelles font défaut. Depuis de nombreux gènes sont largement conservé à travers les 23 microbes, et parce que la sensibilité aux antibiotiques peut être renforcée par certaines perturbations génétiques 34, les relations fonctionnelles éclairés par ESGA peut même potentiellement être exploitée pour concevoir des combinaisons thérapeutiques innovantes.
Déclarations de divulgation
Aucun conflit d'intérêt déclaré.
Remerciements
Ce travail a été soutenu par des fonds de Génome Canada, l'Ontario Genomics Institute, et les Instituts de recherche en santé subventions à JG et AE AG est récipiendaire de bourses d'études supérieures du Canada Vanier.
matériels
| Name | Company | Catalog Number | Comments |
| I. Antibiotics | |||
| Chloramphenicol | Bioshop | #CLR201 | |
| Kanamycin | #KAN201 | ||
| Ampicillin | # AMP201 | ||
| 2. Luria-Bertani medium | |||
| LB powder | Bioshop | #LBL405 | |
| Agar | Bioshop | #AGR003 | |
| 3. Bacterial Strains and Plasmids | |||
| Hfr Cavalli strain λred system (JL238) | Babu et al.14. | ||
| pKD3 | E. coli Genetic Stock Centre, Yale | ||
| Keio E. coli F- recipient collection | National BioResource Project (NBRP) of Japan11 | ||
| Hypomorphic E. coli F- SPA-tag strains | Open biosystems; Babu et al.14 | ||
| 4. Primers | |||
| pKD3-based desalted constant primers | F1: 5'-GGCTGACATGGGAATTAGC-3' R1: 5'-AGATTGCAGCATTACACGTCTT-3' | ||
| Desalted custom primers | Cm-R: 5'-TTATACGCAAGGCGACAAGG-3' Cm-F: 5'- GATCTTCCGTCACAGGTAGG-3' | ||
| Desalted custom primers | F2 and R2: 20 nt constant regions based on pKD3 sequence and 45 nt custom homology regions F2 constant region: 5'-CATATGAATATCCTCCTTA-3' R2 constant region: 5'-TGTGTAGGCTGGAGCTGCTTC-3'S1 and S2: 27 nt constant regions for priming the amplification of the SPA-Cm cassette and 45 nt custom homology regions S1 constant region: 5'AGCTGGAGGATCCATGGAAAAGAGAAG -3' S2 constant region: 5'- GGCCCCATATGAATATCCTCCTTAGTT -3' KOCO-F and KOCO-C: 20 nt primers 200 bp away from the non-essential gene deletion site or the essential gene SPA-tag insertion site | ||
| 5. PCR and Electrophoresis Reagents | |||
| Taq DNA polymerase | Fermentas | # EP0281 | |
| 10X PCR buffer | Fermentas | # EP0281 | |
| 10 mM dNTPs | Fermentas | # EP0281 | |
| 25 mM MgCl2 | Fermentas | # EP0281 | |
| Agarose | Bioshop | # AGA002 | |
| Loading dye | NEB | #B7021S | |
| Ethidium bromide | Bioshop | # ETB444 | |
| 10X TBE buffer | Bioshop | # ETB444.10 | |
| Tris Base | Bioshop | # TRS001 | |
| Boric acid | Sigma | # T1503-1KG | |
| 0.5 M EDTA (pH 8.0) | Sigma | # B6768-500G | |
| DNA ladder | NEB | #N3232L | |
| 6. DNA isolation and Clean-up Kits | |||
| Genomic DNA isolation and purification kit | Promega | #A1120 | |
| Plasmid Midi kit | Qiagen | # 12143 | |
| QIAquick PCR purification kit | Qiagen | #28104 | |
| 7. Equipment for PCR, Transformation and Replica-pinning | |||
| Thermal cycler | BioRad, iCycler | ||
| Agarose gel electrophoresis | BioRad | ||
| Electroporator | Bio-Rad GenePulser II | ||
| 0.2 cm electroporation cuvette | Bio-Rad | ||
| 42 °C water bath shaker | Innova 3100 | ||
| Beckman Coulter TJ-25 centrifuge | Beckman Coulter | ||
| 32 °C shaker | New Brunswick Scientific, USA | ||
| 32 °C plate incubator | Fisher Scientific | ||
| RoToR-HDA benchtop robot | Singer Instruments | ||
| 96, 384 and 1,536 pin density pads | Singer Instruments | ||
| 96 or 384 long pins | Singer Instruments | ||
| 8. Imaging Equipments | |||
| Camera stand | Kaiser | ||
| Digital camera, 10 megapixel | Any Vendor | ||
| Light boxes, Testrite 16" x 24" units | Testrite | ||
| 9. Pads or Plates Recycling | |||
| 10% bleach | Any Vendor | ||
| 70% ethanol | Any Vendor | ||
| Sterile distilled water | Any Vendor | ||
| Flow hood | Any Vendor | ||
| Ultraviolet lamp | Any Vendor | ||
| 10. Labware | |||
| 50 ml polypropylene tubes | Any Vendor | ||
| 1.5 ml micro-centrifuge tubes | Any Vendor | ||
| 250 ml conical flaks | VWR | # 29140-045 | |
| 15 ml sterile culture tubes | Thermo Scientific | # 366052 | |
| Cryogenic vials | VWR | # 479-3221 | |
| Rectangular Plates | Singer Instruments | ||
| 96-well and 384-well microtitre plates | Singer Instruments | Nunc | |
| Plate roller for sealing multi-well | Sigma | #R1275 | |
| plates | ABgene | # AB-0580 | |
| Adhesive plate seals | Fisher Scientific | # 13-990-14 | |
| -80 °C freezer | Any Vendor | ||
Références
- Bandyopadhyay, S., Kelley, R., Krogan, N. J., Ideker, T. Functional maps of protein complexes from quantitative genetic interaction data. PLoS Comput. Biol. 4, e1000065(2008).
- Costanzo, M., Baryshnikova, A., Myers, C. L., Andrews, B., Boone, C. Charting the genetic interaction map of a cell. Curr. Opin. Biotechnol. 22, 66-74 (2011).
- Costanzo, M., et al. The genetic landscape of a cell. Science. 327, 425-4231 (2010).
- Fiedler, D., et al. Functional organization of the S. cerevisiae phosphorylation network. Cell. 136, 952-963 (2009).
- Roguev, A., et al. Conservation and rewiring of functional modules revealed by an epistasis map in fission yeast. Science. 322, 405-4010 (2008).
- Schuldiner, M., et al. Exploration of the function and organization of the yeast early secretory pathway through an epistatic miniarray profile. Cell. 123, 507-519 (2005).
- Wilmes, G. M., et al. A genetic interaction map of RNA-processing factors reveals links between Sem1/Dss1-containing complexes and mRNA export and splicing. Mol. Cell. 32, 735-746 (2008).
- Babu, M., et al. Systems-level approaches for identifying and analyzing genetic interaction networks in Escherichia coli and extensions to other prokaryotes. Mol. Biosyst. 12, 1439-1455 (2009).
- Butland, G., et al. coli synthetic genetic array analysis. Nat. Methods. 5, 789-7895 (2008).
- Typas, A., et al. Regulation of peptidoglycan synthesis by outer-membrane proteins. Cell. 143, 1097-10109 (2010).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Mol. Syst. Biol. 2, 2006-200008 (2006).
- Babu, M., et al. Genetic interaction maps in Escherichia coli reveal functional crosstalk among cell envelope biogenesis pathways. PLoS Genet. 7, e1002377(2011).
- Nichols, R. J., et al. Phenotypic landscape of a bacterial cell. Cell. 144, 143-156 (2011).
- Babu, M., Gagarinova, A., Greenblatt, J., Emili, A. Array-based synthetic genetic screens to map bacterial pathways and functional networks in Escherichia coli. Methods Mol Biol. 765, 125-153 (2011).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc. Natl. Acad. Sci. U.S.A. 97, 6640-665 (2000).
- Yu, D., et al. An efficient recombination system for chromosome engineering in Escherichia coli. Proc. Natl. Acad. Sci. U.S.A. 97, 5978-5983 (2000).
- Typas, A., et al. quantitative analyses of genetic interactions in. E. coli. Nat. Methods. 5, 781-787 (2008).
- Zeghouf, M., et al. Sequential Peptide Affinity (SPA) system for the identification of mammalian and bacterial protein complexes. J. Proteome Res. 3, 463-468 (2004).
- Davierwala, A. P., et al. , 1147-1152 (2005).
- Breslow, D. K., et al. A comprehensive strategy enabling high-resolution functional analysis of the yeast genome. Nat. Methods. 5, 711-718 (2008).
- Babu, M., et al. Sequential peptide affinity purification system for the systematic isolation and identification of protein complexes from Escherichia coli. Methods Mol. Biol. 564, 373-400 (2009).
- Butland, G., et al. Interaction network containing conserved and essential protein complexes in Escherichia coli. Nature. 433, 531-537 (2005).
- Hu, P., Janga, S. C., Babu, M., Diaz-Mejia, J. J., Butland, G., et al. Global functional atlas of Escherichia coli encompassing previously uncharacterized proteins. PLoS Biol. 7, (2009).
- Anderson, R. P., Roth, J. R. Tandem genetic duplications in phage and bacteria. Annu. Rev. Microbiol. 31, 473-505 (1977).
- Boone, C., Bussey, H., Andrews, B. J. Exploring genetic interactions and networks with yeast. Nat. Rev. Genet. 8, 437-449 (2007).
- Collins, S. R., et al. Functional dissection of protein complexes involved in yeast chromosome biology using a genetic interaction map. Nature. 446, 806-8010 (2007).
- Le Meur, N., Gentleman, R. Modeling synthetic lethality. Genome Biol. 9, R135(2008).
- Tong, A. H., et al. Systematic genetic analysis with ordered arrays of yeast deletion mutants. Science. 294, 2364-2368 (2001).
- Tong, A. H., et al. Global mapping of the yeast genetic interaction network. Science. 303, 808-813 (2004).
- Wong, S. L., et al. Combining biological networks to predict genetic interactions. Proc. Natl. Acad. Sci. U.S.A. 101, 15682-15687 (2004).
- van Opijnen, T., Bodi, K. L., Camilli, A. Tn-seq: high-throughput parallel sequencing for fitness and genetic interaction studies in microorganisms. Nat. Methods. 6, 767-772 (2009).
- Gagarinova, A., Emili, A. Genome-scale genetic manipulation methods for exploring bacterial molecular biology. Mol. Biosyst. 8, 1626-1638 (2012).
- Dewey, C. N., et al. Positional orthology: putting genomic evolutionary relationships into context. Brief Bioinform. 12, 401-412 (2011).
- St Onge, R. P., et al. Systematic pathway analysis using high-resolution fitness profiling of combinatorial gene deletions. Nat. Genet. 39, 199-206 (2007).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon
Nous utilisons des cookies afin d'améliorer votre expérience sur notre site web.
En continuant à utiliser notre site ou en cliquant sur le bouton ''continuer'', vous acceptez l'utilisation de cookies.
