Method Article
細菌機能ネットワークとPathwaysでのマッピング
要約
体系的、大規模合成遺伝子(遺伝子遺伝子またはエピスタシス)対話画面が遺伝的冗長性と経路のクロストークを探索するために使用することができます。ここで、我々は上位性の関係を解明するとの遺伝的相互作用ネットワークを探索するために開発されeSGAと呼ばれるハイスループット定量合成遺伝子配列のスクリーニング技術を記述大腸菌。
要約
表現型は、物理(蛋白質-蛋白質など )と機能( 例えば遺伝子遺伝子または遺伝的)相互作用(GI)1一連の複雑によって決定されます。物理的な相互作用が細菌のタンパク質が複合体として関連付けられているかを示すことができますが、それらは必ずしも経路レベルの機能relationships1を明らかにしない。 2削除または不活性化された遺伝子をもつ二重変異株の増殖を測定し、対応する単一の変異体と比較されるGIの画面は、遺伝子座の間の上位性の依存性を照らすそれゆえ小説機能的関係2を照会し、発見するための手段を提供することができる。大規模GIマップは酵母3月7日のような真核生物のために報告したが、GIの情報は、細菌ゲノムの機能アノテーションを妨げる原核8日のための疎のままされています。この目的のために、我々と他の人は、ハイスループット定量的な細菌性胃腸スクリーニング方法(9)(10)を開発しましたここでは、定量的なEを実行するために必要な主要な手順を提示全身的に生成し、コロニー配列形式の二重変異体の多数の適合度を測定するために自然な細菌接合と相同組換えを用いて大腸菌ゲノムスケールの9の合成遺伝子アレイ(eSGA)スクリーニング手順は、 簡単に述べると、ロボットを転送するために使用され、共役を介して、クロラムフェニコール(Cm) - 著しいF-レシピエント株 - (組換えの高周波)エンジニアリングHFRから変異アレルをマーク 'ドナー株'カナマイシン(菅)の規則正しい配列にします。一般的に、我々はに機能喪失型の非必須遺伝子欠失をもつ単一突然変異( '慶應義塾'コレクション11 など )と必須遺伝子hypomorphic変異(還元タンパク質の発現、安定性、または活動9、12、13を付与する 、すなわち対立遺伝子)を使用非必須と必須遺伝子の機能アソシエーション、resを問い合わせるpectively。相同組換えによる共役とその後の遺伝子交換を媒介した後、得られた二重変異体は、両方の抗生物質を含有する固体培地上で選択されています。伸長した後、プレートをデジタル画像化されるとコロニーサイズを定量的に社内自動画像処理システム14を用いて採点されます。二重変異体の成長率はどちら有意に良好または9予想以上に悪化しているときにGISは明らかにされています。悪化(または負)GISは、多くの場合、同じ基本的なプロセス2に当たる代償経路からの遺伝子のペアにおける機能喪失型変異との間に生じる。ここでは、単一の遺伝子の損失は、単一の変異が生存可能であるように、バッファリングされます。しかし、両経路の損失は有害であると合成致死性または病気( すなわち低成長)が返されます。逆に、緩和(または正)の相互作用と同じ経路またはタンパク質複合体2の遺伝子の間に発生する可能性がありますいずれかの遺伝子の欠失だけでは、多くの場合、追加摂動はさらに、活性を低下させるため、成長しないような経路または複合体の正常な機能を混乱させるのに十分である。全体的に、体系的にGIのネットワークを識別し、分析することは、他の手法では見逃さ経路レベルの情報が9推測することができ、そこから多数の遺伝子間の機能的関係の公平、グローバルマップを提供することができます。
プロトコル
1。 15遺伝子改変技術によりHFRカヴァッリドナー変異株を構築し、16
eSGAドナー汚れを構築するための手順を以下に説明します。簡単に言えば、我々は対象とλを使う-赤は非必須遺伝子の欠失変異体(1.1項)またはHUPとして使用されている必須遺伝子hypomorphic変異ドナー株(セクション1.2)を作成し、PCRによって生成された増幅選択可能なDNAマーカーカセットの相同組換え16フラグメントを媒介GIのネットワークを定義するために 'クエリ'。
注:技術開発の過程で、我々は明確に定義されたHFRのドナー株(HFRカヴァッリ、HFRヘイズ、とHFR 3000)を使用して突然変異を結合するためのHFR媒介接合伝達を使用しての有効性を評価した。我々は、(i)効率的Yuらによって開拓コンビメソッドを使ってドナー突然変異体を作る能力を調べた。 (2000)16、(ii)の相対効率共役DNAの異なる染色体マーカーの転送、および(iii)クエリ遺伝子の位置とHFR転送軌跡、点の他に相対的な染色体の向きの効果。の我々は、λを発見- Red媒介相同組換えの効率がHFRヘイズまたはHfr3000よりHFRカヴァッリではるかに高かった。必要な場合は、P1形質導入または疑似HFR株17は 、変異体ドナーを作成するために使用できます。我々は、正常ドナーの多数を作ってスクリーニングするためのすべてのこれらの方法を適応している。我々の試験抱合実験で、転送と観察EX-conjugants数の全体的な効率はHFRカヴァッリと有意に高かったので、この特定の株の背景は、ドナーの建設や大規模eSGAに選ばれました。 HFRカヴァッリドナーを用いeSGA画面のすべての手順が記載されている。
- 非必須遺伝子を削除し、その後のコンビのためのDNA断片を増幅する
2段階のネストされたPCR増幅を採用15:;:プラスミドpKD3(AAL02033.1プロテインID 15554330 GI)からCM耐性マーカーを使用してターゲットのオープンリーディングフレームを交換するための遺伝子欠失カセットを作成するfication( 図1)。マーカーは実験をフォローアップにおける耐性遺伝子を除去することを、必要に応じて、できるように、FRT部位11に隣接している。
注:私達は、細菌細胞を形質転換するために続いて使用された増幅産物からプラスミドを除去するために2段階のPCRを入れ子ん。プラスミドを除去する(最初のPCRで)、次いで第2のPCRでの遺伝子ノックアウトカセットを作成すると、プラスミドによる形質転換の方がはるかに効率的であるため、コンビよりも必要であり、目標は、取得することです-できるだけ-持っている唯一の株正常に選択マーカーで標的ゲノムDNAを取り替えた。 nested PCR法に代わる第二のPCRで増幅された領域の外側にプラスミドの制限酵素消化を行うことですステップ。その後、制限酵素消化の産物を精製し、ネステッドPCRステップ(1.1.2)でのテンプレートとして使用されます。また、このオプションはシンプルですが、タイムアップより多くの作業が必要です。- 1070 bpの製品を生産するために前方にF1とR1の逆プライマーを用いたPCRの鋳型としてpKD3の約45 ngを使用します。キアゲンPCR精製プロトコールを用いたPCR断片を精製し、5 ng /μlに濃度になるように滅菌蒸留水で製品を希釈します。
- できるように、5 '末端において遺伝子特異的相同性領域の(i)は45 ntを持っている遺伝子特異的な( 例えばノックアウト)ネストしたプライマーは、F2とR2との二回目のPCRを設定し、テンプレートとして精製したPCR産物を用いて相同組換えのためのすぐ上流と下流のCMのマーカーの合成をプライミングするために3 '末端におけるCm耐性マーカー、及び(ii)20 NTとそれを置き換えるために、標的遺伝子の。生産フラグメントの大きさは1123 bpである。遺伝子特異的な相同性領域は、標的opを除去するように設計されて任意の隣接する、または部分的に重複コードセグメントをそのまま残しながら読み枠専用。この増幅した後、1.3に進みます。
- 必須遺伝子hypomorphic突然変異を作成するためのタグ付けカセットを増幅
- hypomorphicドナー変異株を構築するために、しばしばやや不安定な転写産物量( すなわちタンパク質のコピー数)またはタンパク質フォールディング/活動によって本質的なタンパク質の機能に影響を与え、C末端シーケンシャルペプチドアフィニティー(SPA)を融合タグを追加します。この目的を達成するために、pKD3 15 pJL148で菅マーカーに隣接する同一の塩基配列とCmマーカー間コンビを使用してCMにpJL148 18に菅耐性マーカーを変換することによって、選択可能なSPA-タグ(SPA-CM)DNAテンプレートを作成します。結果のテンプレートは、Cm耐性マーカーが続くインフレームSPA-タグで構成されており、PCR増幅、コンビと後続eSGA 12に適しています。
注:</ strong>は必須遺伝子が潜在的に遺伝子機能研究のための最も興味深いものです。これらの遺伝子を削除することはできませんので、しかし、革新的な方法は、必須遺伝子の機能的関係を調査するために開発されなければならなかった。いくつかの適切なアプローチは非常に成功した酵母で最初に開発されてきた。たとえば、Davierwalaら。 (2005)19 575温度感受性必須遺伝子変異体のパネルを使用して、GIの画面を実行しているため、本質的な遺伝子は非必須遺伝子としてGISの約5倍の数値を示したことが分かった。同様に、ブレスローら(2008)20(湿った)経路と複合体を研究するために不可欠な酵母遺伝子の約82%をカバーするhypomorphic対立遺伝子のライブラリーを作製するためのアプローチ"のmRNA摂動減豊富"を開発した。湿ったアプローチは、可能性の高い原因は、不安定化をmRNAに標的遺伝子6のレベルを減少させる、発現されるmRNAの3 '末端を変更します。 DAMPアプローチ6、私たちのstrategに似てyは必須タンパク質の発現されるmRNAの3 '末端を乱すの微妙な効果を利用してきました。
我々は以下に説明するように、それ自体でタグはおそらくタンパク質の折りたたみや機能には影響しませんが、環境ストレスや他の変異と組み合わせるとタグによるmRNAの微妙な摂動は、十分であるが、機能的に有益な遺伝子-環境13と遺伝子を明らかにする遺伝子9,12の相互作用。
まず、これまでに、我々は、本質的なタンパク質は私たちのSPA-タグ株8の間で表現されている307大腸菌の80%以上で、〜3000必須及び非必須大腸菌タンパク質21-23を精製するために、SPA-タグを使用していました。タグが正常に相互に検証された既知のタンパク質複合体を、(同じ錯体がタギングによって単離することができるIEと同じコンプレックス内の異なるタンパク質を精製)を単離するために使用されたので、我々は理由孤立した複合体は、生物学的に妥当であったことタグやる通常、タンパク質のフォールディングや機能12に影響を与えない。同様に、私たちの未発表の観察は、それ自体でタグは通常、歪みの成長に影響を及ぼさないことを明らかにした。しかし、我々は、湿った菌株6について報告されたように、タグとマーカーカセットを統合することによって、3'-UTRを変更することにより、RNAレベルで特定の転写産物が不安定になったり、まれにの適切な折りたたみ又は機能を妨げる可能性が理路融合タンパク質。したがって、我々はそのような他の場所でゲノム中の他の機能的に関連する突然変異に不可欠な遺伝子の対立遺伝子を摂動組み合わせると実に9、12が観察された有益なGIS、になるだろうと予測した。さらに、必須遺伝子のSPAのタグ付き対立遺伝子はニコルズらの表現型プロファイリング作業に応じて( 例えば、薬物過敏症)遺伝子と環境の相互作用を示した。成長の様々な条件にSPAのタグ付き必須の株の一部を受ける(2011)13。同じ研究confirmeDそのSPA-タグ(58テスト済みの54のうち)最も重要な株は、SPA-タグはゲノムワイドに有用である必須遺伝子の軽度hypomorphic対立遺伝子を頻繁に作成するという考えを支持する1つ以上の培養条件13で損なわ適性を示したeSGA含むGIのスクリーンアプリケーション。 - 相同(ⅱ)に続いて、5 '末端に遺伝子改変のためにS1とS2の遺伝子特異的プライマーは、それぞれの含有(i)は45 ntの遺伝子特異的な領域を使用してSPA-CMテンプレートからタギングカセットを増幅27 bpのSPA- 3 '末端に特異的な配列センチ。
- hypomorphicドナー変異株を構築するために、しばしばやや不安定な転写産物量( すなわちタンパク質のコピー数)またはタンパク質フォールディング/活動によって本質的なタンパク質の機能に影響を与え、C末端シーケンシャルペプチドアフィニティー(SPA)を融合タグを追加します。この目的を達成するために、pKD3 15 pJL148で菅マーカーに隣接する同一の塩基配列とCmマーカー間コンビを使用してCMにpJL148 18に菅耐性マーカーを変換することによって、選択可能なSPA-タグ(SPA-CM)DNAテンプレートを作成します。結果のテンプレートは、Cm耐性マーカーが続くインフレームSPA-タグで構成されており、PCR増幅、コンビと後続eSGA 12に適しています。
- 確認し、PCR産物を精製
- タギングカセットが正しくアガロースゲル電気泳動にPCR産物の2から5μlを付すことによって増幅されていることを確認し、製造者の標準的なPCR精製プロトコール(キアゲン)に続く残りのDNAを精製する。滅菌蒸留水を30μlでPCR産物を溶出し、〜50 ng /μlに濃度に希釈します。 purifiedの製品は変換ですぐに使用し(ステップ1.5)または6ヶ月まで、使用するまで-20℃で保存することができます。
- ドナーの建設のためHFRカヴァッリコンピテントセルを調製し
- 250mlのフラスコに50μg/ mlアンピシリン35μlの持つ新鮮なルリア - ベルターニ(LB)培地70ml中にHFRカヴァッリ株の飽和一晩培養液1ミリリットルを接種する。 (〜2時間)が得られる光学密度(OD)まで、220 rpmで〜0.5から0.6の600nmを振とうしながら32℃で培養をインキュベートする。
- 42でのλ 赤組換え系16°160 rpmで振盪しながら15分間Cの熱を誘導するための水浴に文化を転送します。 160rpmで10〜20分間冷やした氷スラリーの水浴に文化を転送することによって誘導を停止します。使用前に使用するチューブと、少なくとも15分間氷上で冷却されたキュベットを、変換後まで、この点から細胞を冷たくしておくようにしてください。
- cを分ける均等に2予冷50mlポリプロピレンチューブ(チューブあたり〜35 mlの培養液)と4℃で6分間4400 xgで遠心℃にulture
- 氷冷滅菌蒸留水〜50mlに穏やかに転倒することにより、細胞ペレットを上清を廃棄し、再懸濁する。 4℃で6分間4400 xgで再び細胞を遠心上清を捨てて、もう一度、上記のように氷冷滅菌10%グリセロールおよび遠心20mlに再懸濁させ、各細胞ペレット。
- 上清を除去し、氷冷した10%グリセロール500μlの細胞ペレットを再懸濁する。即時の形質転換のための個々の、予冷1.5mlマイクロ遠心チューブに50μlの体積でアリコート調製したコンピテントセルを。最大3ヶ月までのための細胞はドライアイスや液体窒素で凍結させ、-80℃で保存することができる。しかし、形質転換効率は、新たに調製したコンピテント細胞と最も高くなっている。
- 変態
- からPCR産物を精製した100 ngの追加コンピテントセルへのステップ1.3.1。チューブをフリックし、懸濁液を氷上で5分間座ってすることができます。
- 予め冷却エレクトロポレーション用キュベットに氷のように冷たいテント細胞とDNAの懸濁液を移す。 2ミリメートルギャップキュベットに設定2.5kVで、25μF、200オームと細胞混合物をエレクトロポ( すなわち異なるキュベットを使用している場合は、エレクトロポレーションの設定については、製造元の指示を参照してください)、すぐに室温SOC培地1mlを加える。軌道を220 rpmで振盪しながら1時間32℃で15 mlの培養液管とインキュベートにSOC培地でエレクトロポレーションした細胞を転送します。
- インキュベーション後、室温で5分間4400 xgで細胞を遠心分離します。清の約850μlを外し、残りの液に細胞ペレットを再懸濁する。
- 32℃で一晩17μg/ mlのCmとインキュベートを含むあらかじめ温めておいたLBプレート上で細胞を広げた。 LBの上に2つの個々の形質転換体のコロニーと連勝を選び出す-CM変異確認のためのプレート。我々は、珍しいサプレッサー変異と系統をピッキングの可能性を低減するために、最大の形質転換体のコロニーを避けることをお勧め。また、連勝はLB館で同じ形質転換体は、株が菅耐性でないことを確認します。
注:Cm耐性マーカーがドナー株を作るために使用されているので、我々はドナー変異体は菅耐性があることを期待しないでください。 LB-菅とLB-CMプレート両方に形質転換体をストリーキングすることによって、我々はドナー変異自体は何とか菅抵抗を引き起こさなかったことを確認してください。このステップを実行すると、ターゲット変異が菅に生き残るためには株の能力に影響を及ぼすことがあっていなかった場合、二重変異体は、ドナー株はすべての選択手順を生き残るになるので効果的にeSGAを使用して行われないであろう。これは、単に予想外の表現型またはeSGA画面の完了を妨げる可能性のある他の問題をキャッチするための予防の論理的なステップである。このステップはスキップされた場合、他のコントロールを上げ得るそれでも失敗した画面を識別すると思います。しかし、菅に耐性のあるドナーの早期検出は非生産的と不必要な一連の手順を排除するでしょう。菅に成長する株の他の考えられる理由には、例えば、可能性が間違ったPCR産物または変換のための株の使用だけでなく、抗生物質の有効期限が切れました。すべてのこれらの問題は必要ですが、eSGAにはお勧めできません、このコントロールの助けと手続きの早い段階で識別し、対処することができます。
- 非必須遺伝子欠失(ステップ1.6.1)または必須遺伝子hypomorphic突然変異(セクション1.6.2)を確認する
注:PCRによる遺伝子欠失を確認すると、野生型株からゲノムDNAを対照として使用する必要があります。- 非必須遺伝子の欠失を確認
- PCRにより遺伝子欠失を確認するには、manufactに続いて、液体LB-CM培地で増殖させ、変換、推定上のノックアウト株からゲノムDNAを単離urerの指示(Promega社)。
- 成功したコンビを確認するには、ノックアウト確認プライマーの3つの異なるセットを使用して、各形質転換体からDNAを増幅する。ゲノムDNAテンプレートの〜150 ngの反応を行い、アガロースゲルでPCR-合成製品を実行して、正しい断片のサイズを確認してください。
- 第1のプライマーセットは、CMカセット配列に相補的な20-NT隣接標的領域(KOCO-F)の200 bp上流に位置するプライマー、及びCm-Rプライマー、で構成されています。この増幅は変異体ではなく、野生型株( 図1)445 ntのアンプリコンを生成することが期待されています。
- 第二セットは、3 '末端の200bpの下流にアニールするように設計されているCMのカセット配列にアニーリングフォワードプライマー(CM-F)、および確認プライマーに隣接する20-ntのリバース(KOCO-R)を、含まれています削除された遺伝子。この増幅反応は309 ntの変異体でアンプリコンが、それを生成しないことが期待される野生型株のt( 図1)。
- 第三PCRはKOCO KOCO-Fおよび-Rプライマーを含んでいます。この反応は、一つの遺伝子座がカセットで置き換えられ、別の重複しますが、それ以外の野生型遺伝子のコピーがまだ存在してきたことで選択された菌株は、merodiploidでないことを確認するために必要です。正しい欠失変異体からこの増幅は1.4kbの生成物( 図1)を生成するために期待されています。
注:ターゲット遺伝子が遺伝子欠失カセットとして塩基対でほぼ同じ長さである場合、1はカセットに置き換えられている遺伝子座ではなく、持って座の間の違いに気付くことはありません。そのような場合には決定的な確認のために、我々は、明示的に削除遺伝子内のDNA断片を増幅するための追加のプライマーを使用することをお勧めします。遺伝子が削除されている場合この場合には、増幅産物は、野生型コントロール株ではなく、突然変異体で観察されます。これらのプライマー手動で、または、計算明示的に削除領域内のDNA断片を増幅するように設計することができる。我々は一般的に140 bpの領域を増幅する。
- 必須遺伝子hypomorphic変異を確認
- PCRによるhypomorphic変異を確認するために、遺伝子特異的な20-NTフォワード(KOCO-F)およびSPA-タグの挿入部位のそれぞれ200 bp上流と下流に位置するリバースプライマー(KOCO-R)は、( 図2)を使用します 。
注:この増幅工程がターゲット必須遺伝子の非タグ付けをされたコピーが存在しないことを確実にするためにコントロールとして使用される。タグ付けされたバージョンのみが存在する場合、SPA-CMアンプリコンより大きい単一のバンド310 bpのが観察される。 400bpの生成物は、遺伝子のタグなしバージョンの存在を示すであろう。 - PCRの確認と並行して、SPのFLAGエピトープに特異的な抗FLAG M2抗体を用いたウエスタンはSPA-tagのインフレーム挿入を確認するブロッティング使用タグ21。
- PCRによるhypomorphic変異を確認するために、遺伝子特異的な20-NTフォワード(KOCO-F)およびSPA-タグの挿入部位のそれぞれ200 bp上流と下流に位置するリバースプライマー(KOCO-R)は、( 図2)を使用します 。
- 非必須遺伝子の欠失を確認
- 確認されたドナー株を保存する
- 、標識されたクライオバイアルに確認換えドナー変異株の一晩培養物を移し15%グリセロールを最終濃度を補完し、よく混ぜる。長期保存には、-80℃で凍結バイアルを保つ℃に
2。 Eを並べるeSGAスクリーン用の大腸菌のF-受取変異体
- レプリカピンE.森らによって生成された大腸菌慶應義塾単一の非必須遺伝子の欠失変異体コレクション。ウェル当たり液 体LB培地の80μlを含む二〇から四384ウェルマイクロプレートにロボット(3968株11;、F-BW25113は、Open Biosystems社から入手できます) 、50μg/ mlのカンザスで補充
注:系統的に不可欠な遺伝子とGISを評価するために、受信者慶應義塾コレクション11は 、F-菅マーキング必須遺伝子わい小体型を補充することができますC末端のSPA-22タグ付きIC変異株、23。 - ポジティブコントロールとして機能する国境管理菌株のためのスペースを確保するためだけでなく、自動化されたコロニーの量子化のように、内とプレートの正規の間での援助は、上記のステップとへの転送から各プレートの最も外側の井戸から接種メディアを削除新しいプレートは、再び最も外側のウェルが空のまま。
注:慶應義塾コレクション11から国境管理菌株JW5028が、私たちの全ゲノムの画面で、任意のドナーとGIを発揮していないべきではない小さな偽遺伝子のために削除されます。したがって、これは、接合、固定、または選択が失敗したときに存在しないか、または散発国境株の成長で、その結果、まれに識別するための優れたポジティブコントロール株である。国境管理のコロニーが二重変異体選択後に成長しない場合などに、クエリー株が共役することが可能ではないかもしれないが、結合は、iを試みられている可能性があります場所(抗生物質含有プレート上など )、または間違った抗生物質を服用することを妨げるNA条件が選択プレートに追加されている可能性があります。非常に少数の、散発コロニーが境界線を含む全プレート、上認められた場合には同様に、接合またはpinningは失敗している可能性があり、画面が繰り返される必要があります。この散発的な国境の成長は再現性があるなら、それは一貫して貧しいドナー共役を示すことがありますので、二重変異体が観察されていないインスタンスはおそらく抱合障害ではなく、真の兵によるものである。このような画面がさらなる分析から破棄される必要があるでしょう。さらに、すべてのプレート上に存在していることにより、国境管理株はコロニー量子化ソフトウェアが自動的に二重変異体プレートとこうして植民地の位置は手動で区画されている必要はありません上のコロニーの位置を決定することができます。最後に、国境管理ひずみ内のコロニーの大きさの正規化における助剤( 例えば、プレート内の異なる行)とbetwEEN( 例えば、異なった受取人のプレートが異なる時間に同じドナーで固定)プレート。 - 同様に、各プレート内の別の場所(ない境界)から、任意の2つの系統を除去することによってネガティブコントロールスポットを作成し、新しいプレートに株を譲渡する。 Cmの17μg/ mlを含むLB培地で、これらの空の井戸を埋める。これらのネガティブコントロールスポットが受取人、したがって二重変異体プレートで空になり、イメージング、ナンバリング、またはプレートを固定するときに処理やプレートハンドリングエラーがなかったことを確認するために期待されています。
注意:これらの空のスポットがネガティブコントロールを要求される。陰性対照株は選択中に育つとき-ネガティブコントロールは選択が失敗したときにインスタンスを識別するのに役立ちます。私たちは、レシピエントプレートの各々のためのユニークなネガティブコントロールスポットを選択するだけでなく、プレートの同じ象限内のネガティブコントロールスポットを(右下など)を選択することをお勧めします。このように、パターン陰性コントロールスポットの可能プレート取り扱いミスエラー( 例えばプレートはラベルが間違っているか、または左上コロニーは右下の位置に固定されている場合には、上下逆さまに固定されている)を識別する。 - 私たちの全ゲノムeSGA画面に基づいて菅の50μg/ mlを含む200mlのLBと500ミリリットルのフラスコに、偽遺伝子欠失、慶應義塾株JW5028 11を接種し、この特定の変異体の成長は野生に最も近いタイプBW25113そしてそれは、このように国境をコントロールとして使用されています。
- 600nmで0.6から32で一晩アレイ菌株ならびにJW5028文化℃〜190回転軌道で〜0.4のODに揺れを栽培しています。一晩増殖させた後、JW5028一晩培養した培養液の約80μlの国境井戸を埋める。
- 組み立てられたプレートは、ステップ3.2で使用することができる。あるいは、長期保存のために、15%グリセロールを持つ受信プレートの各ウェルを補完するメディアとグリセリンを混ぜて、プレートを保つ-80℃
3。 Eを生成するための高密度ひずみ嵌合手順コリ二重変異体
- 32℃で一晩CM、°CリッチLB-CM液体培地(CMの17μg/ ml)の中で、220rpmで振盪しながらでマーククエリー変異を、ベアリング、HFRのドナー株を栽培しています。
- 並行してカンザスの50μg/ mlのを補った固体LBプレート上に384コロニー密度で並べ受信者変異コレクションをピン、LBプレートと同じ数の上に、384コロニー密度のドナークエリ変異株をピン、17μgのを補ったCmの/ mlである。 32℃で一晩、プレートをインキュベート
注:ピン止めするため使用される受信者板は、後続のステップのために十分に大きいコロニーを得るための36時間を必要とすることがあります。平均ボーダーコロニーの大きさは直径約2mm(またはそれ以上)の場合、プレートが固定される準備ができている。 - 株を結合するために、上の384コロニー一夜ドナープレートからドナーをピン固体LBプレートに。その後接合プレート上たて固定ドナー上の単一の384コロニーの受取プレートをピン。すべてのドナーレシピエントプレートが固体LB抱合プレート上に固定することによって共役されるまでピン止め続ける。 16から24時間32℃で固定された接合プレートをインキュベートする。
注:個々の単一突然変異ドナー系統が原因で共役で突然変異、DNA導入効率、または適合性の潜在的な影響の接合効率で遺伝子対遺伝子変動を示すことがあります。成長は16と十分に大きいコロニーは、後続のステップのために固定が得られている24時間の間にいつでも停止することができます。 16から24時間持続する動詞は若干低い適性を持つ後期転送遺伝子または変異体の変異体のためにも再現性のある結果を得るためにeSGA EX-conjugants十分な数を生成します。 - 、単一の固体LBプレートに各384密度共役プレートをピン含むCM(17μg/ ml)を、菅(50μg/ ml)のすべてのコンジュゲーションプレートが固定されるまで。 32℃で16から36時間、たて固定第一選択プレートをインキュベート
注:選択ステップに関しては、異なる時間、異なるドナーの画面に必要であるかもしれません。指定された画面ですべての二重変異体は同じドナーの変異を持っているので、平均的には、遅い成長ドナー変異体は、より遅い成長二重変異体を生じさせる。したがって、ドナーの適性に応じて、選択ステップは、16〜36時間の間に必要となる場合があります。二重変異体は、後の工程は、2番目の後の最初の選択またはイメージング後に固定するために十分に大きい場合、二重変異体の成長を停止させることができる。これは通常、少なくとも2ミリメートル直径の平均で国境コントロールのコロニーに変換されます。 - それぞれの第一選択のコロニーは上の4つのコロニーで表されるように、第二、二重薬(CMおよびKan)、1536 - コロニー形式の選択プレート上にそれぞれの最初の選択プレートを再度ピン第二の選択プレート。 32℃で16から36時間、プレートをインキュベート定量的変異体の成長適性を測定するために、遺伝子のペア( 図3)との間の相互作用を分析するために最後の二重変異体選択プレートを撮影。
注:merodiploidy(部分染色体重複)の周波数は約1/1、000のセル24で低いですが、merodiploidyがGIをマスクすることができます。したがって、私たちは、生物すべてeSGA画面で複製を含めることをお勧め。幸いなことに、商業使い捨ての皿やプラスチック製のピン止めパッドは次のように材料を滅菌することにより3回まで再利用することができます:プレートから培地を廃棄し、プレートだけでなく、パッドは一晩10%漂白剤でそれらを浸漬することによって滅菌され、続いて70%エタノールで洗浄し、紫外線下流フード内のプラスチック容器を空気乾燥、蒸留水ですすいでください。滅菌パッドとプレートは使用するまで密閉された無菌のビニール袋に格納されています。
4。データの処理とGIスコアを導出
- バッチ·モードでの各プレート上のコロニーを測定したり、個々に特化したコロニーのイメージングソフトウェア3,9を使用しています 。
注:適切なJavaベースのハイスループットコロニーイメージングと得点アプリケーションはパブリック·アクセス(自由に利用可能ですhttp://srcollins77.users.sourceforge.net/ )。ソフトウェアは、異なるプラットフォーム(MacまたはPC)上で動作し、実行可能ファイルとして、またはターミナルウィンドウのいずれかから起動することができます。 - このようなプレートのエッジ効果、プレート間の変動の影響、画像ムラの照明、寒天表面の物理的な曲率によるアーティファクト、近隣コロニーのための競争の効果と可能性ピンニング欠陥などのプレート内との間に系統的にバイアスを補正することで生のコロニーの大きさの測定値を正規化するだけでなく、成長時間の違い P> 9。これらの体系的なアーティファクトは二重変異フィットネス独立しており、彼らが偽り成長フィットネス見積もりを生じさせるように修正する必要があります。
- エッジの行と列にコロニーがプレートの中央に見られるよりも、栄養素の低い競争のためにサイズが大きくなる傾向があります。したがって、この効果を補正するために、エッジのコロニーの大きさは、その行または列内のメジアン径がプレートの中央にあるコロニーのメジアン径に等しくなるようにする拡張。
- 考慮複写コロニー測定の中央値サイズからの再現性および標準偏差を取る統計的に結果を分析します。少なくとも3つの独立した生物学的反復実験は、実験の再現性を評価するために必要である。
- 最後に、以下の式を用いて各遺伝子対のGIスコア(S)を生成するために正規化された中央値コロニーの増殖フィットネス·サイズを使用する:
nが1の "src =" / files/ftp_upload/4056/4056eq1.jpg "/>
ここで、S = VAR(VAR経験のx(n Exp に -1)+ VAR 写 X(N 連写 -1))/(n個の経験 + N 写 -2); varのExpの正規化されたコロニーの大きさの最大値=分散二重変異体、参照セットから正規化された二重変異コロニーサイズの差異のvar CONT =中央値であり、n Exp に二重変異コロニーサイズの測定=数であり、n CONT =実験の数の中央値は、すべての実験を介して複製します。μの経験 =中央値正規化された二重変異体のコロニーサイズ;とμCONT =単一ドナー変異株に起因する全ての二重変異体のために正規化されたコロニーの大きさの中央値。 S-スコアは、推定二遺伝子間相互作用の統計的信頼性だけでなく、相互作用の生物学的な強さの両方を反映している。強い陽性、S-スコアは軽減効果は、sを示すS-スコアに有意な負のは、多くの場合、並列冗長経路9のメンバーシップを示唆している合成病気や致死性を反映しながら、相互作用の遺伝子が同じ経路9に参加することuggesting。経路レベルの機能的関係を決定するために、試験した遺伝子のペアごとに単一のS-スコアは最終データセットから作られています。
注:我々の以前の研究9では、我々は組換えが互いの30 kbpの範囲内の遺伝子間でそれほど頻繁に発生する傾向があることがわかった。したがって、下流の解析のために、正規化およびスコア生成後に、私たちはお互いの30 kbpの範囲内の遺伝子間の相互作用を除去します。兵自身に加えて、遺伝子の組の間の機能的関係は二つの遺伝子のGIプロファイルがどの程度似ているかを見て調べることができます。遺伝子のGIプロファイルは外のゲノム内のすべての他の遺伝子と、その遺伝子のすべての相互作用の集合である遺伝子の連鎖エリア。機能的に同様の遺伝子は遺伝的相互作用プロファイル12、25の高い相関を持つ傾向にあります。このように、実験的なデータセット内のすべての遺伝子の相関係数を計算することにより、1つでも染色体上に互いに近接して横たわっている遺伝子間の機能的関係を調査するためeSGAを使用することができます。
結果
GIs reveal functional relationships between genes. Similarly, since genes in the same pathway display similar GI patterns and the GI profile similarity represents the congruency of phenotypes, we can group functionally related genes into pathways by clustering their GI profiles. Integrating GI and GI correlation networks with physical interaction information or other association data, such as genomic context (GC) relationships can also reveal the organization of higher-order functional modules that define core biological systems (and system crosstalk) in bacteria. For example, as with yeast4, 6, 7, 26, E. coli gene pairs exhibiting alleviating interactions that also have highly correlated GI profiles tend to encode proteins that are either physically associated (e.g. form a complex; Figure 4a) or that act coherently in a common biochemical pathway (e.g. components in a linear cascade). A representative example is shown in Figure 4b, where the components of the functionally redundant Isc and Suf pathways, which jointly participate in the essential Fe-S biosynthesis process, form distinct clusters that are linked together by extensive aggravating interactions (i.e. synthetic lethality). A statistical measure (e.g. hypergeometric distribution function27) can be used on GI or GI correlation data to find significant enrichment for interactions between and within pathways (Figure 4c). Cluster analysis can also be applied to functional networks derived using eSGA to predict the functions of genes lacking annotations5, 6, 9, 28-30 (Figure 4d). Since clustering algorithms vary, however, putative functional assignments determined through clustering require independent experimental verification.
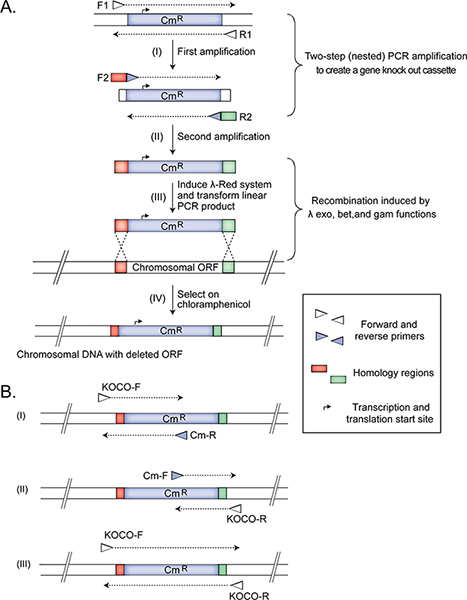
Figure 1. The construction and confirmation of Hfr Cavalli non-essential single gene deletion donor strains. The panels (adapted from14) illustrate the deletion of E. coli chromosomal ORF (A) and the three primer sets used for confirming (B) the correctly generated mutant Hfr Cavalli donor strain. Amplifications from a correctly constructed deletion donor strain should produce (i) 445 bp, (ii) 309 bp, and (iii) 1.4 kb products. See protocol text for details. Click here to view larger figure.

Figure 2. The construction and confirmation strategies of essential hypomorphic donor mutant strains in an Hfr Cavalli genetic background. The panels illustrate the creation of hypomorphic mutations of essential genes through recombineering technology (A) and the primer sets (B) used for the PCR confirmation of essential gene hypomorphic mutations. See protocol text for details.

Figure 3. Schematic summary of key eSGA steps. The Hfr donor (marked with chloramphenicol resistance, CmR) and recipient F- (marked with kanamycin resistance, KanR) mutant strains are grown in 384-colony density on LB-Cm and LB-Kan plates, respectively. The donor and the recipient strains are conjugated by pinning them over each other onto an LB plate, which is then incubated overnight at 32 °C. Consequently, the conjugants are pinned onto plates containing both Kan and Cm to select double mutants. The double mutant plates are digitally imaged and the growth fitness of the colony sizes is quantitatively scored to identify aggravating and alleviating interactions.

Figure 4. Representative computational analyses of genetic interaction (GI) scores for determining pathway-level functional relationships. (A) Panel I, Distribution of correlation coefficients between the GI profiles of gene pairs encoding proteins linked by protein-protein interactions (PPI) versus randomly drawn gene pairs. The p-value was computed using the two-sample Kolmogorov-Smirnov (KS) test; Panel II, Scatter plot of correlated genetic profiles from rich media (RM) for two transporters (mdtI, mdtJ) that form a heterodimeric complex required for spermidine excretion. (B) The hierarchical clustering of the GI sub-network adapted from Butland et al.9 highlights the functional connectivity between components of the previously known Isc and Suf pathways with similar GI patterns. Pink represents aggravating (negative S-score) interactions, green represents alleviating (positive S-score) interactions and black represents absence of GI. A predicted novel component of the SUF pathway, ydhD, displays GIs with the members of the Isc pathway. (C) Pathway cross-talk recorded among cell envelope processes that are significantly enriched for aggravating or alleviating GI according to the hypergeometric enrichment analysis27. (D) A GI sub-network predicts the role of two functionally unannotated genes, yceG and yebA, in peptidoglycan splitting based on their pattern of strong alleviating interactions with well-known cell division peptidoglycan hydrolases. The figures shown in panels A, C and D are adapted from Babu et al. (2011)12. Click here to view larger figure.
ディスカッション
我々は、GIを問い合わせることによって経路レベルで細菌の遺伝子の機能を調べるためにロボットeSGAスクリーニングを使用するための段階的なプロトコルについて概説しました。このアプローチは、 大腸菌内の個々の遺伝子だけでなく、全体のシステム生物学を研究するために使用することができます大腸菌 。慎重に実験手順を実行すると、すべての適切なコントロールを含め、上記のように、そして厳格に分析し、独立して、GIのデータが新しい機能の発見を行う際にeSGAの成功のための重要な側面で検証する。 eSGA、 大腸菌にGIを研究するための概念的に類似した方法に加えて、 大腸菌は 、巨大大腸菌17と称され、しばしばこのようなプロテオーム23または単独で表現型の13スクリーンなど他のアプローチでは見逃される新規機能的な関係を照らすために使用することができます。それにもかかわらず、任意のゲノムワイドなアプローチと同様に、制限は、例えば、他の細菌種に、eSGAまたは巨大大腸菌の適用に存在しない。これは部分的であることです特に標的突然変異誘発は、相同組換えの低い固有振動数によって妨げられている種については、ほとんどの細菌種のゲノムワイドな単一遺伝子欠失変異株のコレクションの使用不能の原因となります。このような場合には、そのようなTnSeq 31のようなランダムな突然変異誘発追跡ベースアッセイなどの方法を用いて遺伝子破壊は、潜在的に細菌の遺伝子の機能を調べるために使用することができる。新興の代替細菌遺伝子スクリーニング法の概要については、Gagarinovaとエミリ(2012)32でレビュー論文を参照してください。
GIの方法は、多くの場合、新規機序のリンクを示唆する多くの興味深い接続を明らかにするような表現型プロファイリング13、物理的な相互作用ネットワーク22のような他の情報と、これらのデータの統合、23、およびGC-推論団体は特に有益なことができます。ものの例えば、保全に基づいており、GCの団体、GIのネットワークと同様に遺伝子順序(オペロン)、細菌遺伝子融合により、遺伝子間のゲノム全体で予測オペロンの距離だけでなく、系統学的プロファイリング33由来オペロンの組換え頻度は、メジャーを仲介し、調整するために、細菌細胞が機能的経路へのタンパク質複合体を編成する方法への追加的な洞察を提供することができます細胞プロセス12、23。
大腸菌は他のグラム陰性菌の分子生物学を理解するための主力モデルである。この目的を達成するために、eSGA GIのデータは、他のプロテオバクテリア種を越えてeSGAによって発見された機能的関係および原核生物の分類群の進化的保存性を調査するために、比較ゲノム情報( 例えば、系統学的プロファイリング、遺伝子発現プロファイリング)と組み合わせて使用することができます。また、相互作用のデータは、E.のために生成大腸菌は metagenoによって発見された微生物の経路アーキテクチャへの洞察を得るために用いることができるマイクは、そのための機能注釈が不足している。多くの遺伝子が広くすべての微生物が23を越えて保存されているので、抗生物質感受性が特定の遺伝的摂動34によって向上させることができるので、eSGAによって照らさ機能的な関係であっても潜在的に革新的な組み合わせの薬物療法を設計するために悪用される可能性があります。
開示事項
特別な利害関係は宣言されません。
謝辞
この作品は、ゲノムカナダ、オンタリオ州ゲノミクス研究所、保健研究助成金のカナダの協会からJGとAE社への資金によってサポートされていましたヴァニエ·カナダ大学院奨学金の受賞者です。
資料
| Name | Company | Catalog Number | Comments |
| I. Antibiotics | |||
| Chloramphenicol | Bioshop | #CLR201 | |
| Kanamycin | #KAN201 | ||
| Ampicillin | # AMP201 | ||
| 2. Luria-Bertani medium | |||
| LB powder | Bioshop | #LBL405 | |
| Agar | Bioshop | #AGR003 | |
| 3. Bacterial Strains and Plasmids | |||
| Hfr Cavalli strain λred system (JL238) | Babu et al.14. | ||
| pKD3 | E. coli Genetic Stock Centre, Yale | ||
| Keio E. coli F- recipient collection | National BioResource Project (NBRP) of Japan11 | ||
| Hypomorphic E. coli F- SPA-tag strains | Open biosystems; Babu et al.14 | ||
| 4. Primers | |||
| pKD3-based desalted constant primers | F1: 5'-GGCTGACATGGGAATTAGC-3' R1: 5'-AGATTGCAGCATTACACGTCTT-3' | ||
| Desalted custom primers | Cm-R: 5'-TTATACGCAAGGCGACAAGG-3' Cm-F: 5'- GATCTTCCGTCACAGGTAGG-3' | ||
| Desalted custom primers | F2 and R2: 20 nt constant regions based on pKD3 sequence and 45 nt custom homology regions F2 constant region: 5'-CATATGAATATCCTCCTTA-3' R2 constant region: 5'-TGTGTAGGCTGGAGCTGCTTC-3'S1 and S2: 27 nt constant regions for priming the amplification of the SPA-Cm cassette and 45 nt custom homology regions S1 constant region: 5'AGCTGGAGGATCCATGGAAAAGAGAAG -3' S2 constant region: 5'- GGCCCCATATGAATATCCTCCTTAGTT -3' KOCO-F and KOCO-C: 20 nt primers 200 bp away from the non-essential gene deletion site or the essential gene SPA-tag insertion site | ||
| 5. PCR and Electrophoresis Reagents | |||
| Taq DNA polymerase | Fermentas | # EP0281 | |
| 10X PCR buffer | Fermentas | # EP0281 | |
| 10 mM dNTPs | Fermentas | # EP0281 | |
| 25 mM MgCl2 | Fermentas | # EP0281 | |
| Agarose | Bioshop | # AGA002 | |
| Loading dye | NEB | #B7021S | |
| Ethidium bromide | Bioshop | # ETB444 | |
| 10X TBE buffer | Bioshop | # ETB444.10 | |
| Tris Base | Bioshop | # TRS001 | |
| Boric acid | Sigma | # T1503-1KG | |
| 0.5 M EDTA (pH 8.0) | Sigma | # B6768-500G | |
| DNA ladder | NEB | #N3232L | |
| 6. DNA isolation and Clean-up Kits | |||
| Genomic DNA isolation and purification kit | Promega | #A1120 | |
| Plasmid Midi kit | Qiagen | # 12143 | |
| QIAquick PCR purification kit | Qiagen | #28104 | |
| 7. Equipment for PCR, Transformation and Replica-pinning | |||
| Thermal cycler | BioRad, iCycler | ||
| Agarose gel electrophoresis | BioRad | ||
| Electroporator | Bio-Rad GenePulser II | ||
| 0.2 cm electroporation cuvette | Bio-Rad | ||
| 42 °C water bath shaker | Innova 3100 | ||
| Beckman Coulter TJ-25 centrifuge | Beckman Coulter | ||
| 32 °C shaker | New Brunswick Scientific, USA | ||
| 32 °C plate incubator | Fisher Scientific | ||
| RoToR-HDA benchtop robot | Singer Instruments | ||
| 96, 384 and 1,536 pin density pads | Singer Instruments | ||
| 96 or 384 long pins | Singer Instruments | ||
| 8. Imaging Equipments | |||
| Camera stand | Kaiser | ||
| Digital camera, 10 megapixel | Any Vendor | ||
| Light boxes, Testrite 16" x 24" units | Testrite | ||
| 9. Pads or Plates Recycling | |||
| 10% bleach | Any Vendor | ||
| 70% ethanol | Any Vendor | ||
| Sterile distilled water | Any Vendor | ||
| Flow hood | Any Vendor | ||
| Ultraviolet lamp | Any Vendor | ||
| 10. Labware | |||
| 50 ml polypropylene tubes | Any Vendor | ||
| 1.5 ml micro-centrifuge tubes | Any Vendor | ||
| 250 ml conical flaks | VWR | # 29140-045 | |
| 15 ml sterile culture tubes | Thermo Scientific | # 366052 | |
| Cryogenic vials | VWR | # 479-3221 | |
| Rectangular Plates | Singer Instruments | ||
| 96-well and 384-well microtitre plates | Singer Instruments | Nunc | |
| Plate roller for sealing multi-well | Sigma | #R1275 | |
| plates | ABgene | # AB-0580 | |
| Adhesive plate seals | Fisher Scientific | # 13-990-14 | |
| -80 °C freezer | Any Vendor | ||
参考文献
- Bandyopadhyay, S., Kelley, R., Krogan, N. J., Ideker, T. Functional maps of protein complexes from quantitative genetic interaction data. PLoS Comput. Biol. 4, e1000065 (2008).
- Costanzo, M., Baryshnikova, A., Myers, C. L., Andrews, B., Boone, C. Charting the genetic interaction map of a cell. Curr. Opin. Biotechnol. 22, 66-74 (2011).
- Costanzo, M., et al. The genetic landscape of a cell. Science. 327, 425-4231 (2010).
- Fiedler, D., et al. Functional organization of the S. cerevisiae phosphorylation network. Cell. 136, 952-963 (2009).
- Roguev, A., et al. Conservation and rewiring of functional modules revealed by an epistasis map in fission yeast. Science. 322, 405-4010 (2008).
- Schuldiner, M., et al. Exploration of the function and organization of the yeast early secretory pathway through an epistatic miniarray profile. Cell. 123, 507-519 (2005).
- Wilmes, G. M., et al. A genetic interaction map of RNA-processing factors reveals links between Sem1/Dss1-containing complexes and mRNA export and splicing. Mol. Cell. 32, 735-746 (2008).
- Babu, M., et al. Systems-level approaches for identifying and analyzing genetic interaction networks in Escherichia coli and extensions to other prokaryotes. Mol. Biosyst. 12, 1439-1455 (2009).
- Butland, G., et al. coli synthetic genetic array analysis. Nat. Methods. 5, 789-7895 (2008).
- Typas, A., et al. Regulation of peptidoglycan synthesis by outer-membrane proteins. Cell. 143, 1097-10109 (2010).
- Baba, T., et al. Construction of Escherichia coli K-12 in-frame, single-gene knockout mutants: the Keio collection. Mol. Syst. Biol. 2, 2006-200008 (2006).
- Babu, M., et al. Genetic interaction maps in Escherichia coli reveal functional crosstalk among cell envelope biogenesis pathways. PLoS Genet. 7, e1002377 (2011).
- Nichols, R. J., et al. Phenotypic landscape of a bacterial cell. Cell. 144, 143-156 (2011).
- Babu, M., Gagarinova, A., Greenblatt, J., Emili, A. Array-based synthetic genetic screens to map bacterial pathways and functional networks in Escherichia coli. Methods Mol Biol. 765, 125-153 (2011).
- Datsenko, K. A., Wanner, B. L. One-step inactivation of chromosomal genes in Escherichia coli K-12 using PCR products. Proc. Natl. Acad. Sci. U.S.A. 97, 6640-665 (2000).
- Yu, D., et al. An efficient recombination system for chromosome engineering in Escherichia coli. Proc. Natl. Acad. Sci. U.S.A. 97, 5978-5983 (2000).
- Typas, A., et al. quantitative analyses of genetic interactions in. E. coli. Nat. Methods. 5, 781-787 (2008).
- Zeghouf, M., et al. Sequential Peptide Affinity (SPA) system for the identification of mammalian and bacterial protein complexes. J. Proteome Res. 3, 463-468 (2004).
- Davierwala, A. P., et al. . , 1147-1152 (2005).
- Breslow, D. K., et al. A comprehensive strategy enabling high-resolution functional analysis of the yeast genome. Nat. Methods. 5, 711-718 (2008).
- Babu, M., et al. Sequential peptide affinity purification system for the systematic isolation and identification of protein complexes from Escherichia coli. Methods Mol. Biol. 564, 373-400 (2009).
- Butland, G., et al. Interaction network containing conserved and essential protein complexes in Escherichia coli. Nature. 433, 531-537 (2005).
- Hu, P., Janga, S. C., Babu, M., Diaz-Mejia, J. J., Butland, G., et al. Global functional atlas of Escherichia coli encompassing previously uncharacterized proteins. PLoS Biol. 7, (2009).
- Anderson, R. P., Roth, J. R. Tandem genetic duplications in phage and bacteria. Annu. Rev. Microbiol. 31, 473-505 (1977).
- Boone, C., Bussey, H., Andrews, B. J. Exploring genetic interactions and networks with yeast. Nat. Rev. Genet. 8, 437-449 (2007).
- Collins, S. R., et al. Functional dissection of protein complexes involved in yeast chromosome biology using a genetic interaction map. Nature. 446, 806-8010 (2007).
- Le Meur, N., Gentleman, R. Modeling synthetic lethality. Genome Biol. 9, R135 (2008).
- Tong, A. H., et al. Systematic genetic analysis with ordered arrays of yeast deletion mutants. Science. 294, 2364-2368 (2001).
- Tong, A. H., et al. Global mapping of the yeast genetic interaction network. Science. 303, 808-813 (2004).
- Wong, S. L., et al. Combining biological networks to predict genetic interactions. Proc. Natl. Acad. Sci. U.S.A. 101, 15682-15687 (2004).
- van Opijnen, T., Bodi, K. L., Camilli, A. Tn-seq: high-throughput parallel sequencing for fitness and genetic interaction studies in microorganisms. Nat. Methods. 6, 767-772 (2009).
- Gagarinova, A., Emili, A. Genome-scale genetic manipulation methods for exploring bacterial molecular biology. Mol. Biosyst. 8, 1626-1638 (2012).
- Dewey, C. N., et al. Positional orthology: putting genomic evolutionary relationships into context. Brief Bioinform. 12, 401-412 (2011).
- St Onge, R. P., et al. Systematic pathway analysis using high-resolution fitness profiling of combinatorial gene deletions. Nat. Genet. 39, 199-206 (2007).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved