Method Article
Um Protocolo de Chelex simples para extração de DNA de
Neste Artigo
Resumo
Uma maneira rápida e acessível para extrair qualidade parasita da malária e de DNA a partir de amostras do mosquito vetor é descrito. Capitalizando propriedades quelantes de resina Chelex, o método simples que permite a determinação do genótipo de parasitas da malária, em fases de glândulas de mosquitos mid-intestinal e de saliva, bem como a identificação molecular da Anopheles Sibling espécies por PCR.
Resumo
Países endêmicos estão adotando cada vez mais ferramentas moleculares para tipagem eficiente, identificação e vigilância contra os parasitas da malária e mosquitos vetores, como parte integrante de seus programas de controlo 1,2,3,4,5. Para o estabelecimento sustentável dessas abordagens precisos nas operações de pesquisa para fortalecer o controle da malária e os esforços de eliminação, métodos simples e acessíveis, com o reagente parcimoniosa e requisitos de equipamentos são essenciais 6,7,8. Aqui apresentamos uma técnica baseada Chelex simples para extrair parasita da malária e DNA vetor de espécimes coletados no campo de mosquito.
Nós identificados morfologicamente 72 Anopheles gambiae sl. Das 156 mosquitos capturados por piretro spray de capturas em quartos de dormir das famílias dentro de uma vizinhança 2.000 km 2, do Instituto Malária em Macha. Após a dissecção de separar a cabeça eo tórax do abdômen para todas as 72 Anopheles gambiae sl. mosquitos, os dois cortes foram colocados individualmente em tubos de microcentrífuga de 1,5 ml e imerso em 20 ul de água desionizada. Utilizando uma ponta de pipeta estéril, cada secção de mosquito foi separadamente homogeneizados a uma suspensão uniforme na água desionizada. Do homogeneizado resultante a partir de cada secção de mosquito, 10 ul foi retida enquanto que o outro 10 ul foram transferidas para um tubo separado ml autoclavado 1.5. As alíquotas separadas foram submetidas à extração de DNA por qualquer Chelex o simplificado ou o padrão de salga a extração protocolo 9,10. O protocolo de salting out é chamado e amplamente utilizado porque emprega altas concentrações de sal em lugar de solventes orgânicos perigosos (tais como fenol e clorofórmio) para o passo de precipitação de proteína durante a extracção de ADN 9.
Os extractos foram utilizados como moldes para a amplificação por PCR utilizando iniciadores com alvo mitocondrial artrópode dehydroge nicotinamida-adenina-dinucleótidonase (NADH) subunidade 4 gene (ND4) para verificar a qualidade do DNA 11, um PCR para identificação da espécie Anopheles gambiae 10 irmãos e um PCR para a digitação de infecção por Plasmodium falciparum 12. Comparação com a qualidade do DNA (ND4) PCR mostrou sensibilidade de 93% e especificidade de 82% para a abordagem Chelex relação ao estabelecido protocolo de salting out. Os correspondentes valores de sensibilidade e especificidade foram de 100% e 78%, respectivamente, utilizando-se a identificação da espécie irmão PCR e 92% e 80%, respectivamente, para P. falciparum detecção PCR. Não houve diferenças significativas na percentagem de amostras que deram sinal amplicon com o Chelex ou a regular as trocas salinas protocolo em todas as três aplicações de PCR. A abordagem Chelex necessárias três reagentes simples e 37 min para completar, enquanto o. Salting out protocolo envolveu 10 diferentes reagentes e 2 horas e 47 minutos de tempo 'de processamento, incluindo uma etapa durante a noite Nossos resultados mostram ªno método Chelex é comparável à extração salting out existente e pode ser substituído como uma abordagem simples e sustentável de recursos limitados, onde uma cadeia de fornecimento constante de reagente é muitas vezes difícil de manter.
Protocolo
1. Dissecção Preparatória de espécimes do mosquito
- Mosquito lugar em um corte pequeno de parafilme e montagem em microscópio de dissecção.
- Cobrir a gota de água desionizada, para amaciar o tecido.
- Faça uma incisão na carcaça mosquito precisamente na articulação entre o tórax eo abdômen, assim nicking fora a cabeça eo tórax da seção abdominal.
- Transfira cada seção mosquito dissecado em um tubo de microcentrífuga limpo autoclavado de 1,5 ml prelabeled com detalhes de identificação do mosquito número e adequadamente marcados para indicar seção do corpo (por exemplo, "m" para meados de intestino seção; "ht" para a cabeça e tórax seção).
- Descarte parafilme e limpe cuidadosamente estágio do microscópio com Kimwipe 70% de álcool-umedecidos antes de processar o mosquito seguinte.
2. Extração de DNA a partir de amostras de mosquitos
- Pipetar 20 ul de água desionizada num tubo de amostra e a ponta da pipeta para moer o uso a secção de mosquito submerso num unsuspensão iForm.
- Adicionar 100 ul de autoclavado 1X PBS / solução de saponina 1% a homogeneizado amostra e misturar por vórtex momentânea suave.
- Incubar à temperatura ambiente durante 20 min.
- Centrifugar a 20.000 xg durante 2 minutos e descartar o sobrenadante.
- Ressuspender o sedimento em 100 ul de PBS 1X.
- Centrifugar novamente a 20000 xg durante 2 min e descartar o sobrenadante.
- Por centrifugação suave (5 segundos), pellet ressuspender em 75 uL de água estéril desionizada e 25 uL de 20% w / v de suspensão de resina Chelex-100, em água desionizada.
- Pierce buraco na tampa do tubo de amostra usando agulha hipodérmica estéril 23G inflamado no bico de Bunsen.
- Suspensão fervura da amostra em banho-maria na cremalheira de flutuação (ou no bloco de aquecimento), durante 10 min.
- Centrifugar a 20.000 xg, durante 1 min e uma solução de transferência de DNA que se seguiu no frasco de armazenamento prelabeled para utilização como molde de PCR em aplicações.
3. Plasmodium falciparum Genotipagem e Anophelesspp. Identificação Molecular
- Adicionar 2,5 ul do extrato de DNA Chelex em 25 ul de reações de PCR para verificar a qualidade do DNA 11, identificar a espécie Anopheles 10 e ao genótipo P. falciparum 12 meados de intestino e infecções da glândula salivar.
- Para maximizar o rendimento de amplicon, especialmente para P. falciparum, detecção incluem 1.5X albumina de soro bovino (BSA) no ensaio de PCR. A composição de reacção que se segue é recomendada: (2,5 ul template, primers 0,25 uM, 1,5 uM de cloreto de magnésio, 200 uM dNTPs, 1X tampão de PCR, 1,5 X e BSA 1.0u polimerase de ADN Taq, em 25 volumes uL).
- Para verificar a qualidade do DNA em extrato, amplificar região do mitocondrial desidrogenase nicotinamida adenina dinucleotídeo artrópodes (NADH) subunidade 4 (ND4) gene (primers ND4FW [5'-GTD YAT TTA TGA TTR CCT AA-3 '] e ND4RV [5' -CTT CGD CTT CCW ADW CGT TC-3 ']; esperar produto 400pb) 11,13.
- Para diferenciar Anopheles gambiade e sl espécies. irmãos (An. gambiae ss e Uma arabiensis. apenas), amplificar região flanqueando SNPs no espaçador intergênica (IGS) região (primers ONU [5'-GTGTGCCCCTTCCTCGATGT-3 '], GA [5'-CTGGTTTGGTCGGCACGTTT- 3 '], AR [5'-AAGTGTCCTTCTCCATCCTA-3']; esperar 390bp Um produto ss gambiae, 315bp Uma arabiensis) 10,11.
- Para a tipagem P. falciparum antifolato polimorfismos de resistência a drogas, realizar PCR aninhada para amplificar região flanqueadora codões de aminoácidos 108, 51, 59, 16 e 164 no parasita diidrofolato redutase (DHFR), gene:
Primers redondos primárias: M1 [5'-TTTATGATGGAACAAGTCTGC-3 '] e M5 [5'-AGTATATACATCGCTAACAGA-3']
Secundárias primers redondos: M3 [5'-TTTATGATGGAACAAGTCTGCGACGTT-3 '] com F / [5'-AAATTCTTGATAAACAACGGAAACCTTTTA-3']
Ou
F [5'-GAAATGTAATTCCCTAGATATGGAATATT-3] com M4 [5'-TTAATTTCCCAAGTAAAACTATTAGAGCTTC-3 ']).
Também amplificar região contendo codões de aminoácidos 436, 437, 540, 581 umnd 613 no P. falciparum dihidropteroato sintetase gene (dhps):
Primário redondo primers R2 [5'-AACCTAAACGTGCTGTTCAA-3 '] com R / [5'-AATTGTGTGATTTGTCCACAA3'];
Secundárias primers redondas
J: [5'-'TGCTAGTGTTATAGATATAGGTGGAGAAAGC-3'] com K / [5'-CTATAACGAGGTATTGCATTTATTGCAAGAA-3 ']
Ou
K: [5'-TGCTAGTGTTATAGATATAGGATGAGCATC-3 '] com K /,
Ou
L [5'-ATAGGATACTATTTGATATTGATATTGGACCAGGATTCG-3 '] com L / [5'-5TATTACAACATTTTGATCATTCGCGCAACCGG-3'] 12. - Amplicon Sujeito 4 ul de alelo-específicos digestões com enzimas de restrição, em 30 ul de reacções seguindo as instruções do fabricante, para detectar drogas antifolato associadas à resistência polimorfismos 12.
- Ul sujeito 5 de PCR amplicon (ou 15 ul de digestão de restrição) por pista de etídio corado com brometo de gel de agarose a 2% para electroforese (100 - 120V) e visualizar as bandas num transiluminador UV.
Resultados
Exemplos de resultados de ensaios de PCR para a qualidade do extracto mosquito DNA (Figura 1), Anopheles arabiensis identificação molecular (Figura 2) e P. falciparum de detecção (Figura 3) mostra que o método de Chelex simplificada dá resultados semelhantes ao padrão de salting out protocolo 10, apesar de muito menos passos (Tabela 1). Com uma qualidade comparável DNA nos respectivos extractos, não é surpreendente que a taxas de positividade de amostra em relação a um. espécies crípticas gambiae, bem como as taxas de infecção do parasita não foram estatisticamente diferentes com base no teste de McNemar qui-quadrado (Figura 3).
Sensibilidade (%) foi calculada como TP / (TP + FN) * 100, onde TP denota verdadeiros positivos e falsos negativos FN indica. Especificidade (%) foi determinada como TN / (TN + FP) * 100, onde TN indica verdadeiros negativos e FP denota falsos positivos. A qualidade do ADN (ND4) PCR mostrou sensibilidade de 93% e especificidade de 82% para a abordagem Chelex em comparação com o padrão estabelecido ouro salting out protocolo como. Os correspondentes valores de sensibilidade e especificidade foram de 100% e 78%, respectivamente, utilizando-se a identificação da espécie irmão PCR e 92% e 80%, respectivamente, para P. falciparum detecção PCR.
A adição de BSA para misturas de reacção resultou em um aumento geral de positivos de PCR (Figura 3), devido ao alívio da PCR inibidores 14, tanto para o Chelex simplificada e regular salting out protocolo. No entanto, este aumento não foi estatisticamente significativa, exceto para a qualidade do DNA (ND4 PCR) em extratos Chelex (p = 0,039). Específica de alelo de digestão com enzimas de restrição em P. falciparum DHFR (ou outro gene alvo) amplicon permite a genotipagem de infecções da glândula do intestino médio e salivares malária para os alelos de resistência da droga (Figura 4, os dados de glândulas salivares não mostrado).
| Procedimento simples Chelex | Padrão Salga fora Procedimento | ||
| Passos | Reagentes | Passos | Reagentes |
| PBS
Chelex-100 esferas |
| Dietilpirocarbonato (DEPC)
8 de acetato de potássio M Etanol 0,1 X salina tampão de citrato de sódio (SSC) RNAse (10 ug / ml) |
| Tempo total: 37 min | Tempo total: 2 horas 47 min, mais durante a noite | ||
Tabela 1. Passo a passo de comparação dos reagentes e requisitos de tempo para a Chelex protocolo simples e ficarard salting out método de extração de DNA a partir de amostras de mosquitos.

Figura 1. ND4 mitocondrial comparação PCR de DNA de qualidade simplificada Chelex "C" e padrão salting out "M" extractos de amostras de campo Anopheles arabiensis. NC, controlo negativo; M1 e C1, C2 e M2, M3 e C3, e C4 e M4 são emparelhados salting out e extractos a partir de uma mesma Chelex. espécimes do mosquito arabiensis. L, 100 bp ADN.
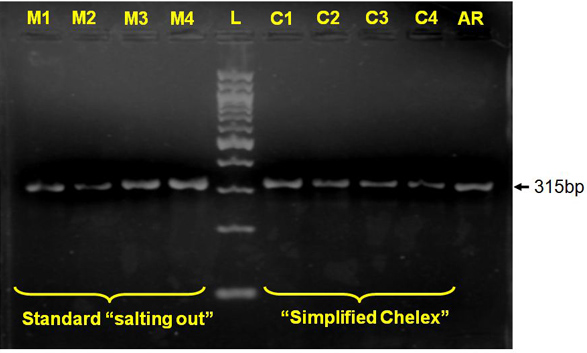
Figura 2 molecular de PCR para identificação arabiensis Anopheles C1 e M1, M2 e C2, C3 e M3, M4 e C4 denotam amplicon respectiva de salting out "M" e simplificado Chelex "C" extratos de DNA para amostras de mosquitos mesmos;.. AR, Anopheles arabiensiscontrole positivo; L, 100 bp ADN.

Figura 3. Detecção de positivos sobre Chelex e salga fora extratos de DNA de amostras de campo do mosquito, com ensaios de PCR para a identificação molecular de Anopheles arabiensis (AR) 10, artrópode gene NADH desidrogenase 4 (ND4) teste de DNA de qualidade 11 e P resistência antifolato droga . falciparum DHFR genotipagem 12 (F-M4). Os resultados apresentados para os testes realizados com ou sem BSA na mistura de reacção. O teste de McNemar de qui-quadrado foi usado para determinar se as diferenças entre cento dos PCRs positivas de DNA extraído a partir da Chelex simplificada e o padrão de salting out protocolos foram estatisticamente significativas.
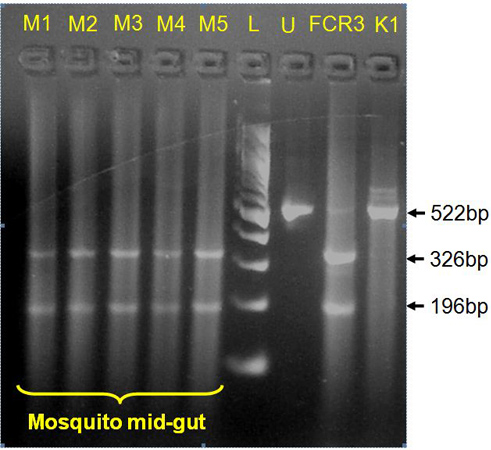
Figura 4. Aplicação da simplificado Chelex protocolo em genotipagem P. falciparum em mosquitos (meados de intestino dados mostrados) para os alelos de resistência a drogas. BstN I em digestão amplicon flanqueando codão 108 do P. falciparum gene DHFR 12 mostra os mutantes resistentes cicloguanilo S108T em amostras de mosquitos (M1-M5; mid-intestinal dados apresentados). U, 522 pb digerido amplicon; FCR3, padrão de laboratório P. falciparum clone controle positivo levando S108T; K1, P. falciparum laboratório padrão de controle clone contábil negativo cicloguanilo sensível S108N, L, 100 bp ADN.
Discussão
O método aqui apresentado Chelex simplificado permite que a extracção de qualidade Anopheles spp e P. falciparum a partir de amostras de DNA de mosquitos passíveis de diversas aplicações de PCR. Esta técnica pode ser utilizada para a identificação molecular de mosquitos vectores da malária e de vigilância de fármaco-resistente P. alelos falciparum em mosquitos para os programas nacionais de controlo da malária. As vantagens da técnica Chelex simplificado incluem a simplicidade, os reagentes menos e, portanto, o custo, a segurança e o tempo de processamento mais curto (37 min) do que os protocolos standard, tais como o método de salting out 10 (2 h 47 min e uma etapa durante a noite, a Tabela 1). As vantagens e os requisitos acima mencionados reagentes mínimos (3 reagentes) em relação ao protocolo padrão atual (10 reagentes, Tabela 1) 11,15, fazer o protocolo simplificado Chelex particularmente amigável para os laboratórios de países endêmicos, onde reagentes uma constantecadeia de fornecimento é muitas vezes difícil de manter. Uma limitação deste método é que, como o protocolo padrão, foi também sujeito a inibidores de PCR conhecidos para ocorrer no tegumento mosquito 16. No entanto, este é prontamente aliviada pela inclusão de BSA nos ensaios. BSA também tem sido empregada com sucesso como um realçador contra os inibidores da amplificação em outras aplicações de PCR 17,18.
Divulgações
Não há conflitos de interesse declarados.
Agradecimentos
Os autores estão em dívida com os chefes, chefes e comunidades de Macha para seu unwaveing cooperar durante as coletas de amostras de mosquitos de campo. Dr. Doug Norris e Rebeca Kent ofereceu conhecimentos inestimáveis sobre o protocolo de salting out. Este trabalho foi financiado pela malária Hopkins Johns Research Institute piloto sistema de concessão. Mosquito e padrões de DNA do parasita laboratoriais foram gentilmente doado por MR4 Tipo, American & Culture Collection.
Materiais
| Name | Company | Catalog Number | Comments |
| Nome do reagente | Companhia | Número de catálogo | Comentários (opcional) |
| Chelex-100, 50-100 mesh seca | BioRad | # 143-2832 | |
| Saponina | SIGMA | 142-7425 | |
| BSA | New England Biolabs | B9001S |
Referências
- Bonnet, M., et al. Efficacy of antimalarial treatment in Guinea: in vivo study of two artemisinin combination therapies in Dabola and molecular markers of resistance to sulphadoxine-pyrimethamine in N'Zerekore. Malar. J. 6, 54 (2007).
- Nsimba, B., et al. Sulphadoxine/pyrimethamine versus amodiaquine for treating uncomplicated childhood malaria in Gabon: a randomized trial to guide national policy. Malar. J. 7, 31 (2008).
- Oyewole, I. O., et al. Behaviour and population dynamics of the major anopheline vectors in a malaria endemic area in southern Nigeria. J. Vector Borne Dis. 44, 56-64 (2007).
- Harris, I., et al. A large proportion of asymptomatic Plasmodium infections with low and sub-microscopic parasite densities in the low transmission setting of Temotu Province, Solomon Islands: challenges for malaria diagnostics in an elimination setting. Malar. J. . 9, 254 (2010).
- Ouedraogo, A. L., et al. Substantial contribution of submicroscopical Plasmodium falciparum gametocyte carriage to the infectious reservoir in an area of seasonal transmission. PLoS ONE. 4, e8410 (2009).
- Birx, D., de Souza, M., Nkengasong, J. N. Laboratory challenges in the scaling up of HIV, TB, and malaria programs: The interaction of health and laboratory systems, clinical research, and service delivery. Am. J. Clin. Pathol. 131, 849-851 (2009).
- Ishengoma, D. R., et al. Health laboratories in the Tanga region of Tanzania: the quality of diagnostic services for malaria and other communicable diseases. Ann. Trop. Med. Parasitol. 103, 441-453 (2009).
- Alonso, P. L., et al. A research agenda for malaria eradication: vector control. PLoS Med. 8, e1000401 (2011).
- Grimberg, J., et al. A simple and efficient non-organic procedure for the isolation of genomic DNA from blood. Nucleic Acids Res. 17, 8390 (1989).
- Scott, J. A., Brogdon, W. G., Collins, F. H. Indentification of single specimens of the Anopheles gambiae complex by polymerase chain reaction. American Journal of Tropical Medicine and Hygiene. 49, 520-529 (1993).
- Kent, R. J., Norris, D. E. Identification of mammalian blood meals in mosquitoes by a multiplexed polymerase chain reaction targeting cytochrome B. Am. J. Trop. Med. Hyg. 73, 336-342 (2005).
- Duraisingh, M. T., Curtis, J., Warhurst, D. C. Plasmodium falciparum: detection of polymorphisms in the dihydrofolate reductase and dihydropteroate synthetase genes by PCR and restriction digestion. Exp. Parasitol. 89, 1-8 (1998).
- Gorrochotegui-Escalante, N., Munoz, M. L., Fernandez-Salas, I., Beaty, B. J., Black, W. C. T. Genetic isolation by distance among Aedes aegypti populations along the northeastern coast of Mexico. Am. J. Trop. Med. Hyg. 62, 200-209 (2000).
- Kreader, C. A. Relief of amplification inhibition in PCR with bovine serum albumin or T4 gene 32 protein. Appl. Environ. Microbiol. 62, 1102-1106 (1996).
- Norris, D. E., Shurtleff, A. C., Toure, Y. T., Lanzaro, G. C. Microsatellite DNA polymorphism and heterozygosity among field and laboratory populations of Anopheles gambiae ss (Diptera Culicidae). J. Med. Entomol. 38, 336-340 (2001).
- Schriefer, M. E., Sacci, J. B., Wirtz, R. A., Azad, A. F. Detection of polymerase chain reaction-amplified malarial DNA in infected blood and individual mosquitoes. Exp. Parasitol. 73 (91), 311-316 (1991).
- Al-Soud, W. A., Radstrom, P. Purification and characterization of PCR-inhibitory components in blood cells. J. Clin. Microbiol. 39, 485-493 (2001).
- Hyun, C., Filippich, L. J., Hughes, I. The inhibitory effect of pentobarbitone on reverse transcription-PCR. J. Biochem. Biophys. Methods. 62, 63-68 (2005).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados