Method Article
Un protocollo Chelex semplice per l'estrazione del DNA da
In questo articolo
Riepilogo
Un modo rapido e conveniente per estrarre parassita della malaria qualità e il DNA da campioni di zanzara vettore è descritto. Capitalizzando proprietà chelanti di resina Chelex, il semplice metodo consente genotipizzazione dei parassiti della malaria in fasi zanzara ghiandole mid-intestinali e salivare, nonché identificazione molecolare del Anopheles Sibling specie mediante PCR.
Abstract
Paesi endemici stanno sempre più adottando strumenti molecolari per la tipizzazione efficiente, identificazione e sorveglianza contro i parassiti della malaria e le zanzare vettore, come parte integrante dei loro programmi di controllo 1,2,3,4,5. Per la creazione sostenibile di questi approcci precisi nelle operazioni di ricerca per rafforzare il controllo della malaria e gli sforzi di eliminazione, metodi semplici e convenienti, con reagente parsimoniosa e alle attrezzature sono essenziali 6,7,8. Qui vi presentiamo una semplice Chelex tecnica basata per l'estrazione parassita della malaria e il DNA da campioni di zanzara vettore raccolti sul campo.
Abbiamo identificato morfologicamente 72 Anopheles gambiae sl. Da 156 zanzare catturate dalla piretro spruzzo catture accessorie nelle camere da letto delle famiglie all'interno di un km 2.000 2 pressi dell'Istituto malaria a Macha. Dopo la dissezione, per separare la testa e il torace dall'addome per tutti Anopheles 72 gambiae sl. zanzare, le due sezioni sono state singolarmente messo in tubi microcentrifuga da 1,5 ml e immerso in 20 ml di acqua deionizzata. Usando una pipetta sterile, ogni sezione zanzara stato omogeneizzato separatamente ad una sospensione uniforme in acqua deionizzata. Dell'omogenato derivanti da ciascuna sezione zanzara, 10 microlitri è stata conservata mentre l'altro 10 microlitri è stato trasferito ad una provetta separata autoclavata 1,5 ml. Le aliquote separate sono stati sottoposti ad estrazione del DNA sia dal Chelex semplificata o lo standard salting out estrazione protocollo 9,10. Il protocollo salting out è il cosiddetto e ampiamente utilizzati perché impiega elevate concentrazioni saline in luogo di pericolosi solventi organici (come fenolo e cloroformio) per la fase di precipitazione della proteina durante l'estrazione del DNA 9.
Gli estratti sono stati utilizzati come modelli per l'amplificazione PCR utilizzando primers mirati artropodi mitocondriale dehydroge nicotinammide-adenina-dinucleotidenase (NADH) subunità 4 gene (ND4) per verificare la qualità del DNA 11, una PCR per l'identificazione di specie Anopheles gambiae fratelli 10 e un nested PCR per la tipizzazione di infezione da Plasmodium falciparum 12. Confronto con qualità del DNA (ND4) PCR ha mostrato il 93% di sensibilità e specificità 82% per l'approccio Chelex relativo alla stabilita salatura fuori protocollo. Corrispondenti valori di sensibilità e specificità sono state del 100% e 78%, rispettivamente, con fratello identificazione delle specie PCR e il 92% e 80%, rispettivamente, per P. falciparum rilevamento PCR. Non ci sono state differenze significative nella percentuale di campioni che con il segnale amplificato Chelex o il regolare salatura su protocollo in tutte e tre le applicazioni PCR. L'approccio Chelex richiesto tre reagenti semplici e 37 min per completare, mentre la salatura su protocollo ha comportato 10 diversi reagenti e 2 ore e 47 min il tempo di elaborazione ', tra cui un passo durante la notte. I nostri risultati mostrano °al metodo Chelex è paragonabile a quella esistente salting out estrazione e può essere sostituito da un approccio semplice e sostenibile in risorse limitate, dove una costante filiera reagente è spesso difficile da mantenere.
Protocollo
1. Dissezione di preparazione dei campioni Mosquito
- Zanzara Posto su un piccolo taglio di parafilm e supporto sulla dissezione microscopio.
- Copertina in goccia di acqua deionizzata per ammorbidire i tessuti.
- Incidere la carcassa zanzara proprio in corrispondenza della giunzione tra il torace e l'addome, in modo da intaccare la testa e il torace dalla sezione addominale.
- Trasferire ogni sezione zanzara sezionato in un ambiente pulito in autoclave 1,5 ml tubetto microcentrifuga prelabeled con dettagli zanzare numero ID e che portano l'etichetta per indicare la sezione del corpo (ad esempio, "m" per la metà-gut sezione, "ht" per testa e torace sezione).
- Eliminare parafilm e asciugare con cura tavolino del microscopio con il 70% di alcool-imbevuto Kimwipe prima di elaborare la zanzara successivo.
2. Estrazione del DNA da campioni Mosquito
- Pipettare 20 ml di acqua deionizzata in provetta e l'uso della punta di pipetta per macinare la sezione zanzara sommerso in una ONUsospensione iForm.
- Aggiungere 100 ml di PBS 1X autoclavato / soluzione di 1% di saponina omogenato del campione e mescolare dolce vortex momentanea.
- Incubare a temperatura ambiente per 20 min.
- Centrifugare a 20.000 xg per 2 minuti ed eliminare il surnatante.
- Risospendere il pellet in 100 ul di PBS 1X.
- Centrifugare nuovamente a 20.000 xg per 2 minuti ed eliminare il surnatante.
- Vortexando gentilmente (5 sec), risospendere il pellet in 75 microlitri di acqua sterile deionizzata e 25 ml di 20% w / v Chelex-100 sospensione resina in acqua deionizzata.
- Pierce foro nel coperchio della provetta sterile con ago ipodermico 23G fiammato in becco Bunsen.
- Ebollizione sospensione del campione in bagnomaria a cremagliera galleggiante (o in blocco di riscaldamento) per 10 min.
- Centrifugare a 20.000 xg per 1 min e soluzione di DNA conseguente trasferimento nella fiala stoccaggio prelabeled per uso come modello in applicazioni PCR.
3. Plasmodium falciparum Genotipizzazione e Anophelesspp. Identificazione molecolare
- Aggiungere 2,5 ml di estratto DNA Chelex in 25 ul reazioni PCR per verificare la qualità del DNA 11, identificare le specie Anopheles 10 e al genotipo P. falciparum 12 infezioni della ghiandola metà del budello e salivari.
- Per massimizzare la resa amplicone, soprattutto per P. rilevamento falciparum, includono 1.5X albumina di siero bovino (BSA) in PCR. La composizione seguente reazione è raccomandato: (2,5 template pl, 0,25 primers pm, 1,5 mM di cloruro di magnesio, 200 microM dNTPs, 1X PCR Buffer, 1.5X BSA e 1.0U Taq DNA polimerasi, in 25 volumi pl).
- Per verificare la qualità del DNA in estratto, amplificano regione del artropodi deidrogenasi mitocondriale nicotinammide adenin dinucleotide (NADH) subunità 4 (ND4) gene (primer ND4FW [5'-GTD YAT TTA TGA TTR CCT AA-3 '] e ND4RV [5' CTT-CGD CTT CCW ADW CGT TC-3 ']; aspettiamo prodotto 400BP) 11,13.
- Per differenziare Anopheles gambiae SL. specie sorella (An. gambiae ss e An. arabiensis solo), amplificare affiancano la regione SNPs nel distanziale intergenica (IGS) regione (primer ONU [5'-GTGTGCCCCTTCCTCGATGT-3 '], GA [5'-CTGGTTTGGTCGGCACGTTT- 3 '], AR [5'-AAGTGTCCTTCTCCATCCTA-3']; aspettiamo 390bp prodotto una SS gambiae, 315bp arabiensis An) 10,11.
- Per la tipizzazione P. falciparum antifolato polimorfismi di resistenza ai farmaci, eseguire nested PCR per amplificare regione fiancheggiante codoni amminoacidici 108, 51, 59, 16 e 164 nella parassita diidrofolato reduttasi (DHFR) gene:
Primer rotonde primari: M1 [5'-TTTATGATGGAACAAGTCTGC-3 '] e M5 [5'-AGTATATACATCGCTAACAGA-3']
Secondari primer rotonde: M3 [5'-TTTATGATGGAACAAGTCTGCGACGTT-3 '] con F / [5'-AAATTCTTGATAAACAACGGAAACCTTTTA-3']
O
F [5'-GAAATGTAATTCCCTAGATATGGAATATT-3] con M4 [5'-TTAATTTCCCAAGTAAAACTATTAGAGCTTC-3 ']).
Anche amplificare regione contenente codoni amminoacidici 436, 437, 540, 581 And 613 nel P. falciparum diidropteroato sintetasi (DHPS) gene:
Primaria tutto primer R2 [5'-AACCTAAACGTGCTGTTCAA-3 '] con R / [5'-AATTGTGTGATTTGTCCACAA3'];
Secondari primer rotonde
J: [5'-'TGCTAGTGTTATAGATATAGGTGGAGAAAGC-3'] con K / [5'-CTATAACGAGGTATTGCATTTATTGCAAGAA-3 ']
O
K: [5'-TGCTAGTGTTATAGATATAGGATGAGCATC-3 '] con K /,
O
L [5'-ATAGGATACTATTTGATATTGATATTGGACCAGGATTCG-3 '] con L / [5'-5TATTACAACATTTTGATCATTCGCGCAACCGG-3'] 12. - Soggetto 4 amplicone microlitri di digestioni di restrizione allele-specifici enzimi in 30 ul reazioni seguendo le istruzioni del fabbricante, di rilevare antifolato farmaci associate a resistenza polimorfismi 12.
- Soggetto 5 pl di ampliconi PCR (o 15 ml di digestione di restrizione) per pista di bromuro di etidio-colorato gel di agarosio al 2% per elettroforesi (100 - 120V) e visualizzare le bande con transilluminazione UV.
Risultati
Esempi di risultati di saggi di PCR per la qualità zanzara estratto DNA (Figura 1), arabiensis Anopheles identificazione molecolare (Figura 2) e P. falciparum rilevamento (Figura 3) mostra che il metodo semplificato Chelex produce risultati simili allo standard salting out protocollo 10, nonostante passi molto minori (Tabella 1). Con la qualità del DNA comparabile nei rispettivi estratti non è sorprendente che la positività frequenze di campionamento per quanto riguarda An. specie sorella gambiae così come i tassi di infezione del parassita non erano statisticamente differenti in base a McNemar test Chi-quadrato (Figura 3).
Sensibilità (%) è stata calcolata come VP / (VP + FN) * 100, dove TP denota veri positivi e falsi negativi FN indica. Specificità (%) è stato determinato come TN / (TN + FP) * 100, dove TN denota veri negativi e FP denota falsi positivi. La qualità del DNA (ND4) PCR ha mostrato il 93% di sensibilità e specificità 82% per l'approccio Chelex rispetto alla norma stabilita salatura fuori protocollo come l'oro. Corrispondenti valori di sensibilità e specificità sono state del 100% e 78%, rispettivamente, con fratello identificazione delle specie PCR e il 92% e 80%, rispettivamente, per P. falciparum rilevamento PCR.
L'aggiunta di BSA a miscele di reazione determinato un generale aumento di PCR positivi (Figura 3) dovute ai rilievi inibitori della PCR 14, sia per il Chelex semplificato e regolare salting out protocollo. Tuttavia, questo aumento non era statisticamente significativo tranne che per qualità del DNA (ND4 PCR) su estratti Chelex (p = 0.039). Allele-specifica digestione con enzimi di restrizione P. falciparum DHFR (o altro gene target) amplicon consente la genotipizzazione di infezioni metà ghiandola salivare intestinale e malaria per alleli di resistenza ai farmaci (Figura 4; ghiandole salivari dati non mostrati).
| Chelex procedura semplice | Standard salatura fuori procedura | ||
| Passi | Reagenti | Passi | Reagenti |
| PBS
Chelex-100 perle |
| Dietilpirocarbonato (DEPC)
8 M di acetato di potassio Etanolo 0,1 X. Saline di sodio citrato (SSC) tampone RNAsi (10 mg / ml) |
| TEMPO TOTALE: 37 min | TEMPO TOTALE: 2 h 47 min, più durante la notte | ||
Tabella 1. Confronto passo per passo dei reagenti e dei tempi necessari per il protocollo Chelex semplice e standard salatura metodo di estrazione del DNA da campioni di zanzara.

Figura 1. ND4 mitocondriale confronto PCR di DNA di qualità semplificato Chelex "C" e standard salting out "M" su campioni estratti arabiensis campo Anopheles. NC, controllo negativo, M1 e C1, C2 e M2, M3 e C3, C4 e M4 e sono accoppiati salatura ed estratti Chelex da An stessa. arabiensis zanzara campioni. L, 100 bp DNA ladder.
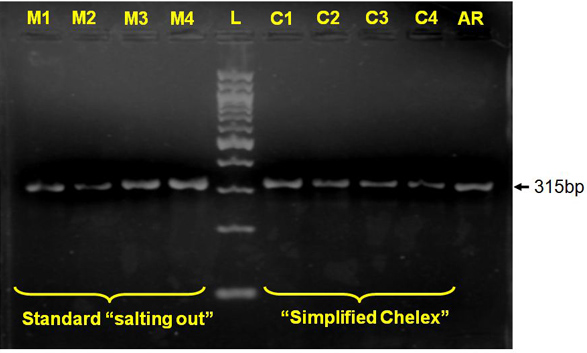
Figura 2 molecolare PCR per l'identificazione arabiensis Anopheles C1 e M1, M2 e C2, C3 e M3, M4 C4 e denotano rispettivamente da amplicon salting out "M" e semplificato Chelex "C" estratti di DNA per gli esemplari di zanzara stessi,.. AR, Anopheles arabiensiscontrollo positivo, L, 100 bp DNA ladder.

Figura 3. Rilevamento di positivi su Chelex e salatura su DNA estratto da campioni prelevati sul campo di zanzara, con saggi di PCR per l'identificazione molecolare di Anopheles arabiensis (AR) 10, artropodi gene NADH deidrogenasi 4 (ND4) qualità del DNA test 11, e antifolato P resistenza ai farmaci . falciparum DHFR genotipizzazione 12 (F-M4). Risultati visualizzati per dosaggi con o senza BSA nella miscela di reazione. McNemar chi-square test è stato utilizzato per determinare se le differenze tra cento di PCR positivi da DNA estratto dal Chelex semplificata e lo standard salting out protocolli erano statisticamente significative.
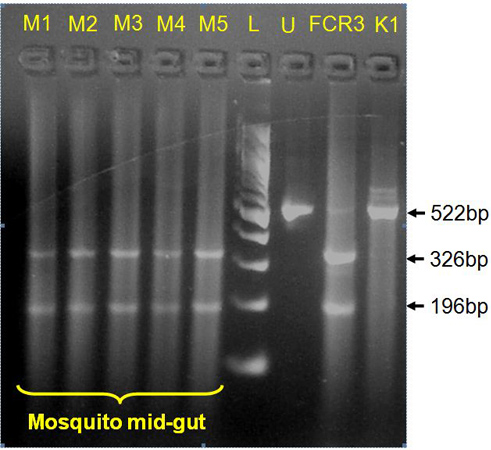
Figura 4. Applicazione del protocollo semplificato Chelex genotipizzazione P. infezioni falciparum in zanzare (mid-gut dati illustrati) per gli alleli di resistenza ai farmaci. BstN ho la digestione su amplicone accompagnamento codone 108 del P. falciparum gene DHFR 12 mostra cicloguanile mutanti resistenti S108T in campioni di zanzara (M1-M5; metà gut dati riportati). U, non digerito 522 bp ampliconi, FCR3, standard di laboratorio P. falciparum clone controllo positivo svolgimento S108T, K1, P. falciparum standard di laboratorio di controllo clone negativo portando cicloguanile sensibile S108N, L, 100 bp DNA ladder.
Discussione
Il metodo semplificato Chelex qui presentato permette l'estrazione di qualità Anopheles spp e P. falciparum DNA da campioni di zanzara suscettibili di diverse applicazioni PCR. Questa tecnica può essere utilizzata per l'identificazione molecolare di zanzare vettore malaria e la sorveglianza della farmaco-resistenti P. alleli falciparum in zanzare per i programmi nazionali di controllo della malaria. I vantaggi della tecnica Chelex semplificata includono semplicità, meno reattivi e quindi dei costi, sicurezza e tempi di lavorazione più brevi (37 min) di protocolli standard quali il salting out metodo 10 (2 h 47 min e un passo overnight, Tabella 1). I vantaggi di cui sopra ed i requisiti minimi dei reagenti (3 reagenti) rispetto al protocollo standard attuale (10 reagenti, Tabella 1) 11,15, rendono il protocollo Chelex semplificato particolarmente cordiale ai laboratori dei paesi endemici in cui un reagente costantefiliera è spesso difficile da mantenere. Una limitazione del metodo è che, come il protocollo standard, è stato anche oggetto di inibitori della PCR noti a verificarsi nel tegumento zanzara 16. Tuttavia, questo è facilmente alleviato inclusione di BSA nei saggi. BSA è stata utilizzata con successo come amplificazione enhancer contro inibitori in altre applicazioni PCR 17,18.
Divulgazioni
Nessun conflitto di interessi dichiarati.
Riconoscimenti
Gli autori sono in debito con i capi tribù, capi e le comunità di Macha per la loro collaborazione nel corso unwaveing collezioni zanzara campione sul campo. Il dottor Doug e Rebecca Norris Kent offerto competenze di alta qualità sul protocollo salatura fuori. Questo lavoro è stato finanziato dalla Johns Hopkins Malaria Research Institute sistema pilota di sovvenzione. Mosquito e parassiti del DNA standard di laboratorio sono stati gentilmente donati da MR4, tipo American & Culture Collection.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti (opzionale) |
| Chelex-100, 50-100 mesh a secco | BioRad | # 143-2832 | |
| Saponina | SIGMA | 142-7425 | |
| BSA | New England Biolabs | B9001S |
Riferimenti
- Bonnet, M., et al. Efficacy of antimalarial treatment in Guinea: in vivo study of two artemisinin combination therapies in Dabola and molecular markers of resistance to sulphadoxine-pyrimethamine in N'Zerekore. Malar. J. 6, 54 (2007).
- Nsimba, B., et al. Sulphadoxine/pyrimethamine versus amodiaquine for treating uncomplicated childhood malaria in Gabon: a randomized trial to guide national policy. Malar. J. 7, 31 (2008).
- Oyewole, I. O., et al. Behaviour and population dynamics of the major anopheline vectors in a malaria endemic area in southern Nigeria. J. Vector Borne Dis. 44, 56-64 (2007).
- Harris, I., et al. A large proportion of asymptomatic Plasmodium infections with low and sub-microscopic parasite densities in the low transmission setting of Temotu Province, Solomon Islands: challenges for malaria diagnostics in an elimination setting. Malar. J. . 9, 254 (2010).
- Ouedraogo, A. L., et al. Substantial contribution of submicroscopical Plasmodium falciparum gametocyte carriage to the infectious reservoir in an area of seasonal transmission. PLoS ONE. 4, e8410 (2009).
- Birx, D., de Souza, M., Nkengasong, J. N. Laboratory challenges in the scaling up of HIV, TB, and malaria programs: The interaction of health and laboratory systems, clinical research, and service delivery. Am. J. Clin. Pathol. 131, 849-851 (2009).
- Ishengoma, D. R., et al. Health laboratories in the Tanga region of Tanzania: the quality of diagnostic services for malaria and other communicable diseases. Ann. Trop. Med. Parasitol. 103, 441-453 (2009).
- Alonso, P. L., et al. A research agenda for malaria eradication: vector control. PLoS Med. 8, e1000401 (2011).
- Grimberg, J., et al. A simple and efficient non-organic procedure for the isolation of genomic DNA from blood. Nucleic Acids Res. 17, 8390 (1989).
- Scott, J. A., Brogdon, W. G., Collins, F. H. Indentification of single specimens of the Anopheles gambiae complex by polymerase chain reaction. American Journal of Tropical Medicine and Hygiene. 49, 520-529 (1993).
- Kent, R. J., Norris, D. E. Identification of mammalian blood meals in mosquitoes by a multiplexed polymerase chain reaction targeting cytochrome B. Am. J. Trop. Med. Hyg. 73, 336-342 (2005).
- Duraisingh, M. T., Curtis, J., Warhurst, D. C. Plasmodium falciparum: detection of polymorphisms in the dihydrofolate reductase and dihydropteroate synthetase genes by PCR and restriction digestion. Exp. Parasitol. 89, 1-8 (1998).
- Gorrochotegui-Escalante, N., Munoz, M. L., Fernandez-Salas, I., Beaty, B. J., Black, W. C. T. Genetic isolation by distance among Aedes aegypti populations along the northeastern coast of Mexico. Am. J. Trop. Med. Hyg. 62, 200-209 (2000).
- Kreader, C. A. Relief of amplification inhibition in PCR with bovine serum albumin or T4 gene 32 protein. Appl. Environ. Microbiol. 62, 1102-1106 (1996).
- Norris, D. E., Shurtleff, A. C., Toure, Y. T., Lanzaro, G. C. Microsatellite DNA polymorphism and heterozygosity among field and laboratory populations of Anopheles gambiae ss (Diptera Culicidae). J. Med. Entomol. 38, 336-340 (2001).
- Schriefer, M. E., Sacci, J. B., Wirtz, R. A., Azad, A. F. Detection of polymerase chain reaction-amplified malarial DNA in infected blood and individual mosquitoes. Exp. Parasitol. 73 (91), 311-316 (1991).
- Al-Soud, W. A., Radstrom, P. Purification and characterization of PCR-inhibitory components in blood cells. J. Clin. Microbiol. 39, 485-493 (2001).
- Hyun, C., Filippich, L. J., Hughes, I. The inhibitory effect of pentobarbitone on reverse transcription-PCR. J. Biochem. Biophys. Methods. 62, 63-68 (2005).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon