Method Article
녹농균(Pseudomonas aeruginosa)에 의해 생산된 Mono-Rhamnolipids 및 Di-Rhamnolipids의 검출 및 정량화
요약
녹농균(Pseudomonas aeruginosa)은 람놀리피드 생물 계면활성제를 생산합니다. 박층 크로마토그래피는 각 균주에 의해 생성된 mono- rhamnolipid와 di-rhamnolipid의 비율을 검출하고 측정합니다. 총 람놀리피드의 정량화에는 오르시놀 방법을 사용하여 배양 상층액에서 추출한 이러한 생물계면활성제에 존재하는 람노스 등가물을 평가하는 것이 포함됩니다.
초록
환경 박테리아 녹농균(Pseudomonas aeruginosa )은 항생제 내성이 높아 건강에 위험을 초래하는 기회 감염 병원체입니다. 이 박테리아는 람놀라이피드(RL)로 알려진 생물 계면활성제를 고농도로 생성하는데, 이 계면활성제는 생명공학적 가치가 있는 분자이지만 독성 특성과도 관련이 있습니다. 이러한 점에서, RL의 검출 및 정량화는 생명공학 응용 분야와 생물 의학 연구 프로젝트 모두에 유용할 수 있습니다. 이 기사에서는 박층 크로마토그래피(TLC)를 사용하여 녹농균에 의해 생성되는 두 가지 형태의 RL의 생성을 검출하는 기술을 단계별로 보여줍니다: 단일 람인지질(mRL), 하나의 람노스 부분에 연결된 지방산 이량체(주로 C10-C10)로 구성된 분자 및 두 개의 람노스 부분에 연결된 유사한 지방산 이량체로 구성된 분자인 디-람놀리피드(dRL). 또한, 녹농 균 배양 상층액에서 추출한 이러한 생물계면활성제의 산 가수분해와 오르시놀과 반응하는 람노스 농도의 후속 검출을 기반으로 RL의 총량을 측정하는 방법을 제시합니다. 두 기법의 조합은 유형 균주 PAO1(계통군 1), PA14(계통군 2) 및 PA7(계통군 3)과 함께 여기에 예시된 바와 같이 특정 균주에 의해 생성된 mRL 및 dRL의 대략적인 농도를 추정하는 데 사용할 수 있습니다.
서문
녹농균(Pseudomonas aeruginosa)은 환경 박테리아이며 독성 관련 형질의 생성과 높은 항생제 내성으로 인해 큰 우려가 있는 기회 감염 병원체입니다 1,2. 이 박테리아에 의해 생성되는 특징적인 2차 대사 산물은 생물 계면활성제 RL이며, 이는 산화 환원 활성을 가진 항생제인 페나진 피오시아닌(phenazine pyocyanin) 및 프로테아제 엘라스타제3와 같은 여러 독성 관련 특성과 조화롭게 생산됩니다. RL의 텐시오 활성 및 유화 특성은 다양한 산업 응용 분야에서 활용되었으며 현재 상용화되어 있습니다4.
계통군 1 및 2에 속하는 대부분의 녹농균 균주는 두 가지 유형의 RL을 생성한다: 주로 10개의 탄소로 구성된 지방산 이량체에 연결된 하나의 람노스 부분으로 구성된 mRL과 첫 번째 람노스4에 연결된 추가적인 람노스 부분을 포함하는 dRL. 그러나 두 개의 작은 녹농균 계통군(그룹 3 및 5)은 mRL 5,6만 생성하는 것으로 보고되었습니다. RL의 두 가지 유형은 언급한 바와 같이 주로 C10-C10인 지방산 이량체의 혼합물을 포함하지만 C12-C10, C12-C12 및 C10-C12:1 이량체를 포함하는 분자의 더 작은 비율도 생성됩니다. HPLC MS/MS를 사용하여 다양한 균주에 의해 생성된 RL 동족체의 특성화가 보고되었습니다 7,8. 이 연구에서 기술된 방법은 mRL과 dRL만 구별할 수 있을 뿐 RL 동족체의 특성 분석에는 사용할 수 없습니다.
녹농균(P. aeruginosa)과 일부 버크홀데리아(Burkholderia) 종은 RL9의 자연 생산자이지만, 이전의 박테리아가 가장 효율적인 생산자입니다. 그러나, 상업적으로 사용되는 RL은 현재 이러한 기회주의적 병원체의 사용을 피하기 위해 녹농균 유전자를 발현하는 슈도모나스 푸티다 KT2440 유도체에서 생산되고 있다(10,11). 녹농균(P. aeruginosa)에 의해 생성된 RL의 검출 및 정량화는 독성 관련 형질(12)의 발현에 관여하는 분자 메커니즘을 연구하고, 군(clade) 3 또는 5에 속하는 균주의 특성 분석(13)하며, 독성을 감소시키면서 이러한 생물계면활성제를 과도하게 생산하는 녹농균 유도체(P. aeruginosa derivatives)를 구성하는 데 매우 중요하다14. 다양한 미생물에 의한 생물 계면 활성제의 생산은 붕괴 방울 방법 또는 유화 지수15와 같은 이러한 화합물의 일부 일반적인 특성에 기초하여 검출되었지만, 이러한 방법은 정확하지도 않고 특이적이지도 않다16.
여기에서는 다양한 녹농균 유형 균주의 배양 상층액에서 총 RL을 액체 추출하고 TLC를 사용하여 두 유형의 RL을 분리하여 mRL 및 dRL을 검출하는 프로토콜에 대해 설명합니다. 이 방법에서는 배양 상층액에서 추출한 RL을 TLC에 사용되는 용매의 용해도에 따른 차등 용해도에 의해 분리하여 실리카겔 플레이트에서 차등 이동을 일으킵니다. 따라서 mRL은 dRL보다 더 빠른 이동을 가지며 플레이트를 건조하고 α-나프톨로 염색할 때 별도의 반점으로 검출될 수 있습니다.
TLC에 의한 mRL 및 dRL을 검출하기 위해 여기에 설명된 방법은 이전에 발표된 논문17을 기반으로 하며, 이는 수행하기 쉽고 고가의 장비를 필요로 하지 않는다. 이 방법은 RL을 생산할 수 없는 P. aeruginosa-유래 돌연변이와 같은 적절한 대조군을 사용하여 다양한 P. aeruginosa 분리체(13)에서 RL을 검출하는 데 유용하다. 그러나, 이는 특이성이 부족하기 때문에 녹농균(Pseudomonas aeruginosa) 이외의 박테리아에 의해 생성되는 새로운 생물 계면활성제를 특성화하는 데 선호되는 방법은 아닙니다.
또한, 녹농균 배양 상층액에서 추출한 총 RL의 람노스 등가물을 정량화하는 방법을 제시합니다. 이 방법은 오르시놀과 환원당의 반응에 기초하여 이러한 생물계면활성제를 정량화하며, 그 결과 앞서 설명한 바와 같이 421nm에서 분광광도계로 측정할 수 있는 생성물이 생성된다18. 오르시놀과의 반응은 람노스에 특이적이지 않기 때문에, 리포다당류(LPS)와 같은 다른 당 함유 분자를 상당량 함유하지 않는 배양 상청액에서 추출한 RL로 이 방법을 수행하는 것이 중요합니다. 산성화된 클로로포름/메탄올 혼합물은 RL18의 액체 추출을 위해 여기에 사용되지만, 에틸 아세테이트도 사용할 수 있으며, 고체상 추출(SPE)은 매우 우수한 결과를 산출한다19. 여기에 설명된 오르시놀 방법은 정교한 장비를 필요로 하지 않으며, 논의된 바와 같이 분석된 샘플을 준비할 때 특별한 주의를 기울여 수행할 경우 신뢰할 수 있는 결과를 제공할 수 있습니다. 적절한 시료 전처리를 위해서는 RL20을 생성할 수 없는 녹농균 rhlA 돌연변이를 포함하고 각 측정에 대해 3번의 생물학적 복제와 3번의 기술적 복제를 수행하는 것이 중요합니다.
문헌16,21에서 오르시놀 방법에 의한 RL 측정과 관련하여 상당한 논란이 있었으며, 일부 연구에서는 RL 생산이 과대평가되고 분석법이 람노스에 대한 특이성이 부족하여 잠재적으로 다른 당을 검출할 수 있다고 제안했습니다. 그러나 여기에서 설명된 방법이 적절한 조건에서 정확하고 구체적일 수 있음을 보여줍니다. 또한 이 기사에 설명된 절차와 비교하기 위해 dRL 표준물질의 UPLC-MS/MS 검출을 활용하고 orcinol 분석법으로도 유사한 결과를 얻을 수 있음을 입증합니다. 이 방법을 사용하여 RL을 정량화하기 위한 자세한 프로토콜은 보충 파일 1에 포함되어 있습니다.
이들 프로토콜은 유형 균주 PAO1(계통군 1), PA14(계통군 2) 및 PA7(계통군 3)을 사용하여 예시된다. 이러한 균주는 특성이 잘 규명되고 다양한 RL 프로필을 생성하기 때문에 선택되었습니다.
프로토콜
이 절차는 보충 그림 1에 도식화되어 있습니다. 연구에 사용된 시약 및 장비는 재료 표에 나열되어 있습니다.
1. TLC를 사용한 녹농균(P. aeruginosa)의 배양 상층액에서 mRL 및 dRL 검출
- 관심 있는 P. aeruginosa 균주의 원심 분리된 육수로 시작하여 액체 배지에서 24시간 동안 배양합니다(RL이 생성되는 고정 성장 단계에 도달하기 위해). 일반적으로 이러한 배양물에는 mL당 1 x 109 개의 박테리아가 포함되어 있습니다.
- 농축된 HCl을 사용하여 배양액의 pH를 2로 조정합니다.
- 50mL 폴리프로필렌 튜브에 산성화된 배양 상층액 5mL를 넣고 5mL의 2:1 클로로포름: 메탄올 혼합물을 추가합니다.
- 매번 10초 동안 뒤집어서 튜브를 세 번 교반하고 각 교반 사이에 2분 동안 튜브를 반전 없이 그대로 둡니다.
- 두 상이 분리될 때까지 약 3시간 동안 튜브를 교반하지 않고 그대로 두거나 4°C에서 3,000 x g 에서 10분 동안 튜브를 원심분리합니다.
- 유기상(바닥층)을 깨끗한 튜브로 옮기고 건조가 발생할 때까지 튜브를 추출 후드에 그대로 둡니다.
- 1.3 단계부터 시작하여 두 번째 클로로포름 : 메탄올 추출의 유기 상을 첫 번째 추출에 사용 된 튜브에 넣습니다.
- 1mL 또는 1.5mL가 남을 때까지 유기상을 증발시킵니다. 이 부피를 1.5mL 원심분리 튜브로 옮기고 용매를 밤새 건조시킵니다.
- 건조된 튜브에 50μL의 메탄올을 첨가하여 RL을 재현탁시킵니다.
- 실리카겔 플레이트에서 박층 크로마토그래피를 수행합니다.
알림: TLC 플레이트의 크기는 분석할 샘플의 수에 따라 절단해야 합니다. 각 샘플은 1.5cm에 적용해야 하며 적용 지점은 플레이트 가장자리에서 1cm 떨어져 있어야 합니다(연필로 수평선 그리기). - 10 μL 마이크로피펫을 사용하여 각 샘플에 5 μL를 적용합니다.
- TLC 액상은 클로로포름: 메탄올: 아세트산의 65:15:2 혼합물로 구성됩니다. 이 혼합물 35mL를 제조하려면 클로로포름 26mL, 메탄올 6mL, 아세트산 20% 원액 0.8mL를 혼합합니다. 이러한 용매를 혼합하고 크로마토그래피를 시작하기 전에 최소 10분 동안 밀폐된 TLC 챔버에 넣어 대기가 휘발성 용매로 포화되도록 합니다.
- 샘플이 적용된 지점과의 접촉을 피하고 TLC 플레이트를 챔버에 놓습니다.
- 챔버를 닫고 용매가 플레이트 가장자리 1cm 전에 도달할 때까지 TLC를 그대로 둡니다. 이 시점에서 플레이트를 제거하고 건조시키십시오 (공기 흐름을 적용하여 프로세스를 가속화 할 수 있음).
- 이 화합물 5g을 에탄올 33mL에 녹여 α-나프톨 용액을 준비합니다. 용해되면 에탄올 127.5mL, 물 12.6mL, 차가운 H2SO4 20.5mL를 첨가합니다.
- mRL 및 dRL의 존재를 감지하려면 추출 후드의 플레이트에 α-나프톨 용액을 분무하고 RL의 분홍빛이 도는 표시가 분명해질 때까지 분무된 플레이트를 85°C의 오븐에 몇 분 동안 넣습니다.
- TLC의 그림을 사용하여 각 스폿의 밀도를 감지하는 소프트웨어를 사용하여 각 샘플에 존재하는 mono- 및 di-RL의 비율을 계산합니다.
2. 생물 계면활성제에 존재하는 람노스 등가물을 측정하는 RL의 총량 정량화
- 1.2mL의 고정상 배양액(24시간 동안 성장)을 1.5mL 원심분리 튜브에 넣고 4°C에서 3,000 x g 에서 3분 동안 원심분리합니다. 깨끗한 튜브에 상층액을 수집합니다.
- 333μL를 깨끗한 튜브에 옮기고(이 단계를 3번 수행) 1mL의 에테르를 추가합니다.
- 30초 동안 소용돌이에서 격렬하게 섞습니다. 한 번에 하나의 튜브로 이 절차를 반복합니다.
- 4 °C에서 3,000 x g 에서 2분 동안 원심분리합니다. 유기상(상부)을 채취하여 깨끗한 튜브에 옮기고 건조가 발생할 때까지 추출 후드에 열어 둡니다.
- 2.2단계에서 설명한 대로 에테르로 추출을 반복합니다. 2.3단계와 2.4단계를 반복합니다.
- 완전히 건조되면 각 튜브에 1mL의 물을 추가합니다. RL이 수화될 수 있도록 튜브를 12시간 동안 그대로 둔 다음 소용돌이에서 격렬하게 교반합니다.
- 깨끗한 플라스크를 얼음에 5분 동안 넣습니다(발열 반응이므로). 60 % H2SO4 (플라스크를 빛으로부터 보호)의 용액을 준비하십시오.
- 깨끗한 튜브에 증류수에 1.6% 오르시놀 용액을 준비합니다. 오르시놀 시약을 준비하려면 4.4 % H2SO4 용액 40 mL와 1.6 % 오르시놀 용액 0.6 mL를 섞는다.
- 필요한 오르시놀 시약의 최종 부피를 계산합니다. 이 시약 900 μL를 각 샘플과 보정 곡선의 각 농도에 첨가하고, 서로 다른 람노스 농도(일반적으로 9 농도의 람노스는 1 μg/mL에서 9 μg/mL 범위에서 사용됨)를 사용하고, 1 농도는 100 μL의 물과 함께 추가하여 모든 것이 삼중으로 수행되도록 합니다.
- 유리관에 있는 오르시놀 시약 900μL에 각 RL 샘플 100μL를 추가하고 두 용액을 혼합합니다.
- 80 °C로 예열된 수조에서 30분 동안 배양합니다.
- 튜브를 실온으로 식히십시오.
- quartz cell을 사용하여 421nm에서 샘플의 흡광도와 보정 곡선을 판독합니다.
- 보정 곡선에서 흡광도를 보간하고 측정에 사용된 부피를 고려하여 각 샘플의 람노스 농도를 계산합니다.
- μM 농도를 계산하려면 μg/mL 단위로 얻은 농도를 182.2(람노스의 분자량)로 나누고 1000을 곱합니다.
결과
이 논문에서는 3개의 서로 다른 P. aeruginosa 유형 균주를 사용하여 각각 다양한 RL 생산 수준과 mRL 및 dRL의 비율을 가진 3개의 계통군을 표현했습니다. 이러한 균주에는 PAO1(호주에서 분리한 상처, 195522), PA14(미국에서 분리한 식물, 197723) 및 PA7(아르헨티나에서 임상적으로 분리한 것, 201024)이 포함된다. 음성 대조군으로는 RL 생산이 불가능한 PAO1 rhlA 돌연변이가 사용되었습니다. 모든 균주는 37°C에서 높은 RL 수준(25)을 촉진하도록 특별히 설계된 PPGAS 배지에서 24시간 동안 배양하였다. PPGAS 배양액은 LB 배지26 오버나이트 배양을 사용하여 600nm에서 0.05의 광학 밀도로 접종하였다. 일반적으로 녹농균 균주는 24시간 후 PPGAS 배지에서 600nm에서 측정된 2의 광학 밀도를 달성하며, 이는 대략 mL당 1 x 109개의 박테리아에 해당합니다. RL 검출 및 정량화를 위해 배양 상층액을 수집하기 위해 배양액을 4°C에서 3,000 x g에서 15분 동안 원심분리하고 세포 펠릿을 폐기했습니다. 이 두 가지 방법(그림 2 및 그림 3)을 사용하여 얻은 결과는 각 균주에 의해 생성된 RL 유형과 생산량이 모두 분석된 세 가지 균주 간에 다르다는 것을 보여줍니다. 그림 2에서 볼 수 있듯이 PAO1은 약 30% mRL 및 70% dRL을 생성하는 반면, PA14는 50% mRL과 50% dRL의 동일한 비율을 생성하고, PA7은 독점적으로 mRL을 생성합니다. 이 RL 생산 프로필은 이전 보고서12,14와 일치합니다.
그림 3A 는 이러한 세 가지 유형의 균주 각각에 의해 생성된 RL에 존재하는 람노스 등가물의 양을 보여줍니다. 검출된 람노스의 양으로부터, PA14 균주가 가장 많은 양의 RL을 생성하는 반면, PA7 균주가 가장 적은 양을 생성한다는 것이 분명하다. 그러나 이러한 결과만으로는 각 균주에 의해 생성된 RL의 μM 농도를 추정하는 데 사용할 수 없습니다. 또한, 오르시놀 분석법만으로는 각 관심 균주에 의해 생성된 mRL 및 dRL의 비율을 특성화하는 데 의존하기 때문에 대략적인 RL 농도를 제공할 수 없습니다. 따라서 RL의 μM 농도 추정치를 얻으려면 TLC에서 측정된 각 유형의 RL 비율을 고려해야 하며 람노스 농도가 다른 표준 곡선을 포함해야 합니다(그림 3B).
오르시놀 방법은 실제로 이 생물 계면활성제의 한 가지 유형만 생산될 때 RL의 농도를 정량화하는 데 사용할 수 있습니다. 이러한 경우, 검출된 람노스의 μM 농도는 mRL의 μM 농도와 직접적으로 일치한다. dRL의 경우 각 dRL 분자의 가수분해에 의해 두 개의 람노스가 생성되기 때문에 RL에서 검출된 람노스의 μM 농도를 2로 나누어 μM 농도를 얻어야 합니다( 그림 4 참조).
그러나 대부분의 녹농균 균주는 다양한 비율로 두 가지 유형의 RL을 모두 생성하므로( 그림 2 참조) 총 RL의 대략적인 농도만 확인할 수 있습니다. 이를 고려하여 각 균주에 의한 RL 생산량을 다음과 같이 추정했습니다: PA7 균주의 경우 mRL만 생성하기 때문에 RL의 일부를 형성하는 람노스(44.6 μg/mL + 4.5 μg/mL)는 μM 농도(244.78)에 직접 해당하며, 이는 각 RL 분자에 하나의 람노스 부분이 포함되어 있으므로 배양 상층액에서 이 생물 계면활성제의 농도를 나타냅니다. 그러나 PAO1 균주의 경우 검출된 람노스 농도가 111.55μg/mL + 11.41μg/mL이지만 mRL에 해당하는 비율은 30%에 불과합니다. 따라서 RL 분자의 70%는 분자당 2개의 람노스를 포함합니다. RL의 농도를 추정하기 위해, 1/5은 mRL(122.49)의 μM 농도에 해당하고 2/5(244.89)는 dRL에 해당한다고 고려하여 μM 람노스 농도(612.24)를 5로 나눴습니다. 따라서 이 균주에 의해 생성된 RL의 대략적인 μM 농도는 367.39입니다.
PA14 균주의 경우 검출된 람노스 농도는 194.39μg/mL + 11.5μg/mL이며, 이를 μM 농도(1,066.9)로 변환하고 3으로 나눴습니다. 결과는 각 유형이 50%에서 생성되었고 dRL이 분자당 2개의 람노스를 포함한다는 점을 고려하면 각 RL 유형의 농도를 나타냅니다. 따라서 이 균주는 355.63μM의 mRL과 동일한 농도의 dRL을 생성하여 RL의 대략적인 μM 농도가 711.27이며, 이는 균주 PAO1의 거의 두 배이고 PA7 균주에 의해 생성된 농도의 거의 세 배입니다.

그림 1: mono-rhamnolipids 및 di-rhamnolipids의 주요 동족체의 화학적 조성. (A) 모노-람놀라이질. (B) 디-람놀리피드. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 2: 박층 크로마토그래피를 이용한 mono-rhamnolipids 및 di-rhamnolipids 검출. (A) RL 표준물질과 PAO1, PA14, PA7 및 PAO1 유래 ΔrhlA 돌연변이에 의해 생성된 RL을 보여주는 TLC 플레이트의 그림. (B) ImageJ 소프트웨어를 사용하여 (A)에서 테스트한 각 샘플에서 각 RL 유형(mRL 및 dRL)의 비율 추정. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: orcinol 방법을 사용한 RL 농도 정량화. (A) PAO1(검은색 막대), PA14(밝은 회색 막대) 및 PA7(짙은 회색 막대) 균주의 배양 상층액에서 추출한 RL에 포함된 람노스 농도(μg/mL). 막대는 표준 편차를 나타냅니다. (B) (A)에 표시된 실험에 대한 람노스 보정 곡선. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
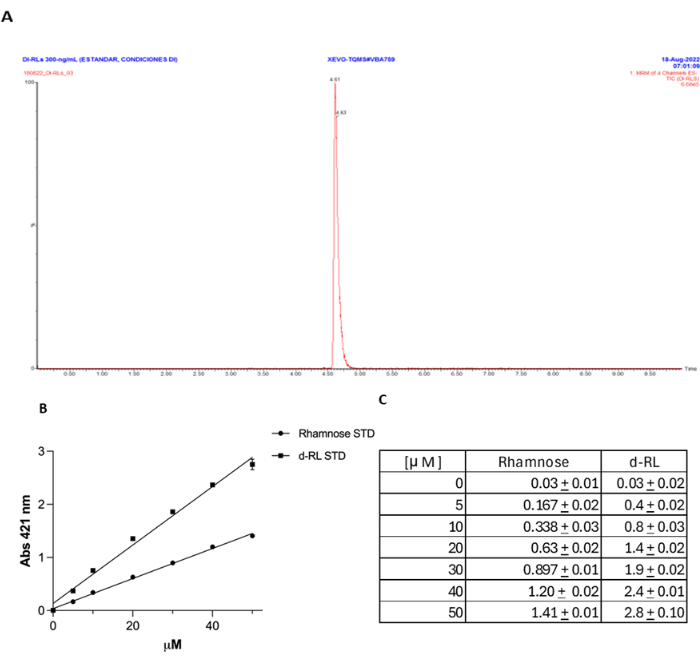
그림 4: dRL 정량을 위해 UPLC-MS/MS와 비교한 orcinol 분석법의 검증. (A) dRL 표준물질은 UPLC-MS/MS를 사용하여 정량화하였다. (B) mM으로 표시되는 람노스 농도와 비교한 오르시놀 방법. (C) 람노스와 동일한 mM 농도의 dRL은 예상대로 421nm에서 약 두 배의 흡광도를 제공합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 그림 1: 프로토콜의 개략적인 표현. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 파일 1: UPLC-MS/MS로 dRL을 정량화하기 위한 세부 프로토콜. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
RL을 검출하고 정량화하는 가장 정확한 방법은 질량 분석법(MS)7,8,27과 결합된 HPLC입니다. 그러나 많은 연구자가 접근하기 어려울 수 있는 전문적이고 값비싼 장비가 필요합니다. 여기에 설명된 방법은 RL 농도를 검출하고 추정하기 위해 기본 실험실 재료 및 장비를 사용하여 일상적으로 수행할 수 있지만, 몇 가지 제한 사항, 특히 mRL 및 dRL 혼합물을 측정하는 데 부정확성이 있습니다. 또한 RL 정량화에 사용되는 시료 준비는 신중하게 실행되어야 합니다. RL 검출 및 정량화에 사용되는 배양은 세포가 응집되거나 용해될 수 있으므로 얻은 결과에 변동을 일으킬 수 있는 비균질성 세포 현탁액이 발생할 수 있으므로 후기 고정상에 있어서는 안 됩니다. 또한 세포 파편이 오르시놀 방법을 방해할 수 있습니다.
RL을 생성할 수 없는 녹농균 균주(예: 본 연구에서 사용된 PAO1 ΔrhlA 돌연변이20, 그림 2)와 같은 적절한 대조군과 함께 설명된 프로토콜을 수행할 때 정확한 결과를 얻을 수 있습니다. 이러한 결과는 다양한 녹농균 분리체(12,13)를 특성화하거나, RL 생산을 증가시키기 위해 균주를 유전적으로 변형하거나14 다양한 녹농균 분리체(12)에 의한 RL 생산의 분자 유전학을 연구하는 데 유용하다.
따라서, 여기에 기술된 방법은 독성 인자 생성에 대한 녹농균 균주를 특성화하고 특히 연구의 초기 단계에서 생명공학적 잠재력을 평가하는 데 사용할 수 있습니다. 이러한 방법은 HPLC/MS와 같은 기술을 대체하기 위한 것이 아니라 녹농균 균주의 초기 균주 특성 분석 및 유전자 조작을 위한 접근 가능한 기술이라는 점에 유의해야 합니다.
또한, 연구가 생물의학적 목적을 위한 RL 생성에 초점을 맞추고 있다면, RL 생성은 녹농균에 의해 생성되는 여러 요인 중 하나일 뿐이라는 점을 인정하는 것이 중요합니다. 또한, 생산된 RL의 유형 또는 그들의 생산량은 특정 균주의 독성과 엄격하게 상관되지 않는데, 이는 독성이 여러 요인에 의해 영향을 받기 때문이다28.
여기에 설명된 방법의 재현성과 관련하여, TLC에 의한 RL 검출(단계 1.6)과 오르시놀 방법(단계 2.4)을 사용한 RL 측정 모두에서 유기상을 얻을 때 특별한 주의를 기울여야 합니다. 추출 중에 다른 단계를 소량 사용해도 얻은 결과에 상당한 변동이 발생할 수 있습니다. 배양 상층액에서 RL을 추출하는 것은 orcinol 분석법을 사용하여 신뢰할 수 있고 재현 가능한 결과를 달성하는 데 중요한 단계입니다. 유기상을 수집할 때 계면상과 액상을 제외하는 것이 중요합니다. 에테르 상이 3시간 이내에 완전히 건조되지 않으면 물이 포함되어 있을 수 있으므로 새로운 추출이 필요합니다.
RL 정량화를 위한 orcinol 분석법의 사용은 그 정확성과 특이성에 대한 논쟁과 함께 논쟁이 되어 왔습니다. RL은 종종 과대 평가된다고 주장되었으며, orcinol은 람노스뿐만 아니라 다른 설탕과 반응 할 수 있기 때문에이 방법은 구체적이지 않습니다. 현재 데이터는 RL이 배양 상층액에서 효율적으로 추출되는 경우, rhlA 돌연변이를 음성 대조군으로 사용할 때 반응이 없다는 것에서 알 수 있듯이 이 기술이 특이적임을 보여줍니다. 그러나, 추출 절차와 무관하게, 검출에 대한 방법의 정확성에 대한 추가적인 증거를 제공하기 위해, 앞서 설명한 바와 같이 UPLC-MS/MS에 의한 dRL 표준물질을 검출하고27, 수득한 결과를 오르시놀 방법(18 )과 비교하였다(보충 파일 1, 그림 4A). 보고된 18과 같이 이 방법을 사용하여 측정된 RL 농도 간에 매우 양호한 상관관계가 있음을 발견했으며, 여기서 검출된 dRL의 각 μM 농도는 421nm에서 동일한 람노스 μM 농도의 흡광도의 약 2배에 해당합니다(그림 4B, C). 이 대조 실험은 RL 정량화에 대한 이 방법의 정확성을 명확하게 보여줍니다.
그러나 orcinol 방법으로 mRL 및 dRL 혼합물의 농도를 측정하는 데 어려움이 있기 때문에 대부분의 간행물에서는 결과를 RL의 람노스 등가물로 보고합니다. 이 값은 여기에 제시된 예에서 볼 수 있듯이 실제 RL 농도와 직접적인 상관관계가 없을 수 있지만, 그럼에도 불구하고 특히 동일한 녹농균 균주를 사용할 때 서로 다른 조건에서 RL 양을 비교하는 유용한 수단으로 사용됩니다.
mRL 및 dRL 정량을 위한 대안적인 방법이 보고되었는데, 이는 볼펜29에 일반적으로 사용되는 소수성 염료 Victoria Pure Blue BO의 용해화를 기반으로 합니다. 이 방법은 빠르고 저렴합니다. 그러나 이 프로토콜은 P. putida 에 의한 RL의 이종 생성을 정량화하기 위해 개발되었으며 청색 독소 pyocyanin의 발현이 이 방법을 방해하기 때문에 P. aeruginosa에 의해 생성된 RL과 함께 사용할 수 없습니다.
요약하면, 여기에 설명된 방법은 녹농균에 의해 생성되는 RL의 검출 및 정량화를 위한 실행 가능한 대안을 제공하며, 정교하고 값비싼 장비가 필요하지 않습니다. 연구자가 다양한 실험 환경에서 RL 생성을 평가할 수 있는 실용적인 접근 방식을 제공합니다.
공개
저자는 공개할 이해 상충이 없습니다.
감사의 말
GSCh 실험실은 Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica (PAPIIT), Dirección General de Asuntos del Personal Académico -UNAM의 보조금 IN201222으로 부분적으로 지원됩니다.
자료
| Name | Company | Catalog Number | Comments |
| 1-NAPHTHOL | SIGMA-ALDRICH | 70442 | |
| ACETIC ACID | J.T. BAKER | 9508-02 | |
| CENTRIFUGE | For centrifuging tubes 1.5 mL and 50 mL | ||
| CHLOROFORM | J.T. BAKER | 9180-02 | |
| DRYING OVEN | |||
| ETHER | J.T. BAKER | 9244-02 | |
| GLASS PIPETTE | SIGMA-ALDRICH | CLS706510 | |
| HYDROCHLORIC ACID | J.T. BAKER | 5622-02 | |
| LB | |||
| L-RHAMNOSE MONOHYDRATE | SIGMA-ALDRICH | R-3875 | |
| METHANOL | J.T. BAKER | 9049-02 | |
| ORCINOL MONOHYDRATE | SIGMA-ALDRICH | O1875 | |
| PPGAS Broth | Tris HCL (0.12M), Potassium Chloride ( 0.02M) Ammonium Chloride (0.02M), Peptone (1%), pH 7.4 Autoclaved. Add Glucose (5%) and Magnesium Sulfate (0.0016M) | ||
| QUARTZ CELL (CUVETTE) | SIGMA-ALDRICH | Z276669 | |
| RECTANGULAR TLC DEVELOPING TANK | FISHER SCIENTIFIC | K4161801020 | |
| RHAMNOLIPIDS | SIGMA-ALDRICH | R-90 | |
| SPECTROPHOTOMETER | VIS | ||
| SPRAYER | SIGMA-ALDRICH | Z529710-1EA | |
| SULFURIC ACID | J.T. BAKER | 9681-02 | |
| TES TUBES 5mL | CORNING | 352002 | |
| TLC SILICA GEL 60 F254 | MERCK | 1.05554.0001 | |
| WATER BATH | > 80 °C |
참고문헌
- Moradali, M. F., Ghods, S., Rehm, B. H. A. Pseudomonas aeruginosa lifestyle: A paradigm for adaptation, survival, and persistence. Frontiers in Cell Infection and Microbiology. 7, 1-29 (2017).
- Gellatly, S. L., Hancock, R. E. W. Pseudomonas aeruginosa: New insights into pathogenesis and host defenses. Pathogens and Disease. 67 (3), 159-173 (2013).
- Williams, P., Cámara, M. Quorum sensing and environmental adaptation in Pseudomonas aeruginosa: a tale of regulatory networks and multifunctional signal molecules. Current Opinion in Microbiology. 12 (2), 182-191 (2009).
- Soberón-Chávez, G., González-Valdez, A., Soto-Aceves, M. P., Cocotl-Yañez, M. Rhamnolipids produced by Pseudomonas: From molecular genetics to the market. Microbial Biotechnology (MBT). 14 (1), 136-146 (2021).
- Freschi, L., et al. The Pseudomonas aeruginosa pan-genome provides new insights on its population structure, horizontal gene transfer, and pathogenicity. Genome Biology and Evolution. 11 (1), 109-120 (2019).
- Quiroz-Morales, S. E., García-Reyes, S., Ponce-Soto, G. Y., Servin-Gonzalez, L. Tracking the origins of Pseudomonas aeruginosa phylogroups by diversity and evolutionary analysis of important pathogenic marker genes. Diversity. 14 (5), 345 (2022).
- Déziel, E., et al. Liquid chromatography/mass spectrometry analysis of mixtures of rhamnolipids produced by Paeudomonas aeruginosa strain 57RP grown on mannitol or naphthalene. Biochemistry and Biophysic Acta. 1440 (2-3), 244-252 (1999).
- Abdel-Mawgoud, A. M., Lépine, F., Déziel, E. Rhamnolipids: Diversity of structures, microbial origins, and roles. Applied Microbiology and Biotechnology. 86 (5), 1323-1336 (2010).
- Toribio, J., Escalante, A. E., Soberón-Chávez, G. Production of rhamnolipids in bacteria other than Pseudomonas aeruginosa. European Journal of Lipid Science and Technology. 112, 1082-1087 (2010).
- Filbig, M., et al., Soberón-Chávez, G., et al. Metabolic and process engineering on the edge-Rhamnolipids are a true challenge: A review. Biosurfactants. Foundations and Frontiers in Enzymology. , 157-181 (2023).
- Noll, P., et al. Limits for sustainable biosurfactant production: Techno-economic and environmental assessment of a rhamnolipid production process. Bioresource Technology. 25, 101767 (2024).
- García-Reyes, S., Cocotl-Yañez, M., González-Valdez, A., Servín-González, L., Soberón Chávez, G. The PqsR-independent quorum-sensing response of Pseudomonas aeruginosa ATCC 9027 outlier-strain reveals new insights on the PqsE effect on RhlR activity. Molecular Microbiology. 116 (4), 1113-1123 (2021).
- Grosso-Becerra, M. V., et al. Pseudomonas aeruginosa ATCC 9027 is a non-virulent strain suitable for mono-rhamnolipids production. Applied Microbiology and Biotechnology. 100 (23), 9995-10004 (2016).
- Gutiérrez-Gómez, U., Soto-Aceves, M. P., Servín-González, L., Soberón-Chávez, G. Overproduction of rhamnolipids in Pseudomonas aeruginosa PA14 by redirection of the carbon flux from polyhydroxyalcanoate synthesis and overexpression of the rhlAB-R operon. Biotechnology Letters. 40 (11), 1561-1566 (2018).
- Zibek, S., Soberón-Chávez, G., Hausmann, R., Henkel, M. Overview on glycosylated lipids produced by bacteria and fungi: Rhamno-, Sophoro-, Mannosylerythritol and Cellobiose Lipids. Biosurfactants for a Biobased. Advances in Biochemical Engineering/Biotechnology. , 181 (2022).
- Twigg, M. S., et al. Microbial biosurfactant research: time to improve the rigour in the reporting of synthesis, functional characterization and process development. Microbial Biotechnology. 14 (1), 147-170 (2021).
- Matsuyama, T., Sogawa, M., Yano, I. Direct colony thin layer chromatography and rapid characterization of Serratia marscescens wetting agents. Applied and Environmental Microbiology. 53 (5), 1186-1188 (1987).
- Chandrasekaran, E. V., Bemiller, J. N. Constituent analyses of glycosaminoglycans. Methods on Carbohydrate Chemistry. 8, 89-96 (1980).
- Behrens, B., Engelen, J., Tiso, T., Blank, L. M., Hayen, H. Characterization of rhamnolipids by liquid chromatography/mass spectrometry after solid-phase extraction. Analytic and Bioanalytic Chemistry. 408 (10), 2505-2514 (2016).
- Rahim, R., et al. Cloning and functional characterization of the Pseudomonas aeruginosa rhlC gene that encodes rhamnosyltransferase 2, an enzyme responsible for di-rhamnolipid biosynthesis. Molecular Microbiology. 40 (3), 708-718 (2001).
- Irorere, V. U., Tripathi, L., Marchant, R., McClean, S., Banat, I. M. Microbial rhamnolipid production: A critical re-evaluation of published data and suggested future publication criteria. Applied Microbiology and Biotechnology. 101 (10), 3941-3951 (2017).
- Holloway, B. W. Genetic Recombination in Pseudomonas aeruginosa. Journal of General Microbiology. 13 (3), 572-581 (1955).
- Mathee, K. Forensic investigaction into the origin of Pseudomonas aeruginosa PA14-old but not lost. Journal of Medical Microbiology. 67 (8), 1019-1021 (2018).
- Roy, P. H., et al. Complete genome sequence of the multiresistant taxonomic outlier Pseudomonas aeruginosa PA7. PLoS ONE. 5, e8842 (2010).
- Zhang, Y., Miller, R. M. Enhanced octadecane dispersion and biodegradation by a Pseudomonas rhamnolipid surfactant (biosurfactant). Applied and Environmental Microbiology. 58 (10), 3276-3282 (1992).
- Miller, J. . Experiments in Molecular Genetics. , 352-355 (1992).
- Abdel-Mawgoud, A. M., Lépine, F., Déziel, E. Liquid chromatography/mass spectrometry for the identification and quantification of rhamnolipids. Pseudomonas Methods and Protocols. 30, 359-373 (2014).
- Lee, D. G., et al. Genomic analysis reveals that Pseudomonas aeruginosa virulence is combinatorial. Genome Biology. 7 (10), 90 (2006).
- Kubicki, S., et al. A straightforward assay for screening and quantification of biosurfactants in microbial culture supernatants. Frontiers in Bioengineering and Biotechnology. 8, 958 (2020).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유