Method Article
緑膿菌によって産生されるモノラムノリピッドおよびジラムノリピッドの検出と定量
要約
緑膿菌はラムノリピッドバイオサーファクタントを産生します。薄層クロマトグラフィーは、各菌株によって産生されるモノラムノ脂質とジラムノリピッドの割合を検出し、決定します。総ラムノリピドの定量には、培養上清から抽出されたこれらのバイオサーファクタントに存在するラムノース当量をオルシノール法を用いて評価することが含まれます。
要約
環境細菌 Pseudomonas aeruginosa は、健康被害を表す高い抗生物質耐性を持つ日和見病原体です。この細菌は、ラムノリピッド(RL)として知られるバイオサーファクタントを高レベルで生成しますが、これはバイオテクノロジー上の大きな価値を持つ分子ですが、病原性形質にも関連しています。この点で、RLの検出と定量は、バイオテクノロジーアプリケーションと生物医学研究プロジェクトの両方に役立つ可能性があります。本稿では、 緑膿菌 が産生する2種類の液性RLの産生を、単鎖脂質(mRL)という1つのラムノース部分に結合した脂肪酸(主にC10-C10)の二量体で構成する分子と、2つのラムノース結合体に結合した類似の脂肪酸二量体で構成する分子であるジラムノリピッド(dRL)の産生を、薄膜クロマトグラフィー(TLC)を用いて検出する手法を段階的に示します。さらに、 緑膿菌 培養上清から抽出したこれらのバイオサーファクタントの酸加水分解と、その後のオルシノールと反応するラムノースの濃度の検出に基づいて、RLの総量を測定する方法を紹介します。両方の手法の組み合わせを使用して、PAO1(系統群1)、PA14(系統群2)、およびPA7(系統群3)のタイプ株でここで例示されるように、特定の株によって産生されるmRLおよびdRLのおおよその濃度を推定することができます。
概要
緑膿菌は環境細菌であり、病原性に関連する形質の産生と高い抗生物質耐性により、非常に懸念される日和見病原体です1,2。この細菌によって産生される特徴的な二次代謝産物はバイオサーファクタントRLであり、これは酸化還元活性を持つ抗生物質であるフェナジンピオシアニンやプロテアーゼエラスターゼ3など、いくつかの病原性関連形質と協調して産生されます。RLの張力活性および乳化特性は、さまざまな産業用途で利用されており、現在商品化されています4。
ほとんどの緑膿菌株は、系統群1および2に属し、主に10個の炭素からなる脂肪酸二量体に結合した1つのラムノース部分からなるmRLと、最初のラムノース4に結合した追加のラムノース部分を含むdRLの2種類のRLを産生します(図1を参照)。しかし、緑膿菌の2つのマイナーな系統群(グループ3および5)は、mRL5,6のみを産生することが報告されています。2種類のRLには、前述のように主にC10-C10である脂肪酸二量体の混合物が含まれていますが、C12-C10、C12-C12、およびC10-C12:1二量体を含む分子の割合も小さくなります。HPLC MS/MSを用いた異なる株によって産生されるRL同族体の特性評価が報告されています7,8。この研究で説明されている方法は、mRLとdRLを区別することしかできず、RL同族体の特性評価には使用できません。
P. aeruginosaと一部のBurkholderia種はRL9の天然生産者ですが、前者の細菌が最も効率的な生産者です。しかし、市販されているRLは、現在、緑膿菌遺伝子を発現するPseudomonas putida KT2440誘導体で産生されており、この日和見病原体の使用を回避しています10,11。緑膿菌が産生するRLの検出と定量は、病原性関連形質の発現に関与する分子メカニズムの研究12、クレード3または5に属する株の特性評価13、およびこれらのバイオサーファクタントを過剰に産生し、病原性を低下させる緑膿菌の誘導体を構築するために非常に重要です14.異なる微生物によるバイオサーファクタントの産生は、これらの化合物のいくつかの一般的な特性、例えば、崩壊滴法や乳化指数15に基づいて検出されてきたが、これらの方法は正確でも特異でもない16。
ここでは、 緑膿菌タイプの異なる菌株の培養上清から全RLを液抽出し、TLCを用いて両タイプのRLを分離する方法を用いて、mRLとdRLを検出するプロトコールについて述べる。この方法では、培養上清から抽出されたRLは、TLCに使用される溶媒への溶解度によって分離され、シリカゲルプレート上での移動差を引き起こします。したがって、mRLはdRLよりも急速に移動し、プレートを乾燥させてα-ナフトールで染色すると、別々のスポットとして検出できます。
ここで説明するTLCによるmRLおよびdRLの検出方法は、以前に公開された記事17に基づいており、実行が容易で高価な機器を必要としません。この方法は、緑膿菌由来の変異体がRLを産生できないなど、適切な制御を用いて種々の緑膿菌分離株13中のRLを検出するのに有用である。しかし、緑膿菌以外の細菌が産生する新規バイオサーファクタントの特性評価には、特異性がないため好ましい方法ではありません。
さらに、緑膿菌培養上清から抽出された総RLのラムノース当量を定量化する方法が提示されます。この方法は、オルシノールと還元糖との反応に基づいてこれらのバイオサーファクタントを定量し、前述のように421nmで分光光度法で測定できる生成物をもたらす18。オルシノールとの反応はラムノースに特異的ではないため、リポ多糖類(LPS)などの他の糖含有分子をあまり含まない培養上清から抽出したRLを使用してこの方法を実行することが重要です。ここでは、RLの液体抽出18に酸性化クロロホルム/メタノール混合物が使用されているが、酢酸エチルも使用でき、固相抽出(SPE)は非常に良好な結果をもたらす19。ここで説明するオルシノール法は、高度な機器を必要とせず、分析されたサンプルの調製に特別な注意を払って行えば、信頼性の高い結果を得ることができます。適切なサンプル調製を確実にするためには、RL20を産生できないPseudomonas aeruginosa rhlA変異体を含め、各測定に対して3回の生物学的反復と3回の技術的反復を行うことが重要です。
文献16,21では、オルシノール法によるRL測定に関して大きな論争があり、一部の研究では、RL産生が過大評価されており、アッセイはラムノースに対する特異性を欠いており、他の糖を検出する可能性があることが示唆されています。ただし、ここでは、説明されている方法が適切な条件下で正確かつ特異的であることを示しています。さらに、本稿で概説した手順との比較のために、dRL標準のUPLC-MS/MS検出を利用し、オルシノール法でも同様の結果が得られることを実証します。この方法を使用してRLを定量するための詳細なプロトコルは、補足ファイル1に含まれています。
これらのプロトコルは、タイプ株PAO1(phylogroup 1)、PA14(phylogroup 2)、およびPA7(phylogroup 3)を用いて例示される。これらの株が選ばれたのは、特性が十分に評価され、異なるRLプロファイルを生成するためです。
プロトコル
この手順は 、補足図 1 に図示されています。本試験に使用した試薬および機器は、 資料表に記載されています。
1. TLCを用いた緑膿菌の培養上清中のmRLおよびdRLの検出
- 目的の 緑膿菌 株の遠心分離したブロスから始め、液体培地で24時間培養します(RLが産生される成長の固定段階に到達するため)。通常、これらの培養物には、1 mLあたり1 x 109 個の細菌が含まれています。
- 濃縮HClを使用して、培養物のpHを2に調整します。
- 50 mLのポリプロピレンチューブに5 mLの酸性化培養上清を入れ、5 mLの2:1クロロホルム:メタノール混合物を加えます。
- チューブを3回、毎回10秒間反転させて攪拌し、各攪拌の間に2分間反転せずにチューブを放置します。
- 2つの相が分離するまでチューブを約3時間攪拌せずに放置するか、チューブを3,000 x g 、4°Cで10分間遠心分離します。
- 有機相(最下層)をきれいなチューブに移し、乾燥するまでチューブを抽出フードに入れておきます。
- ステップ1.3からこのプロセスを繰り返し、2番目のクロロホルムの有機相であるメタノール抽出を、最初の抽出で使用したチューブに入れます。
- 有機相を1mLまたは1.5mLになるまで蒸発させます。この容量を1.5 mLの遠心分離チューブに移し、溶媒を一晩蒸発させて乾燥させます。
- 乾燥したチューブに50 μLのメタノールを加えて、RLを再懸濁します。
- シリカゲルプレート上で薄層クロマトグラフィーを実行します。
注:TLCプレートのサイズは、分析するサンプルの数に応じてカットする必要があります。各サンプルは1.5 cmで適用し、適用点はプレートの端から1 cmにする必要があります(鉛筆で水平線を引きます)。 - 10 μLのマイクロピペットを使用して、各サンプルに5 μLを塗布します。
- TLC液相は、クロロホルム:メタノール:酢酸の65:15:2混合物で構成されています。この混合物を35 mL調製するには、26 mLのクロロホルム、6 mLのメタノール、および0.8 mLの20%酢酸原液を混合します。これらの溶媒を混合し、クロマトグラフィーを開始する前に少なくとも10分間密閉TLCチャンバーに入れます。
- TLCプレートをチャンバー内に配置し、サンプルが適用されたポイントとの接触を避けます。
- チャンバーを閉じ、溶媒がプレートの端の1cm手前に達するまでTLCを放置します。この時点で、プレートを取り外して乾かします(プロセスを加速するために空気の流れを適用することができます)。
- この化合物の5gを33mLのエタノールに溶解することにより、α-ナフトールの溶液を調製します。溶解したら、127.5 mLのエタノール、12.6 mLの水、および20.5 mLの冷H2SO4を加えます。
- mRLおよびdRLの存在を検出するには、α-ナフトール溶液を抽出フード内のプレートにスプレーし、スプレーしたプレートを85°Cのオーブンに数分間置き、RLのピンクがかったマークが明らかになるまで待ちます。
- TLCの写真を使用して、各スポットの密度を検出するソフトウェアを使用して、各サンプルに存在するモノRLとジRLの割合を計算します。
2. バイオサーファクタント中に存在するラムノース当量を測定するRLの総量の定量化
- 1.2 mLの固定相培養物(24時間培養)を1.5 mLの遠心チューブに入れ、3,000 x g 、4°Cで3分間遠心分離します。 上清をきれいなチューブに集めます。
- 333 μLをきれいなチューブに移し(このステップを3回に分けて行います)、1 mLのエーテルを加えます。
- 渦の中で30秒間激しく混合します。この手順を一度に1本のチューブで繰り返します。
- 3,000 x g 、4°Cで2分間遠心分離します。 有機相(上相)を採取し、きれいなチューブに移し、乾燥するまで抽出フード内で開いたままにしておきます。
- ステップ 2.2 で説明したように、エーテルを使用して抽出を繰り返します。手順 2.3 と 2.4 を繰り返します。
- 完全に乾いたら、各チューブに1mLの水を加えます。チューブを12時間放置してRLが水和するのを待ってから、渦中で激しく攪拌します。
- きれいなフラスコを氷に5分間置きます(発熱反応であるため)。60%H2SO4 の溶液を調製します(フラスコを光から保護します)。
- きれいなチューブに、蒸留水に1.6%オルシノール溶液を調製します。オルシノール試薬を調製するには、60%H2SO4 溶液4.4 mLと1.6%オルシノール溶液0.6 mLを混合します。
- 必要なオルシノール試薬の最終容量を計算します。この試薬900 μLを、異なるラムノース濃度(通常、1 μg/mL から 9 μg/mL の範囲で 9 種類のラムノースを 9 種類使用)と 1 つの 1 種類を 100 μL の水を使用して、各サンプルおよび検量線の各濃度に加え、1 つを 100 μL の水で追加し、すべてが 3 回に実行されるようにします。
- ガラス管内のオルシノール試薬900 μLに各RLサンプル100 μLを加え、2つの溶液を混合します。
- 80°Cに予熱した水浴中で30分間インキュベートします。
- チューブを室温まで冷まします。
- 石英セルを使用して、421 nmでのサンプルの吸光度と検量線を読み取ります。
- 各サンプル中のラムノースの濃度は、検量線上の吸光度を補間し、測定に使用された体積を考慮して計算します。
- μM濃度を計算するには、μg/mLで求めた濃度を182.2(ラムノースの分子量)で割り、1000を掛けます。
結果
この記事では、緑膿菌の3種類の系統群を用いて、それぞれが異なるRL産生レベルとmRLとdRLの割合を持つ3つの系統群を表しました。これらの株には、PAO1(オーストラリアからの創傷分離株、1955年22月)、PA14(米国からの植物分離株、1977年23月)、およびPA7(アルゼンチンからの臨床分離株、2010年24月)が含まれる。ネガティブコントロールとして、RL産生が不可能なPAO1 rhlA変異体を採用しました。すべての菌株は、37°Cで高RLレベル25を促進するように特別に設計されたPPGAS培地で24時間培養しました。 PPGAS培養物は、LB培地26一晩培養物を用いて、600nmで0.05の光学密度で接種した。典型的には、緑膿菌株は、24時間後にPPGAS培地中で600nmで測定された光学密度2を達成し、これは1mLあたり1 x 109個の細菌にほぼ相当する。RLの検出と定量のために培養上清を回収するために、培養物を3,000 x g、4°Cで15分間遠心分離し、細胞ペレットを廃棄しました。これら2つの方法(図2および図3)を使用して得られた結果は、分析された3つの株間で、各菌株によって生成されるRLの種類とその生産量の両方が異なることを示しています。図2に示すように、PAO1は約30%のmRLと70%のdRLを生成し、PA14は50%のmRLと50%のdRLの等しい比率を生成し、PA7はmRLのみを生成します。このRLプロダクションプロファイルは、以前のレポート12,14と一致しています。
図3A は、これら3種類の株のそれぞれによって生成されるRLに存在するラムノース当量の量を示しています。検出されたラムノースの量から、PA14株が最も多くのRLを生成し、PA7株が最も少ない量を生成することが明らかです。ただし、これらの結果だけでは、各菌株によって生成されるRLのμM濃度を推定することはできません。さらに、オルシノール法だけでは、対象の各菌株によって産生されるmRLとdRLの割合を特徴づけることに依存しているため、おおよそのRL濃度を得ることはできません。したがって、RLのμM濃度の推定値を取得するには、TLCで決定された各タイプのRLの割合を考慮し、ラムノースの濃度が異なる標準曲線を含める必要があります(図3B)。
オルシノール法は、このバイオサーファクタントが1種類しか製造されていない場合のRLの濃度を定量するために実際に利用することができます。このような場合、検出されたラムノースのμM濃度は、mRLのμM濃度に直接対応します。dRLの場合、各dRL分子の加水分解によって2つのラムノースが生成されるため、RLで検出されたラムノースのμM濃度を2で割ってμM濃度を求める必要があります( 図4を参照)。
しかし、 緑膿菌 株の大部分は、( 図2に示すように)異なる割合で両方のタイプのRLを産生するため、総RLのおおよその濃度しか決定できません。これを考慮に入れて、各菌株によるRL産生を次のように推定しました:PA7株については、mRLのみを産生するため、RLのラムノース形成部分(44.6 μg/mL + 4.5 μg/mL)はμM濃度(244.78)に直接対応し、各RL分子が1つのラムノース部分を含んでいるため、培養上清中のこのバイオサーファクタントの濃度を表します。しかし、PAO1株については、検出されたラムノース濃度が111.55μg/mL+11.41μg/mLであったにもかかわらず、mRLに対応するのは30%に過ぎません。したがって、RL分子の70%は、分子ごとに2つのラムノースを含んでいます。RLの濃度を推定するために、1/5がmRL(122.49)のμM濃度に対応し、2/5(244.89)がdRLに対応することを考慮して、μMラムノース濃度(612.24)を5で割った。したがって、この株によって生成されるRLのおよそμM濃度は367.39です。
PA14株の場合、検出されたラムノース濃度は194.39μg/mL+11.5μg/mLであり、これをμM濃度(1,066.9)に変換して3で割った。この結果は、各 RL タイプが 50% で生成され、dRL が分子あたり 2 個のラムノースを含んでいることを考慮して、各 RL タイプの濃度を表します。したがって、この株は355.63μMのmRLと同濃度のdRLを生成し、その結果、約μMのRL濃度は711.27となり、これはPAO1株のほぼ2倍、PA7株のほぼ3倍の濃度になります。

図1:モノラムノリピッドとジラムノリピッドの主な同族体の化学組成(A)モノラムノリピッド。(B)ジラムノリピッド。この図の拡大版を表示するには、ここをクリックしてください。

図2:薄層クロマトグラフィーによるモノラムノリピッドおよびジラムノリピッドの検出 (A)RL標準を示すTLCプレートの写真、およびPAO1、PA14、PA7、およびPAO1由来のΔrhlA 変異体によって産生されるRL。(B)ImageJソフトウェアを使用して、(A)でテストした各サンプルの各RLタイプ(mRLおよびdRL)の割合を推定します。 この図の拡大版を表示するには、ここをクリックしてください。

図3:オルシノール法を用いたRL濃度の定量(A)PAO1(黒の棒)、PA14(薄い灰色の棒)、およびPA7(濃い灰色の棒)株の培養上清から抽出したRLに含まれるラムノース濃度(μg/mL)。バーは標準偏差を示します。(B)(A)に示す実験のラムノース検量線。この図の拡大版を表示するには、ここをクリックしてください。
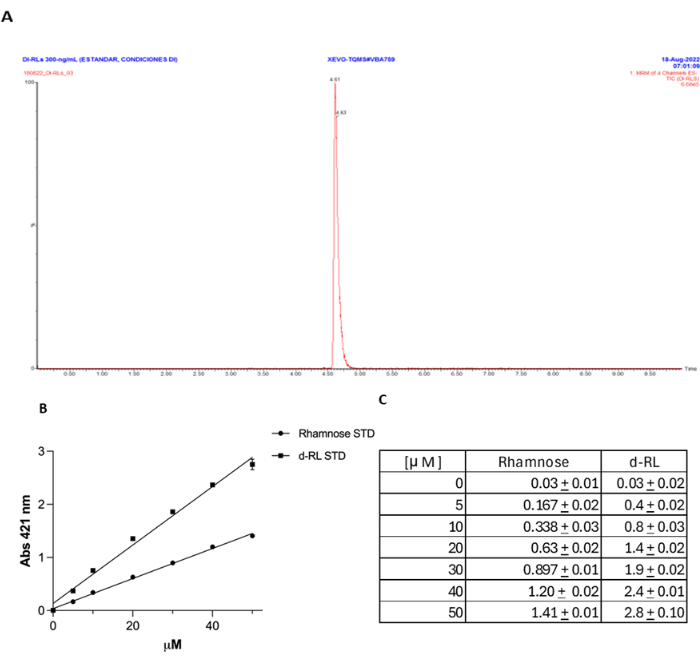
図4:dRL定量におけるオルシノール法とUPLC-MS/MSの比較によるバリデーション (A)dRL標準液はUPLC-MS/MSを用いて定量しました。(B)オルシノール法とラムノース濃度をmMで表した値との比較。(C)ラムノースと同じmM濃度のdRLは、予想通り421nmで約2倍の吸光度を示します。 この図の拡大版を表示するには、ここをクリックしてください。
補足図1:プロトコルの概略図。このファイルをダウンロードするには、ここをクリックしてください。
補足ファイル1:UPLC-MS/MSによるdRL定量のための詳細なプロトコル。このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
RLを検出および定量するための最も正確な方法は、質量分析(MS)と組み合わせたHPLCです7,8,27;ただし、多くの研究者がアクセスできない可能性のある専門的で高価な機器が必要です。ここで説明する方法は、基本的な実験室の材料や機器を使用してRL濃度を検出および推定するために日常的に実行できますが、いくつかの制限があり、特にmRLとdRLの混合物を決定する際の不正確さがあります。さらに、RL定量に使用するサンプルの調製は慎重に実行する必要があります。RLの検出と定量に使用する培養物は、細胞が凝集または溶解され、得られた結果にばらつきを引き起こす可能性のある不均一な細胞懸濁液が生じる可能性があるため、後期固定相にしないでください。さらに、細胞の破片がオルシノール法に干渉する可能性があります。
RLを産生できない緑膿菌株(この研究で使用されたPAO1 ΔrhlA変異体20など)など、記載されたプロトコールが適切なコントロールを用いて実施されると、正確な結果を得ることができる、図2。これらの結果は、異なる緑膿菌分離株12,13の特性評価、RL産生を増加させるための遺伝子改変株14、または様々な緑膿菌分離株12によるRL産生の分子遺伝学の研究に有用である。
したがって、ここで述べる方法は、特に調査の初期段階で、 緑膿菌 の病原性因子の産生を特徴付け、それらのバイオテクノロジーの可能性を評価するために利用することができる。これらの方法は、HPLC/MSのような技術の代替として意図されたものではなく、 緑膿菌 株の初期株特性評価および遺伝子操作のためのアクセス可能な技術として意図されていることに注意することが重要です。
さらに、研究の焦点が生物医学目的でのRL産生にある場合、RL産生は 緑膿菌によって生成されるいくつかの要因の1つにすぎないことを認識することが重要です。さらに、生成されるRLの種類またはその生産量は、病原性が複数の要因によって影響を受けるため、特定の株の病原性と厳密には相関していません28。
ここで説明した方法の再現性については、TLCによるRLの検出(ステップ1.6)とオルシノール法によるRLの測定(ステップ2.4)の両方で有機相を取得する際には特に注意が必要です。抽出中に摂取した他の相の少量でさえ、得られる結果に大きなばらつきをもたらす可能性があります。培養上清からのRLの抽出は、オルシノール法で信頼性と再現性のある結果を達成するための重要なステップです。有機相を採取する際には、間相と液相を除外することが重要です。エーテル相が3時間以内に完全に乾燥していない場合、エーテル相には水が含まれている可能性が高く、新しい抽出が必要になります。
RL定量にオルシノール法を使用することについては議論があり、その精度と特異性に対する議論があります。RLはしばしば過大評価されていると主張されており、オルシノールはラムノースだけでなくさまざまな糖と反応する可能性があるため、この方法は特異的ではありません。現在のデータは、RLが培養上清から効率的に抽出される場合、 rhlA 変異体をネガティブコントロールとして使用した場合の反応の欠如が示すように、この手法が特異的であることを示しています。しかし、この分析法の検出精度に関するさらなる証拠を提供するために、抽出手順とは無関係に、前述のように UPLC-MS/MS により dRL 標準試料を検出しました27、また、オルシノール法 18 で得られた結果を比較しました18 (補足ファイル 1、 図 4A)。報告された18として、この方法を使用して測定されたRLの濃度との間に非常に良好な相関関係があることがわかり、検出された各μM濃度のdRLは、同じラムノースμM濃度の421nmでの吸光度の約2倍に相当します(図4B、C)。この対照実験は、RL定量に対するこの方法の精度を明確に示しています。
しかし、オルシノール法でmRLとdRLの混合物の濃度を測定することは困難であるため、ほとんどの出版物では、結果をRLのラムノース当量として報告しています。この値は、ここで紹介した例に示されているように、実際のRL濃度と直接的な相関関係を持たない場合がありますが、それでも、特に同じ 緑膿菌 株を使用する場合に、異なる条件下でのRL量を比較する有用な手段として機能します。
mRLおよびdRL定量のための代替方法が報告されており、これはボールペン29で一般的に使用される疎水性染料Victoria Pure Blue BOの可溶化に基づいている。この方法は迅速かつ安価です。しかし、このプロトコルは 、P. putida によるRLの異種産生を定量化するために開発されたものであり、青毒素ピオシアニンの発現がこの方法を妨げるため、 P. aeruginosaによって産生されるRLと併用することは不可能である。
要約すると、ここで説明する方法は、 緑膿菌 によって産生されるRLの検出と定量のための実行可能な代替手段を提供し、高度で高価な機器を必要としません。これらは、研究者がさまざまな実験環境でRL産生を評価するための実践的なアプローチを提供します。
開示事項
著者には、開示すべき利益相反はありません。
謝辞
GSChの研究室は、Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica (PAPIIT), Dirección General de Asuntos del Personal Académico -UNAMからの助成金IN201222によって部分的にサポートされています。
資料
| Name | Company | Catalog Number | Comments |
| 1-NAPHTHOL | SIGMA-ALDRICH | 70442 | |
| ACETIC ACID | J.T. BAKER | 9508-02 | |
| CENTRIFUGE | For centrifuging tubes 1.5 mL and 50 mL | ||
| CHLOROFORM | J.T. BAKER | 9180-02 | |
| DRYING OVEN | |||
| ETHER | J.T. BAKER | 9244-02 | |
| GLASS PIPETTE | SIGMA-ALDRICH | CLS706510 | |
| HYDROCHLORIC ACID | J.T. BAKER | 5622-02 | |
| LB | |||
| L-RHAMNOSE MONOHYDRATE | SIGMA-ALDRICH | R-3875 | |
| METHANOL | J.T. BAKER | 9049-02 | |
| ORCINOL MONOHYDRATE | SIGMA-ALDRICH | O1875 | |
| PPGAS Broth | Tris HCL (0.12M), Potassium Chloride ( 0.02M) Ammonium Chloride (0.02M), Peptone (1%), pH 7.4 Autoclaved. Add Glucose (5%) and Magnesium Sulfate (0.0016M) | ||
| QUARTZ CELL (CUVETTE) | SIGMA-ALDRICH | Z276669 | |
| RECTANGULAR TLC DEVELOPING TANK | FISHER SCIENTIFIC | K4161801020 | |
| RHAMNOLIPIDS | SIGMA-ALDRICH | R-90 | |
| SPECTROPHOTOMETER | VIS | ||
| SPRAYER | SIGMA-ALDRICH | Z529710-1EA | |
| SULFURIC ACID | J.T. BAKER | 9681-02 | |
| TES TUBES 5mL | CORNING | 352002 | |
| TLC SILICA GEL 60 F254 | MERCK | 1.05554.0001 | |
| WATER BATH | > 80 °C |
参考文献
- Moradali, M. F., Ghods, S., Rehm, B. H. A. Pseudomonas aeruginosa lifestyle: A paradigm for adaptation, survival, and persistence. Frontiers in Cell Infection and Microbiology. 7, 1-29 (2017).
- Gellatly, S. L., Hancock, R. E. W. Pseudomonas aeruginosa: New insights into pathogenesis and host defenses. Pathogens and Disease. 67 (3), 159-173 (2013).
- Williams, P., Cámara, M. Quorum sensing and environmental adaptation in Pseudomonas aeruginosa: a tale of regulatory networks and multifunctional signal molecules. Current Opinion in Microbiology. 12 (2), 182-191 (2009).
- Soberón-Chávez, G., González-Valdez, A., Soto-Aceves, M. P., Cocotl-Yañez, M. Rhamnolipids produced by Pseudomonas: From molecular genetics to the market. Microbial Biotechnology (MBT). 14 (1), 136-146 (2021).
- Freschi, L., et al. The Pseudomonas aeruginosa pan-genome provides new insights on its population structure, horizontal gene transfer, and pathogenicity. Genome Biology and Evolution. 11 (1), 109-120 (2019).
- Quiroz-Morales, S. E., García-Reyes, S., Ponce-Soto, G. Y., Servin-Gonzalez, L. Tracking the origins of Pseudomonas aeruginosa phylogroups by diversity and evolutionary analysis of important pathogenic marker genes. Diversity. 14 (5), 345(2022).
- Déziel, E., et al. Liquid chromatography/mass spectrometry analysis of mixtures of rhamnolipids produced by Paeudomonas aeruginosa strain 57RP grown on mannitol or naphthalene. Biochemistry and Biophysic Acta. 1440 (2-3), 244-252 (1999).
- Abdel-Mawgoud, A. M., Lépine, F., Déziel, E. Rhamnolipids: Diversity of structures, microbial origins, and roles. Applied Microbiology and Biotechnology. 86 (5), 1323-1336 (2010).
- Toribio, J., Escalante, A. E., Soberón-Chávez, G. Production of rhamnolipids in bacteria other than Pseudomonas aeruginosa. European Journal of Lipid Science and Technology. 112, 1082-1087 (2010).
- Filbig, M., et al. Metabolic and process engineering on the edge-Rhamnolipids are a true challenge: A review. Biosurfactants. Foundations and Frontiers in Enzymology. Soberón-Chávez, G., et al. , Academic Press. New York. Chapter 8 157-181 (2023).
- Noll, P., et al. Limits for sustainable biosurfactant production: Techno-economic and environmental assessment of a rhamnolipid production process. Bioresource Technology. 25, 101767(2024).
- García-Reyes, S., Cocotl-Yañez, M., González-Valdez, A., Servín-González, L., Soberón Chávez, G. The PqsR-independent quorum-sensing response of Pseudomonas aeruginosa ATCC 9027 outlier-strain reveals new insights on the PqsE effect on RhlR activity. Molecular Microbiology. 116 (4), 1113-1123 (2021).
- Grosso-Becerra, M. V., et al. Pseudomonas aeruginosa ATCC 9027 is a non-virulent strain suitable for mono-rhamnolipids production. Applied Microbiology and Biotechnology. 100 (23), 9995-10004 (2016).
- Gutiérrez-Gómez, U., Soto-Aceves, M. P., Servín-González, L., Soberón-Chávez, G. Overproduction of rhamnolipids in Pseudomonas aeruginosa PA14 by redirection of the carbon flux from polyhydroxyalcanoate synthesis and overexpression of the rhlAB-R operon. Biotechnology Letters. 40 (11), 1561-1566 (2018).
- Zibek, S., Soberón-Chávez, G. Overview on glycosylated lipids produced by bacteria and fungi: Rhamno-, Sophoro-, Mannosylerythritol and Cellobiose Lipids. Biosurfactants for a Biobased. Advances in Biochemical Engineering/Biotechnology. Hausmann, R., Henkel, M. , Springer. Berlin, Heidelberg. Chapter 4 181(2022).
- Twigg, M. S., et al. Microbial biosurfactant research: time to improve the rigour in the reporting of synthesis, functional characterization and process development. Microbial Biotechnology. 14 (1), 147-170 (2021).
- Matsuyama, T., Sogawa, M., Yano, I. Direct colony thin layer chromatography and rapid characterization of Serratia marscescens wetting agents. Applied and Environmental Microbiology. 53 (5), 1186-1188 (1987).
- Chandrasekaran, E. V., Bemiller, J. N. Constituent analyses of glycosaminoglycans. Methods on Carbohydrate Chemistry. 8, 89-96 (1980).
- Behrens, B., Engelen, J., Tiso, T., Blank, L. M., Hayen, H. Characterization of rhamnolipids by liquid chromatography/mass spectrometry after solid-phase extraction. Analytic and Bioanalytic Chemistry. 408 (10), 2505-2514 (2016).
- Rahim, R., et al. Cloning and functional characterization of the Pseudomonas aeruginosa rhlC gene that encodes rhamnosyltransferase 2, an enzyme responsible for di-rhamnolipid biosynthesis. Molecular Microbiology. 40 (3), 708-718 (2001).
- Irorere, V. U., Tripathi, L., Marchant, R., McClean, S., Banat, I. M. Microbial rhamnolipid production: A critical re-evaluation of published data and suggested future publication criteria. Applied Microbiology and Biotechnology. 101 (10), 3941-3951 (2017).
- Holloway, B. W. Genetic Recombination in Pseudomonas aeruginosa. Journal of General Microbiology. 13 (3), 572-581 (1955).
- Mathee, K. Forensic investigaction into the origin of Pseudomonas aeruginosa PA14-old but not lost. Journal of Medical Microbiology. 67 (8), 1019-1021 (2018).
- Roy, P. H., et al. Complete genome sequence of the multiresistant taxonomic outlier Pseudomonas aeruginosa PA7. PLoS ONE. 5, e8842(2010).
- Zhang, Y., Miller, R. M. Enhanced octadecane dispersion and biodegradation by a Pseudomonas rhamnolipid surfactant (biosurfactant). Applied and Environmental Microbiology. 58 (10), 3276-3282 (1992).
- Miller, J. Experiments in Molecular Genetics. , Cold Spring Harbor Laboratory. New York. 352-355 (1992).
- Abdel-Mawgoud, A. M., Lépine, F., Déziel, E. Liquid chromatography/mass spectrometry for the identification and quantification of rhamnolipids. Pseudomonas Methods and Protocols. 30, 359-373 (2014).
- Lee, D. G., et al. Genomic analysis reveals that Pseudomonas aeruginosa virulence is combinatorial. Genome Biology. 7 (10), 90(2006).
- Kubicki, S., et al. A straightforward assay for screening and quantification of biosurfactants in microbial culture supernatants. Frontiers in Bioengineering and Biotechnology. 8, 958(2020).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved