Method Article
Nachweis und Quantifizierung von Mono-Rhamnolipiden und Di-Rhamnolipiden, die von Pseudomonas aeruginosa produziert werden
In diesem Artikel
Zusammenfassung
Pseudomonas aeruginosa produziert die Rhamnolipid-Biotenside. Die Dünnschichtchromatographie detektiert und bestimmt den Anteil der Mono- und Dirhamnolipide, die von jedem Stamm produziert werden. Die Quantifizierung der Gesamtrhamnolipide umfasst die Bewertung der Rhamnose-Äquivalente, die in diesen Biotensiden vorhanden sind, die mit Hilfe der Orcinol-Methode aus den Kulturüberständen extrahiert werden.
Zusammenfassung
Das Umweltbakterium Pseudomonas aeruginosa ist ein opportunistischer Erreger mit hoher Antibiotikaresistenz, der eine Gefahr für die Gesundheit darstellt. Dieses Bakterium produziert einen hohen Gehalt an Biotensiden, die als Rhamnolipide (RL) bekannt sind, bei denen es sich um Moleküle mit erheblichem biotechnologischem Wert handelt, die aber auch mit Virulenzmerkmalen in Verbindung gebracht werden. In dieser Hinsicht kann der Nachweis und die Quantifizierung von RL sowohl für biotechnologische Anwendungen als auch für biomedizinische Forschungsprojekte nützlich sein. In diesem Artikel demonstrieren wir Schritt für Schritt die Technik zum Nachweis der Produktion der beiden von P. aeruginosa produzierten RL-Formen mittels Dünnschichtchromatographie (DC): Mono-Rhamnolipide (mRL), Moleküle, die aus einem Dimer von Fettsäuren (hauptsächlich C10-C10) bestehen, das an eine Rhamnose-Einheit gebunden ist, und Di-Rhamnolipide (dRL), Moleküle, die aus einem ähnlichen Fettsäure-Dimer bestehen, das an zwei Rhamnose-Einheiten gebunden ist. Darüber hinaus stellen wir eine Methode zur Messung der Gesamtmenge an RL vor, die auf der sauren Hydrolyse dieser Biotenside basiert, die aus einem Überstand der P . aeruginosa-Kultur extrahiert wurden, und dem anschließenden Nachweis der Konzentration von Rhamnose, die mit Orcin reagiert. Die Kombination beider Techniken kann verwendet werden, um die ungefähre Konzentration von mRL und dRL abzuschätzen, die von einem bestimmten Stamm produziert wird, wie hier am Beispiel der Typstämme PAO1 (Phylogruppe 1), PA14 (Phylogruppe 2) und PA7 (Phylogruppe 3).
Einleitung
Pseudomonas aeruginosa ist ein Umweltbakterium und ein opportunistischer Krankheitserreger, der aufgrund seiner Produktion von Virulenz-assoziierten Merkmalen und seiner hohen Antibiotikaresistenz sehr besorgniserregendist 1,2. Ein charakteristischer Sekundärmetabolit, der von diesem Bakterium produziert wird, ist das Biotensid RL, das in koordinierter Weise mit mehreren Virulenz-assoziierten Merkmalen wie dem Phenazin Pyocyanin, einem Antibiotikum mit Redoxaktivität, und der Protease Elastase3 hergestellt wird. Die tensioaktiven und emulgierenden Eigenschaften von RL wurden in verschiedenen industriellen Anwendungen genutzt und werden derzeit kommerzialisiert4.
Die meisten P. aeruginosa-Stämme, die zu den Phylogruppen 1 und 2 gehören, produzieren zwei Arten von RL: mRL, das aus einer Rhamnose-Einheit besteht, die mit einem Fettsäuredimer verbunden ist, das hauptsächlich aus 10 Kohlenstoffatomen besteht, und dRL, das eine zusätzliche Rhamnose-Einheit enthält, die mit der ersten Rhamnose4 verbunden ist (siehe Abbildung 1). Es wurde jedoch berichtet, dass zwei kleinere P. aeruginosa-Phylogruppen (Gruppen 3 und 5) nur mRL 5,6 produzieren. Die beiden Arten von RL enthalten ein Gemisch von Fettsäuredimeren, die, wie erwähnt, hauptsächlich C10-C10 sind, aber es werden auch kleinere Anteile von Molekülen hergestellt, die C12-C10-, C12-C12- und C10-C12:1-Dimere enthalten. Die Charakterisierung der RL-Kongenere, die von verschiedenen Stämmen unter Verwendung von HPLC MS/MS produziert werden, wurde berichtet 7,8. Die in dieser Arbeit beschriebenen Methoden können nur zwischen mRL und dRL unterscheiden, können aber nicht für die Charakterisierung der RL-Kongenere verwendet werden.
P. aeruginosa und einige Burkholderia-Arten sind natürliche Produzenten von RL9, aber das erstgenannte Bakterium ist der effizienteste Produzent. Kommerziell genutztes RL wird jedoch derzeit in Pseudomonas putida KT2440-Derivaten hergestellt, die P. aeruginosa-Gene exprimieren, um die Verwendung dieses opportunistischen Erregers zu vermeiden10,11. Der Nachweis und die Quantifizierung von RL, die von P. aeruginosa produziert werden, sind von großer Bedeutung für die Untersuchung der molekularen Mechanismen, die an der Ausprägung von Virulenzmerkmalen beteiligt sind12, für die Charakterisierung von Stämmen, die zu den Kladen 3 oder 513 gehören, und für die Konstruktion von P. aeruginosa-Derivaten, die diese Biotenside überproduzieren und gleichzeitig eine verminderte Virulenz aufweisen14. Die Produktion von Biotensiden durch verschiedene Mikroorganismen wurde auf der Grundlage einiger allgemeiner Eigenschaften dieser Verbindungen, wie z. B. der Kollapstropfenmethode oder des Emulgierungsindex15, nachgewiesen, aber diese Methoden sind weder genau noch spezifisch16.
In dieser Arbeit beschreiben wir das Protokoll zum Nachweis von mRL und dRL durch die flüssige Extraktion von Gesamt-RL aus den Kulturüberständen verschiedener P. aeruginosa-Typ-Stämme und die Trennung beider RL-Typen mittels DC. Bei diesem Verfahren werden die aus dem Kulturüberstand extrahierten RL durch ihre unterschiedliche Löslichkeit in den für die DC-Behandlung verwendeten Lösungsmitteln getrennt, was zu einer differentiellen Migration auf der Kieselgelplatte führt. Daher haben mRL eine schnellere Migration als dRL und können als separate Flecken nachgewiesen werden, wenn die Platten getrocknet und mit α-Naphthol gefärbt werden.
Das hier beschriebene Verfahren zum Nachweis von mRL und dRL mittels TLC basiert auf einem bereits veröffentlichten Artikel17, der einfach durchzuführen ist und keine teure Ausrüstung erfordert. Diese Methode hat sich als nützlich für den Nachweis von RL in verschiedenen P. aeruginosa-Isolaten 13 unter Verwendung geeigneter Kontrollen erwiesen, wie z. B. einer von P. aeruginosa abgeleiteten Mutante, die nicht in der Lage ist, RL zu produzieren. Aufgrund ihrer mangelnden Spezifität ist sie jedoch nicht die bevorzugte Methode zur Charakterisierung neuartiger Biotenside, die von anderen Bakterien als Pseudomonas aeruginosa produziert werden.
Darüber hinaus wird eine Methode zur Quantifizierung der Rhamnose-Äquivalente der Gesamt-RL vorgestellt, die aus einem Überstand der P . aeruginosa-Kultur extrahiert wurde. Diese Methode quantifiziert diese Biotenside auf der Grundlage der Reaktion von Orcinol mit reduktiven Zuckern, was zu einem Produkt führt, das spektrophotometrisch bei 421 nm gemessen werden kann, wie zuvor beschrieben18. Da die Reaktion mit Orcinol nicht spezifisch für Rhamnose ist, ist es wichtig, diese Methode mit RL durchzuführen, das aus dem Kulturüberstand extrahiert wurde, der keine signifikanten Mengen anderer zuckerhaltiger Moleküle wie Lipopolysaccharide (LPS) enthält. Für die flüssige Extraktion von RL18 wird hier ein angesäuertes Chloroform/Methanol-Gemisch verwendet, aber auch Ethylacetat kann verwendet werden, und die Festphasenextraktion (SPE) liefert sehr gute Ergebnisse19. Die hier beschriebene Orcin-Methode erfordert keine ausgeklügelte Ausrüstung und kann zuverlässige Ergebnisse liefern, wenn sie, wie besprochen, bei der Vorbereitung der analysierten Proben mit besonderer Sorgfalt durchgeführt wird. Um eine ordnungsgemäße Probenvorbereitung zu gewährleisten, ist es wichtig, eine Pseudomonas aeruginosa rhlA-Mutante einzubeziehen, die nicht in der Lage ist, RL20 zu produzieren, und für jede Bestimmung drei biologische und drei technische Replikationen durchzuführen.
In der Literatur16,21 gab es erhebliche Kontroversen über die RL-Bestimmung mit der Orcin-Methode, wobei einige Studien darauf hindeuten, dass die RL-Produktion überschätzt wird und dass es dem Assay an Spezifität für Rhamnose mangelt, was möglicherweise zum Nachweis anderer Zucker führt. Wir zeigen hier jedoch, dass die beschriebenen Methoden unter den entsprechenden Bedingungen genau und spezifisch sein können. Darüber hinaus verwenden wir zum Vergleich mit den in diesem Artikel beschriebenen Verfahren die UPLC-MS/MS-Detektion eines dRL-Standards und zeigen, dass mit der Orcinol-Methode ähnliche Ergebnisse erzielt werden. Das detaillierte Protokoll zur Quantifizierung der RL mit dieser Methode ist in der Zusatzdatei 1 enthalten.
Diese Protokolle werden am Beispiel der Typstämme PAO1 (Phylogruppe 1), PA14 (Phylogruppe 2) und PA7 (Phylogruppe 3) veranschaulicht. Diese Stämme wurden ausgewählt, weil sie gut charakterisiert sind und unterschiedliche RL-Profile erzeugen.
Protokoll
Dieses Verfahren ist in der ergänzenden Abbildung 1 schematisiert. Die für die Studie verwendeten Reagenzien und Geräte sind in der Materialtabelle aufgeführt.
1. Nachweis von mRL und dRL in Kulturüberständen von P. aeruginosa mittels TLC
- Beginnen Sie mit der zentrifugierten Brühe des interessierenden Stammes P. aeruginosa , die 24 Stunden lang in flüssigem Medium kultiviert wird (um die stationäre Wachstumsphase zu erreichen, in der RL produziert wird). Typischerweise enthalten diese Kulturen 1 x 109 Bakterien pro ml.
- Stellen Sie den pH-Wert der Kultur mit konzentriertem HCl auf 2 ein.
- 5 ml des angesäuerten Kulturüberstands werden in ein 50-ml-Polypropylenröhrchen gegeben und 5 ml eines Chloroform-Methanol-Gemisches von 2:1 hinzugefügt.
- Rühren Sie das Rohr dreimal durch Inversion, jedes Mal für 10 s, und lassen Sie das Rohr zwischen jedem Rühren 2 Minuten lang ohne Inversion.
- Lassen Sie das Röhrchen ca. 3 h lang ohne Rühren, bis die beiden Phasen getrennt sind, oder zentrifugieren Sie das Röhrchen 10 min lang bei 3.000 x g bei 4 °C.
- Übertragen Sie die organische Phase (untere Schicht) in ein sauberes Rohr und lassen Sie das Rohr in einer Extraktionshaube, bis es zu Trockenheit kommt.
- Wiederholen Sie den Vorgang ab Schritt 1.3 und geben Sie die organische Phase aus der zweiten Chloroform-Extraktion: Methanolextraktion in das Röhrchen, das bei der ersten Extraktion verwendet wurde.
- Verdampfen Sie die organische Phase, bis 1 mL oder 1,5 mL übrig sind. Übertragen Sie dieses Volumen in ein 1,5-ml-Zentrifugenröhrchen und verdampfen Sie das Lösungsmittel über Nacht, bis es trocken ist.
- Geben Sie 50 μl Methanol in das getrocknete Rohr, um RL zu resuspendieren.
- Führen Sie eine Dünnschichtchromatographie auf Kieselgelplatten durch.
HINWEIS: Die Größe der DC-Platte sollte entsprechend der Anzahl der zu analysierenden Proben zugeschnitten werden. Jede Probe sollte in einem Abstand von 1,5 cm aufgetragen werden, und der Applikationspunkt sollte 1 cm vom Rand der Platte entfernt sein (zeichnen Sie mit einem Bleistift eine horizontale Linie). - Geben Sie 5 μl jeder Probe mit einer 10-μl-Mikropipette.
- Die flüssige DC-Phase besteht aus einem 65:15:2 Gemisch aus Chloroform: Methanol: Essigsäure. Um 35 ml dieser Mischung herzustellen, mischen Sie 26 ml Chloroform, 6 ml Methanol und 0,8 ml einer 20%igen Stammlösung Essigsäure. Mischen Sie diese Lösungsmittel und legen Sie sie mindestens 10 Minuten lang in die geschlossene DC-Kammer, bevor Sie mit der Chromatographie beginnen, damit die Atmosphäre mit den flüchtigen Lösungsmitteln gesättigt ist.
- Setzen Sie die DC-Platte in die Kammer ein und vermeiden Sie den Kontakt mit der Stelle, an der die Proben aufgetragen wurden.
- Schließen Sie die Kammer und lassen Sie die DC, bis das Lösungsmittel 1 cm vor dem Rand der Platte angelangt ist. Nehmen Sie an dieser Stelle die Platte heraus und lassen Sie sie trocknen (ein Luftstrom kann angewendet werden, um den Vorgang zu beschleunigen).
- Bereiten Sie eine Lösung von α-Naphthol her, indem Sie 5 g dieser Verbindung in 33 ml Ethanol auflösen. Nach dem Auflösen 127,5 ml Ethanol, 12,6 ml Wasser und 20,5 ml kaltes H2SO4 hinzufügen.
- Um das Vorhandensein von mRL und dRL nachzuweisen, sprühen Sie die α-Naphthhol-Lösung auf die Platte in der Extraktionshaube und stellen Sie die besprühte Platte einige Minuten lang bei 85 °C in einen Ofen, bis die rosafarbene Markierung von RL sichtbar ist.
- Verwenden Sie ein Bild der TLC, um den Anteil an Mono- und di-RL in jeder Probe zu berechnen, indem Sie eine Software verwenden, die die Dichte jedes Spots erkennt.
2. Quantifizierung der Gesamtmenge an RL unter Messung der im Biotensid enthaltenen Rhamnoseäquivalente
- 1,2 mL einer stationären Phasenkultur (24 h lang gezüchtet) in ein 1,5 mL Zentrifugenröhrchen geben und 3 min bei 3.000 x g bei 4 °C zentrifugieren. Sammeln Sie den Überstand in einem sauberen Rohr.
- Übertragen Sie 333 μL in ein sauberes Röhrchen (führen Sie diesen Schritt in dreifacher Ausfertigung durch) und fügen Sie 1 mL Ether hinzu.
- 30 s kräftig in einem Wirbel einrühren. Wiederholen Sie diesen Vorgang mit einem Schlauch nach dem anderen.
- 2 min bei 3.000 x g bei 4 °C zentrifugieren. Sammeln Sie die organische Phase (obere Phase), geben Sie sie in ein sauberes Rohr und lassen Sie sie in der Absaughaube offen, bis die Trockenheit eintritt.
- Wiederholen Sie die Extraktion mit Äther wie in Schritt 2.2 beschrieben. Wiederholen Sie die Schritte 2.3 und 2.4.
- Sobald es vollständig getrocknet ist, geben Sie 1 ml Wasser in jedes Röhrchen. Lassen Sie die Röhren 12 Stunden lang stehen, damit das RL hydratisieren kann, und rühren Sie dann kräftig im Wirbel.
- Legen Sie einen sauberen Kolben für 5 Minuten in Eis (da es sich um eine exotherme Reaktion handelt). Eine Lösung von 60 % H2SO4 herstellen (Kolben vor Licht schützen).
- Bereiten Sie in einem sauberen Röhrchen eine 1,6%ige Orcinlösung in destilliertem Wasser vor. Zur Herstellung des Orcin-Reagenzes mischen Sie 4,4 ml der 60%igen H2SO4-Lösung mit 0,6 ml der 1,6 % igen Orcin-Lösung.
- Berechnen Sie das endgültige Volumen des benötigten Orcin-Reagenzes. Geben Sie 900 μl dieses Reagenzes zu jeder Probe und zu jeder Konzentration der Kalibrierkurve unter Verwendung unterschiedlicher Rhamnose-Konzentrationen (typischerweise werden 9 Konzentrationen von Rhamnose im Bereich von 1 μg/ml bis 9 μg/ml verwendet) und einer mit 100 μl Wasser, wobei sichergestellt wird, dass alle Konzentrationen in dreifacher Ausführung durchgeführt werden.
- Geben Sie 100 μl jeder RL-Probe zu 900 μl des Orcin-Reagenzes in ein Glasröhrchen und mischen Sie die beiden Lösungen.
- 30 min in einem auf 80 °C vorgeheizten Wasserbad inkubieren.
- Lassen Sie die Röhren auf Raumtemperatur abkühlen.
- Lesen Sie die Extinktion der Proben und die Kalibrierkurve bei 421 nm mit einer Quarzzelle ab.
- Die Rhamnosekonzentration in jeder Probe wird berechnet, indem die Extinktion auf der Kalibrierkurve interpoliert und das für die Bestimmung verwendete Volumen berücksichtigt wird.
- Um die μM-Konzentration zu berechnen, dividieren Sie die in μg/ml erhaltene Konzentration durch 182,2 (das Molekulargewicht von Rhamnose) und multiplizieren Sie sie mit 1000.
Ergebnisse
In diesem Artikel wurden drei verschiedene Stämme vom Typ P. aeruginosa verwendet, um drei Phylogruppen zu repräsentieren, jede mit unterschiedlichen RL-Produktionsniveaus und Anteilen von mRL und dRL. Zu diesen Stämmen gehören PAO1 (ein Wundisolat aus Australien, 195522), PA14 (ein Pflanzenisolat aus den USA, 197723) und PA7 (ein klinisches Isolat aus Argentinien, 201024). Als Negativkontrolle wurde die PAO1 rhlA-Mutante verwendet, die nicht in der Lage ist, RL zu produzieren. Alle Stämme wurden 24 Stunden lang in PPGAS-Medium kultiviert, das speziell entwickelt wurde, um hohe RL-Werte25 bei 37 °C zu fördern. Die PPGAS-Kulturen wurden mit einer optischen Dichte von 0,05 bei 600 nm unter Verwendung einer LB-Medium26 Übernachtkultur inokuliert. Typischerweise erreichen P. aeruginosa-Stämme nach 24 h eine optische Dichte von 2, gemessen bei 600 nm in PPGAS-Medium, was in etwa 1 x 109 Bakterien pro mL entspricht. Um die Kulturüberstände für den RL-Nachweis und die Quantifizierung zu sammeln, wurden die Kulturen 15 Minuten lang bei 3.000 x g bei 4 °C zentrifugiert und das Zellpellet verworfen. Die mit diesen beiden Methoden erzielten Ergebnisse (Abbildung 2 und Abbildung 3) zeigen, dass sich sowohl die Arten von RL, die von jedem Stamm produziert werden, als auch die Menge ihrer Produktion zwischen den drei analysierten Stämmen unterscheiden. Wie in Abbildung 2 dargestellt, erzeugt PAO1 etwa 30 % mRL und 70 % dRL, während PA14 ein gleiches Verhältnis von 50 % mRL und 50 % dRL erzeugt und PA7 ausschließlich mRL erzeugt. Dieses RL-Produktionsprofil stimmt mit früheren Berichtenüberein 12,14.
Abbildung 3A zeigt die Menge an Rhamnose-Äquivalenten, die in RL vorhanden sind und von jedem dieser drei Arten von Stämmen produziert werden. Aus den nachgewiesenen Rhamnose-Mengen geht hervor, dass der Stamm PA14 die höchste Menge an RL produziert, während der Stamm PA7 die geringste Menge produziert. Diese Ergebnisse allein können jedoch nicht verwendet werden, um die μM-Konzentration von RL abzuschätzen, die von jedem Stamm produziert wird. Darüber hinaus kann die Orcin-Methode allein keine ungefähre RL-Konzentration liefern, da sie auf der Charakterisierung der Anteile von mRL und dRL beruht, die von jedem interessierenden Stamm produziert werden. Um eine Abschätzung der μM-Konzentration von RL zu erhalten, muss daher der Anteil jeder Art von RL berücksichtigt werden, der in der TLC bestimmt wurde, und es sollte eine Standardkurve mit unterschiedlichen Rhamnose-Konzentrationen einbezogen werden (Abbildung 3B).
Die Orcin-Methode kann in der Tat verwendet werden, um die Konzentration von RL zu quantifizieren, wenn nur eine Art dieses Biotensids hergestellt wird. In solchen Fällen entspricht die μM-Konzentration der detektierten Rhamnose direkt der μM-Konzentration von mRL. Da bei dRL zwei Rhamnosen durch die Hydrolyse jedes dRL-Moleküls erzeugt werden, muss die μM-Konzentration der in RL nachgewiesenen Rhamnose durch 2 geteilt werden, um ihre μM-Konzentration zu erhalten (siehe Abbildung 4).
Die Mehrzahl der P. aeruginosa-Stämme produziert jedoch beide Arten von RL in unterschiedlichen Anteilen (wie in Abbildung 2 gezeigt), so dass nur eine ungefähre Konzentration der Gesamt-RL bestimmt werden kann. Unter Berücksichtigung dessen schätzten wir die RL-Produktion durch jeden Stamm wie folgt: Da der PA7-Stamm nur mRL produziert, entspricht die Rhamnose, die Teil der RL ist (44,6 μg/mL + 4,5 μg/ml), direkt der μM-Konzentration (244,78), was der Konzentration dieses Biotensids im Kulturüberstand entspricht, da jedes RL-Molekül eine Rhamnose-Einheit enthält. Für den PAO1-Stamm betrug die nachgewiesene Rhamnose-Konzentration zwar 111,55 μg/ml + 11,41 μg/ml, aber nur 30 % entsprechen mRL. Daher enthalten 70% der RL-Moleküle zwei Rhamnosen pro Molekül. Zur Schätzung der RL Konzentration wurde die μM Rhamnose Konzentration (612,24) durch 5 geteilt, wobei 1/5 der μM Konzentration von mRL (122,49) und 2/5 (244,89) dRL entspricht. Somit beträgt die ungefähre μM-Konzentration von RL, die von diesem Stamm erzeugt wird, 367,39.
Für den PA14-Stamm betrug die nachgewiesene Rhamnose-Konzentration 194,39 μg/ml + 11,5 μg/ml, die in die μM-Konzentration (1.066,9) umgerechnet und durch 3 geteilt wurde. Das Ergebnis stellt die Konzentration jedes RL-Typs dar, wobei man bedenkt, dass jeder Typ zu 50 % hergestellt wurde und dass dRL 2 Rhamnosen pro Molekül enthält. Somit produziert dieser Stamm 355,63 μM mRL und die gleiche Konzentration an dRL, was zu einer ungefähren μM-Konzentration von RL von 711,27 führt, fast doppelt so viel wie der Stamm PAO1 und fast das Dreifache der Konzentration, die vom PA7-Stamm erzeugt wird.

Abbildung 1: Chemische Zusammensetzung der Hauptkongenere von Mono-Rhamnolipiden und Di-Rhamnolipiden. (A) Mono-Rhamnolipide. (B) Di-Rhamnolipide. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 2: Nachweis von Mono-Rhamnolipiden und Di-Rhamnolipiden mittels Dünnschichtchromatographie. (A) Bild einer DC-Platte mit RL-Standards und der von den Stämmen PAO1, PA14, PA7 und der PAO1-abgeleiteten ΔrhlA-Mutante produzierten RL. (B) Schätzung des Anteils jedes RL-Typs (mRL und dRL) in jeder der unter Buchstabe A geprüften Proben unter Verwendung der Software ImageJ. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Quantifizierung der RL-Konzentration mit der Orcin-Methode. (A) Rhamnose-Konzentration (μg/ml) in RL, extrahiert aus den Kulturüberständen der Stämme PAO1 (schwarzer Balken), PA14 (hellgrauer Balken) und PA7 (dunkelgrauer Balken). Die Balken bezeichnen die Standardabweichung. (B) Die Rhamnose-Kalibrierungskurve für das in (A) gezeigte Experiment. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
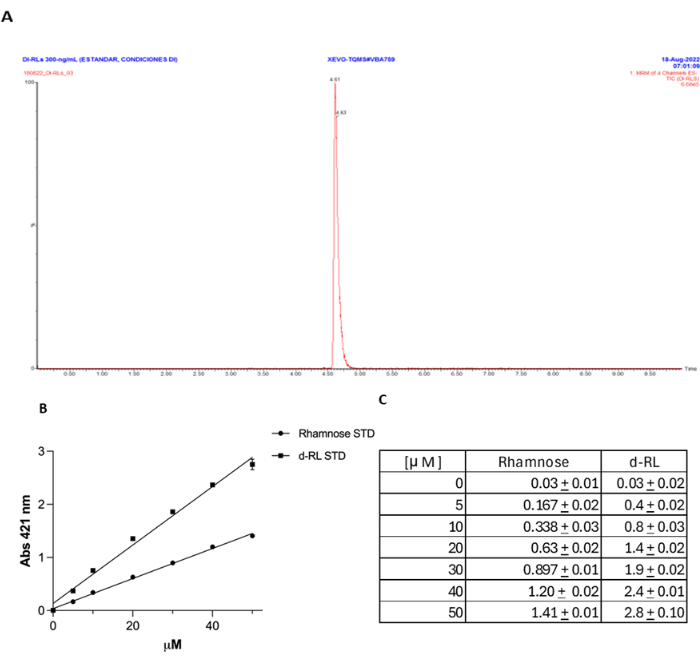
Abbildung 4: Validierung der Orcinol-Methode im Vergleich zu UPLC-MS/MS für die dRL-Quantifizierung. (A) Der dRL-Standard wurde mit UPLC-MS/MS quantifiziert. (B) Die Orcin-Methode im Vergleich zur Rhamnose-Konzentration, ausgedrückt in mM. (C) Die gleiche mM-Konzentration von dRL wie bei Rhamnose ergibt bei 421 nm erwartungsgemäß etwa die doppelte Absorption. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1: Schematische Darstellung des Protokolls. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 1: Detailprotokoll zur Quantifizierung von dRL durch UPLC-MS/MS. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die genaueste Methode zum Nachweis und zur Quantifizierung von RL ist die HPLC gekoppelt mit Massenspektrometrie (MS)7,8,27; Es erfordert jedoch spezielle und teure Geräte, die für viele Forscher möglicherweise nicht zugänglich sind. Die hier beschriebenen Methoden können routinemäßig mit grundlegenden Labormaterialien und -geräten durchgeführt werden, um RL-Konzentrationen zu ermitteln und abzuschätzen, aber sie weisen einige Einschränkungen auf, insbesondere ihre Ungenauigkeit bei der Bestimmung von mRL- und dRL-Gemischen. Darüber hinaus sollte die Vorbereitung der Probe, die für die RL-Quantifizierung verwendet wird, sorgfältig durchgeführt werden. Kulturen, die für den RL-Nachweis und die Quantifizierung verwendet werden, sollten sich nicht in der späten stationären Phase befinden, da Zellen aggregiert oder lysiert werden können, was zu inhomogenen Zellsuspensionen führt, die zu Schwankungen in den erzielten Ergebnissen führen können. Darüber hinaus können Zelltrümmer die Orcinol-Methode beeinträchtigen.
Genaue Ergebnisse können erzielt werden, wenn das beschriebene Protokoll mit geeigneten Kontrollen durchgeführt wird, wie z.B. einem Pseudomonas aeruginosa-Stamm, der nicht in der Lage ist, RL zu produzieren (wie die in dieser Arbeit verwendete PAO1 ΔrhlA-Mutante 20, Abbildung 2). Diese Ergebnisse sind nützlich für die Charakterisierung verschiedener P. aeruginosa-Isolate 12,13, die genetische Modifikation von Stämmen zur Steigerung der RL-Produktion14 oder die Untersuchung der molekularen Genetik der RL-Produktion durch verschiedene P. aeruginosa-Isolate 12.
Somit können die hier beschriebenen Methoden genutzt werden, um Pseudomonas aeruginosa-Stämme hinsichtlich ihrer Produktion von Virulenzfaktoren zu charakterisieren und ihr biotechnologisches Potenzial zu bewerten, insbesondere in den frühen Stadien der Untersuchung. Es ist wichtig zu beachten, dass diese Methoden nicht als Ersatz für Techniken wie HPLC/MS gedacht sind, sondern als zugängliche Techniken für die anfängliche Charakterisierung von Stämmen und die genetische Manipulation von P. aeruginosa-Stämmen .
Wenn der Forschungsschwerpunkt auf der RL-Produktion für biomedizinische Zwecke liegt, ist es außerdem wichtig zu erkennen, dass die RL-Produktion nur einer von mehreren Faktoren ist, die von Pseudomonas aeruginosa produziert werden. Darüber hinaus korreliert die Art der produzierten RL oder die Menge ihrer Produktion nicht streng mit der Virulenz eines bestimmten Stammes, da die Virulenz von mehreren Faktoren beeinflusst wird28.
Hinsichtlich der Reproduzierbarkeit der hier beschriebenen Methoden sollte bei der Gewinnung der organischen Phasen sowohl beim Nachweis von RL durch TLC (Schritt 1.6) als auch bei der Messung von RL mit der Orcin-Methode (Schritt 2.4) besondere Aufmerksamkeit gewidmet werden. Selbst eine kleine Menge der anderen Phase, die während der Extraktion eingenommen wird, kann zu erheblichen Schwankungen der erzielten Ergebnisse führen. Die Extraktion von RL aus dem Kulturüberstand ist ein entscheidender Schritt, um mit der Orcin-Methode zuverlässige und reproduzierbare Ergebnisse zu erzielen. Es ist entscheidend, dass bei der Sammlung der organischen Phase die Zwischenphase und die flüssige Phase ausgeschlossen werden. Wenn eine Ätherphase nicht innerhalb von 3 h vollständig getrocknet ist, enthält sie wahrscheinlich Wasser, was eine erneute Extraktion erforderlich macht.
Die Verwendung der Orcin-Methode zur RL-Quantifizierung wurde diskutiert, wobei Argumente gegen ihre Genauigkeit und Spezifität diskutiert wurden. Es wurde behauptet, dass RL oft überschätzt werden, und die Methode ist nicht spezifisch, da Orcinol mit verschiedenen Zuckern reagieren kann, nicht nur mit Rhamnose. Die aktuellen Daten zeigen, dass diese Technik spezifisch ist, wenn RL effizient aus dem Kulturüberstand extrahiert werden, wie das Fehlen einer Reaktion zeigt, wenn die rhlA-Mutante als Negativkontrolle verwendet wird. Um jedoch einen zusätzlichen Nachweis für die Detektionsgenauigkeit der Methode unabhängig vom Extraktionsverfahren zu erbringen, detektierten wir einen dRL-Standard mittels UPLC-MS/MS, wie zuvor beschrieben27, und verglichen die Ergebnisse mit der Orcinol-Methode18 (Ergänzungsdatei 1, Abbildung 4A). Wir fanden, wie berichtet18, eine sehr gute Korrelation zwischen der mit dieser Methode gemessenen RL-Konzentration, wobei jede detektierte μM-Konzentration von dRL etwa der doppelten Extinktion bei 421 nm entspricht wie die gleiche Rhamnose-μM-Konzentration (Abbildung 4B,C). Dieses Kontrollexperiment zeigt deutlich die Genauigkeit dieser Methode zur RL-Quantifizierung.
Aufgrund der Herausforderung, die Konzentration von mRL und dRL-Gemischen mit der Orcin-Methode zu messen, werden die Ergebnisse in den meisten Publikationen jedoch als Rhamnose-Äquivalente in RL angegeben. Dieser Wert hat möglicherweise keine direkte Korrelation mit der tatsächlichen RL-Konzentration, wie in den hier vorgestellten Beispielen dargestellt, aber er dient dennoch als nützliches Mittel zum Vergleich von RL-Mengen unter verschiedenen Bedingungen, insbesondere bei Verwendung desselben P. aeruginosa-Stammes .
Es wurde über eine alternative Methode zur mRL- und dRL-Quantifizierung berichtet, die auf der Solubilisierung des hydrophoben Farbstoffs Victoria Pure Blue BO basiert, der üblicherweise in Kugelschreibern verwendetwird 29. Diese Methode ist schnell und kostengünstig. Dieses Protokoll wurde jedoch entwickelt, um die heterologe Produktion von RL durch P. putida zu quantifizieren, und ist nicht für die Verwendung mit RL von P. aeruginosa geeignet, da die Expression des blauen Toxins Pyocyanin diese Methode stört.
Zusammenfassend lässt sich sagen, dass die hier beschriebenen Methoden eine praktikable Alternative für den Nachweis und die Quantifizierung von RL aus P . aeruginosa bieten, die keine aufwändige und teure Ausrüstung erfordert. Sie bieten Forschern einen praktischen Ansatz, um die RL-Produktion in einer Vielzahl von experimentellen Umgebungen zu bewerten.
Offenlegungen
Die Autoren haben keinen Interessenkonflikt offenzulegen.
Danksagungen
Das Labor des GSCh wird teilweise durch Zuschüsse IN201222 des Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica (PAPIIT), der Dirección General de Asuntos del Personal Académico -UNAM unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 1-NAPHTHOL | SIGMA-ALDRICH | 70442 | |
| ACETIC ACID | J.T. BAKER | 9508-02 | |
| CENTRIFUGE | For centrifuging tubes 1.5 mL and 50 mL | ||
| CHLOROFORM | J.T. BAKER | 9180-02 | |
| DRYING OVEN | |||
| ETHER | J.T. BAKER | 9244-02 | |
| GLASS PIPETTE | SIGMA-ALDRICH | CLS706510 | |
| HYDROCHLORIC ACID | J.T. BAKER | 5622-02 | |
| LB | |||
| L-RHAMNOSE MONOHYDRATE | SIGMA-ALDRICH | R-3875 | |
| METHANOL | J.T. BAKER | 9049-02 | |
| ORCINOL MONOHYDRATE | SIGMA-ALDRICH | O1875 | |
| PPGAS Broth | Tris HCL (0.12M), Potassium Chloride ( 0.02M) Ammonium Chloride (0.02M), Peptone (1%), pH 7.4 Autoclaved. Add Glucose (5%) and Magnesium Sulfate (0.0016M) | ||
| QUARTZ CELL (CUVETTE) | SIGMA-ALDRICH | Z276669 | |
| RECTANGULAR TLC DEVELOPING TANK | FISHER SCIENTIFIC | K4161801020 | |
| RHAMNOLIPIDS | SIGMA-ALDRICH | R-90 | |
| SPECTROPHOTOMETER | VIS | ||
| SPRAYER | SIGMA-ALDRICH | Z529710-1EA | |
| SULFURIC ACID | J.T. BAKER | 9681-02 | |
| TES TUBES 5mL | CORNING | 352002 | |
| TLC SILICA GEL 60 F254 | MERCK | 1.05554.0001 | |
| WATER BATH | > 80 °C |
Referenzen
- Moradali, M. F., Ghods, S., Rehm, B. H. A. Pseudomonas aeruginosa lifestyle: A paradigm for adaptation, survival, and persistence. Frontiers in Cell Infection and Microbiology. 7, 1-29 (2017).
- Gellatly, S. L., Hancock, R. E. W. Pseudomonas aeruginosa: New insights into pathogenesis and host defenses. Pathogens and Disease. 67 (3), 159-173 (2013).
- Williams, P., Cámara, M. Quorum sensing and environmental adaptation in Pseudomonas aeruginosa: a tale of regulatory networks and multifunctional signal molecules. Current Opinion in Microbiology. 12 (2), 182-191 (2009).
- Soberón-Chávez, G., González-Valdez, A., Soto-Aceves, M. P., Cocotl-Yañez, M. Rhamnolipids produced by Pseudomonas: From molecular genetics to the market. Microbial Biotechnology (MBT). 14 (1), 136-146 (2021).
- Freschi, L., et al. The Pseudomonas aeruginosa pan-genome provides new insights on its population structure, horizontal gene transfer, and pathogenicity. Genome Biology and Evolution. 11 (1), 109-120 (2019).
- Quiroz-Morales, S. E., García-Reyes, S., Ponce-Soto, G. Y., Servin-Gonzalez, L. Tracking the origins of Pseudomonas aeruginosa phylogroups by diversity and evolutionary analysis of important pathogenic marker genes. Diversity. 14 (5), 345 (2022).
- Déziel, E., et al. Liquid chromatography/mass spectrometry analysis of mixtures of rhamnolipids produced by Paeudomonas aeruginosa strain 57RP grown on mannitol or naphthalene. Biochemistry and Biophysic Acta. 1440 (2-3), 244-252 (1999).
- Abdel-Mawgoud, A. M., Lépine, F., Déziel, E. Rhamnolipids: Diversity of structures, microbial origins, and roles. Applied Microbiology and Biotechnology. 86 (5), 1323-1336 (2010).
- Toribio, J., Escalante, A. E., Soberón-Chávez, G. Production of rhamnolipids in bacteria other than Pseudomonas aeruginosa. European Journal of Lipid Science and Technology. 112, 1082-1087 (2010).
- Filbig, M., et al., Soberón-Chávez, G., et al. Metabolic and process engineering on the edge-Rhamnolipids are a true challenge: A review. Biosurfactants. Foundations and Frontiers in Enzymology. , 157-181 (2023).
- Noll, P., et al. Limits for sustainable biosurfactant production: Techno-economic and environmental assessment of a rhamnolipid production process. Bioresource Technology. 25, 101767 (2024).
- García-Reyes, S., Cocotl-Yañez, M., González-Valdez, A., Servín-González, L., Soberón Chávez, G. The PqsR-independent quorum-sensing response of Pseudomonas aeruginosa ATCC 9027 outlier-strain reveals new insights on the PqsE effect on RhlR activity. Molecular Microbiology. 116 (4), 1113-1123 (2021).
- Grosso-Becerra, M. V., et al. Pseudomonas aeruginosa ATCC 9027 is a non-virulent strain suitable for mono-rhamnolipids production. Applied Microbiology and Biotechnology. 100 (23), 9995-10004 (2016).
- Gutiérrez-Gómez, U., Soto-Aceves, M. P., Servín-González, L., Soberón-Chávez, G. Overproduction of rhamnolipids in Pseudomonas aeruginosa PA14 by redirection of the carbon flux from polyhydroxyalcanoate synthesis and overexpression of the rhlAB-R operon. Biotechnology Letters. 40 (11), 1561-1566 (2018).
- Zibek, S., Soberón-Chávez, G., Hausmann, R., Henkel, M. Overview on glycosylated lipids produced by bacteria and fungi: Rhamno-, Sophoro-, Mannosylerythritol and Cellobiose Lipids. Biosurfactants for a Biobased. Advances in Biochemical Engineering/Biotechnology. , 181 (2022).
- Twigg, M. S., et al. Microbial biosurfactant research: time to improve the rigour in the reporting of synthesis, functional characterization and process development. Microbial Biotechnology. 14 (1), 147-170 (2021).
- Matsuyama, T., Sogawa, M., Yano, I. Direct colony thin layer chromatography and rapid characterization of Serratia marscescens wetting agents. Applied and Environmental Microbiology. 53 (5), 1186-1188 (1987).
- Chandrasekaran, E. V., Bemiller, J. N. Constituent analyses of glycosaminoglycans. Methods on Carbohydrate Chemistry. 8, 89-96 (1980).
- Behrens, B., Engelen, J., Tiso, T., Blank, L. M., Hayen, H. Characterization of rhamnolipids by liquid chromatography/mass spectrometry after solid-phase extraction. Analytic and Bioanalytic Chemistry. 408 (10), 2505-2514 (2016).
- Rahim, R., et al. Cloning and functional characterization of the Pseudomonas aeruginosa rhlC gene that encodes rhamnosyltransferase 2, an enzyme responsible for di-rhamnolipid biosynthesis. Molecular Microbiology. 40 (3), 708-718 (2001).
- Irorere, V. U., Tripathi, L., Marchant, R., McClean, S., Banat, I. M. Microbial rhamnolipid production: A critical re-evaluation of published data and suggested future publication criteria. Applied Microbiology and Biotechnology. 101 (10), 3941-3951 (2017).
- Holloway, B. W. Genetic Recombination in Pseudomonas aeruginosa. Journal of General Microbiology. 13 (3), 572-581 (1955).
- Mathee, K. Forensic investigaction into the origin of Pseudomonas aeruginosa PA14-old but not lost. Journal of Medical Microbiology. 67 (8), 1019-1021 (2018).
- Roy, P. H., et al. Complete genome sequence of the multiresistant taxonomic outlier Pseudomonas aeruginosa PA7. PLoS ONE. 5, e8842 (2010).
- Zhang, Y., Miller, R. M. Enhanced octadecane dispersion and biodegradation by a Pseudomonas rhamnolipid surfactant (biosurfactant). Applied and Environmental Microbiology. 58 (10), 3276-3282 (1992).
- Miller, J. . Experiments in Molecular Genetics. , 352-355 (1992).
- Abdel-Mawgoud, A. M., Lépine, F., Déziel, E. Liquid chromatography/mass spectrometry for the identification and quantification of rhamnolipids. Pseudomonas Methods and Protocols. 30, 359-373 (2014).
- Lee, D. G., et al. Genomic analysis reveals that Pseudomonas aeruginosa virulence is combinatorial. Genome Biology. 7 (10), 90 (2006).
- Kubicki, S., et al. A straightforward assay for screening and quantification of biosurfactants in microbial culture supernatants. Frontiers in Bioengineering and Biotechnology. 8, 958 (2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten