Method Article
Detección y cuantificación de mono-ramnolípidos y di-ramnolípidos producidos por Pseudomonas aeruginosa
En este artículo
Resumen
Pseudomonas aeruginosa produce los biosurfactantes ramnolípidos. La cromatografía en capa fina detecta y determina la proporción de mono y di-ramnolípidos producidos por cada cepa. La cuantificación de los ramnolípidos totales implica la evaluación de los equivalentes de ramnosa presentes en estos biosurfactantes extraídos de los sobrenadantes de cultivo mediante el método de orcinol.
Resumen
La bacteria ambiental Pseudomonas aeruginosa es un patógeno oportunista con alta resistencia a los antibióticos que representa un peligro para la salud. Esta bacteria produce altos niveles de biosurfactantes conocidos como ramnolípidos (RL), que son moléculas con un importante valor biotecnológico pero que también se asocian a rasgos de virulencia. En este sentido, la detección y cuantificación de RL puede ser útil tanto para aplicaciones biotecnológicas como para proyectos de investigación biomédica. En este artículo, demostramos paso a paso la técnica para detectar la producción de las dos formas de RL producidas por P. aeruginosa mediante cromatografía en capa fina (TLC): mono-ramnolípidos (mRL), moléculas constituidas por un dímero de ácidos grasos (principalmente C10-C10) unido a una fracción de ramnosa, y di-ramnolípidos (dRL), moléculas constituidas por un dímero de ácido graso similar unido a dos partes de ramnosa. Además, presentamos un método para medir la cantidad total de RL basado en la hidrólisis ácida de estos biosurfactantes extraídos de un sobrenadante de cultivo de P. aeruginosa y la posterior detección de la concentración de ramnosa que reacciona con el orcinol. La combinación de ambas técnicas se puede utilizar para estimar la concentración aproximada de mRL y dRL producida por una cepa específica, como se ejemplifica aquí con las cepas tipo PAO1 (filogrupo 1), PA14 (filogrupo 2) y PA7 (filogrupo 3).
Introducción
Pseudomonas aeruginosa es una bacteria ambiental y un patógeno oportunista de gran preocupación debido a su producción de rasgos asociados a la virulencia y su alta resistencia a los antibióticos 1,2. Un metabolito secundario característico producido por esta bacteria es el biosurfactante RL, que se produce de forma coordinada con varios rasgos asociados a la virulencia como la fenazina piocianina, un antibiótico con actividad redox, y la proteasaelastasa 3. Las propiedades tensioactivas y emulsificantes de RL han sido explotadas en diferentes aplicaciones industriales y actualmente se comercializan4.
La mayoría de las cepas de P. aeruginosa, pertenecientes a los filogrupos 1 y 2, producen dos tipos de RL: mRL, que consiste en una fracción de ramnosa unida a un dímero de ácidos grasos principalmente de 10 carbonos, y dRL, que contiene una fracción de ramnosa adicional unida a la primera ramnosa4 (ver Figura 1). Sin embargo, se ha reportado que dos filogrupos menores de P. aeruginosa (grupos 3 y 5) solo producen mRL 5,6. Los dos tipos de RL contienen una mezcla de dímeros de ácidos grasos, que, como se mencionó, son principalmente C10-C10, pero también se producen proporciones más pequeñas de moléculas que contienen dímeros C12-C10, C12-C12 y C10-C12:1. Se ha reportado la caracterización de los congéneres RL producidos por diferentes cepas utilizando HPLC MS/MS 7,8. Los métodos descritos en este trabajo solo pueden diferenciar entre mRL y dRL, pero no se pueden utilizar para la caracterización de los congéneres de RL.
P. aeruginosa y algunas especies de Burkholderia son productoras naturales de RL9, pero la primera bacteria es la productora más eficiente. Sin embargo, actualmente se produce RL de uso comercial en derivados de Pseudomonas putida KT2440 que expresan genes de P. aeruginosa para evitar el uso de este patógeno oportunista10,11. La detección y cuantificación de RL producida por P. aeruginosa son de gran importancia para el estudio de los mecanismos moleculares implicados en la expresión de rasgos relacionados con la virulencia12, en la caracterización de cepas pertenecientes a los clados 3 o 513, y para la construcción de derivados de P. aeruginosa que sobreproduzcan estos biosurfactantes y tengan una virulencia reducida14. La producción de biosurfactantes por parte de diferentes microorganismos se ha detectado a partir de algunas características generales de estos compuestos, como el método de la gota de colapso o el índice de emulsificación15, pero estos métodos no son precisos ni específicos16.
En este trabajo se describe el protocolo para la detección de mRL y dRL mediante la extracción líquida de RL total a partir de los sobrenadantes de cultivo de diferentes cepas de tipo P. aeruginosa y la separación de ambos tipos de RL mediante TLC. En este método, los RL extraídos del sobrenadante de cultivo se separan por su solubilidad diferencial en los disolventes utilizados para el TLC, lo que provoca una migración diferencial en la placa de gel de sílice. Por lo tanto, los mRL tienen una migración más rápida que los dRL, y se pueden detectar como puntos separados cuando las placas se secan y se tiñen con α-naftol.
El método descrito aquí para la detección de mRL y dRL por TLC se basa en un artículo17 publicado anteriormente, que es fácil de realizar y no requiere equipos costosos. Este método ha sido útil para detectar RL en varios aislados de P. aeruginosa 13 utilizando controles apropiados, como un mutante derivado de P. aeruginosa incapaz de producir RL. Sin embargo, no es el método preferido para caracterizar nuevos biosurfactantes producidos por bacterias distintas de Pseudomonas aeruginosa debido a su falta de especificidad.
Además, se presenta un método para cuantificar los equivalentes de ramnosa de RL total extraído de un sobrenadante de cultivo de P. aeruginosa . Este método cuantifica estos biosurfactantes a partir de la reacción del orcinol con azúcares reductores, dando como resultado un producto que puede medirse espectrofotométricamente a 421 nm, como se ha descrito anteriormente18. Dado que la reacción con orcinol no es específica de la ramnosa, es importante realizar este método con RL extraído del sobrenadante del cultivo que no contenga cantidades significativas de otras moléculas que contengan azúcares, como los lipopolisacáridos (LPS). Aquí se utiliza una mezcla acidificada de cloroformo y metanol para la extracción líquida de RL18, pero también se puede utilizar acetato de etilo, y la extracción en fase sólida (SPE) da muy buenos resultados19. El método de orcinol descrito aquí no requiere equipos sofisticados y puede proporcionar resultados confiables si se realiza con especial cuidado en la preparación de las muestras analizadas, como se discutió. Para garantizar una preparación adecuada de la muestra, es importante incluir un mutante de Pseudomonas aeruginosa rhlA incapaz de producir RL20 y realizar tres réplicas biológicas y tres técnicas para cada determinación.
Ha existido una importante controversia en la literatura16,21 en relación a la determinación de RL por el método de orcinol, con algunos estudios que sugieren que la producción de RL está sobreestimada y que el ensayo carece de especificidad para la ramnosa, potencialmente detectando otros azúcares. Sin embargo, demostramos aquí que los métodos descritos pueden ser precisos y específicos en las condiciones adecuadas. Además, para la comparación con los procedimientos descritos en este artículo, utilizamos la detección UPLC-MS/MS de un estándar dRL y demostramos que se obtienen resultados similares con el método orcinol. El protocolo detallado para cuantificar el RL utilizando este método se incluye en el Archivo Complementario 1.
Estos protocolos se ejemplifican utilizando las cepas tipo PAO1 (filogrupo 1), PA14 (filogrupo 2) y PA7 (filogrupo 3). Se eligieron estas cepas porque están bien caracterizadas y producen diferentes perfiles de RL.
Protocolo
Este procedimiento se esquematiza en la Figura Suplementaria 1. Los reactivos y equipos utilizados para el estudio se enumeran en la Tabla de Materiales.
1. Detección de mRL y dRL en sobrenadantes de cultivo de P. aeruginosa mediante TLC
- Comenzar con el caldo centrifugado de la cepa de interés P . aeruginosa , cultivado en medio líquido durante 24 h (para llegar a la fase estacionaria de crecimiento donde se produce RL). Por lo general, estos cultivos contienen 1 x 109 bacterias por ml.
- Ajuste el pH del cultivo a 2 utilizando HCl concentrado.
- Poner 5 mL del sobrenadante de cultivo acidificado en un tubo de polipropileno de 50 mL y añadir 5 mL de una mezcla 2:1 de cloroformo: metanol.
- Agite el tubo por inversión tres veces, cada vez durante 10 s, y deje el tubo sin inversión durante 2 minutos entre cada agitación.
- Deje el tubo sin agitación durante aproximadamente 3 h hasta que se separen las dos fases, o centrifugue el tubo durante 10 min a 3.000 x g a 4 °C.
- Transfiera la fase orgánica (capa inferior) a un tubo limpio y deje el tubo en una campana de extracción hasta que se seque.
- Repita el proceso a partir del paso 1.3, colocando la fase orgánica del segundo cloroformo: extracción con metanol, en el tubo que se utilizó en la primera extracción.
- Evaporar la fase orgánica hasta que queden 1 mL o 1,5 mL. Transfiera este volumen a un tubo de centrífuga de 1,5 ml y evapore el solvente hasta que se seque durante la noche.
- Agregue 50 μL de metanol al tubo seco para resuspender RL.
- Realice cromatografía en capa fina en placas de gel de sílice.
NOTA: El tamaño de la placa TLC debe cortarse de acuerdo con el número de muestras que se analizarán. Cada muestra debe aplicarse a 1,5 cm, y el punto de aplicación debe estar a 1 cm del borde de la placa (dibuje una línea horizontal con un lápiz). - Aplique 5 μL de cada muestra con una micropipeta de 10 μL.
- La fase líquida de TLC consiste en una mezcla 65:15:2 de cloroformo: metanol y ácido acético. Para preparar 35 mL de esta mezcla, mezcle 26 mL de cloroformo, 6 mL de metanol y 0,8 mL de una solución madre al 20% de ácido acético. Mezcle estos solventes y colóquelos en la cámara TLC cerrada durante al menos 10 minutos antes de comenzar la cromatografía, de modo que la atmósfera esté saturada con los solventes volátiles.
- Coloque la placa TLC en la cámara, evitando el contacto con el punto donde se aplicaron las muestras.
- Cierre la cámara y deje el TLC hasta que el solvente alcance 1 cm antes del borde de la placa. En este punto, retire la placa y déjela secar (se puede aplicar un flujo de aire para acelerar el proceso).
- Prepare una solución de α-naftol disolviendo 5 g de este compuesto en 33 mL de etanol. Una vez disuelto, añadir 127,5 mL de etanol, 12,6 mL de agua y 20,5 mL de H2SO4 frío.
- Para detectar la presencia de mRL y dRL, rocíe la solución de α-naftol sobre la placa en la campana de extracción y coloque la placa rociada en un horno a 85 °C durante varios minutos hasta que la marca rosada de RL sea evidente.
- Utilice una imagen del TLC para calcular la proporción de mono- y di-RL presentes en cada muestra utilizando un software que detecte la densidad de cada mancha.
2. Cuantificación de la cantidad total de RL midiendo los equivalentes de ramnosa presentes en el biosurfactante
- Colocar 1,2 mL de un cultivo en fase estacionaria (cultivado durante 24 h) en un tubo de centrífuga de 1,5 mL y centrifugar durante 3 min a 3.000 x g a 4 °C. Recoja el sobrenadante en un tubo limpio.
- Transfiera 333 μL a un tubo limpio (realice este paso por triplicado) y agregue 1 mL de éter.
- Mezcle vigorosamente en un vórtice durante 30 s. Repita este procedimiento con un tubo a la vez.
- Centrifugar durante 2 min a 3.000 x g a 4 °C. Recoja la fase orgánica (fase superior), transfiérala a un tubo limpio y déjela abierta en la campana de extracción hasta que se produzca la sequedad.
- Repita la extracción con éter como se describe en el paso 2.2. Repita los pasos 2.3 y 2.4.
- Una vez que esté completamente seco, agregue 1 mL de agua a cada tubo. Deje los tubos durante 12 h para permitir que el RL se hidrate, luego agite vigorosamente en el vórtice.
- Coloque un matraz limpio en hielo durante 5 min (ya que es una reacción exotérmica). Preparar una solución de 60% H2SO4 (proteger el matraz de la luz).
- En un tubo limpio, prepare una solución de orcinol al 1,6% en agua destilada. Para preparar el reactivo de orcinol, mezcle 4,4 mL de la solución de H2SO4 al 60% con 0,6 mL de la solución de orcinol al 1,6%.
- Calcular el volumen final del reactivo de orcinol necesario. Añadir 900 μL de este reactivo a cada muestra y a cada concentración de la curva de calibración, utilizando diferentes concentraciones de ramnosa (normalmente, se utilizan 9 concentraciones de ramnosa en el rango de 1 μg/mL a 9 μg/mL), y una con 100 μL de agua, asegurándose de que todas se realicen por triplicado.
- Añada 100 μL de cada muestra de RL a 900 μL del reactivo de orcinol en un tubo de vidrio y mezcle las dos soluciones.
- Incubar durante 30 min en un baño de agua precalentado a 80 °C.
- Deje que los tubos se enfríen a temperatura ambiente.
- Lea la absorbancia de las muestras y la curva de calibración a 421 nm utilizando una celda de cuarzo.
- Calcule la concentración de ramnosa en cada muestra interpolando la absorbancia en la curva de calibración y considerando el volumen utilizado para la determinación.
- Para calcular la concentración en μM, divida la concentración obtenida en μg/mL por 182,2 (el peso molecular de la ramnosa) y multiplíquela por 1000.
Resultados
En este artículo, se utilizaron tres cepas diferentes de P. aeruginosa para representar tres filogrupos, cada uno con diferentes niveles de producción de RL y proporciones de mRL y dRL. Estas cepas incluyen PAO1 (un aislado de herida de Australia, 195522), PA14 (un aislado de planta de los EE. UU., 197723) y PA7 (un aislado clínico de Argentina, 201024). Como control negativo, se empleó el mutante PAO1 rhlA, que es incapaz de producir RL. Todas las cepas se cultivaron durante 24 h en medio PPGAS, específicamente diseñado para promover altos niveles de RL25, a 37 °C. Los cultivos de PPGAS se inocularon a una densidad óptica de 0,05 a 600 nm utilizando un medio LB26 durante la noche. Por lo general, las cepas de P. aeruginosa alcanzan una densidad óptica de 2 medida a 600 nm en medio PPGAS después de 24 h, lo que corresponde aproximadamente a 1 x 109 bacterias por mL. Para recolectar los sobrenadantes de cultivo para la detección y cuantificación de RL, los cultivos se centrifugaron a 3.000 x g a 4 °C durante 15 min, y se desechó el pellet celular. Los resultados obtenidos utilizando estos dos métodos (Figura 2 y Figura 3) ilustran que tanto los tipos de RL producidos por cada cepa como la cantidad de su producción difieren entre las tres cepas analizadas. Como se muestra en la Figura 2, PAO1 produce aproximadamente 30% mRL y 70% dRL, mientras que PA14 produce una proporción igual de 50% mRL y 50% dRL, y PA7 produce exclusivamente mRL. Este perfil de producción de RL es consistente con informes anteriores12,14.
La Figura 3A ilustra la cantidad de equivalentes de ramnosa presentes en la RL producidos por cada uno de estos tres tipos de cepas. A partir de las cantidades de ramnosa detectadas, es evidente que la cepa PA14 produce la mayor cantidad de RL, mientras que la cepa PA7 produce la menor cantidad. Sin embargo, estos resultados por sí solos no pueden utilizarse para estimar la concentración en μM de RL producida por cada cepa. Además, el método del orcinol por sí solo no puede proporcionar una concentración aproximada de RL, ya que se basa en la caracterización de las proporciones de mRL y dRL producidas por cada cepa de interés. Por lo tanto, para obtener una estimación de la concentración en μM de RL, se debe considerar la proporción de cada tipo de RL determinada en el TLC, y se debe incluir una curva estándar con diferentes concentraciones de ramnosa (Figura 3B).
De hecho, el método orcinol se puede utilizar para cuantificar la concentración de RL cuando solo se produce un tipo de este biosurfactante. En tales casos, la concentración en μM de la ramnosa detectada corresponde directamente a la concentración en μM de mRL. En el caso de la dRL, dado que se producen dos ramnosas por la hidrólisis de cada molécula de dRL, la concentración de μM de ramnosa detectada en la RL debe dividirse por 2 para obtener su concentración de μM (consulte la Figura 4).
Sin embargo, la mayoría de las cepas de P. aeruginosa producen ambos tipos de RL en proporciones variables (como se muestra en la Figura 2), lo que permite determinar solo una concentración aproximada del RL total. Teniendo esto en cuenta, estimamos la producción de RL por cada cepa de la siguiente manera: Para la cepa PA7, dado que solo produce mRL, la ramnosa que forma parte de RL (44,6 μg/mL + 4,5 μg/mL) corresponde directamente a la concentración de μM (244,78), representando la concentración de este biosurfactante en el sobrenadante de cultivo, ya que cada molécula de RL contiene un resto de ramnosa. Sin embargo, para la cepa PAO1, aunque la concentración de ramnosa detectada fue de 111,55 μg/mL + 11,41 μg/mL, solo el 30% corresponde a mRL. Por lo tanto, el 70% de las moléculas de RL contienen dos ramnosas por molécula. Para estimar la concentración de RL, se dividió por 5 la concentración de ramnosa en μM (612.24), considerando que 1/5 corresponde a la concentración de μM de mRL (122.49) y 2/5 (244.89) a dRL. Por lo tanto, la concentración aproximada de μM de RL producida por esta cepa es de 367,39.
Para la cepa PA14, la concentración de ramnosa detectada fue de 194,39 μg/mL + 11,5 μg/mL, que se convirtió a la concentración de μM (1.066,9) y se dividió por 3. El resultado representa la concentración de cada tipo de RL, considerando que cada tipo se produjo al 50% y que dRL contiene 2 ramnosas por molécula. Así, esta cepa produce 355,63 μM de mRL y la misma concentración de dRL, lo que da como resultado una concentración aproximada de μM de RL de 711,27, casi el doble que la cepa PAO1 y casi tres veces la concentración producida por la cepa PA7.

Figura 1: Composición química de los principales congéneres de monorhamnolípidos y diramnolípidos. (A) Monorhamnolípidos. (B) Di-ramnolípidos. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Detección de mono-ramnolípidos y di-ramnolípidos por cromatografía en capa fina. (A) Imagen de una placa de TLC que muestra los estándares de RL y la RL producida por las cepas PAO1, PA14, PA7 y el mutante ΔrhlA derivado de PAO1. (B) Estimación de la proporción de cada tipo de RL (mRL y dRL) en cada una de las muestras ensayadas en (A), utilizando el software ImageJ. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Cuantificación de la concentración de RL mediante el método de orcinol. (A) Concentración de ramnosa (μg/mL) contenida en RL extraída de los sobrenadantes de cultivo de las cepas PAO1 (barra negra), PA14 (barra gris claro) y PA7 (barra gris oscuro). Las barras denotan la desviación estándar. (B) La curva de calibración de ramnosa para el experimento que se muestra en (A). Haga clic aquí para ver una versión más grande de esta figura.
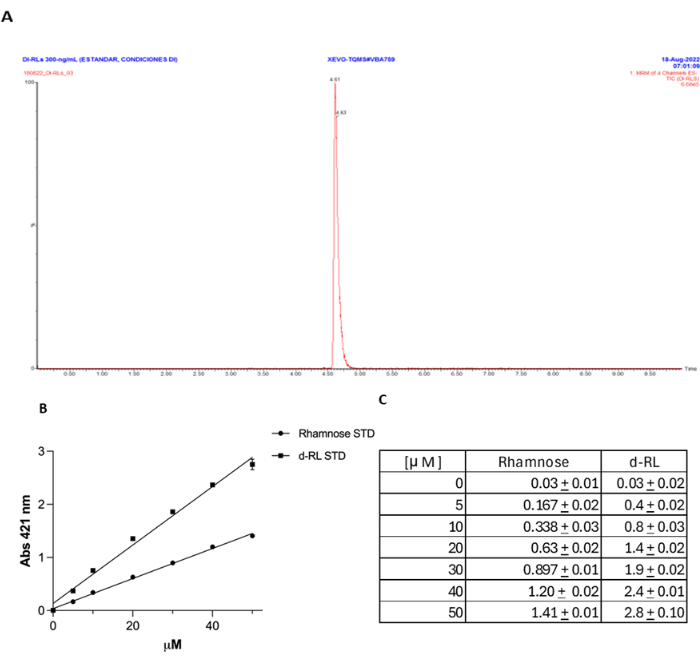
Figura 4: Validación del método de orcinol comparado con UPLC-MS/MS para la cuantificación de dRL. (A) El estándar de dRL se cuantificó utilizando UPLC-MS/MS. (B) El método de orcinol comparado con la concentración de ramnosa expresada en mM. (C) La misma concentración de mM de dRL que la ramnosa da aproximadamente el doble de la absorbancia a 421 nm, como se esperaba. Haga clic aquí para ver una versión más grande de esta figura.
Figura complementaria 1: Representación esquemática del protocolo. Haga clic aquí para descargar este archivo.
Fichero complementario 1: Protocolo de detalle para cuantificar dRL por UPLC-MS/MS. Haga clic aquí para descargar este archivo.
Discusión
El método más preciso para detectar y cuantificar el RL es la HPLC acoplada a la espectrometría de masas (MS)7,8,27; Sin embargo, requiere equipos especializados y costosos que pueden no ser accesibles para muchos investigadores. Los métodos descritos aquí se pueden realizar de forma rutinaria con materiales y equipos básicos de laboratorio para detectar y estimar las concentraciones de RL, pero tienen algunas limitaciones, en particular su inexactitud en la determinación de mezclas de mRL y dRL. Además, la preparación de la muestra utilizada para la cuantificación de RL debe ejecutarse cuidadosamente. Los cultivos utilizados para la detección y cuantificación de RL no deben estar en la fase estacionaria tardía, ya que las células pueden agregarse o lisarse, lo que da lugar a suspensiones celulares no homogéneas que podrían causar variabilidad en los resultados obtenidos. Además, los desechos celulares podrían interferir con el método de orcinol.
Se pueden obtener resultados precisos cuando el protocolo descrito se realiza con controles adecuados, como una cepa de Pseudomonas aeruginosa incapaz de producir RL (como el mutante PAO1 ΔrhlA 20 utilizado en este trabajo, Figura 2). Estos resultados son útiles para caracterizar diferentes aislados de P. aeruginosa 12,13, modificar genéticamente cepas para aumentar la producción de RL14, o estudiar la genética molecular de la producción de RL por varios aislados de P. aeruginosa 12.
Por lo tanto, los métodos aquí descritos pueden utilizarse para caracterizar las cepas de Pseudomonas aeruginosa por su producción de factores de virulencia y evaluar su potencial biotecnológico, especialmente en las primeras etapas de investigación. Es importante tener en cuenta que estos métodos no pretenden ser sustitutos de técnicas como la HPLC/MS, sino más bien como técnicas accesibles para la caracterización inicial de la cepa y la manipulación genética de las cepas de P. aeruginosa .
Además, si la investigación se centra en la producción de RL con fines biomédicos, es crucial reconocer que la producción de RL es solo uno de los varios factores producidos por Pseudomonas aeruginosa. Además, el tipo de RL producido o la cantidad de su producción no está estrictamente correlacionado con la virulencia de una cepa en particular, ya que la virulencia está influenciada por múltiples factores28.
En cuanto a la reproducibilidad de los métodos aquí descritos, se debe prestar especial atención a la obtención de las fases orgánicas tanto en la detección de RL por TLC (paso 1.6) como en la medición de RL por el método de orcinol (paso 2.4). Incluso una pequeña cantidad de la otra fase tomada durante la extracción puede dar lugar a una variación considerable en los resultados obtenidos. La extracción de RL del sobrenadante del cultivo es un paso crítico para lograr resultados fiables y reproducibles con el método de orcinol. Es crucial que al recolectar la fase orgánica, se excluyan la fase intermedia y la fase líquida. Si una fase etérea no se seca completamente en 3 h, es probable que contenga agua, lo que requiere una nueva extracción.
El uso del método de orcinol para la cuantificación de RL ha sido objeto de debate, con argumentos en contra de su precisión y especificidad. Se ha afirmado que las RL a menudo se sobreestiman, y el método no es específico, ya que el orcinol puede reaccionar con diferentes azúcares, no solo con la ramnosa. Los datos actuales demuestran que si las RL se extraen eficientemente del sobrenadante del cultivo, esta técnica es específica, como lo demuestra la falta de reacción cuando el mutante rhlA se utiliza como control negativo. Sin embargo, para proporcionar evidencia adicional de la precisión del método en la detección, independientemente del procedimiento de extracción, detectamos un estándar dRL por UPLC-MS/MS como se describió anteriormente27, y comparamos los resultados obtenidos con el método de orcinol18 (Archivo Suplementario 1, Figura 4A). Encontramos, como se reportó18, una muy buena correlación entre la concentración de RL medida con este método, donde cada concentración de μM de dRL detectada corresponde aproximadamente al doble de la absorbancia a 421 nm que la misma concentración de μM de ramnosa (Figura 4B,C). Este experimento de control demuestra claramente la precisión de este método para la cuantificación de RL.
Sin embargo, debido al desafío de medir la concentración de mezclas de mRL y dRL con el método de orcinol, la mayoría de las publicaciones informan los resultados como equivalentes de ramnosa en RL. Este valor puede no tener una correlación directa con la concentración real de RL, como se ilustra en los ejemplos presentados aquí, pero sin embargo, sirve como un medio útil para comparar las cantidades de RL en diferentes condiciones, especialmente cuando se utiliza la misma cepa de P. aeruginosa .
Se ha descrito un método alternativo para la cuantificación de mRL y dRL, que se basa en la solubilización del colorante hidrofóbico Victoria Pure Blue BO, comúnmente utilizado en bolígrafos29. Este método es rápido y económico. Sin embargo, este protocolo se desarrolló para cuantificar la producción heteróloga de RL por P. putida y no es factible su uso con RL producida por P. aeruginosa, ya que la expresión de la toxina azul piocianina interfiere con este método.
En resumen, los métodos aquí descritos ofrecen una alternativa viable para la detección y cuantificación de RL producida por P. aeruginosa que no requiere de equipos sofisticados y costosos. Proporcionan un enfoque práctico para que los investigadores evalúen la producción de RL en una variedad de entornos experimentales.
Divulgaciones
Los autores no tienen ningún conflicto de intereses que revelar.
Agradecimientos
El laboratorio del GSCh se financia en parte con IN201222 de apoyo del Programa de Apoyo a Proyectos de Investigación e Innovación Tecnológica (PAPIIT) de la Dirección General de Asuntos del Personal Académico -UNAM.
Materiales
| Name | Company | Catalog Number | Comments |
| 1-NAPHTHOL | SIGMA-ALDRICH | 70442 | |
| ACETIC ACID | J.T. BAKER | 9508-02 | |
| CENTRIFUGE | For centrifuging tubes 1.5 mL and 50 mL | ||
| CHLOROFORM | J.T. BAKER | 9180-02 | |
| DRYING OVEN | |||
| ETHER | J.T. BAKER | 9244-02 | |
| GLASS PIPETTE | SIGMA-ALDRICH | CLS706510 | |
| HYDROCHLORIC ACID | J.T. BAKER | 5622-02 | |
| LB | |||
| L-RHAMNOSE MONOHYDRATE | SIGMA-ALDRICH | R-3875 | |
| METHANOL | J.T. BAKER | 9049-02 | |
| ORCINOL MONOHYDRATE | SIGMA-ALDRICH | O1875 | |
| PPGAS Broth | Tris HCL (0.12M), Potassium Chloride ( 0.02M) Ammonium Chloride (0.02M), Peptone (1%), pH 7.4 Autoclaved. Add Glucose (5%) and Magnesium Sulfate (0.0016M) | ||
| QUARTZ CELL (CUVETTE) | SIGMA-ALDRICH | Z276669 | |
| RECTANGULAR TLC DEVELOPING TANK | FISHER SCIENTIFIC | K4161801020 | |
| RHAMNOLIPIDS | SIGMA-ALDRICH | R-90 | |
| SPECTROPHOTOMETER | VIS | ||
| SPRAYER | SIGMA-ALDRICH | Z529710-1EA | |
| SULFURIC ACID | J.T. BAKER | 9681-02 | |
| TES TUBES 5mL | CORNING | 352002 | |
| TLC SILICA GEL 60 F254 | MERCK | 1.05554.0001 | |
| WATER BATH | > 80 °C |
Referencias
- Moradali, M. F., Ghods, S., Rehm, B. H. A. Pseudomonas aeruginosa lifestyle: A paradigm for adaptation, survival, and persistence. Frontiers in Cell Infection and Microbiology. 7, 1-29 (2017).
- Gellatly, S. L., Hancock, R. E. W. Pseudomonas aeruginosa: New insights into pathogenesis and host defenses. Pathogens and Disease. 67 (3), 159-173 (2013).
- Williams, P., Cámara, M. Quorum sensing and environmental adaptation in Pseudomonas aeruginosa: a tale of regulatory networks and multifunctional signal molecules. Current Opinion in Microbiology. 12 (2), 182-191 (2009).
- Soberón-Chávez, G., González-Valdez, A., Soto-Aceves, M. P., Cocotl-Yañez, M. Rhamnolipids produced by Pseudomonas: From molecular genetics to the market. Microbial Biotechnology (MBT). 14 (1), 136-146 (2021).
- Freschi, L., et al. The Pseudomonas aeruginosa pan-genome provides new insights on its population structure, horizontal gene transfer, and pathogenicity. Genome Biology and Evolution. 11 (1), 109-120 (2019).
- Quiroz-Morales, S. E., García-Reyes, S., Ponce-Soto, G. Y., Servin-Gonzalez, L. Tracking the origins of Pseudomonas aeruginosa phylogroups by diversity and evolutionary analysis of important pathogenic marker genes. Diversity. 14 (5), 345 (2022).
- Déziel, E., et al. Liquid chromatography/mass spectrometry analysis of mixtures of rhamnolipids produced by Paeudomonas aeruginosa strain 57RP grown on mannitol or naphthalene. Biochemistry and Biophysic Acta. 1440 (2-3), 244-252 (1999).
- Abdel-Mawgoud, A. M., Lépine, F., Déziel, E. Rhamnolipids: Diversity of structures, microbial origins, and roles. Applied Microbiology and Biotechnology. 86 (5), 1323-1336 (2010).
- Toribio, J., Escalante, A. E., Soberón-Chávez, G. Production of rhamnolipids in bacteria other than Pseudomonas aeruginosa. European Journal of Lipid Science and Technology. 112, 1082-1087 (2010).
- Filbig, M., et al., Soberón-Chávez, G., et al. Metabolic and process engineering on the edge-Rhamnolipids are a true challenge: A review. Biosurfactants. Foundations and Frontiers in Enzymology. , 157-181 (2023).
- Noll, P., et al. Limits for sustainable biosurfactant production: Techno-economic and environmental assessment of a rhamnolipid production process. Bioresource Technology. 25, 101767 (2024).
- García-Reyes, S., Cocotl-Yañez, M., González-Valdez, A., Servín-González, L., Soberón Chávez, G. The PqsR-independent quorum-sensing response of Pseudomonas aeruginosa ATCC 9027 outlier-strain reveals new insights on the PqsE effect on RhlR activity. Molecular Microbiology. 116 (4), 1113-1123 (2021).
- Grosso-Becerra, M. V., et al. Pseudomonas aeruginosa ATCC 9027 is a non-virulent strain suitable for mono-rhamnolipids production. Applied Microbiology and Biotechnology. 100 (23), 9995-10004 (2016).
- Gutiérrez-Gómez, U., Soto-Aceves, M. P., Servín-González, L., Soberón-Chávez, G. Overproduction of rhamnolipids in Pseudomonas aeruginosa PA14 by redirection of the carbon flux from polyhydroxyalcanoate synthesis and overexpression of the rhlAB-R operon. Biotechnology Letters. 40 (11), 1561-1566 (2018).
- Zibek, S., Soberón-Chávez, G., Hausmann, R., Henkel, M. Overview on glycosylated lipids produced by bacteria and fungi: Rhamno-, Sophoro-, Mannosylerythritol and Cellobiose Lipids. Biosurfactants for a Biobased. Advances in Biochemical Engineering/Biotechnology. , 181 (2022).
- Twigg, M. S., et al. Microbial biosurfactant research: time to improve the rigour in the reporting of synthesis, functional characterization and process development. Microbial Biotechnology. 14 (1), 147-170 (2021).
- Matsuyama, T., Sogawa, M., Yano, I. Direct colony thin layer chromatography and rapid characterization of Serratia marscescens wetting agents. Applied and Environmental Microbiology. 53 (5), 1186-1188 (1987).
- Chandrasekaran, E. V., Bemiller, J. N. Constituent analyses of glycosaminoglycans. Methods on Carbohydrate Chemistry. 8, 89-96 (1980).
- Behrens, B., Engelen, J., Tiso, T., Blank, L. M., Hayen, H. Characterization of rhamnolipids by liquid chromatography/mass spectrometry after solid-phase extraction. Analytic and Bioanalytic Chemistry. 408 (10), 2505-2514 (2016).
- Rahim, R., et al. Cloning and functional characterization of the Pseudomonas aeruginosa rhlC gene that encodes rhamnosyltransferase 2, an enzyme responsible for di-rhamnolipid biosynthesis. Molecular Microbiology. 40 (3), 708-718 (2001).
- Irorere, V. U., Tripathi, L., Marchant, R., McClean, S., Banat, I. M. Microbial rhamnolipid production: A critical re-evaluation of published data and suggested future publication criteria. Applied Microbiology and Biotechnology. 101 (10), 3941-3951 (2017).
- Holloway, B. W. Genetic Recombination in Pseudomonas aeruginosa. Journal of General Microbiology. 13 (3), 572-581 (1955).
- Mathee, K. Forensic investigaction into the origin of Pseudomonas aeruginosa PA14-old but not lost. Journal of Medical Microbiology. 67 (8), 1019-1021 (2018).
- Roy, P. H., et al. Complete genome sequence of the multiresistant taxonomic outlier Pseudomonas aeruginosa PA7. PLoS ONE. 5, e8842 (2010).
- Zhang, Y., Miller, R. M. Enhanced octadecane dispersion and biodegradation by a Pseudomonas rhamnolipid surfactant (biosurfactant). Applied and Environmental Microbiology. 58 (10), 3276-3282 (1992).
- Miller, J. . Experiments in Molecular Genetics. , 352-355 (1992).
- Abdel-Mawgoud, A. M., Lépine, F., Déziel, E. Liquid chromatography/mass spectrometry for the identification and quantification of rhamnolipids. Pseudomonas Methods and Protocols. 30, 359-373 (2014).
- Lee, D. G., et al. Genomic analysis reveals that Pseudomonas aeruginosa virulence is combinatorial. Genome Biology. 7 (10), 90 (2006).
- Kubicki, S., et al. A straightforward assay for screening and quantification of biosurfactants in microbial culture supernatants. Frontiers in Bioengineering and Biotechnology. 8, 958 (2020).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados