Method Article
세포 없는 시스템을 사용하여 단백질 프로토타이핑을 위한 최소선형 템플릿의 증폭을 위한 신속하고 효소적인 방법
요약
연구 결과는 복제하거나 살아있는 세포를 사용하지 않고 합성 유전자 단편에서 단백질 검열 캠페인을 위한 DNA의 큰 (μg-mg) 양을 만들기 위한 프로토콜을 기술합니다. 최소 템플릿은 효소적으로 소화되고 원형화된 다음, 이더스말 롤링 서클 증폭을 사용하여 증폭됩니다. 세포 없는 발현 반응은 불정제 된 제품으로 수행 될 수있다.
초록
이 프로토콜은 최소한의 DNA 템플릿의 설계와 효소 증폭을 위한 단계를 설명하며, 세포 없는 발현을 사용하여 24시간 이내에 분석 가능한 단백질의 신속한 프로토타이핑을 가능하게 합니다. 공급 업체로부터 DNA를 받은 후 유전자 단편은 PCR 증폭, 절단, 원형 및 저온 뱅크입니다. 소량의 뱅크된 DNA는 이더스롤링 원 증폭(RCA)을사용하여 현저하게 희석되고 증폭됩니다(최대 106x). RCA는 시작 물질의 피코그램 수준에서 최소한의 발현 템플릿의 마이크로그램 양을 얻을 수 있습니다 (모든 시작 합성 단편이 사용되는 경우 mg 수준). 이 작품에서 시작량은 20pg로 최종 제품의 4μg을 초래했습니다. 생성된 RCA 제품(최소 템플릿의 컨카퍼)은 정화 단계가 없는 세포 없는 반응에 직접 추가할 수 있다. 이 방법은 전적으로 PCR 기반이기 때문에 자동화된 액체 처리 시스템과 결합될 때 미래의 고처리량 선별 작업을 가능하게 할 수 있습니다.
서문
세포 없는 유전자 발현(CFE)은 많은 응용분야와 함께 강력한 도구로 부상하고 있다. 이러한 응용 분야에는질병 검출1, 2,3,4,5,6,미량 영양소 및 소분자 검출7,8,9,10,11,12,바이오 제조13,14,15,16,17 ,18,교육19,20,21,제조 어려운 단백질17,22,23,24,25,26,27,및 변종 스크리닝23,28,29,30, 31,32 ,33. 이는 셀 프리 시스템의 개방적 특성과 그들이 부여하는 유연성 때문입니다. 많은 훌륭한 리뷰 기사는 기술34,35,36,37,38,39,40,41,42, 43,44에대한 역사적 교육과 미래의관점을제공합니다.
전형적인 세포 없는 반응은 세포 추출물, 에너지 혼합 및 유전 템플릿의 세 가지 주요 구성 요소로 구성됩니다. 활성 세포 추출물은 전사 및 번역(TXTL)에 필요한 모든 기계를 포함하고 있으며36의다양한 방법으로 처리될 수 있다. 에너지 믹스의 글리코리틱 중급자, 전해질, 아미노산 및 보조인은 TXTL 공정을 지원합니다. 그것은 세포 없는 실험에 있는 가변성의 주요 근원이고45 및 여러 가지 방법으로 제조될 수 있다34,46. 전통적인 복제 방법은 우수한 발현 특성을 가진 플라스미드를 초래하기 때문에 유전 템플릿의 준비는 적은 개선을 보았다. 이러한 전통적인 방법의 단점은 이를 구성하고 전파하는 데 필요한 처리 시간과 생물학적 전문 지식의 양입니다. 최근 최적화 노력으로 인해 에너지 믹스준비49,50과병행하여 수행할 수 있는 세포 추출물준비(47,48)를 위한 간단한 24시간 워크플로우가 발생했습니다. 그러나, 기존의 복제는 CFE 프로토타이핑 타임라인(표1)23에여러 일을 추가한다. 상용 유전자 단편으로부터 PCR 제품을 빠르게 증폭시키는 것은 직접사용할수 있지만, 이는 약 5개의 반응(전통적인 15μL 부피)에 해당하는 DNA의 1μg만 생성되기 때문에 프로토타이핑 실험의 수를 제한한다. 이러한 순환화 및 이더스말 증폭의 추가 단계로, DNA의 밀리그램 수량보다 큰 (~5,000 반응 1 mg). 이것은 극적으로 단백질의 높은 처리량 검열 또는 조합 효소 네트워크 (세포 없는 신진 대사 공학)에서 만들어질 수 있는 시험의 수를 증가시킵니다; 또한 선형 템플릿 라이브러리를 고농도 DNA로 효과적으로 보존할 수 있습니다. 또한 재료 과학 응용 분야(단백질 기반 섬유 및 하이드로겔)에 필요한 대량의 단백질을 시제품화하기 위해 더 많은 양의 템플릿이 필요합니다. 선형 템플릿의 일부 제한은 BL21 DE3 Star에서 추출한 추출을 사용하거나 최근에 발견된 방법을 사용하여 선형 템플릿을 분해52,53,54로부터보호함으로써 극복할 수 있다. 그러나, 이것은 PCR 증폭을 위한 공급 업체 생산 DNA의 제한된 주식을 가진 또는 복제에 필요한 생물학 전문 지식 및 장비의 문제를 다루지 않습니다.
이 작품은 소량의 벤더 생산 유전자 단편(일반적으로 lyophilized 분말의 500-1000 ng ng)에서 얻을 수 있는 발현 템플릿의 양을 증가시키기 위해 명시적으로 설계된 프로토콜을 제시한다. 설명된 방법은 플라스미드에서 전통적인 복제를 수행하거나 살아있는 세포에서 변환 및 전파하는 데 필요한 기술을 필요로하지 않습니다. 메일에서 유전자 단편을 받으면, 사용자는 이더스럽 롤링 원 증폭(RCA)(도 1)23을사용하여 많은 세포 없는 반응에 대한 충분한 템플릿을 생성할 수 있다. 공급 업체로부터 받은 DNA의 양은 제한된 선별 노력에 충분할 수 있지만, 신속하게 고갈되고 유전자 조각을 다시 구입하는 것은 시간이 많이 걸리고 비용이 많이 듭니다. 이 방법은 또한 대장균에서 독성이 있고 복제하기 어려운 유전자에 특히 적합합니다.
프로토콜
1. 유전자 단편 설계
참고: 유전자 단편은 프로모터, 리보솜 결합 부위(RBS), 시작 코돈, 관심 유전자 및 종기 작성기를 포함하여 전사/번역에 필요한 모든 유전 적 요소를 가져야합니다. 종결자는 선형 식 템플릿(LET)에 필요하지 않지만 사용자가 플라스미드에 시퀀스를 삽입하기로 결정하는 경우 중요합니다. 이러한 시퀀스는 T7 프로모터를 사용하는 pJL1-sfGFP 플라스미드55(마이클 제웨트의 실험실에서 받은 선물)에서 해제되었습니다. 이러한 필수 유전적 원소 외에도, 제한 효소 절단 부위는 발기인(5' 절단 부위) 앞에 6개의 염기 쌍을 추가하고, 다른 6개의 기본 쌍은 종료기(3' cut site) 후에 추가되고, 이 경우 힌드III(다른 제한 효소를 사용할 수 있습니다,하지만 라이브러리에 보관하는 데 필요한 수를 줄이기 위해 하나의 높은 충실도 제한 효소로 서열을 표준화하는 데 도움이 된다). 프라이머 사이트는 5'컷 사이트의 상류 10개의 기본 쌍과 3' 컷 사이트의 하류 10개의 기본 쌍을 추가하며, 이 경우 표준화된 M13 프라이머 시퀀스(프라이머는 저렴한 재고 항목)를 사용합니다. 사용되는 제한 효소 사이트및 프라이머는 사용자의 재량에 따라 사용됩니다. 그러나 사용자는 서열이 템플릿의 다른 곳에 존재하지 않는지 확인해야 합니다(원치 않는 컷 또는 증폭 개시 사이트를 만들고 싶지 않음). 이 작업에 사용되는 템플릿의 시퀀스는 보충 자료에 자세히 설명되어 있습니다. 이러한 단계는 이 기본 템플릿에서 수정하는 데 사용됩니다.
- 대장균에서 발현된 경우 아미노산 서열 또는 유전서열을 발현하고 얻을 수 있는 원하는 유전자를 결정한다.
- 아미노산 서열인 경우, 많은 표준 공급 업체도구(56)중 하나를 사용하여 대장균에 대한 코돈 최적화를 수행한다. 보충에 제공된 템플릿을 사용하는 경우 최적화된 시퀀스에 힌드III 제한 사이트(AAGCTT)가 없는지 확인합니다. 이 경우 더 이상 HindIII 사이트가 없을 때까지 시퀀스를 계속 최적화합니다.
- 서열을 복사하여 관심 유전자가 표시된 보충 서열 #1에 대한 제공된 템플릿에 붙여 넣습니다. sfGFP를 표현하는 경우 보조 시퀀스 #1을 있는 것처럼 사용하십시오. 서브틸리신을 표현하는 경우, 보충 시퀀스 #2를 사용하십시오.
- 바람직한 DNA 합성 서비스에서 최소한의 템플릿과 필요한 프라이머를 주문한다.
2. 유전자 단편및 프라이머 를 다시 일시 중단
참고: 유전자 단편을 수령하면 제조업체의 프로토콜을 따라 재발 을 방지하거나 이 간단한 가이드를 사용하여 DNA 스톡을 만듭니다.
- 원심 분리 튜브 (300 x g for 5 s) 하단에 DNA 펠릿을 수집합니다.
- 이중 증류수(ddH2O)를 추가하여 DNA 템플릿의 10 ng/μL의 최종 농도를 만듭니다.
- 5-10 초의 중간 설정에 대한 용액을 소용돌이.
- 50°C에서 20분 동안 배양하여 전체 펠릿을 녹입니다.
- 잠시 소용돌이 다시
- 300 x g의 원심분리기는 튜브 의 하단에 있는 용액을 수집합니다.
- -20°C에 보관하거나 PCR에 사용하십시오.
- 뉴클레아제없는 물에 프라이머를 다시 분리하여 100 μM 프라이머 재고를 준비합니다. 첨가물의 양을 결정하기 위해, lyophilized 프라이머의 나노몰수를 10배곱한다. 예를 들어, 튜브에 45nM의 리오필화된 프라이머가 포함되어 있는 경우 ddH2O의 450 μL을 추가하고 용액을 소용돌이시다.
- 프라이머 스톡 용액을 -20°C에 저장하거나 증폭을 계속 수행합니다.
3. PCR을 통해 유전자 단편 증폭
참고: 관심 유전자에 적합한 PCR 키트를 결정합니다. 더 작은 유전자 (<1,000 kb)는 더 싼 Taq 폴리머라아제에 더 순종적 일 수 있으며, 더 큰 유전자 (≥1,000 kb)는 오류를 줄이기 위해 높은 충실도 폴리머라제의 혜택을 누릴 수 있습니다. 사용자가 초기 유전자 단편 보존에 관심이 없는 경우 이 초기 PCR 증폭이 필요하지 않다는 점에 유의하는 것이 중요합니다(순환화에 여러 번 의한 시도를 제공하고 LET 대 RCA 제품의 비교 연구를 허용). 또한 이 PCR 증폭 LET이 반응에 직접 사용할 수 있다는 점에 유의하는 것이 중요합니다. 그러나, 소개에서 언급 한 바와 같이, 그것은 단지 추가 증폭 단계를 무시하는 경우에만 반응의 제한된 수를 허용 할 것이다. 소화 및 결찰은 직접 재중단 된 유전자 단편(57)에서 수행 될 수 있습니다 (확실한 경우, 추가 순환 스톡을 수행하기 위해 더 많은 LET이 필요하지 않습니다). 이 경우 섹션 3을 건너뛰고 섹션 4를 계속합니다. PCR을 수행하면 다음 단계를 따르십시오.
- 2.8 단계에서 100 μM 스톡을 사용하여 10 μM 작업 솔루션을 만듭니다. 많은 PCR 키트 프로토콜은 프라이머의 10 μM 솔루션을 요구합니다.
- 키트 제조업체의 프로토콜에 따라 반응을 수행하도록 열 사이클러를 프로그래밍합니다. 다른 키트는 약간 다양한 사이클링 매개 변수를 요구합니다. 재료의 표에나열된 키트의 경우, 조건은 초기 변성 30 s에 대한 94 °C입니다; 30초동안 94°C의 30사이클, 30초의 프라이머 어닐링, 68°C의 연장; 5 분 동안 68 °C에서 최종 연장; 그리고 마지막으로, 10 °C 무기한 홀드.
- 올바른 신장 시간을 선택해야 합니다(증폭되는 유전자의 길이에 따라 변수). 1,000 bp마다 신장 시간이 1분입니다.
- 프라이머에 대한 올바른 어닐링 온도를 입력해야 합니다. 두 프라이머를 입력으로 사용하여 최고의 어닐링 온도58을결정하는 온라인 TM 계산기를 사용합니다. M13 프라이머를 사용할 때 45°C의 어닐링 온도가 충분합니다.
- 사이클 수를 결정할 때 제조업체의 프로토콜을 참조하지만 30사이클은 대부분 충분한 증폭을 초래합니다.
- PCR을 수행하는 경우 dNTP를 해동하고 소용돌이시다. 키트에 제공된 PCR 버퍼를 사용합니다.
- 단일 PCR 튜브에서 제조업체의 프로토콜에 지시된 대로 모든 키트 구성 요소를 결합합니다. 성공적인 증폭을 위해, 1 μL의 암페어 DNA 스톡(2.6단계)을 추가합니다.
- 5-10s의 중간 설정에 대한 소용돌이를 통해 혼합물을 부드럽게 균질화합니다. 또는 피펫의 부피를 10-20배 위아래로 소용돌이로 반쪽.
- PCR 반응을 수행합니다.
- PCR 프로토콜에 최종 냉각 단계가 포함되지 않은 경우, 튜브의 바닥으로 응축을 구동하기 위해 제거하기 전에 10 °C에서 5 분 동안 반응을 냉각 할 수 있습니다.
- 공급업체의 지시에 따라 PCR 정리 키트를 사용하여 반응을 정화합니다.
- 1.5 mL 튜브에서 DNA 결합 버퍼 및 PCR 샘플을 각각 5:1의 비율로 추가합니다.
- 이 혼합물을 스핀 컬럼과 원심분리기로 1분 간 16,000 x g로 옮긴다. 흐름을 삭제합니다.
- 200 μL의 DNA 세척 버퍼를 컬럼에 넣고 실온에서 1분 동안 배양합니다.
- 원심분리기는 16,000 x g에서 1분 동안 원심분리기를 제거하고 흐름을 폐기합니다.
- 1분 배양 단계 없이 2.8.3 및 2.8.4 단계를 반복합니다.
- 16,000 x g에서 1-2분 추가로 원심분리기를 사용하여 남은 버퍼를 제거합니다.
- ddH2O의 46 μL에서 DNA를 엘테.
- 분광광계를 사용하여 정제된 DNA를 정량화합니다.
- 정제된 DNA를 -20°C에 저장하거나 다음 단계로 진행한다.
4. 소화 및 순환화
참고: RCA 다음에 DNA를 순환시킴으로써 추가 증폭을 달성할 수 있다. DNA를 소화하여 순환화를 위한 템플릿을 준비합니다. 이렇게 하면 프라이머 시퀀스가 제거되고 템플릿의 5'와 3'끝에 끈적끈적한 끝이 생성됩니다. 결찰 반응을 통해 이러한 끝을 다시 부착합니다.
- PCR 튜브에서 필요한 완충제의 5 μL, 힌드III 20U, 정제된 DNA45를 3.8단계에서 결합한다.
- 이 혼합물을 파이펫과 부드럽게 균질화합니다.
- 37°C에서 15분 동안 열 사이클러에 혼합물을 배양합니다.
- 열은 80 °C에서 20 분 동안 배양하여 힌드III를 비활성화합니다.
- 튜브의 바닥으로 응축을 구동하기 위해 제거하기 전에 10 °C로 반응을 냉각 할 수 있습니다.
- 새로 소화된 DNA에 T4 리구아제 완충제 5μL과 T4 리개세 800 U를 추가합니다.
- 원하는 경우 T7 리개세사용하십시오.
- 이 혼합물을 파이펫과 부드럽게 균질화합니다.
- 순환화 반응을 수행하기 위해 25°C에서 1시간 동안 혼합물을 배양한다.
- 공급업체의 지시에 따라 PCR 정리 키트를 사용하여 반응을 정화합니다. 3.8 단계에서 자세히 설명된 동일한 프로토콜을 사용합니다.
- 분광계를 사용하여 DNA를 정량화합니다. 예상 값은 ~20 ng/μL입니다.
- -20°C에 보관하거나 다음 단계로 진행하십시오.
5. 이더말 롤링 서클 증폭
참고: 롤링 서클 증폭(RCA)은 상용 키트 또는 개별적으로 구입한 구성 요소를 사용하여 수행할 수 있습니다. 제조업체의 프로토콜을 따라 성공적인 증폭을 보장합니다. 키트는 전형적으로 φ29 폴리머라제와 같은 시료 완충, 반응 버퍼 및 가닥 변위 폴리머라제를 함유하고 있다. 다중 반응 튜브는 세포 없는 발현을 위해 다량의 DNA를 생성하기 위해 결합될 수 있다(시작 물질의 20 pg로부터 4 μg). 다음 프로토콜은 효율적으로 작동합니다.
- 단일 튜브에서, 샘플 버퍼의 20 μL, 반응 버퍼의 20 μL, 효소의 0.8 μL, 및 4.9 단계에서 원형 발현 템플릿(CET)의 1 μL을 결합한다.
참고: 이것은 ~20 ng의 총 DNA 질량을 가질 것이지만, RCA는 picogram 양으로 작동할 수 있으므로 높은 처리량 스크리닝에서 물질에 대한 상당한 필요성이 있는 경우 CET 및 극단적 인 효소 증폭의 희석을 허용합니다. - 혼합물의 피펫과 알리쿼트 10 μL을 4개의 개별 튜브로 균질화한다.
- 4-18 h에 대한 30 °C에서 배양.
- 열은 10 분 동안 65 °C에서 배양하여 효소를 비활성화합니다. 튜브 의 바닥에 응축을 장려하기 위해 5 분 동안 온도를 12 °C로 줄입니다.
참고: 열 사이클러의 자동화된 프로토콜의 모든 온도 단계를 결합하는 것이 가장 쉽습니다. - 각 튜브에 ddH2O의 15 μL을 추가하여 결과 용액을 희석시 희석시.
- 모든 튜브를 결합하고 세포없는 반응에 직접 추가하십시오.
- 원하는 경우 PCR 정리 키트를 사용하여 제품을 정화하고 ddH2O의 36 μL로 정량화하십시오. 템플릿 농도가 ~100 ng/μL인지 확인합니다.
6. 세포 없는 반응
참고: 에너지 버퍼, 추출 및 RCA 템플릿을 결합하여 셀 프리 식을 수행합니다. PANOx-SP 에너지 버퍼를 이용한 전형적인 세포 없는 반응은 각각 1.2mM ATP, 0.85mMMM로 구성되어 있으며, GMP, UMP, 및 CMP, 30 mM 인포에놀피루바테, 130mM 칼륨 글루타민트, 10mM 암모늄 글루타메이트, 12m 마그네슘 글루타민, 1.5 mM 스미디딘, 1mM 퍼프레슈민, 34 μg/mL 의 폴리닉 산, 171 μg/mL 2mMM 각각 20개의 라벨이 없는 아미노산, 0.33m NAD, 0.27 mM 코엔자임 A(CoA), 4m 화소칼륨, 57m HEPES-KOH 버퍼(pH 7.5), 대장균 추출물의 0.24% 부피, 및 가변량 DNA23,49. 반응의 부피는 다를 수 있지만 15 μL 반응은 시약 사용에 저장할 수 있으며 384 개의 검은 벽 마이크로 플레이트49,50에사용하기에 충분히 작습니다.
- sfGFP와 같은 형광 단백질을 표현하는 경우, 플레이트 판독기를 준비하여 원하는 흥분/방출, 온도 및 동요에서 읽을 수 있습니다.
- 384웰 플레이트를 사용하는 경우, 습도를 유지하고 가장자리 효과를 줄이기 위해 빈 샘플에 잘 접한 우물에 H2O의 알리쿼트 60 μL을 제공합니다.
- 각 샘플에 대해 다양한 필수 구성 요소를 튜브에 추가합니다. 삼중을 수행 하기에 충분히 추가 합니다. 플레이트 내에서 복제하면 가변성의 원인을 식별하는 데 도움이 될 수 있습니다.
- 추출물, 에너지 버퍼 및 DNA를 추가합니다.
- ddH2O로 최종 원하는 볼륨에 희석합니다.
- 솔루션 볼륨의 절반을 10-20배 위아래로 파이프팅하여 이 솔루션을 완전히 혼합합니다.
- 15 μL 알리쿼트에서 반응 혼합물을 마이크로티터 플레이트의 원하는 우물로 옮기.
- 습도를 유지하고 증발을 방지하기 위해 무색 밀봉 필름으로 접시를 밀봉하십시오.
- 밀봉된 플레이트를 플레이트 판독기에 놓고 반응을 완료할 수 있습니다.
- 라이브 모니터링 능력이 없는 단백질을 발현하는 경우, 열막과 같은 다른 온도 조절 장치를 사용하여 플레이트를 배양한다.
7. 수틸리신 분석
참고: 보충 서열 #2에서서브틸리신 BPN(SBT(n)) 유전자를 발현하는 경우 이 프로토콜을 따라 활성을 분석합니다.
- 디메틸포르마미드(DMF)에서 N-수치닐-알라-알라-프로-페-니트로아닐드의 10μM 스톡 솔루션을 준비한다.
- 플레이트 판독기를 설정하여 25°C의 온도를 유지하면서 20초마다 410nm에서 흡광도를 측정합니다.
- 평평한 바닥, 무색 96 웰 플레이트, ddH2O의 알리쿼트 94 μL 및 N-수치닐-알라-알라-프로-페-프로-니트로아닐드의 7.1 단계.
- 6.7 단계에서 완성된 세포 없는 반응의 5μL을 추가하고 7.2 단계에서 설명된 프로토콜로 설정된 플레이트 판독기를 사용하여 읽는다.
결과
RCA 템플릿으로부터 sfGFP의 발현은 15 μL반응(도 2A)에서무정제 RCA DNA의 0.30 μL만을 사용할 때 pJL1 플라스미드의 것과 비교할 수 있었다. 사실, 템플릿의 양을 두 배로 늘리고 세 배로 늘리면 BL21 DE3 Star 추출물에 아무런 이점이 없는 것으로 보이며, 이는 반응당 0.30 μL로 템플릿의 이미 포화 수준을 시사합니다. 반대로, SHuffle 균주로부터 공급되는 세포 추출물에 첨가할 때 RCA 템플릿의 양을 증가시켜 주는 이점이 있는 것으로보인다(도 2B)28. 일부 단백질의 경우 결과를 매우 빠르게 관찰할 수 있으며 전체 워크플로우(증폭 및 속담)를 24시간 미만으로 압축합니다. 그러나, 몇몇 단백질은 더 낮은 온도를 요구하거나 더 느린 접이식 시간이 있습니다, 결과가 얻어질 때까지 시간을 증가하되 여기에 제시된 워크플로우에 영향을 미칠 것입니다. 이는 4h의 식 후 SBT(n) 성숙에 대해 충분히 길지 않은 서브틸리신(SBT(n)을 표현할 때 관찰될 수있다(도 3A,실패한 결과의 예). 반응을 계속 하도록 허용 16 h SBT의 검출 가능한 수준으로 이어질 수 있습니다 (n)(그림 3B). 이러한 개선은 최적화된 온도 조건이59,60을탐구한 문헌에서 관찰된 바와 같이 온도에 따라 달라질 수 있다.

도 1: 최소 유전 템플릿의 대표적인 회로도 및 초기 PCR 증폭 단계 후에 겪는 과정. 템플릿은 힌드III로 소화되고 T4 리게아제로 원형화되고 φ29 폴리머라제로 증폭되어 큰 컨케이트머를 만듭니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
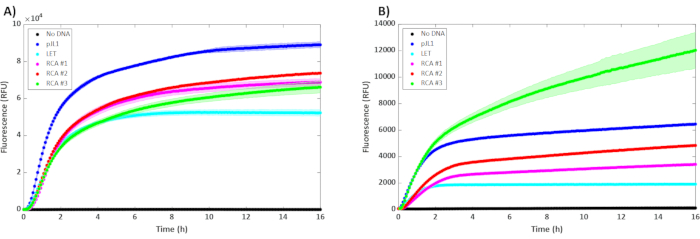
그림 2: 플라스미드(pJL1)의 불순화5nM, 선형 템플릿(LET) 5nM, 불순되지 않은 RCA의 다양한 농도를 가진 세포없는 반응의 결과. RCA #1, #2 및 #3에는 30°C(n=3)에서 배양된 15μL 반응에서 무정제 RCA 제품의 0.3 μL, 0.6 μL 및 0.9 μL(각각)이 포함되었다. 오류 막대는 평균에서 ± 1 SD를 나타냅니다. y축은 형광이며 x축은 반응 중에 경과한 시간입니다. sfGFP 발현의 운동은(A)BL21 DE3 스타 및(B)T7 SHuffle로 표현된다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: SBT (n) LET의 5 nM과 무분별한 RCA 제품의 다양한 농도를 가진 세포없는 반응. RCA ng 및 pg는 롤링 원 증폭을 수행하는 데 사용되는 DNA의 농도에 해당합니다. 무정화 RCA 제품은 30°C(n=3)에서 배양된 15μL 반응에 사용되었다. 오류 막대는 평균에서 ± 1 SD를 나타냅니다. y축은 410nm의 흡광도이며 x축은 분석에서 경과한 시간입니다. 반응은(A)4h 및(B)16 h. 이 그림의 더 큰 버전을 보려면 여기를 클릭하여 수행하였다.
| 복제 방법 | |
| PCR | 2 -4 h |
| 플라스미드 소화 | 약 35분 |
| 리기션 | 1h |
| 변형 | 2 h |
| 하룻밤 배양 | 16 h |
| 시퀀싱 | 24 - 48 h |
| 글리세롤 스톡 프렙 | 16 h |
| 성장과 정화 | 16 h |
| 총 시간 | 46 - 72 h |
| RCA 방법 | |
| PCR | 2 - 4 시간 |
| 소화 | 약 35분 |
| 리기션 | 1h |
| RCA | 4 - 18 h |
| CFE | 4 - 16 h |
| 총 시간 | 12 - 40 시간 |
표 1: 단순화된 기존 복제 프로토콜과 본원에 포함된 RCA 프로토콜 간의 타임라인 비교입니다.
보충 파일: 추가 파일에시 시퀀스가 나열됩니다. 시퀀스 #1은 sfGFP (999 베이스 쌍)이며 시퀀스 #2는 서브틸리신 BPN '(1344 bp)입니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
관심의 유전자는 원하는 단백질이 될 수 있지만, 이 방법의 새로운 채택자를위한 웰 플레이트 판독기에서 실시간 또는 엔드 포인트 판독을위한 편리한 리포터로서 형광 단백질로 시작하는 것이 가장 좋습니다. 새로운 단백질 서열의 경우 원하는 단백질의 아미노산 서열을 복사하여 원하는 코돈 최적화도구(61,62)에붙여 넣습니다. 코돈 최적화 도구에는 일반적으로 많은 사용 가능한 유기체와 대장균균균이 있지만 일반 대장균 옵션을 선택하는 것이 적합합니다. 최적화 후, 이전에 선택된 절단 부위및 프라이머 서열이 존재하지 않도록 유전자를 다시 확인한다. 그렇다면 시퀀스가 더 이상 존재하지 않을 때까지 시퀀스를 최적화할 수 있습니다. 경우에 따라 반복 최적화가 지속적으로 내부 HindIII 절단 사이트를 초래하는 경우 HindIII 절단 사이트를 다른 제한 사이트로 교체해야 할 수 있습니다. 공정을 표준화하고 여러 제한 효소를 유지하는 비용을 줄이기 위해 가능한 한 자주 동일한 절단 부위를 사용하는 것이 가장 좋습니다.
증폭을 시작하기 전에 LET에 가장 적합한 PCR 키트를 선택하십시오. <1,000개의 염기 쌍을 가진 LET은 간단한 Taq 폴리머라제로 증폭될 수 있으며, ≥ 1,000개의 염기 쌍을 가진 LET는 더 높은 충실도폴리머라제(63)를필요로 할 수 있다. 키트 제조업체및 원하는 프라이머의 어닐링 온도에 따라 증폭을 위한 프로토콜을 설정합니다. 어닐링 온도는 성공적인 증폭에 매우 중요합니다. 엄지 손가락의 일반적인 규칙은 프라이머의 Tm보다 5 °C 낮은 어닐링 온도를 선택하는 것입니다. 프라이머58의시퀀스에 따라 최적화된 어닐링 온도를 제공하는 무료 온라인 도구가 있습니다. 올바른 어닐링 온도를 사용하는 것은 고품질 의 DNA 템플릿을 생성하는 데 중요합니다.
세포 없는 유전자 발현은 합성 생물학 프로토타이핑을 위한 속도, 단순성 및 유용성 때문에 최근 몇 년 동안 르네상스를 보았습니다. 이 연구는 새로운 기능성 단백질의 큰 라이브러리를 테스트 할 때 속도와 용이성을 증가시키는 방법을 설명했다. 기존의 복제 방법은 며칠 또는 몇 주가 걸릴 수 있지만 이 프로토콜은 24시간 미만에서 수행할 수 있습니다. 표 1은 기존 복제 및 RCA 프로토콜 모두에 대한 일반적인 시간 범위를 간략하게 설명합니다. 복제 프로토콜이 단순화되었으며 젤 격리와 같은 몇 가지 선택적 단계가 제외되었습니다. 표 1은 또한 일반적인 제한 소화 프로토콜을 말합니다. 다른 많은 복제 방법이 있지만 비슷한 시간이 필요합니다. 이 효소 증폭 프로토콜의 상범위는 특히 8시간 근무일을 고려할 때 복제 방법의 낮은 범위보다 적은 시간이 필요합니다. 이것은 주로 하룻밤 잠복의 제거 및 시퀀싱 확인의 부족 때문입니다. RCA 프로토콜은 전적으로 PCR 및 RCA를 기반으로 하며, 기존의 복제, 변환 및 세포 배양 기술보다 덜 전문적인 기술이 필요합니다. 이 방법을 사용하면 셀을 변환하고 성장하는 데 필요한 복제 또는 자본에 대한 사전 경험이 없는 실험실에서 세포 없는 식에 액세스할 수 있습니다. 이 방법은 또한 세포 독성 단백질에 초점을 맞춘 프로젝트에 적합, 여기서 복제 및 전통적인 복제 유전자의 전파는 살아있는 세포에 독성으로 인해 어렵다. 이 RCA 프로토콜은 또한 CFE에 필요한 수준으로 매우 적은 양의 원형 DNA (DNA의 picograms)를 증폭 할 수 있습니다. 도 3B에서,원형화된 SBT(n) 템플릿은 RCA 이전에 20 ng/μL 및 20 pg/uL로 희석되었다. 분해율이 관찰된 반면, 두 반응 모두 10분 이내에 동일한 양의 기질 분해를 초래했습니다. 제안된 메서드는 복제를 대체하기 위한 것이 아닙니다. 플라스미드 전파는 일치할 수 없는 비독성 유전자에 대한 DNA의 양을 생성할 수 있습니다. 오히려, 이것은 대규모 라이브러리 스크리닝 단계에서 편리한 프로토타이핑 도구입니다 (효소 단계는 표준 유체 처리기로 자동화할 수 있음) 보관 및 추가 전파를 위해 복제해야 하는 시퀀스를 식별하는 데 도움이 됩니다.
세포 없는 반응을 설계할 때 많은 유연성이 있지만 성공을 보장하기 위해 고려해야 할 몇 가지 요인이 있습니다. 반응이 작을수록 체적 비율이 더 크며 가스 교환에 적합하지만 정확한 형광 측정을 위해 반응은 우물 바닥을 완전히 커버해야 합니다. 반응의 온도는 또한 관심있는 유전자에 따라 달라질 수있습니다(59). 에너지 버퍼34,46의선택에 관해서 사용자에 대 한 여러 선택이 있다. 일부 조리법은 다른 조리법보다 더 비싸지 만 결정은 사용자에게 맡지 않습니다. 또한 대장균 추출물(36)에대한 많은 잠재적인 소스가 있다. 사용자는 추출 생산 프로토콜에 익숙해지고 자신의 목적에 가장 적합한 결정해야한다28,48,50,64,65,66,67.
RCA를 사용하여 세포 없는 발현을 위한 템플릿을 증폭할 때 세균균균의 선택은 매우 중요합니다. 식 프로파일은 LED보다 더 나은 경향이 있습니다. 인기 있는 BL21 DE3 스타 스트레인은 RCA가 LET보다 ~1.4배 더 우수하며 pJL1 플라스미드가 최고 RCA농도(도 2A)보다1.2배 더 우수하다. 한편, 일부 균주는 템플릿 분해를 나타내고 네이티브 뉴클레아제(23,28)의존재로 인해 제대로 수행되지 않았다. 이 경우 SHuffle 변형이 증가된 RCA 템플릿의 이점을 제공하는 것으로 보입니다. 이전 문헌은 SHuffle 추출물이 PCR 제품과 잘 작동하지 않지만, 본 연구에서 사용되는 무정제 RCA 제품의 가장 높은 농도가 pJL1 플라스미드(도2B)를능가하는 것으로 나타났다. 기능성 SBT(n)(도3)의발현은 독성으로 인해 살아있는 세포에서 시제품하기 어렵지만 세포가 없지만 편리하게 만들어지지만(이 발현 템플릿을 염균에서 복제및 대장균에서 전파할 수 없음)의 예이다. sfGFP와 달리 SBT(n) 활동은 4h의식(도 3A)만후에 관찰할 수 없습니다. 신호는 발현 및 성숙의 16 h 후 감지(도 3B). 이 예에 사용된 무정제 DNA는 100 μL에서 나왔으며, 이는 4개의 10 μL RCA 반응이 희석되고 결합되었다. 이 한 스톡은 반응당 0.30 μL만 사용하는 경우 333개의 세포 없는 반응을 지원할 수 있습니다.
CFE와 RCA 템플릿의 호환성은 다양한 추출과 함께 더 탐구할 필요가 있습니다. 선형 제품을 가진 잠재적인 합병증은 대장균 치 서열 또는 GamS단백질(52,53)을포함하는 짧은 올리고를 첨가함으로써 완화될 수 있다. 문헌은 치 올리고스를 첨가하면 GamS단백질(53)보다선형 템플릿에 더 큰 보호를 제공할 수 있음을 시사한다. 선형 템플릿에서 낮은 수율을 제공하는 것으로 알려진 추출을 사용하는 경우 사용자는 각 보호 제의 비용을 분석하고 자신의 목적에 가장 적합한 추출을 사용해야 합니다. 또 다른 제한은 결과 컨케이트의 특성으로 인해 RCA 템플릿 농도를 어금니로 변환할 수 없다는 것입니다. 즉, 질량에 따라 동일한 수의 최소 템플릿을 추가할 수 있지만 표현식 수준에 영향을 줄 수 있는 다양한 연결 길이가 있습니다. 저자는 각 우물에서 평균 적인 표현 수준이 동일하기 때문에 선별/프로토타이핑에 있는 문제점이 찾아낸 것은 (낮은 분산) 그러나 작은 볼륨 (예를 들면, microdroplets)에서 표현하는 경우에 문제가 될 수 있었습니다.
세포 없는 유전자 발현은 단백질 프로토타이핑 및 설계-빌드 테스트 워크플로우에서 중요한 도구로 사용될 가능성이 있습니다. 그러나 대부분의 세포 없는 발현 워크플로우는 여전히 전통적인 플라스미드에 유전 적 템플릿으로 의존합니다. 이렇게 하면 프로세스가 느려지고 세포 없는 표현이 스크리닝/프로토타이핑 목적으로 최대한 활용되지 않도록 합니다. 템플릿을 증폭하고 이후에 RCA를 수행하면 많은 반응을 위한 충분한 유전 템플릿을 신속하게 생성하여 다운스트림 특성화 및 기능 성 테스트를 위한 충분한 단백질을 생성할 수 있습니다.
공개
Nigel Reuel은 항체 설계를 위해 세포없는 시스템을 사용하는 회사인 BigHat Biosciences Inc.의 과학 자문 위원회에서 근무하고 있습니다.
감사의 말
저자는 NIH 1R35GM138265-01 및 NSF 2029532 이 프로젝트의 부분적인 지원을 인정합니다.
자료
| Name | Company | Catalog Number | Comments |
| Alaline | Formedium | DOC0102 | |
| Ammonium glutamate | MP Biomedicals | MP21805951 | |
| Arginine | Formedium | DOC0106 | |
| Asparagine | Formedium | DOC0114 | |
| Aspartic Acid | Formedium | DOC0118 | |
| ATP | Sigma | A2383 | |
| Axygen Sealing Film | Corning | PCR-SP | |
| CMP | Sigma | C1006 | |
| Coenzyme A | Sigma | C3144 | |
| CutSmart Buffer | NEB | B7204S | Provided with HindIII |
| Cysteine | Formedium | DOC0122 | |
| DNA Clean and Concentrator Kit | Zymo Research | D4004 | Used for purifying DNA |
| dNTPs | NEB | N0447 | |
| E. coli tRNA | Sigma (Roche) | 10109541001 | |
| Folinic Acid | Sigma | 47612 | |
| Gene Fragment | IDT | ||
| Glutamic Acid | Formedium | DOC0134 | |
| Glutamine | Formedium | DOC0130 | |
| Glycine | Formedium | DOC0138 | |
| GMP | Sigma | G8377 | |
| HEPES | Sigma | H3375 | |
| HindIII-HF | NEB | R3104L | |
| Histidine | Formedium | DOC0142 | |
| Isoleucine | Formedium | DOC0150 | |
| Leucine | Formedium | DOC0154 | |
| Lysine | Formedium | DOC0158 | |
| Magnesium glutamate | Sigma | 49605 | |
| Methionine | Formedium | DOC0166 | |
| Microtiter Plate (384 well) | Greiner | 781906 | |
| Microtiter Plate (96 well) | Greiner | 655809 | |
| Multimode Plate Reader | BioTek | Synergy Neo2 | |
| NAD | Sigma | N8535 | |
| NanoPhotometer | Implen | NP80 | |
| OneTaq DNA Polymerase | NEB | M0480 | |
| PCR Tube | VWR | 20170-012 | |
| Phenylalanine | Formedium | DOC0170 | |
| Phosphoenolpyruvate | Sigma (Roche) | 10108294 | |
| Potassium glutamate | Sigma | G1501 | |
| Potassium oxalate | Fisher Scientific | P273 | |
| Proline | Formedium | DOC0174 | |
| Putrescine | Sigma | P5780 | |
| Serine | Formedium | DOC0178 | |
| Spermidine | Sigma | S0266 | |
| T4 DNA Ligase | NEB | M0202S | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | Provided with T4 DNA Ligase |
| TempliPhi Amplification Kit | Cytiva | 25640010 | Used for RCA |
| Thermal Cycler | Biorad | C1000 Touch | |
| Thermoblock | Eppendorf | ThermoMixer FP | |
| Threonine | Formedium | DOC0182 | |
| Tryptophan | Formedium | DOC0186 | |
| Tyrosine | Formedium | DOC0190 | |
| UMP | Sigma | U6375 | |
| Valine | Formedium | DOC0194 |
참고문헌
- Sun, Q., et al. A simple and low-cost paper-based colorimetric method for detecting and distinguishing the GII.4 and GII.17 genotypes of norovirus. Talanta. 225, 121978 (2021).
- Pardee, K., et al. Low-Cost Detection of Zika Virus Using Programmable Biomolecular Components. Cell. 165 (5), 1255-1266 (2016).
- Pardee, K., et al. Paper-based synthetic gene networks. Cell. 159 (4), 940-954 (2014).
- Ma, D., Shen, L., Wu, K., Diehnelt, C. W., Green, A. A. Low-cost detection of norovirus using paper-based cell-free systems and synbody-based viral enrichment. Synthetic Biology. 3 (1), (2018).
- Park, S., Lee, J. W. Detection of coronaviruses using rna toehold switch sensors. International Journal of Molecular Sciences. 22 (4), 1772 (2021).
- Cao, M., Sun, Q., Zhang, X., Ma, Y., Wang, J. Detection and differentiation of respiratory syncytial virus subgroups A and B with colorimetric toehold switch sensors in a paper-based cell-free system. Biosensors and Bioelectronics. 182, 113173 (2021).
- Mcnerney, M. P., et al. Point-of-care biomarker quantification enabled by sample-specific calibration. Science Advances. 5 (9), (2019).
- Silverman, A. D., Akova, U., Alam, K. K., Jewett, M. C., Lucks, J. B. Design and optimization of a cell-free atrazine biosensor. ACS Synthetic Biology. 9 (3), 671-677 (2020).
- Salehi, A. S. M., et al. Cell-free protein synthesis approach to biosensing hTRβ-specific endocrine disruptors. Analytical Chemistry. 89 (6), 3395-3401 (2017).
- Garamella, J., Majumder, S., Liu, A. P., Noireaux, V. An adaptive synthetic cell based on mechanosensing, biosensing, and inducible gene circuits. ACS Synthetic Biology. 8 (8), 1913-1920 (2019).
- Glasscock, C. J., et al. Dynamic control of pathway expression with riboregulated switchable feedback promoters. ACS Synthetic Biology. 16, (2019).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- Pardee, K., et al. Portable, on-demand biomolecular manufacturing. Cell. 167 (1), 248-259 (2016).
- Nelson, J. A. D., et al. Hydrofoam and oxygen headspace bioreactors improve cell-free therapeutic protein production yields through enhanced oxygen transport. Biotechnology Progress. 37 (2), 3079 (2020).
- Cai, Q., et al. A simplified and robust protocol for immunoglobulin expression in Escherichia coli cell-free protein synthesis systems. Biotechnology Progress. 31 (3), 823-831 (2015).
- Ogonah, O. W., Polizzi, K. M., Bracewell, D. G. Cell free protein synthesis: a viable option for stratified medicines manufacturing? A brief history of cell free synthesis systems. Current Opinion in Chemical Engineering. 18, 77-83 (2017).
- Zawada, J. F., et al. Microscale to manufacturing scale-up of cell-free cytokine production-a new approach for shortening protein production development timelines. Biotechnology and Bioengineering. 108 (7), 1570-1578 (2011).
- Stark, J. C., et al. On-demand biomanufacturing of protective conjugate vaccines. Science Advances. 7 (6), (2021).
- Huang, A., et al. BioBitsTM Explorer: A modular synthetic biology education kit. Science Advances. 4 (8), 1-11 (2018).
- Stark, J. C., et al. BioBits health: classroom activities exploring engineering, biology, and human health with fluorescent readouts. ACS Synthetic Biology. 8 (5), 1001-1009 (2019).
- Stark, J. C., et al. BioBitsTM Bright: A fluorescent synthetic biology education kit. Science Advances. 4 (8), 33 (2018).
- Shinoda, T., et al. Cell-free methods to produce structurally intact mammalian membrane proteins. Scientific Reports. 6, (2016).
- Dopp, J. L., Rothstein, S. M., Mansell, T. J., Reuel, N. F. Rapid prototyping of proteins: Mail order gene fragments to assayable proteins within 24 hours. Biotechnology and Bioengineering. 116 (3), 667-676 (2019).
- Sachse, R., Dondapati, S. K., Fenz, S. F., Schmidt, T., Kubick, S. Membrane protein synthesis in cell-free systems: From bio-mimetic systems to bio-membranes. FEBS Letters. 588 (17), 2774-2781 (2014).
- Salehi, A. S. M., et al. Cell-free protein synthesis of a cytotoxic cancer therapeutic: Onconase production and a just-add-water cell-free system. Biotechnology Journal. 11 (2), 274-281 (2016).
- Georgi, V., et al. On-chip automation of cell-free protein synthesis: New opportunities due to a novel reaction mode. Lab on a Chip. 16 (2), 269-281 (2016).
- Thoring, L., et al. Cell-free systems based on CHO cell lysates: Optimization strategies, synthesis of "difficult-to-express" proteins and future perspectives. PLoS One. 11 (9), (2016).
- Dopp, J. L., Reuel, N. F. Simple, functional, inexpensive cell extract for in vitro prototyping of proteins with disulfide bonds. Biochemical Engineering Journal. 164, 107790 (2020).
- Isaksson, L., Enberg, J., Neutze, R., Göran Karlsson, B., Pedersen, A. Expression screening of membrane proteins with cell-free protein synthesis. Protein Expression and Purification. 82 (1), 218-225 (2012).
- Techner, J. M., et al. High-throughput synthesis and analysis of intact glycoproteins using SAMDI-MS. Analytical Chemistry. 92 (2), 1963-1971 (2020).
- Kim, H. C., et al. Implementing bacterial acid resistance into cell-free protein synthesis for buffer-free expression and screening of enzymes. Biotechnology and Bioengineering. 112 (12), 2630-2635 (2015).
- Rolf, J., Siedentop, R., Lütz, S., Rosenthal, K. Screening and identification of novel cGAS homologues using a combination of in vitro and in vivo protein synthesis. International Journal of Molecular Sciences. 21 (1), (2020).
- Haslinger, K., Hackl, T., Prather, K. L. J. Rapid in vitro prototyping of O-methyltransferases for pathway applications in Escherichia coli. bioRxiv. , (2020).
- Dopp, J. L., Tamiev, D. D., Reuel, N. F. Cell-free supplement mixtures: Elucidating the history and biochemical utility of additives used to support in vitro protein synthesis in E. coli extract. Biotechnology Advances. 37 (1), 246-258 (2018).
- Gregorio, N. E., Levine, M. Z., Oza, J. P. A user's guide to cell-free protein synthesis. Methods and Protocols. 2 (1), 24 (2019).
- Cole, S. D., Miklos, A. E., Chiao, A. C., Sun, Z. Z., Lux, M. W. Methodologies for preparation of prokaryotic extracts for cell-free expression systems. Synthetic and Systems Biotechnology. 5 (4), 252-267 (2020).
- Chiba, C. H., Knirsch, M. C., Azzoni, A. R., Moreira, A. R., Stephano, M. A. Cell-free protein synthesis: advances on production process for biopharmaceuticals and immunobiological products. BioTechniques. 70, (2021).
- Laohakunakorn, N. Cell-free systems: A proving ground for rational biodesign. Frontiers in Bioengineering and Biotechnology. 8, 788 (2020).
- Dondapati, S. K., Stech, M., Zemella, A., Kubick, S. Cell-free protein synthesis: A promising option for future drug development. BioDrugs. , 1-22 (2020).
- Noireaux, V., Liu, A. P. The new age of cell-free biology. Annual Review of Biomedical Engineering. 22, 51-77 (2020).
- Khambhati, K., et al. Exploring the Potential of Cell-Free Protein Synthesis for Extending the Abilities of Biological Systems. Frontiers in Bioengineering and Biotechnology. 7, (2019).
- Carlson, E. D., Gan, R., Hodgman, C. E., Jewett, M. C. Cell-free protein synthesis: Applications come of age. Biotechnology Advances. 30 (5), 1185-1194 (2012).
- Rosenblum, G., Cooperman, B. S. Engine out of the chassis: Cell-free protein synthesis and its uses. FEBS Letters. 588 (2), 261-268 (2014).
- Swartz, J. R. Transforming biochemical engineering with cell-free biology. AIChE Journal. 58 (1), 5-13 (2012).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Caschera, F., Noireaux, V. A cost-effective polyphosphate-based metabolism fuels an all E. coli cell-free expression system. Metabolic Engineering. 27, 29-37 (2015).
- Levine, M. Z., et al. Activation of energy metabolism through growth media reformulation enables a 24-hour workflow for cell-free expression. ACS Synthetic Biology. 9 (10), 2765-2774 (2020).
- Hunt, J. P., et al. Streamlining the preparation of "endotoxin-free" ClearColi cell extract with autoinduction media for cell-free protein synthesis of the therapeutic protein crisantaspase. Synthetic and Systems Biotechnology. 4 (4), 220-224 (2019).
- Dopp, J. L., Jo, Y. R., Reuel, N. F. Methods to reduce variability in E. Coli-based cell-free protein expression experiments. Synthetic and Systems Biotechnology. 4 (4), 204-211 (2019).
- Sun, Z. Z., et al. Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology. Journal of Visualized Experiments: JoVE. , e50762 (2013).
- Schinn, S. M., Broadbent, A., Bradley, W. T., Bundy, B. C. Protein synthesis directly from PCR: Progress and applications of cell-free protein synthesis with linear DNA. New Biotechnology. 33 (4), 480-487 (2016).
- Sitaraman, K., et al. A novel cell-free protein synthesis system. Journal of Biotechnology. 110 (3), 257-263 (2004).
- Marshall, R., Maxwell, C. S., Collins, S. P., Beisel, C. L., Noireaux, V. Short DNA containing χ sites enhances DNA stability and gene expression in E. coli cell-free transcription-translation systems. Biotechnology and Bioengineering. 114, 2137-2141 (2017).
- Sun, Z. Z., Yeung, E., Hayes, C. A., Noireaux, V., Murray, R. M. Linear DNA for rapid prototyping of synthetic biological circuits in an escherichia coli based TX-TL cell-free system. ACS Synthetic Biology. 3 (6), 387-397 (2014).
- . Addgene: pJL1 Available from: https://www.addgene.org/69496/ (2021)
- . IDT Codon Optimization Tool Available from: https://www.idtdna.com/pages/tools/codon-optimization-tool (2021)
- Hadi, T., et al. Rolling circle amplification of synthetic DNA accelerates biocatalytic determination of enzyme activity relative to conventional methods. Scientific Reports. 10 (1), 10279 (2020).
- . New England Biolabs Tm Calculator Available from: https://tmcalculator.neb.com/#!/main (2021)
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. Coli RNA polymerase and sigma factor 70. Journal of Biological Engineering. 4, (2010).
- Colant, N., et al. A rational approach to improving titer in Escherichia coli-based cell-free protein synthesis reactions. Biotechnology Progress. 37 (1), 3062 (2021).
- Burgess-Brown, N. A., et al. Codon optimization can improve expression of human genes in Escherichia coli: A multi-gene study. Protein Expression and Purification. 59, 94-102 (2008).
- Maertens, B., et al. Gene optimization mechanisms: A multi-gene study reveals a high success rate of full-length human proteins expressed in Escherichia coli. Protein Science. 19 (7), 1312-1326 (2010).
- Eckert, K. A., Kunkel, T. A. DNA polymerase fidelity and the polymerase chain reaction. Genome Research. 1 (1), 17-24 (1991).
- Dopp, J. L., Reuel, N. F. Process optimization for scalable E. coli extract preparation for cell-free protein synthesis. Biochemical Engineering Journal. 138, 21-28 (2018).
- Liu, D. V., Zawada, J. F., Swartz, J. R. Streamlining Escherichia Coli S30 extract preparation for economical cell-free protein synthesis. Biotechnology Progress. 21 (2), 460-465 (2005).
- Levine, M. Z., Gregorio, N. E., Jewett, M. C., Watts, K. R., Oza, J. P. Escherichia coli-Based Cell-Free Protein Synthesis: Protocols for a robust, flexible, and accessible platform technology. Journal of Visualized Experiments: JoVE. (144), e58882 (2019).
- Kwon, Y. C., Jewett, M. C. High-throughput preparation methods of crude extract for robust cell-free protein synthesis. Scientific Reports. 5, (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
ISSN 2578-2614
Copyright © 2025 MyJoVE Corporation. 판권 소유
당사 웹 사이트에서는 사용자의 경험을 향상시키기 위해 쿠키를 사용합니다.
당사 웹 사이트를 계속 사용하거나 '계속'을 클릭하는 것은 당사 쿠키 수락에 동의하는 것을 의미합니다.
