Method Article
Schnelle, enzymatische Methoden zur Amplifikation minimaler, linearer Templates für das Protein-Prototyping mit zellfreien Systemen
In diesem Artikel
Zusammenfassung
Die Studie beschreibt ein Protokoll zur Erzeugung großer (μg-mg) Mengen an DNA für Protein-Screening-Kampagnen aus synthetischen Genfragmenten ohne Klonen oder Verwendung lebender Zellen. Die minimale Schablone wird enzymatisch verdaut und zirkuliert und dann mit isothermer Rollkreisamplifikation amplifiziert. Zellfreie Expressionsreaktionen könnten mit dem ungemeinigten Produkt durchgeführt werden.
Zusammenfassung
Dieses Protokoll beschreibt das Design einer minimalen DNA-Schablone und die Schritte zur enzymatischen Amplifikation, die ein schnelles Prototyping von assaybaren Proteinen in weniger als 24 h unter verwendung zellfreier Expression ermöglicht. Nach Erhalt der DNA von einem Anbieter wird das Genfragment PCR-amplifiziert, geschnitten, zirkuliert und kryo-bankiert. Eine kleine Menge der bankierten DNA wird dann verdünnt und signifikant (bis zu 106x) mittels isothermer Rollkreisamplifikation (RCA) amplifiziert. RCA kann Mikrogrammmengen der minimalen Expressionsvorlage aus Pikogrammwerten des Ausgangsmaterials liefern (mg-Werte, wenn alle synthetischen Ausgangsfragmente verwendet werden). Bei dieser Arbeit ergab eine Ausgangsmenge von 20 pg 4 μg des Endprodukts. Das resultierende Cinch-Produkt (Konatemer der minimalen Schablone) kann ohne Reinigungsschritte direkt zu einer zellfreien Reaktion hinzugefügt werden. Da diese Methode vollständig PCR-basiert ist, kann sie in Verbindung mit automatisierten Liquid-Handling-Systemen zukünftige Hochdurchsatz-Screening-Bemühungen ermöglichen.
Einleitung
Die zellfreie Genexpression (CFE) hat sich zu einem leistungsstarken Werkzeug mit vielen Anwendungen entwickelt. Solche Anwendungen umfassen krankheitsnachweis1,2,3,4,5,6, Mikronährstoff- und niedermolekulare Detektion7,8,9,10,11,12 , Bioproduktion13,14,15,16,17 ,18, Ausbildung19,20,21, Herstellung schwieriger Proteine17,22,23,24,25,26,27, und Variantenscreening23,28,29,30,31,32 ,33. Dies liegt an der Offenheit zellfreier Systeme und der Flexibilität, die sie bieten. Viele großartige Übersichtsartikel bieten historische Bildung und Zukunftsperspektiven auf die Technologie34,35,36,37,38,39,40,41,42,43,44.
Eine typische zellfreie Reaktion besteht aus drei Hauptkomponenten: Zellextrakt, Energiemix und genetische Vorlage. Aktiver Zellextrakt enthält alle notwendigen Maschinen für Transkription und Translation (TXTL) und kann auf vielfältige Weise verarbeitet werden36. Glykolytische Zwischenprodukte, Elektrolyte, Aminosäuren und Cofaktoren im Energiemix unterstützen den TXTL-Prozess. Es ist eine Hauptquelle der Variabilität in zellfreien Experimenten45 und kann auf viele Arten hergestellt werden34,46. Die Präparation der genetischen Vorlage hat weniger Verbesserungen erfahren, da traditionelle Klonierungsmethoden zu Plasmiden mit hervorragenden Expressionseigenschaften führen. Der Nachteil dieser traditionellen Methoden ist die Durchlaufzeit und die Menge an biologischem Fachwissen, die erforderlich sind, um sie zu konstruieren und zu vermehren. Jüngste Optimierungsbemühungen haben zu einfachen 24-Stunden-Workflows für die Zellextraktaufbereitung47,48 geführt, die parallel zur Energiemixaufbereitung49,50durchgeführt werden können. Das herkömmliche Klonen fügt jedoch dem CFE-Prototyping-Zeitplan mehrere Tage hinzu (Tabelle 1)23. Schnell amplifizierte PCR-Produkte aus dem kommerziellen Genfragment können direkt verwendet werden51, dies begrenzt jedoch die Anzahl der Prototyping-Experimente, da nur 1 μg DNA produziert wird, was etwa fünf Reaktionen entspricht (traditionelle 15 μL-Volumina). Mit diesen zusätzlichen Schritten der Zirkularisierung und isothermen Amplifikation sind größere Mengen als Milligramm der DNA möglich (~5.000 Reaktionen für 1 mg). Dies erhöht die Anzahl der Tests, die im Hochdurchsatz-Screening von Proteinen oder kombinatorischen Enzymnetzwerken (zellfreies Metabolic Engineering) durchgeführt werden können, drastisch; Es ermöglicht auch eine effektive Erhaltung der linearen Template-Bibliothek als hochkonzentrierte DNA. Darüber hinaus wäre eine erhöhte Menge an Vorlagen erforderlich, um größere Mengen an Protein zu prototypisieren, die für materialwissenschaftliche Anwendungen benötigt werden (proteinbasierte Fasern und Hydrogele). Einige Einschränkungen linearer Schablonen können überwunden werden, indem ein Extrakt aus BL21 DE3 Star verwendet wird oder kürzlich entdeckte Methoden verwendet werden, um lineare Schablonen vor Degradation zu schützen52,53,54. Dies betrifft jedoch nicht den begrenzten Bestand an vom Hersteller produzierter DNA für die PCR-Amplifikation oder die Frage des biologischen Fachwissens und der für das Klonen erforderlichen Ausrüstung.
Diese Arbeit stellt ein Protokoll vor, das explizit entwickelt wurde, um die Menge an Expressionsvorlage zu erhöhen, die aus kleinen Mengen von vom Hersteller produzierten Genfragmenten (typischerweise 500-1000 ng lyophilisiertem Pulver) gewonnen werden kann. Die beschriebene Methode erfordert nicht die notwendigen Fähigkeiten, um traditionelles Klonen in Plasmiden oder Transformieren und Vermehren in lebenden Zellen durchzuführen. Nach Erhalt eines Genfragments in der Post kann ein Benutzer genügend Vorlagen für viele zellfreie Reaktionen erstellen, indem er die isotherme Rollkreisamplifikation (RCA) einsetzt (Abbildung 1)23. Während die Menge an DNA, die vom Anbieter erhalten wird, für begrenzte Screening-Bemühungen ausreichen kann, ist sie schnell erschöpft, und der erneute Kauf von Genfragmenten ist zeitaufwendig und kostspielig. Die Methode eignet sich auch besonders gut für Gene, die in E. colitoxisch und schwer zu klonen sind.
Protokoll
1. Gestaltung des Genfragments
HINWEIS: Das Genfragment sollte alle notwendigen genetischen Elemente für die Transkription/Translation enthalten, einschließlich Promotor, Ribosomenbindungsstelle (RBS), Startcodon, das interessierende Gen und Terminator. Während der Terminator für eine lineare Ausdrucksvorlage (LET) nicht erforderlich ist, ist es wichtig, wenn der Benutzer beschließt, die Sequenz in ein Plasmid einzufügen. Diese Sequenzen wurden aus dem pJL1-sfGFP-Plasmid55 (Geschenk aus dem Labor von Michael Jewett) entnommen, das einen T7-Promotor verwendet. Zusätzlich zu diesen notwendigen genetischen Elementen wird eine Restriktionsenzym-Schnittstelle sechs Basenpaare vor dem Promotor (5'-Schnittstelle) und weitere sechs Basenpaare nach dem Terminator (3'-Schnittstelle) hinzugefügt, in diesem Fall unter Verwendung von HindIII (andere Restriktionsenzyme können verwendet werden, aber es ist hilfreich, die Sequenzen mit einem High-Fidelity-Restriktionsenzym zu standardisieren, um die Anzahl zu reduzieren, die in der Bibliothek aufbewahrt werden muss). Primer-Sites werden zehn Basenpaare stromaufwärts der 5'-Cut-Site und zehn Base-Paare stromabwärts der 3'-Cut-Site hinzugefügt, in diesem Fall unter Verwendung standardisierter M13-Primer-Sequenzen (Primer sind preiswerte Lagerartikel). Die verwendete Restriktionsenzymstelle und die verwendeten Primer liegen im Ermessen des Benutzers. Der Benutzer muss jedoch sicherstellen, dass die Sequenzen an keiner anderen Stelle in der Vorlage vorhanden sind (Sie möchten keine unerwünschten Schnitte oder Orte der Verstärkungsinitiierung erstellen). Die Sequenzen für die in dieser Arbeit verwendeten Vorlagen sind im Ergänzungsmaterial detailliert beschrieben. Diese Schritte werden verwendet, um von dieser Basisvorlage aus zu ändern.
- Bestimmen Sie das gewünschte Gen, das exprimiert werden soll, und erhalten Sie die Aminosäuresequenz oder die genetische Sequenz, wenn es in E. coliexprimiert wurde.
- Wenn es sich um eine Aminosäuresequenz handelt, führen Sie eine Codonoptimierung für E. coli mit einem der vielen Standard-Tools des Anbietersdurch 56. Wenn Sie die in der Ergänzung bereitgestellte Vorlage verwenden, stellen Sie sicher, dass die optimierte Sequenz keine HindIII-Einschränkungsstellen (AAGCTT) aufweist. Falls dies der Fall ist, optimieren Sie die Sequenz so lange, bis keine HindIII-Site mehr vorhanden ist.
- Kopieren Sie die Sequenz und fügen Sie sie in die bereitgestellte Vorlage für Supplementary Sequence #1 ein, in der das interessierende Gen angegeben ist. Wenn Sie sfGFP ausdrücken, verwenden Sie Die ergänzende Sequenz Nr. 1 unverändert. Wenn Sie Subtilisin ausdrücken, verwenden Sie die ergänzende Sequenz #2 so, wie sie ist.
- Bestellen Sie die minimale Schablone und die notwendigen Primer beim bevorzugten DNA-Synthesedienst.
2. Wiederverwendung des Genfragments und der Primer
HINWEIS: Befolgen Sie nach Erhalt des Genfragments die Protokolle des Herstellers für die Resuspension oder verwenden Sie diese einfache Anleitung, um einen DNA-Bestand zu erstellen.
- Zentrifugieren Sie das Röhrchen (300 x g für 5 s), um das DNA-Pellet am Boden zu sammeln.
- Fügen Sie doppelt destilliertes Wasser (ddH2O) hinzu, um eine Endkonzentration von 10 ng / μL DNA-Vorlage zu erhalten.
- Wirbeln Sie die Lösung auf einer mittleren Einstellung für 5-10 s.
- Lösen Sie das gesamte Pellet durch Inkubation bei 50 °C für 20 min auf.
- Wieder kurz Wirbel
- Zentrifuge bei 300 x g für 5 s, um die Lösung am Boden des Röhrchens zu sammeln.
- Bei -20 °C lagern oder in PCR anwenden.
- Bereiten Sie einen 100 μM Primervorrat vor, indem Sie die Primer in nukleasefreiem Wasser wiederverwenden. Um die Menge an hinzuzufügendem Wasser zu bestimmen, multiplizieren Sie die Anzahl der Nanomole lyophilisierter Primer mit 10. Zum Beispiel, wenn das Röhrchen 45 nM lyophilisierte Primer enthält,fügen Sie 450 μL ddH2 O hinzu und wirbeln Sie die Lösung.
- Lagern Sie die Primer-Standardlösungen bei -20 °C oder führen Sie die Verstärkung fort.
3. Amplifikation des Genfragments mittels PCR
HINWEIS: Entscheiden Sie, welches PCR-Kit für das interessierende Gen geeignet ist. Kleinere Gene (<1.000 kb) können für eine billigere Taq-Polymerase zugänglicher sein, während größere Gene (≥1.000 kb) von High-Fidelity-Polymerase profitieren können, um Fehler zu reduzieren. Es ist wichtig zu beachten, dass diese anfängliche PCR-Amplifikation nicht notwendig ist, wenn der Benutzer nicht mit der Erhaltung des ursprünglichen Genfragments befasst ist (Sie bietet mehrere Versuche der Zirkularisierung und ermöglicht vergleichende Studien von LET vs. RCA-Produkt). Es ist auch wichtig zu beachten, dass diese PCR-amplifizierte LET direkt in Reaktionen verwendet werden kann; wie in der Einleitung erwähnt, würde es jedoch nur eine begrenzte Anzahl von Reaktionen zulassen, wenn die weiteren Amplifikationsschritte außer Acht gelassen würden. Verdauung und Ligatur können direkt auf dem resuspendierten Genfragment durchgeführt werden57 (wenn man sicher ist, benötigen sie nicht mehr LET, um zusätzliche Zirkularisierungsbestände durchzuführen). Wenn dies der Fall ist, überspringen Sie Abschnitt 3 und fahren Sie mit Abschnitt 4 fort. Gehen Sie folgendermaßen vor, um eine PCR durchzuführen.
- Verwenden Sie die 100 μM-Bestände aus Schritt 2.8, um 10 μM-Arbeitslösungen zu erstellen. Viele PCR-Kit-Protokolle erfordern 10-μM-Lösungen von Primern.
- Programmieren Sie den thermischen Cycler so, dass er die Reaktion gemäß den Protokollen des Kit-Herstellers durchführt. Verschiedene Kits erfordern leicht unterschiedliche Zyklusparameter. Für das in der Materialtabelle aufgeführteKit sind die Bedingungen 94 °C für 30 s anfängliche Denaturierung; 30 Zyklen von 94 °C für 30 s Denaturierung, 45 °C für 30 s Primerglühen und 68 °C für 60 s Verlängerung; mit einer Endverlängerung bei 68 °C für 5 min; und schließlich ein 10 °C unbestimmter Halt.
- Stellen Sie sicher, dass Sie die richtige Dehnungszeit auswählen (variabel in Abhängigkeit von der Länge des zu amplierenden Gens). Haben Sie eine Dehnungszeit von 1 min pro 1.000 bp.
- Stellen Sie sicher, dass Sie die richtige Glühtemperatur für die Grundierungen eingeben. Verwenden Sie einen Online-Tm-Rechner, der beide Primer als Eingaben verwendet, um die beste Glühtemperatur58zu bestimmen. Bei Verwendung von M13-Primern ist eine Glühtemperatur von 45 °C ausreichend.
- Beziehen Sie sich bei der Bestimmung der Anzahl der Zyklen auf das Protokoll des Herstellers, aber 30 Zyklen führen meistens zu einer ausreichenden Verstärkung.
- Bei PCR die dNTPs auftauen und wirbeln. Verwenden Sie den im Kit enthaltenen PCR-Puffer.
- Kombinieren Sie in einem einzigen PCR-Röhrchen alle Kit-Komponenten gemäß den Anweisungen im Protokoll des Herstellers. Um eine erfolgreiche Amplifikation zu gewährleisten, fügen Sie 1 μL resuspendierten DNA-Bestand hinzu (Schritt 2.6).
- Homogenisieren Sie die Mischung vorsichtig durch Vortexing auf Mediumseinstellung für 5-10 s. Alternativ können Sie die Hälfte des Volumens 10-20 Mal nach oben und unten pipettieren, um sich zu wirbeln.
- Führen Sie die PCR-Reaktion durch.
- Wenn das PCR-Protokoll keinen abschließenden Kühlschritt enthielt, lassen Sie die Reaktion 5 Minuten bei 10 °C abkühlen, bevor Sie sie entfernen, um Kondensation auf den Boden des Rohres zu treiben.
- Reinigen Sie die Reaktion mit einem PCR-Reinigungskit gemäß den Anweisungen des Herstellers.
- In einem 1,5-ml-Röhrchen DNA-Bindungspuffer und PCR-Probe im Verhältnis 5:1 hinzufügen.
- Dieses Gemisch in die Spinnsäule geben und bei 16.000 x g für 1 min zentrifugieren. Verwerfen Sie den Durchfluss.
- 200 μL DNA-Waschpuffer in die Säule geben und bei Raumtemperatur für 1 min inkubieren.
- 1 min bei 16.000 x g zentrifugieren und den Durchfluss verwerfen.
- Wiederholen Sie die Schritte 2.8.3 und 2.8.4 ohne den 1-minütigen Inkubationsschritt.
- Zentrifuge für weitere 1-2 min bei 16.000 x g, um den verbleibenden Puffer zu entfernen.
- Eluieren Sie die DNA in 46 μL ddH2O.
- Quantifizieren Sie die gereinigte DNA mit einem Spektralphotometer.
- Lagern Sie die gereinigte DNA bei -20 °C oder fahren Sie mit dem nächsten Schritt fort.
4. Verdauung und Zirkularisierung
HINWEIS: Eine weitere Amplifikation kann durch Zirkularisierung der DNA gefolgt von RCA erreicht werden. Verdauen Sie die DNA, um die Schablone für die Zirkularisierung vorzubereiten. Dadurch werden die Primersequenzen entfernt und klebrige Enden sowohl am 5'- als auch am 3'-Ende der Vorlage erstellt. Befestigen Sie diese Enden über eine Ligationsreaktion wieder.
- In einem PCR-Röhrchen werden 5 μL des notwendigen Puffers, 20 U HindIII und 45 μL der gereinigten DNA aus Schritt 3.8 kombiniert.
- Homogenisieren Sie diese Mischung vorsichtig mit einer Pipette.
- Die Mischung in einem Thermocycler für 15 min bei 37 °C inkubieren.
- Hitzeinaktivieren Sie HindIII durch Inkubation für 20 min bei 80 °C.
- Lassen Sie die Reaktion auf 10 °C abkühlen, bevor Sie sie entfernen, um Kondenswasser auf den Boden des Rohres zu treiben.
- Fügen Sie 5 μL T4-Ligasepuffer und 800 U T4-Ligase zur neu verdauten DNA hinzu.
- Verwenden Sie die T7-Ligase, falls gewünscht.
- Homogenisieren Sie diese Mischung vorsichtig mit einer Pipette.
- Inkubieren Sie die Mischung für 1 h bei 25 °C, um die Zirkularisierungsreaktion durchzuführen.
- Reinigen Sie die Reaktion mit einem PCR-Reinigungskit gemäß den Anweisungen des Herstellers. Verwenden Sie dasselbe Protokoll, das in Schritt 3.8 beschrieben wurde.
- Quantifizieren Sie die DNA mit einem Spektralphotometer. Die erwarteten Werte sind ~20 ng/μL.
- Bei -20 °C lagern oder mit dem nächsten Schritt fortfahren.
5. Isotherme Rollkreisverstärkung
HINWEIS: Die Rolling Circle Amplification (RCA) kann mit einem handelsüblichen Kit oder mit einzeln zugekauften Komponenten durchgeführt werden. Die Befolgung des Herstellerprotokolls gewährleistet eine erfolgreiche Verstärkung. Kits enthalten typischerweise einen Probenpuffer, einen Reaktionspuffer und eine strangverdrängende Polymerase, wie z. B. φ29-Polymerase. Mehrere Reaktionsröhrchen können kombiniert werden, um eine große Menge an DNA für die zellfreie Expression zu produzieren (4 μg aus 20 pg Ausgangsmaterial). Das folgende Protokoll arbeitet effizient.
- In einem einzigen Röhrchen werden 20 μL des Probenpuffers, 20 μL des Reaktionspuffers, 0,8 μL des Enzyms und 1 μL der zirkulären Expressionsvorlage (MEZ) aus Schritt 4.9 kombiniert.
HINWEIS: Dies wird eine Gesamt-DNA-Masse von ~ 20 ng haben, aber RCA kann mit Pikogrammmengen arbeiten, wodurch die Verdünnung des CET und eine extreme enzymatische Amplifikation ermöglicht werden, wenn im Hochdurchsatz-Screening ein erheblicher Materialbedarf besteht. - Homogenisieren Sie die Mischung mit einer Pipette und aliquot 10 μL der Mischung in vier separate Röhrchen.
- Bei 30 °C für 4-18 h inkubieren.
- Hitze inaktiviert das Enzym durch Inkubation bei 65 °C für 10 min. Reduzieren Sie die Temperatur für 5 min auf 12 °C, um kondensierend am Boden des Rohres zu fördern.
HINWEIS: Es ist am einfachsten, alle Temperaturschritte in einem automatisierten Protokoll auf einem thermischen Cycler zu kombinieren. - Verdünnen Sie die resultierende Lösung durch Zugabe von 15 μL ddH2O zu jedem Röhrchen.
- Kombinieren Sie alle Röhrchen und fügen Sie direkt zu einer zellfreien Reaktion hinzu.
- Falls gewünscht, verwenden Sie ein PCR-Reinigungskit, um das Produkt zu reinigen, und eluieren Sie es in 36 μL ddH2O zur Quantifizierung. Stellen Sie sicher, dass die Schablonenkonzentration ~100 ng/μL beträgt.
6. Zellfreie Reaktion
HINWEIS: Führen Sie einen zellfreien Ausdruck durch, indem Sie Energiepuffer, Extrakt und RCA-Vorlage kombinieren. Eine typische zellfreie Reaktion unter Verwendung des PANOx-SP-Energiepuffers besteht aus 1,2 mM ATP, je 0,85 mM GMP, UMP und CMP, 30 mM Phosphoenolpyruvat, 130 mM Kaliumglutamat, 10 mM Ammoniumglutamat, 12 mM Magnesiumglutamat, 1,5 mM Spermidin, 1 mM Putrescin, 34 μg/ml Folinsäure, 171 μg/mL E. coli tRNA-Gemisch, je 2 mM von 20 nicht markierten Aminosäuren, 0,33 mM NAD, 0,27 mM Coenzym A (CoA), 4 mM Kaliumoxalat, 57 mM HEPES-KOH-Puffer (pH 7,5), 0,24% Volumen des E. coli-Extrakts und variable Mengen an DNA23,49. Das Reaktionsvolumen kann variieren, aber 15 μL-Reaktionen können den Reagenzienverbrauch einsparen und sind klein genug für die Verwendung in einer 384 schwarzwandigen Mikroplatte49,50.
- Wenn Sie ein fluoreszierendes Protein wie sfGFP exprimieren, bereiten Sie einen Plattenleser vor, um bei der gewünschten Anregung / Emission, Temperatur und Bewegung zu lesen.
- Wenn Sie eine 384-Well-Platte verwenden, aliquotieren Sie 60 μLH2O in die Vertiefungen, die an einen leeren Probenschacht grenzen, um die Luftfeuchtigkeit aufrechtzuerhalten und den Kanteneffekt zu reduzieren.
- Fügen Sie die verschiedenen erforderlichen Komponenten für jede Probe in ein Röhrchen hinzu. Fügen Sie genug hinzu, um Triplikate durchzuführen. Replikate innerhalb der Platte können helfen, Ursachen für Variabilität zu identifizieren.
- Fügen Sie den Extrakt, den Energiepuffer und dann die DNA hinzu.
- MitddH2Oauf das gewünschte Endvolumen verdünnen.
- Mischen Sie diese Lösung gründlich, indem Sie die Hälfte des Lösungsvolumens 10-20 Mal nach oben und unten pipettieren.
- Das Reaktionsgemisch in 15 μL Aliquots in die gewünschten Vertiefungen in der Mikrotiterplatte überführen.
- Versiegeln Sie die Platte mit einem farblosen Dichtungsfilm, um die Luftfeuchtigkeit aufrechtzuerhalten und Verdunstung zu verhindern.
- Legen Sie die versiegelte Platte in den Plattenleser und lassen Sie die Reaktion abschließen.
- Wenn Sie ein Protein exprimieren, das nicht live überwacht werden kann, verwenden Sie eine andere temperaturgesteuerte Vorrichtung wie einen Thermoblock, um die Platte zu inkubieren.
7. Subtilisin-Assay
HINWEIS: Wenn Sie das Subtilisin BPN' (SBT(n)))-Gen in der Ergänzungssequenz Nr. 2exprimieren, befolgen Sie dieses Protokoll, um die Aktivität zu testen.
- Herstellen einer 10 μM Stammlösung von N-Succinyl-Ala-Ala-Pro-Phe p-nitroanilide in Dimethylformamid (DMF).
- Stellen Sie ein Plattenlesegerät so ein, dass die Absorption bei 410 nm alle 20 s für 10 minuten gemessen wird, während eine Temperatur von 25 ° C beibehalten wird.
- In einer flachen Bodenplatte, farbloser 96-Well-Platte, aliquot94 μL ddH2 O und 1 μL N-Succinyl-Ala-Ala-Pro-Phe p-nitroanilide aus Schritt 7.1.
- Fügen Sie 5 μL der fertigen zellfreien Reaktion aus Schritt 6.7 hinzu und lesen Sie sie mit einem Plattenleser, der auf das in Schritt 7.2 beschriebene Protokoll eingestellt ist.
Ergebnisse
Die Expression von sfGFP aus RCA-Vorlagen war vergleichbar mit der des pJL1-Plasmids, wenn nur 0,30 μL ungereinigte RCA-DNA in einer 15-μL-Reaktion verwendet wurden (Abbildung 2A). Tatsächlich scheint die Verdoppelung und Verdreifachung der Menge der Vorlage keinen Nutzen im BL21 DE3 Star-Extrakt zu bieten, was auf bereits gesättigte Konzentrationen der Vorlage bei 0,30 μL pro Reaktion hindeutet. Umgekehrt scheint es von Vorteil zu sein, die Menge an RCA-Vorlage zu erhöhen, wenn sie zu Zellextrakt aus dem SHuffle-Stamm hinzugefügt wird (Abbildung 2B)28. Bei einigen Proteinen können Ergebnisse sehr schnell beobachtet werden, was den gesamten Workflow (Amplifikation und Assaying) auf unter 24 h komprimiert. Einige Proteine benötigen jedoch eine niedrigere Temperatur oder haben langsamere Faltungszeiten, was die Zeit bis zum Erhalt der Ergebnisse verlängert, sich jedoch auf den hier dargestellten Workflow auswirkt. Dies kann beobachtet werden, wenn Subtilisin (SBT(n)) exprimiert wird, wo die Untersuchung nach 4 h Expression nicht lang genug für die SBT(n)-Reifung war(Abbildung 3A, Beispiel für ein fehlgeschlagenes Ergebnis). Die Fortsetzung der Reaktion auf 16 h kann zu nachweisbaren SBT(n)-Werten führen (Abbildung 3B). Diese Verbesserung kann temperaturabhängig sein, wie in der Literatur beobachtet wurde, wo optimierte Temperaturbedingungen untersucht wurden59,60.

Abbildung 1: Ein repräsentativer Schaltplan der minimalen genetischen Vorlage und des Prozesses, den sie nach dem ersten PCR-Amplifikationsschritt durchläuft. Die Schablonen werden mit HindIII verdaut, mit T4-Ligase zirkuliert und mit φ29-Polymerase weiter amplifiziert, um große Konateramere zu erzeugen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
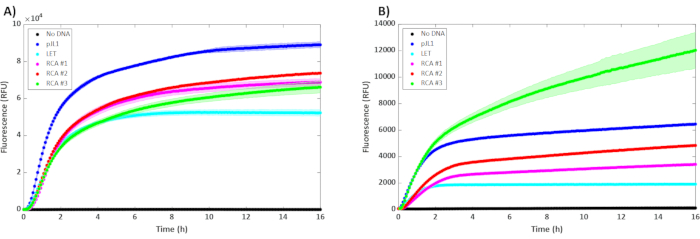
Abbildung 2: Ergebnisse der zellfreien Reaktionen mit ungemeinigten 5 nM Plasmid (pJL1), 5 nM linearer Schablone (LET) und unterschiedlichen Konzentrationen ungereinigter Cinch. RCA #1, #2 und #3 enthielten 0,3 μL, 0,6 μL bzw. 0,9 μL ungereinigtes RCA-Produkt in einer 15 μL-Reaktion, die bei 30 °C inkubiert wurde (n = 3). Fehlerindikatoren stellen ± 1 SD vom Mittelwert dar. Die y-Achse ist Fluoreszenz, und die x-Achse ist die Zeit, die während der Reaktion vergangen ist. Die Kinetik der sfGFP-Expression ist in (A) BL21 DE3 Star und (B) T7 SHuffle dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Zellfreie Reaktionen mit 5 nM SBT(n) LET und unterschiedlichen Konzentrationen von ungemeinigtem RCA-Produkt. RCA ng und pg entsprechen der Konzentration der DNA, die zur Durchführung der Rollkreisamplifikation verwendet wird. Ungereinigtes RCA-Produkt wurde in einer 15-μL-Reaktion verwendet, die bei 30 °C inkubiert wurde (n = 3). Fehlerindikatoren stellen ± 1 SD vom Mittelwert dar. Die y-Achse ist die Absorption bei 410 nm und die x-Achse ist die Zeitspanne, die im Assay vergangen ist. Die Reaktionen wurden für (A) 4 h und (B) 16 h durchgeführt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Klonmethode | |
| PCR | 2 -4 h |
| Plasmidverdauung | 35 Minuten |
| Gebundenheit | 1 Std. |
| Transformation | 2 Std. |
| Inkubation über Nacht | 16 Std. |
| Sequenzierung | 24 - 48 Uhr |
| Glycerin Stock Prep | 16 Std. |
| Wachstum und Reinigung | 16 Std. |
| Gesamtzeit | 46 - 72 Uhr |
| RCA-Methode | |
| PCR | 2 - 4 h |
| Verdauung | 35 Minuten |
| Gebundenheit | 1 Std. |
| RCA | 4 - 18 Uhr |
| CFE | 4 - 16 Uhr |
| Gesamtzeit | 12 - 40 Uhr |
Tabelle 1: Ein Vergleich des Zeitplans zwischen einem vereinfachten traditionellen Klonprotokoll und dem hier behandelten RCA-Protokoll.
Ergänzende Datei: Die ergänzende Datei listet die Sequenzen auf. Sequenz #1 ist sfGFP (999 Basenpaare) und Sequenz #2 ist Subtilisin BPN' (1344 bp). Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Das Gen von Interesse kann jedes gewünschte Protein sein, aber es ist am besten, mit einem fluoreszierenden Protein als bequemer Reporter für echtzeit- oder endpunkte Auslesung auf einem Well Plate Reader für neue Anwender dieser Methode zu beginnen. Für neue Proteinsequenzen kopieren Sie die Aminosäuresequenz des gewünschten Proteins und fügen Sie sie in das gewünschte Codon-Optimierungswerkzeug61,62ein. Es gibt normalerweise viele verfügbare Organismen und Stämme von E. coli im Codon-Optimierungswerkzeug, aber die Wahl der allgemeinen Escherichia coli-Option ist geeignet. Überprüfen Sie nach der Optimierung das Gen, um sicherzustellen, dass die zuvor ausgewählte Schnittstelle und die Primersequenzen nicht vorhanden sind. Wenn ja, kann die Sequenz optimiert werden, bis die Sequenzen nicht mehr vorhanden sind. In einigen Situationen kann es erforderlich sein, die HindIII-Schnittstellen durch eine andere Einschränkungswebsite zu ersetzen, wenn wiederholte Optimierungen konsistent zu einer internen HindIII-Schnittstelle führen. Es ist am besten, die gleiche Schnittstelle so oft wie möglich zu verwenden, um den Prozess standardisiert zu halten und die Kosten für die Bereitstellung mehrerer Restriktionsenzyme zu senken.
Bevor Sie mit der Amplifikation beginnen, wählen Sie das PCR-Kit, das am besten zum LET passt. Ein LET mit <1.000 Basenpaaren kann mit einer einfachen Taq-Polymerase verstärkt werden, während ein LET mit ≥ 1.000 Basenpaaren eine Polymerase mit höherer Wiedergabetreue63erfordern kann. Richten Sie das Protokoll für die Verstärkung entsprechend dem Kit-Hersteller und der Glühtemperatur der gewünschten Primer ein. Die Glühtemperatur ist entscheidend für eine erfolgreiche Verstärkung. Als Faustregel gilt, eine Glühtemperatur zu wählen, die 5 °C niedriger ist als die Tm der Primer. Es gibt kostenlose Online-Tools, die eine optimierte Glühtemperatur basierend auf der Reihenfolge der Primer58bieten. Die Verwendung der richtigen Glühtemperatur ist entscheidend für die Herstellung einer qualitativ hochwertigen DNA-Schablone.
Die zellfreie Genexpression hat in den letzten Jahren aufgrund ihrer Geschwindigkeit, Einfachheit und nützlichkeit für das Prototyping der synthetischen Biologie eine Renaissance erlebt. Diese Arbeit hat eine Methode skizziert, um die Geschwindigkeit und Leichtigkeit beim Testen einer großen Bibliothek neuer, funktioneller Proteine zu erhöhen. Während herkömmliche Klonmethoden Tage oder Wochen dauern können, kann dieses Protokoll in weniger als 24 Stunden durchgeführt werden. Tabelle 1 beschreibt typische Zeitbereiche sowohl für das herkömmliche Klonen als auch für das RCA-Protokoll. Beachten Sie, dass das Klonprotokoll vereinfacht wurde und einige optionale Schritte, wie z. B. die Gelisolierung, ausgelassen wurden. Tabelle 1 bezieht sich auch nur auf die üblichen Restriktionsverdauungsprotokolle; Es gibt viele andere Klonmethoden, aber diese benötigen eine ähnliche Zeit. Der obere Bereich dieses enzymatischen Amplifikationsprotokolls benötigt weniger Zeit als der untere Bereich der Klonmethode, insbesondere wenn ein 8-Stunden-Arbeitstag in Betracht gezogen wird. Dies ist weitgehend auf die Entfernung von Inkubationen über Nacht und die fehlende Sequenzierungsbestätigung zurückzuführen. Das RCA-Protokoll basiert vollständig auf PCR und RCA, die weniger spezielle Fähigkeiten erfordern als herkömmliche Klonierungs-, Transformations- und Zellkulturtechniken. Diese Methode macht die zellfreie Expression für Labore zugänglich, die keine Vorerfahrung im Klonen oder Zugang zu Kapital haben, das für die Transformation und das Wachstum von Zellen erforderlich ist. Diese Methode eignet sich auch gut für Projekte, die sich auf zytotoxische Proteine konzentrieren, bei denen das Klonen und die Vermehrung der Gene mit herkömmlichem Klonen aufgrund der Toxizität in der lebenden Zelle schwierig ist. Dieses RCA-Protokoll ist auch in der Lage, sehr kleine Mengen an zirkulärer DNA (Pikogramm dna) auf das für CFE notwendige Niveau zu amplifizieren. In Abbildung 3Bwurde die zirkularisierte SBT(n)-Schablone vor RCA auf 20 ng/μL und 20 pg/uL verdünnt. Während die beobachteten Abbauraten unterschiedlich waren, führten beide Reaktionen innerhalb von 10 Minuten zu der gleichen Menge an Substratabbau. Die vorgeschlagene Methode soll das Klonen nicht ersetzen; Die Plasmidvermehrung kann DNA-Mengen für ungiftige Gene produzieren, die nicht erreicht werden können. Vielmehr handelt es sich um ein praktisches Prototyping-Tool in einer massiven Bibliotheksscreening-Phase (enzymatische Schritte sind mit einem Standard-Fluidhandler für die Automatisierung zugänglich), das helfen würde, zu identifizieren, welche Sequenzen für die Archivierung und weitere Verbreitung geklont werden sollten.
Bei der Entwicklung einer zellfreien Reaktion gibt es viel Flexibilität, aber es gibt einige Faktoren, die berücksichtigt werden müssen, um den Erfolg sicherzustellen. Kleinere Reaktionen haben ein größeres Verhältnis von Oberfläche zu Volumen, was sich hervorragend für den Gasaustausch eignet, aber die Reaktion muss den Boden des Bohrlochs für genaue Fluoreszenzmessungen vollständig abdecken. Die Temperatur der Reaktion kann auch in Abhängigkeit vom interessierenden Genvariieren 59. Es gibt mehrere Möglichkeiten für Benutzer, wenn es um die Auswahl eines Energiepuffers34,46geht. Einige Rezepte sind teurer als andere, aber die Entscheidung bleibt dem Benutzer überlassen. Es gibt auch viele mögliche Quellen für E. coli-Extrakt 36. Benutzer sollten sich mit den Produktionsprotokollen für Extrakte vertraut machen und entscheiden, welches für ihre Zwecke am besten geeignet ist28,48,50,64,65,66,67.
Bei der Verwendung von RCA zur Amplifikation von Schablonen für die zellfreie Expression ist die Wahl des Bakterienstamms sehr wichtig. Das Ausdrucksprofil ist tendenziell besser als das von LETs. Der beliebte BL21 DE3 Star-Stamm bewältigt dies gut, wobei RCA ~ 1,4x besser als LET und das pJL1-Plasmid ~ 1,2x besser als die beste RCA-Konzentration ist (Abbildung 2A). Auf der anderen Seite weisen einige Stämme einen Schablonenabbau auf und schneiden aufgrund des Vorhandenseins nativer Nukleasen schlecht ab23,28. In diesem Fall scheint es, dass der SHuffle-Stamm von einer erhöhten RCA-Vorlage profitiert. Frühere Literatur hat gezeigt, dass SHuffle-Extrakt mit PCR-Produkten nicht gut funktioniert, aber die höchste Konzentration an ungereinigtem RCA-Produkt, die in dieser Studie verwendet wurde, übertraf das pJL1-Plasmid (Abbildung 2B). Die Expression von funktionellem SBT(n) (Abbildung 3) ist ein Beispiel für ein zytotoxisches Protein, das praktischerweise aus zellfreien, aber aufgrund der Toxizität in lebenden Zellen schwer zu prototypisieren ist (nicht in der Lage, diese Expressionsvorlage im Plasmid zu klonen und sich in E. coli zu vermehren). Im Gegensatz zu sfGFP kann die SBT(n)-Aktivität nicht nach nur 4 h Expression beobachtet werden (Abbildung 3A). Das Signal ist nach 16 h Expression und Reifung nachweisbar (Abbildung 3B). Die in diesen Beispielen verwendete ungereinigte DNA stammte aus 100 μL, was vier 10 μL RCA-Reaktionen war, die verdünnt und kombiniert wurden. Dieser eine Bestand kann 333 zellfreie Reaktionen unterstützen, wenn nur 0,30 μL pro Reaktion verwendet werden.
Die Kompatibilität von RCA-Vorlagen mit CFE muss mit verschiedenen Auszügen weiter untersucht werden. Mögliche Komplikationen bei linearen Produkten können durch Zugabe von kurzen Oligos, die die E. coli Chi-Sequenz oder das GamS-Protein52,53enthalten,gelindert werden. Die Literatur legt nahe, dass die Zugabe von Chi-Oligos linearen Schablonen einen größeren Schutz bieten kann als das GamS-Protein53. Wenn Sie einen Extrakt verwenden, von dem bekannt ist, dass er geringe Erträge aus linearen Schablonen liefert, sollte der Benutzer die Kosten jedes Schutzmittels analysieren und dasjenige verwenden, das seinen Zwecken am besten entspricht. Eine weitere Einschränkung ist die Unfähigkeit, RCA-Template-Konzentrationen aufgrund der Art des resultierenden Concatemers in Molarität umzuwandeln. Dies bedeutet, dass man die gleiche Anzahl von minimalen Vorlagen basierend auf der Masse hinzufügen kann, aber sie haben unterschiedliche Konkatetomlängen, was sich auf die Expressionsniveaus auswirken kann. Die Autoren haben nicht festgestellt, dass dies ein Problem beim Screening / Prototyping ist, da die gemittelten Expressionsniveaus aus jeder Vertiefung gleich sind (geringe Varianz), aber ein Problem sein könnten, wenn sie in kleineren Volumina (z. B. Mikrotröpfchen) ausgedrückt werden.
Die zellfreie Genexpression hat das Potenzial, als bedeutendes Werkzeug im Protein-Prototyping und in Design-Build-Test-Workflows eingesetzt zu werden. Die meisten zellfreien Expressions-Workflows verlassen sich jedoch immer noch auf traditionelle Plasmide als genetische Vorlage. Dies verlangsamt den Prozess und verhindert, dass die zellfreie Expression für Screening- / Prototyping-Zwecke in vollem Umfang genutzt wird. Die Verstärkung von Vorlagen und die anschließende Durchführung von RCA können schnell genügend genetische Vorlagen für viele Reaktionen erzeugen und genügend Protein für die nachgelagerte Charakterisierung und Funktionstests produzieren.
Offenlegungen
Nigel Reuel ist Mitglied des wissenschaftlichen Beirats von BigHat Biosciences Inc., einem Unternehmen, das zellfreie Systeme für das Design von Antikörpern einsetzt.
Danksagungen
Die Autoren erkennen NIH 1R35GM138265-01 und NSF 2029532 für die teilweise Unterstützung dieses Projekts an.
Materialien
| Name | Company | Catalog Number | Comments |
| Alaline | Formedium | DOC0102 | |
| Ammonium glutamate | MP Biomedicals | MP21805951 | |
| Arginine | Formedium | DOC0106 | |
| Asparagine | Formedium | DOC0114 | |
| Aspartic Acid | Formedium | DOC0118 | |
| ATP | Sigma | A2383 | |
| Axygen Sealing Film | Corning | PCR-SP | |
| CMP | Sigma | C1006 | |
| Coenzyme A | Sigma | C3144 | |
| CutSmart Buffer | NEB | B7204S | Provided with HindIII |
| Cysteine | Formedium | DOC0122 | |
| DNA Clean and Concentrator Kit | Zymo Research | D4004 | Used for purifying DNA |
| dNTPs | NEB | N0447 | |
| E. coli tRNA | Sigma (Roche) | 10109541001 | |
| Folinic Acid | Sigma | 47612 | |
| Gene Fragment | IDT | ||
| Glutamic Acid | Formedium | DOC0134 | |
| Glutamine | Formedium | DOC0130 | |
| Glycine | Formedium | DOC0138 | |
| GMP | Sigma | G8377 | |
| HEPES | Sigma | H3375 | |
| HindIII-HF | NEB | R3104L | |
| Histidine | Formedium | DOC0142 | |
| Isoleucine | Formedium | DOC0150 | |
| Leucine | Formedium | DOC0154 | |
| Lysine | Formedium | DOC0158 | |
| Magnesium glutamate | Sigma | 49605 | |
| Methionine | Formedium | DOC0166 | |
| Microtiter Plate (384 well) | Greiner | 781906 | |
| Microtiter Plate (96 well) | Greiner | 655809 | |
| Multimode Plate Reader | BioTek | Synergy Neo2 | |
| NAD | Sigma | N8535 | |
| NanoPhotometer | Implen | NP80 | |
| OneTaq DNA Polymerase | NEB | M0480 | |
| PCR Tube | VWR | 20170-012 | |
| Phenylalanine | Formedium | DOC0170 | |
| Phosphoenolpyruvate | Sigma (Roche) | 10108294 | |
| Potassium glutamate | Sigma | G1501 | |
| Potassium oxalate | Fisher Scientific | P273 | |
| Proline | Formedium | DOC0174 | |
| Putrescine | Sigma | P5780 | |
| Serine | Formedium | DOC0178 | |
| Spermidine | Sigma | S0266 | |
| T4 DNA Ligase | NEB | M0202S | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | Provided with T4 DNA Ligase |
| TempliPhi Amplification Kit | Cytiva | 25640010 | Used for RCA |
| Thermal Cycler | Biorad | C1000 Touch | |
| Thermoblock | Eppendorf | ThermoMixer FP | |
| Threonine | Formedium | DOC0182 | |
| Tryptophan | Formedium | DOC0186 | |
| Tyrosine | Formedium | DOC0190 | |
| UMP | Sigma | U6375 | |
| Valine | Formedium | DOC0194 |
Referenzen
- Sun, Q., et al. A simple and low-cost paper-based colorimetric method for detecting and distinguishing the GII.4 and GII.17 genotypes of norovirus. Talanta. 225, 121978 (2021).
- Pardee, K., et al. Low-Cost Detection of Zika Virus Using Programmable Biomolecular Components. Cell. 165 (5), 1255-1266 (2016).
- Pardee, K., et al. Paper-based synthetic gene networks. Cell. 159 (4), 940-954 (2014).
- Ma, D., Shen, L., Wu, K., Diehnelt, C. W., Green, A. A. Low-cost detection of norovirus using paper-based cell-free systems and synbody-based viral enrichment. Synthetic Biology. 3 (1), (2018).
- Park, S., Lee, J. W. Detection of coronaviruses using rna toehold switch sensors. International Journal of Molecular Sciences. 22 (4), 1772 (2021).
- Cao, M., Sun, Q., Zhang, X., Ma, Y., Wang, J. Detection and differentiation of respiratory syncytial virus subgroups A and B with colorimetric toehold switch sensors in a paper-based cell-free system. Biosensors and Bioelectronics. 182, 113173 (2021).
- Mcnerney, M. P., et al. Point-of-care biomarker quantification enabled by sample-specific calibration. Science Advances. 5 (9), (2019).
- Silverman, A. D., Akova, U., Alam, K. K., Jewett, M. C., Lucks, J. B. Design and optimization of a cell-free atrazine biosensor. ACS Synthetic Biology. 9 (3), 671-677 (2020).
- Salehi, A. S. M., et al. Cell-free protein synthesis approach to biosensing hTRβ-specific endocrine disruptors. Analytical Chemistry. 89 (6), 3395-3401 (2017).
- Garamella, J., Majumder, S., Liu, A. P., Noireaux, V. An adaptive synthetic cell based on mechanosensing, biosensing, and inducible gene circuits. ACS Synthetic Biology. 8 (8), 1913-1920 (2019).
- Glasscock, C. J., et al. Dynamic control of pathway expression with riboregulated switchable feedback promoters. ACS Synthetic Biology. 16, (2019).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- Pardee, K., et al. Portable, on-demand biomolecular manufacturing. Cell. 167 (1), 248-259 (2016).
- Nelson, J. A. D., et al. Hydrofoam and oxygen headspace bioreactors improve cell-free therapeutic protein production yields through enhanced oxygen transport. Biotechnology Progress. 37 (2), 3079 (2020).
- Cai, Q., et al. A simplified and robust protocol for immunoglobulin expression in Escherichia coli cell-free protein synthesis systems. Biotechnology Progress. 31 (3), 823-831 (2015).
- Ogonah, O. W., Polizzi, K. M., Bracewell, D. G. Cell free protein synthesis: a viable option for stratified medicines manufacturing? A brief history of cell free synthesis systems. Current Opinion in Chemical Engineering. 18, 77-83 (2017).
- Zawada, J. F., et al. Microscale to manufacturing scale-up of cell-free cytokine production-a new approach for shortening protein production development timelines. Biotechnology and Bioengineering. 108 (7), 1570-1578 (2011).
- Stark, J. C., et al. On-demand biomanufacturing of protective conjugate vaccines. Science Advances. 7 (6), (2021).
- Huang, A., et al. BioBitsTM Explorer: A modular synthetic biology education kit. Science Advances. 4 (8), 1-11 (2018).
- Stark, J. C., et al. BioBits health: classroom activities exploring engineering, biology, and human health with fluorescent readouts. ACS Synthetic Biology. 8 (5), 1001-1009 (2019).
- Stark, J. C., et al. BioBitsTM Bright: A fluorescent synthetic biology education kit. Science Advances. 4 (8), 33 (2018).
- Shinoda, T., et al. Cell-free methods to produce structurally intact mammalian membrane proteins. Scientific Reports. 6, (2016).
- Dopp, J. L., Rothstein, S. M., Mansell, T. J., Reuel, N. F. Rapid prototyping of proteins: Mail order gene fragments to assayable proteins within 24 hours. Biotechnology and Bioengineering. 116 (3), 667-676 (2019).
- Sachse, R., Dondapati, S. K., Fenz, S. F., Schmidt, T., Kubick, S. Membrane protein synthesis in cell-free systems: From bio-mimetic systems to bio-membranes. FEBS Letters. 588 (17), 2774-2781 (2014).
- Salehi, A. S. M., et al. Cell-free protein synthesis of a cytotoxic cancer therapeutic: Onconase production and a just-add-water cell-free system. Biotechnology Journal. 11 (2), 274-281 (2016).
- Georgi, V., et al. On-chip automation of cell-free protein synthesis: New opportunities due to a novel reaction mode. Lab on a Chip. 16 (2), 269-281 (2016).
- Thoring, L., et al. Cell-free systems based on CHO cell lysates: Optimization strategies, synthesis of "difficult-to-express" proteins and future perspectives. PLoS One. 11 (9), (2016).
- Dopp, J. L., Reuel, N. F. Simple, functional, inexpensive cell extract for in vitro prototyping of proteins with disulfide bonds. Biochemical Engineering Journal. 164, 107790 (2020).
- Isaksson, L., Enberg, J., Neutze, R., Göran Karlsson, B., Pedersen, A. Expression screening of membrane proteins with cell-free protein synthesis. Protein Expression and Purification. 82 (1), 218-225 (2012).
- Techner, J. M., et al. High-throughput synthesis and analysis of intact glycoproteins using SAMDI-MS. Analytical Chemistry. 92 (2), 1963-1971 (2020).
- Kim, H. C., et al. Implementing bacterial acid resistance into cell-free protein synthesis for buffer-free expression and screening of enzymes. Biotechnology and Bioengineering. 112 (12), 2630-2635 (2015).
- Rolf, J., Siedentop, R., Lütz, S., Rosenthal, K. Screening and identification of novel cGAS homologues using a combination of in vitro and in vivo protein synthesis. International Journal of Molecular Sciences. 21 (1), (2020).
- Haslinger, K., Hackl, T., Prather, K. L. J. Rapid in vitro prototyping of O-methyltransferases for pathway applications in Escherichia coli. bioRxiv. , (2020).
- Dopp, J. L., Tamiev, D. D., Reuel, N. F. Cell-free supplement mixtures: Elucidating the history and biochemical utility of additives used to support in vitro protein synthesis in E. coli extract. Biotechnology Advances. 37 (1), 246-258 (2018).
- Gregorio, N. E., Levine, M. Z., Oza, J. P. A user's guide to cell-free protein synthesis. Methods and Protocols. 2 (1), 24 (2019).
- Cole, S. D., Miklos, A. E., Chiao, A. C., Sun, Z. Z., Lux, M. W. Methodologies for preparation of prokaryotic extracts for cell-free expression systems. Synthetic and Systems Biotechnology. 5 (4), 252-267 (2020).
- Chiba, C. H., Knirsch, M. C., Azzoni, A. R., Moreira, A. R., Stephano, M. A. Cell-free protein synthesis: advances on production process for biopharmaceuticals and immunobiological products. BioTechniques. 70, (2021).
- Laohakunakorn, N. Cell-free systems: A proving ground for rational biodesign. Frontiers in Bioengineering and Biotechnology. 8, 788 (2020).
- Dondapati, S. K., Stech, M., Zemella, A., Kubick, S. Cell-free protein synthesis: A promising option for future drug development. BioDrugs. , 1-22 (2020).
- Noireaux, V., Liu, A. P. The new age of cell-free biology. Annual Review of Biomedical Engineering. 22, 51-77 (2020).
- Khambhati, K., et al. Exploring the Potential of Cell-Free Protein Synthesis for Extending the Abilities of Biological Systems. Frontiers in Bioengineering and Biotechnology. 7, (2019).
- Carlson, E. D., Gan, R., Hodgman, C. E., Jewett, M. C. Cell-free protein synthesis: Applications come of age. Biotechnology Advances. 30 (5), 1185-1194 (2012).
- Rosenblum, G., Cooperman, B. S. Engine out of the chassis: Cell-free protein synthesis and its uses. FEBS Letters. 588 (2), 261-268 (2014).
- Swartz, J. R. Transforming biochemical engineering with cell-free biology. AIChE Journal. 58 (1), 5-13 (2012).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Caschera, F., Noireaux, V. A cost-effective polyphosphate-based metabolism fuels an all E. coli cell-free expression system. Metabolic Engineering. 27, 29-37 (2015).
- Levine, M. Z., et al. Activation of energy metabolism through growth media reformulation enables a 24-hour workflow for cell-free expression. ACS Synthetic Biology. 9 (10), 2765-2774 (2020).
- Hunt, J. P., et al. Streamlining the preparation of "endotoxin-free" ClearColi cell extract with autoinduction media for cell-free protein synthesis of the therapeutic protein crisantaspase. Synthetic and Systems Biotechnology. 4 (4), 220-224 (2019).
- Dopp, J. L., Jo, Y. R., Reuel, N. F. Methods to reduce variability in E. Coli-based cell-free protein expression experiments. Synthetic and Systems Biotechnology. 4 (4), 204-211 (2019).
- Sun, Z. Z., et al. Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology. Journal of Visualized Experiments: JoVE. , e50762 (2013).
- Schinn, S. M., Broadbent, A., Bradley, W. T., Bundy, B. C. Protein synthesis directly from PCR: Progress and applications of cell-free protein synthesis with linear DNA. New Biotechnology. 33 (4), 480-487 (2016).
- Sitaraman, K., et al. A novel cell-free protein synthesis system. Journal of Biotechnology. 110 (3), 257-263 (2004).
- Marshall, R., Maxwell, C. S., Collins, S. P., Beisel, C. L., Noireaux, V. Short DNA containing χ sites enhances DNA stability and gene expression in E. coli cell-free transcription-translation systems. Biotechnology and Bioengineering. 114, 2137-2141 (2017).
- Sun, Z. Z., Yeung, E., Hayes, C. A., Noireaux, V., Murray, R. M. Linear DNA for rapid prototyping of synthetic biological circuits in an escherichia coli based TX-TL cell-free system. ACS Synthetic Biology. 3 (6), 387-397 (2014).
- . Addgene: pJL1 Available from: https://www.addgene.org/69496/ (2021)
- . IDT Codon Optimization Tool Available from: https://www.idtdna.com/pages/tools/codon-optimization-tool (2021)
- Hadi, T., et al. Rolling circle amplification of synthetic DNA accelerates biocatalytic determination of enzyme activity relative to conventional methods. Scientific Reports. 10 (1), 10279 (2020).
- . New England Biolabs Tm Calculator Available from: https://tmcalculator.neb.com/#!/main (2021)
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. Coli RNA polymerase and sigma factor 70. Journal of Biological Engineering. 4, (2010).
- Colant, N., et al. A rational approach to improving titer in Escherichia coli-based cell-free protein synthesis reactions. Biotechnology Progress. 37 (1), 3062 (2021).
- Burgess-Brown, N. A., et al. Codon optimization can improve expression of human genes in Escherichia coli: A multi-gene study. Protein Expression and Purification. 59, 94-102 (2008).
- Maertens, B., et al. Gene optimization mechanisms: A multi-gene study reveals a high success rate of full-length human proteins expressed in Escherichia coli. Protein Science. 19 (7), 1312-1326 (2010).
- Eckert, K. A., Kunkel, T. A. DNA polymerase fidelity and the polymerase chain reaction. Genome Research. 1 (1), 17-24 (1991).
- Dopp, J. L., Reuel, N. F. Process optimization for scalable E. coli extract preparation for cell-free protein synthesis. Biochemical Engineering Journal. 138, 21-28 (2018).
- Liu, D. V., Zawada, J. F., Swartz, J. R. Streamlining Escherichia Coli S30 extract preparation for economical cell-free protein synthesis. Biotechnology Progress. 21 (2), 460-465 (2005).
- Levine, M. Z., Gregorio, N. E., Jewett, M. C., Watts, K. R., Oza, J. P. Escherichia coli-Based Cell-Free Protein Synthesis: Protocols for a robust, flexible, and accessible platform technology. Journal of Visualized Experiments: JoVE. (144), e58882 (2019).
- Kwon, Y. C., Jewett, M. C. High-throughput preparation methods of crude extract for robust cell-free protein synthesis. Scientific Reports. 5, (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
ISSN 2578-2614
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten
Wir verwenden Cookies, um Ihre Erfahrung auf unserer Website zu verbessern.
Indem Sie unsere Website weiterhin nutzen oder auf „Weiter“ klicken, stimmen Sie zu, unsere Cookies zu akzeptieren.