Method Article
使用无细胞系统对用于蛋白质原型的最小线性模板进行扩增的快速酶促方法
摘要
该研究描述了一种方案,用于在不克隆或使用活细胞的情况下从合成基因片段中创建大量(μg-mg)DNA以进行蛋白质筛选活动。将最小模板酶消化并循环化,然后使用等温滚动循环扩增进行扩增。无细胞表达反应可以用未纯化的产物进行。
摘要
该协议描述了最小DNA模板的设计以及酶扩增的步骤,从而可以使用无细胞表达在不到24小时的时间内快速对可测定的蛋白质进行原型设计。从供应商处接收DNA后,对基因片段进行PCR扩增、切割、循环和冷冻储存。然后使用等温滚动圆扩增(RCA)稀释和显着扩增少量储存的DNA(高达106x)。RCA可以从起始物质的皮克水平(如果使用所有起始合成片段,则为mg水平)产生微克量的最小表达模板。在这项工作中,起始量为20 pg导致4μg的最终产物。所得的RCA产物(最小模板的串联体)可以直接添加到无细胞反应中,无需纯化步骤。由于这种方法完全基于PCR,因此当与自动化液体处理系统结合使用时,它可能会实现未来的高通量筛选工作。
引言
无细胞基因表达(CFE)已成为一种具有许多应用的强大工具。这些应用包括疾病检测1,2,3,4,5,6, 微量营养素和小分子检测7,8,9,10,11,12, 生物制造13,14,15,16,17 ,18, 教育19,20,21, 制造难解蛋白17,22,23,24,25,26,27, 和变异筛选23,28,29,30,31,32 ,33.这是由于无细胞系统的开放性和它们赋予的灵活性。许多伟大的评论文章提供了对技术的历史教育和未来展望34,35,36,37,38,39,40,41,42,43,44。
典型的无细胞反应由三个主要成分组成:细胞提取物,能量组合和遗传模板。活性细胞提取物包含转录和翻译(TXTL)所需的所有机器,并且可以以多种方式进行处理36。能量组合中的糖酵解中间体、电解质、氨基酸和辅因子支持 TXTL 过程。它是无细胞实验中变异性的主要来源45,并且可以以多种方式制备34,46。由于传统的克隆方法产生具有优异表达特性的质粒,因此遗传模板的制备得到了较少的改进。这些传统方法的缺点是构建和传播它们所需的周转时间和生物专业知识的数量。最近的优化工作已经为细胞提取物制备47,48提供了简单的24小时工作流程,可以与能量混合制备49,50并行进行。然而,传统的克隆为CFE原型设计时间增加了多天(表1)23。从商业基因片段中快速扩增的PCR产物可以直接使用51,但这限制了原型实验的数量,因为只产生1μgDNA,这对应于大约五个反应(传统的15μL体积)。通过这些额外的循环化和等温扩增步骤,DNA的量可能大于毫克(1毫克约5,000次反应)。这大大增加了在蛋白质或组合酶网络的高通量筛选(无细胞代谢工程)中可以进行的测试数量;它还允许将线性模板库有效保存为高浓度DNA。此外,需要增加模板的数量,以原型化材料科学应用所需的大量蛋白质(基于蛋白质的纤维和水凝胶)。线性模板的一些限制可以通过使用BL21 DE3 Star的提取物或使用最近发现的方法来保护线性模板免受退化52,53,54来克服。然而,这并不能解决供应商生产的用于PCR扩增的DNA库存有限或克隆所需的生物学专门知识和设备问题。
这项工作提出了一种方案,明确设计用于增加可以从供应商生产的少量基因片段(通常为500-1000ng冻干粉末)中获得的表达模板的数量。所描述的方法不需要在质粒中进行传统克隆或在活细胞中进行转化和繁殖所需的技能。在邮件中收到基因片段后,用户可以通过采用等温滚动圆扩增(RCA)(图1)23来产生足够的模板来进行许多无细胞反应。虽然从供应商处获得的DNA数量可能足以进行有限的筛选工作,但它很快就会耗尽,重新购买基因片段既耗时又昂贵。该方法也特别适用于大肠杆菌中有毒且难以克隆的基因。
研究方案
1. 设计基因片段
注意:基因片段应具有转录/翻译所需的所有遗传元件,包括启动子,核糖体结合位点(RBS),起始密码子,目的基因和终止子。虽然终止子对于线性表达模板(LET)不是必需的,但如果用户决定将序列插入质粒中,则这一点很重要。这些序列是从pJL1-sfGFP质粒55( 来自Michael Jewett实验室的礼物)中取出的,该质粒使用T7启动子。除了这些必要的遗传元件外,在启动子(5'切位点)之前添加六个碱基对的限制性内切位点,在终止子(3'切位点)之后再添加六个碱基对,在这种情况下使用HindIII(可以使用其他限制性内切酶,但使用一种高保真限制性内切酶标准化序列以减少保存在文库中所需的数量是有帮助的)。在5'切割位点的上游添加10个碱基对,在3'切割位点的下游添加10个碱基对,在这种情况下使用标准化的M13引物序列(引物是廉价的库存项目)。所使用的限制性内切酶位点和引物由使用者自行决定。但是,用户必须确保序列不存在于模板中的其他任何位置(不想创建不需要的切割或扩增起始位点)。本工作中使用的模板的顺序在补充材料中进行了详细介绍。这些步骤用于从此基本模板进行修改。
- 确定要表达的所需基因,并获得氨基酸序列或基因序列(如果已在 大肠杆菌中表达)。
- 如果是氨基酸序列,请使用许多标准供应商工具之一对 大肠杆菌 进行密码子优化56。如果使用补充剂中提供的模板,请确保优化的序列没有HindIII限制性位点(AAGCTT)。如果是这样,请继续优化序列,直到不再有 HindIII 站点。
- 复制序列并将其粘贴到提供的补充序列#1模板中,其中指示了目的基因。如果表达 sfGFP,请按原样使用补充序列 #1。如果表达枯草杆菌蛋白酶,请按原样使用补充序列#2。
- 从首选的DNA合成服务中订购最小的模板和必要的引物。
2. 重用基因片段和引物
注意:收到基因片段后,请遵循制造商的重悬协议或使用本简单指南创建DNA储备。
- 离心管(300×g 5秒)以收集底部的DNA沉淀。
- 加入双蒸馏水(ddH2O)制成终浓度为10 ng / μL的DNA模板。
- 在中等设置下涡旋溶液5-10秒。
- 通过在50°C下孵育20分钟来溶解整个沉淀。
- 再次短暂涡旋
- 以300×g离心5秒,以收集管底部的溶液。
- 储存在-20°C或用于PCR。
- 通过将引物重用无核酸酶水中来制备100μM引物储备。为了确定要添加的水量,将冻干底漆的纳摩尔数乘以10。例如,如果管中含有45 nM冻干引物,则加入450μLdH2O并涡旋溶液。
- 将底漆原液储存在-20°C或继续进行扩增。
3. 通过PCR扩增基因片段
注意:确定哪种PCR试剂盒适合目的基因。较小的基因(<1,000 kb)可能更适合更便宜的Taq聚合酶,而较大的基因(≥1,000 kb)可能受益于高保真聚合酶以减少错误。重要的是要注意,如果使用者不关心保留初始基因片段,则不需要进行初始PCR扩增(它提供了多次循环化尝试,并允许对LET与RCA产物进行比较研究)。同样重要的是要注意,这种PCR扩增的LET可以直接用于反应;然而,正如引言中提到的,如果忽略进一步的扩增步骤,它只允许有限数量的反应。消化和连接可以直接在重悬的基因片段上进行57( 如果确定,它们将不需要更多的LET来执行额外的循环储备)。如果是这种情况,请跳过第 3 节并继续执行第 4 节。要进行 PCR,请按照下列步骤操作。
- 使用步骤 2.8 中的 100 μM 储液来创建 10 μM 工作解决方案。许多PCR试剂盒方案需要10μM引物溶液。
- 对热循环仪进行编程,使其根据试剂盒制造商的协议进行反应。不同的套件需要稍微不同的循环参数。对于 材料表中列出的试剂盒,条件为94°C,初始变性为30秒;30次循环,94°C变性30秒,45°C下底漆退火30秒,68°C延伸60秒;在68°C下最终延伸5分钟;最后,10°C无限期保持。
- 确保选择正确的伸长时间(取决于要扩增的基因的长度)。每1,000 bp的伸长时间为1分钟。
- 确保为底漆输入正确的退火温度。使用在线Tm计算器,该计算器使用两种引物作为输入来确定最佳退火温度58。使用M13引物时,45°C的退火温度就足够了。
- 在确定循环次数时,请参阅制造商的协议,但30个循环通常会导致足够的扩增。
- 如果进行 PCR,请解冻并涡旋 dNTPs。使用试剂盒中提供的 PCR 缓冲液。
- 在单个PCR管中,按照制造商方案中的指示将所有试剂盒组件组合在一起。为确保成功扩增,加入1μL重悬的DNA储备(步骤2.6)。
- 通过在介质设置下涡旋5-10秒来轻轻地均匀混合物。或者,将体积的一半上下移液10-20次以涡旋。
- 进行 PCR 反应。
- 如果PCR方案不包括最后的冷却步骤,则在除去之前,让反应在10°C下冷却5分钟以驱动冷凝到管的底部。
- 按照供应商的说明使用PCR纯化试剂盒纯化反应。
- 在1.5 mL试管中,分别以5:1的比例加入DNA结合缓冲液和PCR样品。
- 将该混合物转移到离心柱中,并以16,000×g离心1分钟。 丢弃流出。
- 向色谱柱中加入200μLDNA洗涤缓冲液,并在室温下孵育1分钟。
- 在16,000×g下离心1分钟,并丢弃流出物。
- 重复步骤2.8.3和2.8.4,不进行1分钟的孵育步骤。
- 在16,000×g下再离心1-2分钟,以除去任何剩余的缓冲液。
- 在46μLdH2O中洗脱DNA。
- 使用分光光度计定量纯化的DNA。
- 将纯化的DNA储存在-20°C或继续下一步。
4. 消化和循环
注意:进一步的扩增可以通过循环DNA然后进行RCA来实现。消化DNA以准备循环化的模板。这将删除引物序列,并在模板的5'和3'末端创建粘性末端。通过连接反应重新连接这些末端。
- 在PCR管中,结合5μL必要的缓冲液,20U的HindIII和45μL来自步骤3.8的纯化DNA。
- 用移液器轻轻地均质这种混合物。
- 将混合物在37°C下在热循环仪中孵育15分钟。
- 通过在80°C下孵育20分钟来加热灭活HindIII。
- 在除去之前,让反应冷却至10°C,以驱动冷凝到管的底部。
- 向新消化的DNA中加入5μLT4连接酶缓冲液和800U T4连接酶。
- 如果需要,使用 T7 连接酶。
- 用移液器轻轻地均质这种混合物。
- 将混合物在25°C下孵育1小时以进行循环反应。
- 按照供应商的说明使用PCR纯化试剂盒纯化反应。使用步骤 3.8 中详述的相同协议。
- 使用分光光度计定量DNA。预期值为~20纳克/μL。
- 储存在-20°C或继续下一步。
5. 等温轧圆放大
注:滚动圆放大(RCA)可以使用商用套件或单独购买的组件进行。遵循制造商的协议将确保成功扩增。试剂盒通常包含样品缓冲液、反应缓冲液和链置换聚合酶,例如φ29聚合酶。多个反应管可以组合产生大量用于无细胞表达的DNA(从20pg起始材料中产生4μg)。以下协议可以有效地工作。
- 在单管中,结合步骤4.9中的20μL样品缓冲液,20μL反应缓冲液,0.8μL酶和1μL环表达模板(CET)。
注意:这将具有〜20ng的总DNA质量,但RCA可以使用皮克量,因此如果高通量筛选中对材料有显着需求,则允许CET的稀释和极端酶扩增。 - 用移液管将混合物均质化,并将10μL混合物等分到四个单独的管中。
- 在30°C孵育4-18小时。
- 通过在65°C下孵育10分钟来加热灭活酶。将温度降低到12°C5分钟,以促进管底部的冷凝。
注意:最简单的方法是在热循环仪上将所有温度步长组合到一个自动实验方案中。 - 通过向每个管中加入15μLdH2O来稀释所得溶液。
- 将所有试管混合并直接添加到无细胞反应中。
- 如果需要,使用PCR纯化试剂盒纯化产物,并将其洗脱在36μLdh2O中进行定量。确保模板浓度为~100 ng/μL。
6. 无细胞反应
注意:通过组合能量缓冲液、提取物和 RCA 模板来执行无细胞表达。使用PANOx-SP能量缓冲液的典型无细胞反应包括1.2mM ATP,GMP,UMP和CMP各0.85mM,30 mM磷酸烯醇丙酮酸盐,130mM谷氨酸钾,10 mM谷氨酸铵,12mM谷氨酸镁,1.5mM亚精胺,1mM腐胺,34μg/ mL亚叶酸,171μg/ mL大肠杆菌TRNA混合物, 20个未标记氨基酸各2mM,0.33mM NAD,0.27mM辅酶A(CoA),4mM草酸钾,57mM HEPES-KOH缓冲液(pH 7.5),0.24%体积的大肠杆菌提取物和可变量的DNA23,49。反应的体积可以变化,但15μL反应可以节省试剂的使用,并且足够小,可以在384黑壁微孔板49,50中使用。
- 如果表达荧光蛋白,如sfGFP,请准备读板器以在所需的激发/发射,温度和搅拌下读取。
- 如果使用384孔板,将60μLH2O等分到空样品孔边缘的孔中,以保持湿度并减少边缘效应。
- 将每个样品所需的各种组分加入试管中。添加足够多的内容以执行一式三份。板内的重复可以帮助确定变异的原因。
- 加入提取物,能量缓冲液,然后加入DNA。
- 用 ddH2O 稀释至最终所需体积。
- 通过上下移取溶液体积的一半10-20次来彻底混合该溶液。
- 将15μL等分试样中的反应混合物转移到微量滴定板中的所需孔中。
- 用无色密封膜密封板,以保持湿度并防止蒸发。
- 将密封的板放入读板器中,让反应完成。
- 如果表达不具有实时监测能力的蛋白质,请使用另一种温度受控的设备(例如热块)来孵育板。
7. 枯草杆菌蛋白酶测定
注意:如果在 补充序列#2中表达枯草杆菌蛋白酶BPN'(SBT(n))基因,请遵循此方案来测定活性。
- 在二甲基甲酰胺(DMF)中制备10μM的N-琥珀酰基-阿拉-阿拉-丙-脯对硝基苯胺的储备溶液。
- 设置读板器每20秒测量410nm处的吸光度10分钟,同时保持25°C的温度。
- 在平底无色96孔板中,等分94μLdH2O和1μL N-琥珀酰-阿拉-阿拉-丙-对硝基苯胺,步骤7.1。
- 加入步骤6.7中5μL成品无细胞反应,并使用设置为步骤7.2中描述的方案的读板仪读取。
结果
当在15μL反应中仅使用0.30μL未纯化的RCA DNA时,RCA模板中sfGFP的表达与pJL1质粒的表达相当(图2A)。事实上,在BL21 DE3 Star提取物中,将模板的量增加一倍和三倍似乎没有益处,这表明模板的饱和水平已经达到每次反应0.30μL。相反,当添加到来自SHuffle菌株的细胞提取物中时,增加RCA模板的量似乎有好处(图2B)28。对于某些蛋白质,可以非常快速地观察到结果,这会将整个工作流程(扩增和测定)压缩到24小时以下。然而,一些蛋白质需要较低的温度或具有较慢的折叠时间,这将增加获得结果的时间,但会影响此处介绍的工作流程。当表达枯草杆菌蛋白酶(SBT(n))时,可以观察到这一点,其中表达4小时后的测定时间不足以使SBT(n)成熟(图3A,失败结果的示例)。允许反应持续16小时可导致可检测的SBT(n)水平(图3B)。这种改进可能与温度有关,正如在文献中观察到的那样,其中探索了优化的温度条件59,60。

图1:最小遗传模板及其在初始PCR扩增步骤后所经历的过程的代表性示意图。 用HindIII消化模板,用T4连接酶循环,并用φ29聚合酶进一步扩增以产生大连接体。 请点击此处查看此图的放大版本。
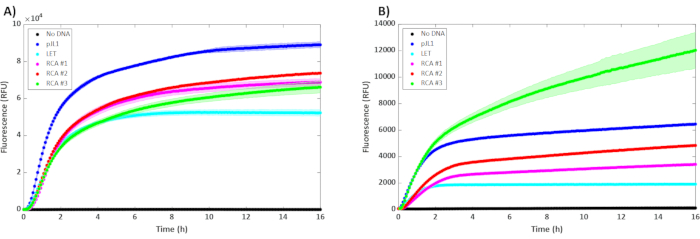
图2:未纯化的5 nM质粒(pJL1),5 nM线性模板(LET)和不同浓度的未纯化RCA的无细胞反应的结果。RCA #1,#2和#3在30°C(n = 3)下孵育的15μL反应中分别含有0.3μL,0.6μL和0.9μL未纯化的RCA产物。误差线表示±平均值的 1 SD。y轴是荧光,x轴是反应过程中经过的时间。sfGFP表达的动力学在(A)BL21DE3星和(B)T7SHuffle中表示。请点击此处查看此图的放大版本。

图3:与5 nM SBT(n)LET和不同浓度的未纯化RCA产物的无细胞反应。 RCA ng和pg对应于用于进行滚动圆扩增的DNA的浓度。未纯化的RCA产物用于在30°C(n = 3)下孵育的15μL反应中。误差线表示±平均值的 1 SD。y轴是410nm处的吸光度,x轴是测定中经过的时间量。反应进行了(A)4小时和(B)16小时。 请单击此处查看此图的放大版本。
| 克隆方法 | |
| 断续器 | 2 -4 小时 |
| 质粒消化 | 35 分 |
| 结扎 | 1 小时 |
| 转型 | 2 小时 |
| 通宵孵化 | 16 小时 |
| 测 序 | 24 - 48 小时 |
| 甘油储备准备 | 16 小时 |
| 生长和纯化 | 16 小时 |
| 总时间 | 46 - 72 小时 |
| RCA 方法 | |
| 断续器 | 2 - 4 小时 |
| 消化 | 35 分 |
| 结扎 | 1 小时 |
| 断续器 | 4 - 18 小时 |
| 欧洲常规武装力量 | 4 - 16 小时 |
| 总时间 | 12 - 40 小时 |
表1:简化的传统克隆方案与本文所涵盖的RCA方案之间的时间线比较。
补充文件:补充文件列出了序列。 序列#1是sfGFP(999个碱基对),序列#2是枯草杆菌蛋白酶BPN'(1344 bp)。 请点击此处下载此文件。
讨论
目的基因可以是任何所需的蛋白质,但最好从荧光蛋白开始,作为方便的报告者,以便在孔板读数仪上进行实时或终点读数,以便新采用该方法。对于新的蛋白质序列,复制所需蛋白质的氨基酸序列并将其粘贴到所需的密码子优化工具61,62中。密码子优化工具中通常有许多可用的大肠杆菌生物和菌株,但选择一般大肠杆菌选项将是合适的。优化后,仔细检查基因以确保先前选择的切点和引物序列不存在。如果是这样,则可以优化序列,直到序列不再存在。在某些情况下,如果重复优化始终导致内部 HindIII 切割位点,则可能需要将 HindIII 切割位点替换为不同的限制位点。最好尽可能多地使用相同的切割部位,以保持过程标准化,并降低手头保留多种限制性内切酶的成本。
在开始扩增之前,选择最适合LET的PCR试剂盒。具有<1,000个碱基对的LET可以使用简单的Taq聚合酶扩增,而具有≥1,000个碱基对的LET可能需要更高保真度的聚合酶63。根据试剂盒制造商和所需引物的退火温度设置扩增方案。退火温度对于成功扩增至关重要。一般的经验法则是选择比底漆Tm低5°C的退火温度。有免费的在线工具,可以根据引物58的顺序提供优化的退火温度。使用正确的退火温度对于生产高质量的DNA模板至关重要。
近年来,无细胞基因表达因其速度,简单性和对合成生物学原型的实用性而复兴。这项工作概述了一种在测试大型新型功能蛋白库时提高速度和易用性的方法。虽然传统的克隆方法可能需要数天或数周的时间,但该协议可以在不到24小时内完成。 表1 概述了传统克隆和RCA协议的典型时间范围。请注意,克隆方案已简化,并且省略了一些可选步骤,例如凝胶分离。 表1 也仅指常见的限制性消化方案;还有许多其他克隆方法,但这些方法需要类似的时间。这种酶促扩增方案的上限范围比克隆方法的较低范围需要更少的时间,特别是在考虑8小时工作日时。这主要是由于去除过夜孵育和缺乏测序确认。RCA方案完全基于PCR和RCA,与传统的克隆,转化和细胞培养技术相比,它们需要较少的专业技能。这种方法使没有克隆经验或获得转化和生长细胞所需资金的实验室可以获得无细胞表达。这种方法也非常适合于专注于细胞毒性蛋白的项目,在这些项目中,由于活细胞中的毒性,使用传统克隆的基因的克隆和繁殖是困难的。该RCA方案还能够将非常少量的环状DNA(DNA的皮克)扩增至CFE所需的水平。在 图3B中,在RCA之前,循环的SBT(n)模板被稀释至20 ng / μL和20 pg / uL。虽然观察到的降解速率不同,但两种反应在10分钟内导致相同量的底物降解。所提出的方法并不是要取代克隆,而是要取代克隆。质粒繁殖可以为无法匹配的无毒基因产生大量DNA。相反,这是一个在大规模文库筛选阶段的方便原型设计工具(酶促步骤适合使用标准流体处理程序进行自动化),这将有助于确定应克隆哪些序列以进行存档和进一步传播。
在设计无细胞反应时,有很大的灵活性,但必须考虑一些因素以确保成功。较小的反应具有较大的表面积与体积比,这对于气体交换非常有用,但反应需要完全覆盖孔的底部以进行准确的荧光测量。反应的温度也可以根据目的基因59而变化。在选择能量缓冲器时,用户有多种选择34,46。有些食谱比其他食谱更昂贵,但决定权留给用户。 大肠杆菌 提取物36也有许多潜在的来源。用户应熟悉数据提取生产协议,并决定哪个最适合他们的目的28,48,50,64,65,66,67.
当使用RCA扩增无细胞表达模板时,细菌菌株的选择非常重要。表达谱往往优于 FET。流行的BL21 DE3 Star菌株处理得很好,RCA的性能比LET好1.4倍,pJL1质粒的性能比最佳RCA浓度好约1.2倍(图2A)。另一方面,由于天然核酸酶23,28的存在,一些菌株表现出模板降解并且表现不佳。在这种情况下,SHuffle菌株似乎受益于增加的RCA模板。先前的文献表明,SHuffle提取物在PCR产物中表现不佳,但本研究中使用的最高浓度的未纯化RCA产物优于pJL1质粒(图2B)。功能性SBT(n)的表达(图3)是细胞毒性蛋白的一个例子,该蛋白由无细胞方便地制成,但由于毒性(无法在质粒中克隆该表达模板并在大肠杆菌中繁殖)而难以在活细胞中进行原型制作。与sfGFP不同,SBT(n)活性仅在表达4小时后就不能观察到(图3A)。在表达和成熟16小时后可检测到信号(图3B)。这些实例中使用的未纯化DNA来自100μL,这是四个稀释和组合的10μL RCA反应。如果每次反应仅使用0.30μL,则该储备液可以支持333个无细胞反应。
RCA模板与CFE的兼容性需要通过各种摘录进一步探索。线性产物的潜在并发症可以通过添加含有大肠杆菌chi序列或GamS蛋白52,53的短寡核苷酸来缓解。文献表明,添加chi寡核苷酸可以为线性模板提供比GamS蛋白53更大的保护。如果使用已知从线性模板中提供低产量的提取物,用户应分析每种保护剂的成本,并使用最适合其用途的提取物。另一个限制是由于所得串联物的性质,无法将RCA模板浓度转换为摩尔浓度。这意味着可以根据质量添加相同数量的最小模板,但它们将具有不同的串联体长度,这可能会影响表达水平。作者没有发现这在筛选/原型设计中是一个问题,因为每个孔的平均表达水平相同(低方差),但如果以较小的体积表达(例如,微滴)可能是一个问题。
无细胞基因表达有可能被用作蛋白质原型设计和设计-构建-测试工作流程中的重要工具。然而,大多数无细胞表达工作流程仍然依赖传统的质粒作为遗传模板。这减慢了该过程,并使无细胞表达无法充分利用用于筛选/原型设计目的。扩增模板并随后执行RCA可以快速产生足够的遗传模板用于许多反应,产生足够的蛋白质用于下游表征和功能测试。
披露声明
Nigel Reuel是BigHat Biosciences Inc.的科学顾问委员会成员,该公司使用无细胞系统来设计抗体。
致谢
作者承认NIH 1R35GM138265-01和NSF 2029532对该项目的部分支持。
材料
| Name | Company | Catalog Number | Comments |
| Alaline | Formedium | DOC0102 | |
| Ammonium glutamate | MP Biomedicals | MP21805951 | |
| Arginine | Formedium | DOC0106 | |
| Asparagine | Formedium | DOC0114 | |
| Aspartic Acid | Formedium | DOC0118 | |
| ATP | Sigma | A2383 | |
| Axygen Sealing Film | Corning | PCR-SP | |
| CMP | Sigma | C1006 | |
| Coenzyme A | Sigma | C3144 | |
| CutSmart Buffer | NEB | B7204S | Provided with HindIII |
| Cysteine | Formedium | DOC0122 | |
| DNA Clean and Concentrator Kit | Zymo Research | D4004 | Used for purifying DNA |
| dNTPs | NEB | N0447 | |
| E. coli tRNA | Sigma (Roche) | 10109541001 | |
| Folinic Acid | Sigma | 47612 | |
| Gene Fragment | IDT | ||
| Glutamic Acid | Formedium | DOC0134 | |
| Glutamine | Formedium | DOC0130 | |
| Glycine | Formedium | DOC0138 | |
| GMP | Sigma | G8377 | |
| HEPES | Sigma | H3375 | |
| HindIII-HF | NEB | R3104L | |
| Histidine | Formedium | DOC0142 | |
| Isoleucine | Formedium | DOC0150 | |
| Leucine | Formedium | DOC0154 | |
| Lysine | Formedium | DOC0158 | |
| Magnesium glutamate | Sigma | 49605 | |
| Methionine | Formedium | DOC0166 | |
| Microtiter Plate (384 well) | Greiner | 781906 | |
| Microtiter Plate (96 well) | Greiner | 655809 | |
| Multimode Plate Reader | BioTek | Synergy Neo2 | |
| NAD | Sigma | N8535 | |
| NanoPhotometer | Implen | NP80 | |
| OneTaq DNA Polymerase | NEB | M0480 | |
| PCR Tube | VWR | 20170-012 | |
| Phenylalanine | Formedium | DOC0170 | |
| Phosphoenolpyruvate | Sigma (Roche) | 10108294 | |
| Potassium glutamate | Sigma | G1501 | |
| Potassium oxalate | Fisher Scientific | P273 | |
| Proline | Formedium | DOC0174 | |
| Putrescine | Sigma | P5780 | |
| Serine | Formedium | DOC0178 | |
| Spermidine | Sigma | S0266 | |
| T4 DNA Ligase | NEB | M0202S | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | Provided with T4 DNA Ligase |
| TempliPhi Amplification Kit | Cytiva | 25640010 | Used for RCA |
| Thermal Cycler | Biorad | C1000 Touch | |
| Thermoblock | Eppendorf | ThermoMixer FP | |
| Threonine | Formedium | DOC0182 | |
| Tryptophan | Formedium | DOC0186 | |
| Tyrosine | Formedium | DOC0190 | |
| UMP | Sigma | U6375 | |
| Valine | Formedium | DOC0194 |
参考文献
- Sun, Q., et al. A simple and low-cost paper-based colorimetric method for detecting and distinguishing the GII.4 and GII.17 genotypes of norovirus. Talanta. 225, 121978 (2021).
- Pardee, K., et al. Low-Cost Detection of Zika Virus Using Programmable Biomolecular Components. Cell. 165 (5), 1255-1266 (2016).
- Pardee, K., et al. Paper-based synthetic gene networks. Cell. 159 (4), 940-954 (2014).
- Ma, D., Shen, L., Wu, K., Diehnelt, C. W., Green, A. A. Low-cost detection of norovirus using paper-based cell-free systems and synbody-based viral enrichment. Synthetic Biology. 3 (1), (2018).
- Park, S., Lee, J. W. Detection of coronaviruses using rna toehold switch sensors. International Journal of Molecular Sciences. 22 (4), 1772 (2021).
- Cao, M., Sun, Q., Zhang, X., Ma, Y., Wang, J. Detection and differentiation of respiratory syncytial virus subgroups A and B with colorimetric toehold switch sensors in a paper-based cell-free system. Biosensors and Bioelectronics. 182, 113173 (2021).
- Mcnerney, M. P., et al. Point-of-care biomarker quantification enabled by sample-specific calibration. Science Advances. 5 (9), (2019).
- Silverman, A. D., Akova, U., Alam, K. K., Jewett, M. C., Lucks, J. B. Design and optimization of a cell-free atrazine biosensor. ACS Synthetic Biology. 9 (3), 671-677 (2020).
- Salehi, A. S. M., et al. Cell-free protein synthesis approach to biosensing hTRβ-specific endocrine disruptors. Analytical Chemistry. 89 (6), 3395-3401 (2017).
- Garamella, J., Majumder, S., Liu, A. P., Noireaux, V. An adaptive synthetic cell based on mechanosensing, biosensing, and inducible gene circuits. ACS Synthetic Biology. 8 (8), 1913-1920 (2019).
- Glasscock, C. J., et al. Dynamic control of pathway expression with riboregulated switchable feedback promoters. ACS Synthetic Biology. 16, (2019).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- Pardee, K., et al. Portable, on-demand biomolecular manufacturing. Cell. 167 (1), 248-259 (2016).
- Nelson, J. A. D., et al. Hydrofoam and oxygen headspace bioreactors improve cell-free therapeutic protein production yields through enhanced oxygen transport. Biotechnology Progress. 37 (2), 3079 (2020).
- Cai, Q., et al. A simplified and robust protocol for immunoglobulin expression in Escherichia coli cell-free protein synthesis systems. Biotechnology Progress. 31 (3), 823-831 (2015).
- Ogonah, O. W., Polizzi, K. M., Bracewell, D. G. Cell free protein synthesis: a viable option for stratified medicines manufacturing? A brief history of cell free synthesis systems. Current Opinion in Chemical Engineering. 18, 77-83 (2017).
- Zawada, J. F., et al. Microscale to manufacturing scale-up of cell-free cytokine production-a new approach for shortening protein production development timelines. Biotechnology and Bioengineering. 108 (7), 1570-1578 (2011).
- Stark, J. C., et al. On-demand biomanufacturing of protective conjugate vaccines. Science Advances. 7 (6), (2021).
- Huang, A., et al. BioBitsTM Explorer: A modular synthetic biology education kit. Science Advances. 4 (8), 1-11 (2018).
- Stark, J. C., et al. BioBits health: classroom activities exploring engineering, biology, and human health with fluorescent readouts. ACS Synthetic Biology. 8 (5), 1001-1009 (2019).
- Stark, J. C., et al. BioBitsTM Bright: A fluorescent synthetic biology education kit. Science Advances. 4 (8), 33 (2018).
- Shinoda, T., et al. Cell-free methods to produce structurally intact mammalian membrane proteins. Scientific Reports. 6, (2016).
- Dopp, J. L., Rothstein, S. M., Mansell, T. J., Reuel, N. F. Rapid prototyping of proteins: Mail order gene fragments to assayable proteins within 24 hours. Biotechnology and Bioengineering. 116 (3), 667-676 (2019).
- Sachse, R., Dondapati, S. K., Fenz, S. F., Schmidt, T., Kubick, S. Membrane protein synthesis in cell-free systems: From bio-mimetic systems to bio-membranes. FEBS Letters. 588 (17), 2774-2781 (2014).
- Salehi, A. S. M., et al. Cell-free protein synthesis of a cytotoxic cancer therapeutic: Onconase production and a just-add-water cell-free system. Biotechnology Journal. 11 (2), 274-281 (2016).
- Georgi, V., et al. On-chip automation of cell-free protein synthesis: New opportunities due to a novel reaction mode. Lab on a Chip. 16 (2), 269-281 (2016).
- Thoring, L., et al. Cell-free systems based on CHO cell lysates: Optimization strategies, synthesis of "difficult-to-express" proteins and future perspectives. PLoS One. 11 (9), (2016).
- Dopp, J. L., Reuel, N. F. Simple, functional, inexpensive cell extract for in vitro prototyping of proteins with disulfide bonds. Biochemical Engineering Journal. 164, 107790 (2020).
- Isaksson, L., Enberg, J., Neutze, R., Göran Karlsson, B., Pedersen, A. Expression screening of membrane proteins with cell-free protein synthesis. Protein Expression and Purification. 82 (1), 218-225 (2012).
- Techner, J. M., et al. High-throughput synthesis and analysis of intact glycoproteins using SAMDI-MS. Analytical Chemistry. 92 (2), 1963-1971 (2020).
- Kim, H. C., et al. Implementing bacterial acid resistance into cell-free protein synthesis for buffer-free expression and screening of enzymes. Biotechnology and Bioengineering. 112 (12), 2630-2635 (2015).
- Rolf, J., Siedentop, R., Lütz, S., Rosenthal, K. Screening and identification of novel cGAS homologues using a combination of in vitro and in vivo protein synthesis. International Journal of Molecular Sciences. 21 (1), (2020).
- Haslinger, K., Hackl, T., Prather, K. L. J. Rapid in vitro prototyping of O-methyltransferases for pathway applications in Escherichia coli. bioRxiv. , (2020).
- Dopp, J. L., Tamiev, D. D., Reuel, N. F. Cell-free supplement mixtures: Elucidating the history and biochemical utility of additives used to support in vitro protein synthesis in E. coli extract. Biotechnology Advances. 37 (1), 246-258 (2018).
- Gregorio, N. E., Levine, M. Z., Oza, J. P. A user's guide to cell-free protein synthesis. Methods and Protocols. 2 (1), 24 (2019).
- Cole, S. D., Miklos, A. E., Chiao, A. C., Sun, Z. Z., Lux, M. W. Methodologies for preparation of prokaryotic extracts for cell-free expression systems. Synthetic and Systems Biotechnology. 5 (4), 252-267 (2020).
- Chiba, C. H., Knirsch, M. C., Azzoni, A. R., Moreira, A. R., Stephano, M. A. Cell-free protein synthesis: advances on production process for biopharmaceuticals and immunobiological products. BioTechniques. 70, (2021).
- Laohakunakorn, N. Cell-free systems: A proving ground for rational biodesign. Frontiers in Bioengineering and Biotechnology. 8, 788 (2020).
- Dondapati, S. K., Stech, M., Zemella, A., Kubick, S. Cell-free protein synthesis: A promising option for future drug development. BioDrugs. , 1-22 (2020).
- Noireaux, V., Liu, A. P. The new age of cell-free biology. Annual Review of Biomedical Engineering. 22, 51-77 (2020).
- Khambhati, K., et al. Exploring the Potential of Cell-Free Protein Synthesis for Extending the Abilities of Biological Systems. Frontiers in Bioengineering and Biotechnology. 7, (2019).
- Carlson, E. D., Gan, R., Hodgman, C. E., Jewett, M. C. Cell-free protein synthesis: Applications come of age. Biotechnology Advances. 30 (5), 1185-1194 (2012).
- Rosenblum, G., Cooperman, B. S. Engine out of the chassis: Cell-free protein synthesis and its uses. FEBS Letters. 588 (2), 261-268 (2014).
- Swartz, J. R. Transforming biochemical engineering with cell-free biology. AIChE Journal. 58 (1), 5-13 (2012).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Caschera, F., Noireaux, V. A cost-effective polyphosphate-based metabolism fuels an all E. coli cell-free expression system. Metabolic Engineering. 27, 29-37 (2015).
- Levine, M. Z., et al. Activation of energy metabolism through growth media reformulation enables a 24-hour workflow for cell-free expression. ACS Synthetic Biology. 9 (10), 2765-2774 (2020).
- Hunt, J. P., et al. Streamlining the preparation of "endotoxin-free" ClearColi cell extract with autoinduction media for cell-free protein synthesis of the therapeutic protein crisantaspase. Synthetic and Systems Biotechnology. 4 (4), 220-224 (2019).
- Dopp, J. L., Jo, Y. R., Reuel, N. F. Methods to reduce variability in E. Coli-based cell-free protein expression experiments. Synthetic and Systems Biotechnology. 4 (4), 204-211 (2019).
- Sun, Z. Z., et al. Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology. Journal of Visualized Experiments: JoVE. , e50762 (2013).
- Schinn, S. M., Broadbent, A., Bradley, W. T., Bundy, B. C. Protein synthesis directly from PCR: Progress and applications of cell-free protein synthesis with linear DNA. New Biotechnology. 33 (4), 480-487 (2016).
- Sitaraman, K., et al. A novel cell-free protein synthesis system. Journal of Biotechnology. 110 (3), 257-263 (2004).
- Marshall, R., Maxwell, C. S., Collins, S. P., Beisel, C. L., Noireaux, V. Short DNA containing χ sites enhances DNA stability and gene expression in E. coli cell-free transcription-translation systems. Biotechnology and Bioengineering. 114, 2137-2141 (2017).
- Sun, Z. Z., Yeung, E., Hayes, C. A., Noireaux, V., Murray, R. M. Linear DNA for rapid prototyping of synthetic biological circuits in an escherichia coli based TX-TL cell-free system. ACS Synthetic Biology. 3 (6), 387-397 (2014).
- . Addgene: pJL1 Available from: https://www.addgene.org/69496/ (2021)
- . IDT Codon Optimization Tool Available from: https://www.idtdna.com/pages/tools/codon-optimization-tool (2021)
- Hadi, T., et al. Rolling circle amplification of synthetic DNA accelerates biocatalytic determination of enzyme activity relative to conventional methods. Scientific Reports. 10 (1), 10279 (2020).
- . New England Biolabs Tm Calculator Available from: https://tmcalculator.neb.com/#!/main (2021)
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. Coli RNA polymerase and sigma factor 70. Journal of Biological Engineering. 4, (2010).
- Colant, N., et al. A rational approach to improving titer in Escherichia coli-based cell-free protein synthesis reactions. Biotechnology Progress. 37 (1), 3062 (2021).
- Burgess-Brown, N. A., et al. Codon optimization can improve expression of human genes in Escherichia coli: A multi-gene study. Protein Expression and Purification. 59, 94-102 (2008).
- Maertens, B., et al. Gene optimization mechanisms: A multi-gene study reveals a high success rate of full-length human proteins expressed in Escherichia coli. Protein Science. 19 (7), 1312-1326 (2010).
- Eckert, K. A., Kunkel, T. A. DNA polymerase fidelity and the polymerase chain reaction. Genome Research. 1 (1), 17-24 (1991).
- Dopp, J. L., Reuel, N. F. Process optimization for scalable E. coli extract preparation for cell-free protein synthesis. Biochemical Engineering Journal. 138, 21-28 (2018).
- Liu, D. V., Zawada, J. F., Swartz, J. R. Streamlining Escherichia Coli S30 extract preparation for economical cell-free protein synthesis. Biotechnology Progress. 21 (2), 460-465 (2005).
- Levine, M. Z., Gregorio, N. E., Jewett, M. C., Watts, K. R., Oza, J. P. Escherichia coli-Based Cell-Free Protein Synthesis: Protocols for a robust, flexible, and accessible platform technology. Journal of Visualized Experiments: JoVE. (144), e58882 (2019).
- Kwon, Y. C., Jewett, M. C. High-throughput preparation methods of crude extract for robust cell-free protein synthesis. Scientific Reports. 5, (2015).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。