Method Article
無細胞システムを用いたタンパク質プロトタイピングのための、最小の線形テンプレートの増幅のための迅速で酵素的な方法
要約
この研究は、クローニングや生きている細胞を使用せずに、合成遺伝子断片からタンパク質スクリーニングキャンペーンのための大量(μg-mg)量のDNAを作成するためのプロトコルを記述しています。最小のテンプレートは酵素的に消化され、円形化され、等熱転がり円増幅を使用して増幅される。無細胞発現反応は、非精製物で行うことができる。
要約
このプロトコルは、最小DNAテンプレートの設計と酵素増幅のステップを説明し、無細胞発現を用いて24時間未満でアッセイ可能タンパク質の迅速なプロトタイピングを可能にする。ベンダーからDNAを受け取った後、遺伝子断片はPCR増幅、切断、円形化、およびクライオバンクされる。少量のバンクDNAは、等温転環増幅(RCA)を使用して、希釈され、有意に増幅されます(最大106x)。RCAは、出発物質のピコグラムレベル(すべての開始合成フラグメントが使用されている場合はmgレベル)から最小発現テンプレートのマイクログラム量を得ることができます。この作業では、20 pgの開始量は最終製品の4 μgをもたらしました。得られたRCA製品(最小鋳型の連結物)を、精製工程なしで無細胞反応に直接添加することができる。この方法は完全にPCRベースであるため、自動液体処理システムと組み合わせることで、将来のハイスループットスクリーニングの取り組みが可能になる可能性があります。
概要
無細胞遺伝子発現(CFE)は、多くの用途を備えた強力なツールとして登場しました。このような用途には、疾患検出1、2、3、4、5、6、微量栄養素および小分子検出7、8、9、10、11、12、バイオマニュファクチャリング13、14、15、16、17が含まれます。 ,18, 教育19,20,21, 製造難しいタンパク質17,22,23,24,25,26,27, およびバリアントスクリーニング23,28,29,30,32 、33.これは、無細胞システムの開放性と柔軟性が与えるためです。多くの偉大なレビュー記事は、技術上の歴史的教育と将来の展望を提供しています34,35,36,37,38,39,40,41,42,43, 44.
典型的な無細胞反応は、細胞抽出物、エネルギーミックス、および遺伝的テンプレートの3つの主要な成分で構成されています。アクティブセル抽出物には、転写と翻訳(TXTL)に必要なすべての機械が含まれ、さまざまな方法で処理することができます36.解糖性中間体、電解質、アミノ酸、およびエネルギーミックスの補因子は、TXTLプロセスをサポートします。これは、無細胞実験45における変動の主要な源であり、多くの方法で34、46を調製することができる。遺伝的テンプレートの調製は、従来のクローニング法が優れた発現特性を有するプラスミドをもたらすため、改善が少なくなっています。これらの伝統的な方法の欠点は、それらを構築し、伝播するために必要な生物学的専門知識のターンアラウンドタイムと量です。最近の最適化の取り組みにより、エネルギーミックス調製49,50と並行して行うことができる細胞抽出製剤47,48の簡単な24時間のワークフローが生まれている。ただし、従来のクローニングは CFE プロトタイピングのタイムラインに複数の日を追加します (表 1)23.市販遺伝子断片から迅速に増幅されたPCR産物は直接51を使用できますが、約5つの反応(従来の15μL量)に相当するDNAの1μgしか生成されていないため、試作実験の数が制限されます。これらの追加の循環化と等温増幅のステップにより、DNAのミリグラム量より大きい(1mgの5,000反応以上)が可能です。これは、タンパク質または組み合わせ酵素ネットワーク(無細胞代謝工学)のハイスループットスクリーニングで行うことができるテストの数を劇的に増加させます。また、高濃度DNAとして線形テンプレートライブラリを効果的に保存することができます。さらに、材料科学の用途(タンパク質ベースの繊維およびヒドロゲル)に必要な大量のタンパク質を試作するために、テンプレートの量を増やす必要があります。線形テンプレートのいくつかの制限は、BL21 DE3 Starからの抽出物を使用するか、最近発見された方法を使用して、リニアテンプレートを劣化52、53、54から保護することで克服できます。しかし、これは、PCR増幅のためのベンダー製DNAの在庫が限られているか、クローン作成に必要な生物学的専門知識と機器の問題を持つことに対処していません。
この研究は、少量のベンダー生産遺伝子断片(通常は500〜1000ngの凍結乾燥粉末)から得ることができる発現テンプレートの量を増やすために明示的に設計されたプロトコルを提示する。記載された方法は、プラスミドでの伝統的なクローニングを行ったり、生きている細胞での変換および伝播を行うために必要なスキルを必要としない。メールで遺伝子断片を受信すると、ユーザは等温転環増幅(RCA)を採用することにより、多くの無細胞反応のための十分なテンプレートを生成することができる(図1)23。ベンダーから受け取ったDNAの量は限られたスクリーニング努力に十分かもしれませんが、すぐに枯渇し、遺伝子断片の再購入には時間とコストがかかります。この方法は、特に毒性があり、大腸菌でクローンを作成することが困難な遺伝子に適しています。
プロトコル
1. 遺伝子断片の設計
注:遺伝子断片は、プロモーター、リボソーム結合部位(RBS)、開始コドン、目的遺伝子、ターミネーターを含む、転写/翻訳に必要なすべての遺伝的要素を有するべきである。ターミネータは線形式テンプレート (LET) には必要ありませんが、ユーザーが配列をプラスミドに挿入することを決定した場合は重要です。これらの配列は、T7プロモーターを使用するpJL1-sfGFPプラスミド55( マイケル・ジュエットの研究室からの贈り物)から持ち上げられた。これらの必要な遺伝的要素に加えて、制限酵素カット部位は、プロモーター(5'カット部位)の前に6塩基対を追加し、ターミネーター(3'カット部位)後に別の6塩基対を追加し、この場合、HindIII(他の制限酵素を使用することができるが、ライブラリに保持するために必要な数を減らすために1つの高忠実度制限酵素で配列を標準化することは有用である)。プライマーサイトは、5'カットサイトの上流に10塩基対を追加し、この場合は標準化されたM13プライマーシーケンスを使用して3'カット部位の下流に10基対を追加します(プライマーは安価なストックアイテムです)。制限酵素部位および使用するプライマーは、ユーザの裁量で行われる。ただし、ユーザーは、シーケンスがテンプレート内の他の場所に存在しないことを確認する必要があります(不要なカットや増幅開始部位を作成したくない)。この作業で使用されるテンプレートの順序は、補足資料で詳述されています。これらの手順は、この基本テンプレートから変更するために使用されます。
- 発現する望ましい遺伝子を決定し、 大腸菌で発現された場合はアミノ酸配列または遺伝的配列を得る。
- アミノ酸配列の場合は、多くの標準的なベンダーツール56の1つを使用して大腸菌のコドン最適化を行う。サプリメントで提供されるテンプレートを使用する場合, 最適化されたシーケンスは、ヒンド III 制限サイト (AAGCTT) がないことを確認します。その場合は、HindIII サイトがなくなるまでシーケンスの最適化を続けます。
- シーケンスをコピーし、目的の遺伝子が示されている 補足シーケンス#1 の提供されたテンプレートに貼り付けます。sfGFP を表現する場合は、 補足シーケンス #1 を使用します。サチリシンを発現する場合は、 補助シーケンス#2 を使用します。
- 最小のテンプレートと必要なプライマーを好ましいDNA合成サービスから注文します。
2. 遺伝子断片とプライマーの再懸濁
注:遺伝子断片を受け取った後、再懸濁液のためのメーカーのプロトコルに従うか、DNAストックを作成するには、この簡単なガイドを使用してください。
- チューブを遠心分離(5sの場合は300 x g)、 底部にDNAペレットを集める。
- 二重蒸留水(ddH2O)を加え、DNAテンプレートの最終濃度を10 ng/μLにします。
- 5~10sの中の設定で溶液をボルテックスする。
- 50°Cで20分間インキュベートしてペレット全体を溶解します。
- 再び簡単に渦
- チューブの底部に溶液を収集するために5 sのための300 x g で遠心分離機。
- -20 °Cで保存するか、PCRで使用してください。
- プライマーをヌクレアーゼフリー水に再懸濁して、100 μMプライマーストックを準備します。添加する水の量を決定するには、凍結乾燥したプライマーのナノモルの数に10を掛けます。例えば、チューブに45 nMの凍結乾燥プライマーが含まれている場合、450 μLのddH2Oを加え、溶液を渦液に加えます。
- プライマーストック溶液を-20°Cで保管するか、増幅を継続します。
3. PCRによる遺伝子断片の増幅
注: 目的の遺伝子に適した PCR キットを決定します。より小さい遺伝子(<1,000 kb)は安価なTaqポリメラーゼに対してより適しているかもしれませんが、より大きな遺伝子(≥1,000 kb)はエラーを減らすために高忠実度ポリメラーゼの恩恵を受けるかもしれません。ユーザーが初期遺伝子断片の保存に関心がない場合、この初期PCR増幅は必要ありません(循環化で複数の試みを提供し、LETとRCA製品の比較研究を可能にします)。また、このPCR増幅LETは反応に直接使用できることに注意することも重要です。しかし、導入で述べたように、さらなる増幅ステップが無視された場合にのみ、反応の数に制限があります。消化およびライゲーションは、再懸濁された遺伝子断片57 に対して直接行うことができる(1つが確実であれば、追加の循環化ストックを実行するためにより多くのLETを必要としない)。この場合は、セクション 3 をスキップしてセクション 4 に進みます。PCR を実行するには、次の手順を実行します。
- ステップ2.8の100μMストックを使用して、10μMの動作ソリューションを作成します。多くの PCR キット プロトコルでは、プライマーの 10 μM ソリューションが必要です。
- サーマルサイクラーをプログラムして、キットメーカーのプロトコルに従って反応を行います。異なるキットは、わずかに変化したサイクリングパラメータを必要とします。 材料表に記載されているキットの場合、条件は初期変性の30 sのための94 °Cです。変性の30 sのための94°Cの30サイクル、プライマーアニーリングの30 sのための45°C、および拡張の60 sのための68°C;5分間68°Cで最終延長。そして最後に、10°Cの無期限保持。
- 正しい伸び時間(増幅される遺伝子の長さに応じて可変)を選択することを確認してください。1,000 bp ごとに 1 分の伸び時間を持ちます。
- プライマーの正しいアニーリング温度を入力するようにしてください。入力として両方のプライマーを使用するオンライン Tm 計算機を使用して、最適なアニーリング温度58を決定します。M13プライマーを使用する場合、45°Cの焼鈍温度で十分です。
- サイクル数を決定する場合は、製造元のプロトコルを参照してください。
- PCRを行う場合は、dNTPsを解凍して渦を出します。キットに用意されている PCR バッファーを使用します。
- 1 つの PCR チューブで、製造元のプロトコルで指示されたキット コンポーネントをすべて組み合わせます。増幅を成功させるには、1 μLの再懸濁したDNAストックを追加します(ステップ2.6)。
- 混合物を5~10sの中で渦を緩やかに均質化する。あるいは、ピペットは体積の半分を渦に10〜20回上下する。
- PCR反応を行います。
- PCR プロトコルに最終冷却ステップが含まれていない場合は、反応を 10 °C で 5 分間冷却してから、チューブの底部に凝縮を駆動させるため取り除きます。
- ベンダーの指示に従って PCR クリーンアップ キットを使用して反応を浄化します。
- 1.5 mLチューブに、DNA結合バッファーとPCRサンプルをそれぞれ5:1の比率で加えます。
- この混合物を16,000 x g でスピンカラムと遠心分離機に1分間移します。フロースルーを破棄します。
- 200 μL の DNA 洗浄バッファーをカラムに加え、室温で 1 分間インキュベートします。
- 16,000 x g で1分間遠心分離機を使用し、フロースルーを廃棄します。
- 1分インキュベーションステップなしでステップ2.8.3と2.8.4を繰り返します。
- 遠心分離機は16,000 x g で1〜2分を追加し、残りのバッファを除去する。
- ddH2Oの46 μLでDNAを溶出する。
- 分光光度計を用いて精製したDNAを定量化する。
- 精製したDNAを-20°Cに保存するか、次の工程に進みます。
4. 消化と回覧
注:さらに増幅は、RCAに続いてDNAを円形化することによって達成することができる。DNAを消化して、テンプレートを循環処理に備えます。これにより、プライマーシーケンスが削除され、テンプレートの 5' と 3' の両端にスティッキーエンドが作成されます。これらの末端をライゲーション反応により再び付着する。
- PCRチューブで、ステップ3.8から必要なバッファー5 μL、20 U の HindIII、精製された DNA の 45 μL を組み合わせます。
- この混合物をピペットで穏やかに均質化します。
- 37°Cで15分間、サーマルサイクラーで混合物をインキュベートします。
- 80°Cで20分間インキュベートすることにより、ヒンドIIIを不活性化します。
- チューブの底部に凝縮を駆動するために除去する前に10°Cに冷却する反応を可能にします。
- 5 μLのT4リガーゼバッファーと800 UのT4リガーゼを新たに消化したDNAに加えます。
- 必要に応じて、T7リガーゼを使用します。
- この混合物をピペットで穏やかに均質化します。
- 25°Cで1時間の混合物をインキュベートし、回覧反応を行う。
- ベンダーの指示に従って PCR クリーンアップ キットを使用して反応を浄化します。手順 3.8 で説明した同じプロトコルを使用します。
- 分光光度計を用いてDNAを定量化する。期待値は約 20 ng/μL です。
- -20°Cで保存するか、次のステップに進みます。
5. 等温圧圧円増幅
注:ローリングサークル増幅(RCA)は、市販のキットを使用するか、個別に購入したコンポーネントを使用して実行できます。メーカーのプロトコルに従うと、増幅が成功します。キットは、典型的には、サンプルバッファー、反応バッファー、およびφ29ポリメラーゼのようなストランド変位ポリメラーゼを含む。複数の反応チューブを組み合わせて、無細胞発現のための大量のDNAを生成することができます(出発物質の20 pgから4 μg)。次のプロトコルは効率的に動作します。
- 1つのチューブで、サンプルバッファーの20 μL、反応バッファーの20 μL、酵素の0.8 μL、およびステップ4.9からの円形発現テンプレート(CET)の1 μLを組み合わせます。
注:これは〜20 ngの総DNA質量を有しますが、RCAはピコグラム量で働くことができるので、高スループットスクリーニングで材料が大幅に必要な場合はCETの希釈と極端な酵素増幅を可能にします。 - 混合物をピペットとアリコート10μLで均質化し、混合物を4つの別々のチューブに分けます。
- 30°Cで4〜18時間インキュベートする。
- 65°Cで10分間インキュベートすることにより、酵素を不活性化する熱。チューブの底部の結露を促すために、温度を12°Cに5分間下げます。
注: サーマルサイクラー上の自動プロトコルですべての温度ステップを組み合わせるのが最も簡単です。 - 得られた溶液を、各チューブに15μLのddH2Oを加えて希釈します。
- すべてのチューブを組み合わせて、無細胞反応に直接追加します。
- 必要に応じて、PCRクリーンアップキットを使用して製品を精製し、36 μLのddH2Oで溶出して定量化します。テンプレート濃度が約100 ng/μLであることを確認します。
6. 無細胞反応
注: エネルギー バッファー、抽出、および RCA テンプレートを組み合わせてセルフリー式を実行します。PANOx-SPエネルギーバッファーを用いた一般的な無細胞反応は、1.2 mM ATP、0.85 mMそれぞれGMPで構成されており、 UMP、およびCMP、30 mMホスホエノールピルビン酸、130 mMカリウムグルタミン酸、10mMアンモニウムグルタミン酸、12mMマグネシウムグルタミン酸、1.5mMスペルミジン、1mMプトレシン、フォリン酸34 μg/mL、171μg/mLのフォリン酸、171 μg/mLのERNA混合物 20個の非標識アミノ酸、0.33 mM NAD、0.27 mM補酵素A(CoA)、4mMシュウ酸カリウム、57 mM HEPES-KOHバッファー(pH 7.5)、0.24%の大腸菌抽出物、および可変量のDNA23、49。反応の量はさまざまですが、15 μLの反応は試薬の使用量を節約でき、384の黒壁マイクロプレート49,50での使用に十分小さいです。
- sfGFPなどの蛍光タンパク質を発現する場合は、希望の励起/放出、温度、攪拌で読み取るプレートリーダーを用意します。
- 384ウェルプレートを使用する場合、湿度を維持し、エッジ効果を低減するために、空のサンプルによく接するウェルにH2Oのアリコート60 μL。
- 各サンプルのチューブに必要なさまざまなコンポーネントを追加します。トリプリケートを実行するのに十分な量を追加します。プレート内の複製は、変動の原因を特定するのに役立ちます。
- 抽出物、エネルギーバッファー、次にDNAを追加します。
- ddH2Oで最終的な必要なボリュームに希釈します。
- 溶液の半分を上下に10~20回ピペット処理して、この溶液を完全に混ぜます。
- 15 μL アリコートで反応混合物をマイクロティタープレート内の所望のウェルに移します。
- 耐湿を維持し、蒸発を防ぐために、無色のシールフィルムでプレートを密封します。
- プレートリーダーに密封プレートを置き、反応を完了させます。
- ライブ監視機能を持たないタンパク質を発現する場合は、熱ブロックなどの別の温度制御装置を用いてプレートをインキュベートする。
7. サブティリンアッセイ
注: 副配列#2でサブチリシンBPN'(SBT(n))遺伝子を発現する場合、このプロトコルに従って活性をアッセイする。
- ジメチルホルムアミド(DMF)でN-スクシニル-アラ-プロフェp-ニトロアニリドの10 μMストック溶液を調製します。
- プレートリーダーをセットし、25°Cの温度を保ちながら、20分ごとに410 nmで吸光度を測定します。
- 平らな底、無色96ウェルプレート、ddH2Oのアリコート94 μLおよびステップ7.1からのN-スクシニルアラアラ-プロ-Phe p-ニトロアニリドの1 μL。
- ステップ6.7から完成した無細胞反応の5μLを加え、ステップ7.2で説明したプロトコルにセットされたプレートリーダーを使用して読み取ります。
結果
RCAテンプレートからのsfGFPの発現は、15μL反応で0.30μLの未精製RCA DNAのみを使用した場合のpJL1プラスミドと同等であった(図2A)。実際、テンプレートの量を倍増して3倍にすることはBL21 DE3 Star抽出物では何のメリットも提供していないようで、反応あたり0.30 μLですでに飽和レベルのテンプレートを示唆しています。逆に、SHuffle株から供給される細胞抽出物に添加するとRCAテンプレートの量を増やす利点があるようです(図2B)28。一部のタンパク質では、結果は非常に迅速に観察することができ、ワークフロー全体(増幅およびアッファー)を24時間下に圧縮します。ただし、一部のタンパク質は温度が低いか、折り畳み時間が遅く、結果が得られるまでの時間は長くなりますが、ここで示すワークフローに影響を与えます。これは、4時間の発現後のアッシングがSBT(n)成熟に十分な長さではなかったサブチリシン(SBT(n))を表現する場合に観察することができます(図3A、失敗した結果の例)。反応を16時間まで続けることを可能にすることで、SBT(n) の検出可能なレベルに至る可能性があります (図 3B)。この改善は、最適化された温度条件が59,60を探索した文献で観察されたように、温度依存性である可能性がある。

図1:最小の遺伝子テンプレートと最初のPCR増幅ステップ後に行うプロセスの代表的な概略図。 テンプレートはHindIIIで消化され、T4リガーゼで円形化され、さらにφ29ポリメラーゼで増幅され、大きな連結体を作り出します。 この図の大きなバージョンを表示するには、ここをクリックしてください。
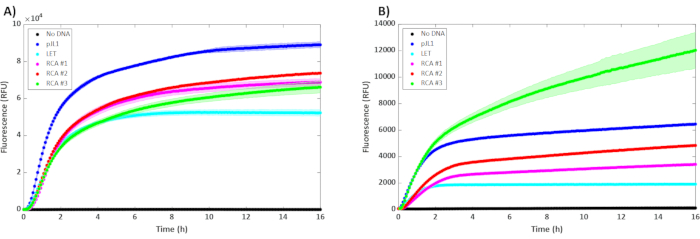
図2: 無精製5 nMのプラスミド(pJL1)、5 nMの線形鋳型(LET)、および未精製RCAの様々な濃度を用いた無細胞反応の結果。RCA #1、#2、#3は、30°C(n = 3)でインキュベートされた15 μLの非精製RCA生成物の0.3 μL、0.6 μL、および0.9 μL(それぞれ)を含んでいた。誤差範囲は平均から±1 SDを表します。y軸は蛍光であり、x軸は反応中に経過した時間です。sfGFP発現の動態は、(A)BL21 DE3スターおよび(B)T7シャッフレで表される。この図の大きなバージョンを表示するには、ここをクリックしてください。

図3:5 nMのSBT(n)LETと不純精製RCA生成物の濃度を変化させる無細胞反応RCA ngおよびpgは、転環増幅を行うために使用されるDNAの濃度に対応する。非精製RCA生成物を、30°C(n=3)でインキュベートした15μL反応で使用した。誤差範囲は平均から±1 SDを表します。y軸は410nmの吸光度であり、x軸はアッセイで経過した時間です。反応は、(A)4時間と(B)16時間に行われました。
| クローニング方法 | |
| PCR | 2 -4時間 |
| プラスミド消化 | 35分 |
| 結紮 | 1時間 |
| 変換 | 2時間 |
| 夜間インキュベーション | 16時間 |
| シークエンシング | 24 - 48時間 |
| グリセロールストック準備 | 16時間 |
| 成長と浄化 | 16時間 |
| 合計時間 | 46 - 72時間 |
| RCAメソッド | |
| PCR | 2 - 4時間 |
| 消化 | 35分 |
| 結紮 | 1時間 |
| RCA | 4 - 18時間 |
| CFE | 4 - 16時間 |
| 合計時間 | 12 - 40時間 |
表1: 簡体となった従来のクローン作成プロトコルと、ここで説明する RCA プロトコルとの間のタイムラインの比較.
補足ファイル: 補足ファイルにはシーケンスがリストされます。 配列#1はsfGFP(999塩基対)であり、配列#2はサブチリンBPN'(1344 bp)である。 このファイルをダウンロードするには、ここをクリックしてください。
ディスカッション
目的の遺伝子は任意の所望のタンパク質であり得るが、この方法の新しい採用者のためのウェルプレートリーダー上のリアルタイムまたはエンドポイント読み出しのための便利なレポーターとして蛍光タンパク質から始めることをお勧めします。新しいタンパク質配列については、所望のタンパク質のアミノ酸配列をコピーし、所望のコドン最適化ツール61,62に貼り付ける。コドン最適化ツールには通常多くの生物や大腸菌の株がありますが、一般的な大腸菌オプションを選択することが適しています。最適化後、遺伝子を再確認して、以前に選択した切断部位とプライマー配列が存在しないことを確認します。その場合、シーケンスが存在しなくなるまでシーケンスを最適化できます。状況によっては、最適化を繰り返し内部 HindIII カット サイトに一貫して発生する場合は、HindIII カット サイトを別の制限サイトに置き換える必要があります。プロセスを標準化し、複数の制限酵素を手元に保持するコストを削減するために、できるだけ頻繁に同じカットサイトを使用するのが最善です。
増幅を開始する前に、LET に最適な PCR キットを選択します。<1,000塩基対を有するLETは、単純なTaqポリメラーゼで増幅することができ、≥1,000塩基対を有するLETは、より高忠実度のポリメラーゼ63を必要とすることができる。キットメーカーと希望プライマーの焼鈍温度に応じて増幅プロトコルを設定します。アニーリング温度は、増幅を成功させるために重要です。一般的な経験則は、プライマーのTmより5°C低いアニーリング温度を選択することです。プライマー58のシーケンスに基づいて最適化されたアニーリング温度を提供する無料のオンライン ツールがあります。正しいアニーリング温度を使用することは、高品質のDNAテンプレートを作り出すために重要です。
無細胞遺伝子発現は、近年、合成生物学のプロトタイピングのスピード、シンプルさ、有用性により、ルネッサンスを見てきました。この研究は、新しい機能性タンパク質の大規模なライブラリをテストする際の速度と容易さを高める方法を概説しました。従来のクローニング方法は数日から数週間かかる場合がありますが、このプロトコルは24時間未満で行 うことができます。 クローニングプロトコルが簡略化され、ゲル分離などのオプションの手順が省略されています。 表 1 は、一般的な制限のダイジェスト プロトコルを参照する場合もあります。他にも多くのクローン作成方法がありますが、これらは同様の時間を必要とします。この酵素増幅プロトコルの上限範囲は、特に8時間の就業日を考慮する場合、クローニング法の下限よりも少ない時間を必要とします。これは主に、夜間のインキュベーションの除去とシーケンシング確認の欠如によるものです。RCA プロトコルは PCR と RCA に基づいており、従来のクローニング、変換、および細胞培養技術よりも専門的なスキルが少なくて済みます。この方法により、細胞の変換や成長に必要な、クローン作成や資本へのアクセスに関する経験がないラボで、セルフリー式にアクセスできるようになります。この方法は、細胞傷害性タンパク質に焦点を当てたプロジェクトにも適しており、従来のクローニングを有する遺伝子のクローニングと増殖は、生細胞の毒性のために困難である。このRCAプロトコルは、CFEに必要なレベルまで、非常に少量の円形DNA(DNAのピコグラム)を増幅することも可能です。 図3Bでは、ラジコン前に20 ng/μLと20 pg/uLに希釈されたSBT(n)テンプレートを回覧しました。観察された分解率は異なっていたが、両方の反応は10分以内に同量の基質分解をもたらした。提案された方法は、クローン作成を置き換えることを意図していません。プラスミドの増殖は、一致しない非毒性遺伝子のDNAの量を生成することができます。むしろ、これは、アーカイブおよびさらなる伝播のためにクローン化されるべきシーケンスを特定するのに役立つ大規模なライブラリスクリーニング段階(酵素的ステップは、標準的な流体ハンドラを使用して自動化に適している)で便利なプロトタイピングツールです。
無細胞反応を設計する場合、柔軟性は非常に高いですが、成功を確実にするために考慮しなければならないいくつかの要因があります。小さな反応は、ガス交換に最適な体積比に対する表面積が大きくなりますが、正確な蛍光測定のために反応が井戸の底部を完全に覆う必要があります。反応の温度は、目的の遺伝子59によっても変化する。エネルギーバッファ34、46の選択に関しては、ユーザーのための複数の選択肢があります。一部のレシピは他のレシピよりも高価ですが、決定はユーザーに委ねされます。大腸菌抽出物36の潜在的なソースもたくさんあります。ユーザーは、抽出の生産プロトコルに慣れ、目的のために最適であるを決定する必要があります28,48,50,64,65,66,67.
RCAを使用して無細胞発現のテンプレートを増幅する場合、細菌株の選択は非常に重要です。式プロファイルは、LED よりも優れている傾向があります。人気のBL21 DE3スター株は、RCAがLETよりも〜1.4倍優れたパフォーマンスを発揮し、pJL1プラスミドが最高のRCA濃度よりも〜1.2倍優れている(図2A)で、これをうまく処理します。一方、いくつかの株は、テンプレートの劣化を示し、ネイティブヌクレアーゼ23、28の存在のために不十分なパフォーマンスを示す。この場合、SHuffle 株は RCA テンプレートの増加によってメリットを得るようです。これまでの文献では、SHuffle抽出物はPCR産物では十分に機能しないことが示されているが、この研究で使用された非精製RCA産物の濃度がpJL1プラスミドを上回った(図2B)。機能的なSBT(n)(図3)の発現は、細胞毒性を持たせた細胞毒性が、毒性のために生細胞での試作が困難である細胞傷害性タンパク質の一例である(この発現テンプレートをプラスミドで複製し、大腸菌で伝播することができない)。sfGFP とは異なり、SBT(n) 活性は、わずか 4 時間の式の後には観察できません (図 3A)。この信号は、16時間の発現および成熟後に検出可能である(図3B)。これらの例で使用された未精製DNAは、100 μLから取得され、10 μL RCA反応が希釈され、組み合わされた4つの反応でした。この1つのストックは、1反応あたり0.30 μLのみを使用する場合、333個の無細胞反応をサポートできます。
RCA テンプレートと CFE の互換性は、さまざまな抽出物でさらに検討する必要があります。リニア製品を伴う潜在的な合併症は、大腸菌カイ配列またはGamSタンパク質52,53を含む短いオリゴを添加することによって緩和され得る。文献は、カイオリゴの添加は、GamSタンパク質53よりも線形テンプレートに対してより大きな保護を提供できることを示唆している。線形テンプレートから低収率を提供することが知られている抽出物を使用する場合、ユーザーは各保護剤のコストを分析し、目的に最も適したものを使用する必要があります。もう一つの制限は、結果として得られる連結体の性質に起因するRCAテンプレート濃度をモルリティに変換できないことである。つまり、質量に基づいて同じ数の最小テンプレートを追加できますが、連結の長さが変化し、式レベルに影響を与える可能性があります。著者らは、各ウェルからの平均発現レベルが同じ(低分散)、小さなボリューム(例えば、微小小滴)で表現する場合、これはスクリーニング/プロトタイピングの問題ではないことを発見していない。
無細胞遺伝子発現は、タンパク質プロトタイピングおよび設計構築テストワークフローにおいて重要なツールとして使用される可能性を秘めています。しかし、ほとんどの無細胞発現ワークフローは、依然として遺伝的テンプレートとして従来のプラスミドに依存しています。これにより、プロセスが遅くなり、スクリーニング/プロトタイピングの目的でセルフリー表現が最大限に活用されるのを防ぐ。テンプレートを増幅し、その後RCAを実行すると、多くの反応に十分な遺伝子テンプレートを迅速に生成し、下流の特性評価と機能テストに十分なタンパク質を生成することができます。
開示事項
ナイジェル・ロイエルは、抗体の設計に無細胞システムを使用するBigHat Biosciences Inc.の科学諮問委員会を務めています。
謝辞
著者らは、このプロジェクトの部分的な支援に関するNIH 1R35GM138265-01およびNSF 2029532を認めている。
資料
| Name | Company | Catalog Number | Comments |
| Alaline | Formedium | DOC0102 | |
| Ammonium glutamate | MP Biomedicals | MP21805951 | |
| Arginine | Formedium | DOC0106 | |
| Asparagine | Formedium | DOC0114 | |
| Aspartic Acid | Formedium | DOC0118 | |
| ATP | Sigma | A2383 | |
| Axygen Sealing Film | Corning | PCR-SP | |
| CMP | Sigma | C1006 | |
| Coenzyme A | Sigma | C3144 | |
| CutSmart Buffer | NEB | B7204S | Provided with HindIII |
| Cysteine | Formedium | DOC0122 | |
| DNA Clean and Concentrator Kit | Zymo Research | D4004 | Used for purifying DNA |
| dNTPs | NEB | N0447 | |
| E. coli tRNA | Sigma (Roche) | 10109541001 | |
| Folinic Acid | Sigma | 47612 | |
| Gene Fragment | IDT | ||
| Glutamic Acid | Formedium | DOC0134 | |
| Glutamine | Formedium | DOC0130 | |
| Glycine | Formedium | DOC0138 | |
| GMP | Sigma | G8377 | |
| HEPES | Sigma | H3375 | |
| HindIII-HF | NEB | R3104L | |
| Histidine | Formedium | DOC0142 | |
| Isoleucine | Formedium | DOC0150 | |
| Leucine | Formedium | DOC0154 | |
| Lysine | Formedium | DOC0158 | |
| Magnesium glutamate | Sigma | 49605 | |
| Methionine | Formedium | DOC0166 | |
| Microtiter Plate (384 well) | Greiner | 781906 | |
| Microtiter Plate (96 well) | Greiner | 655809 | |
| Multimode Plate Reader | BioTek | Synergy Neo2 | |
| NAD | Sigma | N8535 | |
| NanoPhotometer | Implen | NP80 | |
| OneTaq DNA Polymerase | NEB | M0480 | |
| PCR Tube | VWR | 20170-012 | |
| Phenylalanine | Formedium | DOC0170 | |
| Phosphoenolpyruvate | Sigma (Roche) | 10108294 | |
| Potassium glutamate | Sigma | G1501 | |
| Potassium oxalate | Fisher Scientific | P273 | |
| Proline | Formedium | DOC0174 | |
| Putrescine | Sigma | P5780 | |
| Serine | Formedium | DOC0178 | |
| Spermidine | Sigma | S0266 | |
| T4 DNA Ligase | NEB | M0202S | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | Provided with T4 DNA Ligase |
| TempliPhi Amplification Kit | Cytiva | 25640010 | Used for RCA |
| Thermal Cycler | Biorad | C1000 Touch | |
| Thermoblock | Eppendorf | ThermoMixer FP | |
| Threonine | Formedium | DOC0182 | |
| Tryptophan | Formedium | DOC0186 | |
| Tyrosine | Formedium | DOC0190 | |
| UMP | Sigma | U6375 | |
| Valine | Formedium | DOC0194 |
参考文献
- Sun, Q., et al. A simple and low-cost paper-based colorimetric method for detecting and distinguishing the GII.4 and GII.17 genotypes of norovirus. Talanta. 225, 121978 (2021).
- Pardee, K., et al. Low-Cost Detection of Zika Virus Using Programmable Biomolecular Components. Cell. 165 (5), 1255-1266 (2016).
- Pardee, K., et al. Paper-based synthetic gene networks. Cell. 159 (4), 940-954 (2014).
- Ma, D., Shen, L., Wu, K., Diehnelt, C. W., Green, A. A. Low-cost detection of norovirus using paper-based cell-free systems and synbody-based viral enrichment. Synthetic Biology. 3 (1), (2018).
- Park, S., Lee, J. W. Detection of coronaviruses using rna toehold switch sensors. International Journal of Molecular Sciences. 22 (4), 1772 (2021).
- Cao, M., Sun, Q., Zhang, X., Ma, Y., Wang, J. Detection and differentiation of respiratory syncytial virus subgroups A and B with colorimetric toehold switch sensors in a paper-based cell-free system. Biosensors and Bioelectronics. 182, 113173 (2021).
- Mcnerney, M. P., et al. Point-of-care biomarker quantification enabled by sample-specific calibration. Science Advances. 5 (9), (2019).
- Silverman, A. D., Akova, U., Alam, K. K., Jewett, M. C., Lucks, J. B. Design and optimization of a cell-free atrazine biosensor. ACS Synthetic Biology. 9 (3), 671-677 (2020).
- Salehi, A. S. M., et al. Cell-free protein synthesis approach to biosensing hTRβ-specific endocrine disruptors. Analytical Chemistry. 89 (6), 3395-3401 (2017).
- Garamella, J., Majumder, S., Liu, A. P., Noireaux, V. An adaptive synthetic cell based on mechanosensing, biosensing, and inducible gene circuits. ACS Synthetic Biology. 8 (8), 1913-1920 (2019).
- Glasscock, C. J., et al. Dynamic control of pathway expression with riboregulated switchable feedback promoters. ACS Synthetic Biology. 16, (2019).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- Pardee, K., et al. Portable, on-demand biomolecular manufacturing. Cell. 167 (1), 248-259 (2016).
- Nelson, J. A. D., et al. Hydrofoam and oxygen headspace bioreactors improve cell-free therapeutic protein production yields through enhanced oxygen transport. Biotechnology Progress. 37 (2), 3079 (2020).
- Cai, Q., et al. A simplified and robust protocol for immunoglobulin expression in Escherichia coli cell-free protein synthesis systems. Biotechnology Progress. 31 (3), 823-831 (2015).
- Ogonah, O. W., Polizzi, K. M., Bracewell, D. G. Cell free protein synthesis: a viable option for stratified medicines manufacturing? A brief history of cell free synthesis systems. Current Opinion in Chemical Engineering. 18, 77-83 (2017).
- Zawada, J. F., et al. Microscale to manufacturing scale-up of cell-free cytokine production-a new approach for shortening protein production development timelines. Biotechnology and Bioengineering. 108 (7), 1570-1578 (2011).
- Stark, J. C., et al. On-demand biomanufacturing of protective conjugate vaccines. Science Advances. 7 (6), (2021).
- Huang, A., et al. BioBitsTM Explorer: A modular synthetic biology education kit. Science Advances. 4 (8), 1-11 (2018).
- Stark, J. C., et al. BioBits health: classroom activities exploring engineering, biology, and human health with fluorescent readouts. ACS Synthetic Biology. 8 (5), 1001-1009 (2019).
- Stark, J. C., et al. BioBitsTM Bright: A fluorescent synthetic biology education kit. Science Advances. 4 (8), 33 (2018).
- Shinoda, T., et al. Cell-free methods to produce structurally intact mammalian membrane proteins. Scientific Reports. 6, (2016).
- Dopp, J. L., Rothstein, S. M., Mansell, T. J., Reuel, N. F. Rapid prototyping of proteins: Mail order gene fragments to assayable proteins within 24 hours. Biotechnology and Bioengineering. 116 (3), 667-676 (2019).
- Sachse, R., Dondapati, S. K., Fenz, S. F., Schmidt, T., Kubick, S. Membrane protein synthesis in cell-free systems: From bio-mimetic systems to bio-membranes. FEBS Letters. 588 (17), 2774-2781 (2014).
- Salehi, A. S. M., et al. Cell-free protein synthesis of a cytotoxic cancer therapeutic: Onconase production and a just-add-water cell-free system. Biotechnology Journal. 11 (2), 274-281 (2016).
- Georgi, V., et al. On-chip automation of cell-free protein synthesis: New opportunities due to a novel reaction mode. Lab on a Chip. 16 (2), 269-281 (2016).
- Thoring, L., et al. Cell-free systems based on CHO cell lysates: Optimization strategies, synthesis of "difficult-to-express" proteins and future perspectives. PLoS One. 11 (9), (2016).
- Dopp, J. L., Reuel, N. F. Simple, functional, inexpensive cell extract for in vitro prototyping of proteins with disulfide bonds. Biochemical Engineering Journal. 164, 107790 (2020).
- Isaksson, L., Enberg, J., Neutze, R., Göran Karlsson, B., Pedersen, A. Expression screening of membrane proteins with cell-free protein synthesis. Protein Expression and Purification. 82 (1), 218-225 (2012).
- Techner, J. M., et al. High-throughput synthesis and analysis of intact glycoproteins using SAMDI-MS. Analytical Chemistry. 92 (2), 1963-1971 (2020).
- Kim, H. C., et al. Implementing bacterial acid resistance into cell-free protein synthesis for buffer-free expression and screening of enzymes. Biotechnology and Bioengineering. 112 (12), 2630-2635 (2015).
- Rolf, J., Siedentop, R., Lütz, S., Rosenthal, K. Screening and identification of novel cGAS homologues using a combination of in vitro and in vivo protein synthesis. International Journal of Molecular Sciences. 21 (1), (2020).
- Haslinger, K., Hackl, T., Prather, K. L. J. Rapid in vitro prototyping of O-methyltransferases for pathway applications in Escherichia coli. bioRxiv. , (2020).
- Dopp, J. L., Tamiev, D. D., Reuel, N. F. Cell-free supplement mixtures: Elucidating the history and biochemical utility of additives used to support in vitro protein synthesis in E. coli extract. Biotechnology Advances. 37 (1), 246-258 (2018).
- Gregorio, N. E., Levine, M. Z., Oza, J. P. A user's guide to cell-free protein synthesis. Methods and Protocols. 2 (1), 24 (2019).
- Cole, S. D., Miklos, A. E., Chiao, A. C., Sun, Z. Z., Lux, M. W. Methodologies for preparation of prokaryotic extracts for cell-free expression systems. Synthetic and Systems Biotechnology. 5 (4), 252-267 (2020).
- Chiba, C. H., Knirsch, M. C., Azzoni, A. R., Moreira, A. R., Stephano, M. A. Cell-free protein synthesis: advances on production process for biopharmaceuticals and immunobiological products. BioTechniques. 70, (2021).
- Laohakunakorn, N. Cell-free systems: A proving ground for rational biodesign. Frontiers in Bioengineering and Biotechnology. 8, 788 (2020).
- Dondapati, S. K., Stech, M., Zemella, A., Kubick, S. Cell-free protein synthesis: A promising option for future drug development. BioDrugs. , 1-22 (2020).
- Noireaux, V., Liu, A. P. The new age of cell-free biology. Annual Review of Biomedical Engineering. 22, 51-77 (2020).
- Khambhati, K., et al. Exploring the Potential of Cell-Free Protein Synthesis for Extending the Abilities of Biological Systems. Frontiers in Bioengineering and Biotechnology. 7, (2019).
- Carlson, E. D., Gan, R., Hodgman, C. E., Jewett, M. C. Cell-free protein synthesis: Applications come of age. Biotechnology Advances. 30 (5), 1185-1194 (2012).
- Rosenblum, G., Cooperman, B. S. Engine out of the chassis: Cell-free protein synthesis and its uses. FEBS Letters. 588 (2), 261-268 (2014).
- Swartz, J. R. Transforming biochemical engineering with cell-free biology. AIChE Journal. 58 (1), 5-13 (2012).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Caschera, F., Noireaux, V. A cost-effective polyphosphate-based metabolism fuels an all E. coli cell-free expression system. Metabolic Engineering. 27, 29-37 (2015).
- Levine, M. Z., et al. Activation of energy metabolism through growth media reformulation enables a 24-hour workflow for cell-free expression. ACS Synthetic Biology. 9 (10), 2765-2774 (2020).
- Hunt, J. P., et al. Streamlining the preparation of "endotoxin-free" ClearColi cell extract with autoinduction media for cell-free protein synthesis of the therapeutic protein crisantaspase. Synthetic and Systems Biotechnology. 4 (4), 220-224 (2019).
- Dopp, J. L., Jo, Y. R., Reuel, N. F. Methods to reduce variability in E. Coli-based cell-free protein expression experiments. Synthetic and Systems Biotechnology. 4 (4), 204-211 (2019).
- Sun, Z. Z., et al. Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology. Journal of Visualized Experiments: JoVE. , e50762 (2013).
- Schinn, S. M., Broadbent, A., Bradley, W. T., Bundy, B. C. Protein synthesis directly from PCR: Progress and applications of cell-free protein synthesis with linear DNA. New Biotechnology. 33 (4), 480-487 (2016).
- Sitaraman, K., et al. A novel cell-free protein synthesis system. Journal of Biotechnology. 110 (3), 257-263 (2004).
- Marshall, R., Maxwell, C. S., Collins, S. P., Beisel, C. L., Noireaux, V. Short DNA containing χ sites enhances DNA stability and gene expression in E. coli cell-free transcription-translation systems. Biotechnology and Bioengineering. 114, 2137-2141 (2017).
- Sun, Z. Z., Yeung, E., Hayes, C. A., Noireaux, V., Murray, R. M. Linear DNA for rapid prototyping of synthetic biological circuits in an escherichia coli based TX-TL cell-free system. ACS Synthetic Biology. 3 (6), 387-397 (2014).
- . Addgene: pJL1 Available from: https://www.addgene.org/69496/ (2021)
- . IDT Codon Optimization Tool Available from: https://www.idtdna.com/pages/tools/codon-optimization-tool (2021)
- Hadi, T., et al. Rolling circle amplification of synthetic DNA accelerates biocatalytic determination of enzyme activity relative to conventional methods. Scientific Reports. 10 (1), 10279 (2020).
- . New England Biolabs Tm Calculator Available from: https://tmcalculator.neb.com/#!/main (2021)
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. Coli RNA polymerase and sigma factor 70. Journal of Biological Engineering. 4, (2010).
- Colant, N., et al. A rational approach to improving titer in Escherichia coli-based cell-free protein synthesis reactions. Biotechnology Progress. 37 (1), 3062 (2021).
- Burgess-Brown, N. A., et al. Codon optimization can improve expression of human genes in Escherichia coli: A multi-gene study. Protein Expression and Purification. 59, 94-102 (2008).
- Maertens, B., et al. Gene optimization mechanisms: A multi-gene study reveals a high success rate of full-length human proteins expressed in Escherichia coli. Protein Science. 19 (7), 1312-1326 (2010).
- Eckert, K. A., Kunkel, T. A. DNA polymerase fidelity and the polymerase chain reaction. Genome Research. 1 (1), 17-24 (1991).
- Dopp, J. L., Reuel, N. F. Process optimization for scalable E. coli extract preparation for cell-free protein synthesis. Biochemical Engineering Journal. 138, 21-28 (2018).
- Liu, D. V., Zawada, J. F., Swartz, J. R. Streamlining Escherichia Coli S30 extract preparation for economical cell-free protein synthesis. Biotechnology Progress. 21 (2), 460-465 (2005).
- Levine, M. Z., Gregorio, N. E., Jewett, M. C., Watts, K. R., Oza, J. P. Escherichia coli-Based Cell-Free Protein Synthesis: Protocols for a robust, flexible, and accessible platform technology. Journal of Visualized Experiments: JoVE. (144), e58882 (2019).
- Kwon, Y. C., Jewett, M. C. High-throughput preparation methods of crude extract for robust cell-free protein synthesis. Scientific Reports. 5, (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved