Method Article
שיטות מהירות ואנזימטיות להגברה של תבניות ליניאריות מינימליות לאבן טיפוס של חלבונים באמצעות מערכות נטולות תאים
In This Article
Summary
המחקר מתאר פרוטוקול ליצירת כמויות גדולות (מיקרוגרם-מ"ג) של DNA עבור קמפיינים הקרנת חלבונים מרסיסי גנים סינתטיים ללא שיבוט או שימוש בתאים חיים. התבנית המינימלית מתעכלת באופן אנזימטי ומעגלית ולאחר מכן מוגברת באמצעות הגברה איזותרמית מתגלגלת של מעגל. תגובות חופש התא יכול להתבצע עם המוצר שלא שאושר.
Abstract
פרוטוקול זה מתאר את העיצוב של תבנית DNA מינימלית ואת השלבים להגברה אנזימטית, המאפשרת טיפוס מהיר של חלבונים הניתנים לבדיקה בפחות מ-24 שעות באמצעות חופש הביטוי של התא. לאחר קבלת DNA מספק, שבר הגן הוא PCR מוגבר, לחתוך, מעגלי, ו cryo-בנקאות. כמות קטנה של ה- DNA הבנקאי מדוללת ומוגברת באופן משמעותי (עד 106x) באמצעות הגברה איזותרמית מתגלגלת של מעגל (RCA). RCA יכול להניב כמויות מיקרוגרם של תבנית הביטוי המינימלית מרמות פיקוגרמה של חומר התחלתי (רמות מ"ג אם נעשה שימוש בכל הקטע הסינתטי ההתחלתי). בעבודה זו, כמות התחלתית של 20 pg הביא 4 מיקרוגרם של המוצר הסופי. ניתן להוסיף את מוצר RCA המתקבל (ויתור על התבנית המינימלית) ישירות לתגובה נטולת תאים ללא שלבי טיהור. בשל שיטה זו להיות מבוסס PCR לחלוטין, זה עשוי לאפשר מאמצי סינון תפוקה גבוהה בעתיד בשילוב עם מערכות טיפול נוזלי אוטומטי.
Introduction
ביטוי גנים ללא תאים (CFE) הופיע ככלי רב עוצמה עם יישומים רבים. יישומים כאלה כוללים גילוי מחלות1,2,3,4,5,6, מיקרונוטריאנט וזיהוי מולקולות קטנות7,8,9,10,11,12, biomanufacturing13,14,15,16,17 ,18, חינוך19,20,21, ייצור חלבונים קשים17,22,23,24,25,26,27, והקרנהמשתנה 23,28,29,30,31,32 ,33. זאת בשל האופי הפתוח של מערכות ללא תאים והגמישות שהן מעניקות. מאמרי סקירה גדולים רבים מציעים חינוך היסטורי ונקודות מבט עתידיות על הטכנולוגיה34,35,36,37,38,39,40,41,42,43,44.
תגובה טיפוסית ללא תאים מורכבת משלושה מרכיבים עיקריים: תמצית תאים, תערובת אנרגיה ותבנית גנטית. תמצית תא פעיל מכיל את כל המכונות הדרושות עבור שעתוק ותרגום (TXTL) וניתן לעבד במגווןדרכים 36. מתווכים גליקוליטיים, אלקטרוליטים, חומצות אמינו וקופות פחמן מתמהיל האנרגיה תומכים בתהליך TXTL. זהו מקור עיקרי של שונות בניסויים ללא תאים45 והוא יכול להיות מוכן במובנים רבים34,46. הכנת התבנית הגנטית ראתה פחות שיפורים מאז שיטות שיבוט מסורתיות לגרום plasmids עם מאפייני ביטוי מעולה. החיסרון בשיטות מסורתיות אלה הוא זמן התפנית וכמות המומחיות הביולוגית הדרושה כדי לבנות ולהפיץ אותם. מאמצי אופטימיזציה אחרונים הביאו זרימות עבודה פשוטות של 24 שעות להכנת תמצית תאים47,48 שניתן לבצע במקביל להכנת תערובתאנרגיה 49,50. עם זאת, שיבוט מסורתי מוסיף ימים מרובים לציר הזמן של עיצוב-טיפוס של CFE(טבלה 1)23. ניתן להשתמש במוצרי PCR מוגברים במהירות מרסיס הגן המסחרי ישירות51, אך זה מגביל את מספר ניסויי האב-טיפוס שכן מיוצר רק מיקרוגרם אחד של DNA, אשר מתאים לכחמש תגובות (כרכים μL מסורתיים). עם שלבים נוספים אלה של סיבובי הגברה איזותרמית, גדול יותר מכמויות מיליגרם של ה- DNA אפשרי (~ 5,000 תגובות עבור 1 מ"ג). זה מגדיל באופן דרמטי את מספר הבדיקות שניתן לבצע בהקרנה בתפוקה גבוהה של חלבונים או רשתות אנזימים קומבינטוריות (הנדסה מטבולית ללא תאים); זה גם מאפשר שימור יעיל של ספריית התבניות הליניארית כדנ"א בריכוז גבוה. יתר על כן, כמות מוגברת של תבנית יהיה צורך אב טיפוס כמויות גדולות יותר של חלבון הדרוש עבור יישומי מדעי החומר (סיבים מבוססי חלבון הידרוג'ל). ניתן להתגבר על מגבלות מסוימות של תבניות ליניאריות באמצעות תמצית מ- BL21 DE3 Star או באמצעות שיטות שהתגלו לאחרונה כדי להגן על תבניות ליניאריות מפני השפלה52,53,54. עם זאת, אין זה מתייחס למלאי מוגבל של DNA המיוצר על ידי ספק להגברת PCR או לנושא המומחיות הביולוגית והציוד הדרוש לשיבוט.
עבודה זו מציגה פרוטוקול שתוכנן במפורש כדי להגדיל את כמות תבנית הביטוי שניתן להשיג מכמויות קטנות של שברי גנים המיוצרים על ידי ספק (בדרך כלל 500-1000 ננוגרם של אבקת ליופילים). השיטה המתוארת אינה דורשת את הכישורים הדרושים לביצוע שיבוט מסורתי ב plasmids או שינוי והתפשטות בתאים חיים. עם קבלת שבר גנים בדואר, משתמש יכול לייצר מספיק תבניות עבור תגובות רבות ללא תאים על ידי שימוש בהגברת מעגל מתגלגל איזותרמי (RCA) (איור 1)23. בעוד שכמות הדנ"א המתקבלת מהספק עשויה להספיק למאמצי סינון מוגבלים, היא מתרוקנת במהירות, ורכישת שברי גנים מחדש גוזלת זמן ויקר. השיטה גם מתאימה במיוחד לגנים רעילים וקשה לשכפול ב- E. coli.
Protocol
1. עיצוב שבר הגן
הערה: שבר הגן צריך להיות כל האלמנטים הגנטיים הדרושים עבור שעתוק / תרגום, כולל מקדם, אתר כריכה ריבוזום (RBS), להתחיל קודון, גן העניין, ומחסל. בעוד המחסל אינו הכרחי עבור תבנית ביטוי ליניארי (LET), זה יהיה חשוב אם המשתמש מחליט להוסיף את הרצף לתוך plasmid. רצפים אלה הוסרו מן pJL1-sfGFP plasmid55 (מתנה מהמעבדה של מייקל ג'ואט), המשתמשת ביחצ"ן T7. בנוסף לאלמנטים גנטיים הכרחיים אלה, הוסיף אתר אנזים מוגבלת שישה זוגות בסיס לפני המקדם (5 'אתר לחתוך) ועוד שישה זוגות בסיס לאחר המחסל (3 'אתר לחתוך), במקרה זה באמצעות HindIII (אנזימי הגבלה אחרים ניתן להשתמש, אבל זה מועיל לתקנן את הרצפים עם אנזים אחד הגבלת נאמנות גבוהה כדי להפחית את המספר הדרוש כדי לשמור בספרייה). אתרי פריימר מתווספים עשרה זוגות בסיס במעלה הזרם של האתר 5 'לחתוך עשרה זוגות בסיס במורד הזרם של האתר 3 'לחתוך, במקרה זה באמצעות רצפי פריימר M13 סטנדרטי (פריימרים הם פריטי מלאי זולים). אתר האנזים ההגבלה והפריימרים המשמשים נמצאים על פי שיקול דעתו של המשתמש. עם זאת, על המשתמש לוודא שהרצפים אינם קיימים בשום מקום אחר בתבנית (לא רוצה ליצור חתכים לא רצויים או אתרים של אתחול הגברה). הרצפים עבור התבניות המשמשות בעבודה זו מפורטים בחומר המשלים. שלבים אלה משמשים לשינוי מתבנית בסיס זו.
- לקבוע את הגן הרצוי לבוא לידי ביטוי ולקבל את רצף חומצת האמינו או את הרצף הגנטי אם זה בא לידי ביטוי E. coli.
- אם זה רצף חומצת אמינו, לבצע אופטימיזציה קודון עבור E. coli באמצעות אחד מכלים ספק סטנדרטיים רבים56. אם אתה משתמש בתבנית המסופקת בתוספת, ודא שלרצף הממוטב אין אתרי הגבלה HindIII (AAGCTT). במקרה שהוא עושה, להמשיך לייעל את הרצף עד שאין יותר אתר HindIII.
- העתק את הרצף והדבק אותו בתבנית שסופקה עבור רצף משלים #1 שבו מצוין גן העניין. אם אתם מביעים את sfGFP, השתמשו ברצף משלים מס' 1 כפי שהוא. אם מבטאים subtilisin, להשתמש רצף משלים #2 כפי שהוא.
- הזמינו את התבנית המינימלית ואת הפריימרים הדרושים משירות סינתזת הדנ"א המועדף.
2. הכנסה חוזרת של שבר הגן ואת הפריימרים
הערה: עם קבלת שבר הגן, בצע את פרוטוקולי היצרן לחידוש או השתמש במדריך פשוט זה כדי ליצור מלאי DNA.
- צנטריפוגה הצינור (300 x גרם עבור 5 s) כדי לאסוף את כדורי ה- DNA בתחתית.
- הוסף מים מזוקקים כפולים (ddH2O) כדי להפוך את הריכוז הסופי של 10 ננוגרם / μL של תבנית DNA.
- מערבולת הפתרון על הגדרה בינונית עבור 5-10 s.
- ממיסים את כל הכדור על ידי דגירה ב 50 °C (50 °F) במשך 20 דקות.
- מערבולת בקצרה שוב
- צנטריפוגה ב 300 x g עבור 5 s כדי לאסוף את הפתרון בתחתית הצינור.
- יש לאחסן ב- -20 °C (70 °F) או להשתמש ב- PCR.
- הכן מלאי פריימר 100 מיקרומטר על ידי שימוש חוזר בפריימרים במים ללא גרעין. כדי לקבוע את כמות המים להוסיף, להכפיל את מספר nanomoles של פריימר ליופילי על ידי 10. לדוגמה, אם הצינור מכיל 45 ננומטר של פריימר ליופילי, להוסיף 450 μL של ddH2O ומערבולת הפתרון.
- לאחסן את פתרונות מלאי פריימר ב -20 °C (20 °F) או להמשיך לבצע את ההגברה.
3. להגביר את שבר הגן באמצעות PCR
הערה: החלט איזו ערכת PCR מתאימה לגן העניין. גנים קטנים יותר (<1,000 kb) עשויים להיות נוחים יותר לפולימראז טאק זול יותר, בעוד גנים גדולים יותר (≥1,000 kb) עשויים להפיק תועלת מפולימראז אמינות גבוהה כדי להפחית שגיאות. חשוב לציין כי הגברה ראשונית זו של PCR אינה הכרחית אם המשתמש אינו מודאג משימור שבר הגן הראשוני (הוא מספק ניסיונות מרובים להסתמכות ומאפשר מחקרים השוואתיים של מוצר LET לעומת RCA). חשוב גם לציין כי זה PCR מוגבר LET ניתן להשתמש ישירות בתגובות; עם זאת, כפי שהוזכר בהקדמה, זה יאפשר רק מספר מוגבל של תגובות אם צעדי ההגברה הנוספים היו מתעלמים. עיכול וקשירה יכולים להתבצע על שבר הגן resuspended ישירות57 (אם אחד בטוח, הם לא יצטרכו יותר LET לבצע מלאי מעגלי נוסף). במקרה זה, דלג על סעיף 3 והמשיך לסעיף 4. לביצוע PCR, בצע את השלבים הבאים.
- השתמש במניות 100 μM החל בשלב 2.8 כדי ליצור פתרונות עבודה של 10 מיקרומטר. פרוטוקולי ערכת PCR רבים דורשים פתרונות 10 מיקרומטר של פריימרים.
- תכנת את רוכב האופניים התרמי כדי לנהל את התגובה בהתאם לפרוטוקולים של יצרן הערכה. ערכות שונות דורשות פרמטרים מעט מגוונים של רכיבה על אופניים. עבור הערכה המפורטת בטבלת החומרים, התנאים הם 94 °C (75 °F) עבור 30 s של denaturation הראשונית; 30 מחזורים של 94 °C (70 °F) עבור 30 מעלות צלזיוס של denaturing, 45 °C (55 °F) עבור 30 °C (30 ים) של חישול פריימר, ו 68 °C (60 °F) של הרחבה; עם הארכה סופית ב 68 °C (5 דקות; ולבסוף, אחיזה של 10 מעלות צלזיוס ללא הגבלת זמן.
- הקפד לבחור את זמן ההארכה הנכון (משתנה בהתאם לאורך הגן להיות מוגבר). יש זמן התארכות של 1 דקות עבור כל 1,000 bp.
- יש להקפיד על הזנת טמפרטורת הכישול הנכונה עבור הפריימרים. השתמש במחשבון Tm מקוון המשתמש בשני פריימרים כתשומות כדי לקבוע את טמפרטורת חישול הטובה ביותר58. טמפרטורת חישול של 45 °C (70 °F) מספיקה בעת שימוש בפריימרים M13.
- בעת קביעת מספר המחזורים, עיין בפרוטוקול היצרן, אך 30 מחזורים לרוב יביאו להגברה מספקת.
- אם מבצעים PCR, להפשיר ולמערבולת את dNTPs. השתמש במאגר ה- PCR המסופק בערכה.
- בצינור PCR יחיד, שלב את כל רכיבי הערכה לפי ההוראות בפרוטוקול היצרן. כדי להבטיח הגברה מוצלחת, הוסף 1 μL של מלאי DNA resuspended (שלב 2.6).
- בעדינות הומוגניזציה התערובת על ידי מערבולת על הגדרה בינונית עבור 5-10 s. לחלופין, pipette מחצית הנפח למעלה ולמטה 10-20 פעמים למערבולת.
- בצע את תגובת ה- PCR.
- אם פרוטוקול PCR לא כלל שלב קירור סופי, אפשר לתגובה להתקרר במשך 5 דקות ב 10 °C (70 °F) לפני הסרת כדי להניע עיבוי לתחתית הצינור.
- נקה את התגובה באמצעות ערכת ניקוי PCR בהתאם להוראות הספק.
- בצינור 1.5 מ"ל, הוסף חיץ כריכת DNA ודגימת PCR ביחס של 5:1, בהתאמה.
- מעבירים את התערובת לעמודת הספין וצנטריפוגה ב-16,000 x גרם למשך דקה אחת. זרוק את הזרימה דרך.
- הוסף 200 μL של מאגר לשטוף DNA לעמוד ודגור בטמפרטורת החדר במשך 1 דקות.
- צנטריפוגה למשך דקה אחת ב-16,000 x גרם ומחקה את הזרימה.
- חזור על שלבים 2.8.3 ו- 2.8.4 ללא שלב הדגירה של דקה אחת.
- צנטריפוגה למשך 1-2 דקות נוספות ב-16,000 x ז', כדי להסיר כל מאגר שנותר.
- לחמוק DNA ב 46 μL של ddH2O.
- לכמת את הדנ"א המטוהר באמצעות ספקטרופוטומטר.
- לאחסן את ה- DNA המטוהר ב - -20 °C (50 °F) או להמשיך לשלב הבא.
4. עיכול ומעגליות
הערה: הגברה נוספת ניתן להשיג על ידי סיבוב ה- DNA ואחריו RCA. עכל את הדנ"א כדי להכין את התבנית למעגליות. פעולה זו תסיר את רצפי הפריימר ותיצור קצוות דביקים הן בקצוות של 5' ו- 3' של התבנית. לחבר מחדש את הקצוות האלה באמצעות תגובת קשירה.
- בצינור PCR, לשלב 5 μL של המאגר הדרוש, 20 U של HindIII, ו 45 μL של ה- DNA המטוהר משלב 3.8.
- בעדינות הומוגני תערובת זו עם פיפטה.
- לדגור על התערובת ברכב אופניים תרמי במשך 15 דקות ב 37 °C (50 °F).
- החום לאפעיל את הינדי על ידי דגירה במשך 20 דקות ב 80 °C (50 °F).
- אפשר לתגובה להתקרר ל 10 °C (50 °F) לפני הסרת כדי לנהוג עיבוי לתחתית הצינור.
- הוסף 5 μL של חיץ ligase T4 ו 800 U של T4 ligase ל- DNA שעוכל לאחרונה.
- השתמש T7 ligase, אם תרצה.
- בעדינות הומוגני תערובת זו עם פיפטה.
- לדגור על התערובת במשך 1 שעות ב 25 °C (5 °F) כדי לבצע את תגובת המעגליזציה.
- נקה את התגובה באמצעות ערכת ניקוי PCR בהתאם להוראות הספק. השתמש באותו פרוטוקול המפורט בשלב 3.8.
- לכמת את הדנ"א באמצעות ספקטרופוטומטר. הערכים הצפויים הם ~ 20 ng / μL.
- יש לאחסן ב- -20 °C (70 °F) או להמשיך לשלב הבא.
5. הגברה איזותרמית של מעגל מתגלגל
הערה: הגברה מעגל מתגלגל (RCA) ניתן לבצע באמצעות ערכה מסחרית או עם רכיבים שנרכשו בנפרד. ביצוע פרוטוקול היצרן יבטיח הגברה מוצלחת. ערכות מכילות בדרך כלל חיץ מדגם, מאגר תגובה, גדיל עקירת פולימראז, כגון φ29 פולימראז. צינורות תגובה מרובים ניתן לשלב כדי לייצר כמות גדולה של DNA עבור ביטוי חופש התא (4 מיקרוגרם מ 20 pg של חומר התחלתי). הפרוטוקול הבא פועל ביעילות.
- בצינור יחיד, שלב 20 μL של מאגר המדגם, 20 μL של מאגר התגובה, 0.8 μL של האנזים, ו 1 μL של תבנית הביטוי המעגלי (CET) משלב 4.9.
הערה: זה יהיה מסת DNA הכוללת של ~ 20 ננוגרם, אבל RCA יכול לעבוד עם כמויות פיקוגרמה, ובכך לאפשר דילול של CET הגברה אנזימטית קיצונית אם יש צורך משמעותי בחומר בהקרנת תפוקה גבוהה. - הומוגניזציה התערובת עם פיפטה aliquot 10 μL של התערובת לארבעה צינורות נפרדים.
- דגירה ב 30 °C (50 °F) עבור 4-18 שעות.
- חום לנטרל את האנזים על ידי דגירה ב 65 °C (65 °F) במשך 10 דקות. להפחית את הטמפרטורה ל 12 °C (5 דקות) כדי לעודד עיבוי בתחתית הצינור.
הערה: הכי קל לשלב את כל שלבי הטמפרטורה בפרוטוקול אוטומטי על רוכב אופניים תרמי. - לדלל את הפתרון המתקבל על ידי הוספת 15 μL של ddH2O לכל צינור.
- שלב את כל הצינורות והוסף ישירות לתגובה ללא תאים.
- אם תרצה, השתמש בערכת ניקוי PCR כדי לטהר את המוצר ולהתחמק ממנו ב 36 μL של ddH2O כדי לכמת. ודא שריכוז התבנית הוא ~100 ננוגרם/μL.
6. תגובה ללא תאים
הערה: בצע ביטוי חופש תא על-ידי שילוב של מאגר אנרגיה, תמצית ותבנית RCA. תגובה אופיינית ללא תאים באמצעות מאגר האנרגיה PANOx-SP מורכבת מ- ATP של 1.2 מ"מ, 0.85 מ"מ כל אחד מה-GMP, UMP ו-CMP, 30 מ"מ זרחן, 130 מ"מ אשלגן גלוטמט, 10 מ"מ אמוניום גלוטמט, 12 מ"מ מגנזיום גלוטמט, 1.5 מ"מ זרע, 1 mM putrescine, 34 מיקרוגרם/מ"ל של חומצה פולינית, 171 מיקרוגרם/מ"ל של תערובת E. coli tRNA, 2 מ"מ כל אחת מ-20 חומצות אמינו ללא תווית, 0.33 מ"מ NAD, 0.27 מ"מ קואנזים A (CoA), 4 מ"מ אשלגן אוקסלט, 57 מ"מ חיץ HEPES-KOH (pH 7.5), 0.24% נפח תמצית E. coli, וכמויות משתנות של DNA23,49. נפח התגובה יכול להשתנות אבל 15 תגובות μL יכול לחסוך על שימוש ריאגנט והם קטנים מספיק לשימוש 384 מיקרו-לוח שחור49,50.
- אם מבטאים חלבון פלואורסצנטי כגון sfGFP, הכינו קורא לוחות לקריאה בהתרגשות/פליטה הרצויה, בטמפרטורה ובתסיסה.
- אם משתמשים בצלחת 384-well, aliquot 60 μL של H2O לתוך בארות הגובלות בדוגמה ריקה היטב כדי לשמור על לחות ולהפחית את אפקט הקצה.
- הוסף את הרכיבים הנדרשים השונים לתוך צינור עבור כל מדגם. הוסף מספיק כדי לבצע טריפליקאטים. שכפול בתוך הלוח יכול לסייע בזיהוי גורמים לשונות.
- מוסיפים את התמצית, את מאגר האנרגיה ואז את הדנ"א.
- לדלל את הנפח הרצוי הסופי עם ddH2O.
- ערבבו פתרון זה ביסודיות על ידי צנרת מחצית מנפח הפתרון למעלה ולמטה 10-20 פעמים.
- מעבירים את תערובת התגובה ב-15 μL aliquots לבארות הרצויות בצלחת המיקרו-תות.
- אוטמים את הצלחת בסרט איטום חסר צבע לשמירה על לחות ומניעת אידוי.
- מניחים את הצלחת האטומה בקורא הלוחות ומאפשרים לתגובה להסתיים.
- אם מבטאים חלבון שאין לו את היכולת להיות במעקב חי, השתמש במנגנון אחר מבוקר טמפרטורה כגון תרמובלוק כדי לדגור על הצלחת.
7. בדיקה תת-סטיליסין
הערה: אם מבטאים את הגן BPN' (SBT(n)) ברצף משלים מס ' 2, פעל לפי פרוטוקול זה כדי לאבחן את הפעילות.
- הכן פתרון מלאי 10 מיקרומטר של N-סוקציניל-עלא-אלה-פרו-Phe p-nitroanilide בדימתילפורמיד (DMF).
- הגדר קורא צלחת כדי למדוד ספיגה ב 410 ננומטר כל 20 s במשך 10 דקות תוך שמירה על טמפרטורה של 25 °C (70 °F).
- בתחתית שטוחה, צלחת 96-well חסר צבע, aliquot 94 μL של ddH2O ו 1 μL של N-succinyl-Ala-Ala-Pro-Phe p-nitroanilide מהשלב 7.1.
- הוסף 5 μL של התגובה המוגמרת ללא תאים מהשלב 6.7 וקרא באמצעות קורא לוחות המוגדר לפרוטוקול המתואר בשלב 7.2.
תוצאות
ביטוי של sfGFP מתבניות RCA היה דומה לזה של pJL1 plasmid בעת שימוש רק 0.30 μL של DNA RCA לא מתואם בתגובת 15 μL (איור 2A). למעשה, הכפלה ושלשה של כמות התבנית נראה להציע שום תועלת תמצית BL21 DE3 כוכב, מציע רמות רוויות כבר של התבנית ב 0.30 μL לכל תגובה. לעומת זאת, נראה שיש יתרון בהגדלת כמות תבנית RCA כאשר מוסיפים לתמצית תאים שמקורה בזן SHuffle (איור 2B)28. עבור חלבונים מסוימים, ניתן לראות תוצאות מהר מאוד, אשר דוחס את זרימת העבודה כולה (הגברה ובוחן) אל מתחת 24 שעות. עם זאת, חלבונים מסוימים דורשים טמפרטורה נמוכה יותר או שיש להם זמני קיפול איטיים יותר, אשר יגדיל את הזמן עד לתוצאות מתקבלות אבל ישפיע על זרימת העבודה המוצגת כאן. ניתן לצפות באפשרות זו בעת הבעת סטיבליסין משנה (SBT(n)) כאשר בדיקה לאחר 4 שעות של ביטוי לא הייתה ארוכה מספיק עבור התבגרות SBT(n) (איור 3A, דוגמה לתוצאה כושלת). מתן אפשרות לתגובה להמשיך ל- 16 שעות יכול להוביל לרמות הניתנות לזיהוי של SBT(n) (איור 3B). שיפור זה עשוי להיות תלוי טמפרטורה, כפי שנצפה בספרות שבה תנאי טמפרטורה אופטימליים נחקרו59,60.

איור 1: סכמטי מייצג של התבנית הגנטית המינימלית והתהליך שהיא עוברת לאחר שלב ההגברה הראשוני של PCR. התבניות מתעכלות עם HindIII, עגולות עם ligase T4, ומוגברות עוד יותר עם φ29 פולימראז כדי ליצור concatemers גדול. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
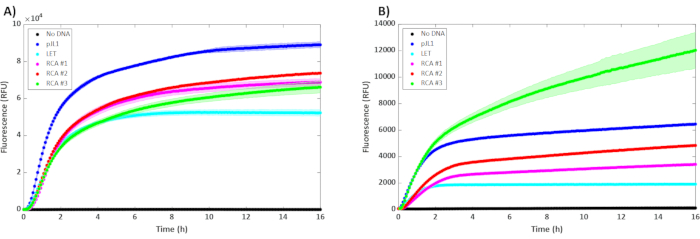
איור 2: תוצאות התגובות נטולות התאים עם 5 ננומטר של פלסמיד (pJL1), 5 ננומטר של תבנית ליניארית (LET) וריכוזים משתנים של RCA לא מנוצל. RCA #1, #2 ו- #3 הכילו 0.3 μL, 0.6 μL ו- 0.9 μL (בהתאמה) של מוצר RCA לא שידן בתגובת 15 μL דגירה ב- 30 °C (n = 3). קווי שגיאה מייצגים ± 1 SD מהממוצע. ציר ה-y הוא פלואורסצנטיות, וציר ה-x הוא הזמן שחלף במהלך התגובה. הקינטיקה של ביטוי sfGFP מיוצגת ב -A) BL21 DE3 Star ו - (B) T7 SHuffle. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 3: תגובות ללא תאים עם 5 ננומטר של SBT(n) LET וריכוזים משתנים של מוצר RCA לא מpurified. RCA ng ו pg תואמים את הריכוז של ה- DNA המשמש לביצוע הגברה מעגלית מתגלגלת. מוצר RCA לא שאושר שימש בתגובת 15 μL דגירה ב 30 °C (n = 3). קווי שגיאה מייצגים ± 1 SD מהממוצע. ציר ה-y הוא הספיגה ב-410 ננומטר, וציר ה-x הוא משך הזמן שחלף בבחיקה. התגובות בוצעו עבור (A) 4 שעות ו (B) 16 שעות. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
| שיטת שכפול | |
| PCR | 2 -4 שעות |
| עיכול פלסמיד | 35 דק' |
| קשירה | שעה 1 |
| שינוי | 2 שעות |
| דגירה לילית | 16 שעות |
| רצף | 24 - 48 שעות |
| הכנה למניות גליצריול | 16 שעות |
| צמיחה וטיהור | 16 שעות |
| זמן כולל | 46 - 72 שעות |
| שיטת RCA | |
| PCR | 2 - 4 שעות |
| עיכול | 35 דק' |
| קשירה | שעה 1 |
| RCA | 4 - 18 שעות |
| CFE | 4 - 16 שעות |
| זמן כולל | 12 - 40 שעות |
טבלה 1: השוואה של ציר הזמן בין פרוטוקול שיבוט מסורתי פשוט לפרוטוקול RCA המכוסה כאן.
קובץ משלים: הקובץ המשלים מפרט את הרצפים. רצף #1 הוא sfGFP (999 זוגות בסיס) ורצף #2 הוא subtilisin BPN' (1344 bp). נא לחץ כאן כדי להוריד קובץ זה.
Discussion
גן העניין יכול להיות כל חלבון רצוי, אבל עדיף להתחיל עם חלבון פלואורסצנטי ככתב נוח לקריאה בזמן אמת או נקודת קצה על קורא צלחת היטב עבור מאמצים חדשים של שיטה זו. עבור רצפי חלבון חדשים, להעתיק את רצף חומצת האמינו של החלבון הרצוי ולהדביק אותו לתוך כלי אופטימיזציה קודון הרצוי61,62. בדרך כלל יש אורגניזמים זמינים רבים זנים של E. coli בכלי אופטימיזציה קודון, אבל בחירת האפשרות הכללית Escherichia coli יהיה מתאים. לאחר אופטימיזציה, בדוק שוב את הגן כדי להבטיח את אתר החיתוך שנבחר בעבר ורצפי פריימר אינם קיימים. אם כן, ניתן למטב את הרצף עד שהרצפים לא יהיו קיימים עוד. במצבים מסוימים, ייתכן שיהיה צורך להחליף את האתרים ההינדיים לחתוך עם אתר הגבלה שונה אם אופטימיזציה חוזרת באופן עקבי תוצאות באתר פנימי HindIII לחתוך. עדיף להשתמש באותו אתר לחתוך לעתים קרובות ככל האפשר כדי לשמור על התהליך סטנדרטי ולהפחית את העלות של שמירה על אנזימי הגבלה מרובים בהישג יד.
לפני תחילת ההגברה, בחר את ערכת ה- PCR המתאימה ביותר ל- LET. ניתן להגביר את ה-LET עם <1,000 זוגות בסיסים עם פולימראז טאק פשוט, בעוד ש-LET עם ≥ 1,000 זוגות בסיסים עשוי לדרוש פולימראז בעל אמינות גבוהה יותר63. הגדר את הפרוטוקול להגברה על פי יצרן הערכה וטמפרטורת חישול של הפריימרים הרצויים. טמפרטורת חישול היא קריטית להגברה מוצלחת. כלל אצבע כללי הוא לבחור טמפרטורת חישול נמוכה ב -5 מעלות צלזיוס מהתמולית של הפריימרים. ישנם כלים מקוונים חינם שיספקו טמפרטורת חישול אופטימלית המבוססת על רצף הפריימרים58. שימוש בטמפרטורת חישול נכונה הוא קריטי לייצור תבנית DNA באיכות גבוהה.
ביטוי גנים ללא תאים ראה רנסנס בשנים האחרונות בשל המהירות, הפשטות והתועלת שלו עבור אבי טיפוס ביולוגי סינתטי. עבודה זו התוותה שיטה להגברת המהירות והקלות בעת בדיקת ספרייה גדולה של חלבונים חדשים ופונקציונליים. בעוד ששיטות שיבוט מסורתיות יכולות להימשך ימים או שבועות, פרוטוקול זה יכול להיעשות בפחות מ- 24 שעות. טבלה 1 מתארת טווחי זמן אופייניים הן עבור השיבוט המסורתי והן עבור פרוטוקול RCA. שים לב שפרוטוקול השיבוט היה פשוט יותר, וכמה שלבים אופציונליים נותרו בחוץ, כגון בידוד ג'ל. טבלה 1 מתייחסת גם לפרוטוקולי העיכול הנפוצים של ההגבלה; ישנן שיטות שיבוט רבות אחרות, אך אלה דורשות פרק זמן דומה. הטווח העליון של פרוטוקול הגברה אנזימטי זה דורש פחות זמן מאשר הטווח התחתון של שיטת השיבוט, במיוחד כאשר שוקלים יום עבודה של 8 שעות. זאת בעיקר בשל הסרת דגירות לילה וחוסר אישור רצף. פרוטוקול RCA מבוסס כולו על PCR ו- RCA, הדורשים מיומנויות פחות מיוחדות מאשר טכניקות שיבוט, טרנספורמציה ותרבות תאים מסורתיות. שיטה זו הופכת את חופש הביטוי לתאים לנגיש למעבדות שאין להן ניסיון קודם בשיבוט או גישה להון הדרוש לשינוי צורה ולגידול תאים. שיטה זו מתאימה גם לפרויקטים המתמקדים בחלבונים ציטוטוקסיים, שבהם שיבוט והפצה של הגנים עם שיבוט מסורתי קשה בשל רעילות בתא החי. פרוטוקול RCA זה מסוגל גם להגביר כמויות קטנות מאוד של DNA מעגלי (פיקוגרמות של DNA) לרמות הדרושות עבור CFE. באיור 3B, תבנית ה-SBT(n) המעגלית מדוללת ל- 20 ננוגה/μL ו- 20 pg/uL לפני RCA. בעוד ששיעורי ההשפלה שנצפו היו שונים, שתי התגובות גרמו לאותה כמות של השפלת מצע תוך 10 דקות. השיטה המוצעת אינה מיועדת להחליף שיבוט; התפשטות plasmid יכול לייצר כמויות של DNA עבור גנים שאינם רעילים שלא ניתן להתאים. במקום זאת, זהו כלי אב טיפוס נוח בשלב סינון מסיבי של הספרייה (שלבים אנזימטיים מקובלים לאוטומציה עם מטפל נוזלים סטנדרטי) שיסייעו לזהות אילו רצפים יש לשכפל עבור אחסון בארכיון והתפשטות נוספת.
בעת עיצוב תגובה ללא תאים, יש גמישות רבה, אך ישנם כמה גורמים שיש לקחת בחשבון כדי להבטיח הצלחה. לתגובות קטנות יותר יש יחס שטח-פנים גדול יותר לנפח, וזה נהדר להחלפת גז, אבל התגובה צריכה לכסות לחלוטין את תחתית הבאר למדידות פלואורסצנטיות מדויקות. טמפרטורת התגובה יכולה גם להשתנות בהתאם לגן של עניין59. ישנן אפשרויות מרובות עבור משתמשים כשמדובר בבחירה של מאגראנרגיה 34,46. מתכונים מסוימים יקרים יותר מאחרים, אך ההחלטה נותרת למשתמש. ישנם גם מקורות פוטנציאליים רבים עבור E. coli לחלץ36. משתמשים צריכים להכיר את פרוטוקולי ייצור לחלץ ולהחליט מה הכי טוב למטרותיהם28,48,50,64,65,66,67.
בעת שימוש RCA כדי להגביר תבניות עבור ביטוי חופש התא, הבחירה של זן חיידקי חשוב מאוד. פרופיל הביטוי נוטה להיות טוב יותר מזה של LETs. זן BL21 DE3 Star הפופולרי מטפל היטב זו, עם ביצוע RCA ~ 1.4x טוב יותר מאשר LET ואת pJL1 plasmid ביצועים ~ 1.2 פעמים טוב יותר מאשר ריכוז RCA הטוב ביותר(איור 2A). מצד שני, זנים מסוימים מציגים השפלה תבנית ולבצע גרוע בשל נוכחות של גרעינים מקומיים23,28. במקרה זה, נראה כי זן SHuffle נהנה מתבנית RCA מוגברת. ספרות קודמת הראתה כי תמצית SHuffle אינה מבצעת היטב עם מוצרי PCR, אבל הריכוז הגבוה ביותר של מוצר RCA לא מנוצל המשמש במחקר זה עולה על pJL1 plasmid (איור 2B). הביטוי של SBT פונקציונלי(n) (איור 3) הוא דוגמה לחלבון ציטוטוקסי המיוצר בנוחות על ידי ללא תאים אך קשה לאב טיפוס בתאים חיים בשל רעילות (לא מסוגל לשכפל תבנית ביטוי זו ב plasmid ולהפיץ ב- E. coli). שלא כמו sfGFP, לא ניתן לצפות בפעילות SBT(n) לאחר 4 שעות בלבד של ביטוי (איור 3A). האות ניתן לזיהוי לאחר 16 שעות של ביטוי והתבגרות (איור 3B). ה- DNA הלא שאושר המשמש בדוגמאות אלה הגיע מ 100 μL, אשר היה ארבע 10 תגובות RCA μL מדולל בשילוב. מניה אחת זו יכולה לתמוך ב-333 תגובות ללא תאים אם רק באמצעות 0.30 μL לכל תגובה.
יש לחקור עוד יותר את התאימות של תבניות RCA עם CFE עם תמציות שונות. סיבוכים פוטנציאליים עם מוצרים ליניאריים עשויים להיות הקלה על ידי הוספת אוליגוס קצר המכיל את רצף E. coli chi או חלבון GamS52,53. הספרות מציעה כי התוספת של צ'י אוליגוס יכול לספק הגנה גדולה יותר לתבניות ליניאריות מאשר חלבון GamS53. אם משתמשים בתמצית הידועה כמספקת תשואות נמוכות מתבניות ליניאריות, על המשתמש לנתח את העלות של כל סוכן מגן ולהשתמש בתמצית המתאימה ביותר למטרותיהם. מגבלה נוספת היא חוסר היכולת להמיר ריכוזי תבנית RCA לאונח בשל אופיו של המתנשא המתקבל. משמעות הדבר היא שניתן להוסיף את אותו מספר של תבניות מינימליות בהתבסס על מסה, אך יהיו להן אורכי תמצית משתנים, דבר שעשוי להשפיע על רמות הביטוי. המחברים לא מצאו כי מדובר בבעיה בהקרנה/אב טיפוס שכן רמות הביטוי הממוצעות מכל באר זהות (שונות נמוכה), אך יכולות להוות בעיה אם היא באה לידי ביטוי בנפחים קטנים יותר (למשל, מיקרו-טיפות).
ביטוי גנים ללא תאים יש פוטנציאל לשמש ככלי משמעותי בתהליך עבודה של אב טיפוס של חלבון ותכנון-בונים-בדיקה. עם זאת, רוב זרימות העבודה של חופש התאים עדיין מסתמכות על פלסמידים מסורתיים כתבנית הגנטית. זה מאט את התהליך ומונע מחופש הביטוי התאי להיות מנוצל במלואו למטרות סינון / טיפוס. תבניות להגברה ולאחר מכן ביצוע RCA יכול לייצר במהירות מספיק תבניות גנטיות עבור תגובות רבות, לייצר מספיק חלבון עבור אפיון במורד הזרם ובדיקות פונקציונליות.
Disclosures
נייג'ל ראול מכהן בוועדה המדעית המייעצת של BigHat Biosciences Inc., חברה המשתמשת במערכות ללא תאים לתכנון נוגדנים.
Acknowledgements
המחברים מכירים NIH 1R35GM138265-01 ו- NSF 2029532 לתמיכה חלקית בפרויקט זה.
Materials
| Name | Company | Catalog Number | Comments |
| Alaline | Formedium | DOC0102 | |
| Ammonium glutamate | MP Biomedicals | MP21805951 | |
| Arginine | Formedium | DOC0106 | |
| Asparagine | Formedium | DOC0114 | |
| Aspartic Acid | Formedium | DOC0118 | |
| ATP | Sigma | A2383 | |
| Axygen Sealing Film | Corning | PCR-SP | |
| CMP | Sigma | C1006 | |
| Coenzyme A | Sigma | C3144 | |
| CutSmart Buffer | NEB | B7204S | Provided with HindIII |
| Cysteine | Formedium | DOC0122 | |
| DNA Clean and Concentrator Kit | Zymo Research | D4004 | Used for purifying DNA |
| dNTPs | NEB | N0447 | |
| E. coli tRNA | Sigma (Roche) | 10109541001 | |
| Folinic Acid | Sigma | 47612 | |
| Gene Fragment | IDT | ||
| Glutamic Acid | Formedium | DOC0134 | |
| Glutamine | Formedium | DOC0130 | |
| Glycine | Formedium | DOC0138 | |
| GMP | Sigma | G8377 | |
| HEPES | Sigma | H3375 | |
| HindIII-HF | NEB | R3104L | |
| Histidine | Formedium | DOC0142 | |
| Isoleucine | Formedium | DOC0150 | |
| Leucine | Formedium | DOC0154 | |
| Lysine | Formedium | DOC0158 | |
| Magnesium glutamate | Sigma | 49605 | |
| Methionine | Formedium | DOC0166 | |
| Microtiter Plate (384 well) | Greiner | 781906 | |
| Microtiter Plate (96 well) | Greiner | 655809 | |
| Multimode Plate Reader | BioTek | Synergy Neo2 | |
| NAD | Sigma | N8535 | |
| NanoPhotometer | Implen | NP80 | |
| OneTaq DNA Polymerase | NEB | M0480 | |
| PCR Tube | VWR | 20170-012 | |
| Phenylalanine | Formedium | DOC0170 | |
| Phosphoenolpyruvate | Sigma (Roche) | 10108294 | |
| Potassium glutamate | Sigma | G1501 | |
| Potassium oxalate | Fisher Scientific | P273 | |
| Proline | Formedium | DOC0174 | |
| Putrescine | Sigma | P5780 | |
| Serine | Formedium | DOC0178 | |
| Spermidine | Sigma | S0266 | |
| T4 DNA Ligase | NEB | M0202S | |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | Provided with T4 DNA Ligase |
| TempliPhi Amplification Kit | Cytiva | 25640010 | Used for RCA |
| Thermal Cycler | Biorad | C1000 Touch | |
| Thermoblock | Eppendorf | ThermoMixer FP | |
| Threonine | Formedium | DOC0182 | |
| Tryptophan | Formedium | DOC0186 | |
| Tyrosine | Formedium | DOC0190 | |
| UMP | Sigma | U6375 | |
| Valine | Formedium | DOC0194 |
References
- Sun, Q., et al. A simple and low-cost paper-based colorimetric method for detecting and distinguishing the GII.4 and GII.17 genotypes of norovirus. Talanta. 225, 121978(2021).
- Pardee, K., et al. Low-Cost Detection of Zika Virus Using Programmable Biomolecular Components. Cell. 165 (5), 1255-1266 (2016).
- Pardee, K., et al. Paper-based synthetic gene networks. Cell. 159 (4), 940-954 (2014).
- Ma, D., Shen, L., Wu, K., Diehnelt, C. W., Green, A. A. Low-cost detection of norovirus using paper-based cell-free systems and synbody-based viral enrichment. Synthetic Biology. 3 (1), Oxford, England. (2018).
- Park, S., Lee, J. W. Detection of coronaviruses using rna toehold switch sensors. International Journal of Molecular Sciences. 22 (4), 1772(2021).
- Cao, M., Sun, Q., Zhang, X., Ma, Y., Wang, J. Detection and differentiation of respiratory syncytial virus subgroups A and B with colorimetric toehold switch sensors in a paper-based cell-free system. Biosensors and Bioelectronics. 182, 113173(2021).
- Mcnerney, M. P., et al. Point-of-care biomarker quantification enabled by sample-specific calibration. Science Advances. 5 (9), (2019).
- Silverman, A. D., Akova, U., Alam, K. K., Jewett, M. C., Lucks, J. B. Design and optimization of a cell-free atrazine biosensor. ACS Synthetic Biology. 9 (3), 671-677 (2020).
- Salehi, A. S. M., et al. Cell-free protein synthesis approach to biosensing hTRβ-specific endocrine disruptors. Analytical Chemistry. 89 (6), 3395-3401 (2017).
- Garamella, J., Majumder, S., Liu, A. P., Noireaux, V. An adaptive synthetic cell based on mechanosensing, biosensing, and inducible gene circuits. ACS Synthetic Biology. 8 (8), 1913-1920 (2019).
- Glasscock, C. J., et al. Dynamic control of pathway expression with riboregulated switchable feedback promoters. ACS Synthetic Biology. 16, (2019).
- Thavarajah, W., et al. Point-of-use detection of environmental fluoride via a cell-free riboswitch-based biosensor. ACS Synthetic Biology. 9 (1), 10-18 (2020).
- Pardee, K., et al. Portable, on-demand biomolecular manufacturing. Cell. 167 (1), 248-259 (2016).
- Nelson, J. A. D., et al. Hydrofoam and oxygen headspace bioreactors improve cell-free therapeutic protein production yields through enhanced oxygen transport. Biotechnology Progress. 37 (2), 3079(2020).
- Cai, Q., et al. A simplified and robust protocol for immunoglobulin expression in Escherichia coli cell-free protein synthesis systems. Biotechnology Progress. 31 (3), 823-831 (2015).
- Ogonah, O. W., Polizzi, K. M., Bracewell, D. G. Cell free protein synthesis: a viable option for stratified medicines manufacturing? A brief history of cell free synthesis systems. Current Opinion in Chemical Engineering. 18, 77-83 (2017).
- Zawada, J. F., et al. Microscale to manufacturing scale-up of cell-free cytokine production-a new approach for shortening protein production development timelines. Biotechnology and Bioengineering. 108 (7), 1570-1578 (2011).
- Stark, J. C., et al. On-demand biomanufacturing of protective conjugate vaccines. Science Advances. 7 (6), (2021).
- Huang, A., et al. BioBitsTM Explorer: A modular synthetic biology education kit. Science Advances. 4 (8), 1-11 (2018).
- Stark, J. C., et al. BioBits health: classroom activities exploring engineering, biology, and human health with fluorescent readouts. ACS Synthetic Biology. 8 (5), 1001-1009 (2019).
- Stark, J. C., et al. BioBitsTM Bright: A fluorescent synthetic biology education kit. Science Advances. 4 (8), 33(2018).
- Shinoda, T., et al. Cell-free methods to produce structurally intact mammalian membrane proteins. Scientific Reports. 6, (2016).
- Dopp, J. L., Rothstein, S. M., Mansell, T. J., Reuel, N. F. Rapid prototyping of proteins: Mail order gene fragments to assayable proteins within 24 hours. Biotechnology and Bioengineering. 116 (3), 667-676 (2019).
- Sachse, R., Dondapati, S. K., Fenz, S. F., Schmidt, T., Kubick, S. Membrane protein synthesis in cell-free systems: From bio-mimetic systems to bio-membranes. FEBS Letters. 588 (17), 2774-2781 (2014).
- Salehi, A. S. M., et al. Cell-free protein synthesis of a cytotoxic cancer therapeutic: Onconase production and a just-add-water cell-free system. Biotechnology Journal. 11 (2), 274-281 (2016).
- Georgi, V., et al. On-chip automation of cell-free protein synthesis: New opportunities due to a novel reaction mode. Lab on a Chip. 16 (2), 269-281 (2016).
- Thoring, L., et al. Cell-free systems based on CHO cell lysates: Optimization strategies, synthesis of "difficult-to-express" proteins and future perspectives. PLoS One. 11 (9), (2016).
- Dopp, J. L., Reuel, N. F. Simple, functional, inexpensive cell extract for in vitro prototyping of proteins with disulfide bonds. Biochemical Engineering Journal. 164, 107790(2020).
- Isaksson, L., Enberg, J., Neutze, R., Göran Karlsson, B., Pedersen, A. Expression screening of membrane proteins with cell-free protein synthesis. Protein Expression and Purification. 82 (1), 218-225 (2012).
- Techner, J. M., et al. High-throughput synthesis and analysis of intact glycoproteins using SAMDI-MS. Analytical Chemistry. 92 (2), 1963-1971 (2020).
- Kim, H. C., et al. Implementing bacterial acid resistance into cell-free protein synthesis for buffer-free expression and screening of enzymes. Biotechnology and Bioengineering. 112 (12), 2630-2635 (2015).
- Rolf, J., Siedentop, R., Lütz, S., Rosenthal, K. Screening and identification of novel cGAS homologues using a combination of in vitro and in vivo protein synthesis. International Journal of Molecular Sciences. 21 (1), (2020).
- Haslinger, K., Hackl, T., Prather, K. L. J. Rapid in vitro prototyping of O-methyltransferases for pathway applications in Escherichia coli. bioRxiv. , (2020).
- Dopp, J. L., Tamiev, D. D., Reuel, N. F. Cell-free supplement mixtures: Elucidating the history and biochemical utility of additives used to support in vitro protein synthesis in E. coli extract. Biotechnology Advances. 37 (1), 246-258 (2018).
- Gregorio, N. E., Levine, M. Z., Oza, J. P. A user's guide to cell-free protein synthesis. Methods and Protocols. 2 (1), 24(2019).
- Cole, S. D., Miklos, A. E., Chiao, A. C., Sun, Z. Z., Lux, M. W. Methodologies for preparation of prokaryotic extracts for cell-free expression systems. Synthetic and Systems Biotechnology. 5 (4), 252-267 (2020).
- Chiba, C. H., Knirsch, M. C., Azzoni, A. R., Moreira, A. R., Stephano, M. A. Cell-free protein synthesis: advances on production process for biopharmaceuticals and immunobiological products. BioTechniques. 70, (2021).
- Laohakunakorn, N. Cell-free systems: A proving ground for rational biodesign. Frontiers in Bioengineering and Biotechnology. 8, 788(2020).
- Dondapati, S. K., Stech, M., Zemella, A., Kubick, S. Cell-free protein synthesis: A promising option for future drug development. BioDrugs. , 1-22 (2020).
- Noireaux, V., Liu, A. P. The new age of cell-free biology. Annual Review of Biomedical Engineering. 22, 51-77 (2020).
- Khambhati, K., et al. Exploring the Potential of Cell-Free Protein Synthesis for Extending the Abilities of Biological Systems. Frontiers in Bioengineering and Biotechnology. 7, (2019).
- Carlson, E. D., Gan, R., Hodgman, C. E., Jewett, M. C. Cell-free protein synthesis: Applications come of age. Biotechnology Advances. 30 (5), 1185-1194 (2012).
- Rosenblum, G., Cooperman, B. S. Engine out of the chassis: Cell-free protein synthesis and its uses. FEBS Letters. 588 (2), 261-268 (2014).
- Swartz, J. R. Transforming biochemical engineering with cell-free biology. AIChE Journal. 58 (1), 5-13 (2012).
- Cole, S. D., et al. Quantification of interlaboratory cell-free protein synthesis variability. ACS Synthetic Biology. 8 (9), 2080-2091 (2019).
- Caschera, F., Noireaux, V. A cost-effective polyphosphate-based metabolism fuels an all E. coli cell-free expression system. Metabolic Engineering. 27, 29-37 (2015).
- Levine, M. Z., et al. Activation of energy metabolism through growth media reformulation enables a 24-hour workflow for cell-free expression. ACS Synthetic Biology. 9 (10), 2765-2774 (2020).
- Hunt, J. P., et al. Streamlining the preparation of "endotoxin-free" ClearColi cell extract with autoinduction media for cell-free protein synthesis of the therapeutic protein crisantaspase. Synthetic and Systems Biotechnology. 4 (4), 220-224 (2019).
- Dopp, J. L., Jo, Y. R., Reuel, N. F. Methods to reduce variability in E. Coli-based cell-free protein expression experiments. Synthetic and Systems Biotechnology. 4 (4), 204-211 (2019).
- Sun, Z. Z., et al. Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology. Journal of Visualized Experiments: JoVE. , e50762(2013).
- Schinn, S. M., Broadbent, A., Bradley, W. T., Bundy, B. C. Protein synthesis directly from PCR: Progress and applications of cell-free protein synthesis with linear DNA. New Biotechnology. 33 (4), 480-487 (2016).
- Sitaraman, K., et al. A novel cell-free protein synthesis system. Journal of Biotechnology. 110 (3), 257-263 (2004).
- Marshall, R., Maxwell, C. S., Collins, S. P., Beisel, C. L., Noireaux, V. Short DNA containing χ sites enhances DNA stability and gene expression in E. coli cell-free transcription-translation systems. Biotechnology and Bioengineering. 114, 2137-2141 (2017).
- Sun, Z. Z., Yeung, E., Hayes, C. A., Noireaux, V., Murray, R. M. Linear DNA for rapid prototyping of synthetic biological circuits in an escherichia coli based TX-TL cell-free system. ACS Synthetic Biology. 3 (6), 387-397 (2014).
- Addgene: pJL1. , Available from: https://www.addgene.org/69496/ (2021).
- IDT Codon Optimization Tool. , Available from: https://www.idtdna.com/pages/tools/codon-optimization-tool (2021).
- Hadi, T., et al. Rolling circle amplification of synthetic DNA accelerates biocatalytic determination of enzyme activity relative to conventional methods. Scientific Reports. 10 (1), 10279(2020).
- New England Biolabs Tm Calculator. , Available from: https://tmcalculator.neb.com/#!/main (2021).
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. Coli RNA polymerase and sigma factor 70. Journal of Biological Engineering. 4, (2010).
- Colant, N., et al. A rational approach to improving titer in Escherichia coli-based cell-free protein synthesis reactions. Biotechnology Progress. 37 (1), 3062(2021).
- Burgess-Brown, N. A., et al. Codon optimization can improve expression of human genes in Escherichia coli: A multi-gene study. Protein Expression and Purification. 59, 94-102 (2008).
- Maertens, B., et al. Gene optimization mechanisms: A multi-gene study reveals a high success rate of full-length human proteins expressed in Escherichia coli. Protein Science. 19 (7), 1312-1326 (2010).
- Eckert, K. A., Kunkel, T. A. DNA polymerase fidelity and the polymerase chain reaction. Genome Research. 1 (1), 17-24 (1991).
- Dopp, J. L., Reuel, N. F. Process optimization for scalable E. coli extract preparation for cell-free protein synthesis. Biochemical Engineering Journal. 138, 21-28 (2018).
- Liu, D. V., Zawada, J. F., Swartz, J. R. Streamlining Escherichia Coli S30 extract preparation for economical cell-free protein synthesis. Biotechnology Progress. 21 (2), 460-465 (2005).
- Levine, M. Z., Gregorio, N. E., Jewett, M. C., Watts, K. R., Oza, J. P. Escherichia coli-Based Cell-Free Protein Synthesis: Protocols for a robust, flexible, and accessible platform technology. Journal of Visualized Experiments: JoVE. (144), e58882(2019).
- Kwon, Y. C., Jewett, M. C. High-throughput preparation methods of crude extract for robust cell-free protein synthesis. Scientific Reports. 5, (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
ISSN 2578-2614
Copyright © 2025 MyJoVE Corporation. All rights reserved
We use cookies to enhance your experience on our website.
By continuing to use our website or clicking “Continue”, you are agreeing to accept our cookies.