Method Article
열토가 마리티마 M42 아미노펩티다제 TmPep1050의 올리고메릭 상태 전환을 연구하는 엑스레이 결정학
요약
이 프로토콜은 구조 수준에서 M42 아미노펩티다아제인 TmPep1050의 디머-도데카메르 전환을 연구하기 위해 개발되었다. 단백질 정제에서 X선 데이터 처리에 이르기까지 간단한 파이프라인입니다. 결정발생, 데이터 세트 인덱싱 및 분자 교체는 연구의 경우, TmPep1050H60A H307A 변이체를 통해 강조되었다.
초록
M42 아미노펩티다스는 12개의 서브유닛으로 이루어진 기능활성 복합체를 형성한다. 그들의 조립 과정은 이머 도데카메르 전환을 유발하는 금속 이온 공동 인자에 의해 규제되는 것으로 보입니다. 금속 이온 바인딩시 활성 부위및 상호 작용 인터페이스에서 여러 가지 구조적 수정이 발생하여 자체 조립을 촉진하기 위해 희미한 모양을 형성합니다. 이러한 수정을 관찰하려면 구조 연구 전에 안정적인 올리고머를 분리해야 합니다. 여기에 보고된 TmPep1050의 안정적인 도데카머와 디머, T. 마리티마의M42 아미노펩티다아제, X선 결정학에 의한 구조 결정의 정화를 허용하는 방법이다. 디머는 킬링 에이전트로 금속 이온을 제거하여 도데카머로부터 제조되었습니다. 그들의 보조 인자가 없으면, dodecamers는 덜 안정되고 가열시 완전히 해리되었다. oligomeric 구조는 간단한 분자 대체 접근에 의해 해결되었습니다. 방법론을 설명하기 위해 금속 이온 결합에 완전히 손상된 TmPep1050 변종의 구조는 단량체에 대한 희미한 기조의 더 이상 분해를 보여주지 않는 것으로 제시된다.
서문
올리고머화는 많은 단백질의 생물학적 기능을 지시하는 지배적인 과정입니다. 대장균에서는단백질의 35%만이 단황1인것으로 추정됩니다. 모르피인이라고 불리는 일부 단백질은 각 올리고메릭 상태2에서뚜렷한 구조를 갖는 하위 단위를 가진 여러 올리고메릭 상태를 채택할 수도 있다. 그들의 올리고메릭 상태 사이 전환은 수시로 각 올리고머 국가가 다른 특정 활동 또는 기능을 가질 수 있기 때문에 단백질 활동을 통제하는 평균입니다. 모페에인의 몇 가지 예는 문학, 특히 포르포빌리노겐 신타제3,HPr 키나아제/포스파타제4,론 프로테아제5,락테이트 탈수소효소6,글리세랄데히드-3-인산염 탈수소효소7,피루바테 키나제8,피루바테 키나제 8, 표루바테 키나테 레카제 8, 피루바테 키나테스 8, 카테카테 키나테스10,카테카테 레테이트에이질성 에서 잘 문서화되어 있다. 최근에는 M42 아미노펩티다아제 TmPep1050을 설명했는데, 모피인과 같은 행동을 가진 효소의 또 다른 예는 그의 활동이 올리고메릭상태(11)에의존한다. 그것의 oligomeric 상태 사이 전환은 하위 단위의 몇몇 구조적인 수정을 유도하는 그것의 금속 공동 인자에 의해 중재됩니다.
M42 아미노펩티다아제 가족은 MH 클랜12,13에속하며 박테리아와고고학(14)에널리 분포되어 있다. M42 아미노펩티다스는 펩티드를 분해하는 정품 이핵 효소로, 길이15의아미노산 잔기는 최대 35개까지 이다. 12개의 서브유닛으로 만들어진 독특한 테트라헤드론 모양의 구조를 채택하고, 내부 캐비티를 지향하는 활성 부위를 가지고 있습니다. 이러한 배열은 종종 제어되지 않는 프로테오리시스를 피하기 위해 활성의 나노 구획화로 설명된다. M42 아미노펩티다스의 생리적 기능은 단백질분해(16,17)에서발생하는 프로테솜, 가수분해 펩타이드와 연관될 수 있다. 피로코커스 호리코시이는 4개의 M42 아미노펩티다아제(aminopeptidases)를 보유하고 있으며, 각각 구별되지만 보완적인특이성(18,19,20,21)을제시한다. 단수적으로, 두 가지 유형의 서브유닛으로 만든 이종복합체는 P. horikoshii에설명되어 있으며, 펩티다섬복합체(22,23)의존재를 시사한다.
M42 아미노펩티다제의 여러 구조는 문헌(11,16,18,19,20,24,25, 25,26)에기재되었다. 하위 단위는 두 개의 고유한 도메인, 촉매 도메인 및 이량화 도메인으로 구성됩니다. 촉매 도메인은 전체 MH 일족에 보존 공통 α /β 접을 채택, 고풍스러운 촉매 도메인인 아미노 펩티다아제 AP1 되는 비브리오 프로테오리틱스27. 이 분화 도메인은 PDZ와 같은 접이식16을 채택하고, 올리고머화에서의 역할 외에도, 내부캐비티(11)에서기판 접근 및 바인딩을 제어하는 역할을 가질 수 있다. 기본 빌딩 블록은 이량자이기 때문에, 도드카메르는 종종 6 개의 희미한 사람의 협회로 설명되며, 각 디머는 테트라 헤드론(16)의각 가장자리에 배치된다. M42 아미노펩티다스의 올리고머화는 금속 보조인의 가용성에 의존한다. 종종 Zn2+ 및 Co2+의희석금속 이온은 펩티드 결합 및 가수분해에 촉매적으로 관여합니다. 그들은 두 개의 별개의 바인딩 사이트에서 발견 됩니다., 즉 M1 및 M2 사이트. 두 금속 이온은 또한 PhTET2, PhTET3, PfTET3 및 TmPep105011,24,28,29에대해 입증된 바와 같이 올리고머화를 정밀하게조정한다. 금속 보조 인자가 고갈되면, 도드카메르는 PhTET2, PhTET3 및 TmPep105011,16,28,또는 단량제와 같이 디머로 분해됩니다.
여기에 TmPep1050 올리고머의 구조를 연구하는 데 사용되는 프로토콜이 여기에 제시된다. 이 프로토콜은 단백질 정제, 단백질 활성 스크리닝, 결정화, X선 회절 및 분자 대체를 포함하는 일반적인 방법의 집합입니다. 메탈로엔자임, 단백질 올리고머화, 단백질 결정화 및 분자 대체를 다루는 데 내재된 미묘함을 강조합니다. 연구의 경우 는 또한 TmPep1050 dodecamers 단량체로 더 해리 수 있는지 여부를 보여주기 위해 제시된다. 이 문제를 해결하기 위해 TmPep1050 변종 TmPep1050H60A H307A는그의-60 (M2 사이트) 및 His-307 (M1 사이트)을 Ala 잔류물으로 돌연변이하여 금속 결합 부위가 손상된 금속 결합 부위를 연구했습니다. 이 프로토콜은 다른 M42 아미노펩티다제 또는 모르피인과 같은 행동을 가진 금속로효소를 연구하기 위해 수용될 수 있다.
프로토콜
1. 재조합 TmPep1050의 생산 및 정화
참고: 이하 복제 절차 및 이전 연구에서 적응된 야생형 TmPep1050의정화(11)를설명한다. 대안적으로, 복제는 합성 유전자를 사용하여 수행 될 수있다. TmPep1050 변종을 생성하기 위해, 사이트 지향 돌연변이 발생은 병렬 프로토콜(SPRINP)방법(30)에서단일 프라이머 반응과 같은 다음 수행될 수 있다. 정제 프로토콜은 TmPep1050 변종에 사용할 수 있습니다. 금속 이온 바인딩을 방해하기 때문에 그분의 꼬리표를 사용하는 것을 피해야 합니다.
- 표현 벡터 디자인
- Thermotoga maritima MSB8 (ATCC 43589) 또는 TmCD00089984 (구조 유전체학을 위한 공동 센터)의 유전체 DNA를 취득합니다.
- 유전체 DNA 또는 템플릿 플라스미드를 사용하여 TM_1050 열린 판독 프레임(ORF)을 증폭하고, 고충실도 DNA 폴리머라제, 그리고 다음 프라이머: ocej419 (5'-TTTAACTTTAAAGGAGA타타카타카타가아가AGAGAGAGAGAGAGAGAGAGAGAGAGAGAGAGAGAGAGAGAGAGCTGG) 및 ocej420 (5'-ATCCGCCAAACAAGAGAGCCGTG가가CCCCCCCCCCCCCCCCCCCCCCAGCCCCCCCCCCCCCCCCCCCCCCCCCCCCCCCCCCAGCCCCCCCCAGCCCCCCCCAGCCCCCCAGCCCCCCAGCCCCAGCCCCAGCCCCCCAGCCCCAGCCCCCCAGCCCCCCAGCCCCCCCCCCAGCCCCAGCCCCCCAGCCCCAGCCCCAGCCCCAGCCCCCCCCAGCCCCCCAGCCCCAGCCCCCCCCAGCCCCCCCCCCCCAGCCCCCCCCAGCCCCCCAGCCCCAGCCCCCCAGCCCCCCAGCCCCAGCCCCCCAGCCCCCCCCCCAGCCCCCCCCAG 다음 계획에 따라 폴리머라제 연쇄 반응(PCR) 스크리닝을 실행: 95°C에서 5분, 3단계의 30사이클(95°C에서 30초, 55°C에서 30초), 72°C에서 90초), 최종 단계로서 72°C에서 10분.
- SLiCE프로토콜(31)에따른 대장균에서 균동재조합(도1)에의해 PCR 단편을 적절한 발현벡터(재료표)로복제한다. 선형 벡터의 50 ng에, 10:1 μL 의 ppY 스트레인 추출물, 50 mM Tris-HCl pH 7.5, 100 mM MgCl2,10mM ATP 및 10 mM 디티오트레이톨(DTT)에 10mM 디티오트리톨(DTT)을 추가하여 10°C°C의 반응에 대해 10:1 μl의 프로스트 비비율로 PCR 단편을 추가한다.
- 재조합 반응의 1 μL로 화학적으로 유능한 대장균 XL1 블루 스트레인(또는 모든 recA-적합한 변형)을 변환합니다. 100 μg/mL 암피실린을 포함하는 LB 배지에 세포를 플레이트. 플레이트를 37°C에서 하룻밤 동안 배양합니다.
- 100 μg/mL 암피실린으로 신선한 LB 플레이트에서 콜로니를 선택하십시오. 플레이트를 37°C에서 최소 8시간 동안 배양한다.
- 적절한 프라이머 쌍을 사용하여 콜로니 PCR에 의해 긍정적 인 후보에 대한 화면 (5'- ATGCCATAGCATTTATCC 및 5'- ATTTAATCTGTATCAC 재료의 표에나열된 권장 벡터를 사용하는 경우). 마이크로팁 끝으로, 고른 콜로니를 긁어내고 각 프라이머의 0.5 μM 및 상업용 Taq DNA 폴리머라제 믹스의 10 μL을 포함하는 반응 혼합의 20 μL로 세포를 이송한다.
- 다음 계획에 따라 PCR 스크리닝을 실행: 5내지 95°C에서 95°C에서 3단계(95°C에서 30초, 55°C에서 30초, 72°C에서 90초), 최종 단계로서 72°C에서 10분.
참고: PCR 반응은 12°C에서 PCR 기계에 하룻밤 동안 저장할 수 있습니다. - 트리스 아세테이트-EDTA(TAE) 버퍼에서 제조된 0.8% 아가로즈 젤에 각 PCR 반응의 10 μL을 적재한다. 전기 전도를 100 V에서 25 분 동안 실행합니다.
참고: 1.1 kbp 앰플리턴이 예상됩니다. - 상용키트(재료표)를사용하여 후보자로부터 플라스미드를 추출하고 1.1.6 단계에서 사용되는 동일한 프라이머 쌍을 사용하여 시퀀스합니다.
- 세포 배양
참고: 적절한 후보가 시퀀싱에 의해 확인된 경우, 권장벡터(재료표)를사용하는 경우 클론을 표현식으로 직접 사용할 수 있다. 이 경우, 발현은 아라비노스 유도PBAD 프로모터(32)에의해 제어된다.- 100 μg/mL 암피실린을 함유한 LB 배지의 10mL을 접종하고 궤도 흔들림 하에서 37°C에서 하룻밤 동안 배양전을 배양한다. 100 μg/mL 암피실린을 사용하여 LB 배지 1L에 프리컬쳐 5mL를 추가합니다. 공기대 액체 비율을 3의 존중하는 마음.
- 세포가 궤도 흔들림 하에서 37°C에서 자랄 수 있도록 합니다. 660 nm (OD660)에서광학 밀도를 모니터링합니다.
- OD660이 0.5-0.6에 도달하면, 얼음에 5 분 동안 문화를 빠르게 냉각하고 18 °C로 설정 인큐베이터로 전송합니다.
- 유전자 발현을 유도하고 18°C에서 12-18h의 배양에 0.2 g/L 아라비노스를 첨가합니다.
- 4°C에서 30분 동안 6,000 x g에서 배양을 원심분리하여 세포를 수확한다. 0.9%(w/v) NaCl의 100mL로 수퍼날레를 버리고 셀을 세척합니다.
- 원심분리기는 4°C에서 15분 동안 6,000 x g에서 다시 원심분리기를 제거하고 상체를 폐기합니다.
참고: 세포 펠릿은 단백질 추출을 위해 직접 사용하거나 -80°C에 저장될 수 있습니다.
- 단백질 정제
- 50mM M의 40mL, 1mM CoCl2,pH 7.2의 세포 펠릿을 재보습한다. EDTA를 포함하지 않는 25 개의 U / μL DNA / RNA 엔도누셀및 프로테아제 억제제 칵테일 1 정육을 1 μL추가하십시오. 냉각 중인 펄스 모드에서 서스펜션을 30분 동안 초음파 처리합니다.
- 4°C에서 30분 동안 20,000 x g의 원유 추출물을 원심분리합니다. 상체를 수집하고 10 분 동안 70 °C에서 수조에서 가열합니다.
- 4°C에서 30분 동안 20,000 x g의 변성 세포 추출물을 원심분리하고 정제를 위한 상퍼를 수집합니다.
- ~15mL의 부피의 기둥에 포장된 적절한 음이온 교환 수지(재료표)를사용하십시오. 작동 유량 및 열 압력 제한에 대한 제조업체의 권장 사항을 참조하십시오. 수지 50m MOPS, 1mM CoCl2,pH 7.2로 수지를 평형화한다.
- 1.3.3 단계에서 수집된 상체를 열에 로드합니다. 280 nm에서 용액의 흡수도를 모니터링합니다. 기준선에 도달하면 용출로 진행합니다.
- 50mM MOPS에서 0~0.5M NaCl, 1mM CoCl2,5열 볼륨(CV)에 pH 7.2의 그라데이션을 적용합니다. 전도도가 안정되고 흡수도가 기준선에 도달할 때까지 기다립니다.
- 최종 그라데이션은 0.5~1M NaCl에서 50mM MOPS, 1mM CoCl2,1CV에 대해 pH 7.2로 적용한다.
- 도데딜 황산염 폴리아크라이아미드 젤 전기포레시스(SDS-PAGE)에 의해 일부 분획(도 2A 참조)을 분석한다.
참고 : TmPep1050 쿠마시 염색 후 36 kDa 밴드로 나타납니다. 대안적으로, TmPep1050의 존재는 활동 분석으로 확인할 수 있습니다(섹션 2.1 참조). 이 단계에서 분획은 하룻밤 사이에 4 °C에서 저장할 수 있습니다. - TmPep1050을 함유한 분획을 풀링하고 1.5M(NH4)2SO4의 농도를 얻기 위해 (NH4)2SO4의 미세하게 접지 분말을 첨가한다. 튜브를 거꾸로 뒤집어 서 완전히 용해될 때까지 부드럽게 섞습니다.
- ~30mL의 부피의 기둥에 포장된 소수성 상호작용수지(재료표)를사용한다. 작동 유량 및 열 압력 제한에 대한 제조업체의 권장 사항을 참조하십시오. 수지 50m M, 1.5M(NH4)2SO4,1mM CoCl2,pH 7.2로 수지를 평형화한다.
- 샘플을 컬럼에 로드하고 280 nm에서 용액의 흡광도를 모니터링합니다. 흡광도가 기준선에 도달하면, 50mM MOPS에서 1.5M ~0M(NH4)2SO4의 그라데이션을 적용하여, 1mM CoCl2,5CV에 대한 pH 7.2를 적용하여 단백질을 결합하였다.
- SDS-PAGE에서 일부 분획(지침그림 2B 참조)을 분석합니다.
참고 : TmPep1050 쿠마시 염색 후 36 kDa 밴드로 나타납니다. 대안적으로, TmPep1050의 존재는 활동 분석으로 확인할 수 있습니다(섹션 2.1 참조). 이 단계에서 분획은 하룻밤 사이에 4 °C에서 저장할 수 있습니다. - TmPep1050을 포함하는 분획을 풀링하고 30 kDa차단장치(재료표)를사용하여 초여과 장치를 사용하여 2mL에 농축한다. 분자량을 결정하기 위해 섹션 1.4로 진행합니다.
- 크기 제외 크로마토그래피
- 크기 배제 수지(재료의 표)를사용 ~ 120 mL의 볼륨의 열에 포장. 작동 유량 및 열 압력 제한에 대한 제조업체의 권장 사항을 참조하십시오. 수지 50m M, 0.5M(NH4)2SO4,1mM CoCl2,pH 7.2로 수지를 평형화한다.
- 샘플을 컬럼에 로드하고 280 nm에서 용액의 흡광도를 모니터링합니다. 열 데드 볼륨(~0.33 CV)에서 용출(1 CV)의 끝까지 분획합니다.
- 관찰된 각 피크에 대해 용출 부피를 측정합니다.
참고: 지도를 위해, 도드카메리크 TmPep1050 elutes ~82 mL(그림 3A)현재 실험 조건하에서, TmPep1050H6050 H60A H307A 변종, ~95 mL(그림 3B)에서엘utes. 일부 TmPep1050은 TmPep1050H60A(도 3C)와같은 올리고머 형태를 모두 채택할 수 있다. - SDS-PAGE를 사용하여 관찰된 피크의 최대값및 꼬리에 해당하는 분수를 분석합니다.
참고 : TmPep1050 쿠마시 염색 후 36 kDa 밴드로 나타납니다. - 각 피크의 분획을 풀과 30 kDa 컷오프(재료의 표)와초여침 단위를 사용하여 농축 - 300 μM의 농도를 얻을 수 있습니다.
- 나노 부피 분광계에 280 nm에서 흡광도를 측정하고 18,910 M-1cm -1의분자 소멸 계수를 사용하여 농도를 계산한다.
- 정제된 단백질을 -18°C에 저장합니다.
- 분자량을 결정하기 위해 분자량 기준(재료 표)을 사용하여 크기 배제 크로마토그래피(SEC)컬럼을 보정합니다. 50mM MOPS, 0.5M(NH4)2SO4,1mM CoCl2 pH 7.2를 실행 버퍼로 사용하여 표준을 분석한다.
2. 활동 분석 및 아포 효소 제제
참고: 원래 아포 효소는 2.1M 말산 pH 7.0의 10부에서 TmPep1050의 1부피를 희석하고 투석11전에1부로 다시 농축하여 제조하였다. 아래는 금속 이온 첼레이터인 1,10-페난트로인을 사용한 대체 절차가 제시되어 있습니다. 이 절차는 단백질 손실을 감소시키고 이전에 발표된 방법보다 동일한 결과를 제공합니다.
- 활동 분석
- 메탄올에 100mM L-류신-p-니트로아닐드(재료표)의 스톡 솔루션을 준비한다.
- 100mM L-류신-p-니트로아닐드의 25 μL을 50m M MOPS의 965 μL, 250 μM CoCl2,pH 7.2, 10% 메탄올에 추가합니다. 마른 목욕에서 75 °C에서 반응 믹스를 미리 배양하십시오.
- 50m MOPS pH 7.2의 농도로 효소를 희석하여 1 μM의 농도로 희석합니다.
- 20% 아세트산의 1mL를 첨가하여 반응을 중지합니다. 소용돌이잘 하고 실온까지 식힙니다.
- 분광광계 세포에서 반응 믹스를 전달합니다. 부정적인 대조군(효소 없이 배양된 반응 믹스)에 대해 410 nm에서 흡광도를 읽습니다.
- 아포 효소 제제
- 에탄올에 1M 1,10-페난트로라인의 스톡 솔루션을 준비한다. 50mM MOPS의 890 μL, 0.5M (NH4)2 SO4,pH 7.2의 1,10-페난트렐 재고 용액10μL을 추가합니다. 정제된 TmPep1050(300 μM-1 mM 농도)의 100 μL을 추가합니다.
- 반응 혼합물에 CoCl2를 추가하지 않고 섹션 2.1에 설명된 활동 분석체를 사용하여 활동 손실을 확인한다.
- 투석 튜브에 샘플을 전송합니다. 50mM M의 200mL, 0.5M(NH4)2SO4,pH 7.2에 대하여 4°C에 대하여 투석한다. 48h 투석 시 신선한 버퍼로 투석을 세 번 교환합니다.
- 투석관에서 샘플을 수집하고 30kDa컷오프(재료표)를이용한 초여통 유닛을 사용하여 100 μL로 다시 농축한다. 나노 부피 분광계를 사용하여 280 nm에서 흡광도를 판독하여 농도를 확인하십시오.
- 디머 준비
- 아포 효소를 50m MOPS, 0.5M(NH4)2SO4,pH 7.2에서 1 μM의 농도로 희석한다. 마른 목욕에서 75 °C에서 2 시간 동안 배양 한 다음 샘플을 실온으로 식힙니다.
- 샘플을 최소 50μM의 효소 농도로 농축합니다. 용출 피크는 ~82mL에서 ~95mL로 이동해야 합니다(현재 실험 조건 하에서).
3. TmPep1050 결정화
참고: 단백질 결정화는 다인자 현상33이기때문에 경험적 과학으로 남아 있다. 일부 파라미터는 식별 및 제어할 수 있지만(예: 온도, pH, 강수제 농도), 다른 매개 변수는 결정화(예: 단백질 및 화학 순도, 프로테오리시스, 샘플 기록)에 영향을 줄 수 있다. 요즘 단백질 결정화는 상업적 결정화 선별 조건 및 자동화의 무리 덕분에 합리적이고 체계적인 방식으로 다루어지고 있습니다. 그러나 결정화 조건의 최적화는 주로 시행 착오 방식에 의존합니다. 이하 단백질결정화에 대한 청사진과 결정화 조건을 최적화하기 위한 몇 가지 팁이 설명되어 있다.
- 결정화 스크리닝
참고: 상업적 결정화 키트를 사용하여 도드카메릭 TmPep1050의 결정은 2.2M DL-malic acid pH 7.0, 0.1 M 비스 트리프로판 pH 7.0 및 0.18 M 트라이 암모늄 질산염, 20% 폴리에틸렌 글리콜(PEG) 350에서 얻어졌다. 디메라 TmPep1050의 결정은 0.1 M 나트륨 구연산 pH 5.6, 0.2 M 암모늄 아세테이트, 30% PEG4000에서 얻어졌다. 도데카운더의 결정은 일주일 이내에 나타나고 한 달에 전체 크기에 도달합니다. 디머의 결정은 일반적으로 24 시간 이내에 나타나고 일주일에 전체 크기로 성장합니다.- 여러 상용 결정화 키트를 획득합니다(예: 재료 표 참조).
- 매달려 있는 낙하 방법에 대한 결정화플레이트(재료 표)를설정합니다. 결정화 스크리닝 키트의 각 용액의 500 μL로 우물을 채웁니다.
- 각 웰에 대해 결정화 지원을 설정합니다. 지지체에, 정제 된 단백질의 1 μL 방울을 입금 (일반적으로 ~10 mg/mL).
- 즉시 우물에서 결정화 용액의 1 μL을 파이프. 단백질 드롭에 조심스럽게 넣고 세 번 거꾸로 피펫을 사용하여 부드럽게 섞습니다. 드롭은 거품없이 반구형으로 유지해야합니다.
- 해당 우물 위에 지지대나사를 나사로 배치합니다. 전체 키트에 대한 작업을 반복합니다.
- 플레이트를 설정한 후 쌍안경으로 각 방울을 관찰합니다. 해석을 위한 결정화 키트 사용자 가이드를 참조하십시오(클리어 드롭, 위상 분리, 침전물, 바늘 등).
- 플레이트를 20°C에서 배양합니다. 첫 주에 하루에 한 번, 그 후 일주일에 한 번 플레이트를 확인하십시오.
- 결정화 키트와 함께 제공되는 점수 시트와 사용자 가이드를 사용하여 각 점수를 매기.
- 결정화 최적화
참고: 도드카메릭 TmPep1050의 초기 결정화 조건은 2.1 M DL-malic acid pH 6.75 및 0.18 M 트라이 암모늄 구연산 pH 7.5에 최적화되었습니다. 40% (w/v) PEG3350의 결정화 상태는 0.1M 디메페1050으로 이동하였으며, 구연산 나트륨 pH 6.0, 10% (w/v) PEG3350으로 이동하였다. 결정성을 향상시키기 위해 시드의 한 주기가 필요했습니다. 이하 TmPep1050H60A H307A 변종의 결정화가 어떻게 최적화되었는지설명되어 있다.- 다른 pH(4.5, 5.2 및 6.0) 및 50% (w/v) PEG3350 용액에서 0.5M 나트륨 구연산 버퍼의 스톡 솔루션을 준비합니다.
- 결정화 플레이트를 pH 대 강수제의 매트릭스로 설정합니다(그림 4A참조).
- 플레이트를 20°C에서 배양합니다. 일주일에 한 번 쌍안경으로 각각 잘 관찰하십시오.
- 크리스탈 크기와 모양에 따라 각각 잘 점수. 최소 50 μm의 결정을 주는 조건을 선택합니다. 마이크로 시드로 진행합니다.
- 마이크로 시드
참고: 마이크로시드는 단백질결정(34)의모양, 크기 및 결정성을 향상시키는 강력한 방법입니다. 더 빠른 파종 방식은 고양이 수염을 사용하여 줄무늬 파종입니다. 결정화 최적화 및 미세 시제가 TmPep1050H60A H307A의결정 모양과 크기를 개선한 예로서 도 4를 참조하십시오.- 3.2.4 단계에서 선택한 잘 사용하여 씨앗을 준비합니다.
- 잘에서 결정화 용액을 추가하여 결정액을 함유한 낙하의 부피를 10 μL로 늘립니다. 드롭을 파이프하고 우물에서 결정화 용액의 90 μL을 추가합니다. 소용돌이를 철저히 하고 씨앗을 얼음 위에 보관하십시오.
- 1x, 10배, 25배, 100x의 씨앗희석을 준비합니다. 소용돌이 잘 파이팅 하기 전에 씨앗. 희석을 얼음 위에 두십시오.
- 각 종자 희석에 대해, pH 대 강수제의 매트릭스로 결정화 플레이트를 설정(도 4B참조). 3.2.1 단계에서 준비된 스톡 솔루션을 사용하십시오.
- 드롭을 할 때, 2 μL 드롭에 대한 씨앗의 0.2 μL을 추가합니다. 플레이트를 20°C에서 배양합니다. 일주일에 한 번 쌍안경으로 각각 잘 관찰하십시오.
참고: 크리스탈 크기 분포 및 모양을 개선해야 하며, TmPep1050H60A H307A의예로 그림 4C를 참조하십시오. 추가 용도로 씨앗은 -20 °C에 보관할 수 있습니다.
4. 엑스레이 회절
- 크리스탈 피킹
참고: 샘플 준비는 X선 소스 시설(홈 시설 대 싱크로트론)에 따라 달라집니다. 그에 따라 저장 장치(바이알 및 바이알 홀더 바구니)를 사용합니다. 냉동 보호제 (글리세롤과 같은)의 첨가는 소금 /강수제 농도에 따라 필요할 수 있습니다. TmPep1050 결정의 경우, 못 또는 완충체 농도가 수결정을 피할 만큼 충분히 높기 때문에 냉동 보호제가 필요하지 않습니다.- 액체 질소로 채워진 목욕을 준비하고, 샘플 처리에 사용되는 바이알이나 바구니를 떨어 지습니다.
- 100, 150 및 200 μm (재료 표)와 같은 다양한 크기의 샘플 따기루프를 설정합니다. 크리스탈 크기에 따라 크기를 선택합니다.
- 쌍안경을 사용하여 결정이 포함된 드롭을 확인하고 격리된 크리스탈(가장 쉽게 선택)을 발견하십시오. 루프를 사용하면 아래에서 크리스탈을 부드럽게 선택합니다. 즉시 액체 질소에 루프를 급락하고 적절한 유리병에 루프를 배치합니다.
- 데이터 수집
참고: 데이터 수집은 X선 소스(홈 시설 대 싱크로트론) 및 검출기 민감도에 따라 크게 달라질 수 있습니다. 수집 전략은 해상도, 스팟 강도, 공간 그룹 등에 따라 샘플과 다른 샘플과 크게 다를 수 있습니다. 이 주제는 Dauter35에의해 광범위하게 검토되었습니다.- 디락토미터의 고니오미터 헤드에 크리스탈을 운반하는 루프를 장착합니다.
- XYZ 축을 따라 고니오미터 헤드를 조정하여 크리스탈을 X선 빔 경로와 정렬합니다.
- 파장을 0.98 Å로 설정하고 검출기를 이동하여 2Å해상도를 얻습니다.
- 적어도 두 개의 서로 다른 결정 방향으로 이미지를 수집하여 짧은 데이터 수집을 시작합니다. 0°및 90°에서 10개의 이미지(0.1°당 1개 이미지)를 취합니다.
- 적절한 소프트웨어(예: ADXV, XDS-뷰어 또는 알불라 뷰어)로 수집된 이미지를 확인합니다. 스팟 강도와 반점이 보이는 가장 높은 해상도를 결정합니다. 단결정성도 확인하고 분리를 찾아보세요.
- 결국, 관측에 따라 검출기 위치를 더 높거나 낮은 해상도및 노출 시간에 대해 변경하여 4.2.3-4.2.5단계를 반복한다.
- 0.1°당 1개의 이미지로 약 360°의 데이터 수집을 시작합니다. 검출기 위치와 노출 시간을 최적으로 설정해야 합니다.
5. 색인, 분자 교체 및 모델 건물
- Indexation
참고: 인덱싱은 회절 반점 강도를 측정하여 구조계수(36)의진폭을 주는 방법입니다. 모즈플름37,HKL200038,다이얼39, XDS40등 4개의 소프트웨어 패키지가 수집된 이미지를 처리하는 데 일반적으로 사용됩니다. 후자는 TmPep1050 크리스탈 회절에서 얻은 데이터 세트를 인덱싱하는 데 사용되었습니다.- XDS 패키지 및 XDSME41을설치합니다. HDF5 파일을 처리하는 경우 XDS Neggia 플러그인을 설치하십시오(데트리스 웹 사이트에서 사용 가능). 자세한 내용은 XDS 위키 웹 페이지 https://strucbio.biologie.uni-konstanz.de/xdswiki/index.php/Main_Page 및 XDSME 웹 페이지 https://github.com/legrandp/xdsme 방문하십시오.
- 데이터를 처리하기 전에 XDS가 실행되는 폴더를 만듭니다. 이미지에 대한 경로를 찾습니다.
- XDSME를 실행하려면 터미널 창에 xdsme /path_to_images/image.extension을 입력합니다.
- XDS가 작업을 종료한 후 올바른 작업을 확인합니다. LP 파일입니다. 공간 그룹 결정, 데이터 완전성, 최고 해상도, 크리스탈 모사이시티 및 데이터 품질의 확률을 기록합니다. 또한 XDS_pointless.log 확인하여 공간 그룹의 가능성을 얻습니다.
참고: 그림 5를 출력의 예로 봅타입니다. - 이전 프로세스를 덮어쓰지 않도록 별도의 폴더에서 XDS에서 제안한 다양한 공간 그룹 솔루션으로 XDSME를 다시 실행합니다. 유형 xdsme-s space_group_name -c "unit_cell_parameters"/path_to_images/image.extension(예: xdsme-s P21 -c "43.295 137.812 61.118 90.000 110.716 90.000).
- 올바른 확인. LP 파일 및 데이터 통계를 기반으로 최상의 솔루션을 선택합니다.
- xscale.py XDS_ASCII 입력하여 XSCALE을 실행합니다. HKL. XSCALE xdsconv.py을 입력하여 XDSCONV를 실행합니다. HKL ccp4.
참고: 경우에 따라 XDSME는 공간 그룹을 식별하지 못하거나 해상도 범위를 제대로 자르지 못하거나 이상한 데이터 통계를 생성합니다. 이러한 문제가 발생하면 기본적으로 XDS를 실행할 가치가 있습니다. XDS에 여러 매개 변수를 도입해야 합니다. INP 개시 파일(XDS 위키 페이지 참조). XDS를 사용하는 경우 CCP4 패키지42의일부인 무의미한 공간을 사용하여 가능한 공간 그룹의 가능성을 확인할 수 있습니다. 데이터 세트 해상도를 줄이기 위해 Rmeas & 60% 및 I/σ ~2는 일반적으로 가장 높은 해상도43을결정하기 위해 허용됩니다. 분자 교체 및 모델 정제는, 그러나, I/σ ~0.5-1.5 및 CC 1/20.2-0.444로해상도를 확장하여 향상될 수 있다.
- 분자 대체
참고: 실험 데이터는 구조적 요인의 진폭에 접근할 수 있지만 위상을 알지 못하면 쓸모가 없습니다. 위상은 비정상적인 신호(예를 들어, 무거운 원자에서) (예를 들어)45에의존하는 다른 방법에 의해 실험적으로 결정될 수 있다. 분자 교체는 비정상적인 산란원자(46)및47없이 위상을 결정하는 또 다른 방법이다. 이 방법은 관련 분자의 좌표를 사용하여 위상을 반복적으로 찾고 개선합니다. 우리는 분자 대체를 위해 페닉스 GUI49에서 Phaser48을 사용합니다.- 4P6Y 좌표를 사용하여 분자 교체를 위한 시작 모델을 준비합니다. pdb 파일에서 모노머 A를 추출하고 Phenix의 PDB 파일 편집기 (모델 도구 탭 에서)를 사용하여 알라닌에 아미노를 잘라.
- XDSCONV(5.1.9)에서 생성된 반사 파일과 입력으로 시퀀스를 사용하여 Phenix(데이터 분석 탭 아래)에서 Xtriage를 실행합니다.
- Xtriage에서 로그 파일을 확인합니다. 완전성, 비대칭 단위의 하위 단위 수, 이불트로피, 얼음 고리의 존재 및 트위닝 발생에 유의하십시오.
- 반사 파일을 이용하여 분자 교체를 위한 페닉스(분자 대체 탭 아래)에서 페이저-MR을 실행하여 폴리 알라닌(step 5.2.1)으로 잘라진 서열 및 시작 4P6Y 모델.
- 완료되면 모델이 발견되었는지 확인하고 분자 대체 점수가 있는지 확인하십시오. 최소 8의 변환 계수 Z 점수(TFZ)는 솔루션이 결정적으로 정확하다는 것을 나타냅니다.
- 모델 빌딩
참고: 분자 대체에 의한 위상을 결정한 후 모델을 구축하고 정제해야 합니다. 이 프로토콜은 Phenix GUI49를 사용하여 자동 건물 및 반복적 개선을 위해, 커트50은 수동 구조 구축 및 개선을 위해 사용합니다.- Phenix에서 Phase-MR을 사용하여 분자 교체 후 자동 빌드 실행을선택합니다. 필요한 모든 파일이 자동으로 추가됩니다. 달리기를 눌러 자동 빌드를 시작하기만 하면 됩니다.
- 완료되면 쿠트에서 모델을 확인하십시오. 쿠트의 전자 밀도 맵에 따라 모델을 수동으로 빌드하고 구체화합니다.
- 모델, 시퀀스 및 회절 데이터를 입력으로 사용하여 Phenix(구체화 탭에서)에서 수동으로 선별된 모델을 구체화합니다. Phenix 도움말을 참조하여 올바른 전략을 선택하십시오.
- 개선 후 결과를 확인하십시오: R무료 및 R작업은 감소해야 하며 Molprobity51 지표는 존중되어야 하며 실제 공간 상관 관계가 낮은 이상값은 제한되어야 합니다.
- 최고의 정제 모델이 생성될 때까지 5.3.2-5.3.4 단계를 반복합니다.
- 서버에서 Molprobity를 실행합니다: http://molprobity.biochem.duke.edu/. Molprobity에서 식별 한 이상값을 확인하십시오.
- 결국 최고의 정제 모델을 얻을 때까지 단계 5.3.2-5.3.6을 반복합니다.
결과
TmPep1050에서 단량체로 가능한 dodecamer 해리를 연구하기 위해, His-60 및 His-307 코돈은 합성 유전자를 사용하여 알라닌 코돈으로 대체되었다. 이 유전자는 TmPep1050 이체의 발현 및 정제를 위한 pBAD 벡터로 복제된 후 TmPep1050H60A H307A라는이름의 이체이다. 크기 배제 크로마토그래피(도3B)는정제된 단백질이 56kDa(단량체의 분자량36.0 kDa)의 명백한 분자량을 가졌다는 것을 보여주었다. 비슷한 명백한 분자량, 52 kDa, TmPep1050 디머11에대 한 보고 되었습니다. 따라서 TmPep1050H60A H307A의 올리고메릭 상태는 희미하게 유추될 수 있습니다. 그것의 특정 활동에 관하여, TmPep1050H60A H307A는 코발트이온의 존재에도, 기판으로 L-Leu-p NA에 완전히 비활성이었습니다. 이 결과는 변형이 금속 이온을 묶을 수 없음을 강력하게 시사합니다.
TmPep1050H60A H307A의 결정화 상태는 이량제(즉, 0.1M 나트륨 구연산 pH 6.0 10% PEG3350)의 상태 주변의 다양한 pH 대 PEG농도(도 4)에의해 최적화되었다. TmPep1050H60A H307A의 최고의 결정은 단결정성 개선을 위한 마이크로시드의 1주기와 함께 0.1M 구연산 나트륨 pH 5.2 20% PEG3350에서 얻어졌다. 해상도 2.36Å(표 1)에서Proxima 2 빔라인(SOLEIL 싱크로트론)에서 전체 데이터 세트를 수집하였다. 데이터 색인은 TmPep1050H60A H307A 결정의 공간 그룹이 C2221이지만 XDS는 또 다른 솔루션인 mP 공간 그룹을 제안한 것으로 나타났다(그림 5참조). 무의미한 에 따르면, C2221 및 P21 우주 그룹의 가능성은 각각 0.711 및 0.149이었다. 데이터 품질 분석에 따르면 비대칭 단위에서 두 개의 단량체가 발견됩니다. Xtriage의 분석에 따르면 데이터 세트는 아마 트윈화되지만 C2221 스페이스 그룹에서 트위닝되는 것은52일가능성이 낮다는 것을 밝혀졌습니다. 여러 명확한 도메인이 서로 평행 한 자신의 격자 방향의 일부를 가지고 결정 성장 이상에서 트위닝 결과53. 트위닝은 또한 잘못된 데이터 인덱스를 나타내는 더 높은 결정 대칭으로 인해 발생할 수 있습니다. 따라서, P21 결정 격자가 C2221처럼보이게 되도록 의사 메로에드랄 쌍둥이가 존재할 수 있다. 데이터 세트는 이후 우주 그룹 P21에서 인덱싱되고 분자 대체에서 테스트되었습니다. P21에서 인덱싱된 데이터 세트의 Xtriage 분석은 쌍둥이 법 h, -k, -h-l에이어 의사 메로에드랄 쌍둥이를 드러냈다.
도드카미드 TmPep1050(PDB 코드 4P6Y)의 단량제 좌표를 사용하여 P21에서만 인덱싱된 데이터 세트에 대해 분자 대체 용액이 발견되었으며, TFZ 점수는 28.9입니다. 따라서 회절 데이터는 모델 빌드를 위한 트윈 데이터 집합으로 처리되었습니다. 분자 교체의 편견을 최소화하기 위해, 첫 번째 모델은 phenix.autobuild54,55를사용하여 제작되었다. TmPep1050H60A H307A의 구조는 페닉스와 쿠트(표 1 및 그림 6A)에서자동화 및 수동 정제의 여러 주기 후에 완료되었습니다. 구조는 PDBe Pisa56에의해 계산된 바와 같이 단량체와 δiG-1 사이의 1,710 Å2의 인터페이스 표면을 가진 올리고메릭 상태를 확인한다. 비교하여, 노름 TmPep10502-mer의 인터페이스 표면 및 ΔiG는 각각 1,673 Å2 및 -16.7 kcalmol-1이다.
TmPep1050H60A H307A의 구조는 정렬 시 0.774 Å의 RMS를 가진 야생 형 이머 구조와 매우 유사합니다. 중요한 것은, α8 및 α10 헬리액의 높은 유연성, 무질서한 활성 부위 Gln-196-Val-202, Lys-229-Ala-235 및 Lys-247-Ser 254의 변위: 두 구조 모두에서 동일한 구조적 변형이 관찰된다. 이러한 수정은 이전에 금속 공동 인자(11)가없는dodecamer 형성의 방해와 상관 관계가 있었다. 그러나 His-60과 His-307의 두 돌연변이는 Asp-168 및 Asp-62의 측면 사슬에 약간의 영향을 미쳤습니다. 그들은 야생 형 이머(도 6B)와다른 형태로 잠겨있는 것처럼 보였다. Asp-168 카박스일레이트는 His-60과 His-307이 없기 때문에 40° 회전합니다. 따라서 두 히스티딘 잔류물은 두 금속 이온을 브리지에 대해 Asp-168 카박스실레이트에 올바르게 배치하는 데 중요합니다. Asp-62 사이드 체인은 촉매 사이트 외부의 Glu-18 카복실레이트를 지향합니다. Asp-62는 His-60의pK를 조절하고, 따라서 M2 부위의 금속 이온 결합에 영향을 미치기 때문에 촉매에 중요한 역할을 할 수 있다. 또한, 도드카메의 형성을 선호하는 금속 이온 결합시 촉매 부위의 안정화에 구조적으로 연루될 수 있다.

그림 1: 동종 재조합에 의한 pBAD 벡터로 복제하는 TM_1050 ORF 클론의 회로도 표현.
ORF는 발기인 BAD 엔드및 서열 업스트림 PmeI 제한 부위에 상동성2개의 30bp 시퀀스에 의해 측면에 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
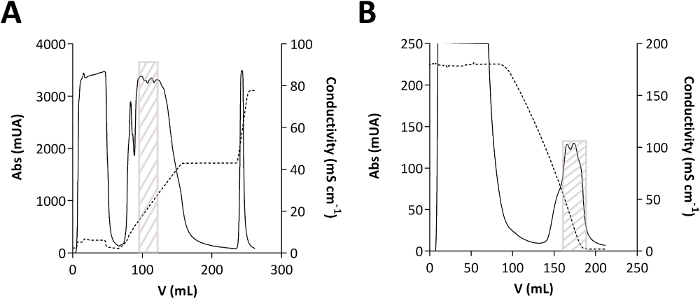
그림 2: TmPep1050 정제의 크로마토그램.
(A)애니온 교환 크로마토그래피. (B)소수성 상호 작용 크로마토그래피. 흡광도(mUA)의 밀리유닛으로 발현된 흡광도(Abs)는 일반 선으로 도시된다. mS cm-1로표현된 전도도는 파선선으로 표시됩니다. 회색 상자는 TmPep1050이 크로마토그램에 용출되는 위치를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 3: (A) TmPep1050 도데카머의 크기 배제 크로마토그래피, (B) TmPep1050H60A H307A,및 (C) TmPep1050H60A.
샘플은 120mL 컬럼에 포장된 SEC 수지를 사용하여 분석하였다. 흡광도(Abs)는 밀리유닛의 흡광도(mUA)로 표현된다. (D)티로글로불린(T), 페리틴(F), 알돌라제(Ald), 콘알부민(C), 알부민(Alb)을 표준으로 사용하여 SEC 컬럼을 교정한다. 상대 질량의 로가릿심과 용출 부피 사이의 상관관계는 선형이며 R2는 0.91이다. 95% 신뢰 구간은 점으로 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4: TmPep1050H60A H307A 결정화의 최적화.
(A)첫 최적화 전략은 PH(4.5~6.0사이) vs PEG3350 농도(5%에서 25% 사이)로 구성됩니다. 결정화 플레이트는 스키마화되고 우물은 침전을 위한 빨간색, 다결정용 노란색, 단결정에 대한 녹색으로 구분됩니다. (B)제2 최적화 전략은 pH 대 PEG3350의 좁은 변형으로 희석된 종자의 사용을 포함한다. (C)크리스탈 형상 및 크기 전(상부 이미지) 및 이후(하부 이미지) 결정화 최적화 및 미세 종자. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 5: 로그 출력 에서 발췌 올바른. TmPep1050H60A H307A 데이터 인덱싱의 LPXDS.
상부 패널, 가능한 Bravais 격자, 가장가능성이 m C, mP 및 oC. 중간 패널, C2221 공간 그룹에서 인덱싱 된 데이터의 전반적인 통계. P21 공간 그룹에서 인덱싱된 데이터의 전체 통계인 낮은 패널입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 6: TmPep1050H60A H307A의구조 .
(A)TmPep1050H6050 H307A 서브유닛(빨간색, PDB 코드 5NE9) 대 도드카메르 서브유닛(흰색, PDB 코드 6NW5) 및 이머 하위유닛(파란색, PDB 코드 5NE6)의 구조 정렬. 화살표는 도드카운퍼와 디머 사이의 구조적 유사성을 나타냅니다. (B)TmPep1050H6050 H307A 활성 부위(빨간색)의 클로즈업은 TmPep1050 디머(blue) 및 도데카메르(흰색)의 활성 부위와 비교된다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| TmPep1050H60A H307A | |
| 데이터 수집 | |
| 온도(K) | 100 |
| 방사선 소스 | 솔레일 프로시마 2 |
| 파장 (Å) | 0.9801 |
| 탐지기 | 데트리스 아이거 X 9M |
| 진동 범위(°) | 0.1 |
| 노출 시간(들) | 0.025 |
| 공간 그룹 | P 1 21 1 |
| 단위 셀 매개변수 | |
| α, β, γ (°) | 90.00, 110.69, 90.00 |
| a, b, c (Å) | 43.24, 137.79, 61.11 |
| 해상도 | 43.99 – 2.37 (2.52-2.37) |
| 고유한 반사 | 26.902 |
| Rmerge (%) | 0.14 |
| 중복 | 6,815 |
| 8.64 (2.12) | |
| 완전성(%) | 99.6 (97.9) |
| CC1/2 (%) | 99.2 (84.1) |
| 구체화 | |
| 해상도 | 43.99 – 2.37 |
| 반사 | 26.9 |
| R자유 세트 테스트 카운트 | 1345 |
| R작업/R무료 | 0.206/0.234 |
| ASU당 단백질 분자 | 2 |
| VM (Å 3/Da) | 2.37 |
| 용매 함량(%) | 49.0 |
| 단백질/용매 원자 | 4,559/96 |
| r.m.s.d. 채권 길이 (Å) | 0.31 |
| r.m.s.d. 본드 각도 (°) | 0.51 |
| 평균 B-요인 (Å2) | 57.0 |
| 선호/허용되지 않는 라마찬드란 φ/ψ(%) | 95.02 / 0.17 |
| 트윈 법 | h, -k, -h-l |
| PDB 코드 | 5NE9 |
표 1: 데이터 수집 및 수정 통계입니다. 괄호의 값은 가장 높은 해상도의 셸을 위한 것입니다.
토론
본 명세서에 기재된 프로토콜은 구조 수준에서 TmPep1050의 디머-도데카메르 전환을 이해할 수 있게 한다. 방법론은 TmPep1050 올리고머11의구조를 결정하기 위해 이전에 경험되었다. 가장 어려운 단계는 안정적인 디머로 도데카운더의 해리를 촉진하는 조건을 찾는 것이었습니다. 이러한 조건은 금속 이온 공동 인자가 추가 될 때 디머가 dodecamers로 재결합 할 수있을만큼 온화했다. 올리고머의 분리는 또한 구조 적인 연구 와 추가 생화학 적 특성화를 조건으로 하는 중요한 단계였다 (예를 들어,Co2+의다양한 용량에서 dodecamer 재결합을 연구). 분자 대체, 단계 측정을 위한 입증된 방법, TmPep1050 올리고머와 그 변이체의 구조를 해결하기 위하여 이용되었습니다. 제안된 프로토콜은 올리고머화 상태가 그들의 금속 공동 요소의 가용성에 의존하는 그밖 금속로 효소를 공부하기 위하여 적응될 수 있습니다.
프로토콜을 설명하기 위해, 연구의 경우, TmPep1050H60A H307A 누구의 금속 바인딩 사이트는 알라닌에 그의-60과 His-307을 돌연변이하여 손상되었다. 이러한 잔류물은 각각 M2 및 M1 사이트에서 Co2+를 바인딩합니다. 금속 결합을 방해하는 것은 올리고머화 상태를 방해하고 단량체로 완전한 해리로 이어질 수 있었다. 이러한 현상의 증거는 PhTET2 및 PfTET3, P. horikoshii및 P. furiosus에서두 개의 M42 아미노펩티다아제, 각각24,29에대해 보고되었다. TmPep1050H60A H307A는 이 변종이 희미한 자를 형성한 것처럼 예상대로 작동하지 않았다. 그 구조는 야생 형 이머와 동일한 수정을 보여 주었지만 두 가지 작은 예외를 보였습니다. 실제로, Asp-168 및 Asp-62의 측면 체인은 활성 부위의 안정화를 방지하는 틀에 얽매이지 않는 방향으로 잠겨 있는 것처럼 보였다. 그들의 방향은 단일 점 돌연변이 변이체에서 그러한 수정이 관찰되지 않았기 때문에 His-60 및 His-307에 의해 부과된 것처럼 보였습니다.
공개
저자는 공개 할 것이 없습니다.
감사의 말
우리는이 논문을 교정하고 건설적인 의견을 주는 마틴 로버에게 감사드립니다. Proxima 2 빔라인(SOLEIL 싱크로트론)에 대한 액세스는 블록 할당 그룹 20151139 내에 있었습니다.
자료
| Name | Company | Catalog Number | Comments |
| 1,10-phenanthroline | Sigma-Aldrich | 13, 137-7 | |
| Amicon Ultra 0.5 ml Centrifugal Filters Ultracel 30K | Merck Millipore | UFC503096 | |
| Amicon Ultra 15 Centrifugal Filters Ultracel 30K | Merck Millipore | UFC903024 | |
| Benzonase Nuclease | Merck Millipore | 70664-3 | |
| CCP4 | N/A | visit http://www.ccp4.ac.uk/ | |
| cOmplete EDTA-free | Roche | 5056489001 | |
| Coot | N/A | visit https://www2.mrc-lmb.cam.ac.uk/personal/pemsley/coot/ | |
| Crystal Screen I | Hampton Research | HR2-110 | |
| Crystal Screen II | Hampton Research | HR2-112 | |
| DreamTaq Green PCR Master Mix | ThermoFisher Scientific | K1082 | |
| EasyXtal 15-well tool | NeXtal | 132007 | |
| Escherichia coli PPY strain | N/A | see reference 31 | |
| Escherichia coli XL1 blue strain | Agilent | 200249 | |
| Gel Filtration Calibration Kit HMW | GE Healthcare Life Sciences | 28-4038-42 | |
| Gel Filtration Calibration Kit LMW | GE Healthcare Life Sciences | 28-4038-41 | |
| Gel Filtration Standard | Biorad | 1511901 | |
| GeneJET Plasmid Miniprep Kit | ThermoFisher Scientific | K0503 | |
| Index | Hampton Research | HR2-144 | |
| Litholoops | Molecular Dimensions | ||
| L-leucine-p-nitroanilide | Bachem AG | 40010720025 | |
| Natrix 1 | Hampton Research | HR2-116 | |
| Natrix 2 | Hampton Research | HR2-117 | |
| Neggia plugin | Dectris | N/A | visit https://www.dectris.com/ |
| NeXtal Tubes JCSG Core Suite I | NeXtal | 130724 | |
| NeXtal Tubes JCSG Core Suite II | NeXtal | 130725 | |
| NeXtal Tubes JCSG Core Suite III | NeXtal | 130726 | |
| NeXtal Tubes JCSG Core Suite IV | NeXtal | 130727 | |
| pBAD-TOPO | ThermoFisher Scientific | K430001 | |
| Phenix | N/A | visit https://www.phenix-online.org/ | |
| Phusion High-Fidelity DNA polymerase | ThermoFisher Scientific | F-530L | |
| Salt RX 1 | Hampton Research | HR2-107 | |
| Salt RX 2 | Hampton Research | HR2-109 | |
| SnakeSkin Dialysis Tubing, 3.5K MWCO | ThermoFisher Scientific | 88242 | |
| Source 15Phe | GE Healthcare Life Sciences | 17014702 | |
| Source 15Q | GE Healthcare Life Sciences | 17094705 | |
| Superdex 200 prep grade | GE Healthcare Life Sciences | 17104301 | |
| Thermotoga maritima MSB8 strain | American Type Culture Collection | ATCC 43589 | |
| TmCD00089984 | DNASU Plasmid Repository | N/A | |
| XDS | N/A | visit http://xds.mpimf-heidelberg.mpg.de/ | |
| xdsme | N/A | visit https://github.com/legrandp/xdsme |
참고문헌
- Levy, E. D., Teichmann, S. A. Structural, Evolutionary, and Assembly Principles of Protein Oligomerization. Progress in Molecular Biology and Translational Science. 117, 25-51 (2013).
- Selwood, T., Jaffe, E. K. Dynamic dissociating homo-oligomers and the control of protein function. Archives of Biochemistry and Biophysics. 519 (2), 131-143 (2012).
- Jaffe, E. K. The Remarkable Character of Porphobilinogen Synthase. Accounts of Chemical Research. 49 (11), 2509-2517 (2016).
- Ramström, H., et al. Properties and Regulation of the Bifunctional Enzyme HPr Kinase/Phosphatase in Bacillus subtilis. Journal of Biological Chemistry. 278 (2), 1174-1185 (2003).
- Rudyak, S. G., Brenowitz, M., Shrader, T. E. Mg2+-Linked Oligomerization Modulates the Catalytic Activiy of the Lon (La) Protease from Mycobacterium smegmatis. Biochemistry. 40 (31), 9317-9323 (2001).
- Yamamoto, S., Storey, K. B. Dissociation-Association of lactate dehydrogenase Isozymes: Influences on the formation of tetramers vs. dimers of M4-LDH and H4-LDH. International Journal of Biochemistry. 20 (11), 1261-1265 (1988).
- Sirover, M. A. Structural analysis of glyceraldehyde-3-phosphate dehydrogenase functional diversity. The International Journal of Biochemistry & Cell Biology. 57, 20-26 (2014).
- Gupta, V., Bamezai, R. N. K. Human pyruvate kinase M2: A multifunctional protein: Multifunctional Human PKM2. Protein Science. 19 (11), 2031-2044 (2010).
- Wiegand, G., Remington, S. J. Citrate synthase: Structure, Control, and Mechanism. Annual Review of Biophysics and Biophysical Chemistry. 15, 97-117 (1986).
- Libonati, M., Gotte, G. Oligomerization of bovine ribonuclease A: structural and functional features of its multimers. Biochemical Journal. 380 (2), 311-327 (2004).
- Dutoit, R., et al. How metal cofactors drive dimer-dodecamer transition of the M42 aminopeptidase TmPep1050 of Thermotoga maritima. Journal of Biological Chemistry. 294 (47), 17777-17789 (2019).
- Rawlings, N. D., et al. The MEROPS database of proteolytic enzymes, their substrates and inhibitors in 2017 and a comparison with peptidases in the PANTHER database. Nucleic Acids Research. 46 (1), 624-632 (2018).
- Neuwald, A. F., Liu, J. S., Lipman, D. J., Lawrence, C. E. Extracting protein alignment models from the sequence database. Nucleic Acids Research. 25 (9), 1665-1677 (1997).
- Dutoit, R., Brandt, N., Legrain, C., Bauvois, C. Functional Characterization of Two M42 Aminopeptidases Erroneously Annotated as Cellulases. PLoS ONE. 7 (11), 50639(2012).
- Franzetti, B., et al. Tetrahedral aminopeptidase: a novel large protease complex from archaea. The EMBO Journal. 21 (9), 2132-2138 (2002).
- Borissenko, L., Groll, M. Crystal Structure of TET Protease Reveals Complementary Protein Degradation Pathways in Prokaryotes. Journal of Molecular Biology. 346 (5), 1207-1219 (2005).
- Appolaire, A., et al. TET peptidases: A family of tetrahedral complexes conserved in prokaryotes. Biochimie. 122, 188-196 (2016).
- Russo, S., Baumann, U. Crystal Structure of a Dodecameric Tetrahedral-shaped Aminopeptidase. Journal of Biological Chemistry. 279 (49), 51275-51281 (2004).
- Schoehn, G., et al. An Archaeal Peptidase Assembles into Two Different Quaternary Structures: A tetrahedron and a giant octahedron. Journal of Biological Chemistry. 281 (47), 36327-36337 (2006).
- Durá, M. A., et al. The structural and biochemical characterizations of a novel TET peptidase complex from Pyrococcus horikoshii reveal an integrated peptide degradation system in hyperthermophilic Archaea: Characterization of P. horikoshii TET3 peptidase. Molecular Microbiology. 72 (1), 26-40 (2009).
- Basbous, H., Appolaire, A., Girard, E., Franzetti, B. Characterization of a Glycyl-Specific TET Aminopeptidase Complex from Pyrococcus horikoshii. Journal of Bacteriology. 200 (17), 00059(2018).
- Appolaire, A., et al. Small-angle neutron scattering reveals the assembly mode and oligomeric architecture of TET, a large, dodecameric aminopeptidase. Acta Crystallographica Section D Biological Crystallography. 70 (11), 2983-2993 (2014).
- Appolaire, A., et al. The TET2 and TET3 aminopeptidases from P yrococcus horikoshii form a hetero-subunit peptidasome with enhanced peptide destruction properties: TET aminopeptidase multi-subunit complex. Molecular Microbiology. 94 (4), 803-814 (2014).
- Colombo, M., Girard, E., Franzetti, B. Tuned by metals: the TET peptidase activity is controlled by 3 metal binding sites. Scientific Reports. 6 (1), 20876(2016).
- Petrova, T. E., et al. Structure of the dodecamer of the aminopeptidase APDkam598 from the archaeon Desulfurococcus kamchatkensis. Acta Crystallographica Section F Structural Biology Communications. 71 (3), 277-285 (2015).
- Kim, D., et al. Structural basis for the substrate specificity of PepA from Streptococcus pneumoniae, a dodecameric tetrahedral protease. Biochemical and Biophysical Research Communications. 391 (1), 431-436 (2010).
- Chevrier, B., et al. Crystal structure of Aeromonas proteolytica aminopeptidase: a prototypical member of the co-catalytic zinc enzyme family. Structure. 2, 283-291 (1994).
- Rosenbaum, E., Ferruit, M., Durá, M. A., Franzetti, B. Studies on the parameters controlling the stability of the TET peptidase superstructure from Pyrococcus horikoshii revealed a crucial role of pH and catalytic metals in the oligomerization process. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1814 (10), 1289-1294 (2011).
- Macek, P., et al. Unraveling self-assembly pathways of the 468-kDa proteolytic machine TET2. Science Advances. 3 (4), 1601601(2017).
- Edelheit, O., Hanukoglu, A., Hanukoglu, I. Simple and efficient site-directed mutagenesis using two single-primer reactions in parallel to generate mutants for protein structure-function studies. BMC Biotechnology. 9 (1), 61(2009).
- Zhang, Y., Werling, U., Edelmann, W. SLiCE: a novel bacterial cell extract-based DNA cloning method. Nucleic Acids Research. 40 (8), 55(2012).
- Schleif, R. AraC protein, regulation of the l-arabinose operon in Escherichia coli, and the light switch mechanism of AraC action. FEMS Microbiology Reviews. 34 (5), 779-796 (2010).
- McPherson, A., Gavira, J. A. Introduction to protein crystallization. Acta Crystallographica Section F Structural Biology Communications. 70 (1), 2-20 (2014).
- Bergfors, T. Seeds to crystals. Journal of Structural Biology. 142 (1), 66-76 (2003).
- Dauter, Z. Collection of X-Ray Diffraction Data from Macromolecular Crystals. Protein Crystallography. 1607, 165-184 (2017).
- Powell, H. R. X-ray data processing. Bioscience Reports. 37 (5), 0227(2017).
- Battye, T. G. G., Kontogiannis, L., Johnson, O., Powell, H. R., Leslie, A. G. W. iMOSFLM a new graphical interface for diffraction-image processing with MOSFLM. Acta Crystallographica Section D Biological Crystallography. 67 (4), 271-281 (2011).
- Otwinowski, Z., Minor, W. Processing of X-ray diffraction data collected in oscillation mode. Methods in Enzymology. 276, 307-326 (1997).
- Clabbers, M. T. B., Gruene, T., Parkhurst, J. M., Abrahams, J. P., Waterman, D. G. Electron diffraction data processing with DIALS. Acta Crystallographica Section D Structural Biology. 74 (6), 506-518 (2018).
- Kabsch, W. XDS. Acta Crystallographica Section D Biological Crystallography. 66 (2), 125-132 (2010).
- Legrand, P. legrandp/xdsme: March 2019 version working with the latest XDS version. , Jan 26, 2018. Zenodo (2019).
- Evans, P. Scaling and assessment of data quality. Acta Crystallographica Section D Biological Crystallography. 62 (1), 72-82 (2006).
- Wlodawer, A., Minor, W., Dauter, Z., Jaskolski, M. Protein crystallography for non-crystallographers, or how to get the best (but not more) from published macromolecular structures: Protein crystallography for non-crystallographers. FEBS Journal. 275 (1), 1-21 (2008).
- Karplus, P. A., Diederichs, K. Assessing and maximizing data quality in macromolecular crystallography. Current Opinion in Structural Biology. 34, 60-68 (2015).
- Taylor, G. L. Introduction to phasing. Acta Crystallographica Section D Biological Crystallography. 66 (4), 325-338 (2010).
- Rossmann, M. G., Blow, D. M. The detection of sub-units within the crystallographic asymmetric unit. Acta Crystallographica. 15, 24-31 (1962).
- Rossmann, M. G. The molecular replacement method. Acta Crystallographica Section A Foundations of Crystallography. 46 (2), 73-82 (1990).
- McCoy, A. J., et al. Phaser crystallographic software. Journal of Applied Crystallography. 40 (4), 658-674 (2007).
- Adams, P. D., et al. PHENIX: a comprehensive Python-based system for macromolecular structure solution. Acta Crystallographica Section D Biological Crystallography. 66 (2), 213-221 (2010).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Crystallographica Section D Biological Crystallography. 66 (4), 486-501 (2010).
- Chen, V. B., et al. MolProbity: all-atom structure validation for macromolecular crystallography. Acta Crystallographica Section D Biological Crystallography. 66 (1), 12-21 (2010).
- Zwart, P. H., Grosse-Kunstleve, R. W., Lebedev, A. A., Murshudov, G. N., Adams, P. D. Surprises and pitfalls arising from (pseudo)symmetry. Acta Crystallographica Section D Biological Crystallography. 64 (1), 99-107 (2008).
- Yeates, T. O. Detecting and overcoming crystal twinning. Methods in Enzymology. 276, 344-358 (1997).
- Terwilliger, T. C. Using prime-and-switch phasing to reduce model bias in molecular replacement. Acta Crystallographica Section D Biological Crystallography. 60 (12), 2144-2149 (2004).
- Terwilliger, T. C., et al. Iterative model building, structure refinement and density modification with the PHENIX AutoBuild wizard. Acta Crystallographica Section D Biological Crystallography. 64 (1), 61-69 (2008).
- Krissinel, E., Henrick, K. Inference of Macromolecular Assemblies from Crystalline State. Journal of Molecular Biology. 372 (3), 774-797 (2007).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유